Method Article
Subclonagem Além disso Inserção (SPI) - Um Método recombineering Novel para a construção rápida de Gene Segmentação Vetores
Neste Artigo
Resumo
Gene targeting methodologies can be used to generate transgenic mice with knockout, knock-in and tagged alleles. Here, we describe an improved method of recombineering in E. coli, that we term ‘subcloning plus insertion’, which can be used to generate custom gene targeting vectors rapidly.
Resumo
Gene targeting refers to the precise modification of a genetic locus using homologous recombination. The generation of novel cell lines and transgenic mouse models using this method necessitates the construction of a ‘targeting’ vector, which contains homologous DNA sequences to the target gene, and has for many years been a limiting step in the process. Vector construction can be performed in vivo in Escherichia coli cells using homologous recombination mediated by phage recombinases using a technique termed recombineering. Recombineering is the preferred technique to subclone the long homology sequences (>4kb) and various targeting elements including selection markers that are required to mediate efficient allelic exchange between a targeting vector and its cognate genomic locus. Typical recombineering protocols follow an iterative scheme of step-wise integration of the targeting elements and require intermediate purification and transformation steps. Here, we present a novel recombineering methodology of vector assembly using a multiplex approach. Plasmid gap repair is performed by the simultaneous capture of genomic sequence from mouse Bacterial Artificial Chromosome libraries and the insertion of dual bacterial and mammalian selection markers. This subcloning plus insertion method is highly efficient and yields a majority of correct recombinants. We present data for the construction of different types of conditional gene knockout, or knock-in, vectors and BAC reporter vectors that have been constructed using this method. SPI vector construction greatly extends the repertoire of the recombineering toolbox and provides a simple, rapid and cost-effective method of constructing these highly complex vectors.
Introdução
O desenvolvimento de tecnologias de segmentação de genes permitiu a construção de linhas de células e modelos de ratos para investigar diferentes sistemas biológicos 1-3. A primeira etapa chave na modificação de uma sequência genômica é a concepção e construção de um gene alvo vetor 4. Vectores de recombinação são as construções de plasmídeos que transportam o alelo de interesse contendo as modificações (s) desejados flanqueadas por um marcador de selecção (por exemplo, neomicina), e regiões genómicas longos necessários para recombinação homóloga eficiente em células de mamíferos 5. Modificação gene preciso que é conseguido através da introdução do vector de direccionamento para estaminais embrionárias (ES) 6 células ou células somáticas 7, em que a recombinação homóloga entre extensões idênticas de sequência de ADN no vector de abordagem selectiva e o locus genómico resultados na transferência da modificação pretendida para a conversão do gene por genoma 8. Tal modified células ES podem ser injectados em blastocistos de ratinho para produzir prole (quimeras) que pode, em seguida, transmitir estes alelos modificados através da linha germinativa 9. Uma via alternativa para produzir ratinhos transgénicos envolve a microinjecção de um vector de expressão de genes individuais em zigotos de ratinho unicelulares, o que leva a uma integração aleatória do vector no genoma do rato 10. Os métodos tradicionais de construção do vector ter invocado convencional "corta e cola" clonagem usando enzimas de restrição e ligases de ADN a clonar o marcador de selecção diferente e fragmentos genómicos em uma estrutura do vector. No entanto, um factor de limitação inerente de clonagem tradicional é o posicionamento e escolha de sítios de restrição, especialmente com sequências de DNA mais longos. Isso muitas vezes requer várias etapas de subclonagem e também apresenta sequências de DNA estranhos no vector, o que muitas vezes leva a uma menor eficiência do gene alvo.
Recombineering (eng recombinagênicoineering) é uma tecnologia de engenharia de ADN que ultrapassa estas limitações através da utilização de recombinação homóloga (HR) mediada por proteínas de recombinação do fago em E. coli 11,12. Uma vez que qualquer região de uma sequência homóloga pode servir como um substrato de recombineering, as restrições sobre disponibilidade de locais de restrição são removidos. Grandes sequências de DNA podem ser facilmente modificados diretamente in vivo, assim também preservar a sua integridade estrutural 13. Recombineering é muito eficiente com homologias curtas (50 pb) 14 e, portanto, braços de homologia (HA) pode ser convenientemente incorporados em sequências oligo sintéticos. Em um experimento recombineering típico, um oligo ou um DNA de cadeia (dsDNA) fragmento duplo contendo HA é electroporados em recombineering E. competente células de E. coli contendo o alvo localizados no cromossoma ou num plasmídeo de 15. O potencial de recombinação é conferido pela expressão induzida do rec Redombination proteínas do fago 16,17 ou as proteínas de RecET do profago 18 rac. A exonuclease Vermelho / RecE converte dsDNA linear a um ADN de cadeia simples (ssDNA) intermediária, que é então ligado pelo seu parceiro, Vermelho / RecT, uma proteína de cadeia simples de recozimento (SSAP) 19. O emparelhamento de um longo ssDNA ou a uma curta oligo com a sua sequência complementar alvo ocorre na costa de retardamento do garfo de replicação e conduz à incorporação da sequência no local alvo. Retardamento ADNcs recombinação é a base da alta eficiência de recombineering e pode ser descrita pela "beta" modelo recombinação 20,21.
Um fluxo de trabalho recombineering típico para construir um vector de abordagem selectiva de genes envolve qualquer uma das duas seguintes vias. Uma via envolve a subclonagem da região genómica desejado a partir de um clone de BAC de rato dentro de um plasmídeo seguido pela inserção sequencial dos locais de recombinação LoxP, um marcador de selecção , 22, etc., ou a rota alternativa envolve segmentação do local genómico BAC com os diferentes elementos do vetor segmentação por várias rodadas de recombineering 10,23 e, em seguida subclonando o locus modificado em um plasmídeo por reparação gap clonagem 24,25. Variações sobre o tema têm sido usados em diferentes condutas de alto rendimento recombineering como parte de programas de produção em grande rato 26,27. No entanto, esses procedimentos envolvem etapas demoradas e complicadas, requerem o uso de vectores especializados e E. coli estirpes (por exemplo, as células que expressam Cre) e utilizar um ou mais passos intermédios de purificação de ADN de vector e re-transformação (Tabela 1). A sub-clonagem de inserção de adição (SPI) é uma técnica recombineering romance que combina beta recombinação e reparação da clonagem num único processo (Figura 1). Montagem vector SPI é simples, rápida e flexível e oferece uma melhoria significativa em recombi padrãonharia abordagens (Tabela 1). Aqui, vamos demonstrar a facilidade e utilidade de usar SPI para diferentes aplicações de construção vector com especial destaque para a construção de projetos de vetores não-padrão e desafiadoras. Os casos de teste incluiu a construção de um vector repórter batendo fluorescente, uma expressão da proteína marcada batendo vector dupla, um vector BAC fluorescente repórter e um vector de knockout condicional. Estes examinados variadamente o requisito de localizar um receptor da superfície da célula, purificar um complexo proteína nuclear ou condicionalmente ablação a expressão de um gene.
Protocolo
1. Gene Segmentação projeto
- Encomendar um clone BAC adequado que cobre a região genômica de interesse. Certifique-se de que este é isogênico com o tipo de células ES de ser modificado por exemplo, clones RPCI-23 e RPCI-24 BAC para gene alvo com C57BL / 6 ES células.
- Aplicar gene convencional critérios de segmentação ao projetar o vetor de direcionamento. Parâmetros-chave incluem se a modificação é necessária para ser constitutiva ou condicional, definindo exons críticos (CE) para eliminação de uma estratégia gene nocaute e espaçamento e colocação de cassetes intrónicas.
NOTA: Cada uma delas tem sido discutida em detalhe em outro lugar 10. - Escolha regiões genómicas cada 5-6 kb de flanqueamento do local de modificação alvo.
NOTA: O tamanho da inserção é, portanto, tipicamente subclonado 10-12 kb, embora o limite superior possa ser tão alta como 80 kb com uma cópia do plasmídeo de subclonagem baixo como p15A, pBR322, etc., e até 200 kb, com um BACvetor.
2. Multiplex recombineering Oligos
- Oligos do projeto para a espinha dorsal plasmídeo (subcloning plasmídeo) e o marcador de selecção (cassete de inserção). Conceber cada oligo de tal modo que ele contém 180 pb HA flanqueando o local alvo genómico e 20 pb da sequência de iniciação específica da cassete de inserção ou o plasmídeo de subclonagem (Figura 2).
NOTA: os braços de homologia não deve conter elementos repetitivos, presença de elementos repetitivos resultará em direcionamento incorreto e subclonagem. Elementos repetitivos pode ser detectada por meio de ferramentas baseadas na web, como o '' Repita Masker ''. - Incluir um local único da enzima de restrição (RE) em um dos oligos de vectores para linearizar o vector de abordagem selectiva de genes para células ES alvo (Figura 2).
- Verifique os parâmetros oligo usando um programa analisador de oligo. Tome cuidado para evitar estruturas secundárias na região primings.
- Shorter HA (50 pb) da cassete de inserção é tolerado e produz menor número de recombinantes, mas garantir que o plasmídeo subcloning HA é sempre mais longo (180 pb).
- Determinar a orientação da inserção de ADN genómico do clone de BAC em particular usando ferramentas com base na web, como '' cloneDB ''. Estabelecer a direcção de replicação de oris, verificando o mapa do esqueleto de plasmídeo BAC usado na construção da biblioteca BAC.
NOTA: oris é geralmente oposta à direcção de transcrição do marcador de cloranfenicol (Chl) para todos os plasmídeos de TAS vulgarmente utilizados. - Adicionar duas fosforotioato (PTO) ligações terminais com a extremidade 5 'do oligonucleótido que é oposta à direcção de replicação no clone de BAC. Adicionar uma modificação 5 'fosfato para o oligo inverso (Figura 2).
NOTA: A cassete PCR phosphorothioated assimétrica sobre a digestão Red gera um ssDNA intermediário que pode iniciar a cadeia atrasadado garfo de replicação.
NOTA: Maximal freqüência de recombinação multiplex é observado com as cassetes protegidas cadeia atrasada. Costa principal protegido ou cassetes protegidas dupla pode não produzir a eficiência de recombinação equivalentes em algum loci. - Encomende oligos com PAGE ou purificação HPLC.
3. BAC Transformation Clone com pSC101 BADgbaA recombineering plasmídeo.
- Para fazer com que a E. estirpe de E. coli que carrega o recombineering BAC proficiente, transformá-lo com o plasmídeo pSC101 BADgbaA contendo os genes vermelhas 16.
NOTA: O plasmídeo pSC101 BADgbaA contém os genes vermelhos e RecA sob o controlo do promotor araC de P-arabinose BAD indutivel, um marcador de selecção de tetraciclina e o replicão pSC101 sensível à temperatura.- Esfaquear uma ponteira estéril na cultura agar BAC e inocular 5,0 ml de caldo lysogeny (LB) pH 8,0 contendo 12,556; g ml -1 Chl. Crescer a 37 ° C durante 5 horas com agitação a 200 rpm.
- Relaxar 10% (v / v) de glicerol, tubos de microcentrífuga e cuvetes de electroporação em gelo. Arrefece-se uma grande centrifugadora de microcentrífuga e refrigerado a 4 ° C.
- Determinar a densidade óptica (OD) da cultura utilizando um espectrofotómetro e medir a absorvância a 600 nm. Preparar células electrocompetentes (como descrito abaixo) quando uma leitura de DO 600 de 0,3 a 0,8 é alcançado.
- Girar as células num tubo de centrífuga de 50 ml em uma grande centrifugadora a 1216 xg durante 5 min a 4 ° C.
- Lavar as células com 1 ml de glicerol a 10% gelada e girar para baixo em células 17.949 xg durante 20 seg a 4 ° C. Realizar a lavagem passo de um total de 3 vezes.
- Ressuspender as células num volume total de 50 ul de glicerol a 10% e adicionar 10-200 ng do plasmídeo pSC101 recombineering BADgbaA. Obter uma suspensão de células individuais pipetandopara cima e para baixo várias vezes e, em seguida, transferir as células para uma pré-refrigerada um milímetro lacuna cuvete de electroporação.
- Electroporate as células com uma configuração de 1,8 kV, 25 mF e 200 Ω.
Nota: Verifique as configurações corretas para cada marca de electroporator. Uma constante de tempo de electroporação menos do que 4 indica a presença de sal e outras impurezas. - Imediatamente recuperar as células em 1 ml de LB e transferência das células para um tubo de centrífuga de 50 ml.
- Crescer a cultura BAC gbaA a 30 ° C durante 2 horas com agitação a 200 rpm.
NOTA: O plasmídeo pSC101 BADgbaA é perdido quando as células são cultivadas a 37 ° C, devido à inactivação do factor de replicação sensível à temperatura repe. Cultivar células gbaA a 30 ° C, para manter as funções recombineering. - Adicionar 9 ml de LB contendo 12,5 ng ml -1 de Chl e 4 ug mL1 de tetraciclina (Tet) para a cultura de BAC gbaA recuperado. Cresça aBAC gbaA cultura O / N em 30 ° C com agitação a 200 rpm.
NOTA: A eficiência de transformação do plasmídeo electroporating gbaA super-enrolado é suficientemente alta para permitir o crescimento saturação de O / N em meios líquidos.
4. Preparação de Inserção Cassetes e Subclonagem Plasmids
- Para incorporar AH à inserção da cassete (s) e o plasmídeo de subclonagem, realizar a reacção em cadeia da polimerase (PCR), utilizando os oligos longo modificados tal como descrito abaixo.
- Opcional: para evitar plasmídeo transição na reacção recombineering, usar uma origem R6K ou molde estreito plasmídeo gama de hospedeiros semelhante para amplificar a cassete de inserção.
- Alternativamente, linearizar o plasmídeo molde utilizando uma digest RE (Tabela 2). Escolha um RE que corta fora da região de amplificação da PCR e é inativada calor. Calor inactivar o RE, como recomendado pelo manufacturer.
- Defina-se reacção em cadeia da polimerase (PCR) utilizando um sistema de DNA polimerase de alta fidelidade HotStart. Prepara-se uma mistura principal de PCR como descrito (Tabela 3). Realizar o ciclo térmico, como mostrado (Tabela 3).
- Analisar os produtos de PCR por electroforese em gel de agarose. Carga de 1-5 uL de cada PCR sobre um 1% (w / v) de gel de agarose contendo 0,5 mg mL-1 de brometo de etídio (EtBr).
NOTA: A presença de produtos de amplificação não-específicos não interfere na reação recombineering. Em alguns casos, no entanto, dimeros de iniciadores pode diminuir a eficiência recombineering. - Purifica-se os produtos de PCR utilizando um kit de purificação de PCR.
- Opcional: remover o plasmídeo molde de PCR por tratamento com Dpnl seguido por PCR clean-up. Eluir ADN num volume mínimo de água desionizada estéril (como recomendado pelo fabricante).
NOTA: A adição de DpnI para não purificados de PCR reações resulta em redução da eficiência de clivagem de the modelo do DNA de plasmídeo desnaturado.
- Opcional: remover o plasmídeo molde de PCR por tratamento com Dpnl seguido por PCR clean-up. Eluir ADN num volume mínimo de água desionizada estéril (como recomendado pelo fabricante).
- Quantificar por PCR de ADN amplificado por análise em gel de agarose de encontro a um conjunto de padrões de ADN conhecidos por exemplo, λ HindIII digest ou usando um espectrofotómetro NanoDrop.
5. Além disso Subclonagem Inserção
- Dilui-se a S / N BAC gbaA cultura de 50 vezes por adição de 200 ul em 10 ml de LB + + Chl Tet. Crescer em 30 ° C com agitação a 200 rpm durante 1 h 50 min. Incluir uma amostra a ser utilizada como controlo negativo.
- Relaxar todos os materiais e equipamentos recombineering a 4 ° C, conforme descrito no passo 3.1.2.
- Pour LB agar pH 8 placas contendo a concentração correcta de o antibiótico selectivo apropriado (s), que irá permitir a selecção do fragmento de ADN que é inserida, assim como para a selecção do vector de subclonagem (Tabela 4).
NOTA: Alguns antibióticos são sensíveis ao pH. Use LBágar pH 8 como uma regra. - Prepara-se uma solução a 10% de L-arabinose (v w /). Filtre através de esterilizar um filtro de seringa de 0,2 um.
NOTA: A arabinose induz a expressão de proteínas a partir do plasmídeo recombineering gbaA. - Verifique o OD da cultura BAC gbaA usando um espectrofotômetro. Uma vez que um OD 600 de 0,25-0,3 é atingido, induzir as proteínas recombineering como descrito no passo seguinte.
- Adicionar 200 ul da solução de arabinose a 10% até 10 ml de cultura de BAC para atingir uma concentração final de arabinose de 0,2%. Incluir uma cultura não induzida (sem arabinose) a ser utilizada como controlo negativo.
- Transferir a cultura para um BAC 37 ° C incubadora de agitação e induzir a expressão vermelho por 45 min com agitação a 230 rpm.
NOTA: Expressão de proteínas vermelhos é ineficiente a 30 ° C. - Girar as células e lava-se com 10% de glicerol, 3 vezes, conforme descrito no passo 3.1.5.
- Adicionar 600-1,000 ng de cada um do plasmídeo de subclonagem e a cassete (s) de inserção para a reacção recombineering. Incluir um vector apenas e vector além de controles individuais de inserção para verificar a proficiência recombinação e integridade do vector e as cassetes.
- Obter uma suspensão de células individuais por pipetagem cima e para baixo. Realizar a electroporação tal como descrito no passo 3.1.7 e recuperação subsequente em 1 ml de LB a 37 ° C durante 1 h para os plasmídeos de multi-cópias ou em 10 ml de LB a 37 ° C durante 3 h para os vectores BAC.
- Placa de diferentes diluições da cultura recuperado por exemplo, 90%, 10%, 1%, nas placas de agar de selecção dupla e crescer a 37 ° C durante 16 horas.
6. Análise de recombinantes
- Opcional: Escolha de 6 a 12 colônias e realizar colônia PCR utilizando um iniciador de flanqueamento HA e um primer específico de inserção. Inclua o plasmídeo subcloning eo cas inserçãoSette plasmídeo assim como o clone de BAC-mãe como controlos negativos.
- Realizar análise em gel de agarose das PCRs colônia. Identificar os clones positivos pela presença de uma banda brilhante com o tamanho esperado.
NOTA: A elevada eficiência do processo de clonagem recombinantes SPI gera principalmente correctas.
- Realizar análise em gel de agarose das PCRs colônia. Identificar os clones positivos pela presença de uma banda brilhante com o tamanho esperado.
- Escolha colónias em 5 ml de LB pH 8 contendo o antibiótico selectivo e crescer O / N a 37 ° C.
- Preparar mini-preparações de ADN utilizando um kit de purificação de coluna de acordo com as instruções do fabricante.
- Prepare BAC ADN miniprep usando isolamento de fenol-clorofórmio padrão ou um protocolo semelhante.
- Execute digere RE no DNA miniprep. DNA separado por eletroforese em gel de agarose. Analisar os padrões de ER para identificar os clones contendo os tamanhos de fragmentos esperados do vector direcionamento correto. Escolha um RE claramente que discrimina entre o vector sem inserto (s) e o vector contendo o inserto (s).
- Optional: a obtenção de células que são desprovidas do BAC, que ainda está presente na E. coli após recombineering, usar o plasmídeo miniprep de ADN para transformar DH5alpha ou DH10B E. células de E. coli.
- Execute sequenciamento de DNA em todo o HA e cassete de inserção para verificar erros de síntese oligo.
Resultados
Vetores Knockin
Vectores de recombinação knockin reflectir a necessidade de introduzir uma característica nova sequência no genoma incluindo substituição único par de bases na região de codificação da proteína, a fusão de um marcador fluorescente ou um marcador de afinidade para uma proteína ou a integração de uma cassete de expressão do gene. Para testar o aplicativo do SPI em estratégias de construção batendo vetor, dois casos de teste diferentes foram examinados. Dnttip1 codifica a deoxynucleotidyltransferase, terminal, interagindo 1A proteína (TDIF1) que, em conjunto com a classe I histonadesacetilase (HDAC) formam um complexo deacetylase mitótico (Midac) 28. Para investigar o papel de Dnttip1 na divisão celular, uma abordagem de marcação de afinidade em tandem foi feita para isolar proteínas que interagem com o TDIF1. As tentativas anteriores para subclonar uma porção do gene Dnttip1 utilizando um vector p15A contendo regiões de homologia de comprimento, resultou em baixa eficiência de reparação uma lacunand frequentes produtos de recombinação aberrantes (dados não mostrados). Assim, a construção de um vector batendo no locus Dnttip1 proporcionado um exercício recombineering desafiador. Uma estratégia de SPI foi concebido para subclonar uma secção de 12 kb do gene Dnttip1 abrangendo o último exão (o exão 13) num vector de cópia baixo p15A e, simultaneamente, para inserir uma cassete de selecção tag de afinidade dupla ao exão 13, substituindo o codão de terminação (Figura 3A ). A proteína de ligação FLAG-calmodulina 2X (CBP), ligado FRT-PGK-Em7-Neomicina (Neo) -BGhpA-FRT cassete (2,0 kb) foi amplificado a partir de um plasmídeo linearizado RE usando dupla PTO oligos modificados que continham 120 bp HA ladeando o Dnttip1 códon de terminação. A p15A zeo Dnttip1 vector subcloning (1,7 kb) foi construído que continha 200 pb regiões homólogas às extremidades da sequência Dnttip1 ser Subclonou. O plasmídeo subclonando Dnttip1 foi re linearizado e amplificado por PCR utilizando 20 pb modificado oligosque gerou um fio protegido principal vetor. Os produtos de PCR foram tratados Dpnl, purificado e co-electroporados em recombineering competente Dnttip1 BAC E. células de E. coli, bem como células de controlo não induzidas. As reacções de SPI foram plaqueadas em zeocina (Zeo) e canamicina (Kan) contendo placas de agar (Figura 3B). SPI produzido o vector Dnttip1 batendo correctamente modificados em todos os 12 recombinantes analisados (Figura 3C). A sequenciação do ADN verificou-se a ausência de quaisquer erros resultantes da síntese de oligo ou amplificação por PCR.
Outro exemplo de SPI envolveu a inserção no quadro de uma proteína fluorescente amarela reforçada (EYFP) ligada cassete de selecção no gene P2rx1. O gene P2rx1 codifica um receptor de G-acoplado a uma proteína que funciona como um canal de iões de ATP 29 fechado. Ligação às YFP fluorescência permite o seguimento do receptor P2X1 na superfície da célula em vaensaios funcionais. rious P2rx1 é outro locus de difícil que tem apresentado problemas significativos com metodologias recombineering convencionais (dados não apresentados). Usando SPI, um segmento de 12 kb do gene P2rx1 abrangendo o exão terminal (exão 12) foi subclonado num vector de p15A zeo e modificado com a inserção de uma cassete de concertada neo flanqueado EYFP -LoxP substituir o codão de paragem no exão 12 para construir o P2rx1-EYFP batendo vetor (Figura 3D). Neste exemplo, o vector de cadeia protegida p15A atraso (1,7 kb) continha 230 pb de HA e a cassete de EYFP continha 50 pb de HA e também foi deixado inalterado. A cassete de inserção EYFP (2,7 kb) foi montado por união de sobreposição de PCR do gene EYFP, amplificado por PCR a partir de pEYFP-C1, e a cassete LoxP- PGK-Em7-Neo-BGhpA-LoxP, amplificado por PCR a partir de pL452 (NCI, Frederick) . A reacção SPI produzido centenas de colónias (dados não mostrados). RE análise de ADNminipreps preparados a partir de 11 clones mostrou que a maioria continha o P2rx1-EYFP batendo vector montado corretamente (Figura 3E). Validação adicional de seqüenciamento de DNA mostraram erro de inserção livre da cassete EYFP. No entanto, alguns dos clones apresentaram perfis do gap não marcado reparado eo fosso reparado plasmídeos marcados na mesma célula (pistas 6-9). Algumas amostras continha também incorrectamente lacuna reparado (pista 2) ou plasmídeos mistargeted (pistas 4 e 5). O fracasso da subcloning concorrente e segmentação em alguns recombinantes SPI destaca os limites da clonagem eficiente SPI ao usar grandes cassetes (> 3 kb), que surgem a partir das limitações sobre processivity Red de fragmentos de DNA longos 30, ou se estiver usando short HA. Com efeito, o aumento da HA da cassete EYFP a 200 pb a maior eficiência SPI e o direccionamento correcto da cassete (dados não mostrados). Gene alvo do P2rx1 - vector batendo EYFP em JM8.N4células do rato ES (linhagem C57BL / 6) gerado seis clones positivos de 96, que continham a sequência P2rx1-EYFP corretamente direcionados (Figura 3F).
BAC Reporter Vetores
Um clone de BAC do gene alvo muitas vezes contém todos os elementos necessários a montante e a jusante de regulação por exemplo, intensificadores, etc., bem UTRs o promotor endógeno para conduzir a expressão de genes a níveis naturais 31. Um vector BAC repórter é, por conseguinte, o veículo preferido para recapitular o padrão de expressão de um gene endógeno de 32. No entanto, o grande tamanho de um plasmídeo BAC (até 200 kb) apresenta problemas práticos significativos com a transfecção de um BAC intacta em células 33. Reduzindo o tamanho da inserção genómica de BAC a BAC através aparar 34,35, mantendo os elementos reguladores necessários de um gene, permite uma manipulação mais fácil do BAC e resulta num mais eficiente transfectino. Atual tecnologia de engenharia BAC envolve várias rodadas de recombineering para atingir esse objetivo 35. Para demonstrar a utilidade de SPI em BAC de corte, um vector BAC pBeloBAC11 foi usado para subclonar uma sequência genómica de 30 kb incluindo o gene de comprimento completo a partir do P2rx1 168 kb P2rx1 BAC em conjunto com a inserção simultânea de uma cassete de EYFP no gene P2rx1 (Figura 4A). O pBeloBAC11 backbone zeo vector (6,5 kb) contendo 180 bp HA foi amplificado por PCR com oligos modificados, que geraram um protegido vector da cadeia atrasada. A mesma cassete neo EYFP (3 kb), utilizado na anterior P2rx1 batendo vector de construção, foi amplificado por PCR utilizando os oligos protegidas fita retardada contendo 180 pb de HA e o exão alvo P2rx1 12 substituindo o codão de paragem. Após a electroporação combinado da cassete e o vector de EYFP zeo pBeloBAC11 em P2rx1 BAC células expressing as proteínas recombineering gbaA, a cultura foi recuperado em 10 ml de LB pH 8, durante 3 horas a 37 ° C para segregar os plasmídeos BAC. Recombinantes puras foram seleccionados com Zeo Kan e placas de agar e foram observadas com uma frequência de 2 x 10 -6. Análise genotipagem Colony PCR revelou bem sucedido BAC recorte nas 3 de 6 clones que foram analisados (Figura 4B). A amplificação por PCR de longo alcance em todo o local de inserção EYFP confirmaram a incorporação cassete correcta nos três clones positivos (Figura 4B). Os três clones faltam o inserto EYFP também foram incorrectamente lacuna reparado no extremo 5 ', embora a causa de mistargeting da cassete EYFP nestes clones podem ser separados para o fecho correcto da extremidade 5' de BAC. Os três clones EYFP alcoolemia positiva foram ainda analisados com digere RE e apresentaram o perfil esperado da corretamente aparado EYFP recombinante BAC (Figura 4C </ Strong>). A análise da sequência revelou ausência de erros na cassete EYFP em apenas um clone dos 3 pontos positivos.
Knockout Condicional (CKO) Vetores
Ablação condicional da expressão do gene é uma ferramenta importante para estudar processos de desenvolvimento ou para estudar sistemas biológicos num ponto de tempo específico. Uma estratégia gene knockout condicional tipicamente envolve a colocação de locais de recombinação LoxP torno de um exão crítica (CE). A deleção de um EC após expressão ou activação Cre produz um desvio de enquadramento e um codão de paragem prematuro, resultando na degradação do mRNA devido à deterioração absurdo mediada (NMD). A construção de um vector gene alvo condicional é uma tarefa complexa e envolve várias etapas de subclonagem, segmentação e transformação 22. SPI oferece uma rota conveniente para simplificar este processo. Como um caso de teste, um alelo condicional do gene Zrsr2 foi construídoutilizando a metodologia SPI (Figura 5A). O gene Zrsr2 codifica um factor de splicing e uma única cópia está localizado no cromossoma X. O estado condicional de deleção do gene é particularmente importante no caso em apreço para controlar a possibilidade de as células adaptam-se à falta de Zrsr2 durante a selecção de células ES em uma estratégia de deleção do gene alvo constitutiva. SPI foi realizada para subclonar uma porção de 10 kb do gene ZrSr2 com inserção simultânea de duas cassetes de selecção diferentes flanqueadas LoxP. A FRT-PGK-Em7-Neo-FRT-LoxP cassete (2 kb) foi amplificado por PCR a partir de RE digerido pL451 usando retardamento protegido oligos que continham 180 pb HA visando o ZrSr2 intron 2. A segunda cassete contendo Rox-PGK-em7- Blasticidin (BSD) -Rox-LoxP (2 kb) foi amplificado por PCR a partir de um plasmídeo R6K com oligos protegidas cadeia atrasada contendo 180 pb HA idêntica a uma região a jusante no intron 3. Os dois sites LoxP ladeado exão 3, a CE cujo deletion sobre condicionais Cre ativação resulta em um desvio de enquadramento e introduz um códon de parada prematura. O plasmídeo de subclonagem Zrsr2 (1,6 kb) foi amplificado por PCR a partir de um plasmídeo linearizado RE p15A zeo usando retardamento protegido oligos contendo 180 pb HA correspondentes às extremidades da sequência Zrsr2 10 kb. SPI reacções foram realizadas tal como descrito antes e plaqueadas em placas de Zeo Neo + + BSD. O direccionamento ZrSr2 condicional vector foi montado com êxito na maioria dos 12 recombinantes analisados (Figura 5B). Uma análise mais aprofundada sequenciamento de DNA mostrou correta inserção de ambos os marcadores de seleção no vetor ZrSr2 cko.

Figura 1. SPI clonagem. Resumo do processo de clonagem SPI combinação de inserção de cassetes e de subclonagem num único passo. (A) O clone de BAC é transformed com o plasmídeo pSC101 BADgbaA e cultivadas a 30C. (B) Após a indução de arabinose para expressar as proteínas vermelhas, a cassete de inserção modificada assimétrica e plasmídeo de subclonagem são introduzidos no clone BAC e selecionados com ambos a inserção e subclonagem marcadores para gerar o vector final. Por favor, clique aqui para ver uma versão maior esta figura.
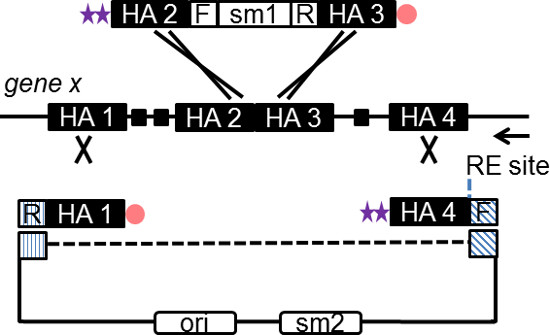
Figura 2. Diagrama esquemático que ilustra o desenho de oligos recombineering SPI. O plasmídeo de subclonagem e cassete de inserção são ambos gerados por PCR usando uma combinação de terminal de tomada de força e fosfato oligos modificados. O fragmento de PCR por digestão com vermelho in vivo, produz um intermediário de ADNcs, que hibrida com a cadeia atrasada do garfo de replicação. Gene HA específico de 50-180 pb é incorporado em cada oligo, como mostrado. A seta indica o sentido da replicação do ADN através de um gene candidato. A linha tracejada representa seqüência plasmídeo subcloning não incorporado no produto PCR. Site de RE, local da enzima de restrição da linearização de vector final; F, sequência para a frente (20 pb) específico para o plasmídeo de subclonagem ou cassete de inserção; R, sequência complementar reverso (20 pb) do plasmídeo ou cassete; sm, marcador de seleção. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 3. SPI permite a construção de vectores knockin difíceis. (A) Representação esquemática do SPI estratégia utilizada na construção do vector de Dnttip1 dupla etiquetado. Seta indicates a direção de replicação no clone BAC. resultados (B) chapeamento das amostras não induzidos e induzidos da experiência Dnttip1 SPI. (C) EcoRI de clones Dnttip1 SPI. M, 1 kb escada (NEB); C, p15A Dnttip1 lacuna reparado plasmídeo sem a cassete de marcação dupla. O tamanho dos fragmentos são as seguintes: marcado, 9,2 + 4,4 + 2,2 kb; controle; 9,2 + 4,6 kb. (D) SPI baseado P2rx1-EYFP batendo construção vector. (E) EcoRI de clones P2rx1-EYFP SPI. M, 1 kb + escada (Invitrogen); C, p15A P2rx1 lacuna reparado plasmídeo sem a cassete de EYFP. Tamanhos dos fragmentos são: marcado, 8,3 + 4,4 + 3,1 + 0,03 kb; controle; 8,3 + 3,1 + 1,8 + 0,03 kb (F) Southern blot de P2rx1 -. EYFP gene alvo em células celulares JM8.N4 ES. Top painel, Southern blot com a sonda 3 'usando PshAI digerir. É mostrado o resultado do rastreio de 5 clones. Painel inferior, Southern blot usando5 'sonda final e Spel resumo dos pontos positivos identificados a partir do 3' triagem final. Local PshAI RE; S, local Spel RE. A linha tracejada representa o fim do vector de HA. Caixa preta indica a sonda do sul. Fragmentos de restrição são esperados, PshAI: WT, 8,9 kb; EYFP-neo, 11,5 kb; Spel:. WT, 6,9 kb; EYFP-neo, 7,6 kb por favor, clique aqui para ver uma versão maior desta figura.
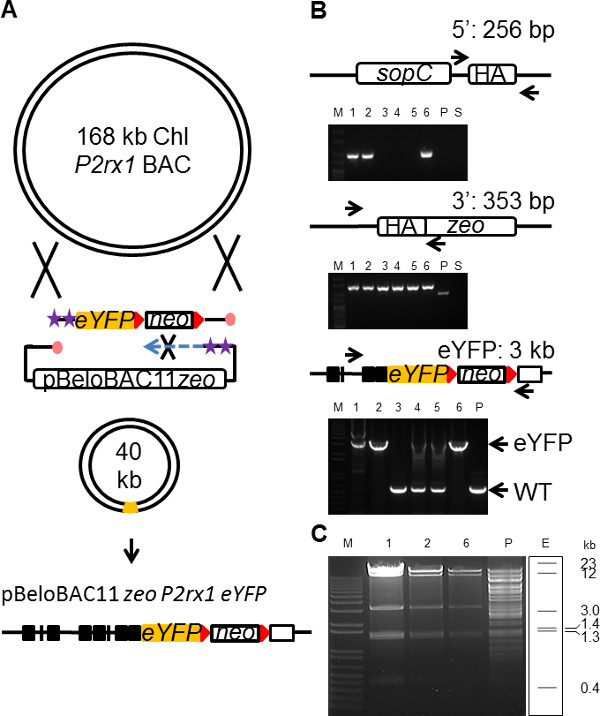
Figura 4. Simplificado BAC aparar usando SPI. (A) Esquema ilustrando o conceito de BAC aparar usando SPI. Legenda dos símbolos é descrito na Figura 1. Amplificação (B) através de PCR a 5 'e 3' da inserção e subclonado entre os insertio P2rx1-EYFPn local. A estratégia de rastreio são mostrados para cada tipo de PCR. M (painel superior), Hyperladder 25 pb, (painéis inferiores), 1kb +; P, P2rx1 BAC; . S, pBeloBAC11 zeo subclonando plasmídeo (C) HindIII digerir dos três EYFP positivos clones SPI TAS de (B); E, padrão de restrição HindIII do aparadas EYFP BAC esperado. Por favor, clique aqui para ver uma versão maior desta figura.
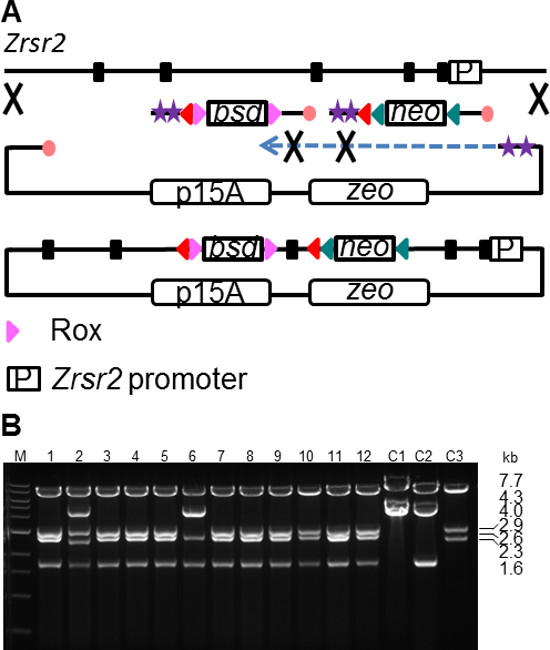
Figura 5. condicional geração nocaute vector de clonagem utilizando SPI. (A) Esquema da inserção simultânea de duas cassetes de selecção diferentes LoxP flanqueado durante a subclonagem do alelo Zrsr2. Legenda dos símbolos é descrito na Figura 1. (B) </ Digere strong> EcoRI de clones Zrsr2 SPI. L, 1 kb escada (NEB); C1, p15A ZrSr2 lacuna reparado plasmídeo, C2, p15A ZrSr2 lacuna plasmídeo contendo a inserção neo reparado; C3, p15A ZrSr2 lacuna plasmídeo contendo a inserção deb reparado. O tamanho dos fragmentos são as seguintes: cko, 7,7 + 2,9 + 2,6 + 1,6 kb; C1, 7,7 + 4,0 kb; C2, 7,7 + 4,3 + 1,6 kb; C3, 7,7 + 2,9 + 2,3 kb. Por favor, clique aqui para ver uma versão maior desta figura.
| Nenhuma das etapas | Recombineering convencional oleoduto uma | Multiplex recombineering b |
| Passo 1 | Transformação de recombineering plasmídeo no hospedeiro BAC | Transformação de recombineering plasmídeo no hospedeiro BAC. Preparação das cassetes de segmentação e de subclonagem vetores. |
| Passo2 | A inserção da cassete de gateway R1 R2 / | Gap Multiplex reparação clonagem |
| Passo 3 | Inserção de cassetes Kan floxed | O / N a cultura a partir de colónias individuais |
| Passo 4 | Reparação Gap em R3 / R4 plasmídeo | Preparação de plasmídeo e verificação |
| Passo 5 | A transformação em E. Cre + coli | |
| Passo 6 | Preparação de plasmídeo e verificação | |
| Etapa 7 | O / N três vias reação gateway | |
| Passo 8 | Transformação de três vias reação gateway em DH10B E. células de E. coli | |
| Passo 9 | Cultura durante a noite de colónias individuais | |
| Passo 10 | Preparação de plasmídeo e a verificação da sequência | |
| b duração média de 4 dias para clone verificado | ||
Tabela 1: Comparação de recombineering convencional com SPI na construção de vectores knockout condicionais.
| Tampão RE | 5 ul |
| DNA | 1 ug de plasmídeo ou de produtos de PCR purificados |
| RE | 1 ml (5 unidades ou mais) |
| TE | até 50 ul |
| Incubar a 37 ° C durante pelo menos 1 hr. Aquecer inacitvate de acordo com as instruções do fabricante | |
Tabela 2: RE digerir.
| Materiais de PCR | A concentração final |
| O tampão de RCP | 1x |
| dNTP | 200 nM |
| MgSO4 | 1,5 mM |
| Betaine | 1,3 M |
| DMSO | 1% |
| Iniciador directo | 200 nM |
| Iniciador inverso | 200 nM |
| DNA polimerase | 1 L |
| Modelo | 10 ng de plasmídeos multicópia ou 2,5 ul de ADN de miniprep de genotipagem para PCRs |
| Água | até 50 ul um |
| um Para uma reacção de PCR de 50 ul padrão com plasmídeos multicópia. PCRs de genotipagem de longo alcance foram criados em 25 reações de PCR ul. | |
| As condições de PCR | |
| 95 ° C | 2 min |
| 92 ° C | 10 seg |
| 55 ° C | 30 seg |
| 72 ° C | 30 seg |
| 30 ciclos b | |
| b ciclo não pode ser alargada a 35 para BAC PCR genotipagem | |
Tabela 3: PCR Set-up e Condições.
| Antibióticos | Concentração A (ug mL-1) |
| Ampicllin | 50 |
| Blasticidin b | 40 |
| Cloranfenicol | 12.5 |
| A gentamicina | 2 |
| Higromicina c | 30 |
| Kanamycin b | 15 |
| Tetraciclina | 4 |
| Trimetoprima c | 10 |
| Zeocin | 5 |
| um Recomendado para uso com os BACs e plasmídeos multicópia quando usado em combinações em multiplex recombineering | |
| b Blasticidina (35 ug mL-1) e canamicina (6 ug mL-1), quando utilizados em conjunto em combinação | |
| c Higromicina e Trimetoprima não são recomendados para a seleção com BACs de cópia única. | |
Tabela 4. Recomendado concentrações de antibiótico para uso em experimentos de SPI.
Discussão
Construção de linhas de células ES e modelos de mouse tem historicamente envolvidos gene alvo usando construções de plasmídeos que continham o alelo 4 modificado. No entanto, a construção destes vectores de direccionamento de genes complexos provou ser um gargalo significativo na produção tempo de tais modelos. O desenvolvimento de estratégias de construção vector recombineering baseado permitiu melhores desenhos vetoriais e montagem do vetor mais eficiente. No entanto, os protocolos recombineering atuais ainda envolvem várias etapas, exigem intermediário de purificação de plasmídeos e usar diferentes estirpes bacterianas. A sub-clonagem de inserção mais oferece uma nova abordagem para a construção do vector que pode ser realizada em um evento de electroporação na estirpe hospedeira BAC residente. O utilitário de SPI em segmentação gene foi aqui testado numa variedade de aplicações de construção de vector. Em todos os casos aqui examinados, SPI mostrou-se eficiente e o plasmídeo recombinante correta foi produzido.Na maioria dos casos, as múltiplas cassetes diferentes foram correctamente inserido no vector de direccionamento. Grandes cassetes de ADN e vectores foram facilmente acomodados no protocolo SPI e demonstrou a flexibilidade do presente sistema.
SPI baseia-se na utilização de sequências longas de homologia e fosforotioato (PTO) protecção das cassetes de ADN linear. Modificação PTO confere protecção contra exonucleases para o ADN linear e a longo 20,21 HA aumenta a eficiência de recombinação para permitir a multiplexação. No entanto, a síntese de sequências oligo mais longos aumenta as possibilidades de acumulação de erros especialmente deleções. Mutações no oligo pode ser particularmente prejudicial se eles cobrem regiões de proteína codificação. Sequenciamento de DNA em todo o HA e cobrindo todo o comprimento da fita inserida é altamente recomendável para eliminar clones com quaisquer alterações de sequência. Uso de um sistema de polimerase de ADN de alta fidelidade também é sugerido para evitar a introdução de umy erros de PCR. O comprimento e composição do HA do plasmídeo de subclonagem é mais crítico em relação à inserção da cassete (dados não mostrados). Para aplicações particularmente sensíveis, tais como a construção de vectores knockin, onde quaisquer mutações em regiões de exão não são tolerados, o HA da cassete de inserção pode ser reduzido (pb 50-120) para evitar problemas associados com os oligos longos. A cassete de inserção também pode ser não modificado ou deixado phosphorothioated dupla (onde o conhecimento do sentido de replicação não está disponível). Mas multiplexing, nestes casos, ainda requer longo protegido HA plasmídeos subclonagem. Uma advertência desta estratégia particular é a diminuição da eficiência de multiplexação que podem potencialmente impactar SPI clonagem em loci diferentes.
O comprimento da inserção da cassete e do plasmídeo de subclonagem é um outro parâmetro importante na SPI clonagem. Moléculas de ADN maiores electroporate menos eficientemente e o efeito cumulativo é, dado o reexi- para introduzir todos os alimentadores na mesma célula (resultados não apresentados). Multiplexing é mais eficiente com cassetes de inserção menores. DNA fragmentos maiores que 3 kb também colocar um limite no processamento ssDNA Red mediada, que é mais eficiente até 3 kb 30. Cassetes de inserção superiores a 3 kb são dual ressecada e recombinar com menos eficiência através de um beta via independente 20. Portanto, a seleção de colônias suficientes para identificar o clone correto torna-se importante nestes casos. A longa duração da recombinação pós eletroporação também aumenta as chances de recuperação do clone correto em exercícios SPI difíceis. Até quatro pequenas cassetes (<1,5 kb) podem ser inseridos simultaneamente com o processo de SPI, embora a eficiência de multiplexação diminui com cada cassete adicional (dados não mostrados). No entanto, isso reflete o resultado de um experimento SPI óptima e é aconselhável a considerar os limites de multiplexação ao usar muitas grandes cassetes.
O desenvolvimento de ferramentas de edição de genoma como cluster repete palindrômicas curtas regularmente intercaladas (CRISPR) sistema endonuclease -cas9 permitiu a criação de novas modificações do genoma e tem levado a mais eficiente engenharia genoma 36. No entanto, essas tecnologias novas complementaram vectores de genes alvo em vez de suplantado eles. Prevê-se que o sistema CRISPR-cas poderia substituir seleção antibiótico e aperfeiçoar o protocolo recombineering multiplex.
Divulgações
T.R.R is the inventor of a British patent application on multiplex recombineering technology.
Agradecimentos
T.R.R conceived the project, designed and performed experiments and prepared the manuscript. E.J.K and S.E.M performed experiments. All authors contributed intellectually to the final manuscript. This work was funded by a University of Leicester Innovation Fellowship to SEM and by a UK Medical Research Council (MRC) Senior Research Fellowship to S.M.C (MR/J009202/1). DNA sequencing was performed by Protein and Nucleic Acid Laboratories (PNACL), Centre for Core Biotechnology Services, University of Leicester. We wish to thank A. Francis Stewart for providing the Rox and Dre plasmids. pL451 and pL452 plasmids were obtained from National Cancer Institute (NCI), Frederick, USA.
Materiais
| Name | Company | Catalog Number | Comments |
| Antibiotics except Zeocin | Sigma: http://www.sigmaaldrich.com | Ampicillin, A9518-5G; Chloramphenicol , C1919-5G; Blasticidin, 15205-25MG; Gentamicin, G1264-50MG; Hygromycin, H3274-50MG; Kanamycin,: K1876-1G; Tetracycline hydrochloride, T7660-5G; Trimethoprim, 92131-1G | |
| Zeocin | Invivogen: | ant-zn-1 | Zeocin is also available from Life technologies/Fisher |
| C57BL/6 BAC clones | CHORI: https://bacpac.chori.org/ | RPCI-23 or RPCI-24 BAC libraries | 129/Sv BAC clones can be obtained from Invivogen or Life technologies/Fisher |
| KOD hotstart DNA polymerase system | Millipore: https://www.merckmillipore.com | 71086-3 | |
| L-Arabinose | Sigma: http://www.sigmaaldrich.com | A3256-25G | |
| Long oligos | IDT: https://www.idtdna.com; Gene link: https://www.genelink.com | IDT Ultramers or Gene Link long oligos | PTO and Phosphosphate available as custom modifications. PAGE purification strongly recommended for long oligos. |
| MinElute PCR purification kit | Qiagen: http://www.qiagen.com | 28004 | Minimum elution PCR purifications kits are preferred. |
| QIAPrep Spin Mini Prep kit | Qiagen: http://www.qiagen.com | 27104 | Silica column or similar matrix kits are preferred. |
| Restriction enzymes | NEB: https://www.neb.com | DpnI: R0176L | See NEB website for a list of RE. |
Referências
- Dovey, O. M., Foster, C. T., Cowley, S. M. Histone deacetylase 1 (HDAC1), but not HDAC2, controls embryonic stem cell differentiation. Proc. Natl. Acad. Sci. USA. 107, 8242-8247 (2010).
- Harvey, M., et al. Spontaneous and carcinogen-induced tumorigenesis in p53-deficient mice. Nat. Genet. 5, 225-229 (1993).
- Waterhouse, P., et al. Lymphoproliferative disorders with early lethality in mice deficient in Ctla-4. Science. 270, 985-988 (1995).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51, 503-512 (1987).
- Hasty, P., Rivera-Pérez, J., Bradley, A. The length of homology required for gene targeting in embryonic stem cells. Mol. Cell. Biol. 11, 5586-5591 (1991).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. Nature. 317, 230-234 (1985).
- Hirata, R., Chamberlain, J., Dong, R., Russell, D. W. Targeted transgene insertion into human chromosomes by adeno-associated virus vectors. Nat. Biotechnol. 20, 735-738 (2002).
- Szostak, J. W., Orr-Weaver, T. L., Rothstein, R. J., Stahl, F. W. The double-strand-break repair model for recombination. Cell. 33, 25-35 (1983).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309, 255-256 (1984).
- Fu, J., Teucher, M., Anastassiadis, K., Skarnes, W., Stewart, A. F. A recombineering pipeline to make conditional targeting constructs. Methods Enzymol. Soriano, P. M., Wassarman, P. M. 477, Academic Press. 125-144 (2010).
- Zhang, Y., Buchholz, F., Muyrers, J., Stewart, A. A new logic for DNA engineering using recombination in Escherichia coli. Nat. Genet. 20, 123-128 (1998).
- Copeland, N., Jenkins, N., Court, D. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Muyrers, J., Zhang, Y., Testa, G., Stewart, A. Rapid modification of bacterial artificial chromosomes by ET-recombination. Nucleic Acids Res. 27, 1555-1557 (1999).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. USA. 97, 5978-5983 (2000).
- Sharan, S. K., Thomason, L. C., Kuznetsov, S. G., Court, D. L. Recombineering: a homologous recombination-based method of genetic engineering. Nature Protoc. 4, 206-223 (2009).
- Wang, J., et al. An improved recombineering approach by adding RecA to λ red recombination. Mol. Biotechnol. 32, 43-53 (2006).
- Datta, S., Costantino, N., Court, D. L. A set of recombineering plasmids for gram-negative bacteria. Gene. 379, 109-115 (2006).
- Fu, J., et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting. Nat. Biotechnol. 30, 440-446 (2012).
- Muyrers, J., Zhang, Y., Buchholz, F., Stewart, A. RecE/RecT and Redalpha/Redbeta initiate double-stranded break repair by specifically interacting with their respective partners. Genes Develop. 14, 1971-1982 (2000).
- Maresca, M., et al. Single-stranded heteroduplex intermediates in lambda Red homologous recombination. BMC Mol. Biol. 11, 54(2010).
- Mosberg, J. A., Lajoie, M. J., Church, G. M. Lambda Red recombineering in Escherichia coli occurs through a fully single-stranded intermediate. Genetics. 186, 791-799 (2010).
- Liu, P., Jenkins, N. A., Copeland, N. G. A highly efficient recombineering-based method for generating conditional knockout mutations. Genome Res. 13, 476-484 (2003).
- Chan, W., et al. A recombineering based approach for high-throughput conditional knockout targeting vector construction. Nucleic Acids Res. 35, e64(2007).
- Zhang, Y., Muyrers, J. P. P., Testa, G., Stewart, A. F. DNA cloning by homologous recombination in Escherichia coli. Nat. Biotechnol. 18, 1314-1317 (2000).
- Lee, E. C., et al. A highly efficient Escherichia coli-based chromosome engineering system adapted for recombinogenic targeting and subcloning of BAC DNA. Genomics. 73, 56-65 (2001).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Valenzuela, D. M., et al. High-throughput engineering of the mouse genome coupled with high-resolution expression analysis. Nat. Biotechnol. 21, 652-659 (2003).
- Bantscheff, M., et al. Chemoproteomics profiling of HDAC inhibitors reveals selective targeting of HDAC complexes. Nat. Biotechnol. 29, 255-265 (2011).
- Mulryan, K., et al. Reduced vas deferens contraction and male infertility in mice lacking P2X1 receptors. Nature. 403, 86-89 (2000).
- Subramanian, K., Rutvisuttinunt, W., Scott, W., Myers, R. The enzymatic basis of processivity in lambda exonuclease. Nucleic Acids Res. 31, 1585-1596 (2003).
- Antoch, M. P., et al. Functional identification of the mouse circadian Clock gene by transgenic BAC rescue. Cell. 89, 655-667 (1997).
- Rostovskaya, M., et al. Transposon-mediated BAC transgenesis in human ES cells. Nucleic Acids Res. 40, e150(2012).
- Giraldo, P., Montoliu, L. Size matters: use of YACs, BACs and PACs in transgenic animals. Transgenic Res. 10, 83-103 (2001).
- Hill, F., et al. BAC trimming: minimizing clone overlaps. Genomics. 64, 111-113 (2000).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36(2005).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339, 819-823 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados