Method Article
Subcloning più Insertion (SPI) - A Novel Metodo recombineering per la costruzione rapida di gene targeting Vettori
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Gene targeting methodologies can be used to generate transgenic mice with knockout, knock-in and tagged alleles. Here, we describe an improved method of recombineering in E. coli, that we term ‘subcloning plus insertion’, which can be used to generate custom gene targeting vectors rapidly.
Abstract
Gene targeting refers to the precise modification of a genetic locus using homologous recombination. The generation of novel cell lines and transgenic mouse models using this method necessitates the construction of a ‘targeting’ vector, which contains homologous DNA sequences to the target gene, and has for many years been a limiting step in the process. Vector construction can be performed in vivo in Escherichia coli cells using homologous recombination mediated by phage recombinases using a technique termed recombineering. Recombineering is the preferred technique to subclone the long homology sequences (>4kb) and various targeting elements including selection markers that are required to mediate efficient allelic exchange between a targeting vector and its cognate genomic locus. Typical recombineering protocols follow an iterative scheme of step-wise integration of the targeting elements and require intermediate purification and transformation steps. Here, we present a novel recombineering methodology of vector assembly using a multiplex approach. Plasmid gap repair is performed by the simultaneous capture of genomic sequence from mouse Bacterial Artificial Chromosome libraries and the insertion of dual bacterial and mammalian selection markers. This subcloning plus insertion method is highly efficient and yields a majority of correct recombinants. We present data for the construction of different types of conditional gene knockout, or knock-in, vectors and BAC reporter vectors that have been constructed using this method. SPI vector construction greatly extends the repertoire of the recombineering toolbox and provides a simple, rapid and cost-effective method of constructing these highly complex vectors.
Introduzione
Lo sviluppo di tecnologie di targeting dei geni ha consentito la costruzione di linee cellulari e modelli di topo per studiare diversi sistemi biologici 1-3. Una chiave prima fase nella modifica di una sequenza genomica è il progetto e la costruzione di un gene targeting vettore 4. Vettori targeting sono costrutti plasmide che portano l'allele di interesse contenente le modifiche desiderate (s) affiancato da un marcatore di selezione (ad esempio, la neomicina), e lunghe regioni genomiche necessarie per un efficiente ricombinazione omologa in cellule di mammifero 5. Modificazione genetica precisa si ottiene l'introduzione del vettore targeting in staminali embrionali (ES) cellule o cellule somatiche 6 7, in cui la ricombinazione omologa tra tratti identici di sequenza di DNA sul vettore targeting ei risultati locus genomico nel trasferimento della modificazione previsto al genoma mediante conversione genica 8. Tale modificellule ed ES possono essere iniettati in blastocisti di topo per produrre prole (chimere) che possono poi trasmettere questi alleli modificati tramite la linea germinale 9. Un percorso alternativo per produrre topi transgenici comporta la microiniezione di un vettore di espressione genica in singoli zigoti unicellulari topo, che porta l'integrazione casuale del vettore nel genoma del mouse 10. I metodi tradizionali di costruzione del vettore si sono basati su tradizionali 'copia e incolla' clonazione utilizzando enzimi di restrizione e ligasi del DNA per clonare le diverse marcatore di selezione e frammenti genomici in un vettore backbone. Tuttavia, un fattore limitante intrinseca della clonazione tradizionale è il posizionamento e la scelta di siti di restrizione, soprattutto con sequenze di DNA più lunghe. Ciò richiede spesso più passaggi subcloning ed introduce anche sequenze di DNA estraneo nel vettore, che spesso porta ad una minore efficienza di gene targeting.
Recombineering (eng recombinogenicineering) è una tecnologia di ingegneria DNA che supera queste limitazioni utilizzando la ricombinazione omologa (HR) mediato da proteine fago ricombinazione a E. coli 11,12. Da una regione di una sequenza omologa può servire come substrato di recombineering, i vincoli di disponibilità di siti di restrizione vengono rimossi. Le grandi sequenze di DNA possono essere facilmente modificati direttamente in vivo, quindi anche preservare la loro integrità strutturale 13. Recombineering è molto efficiente con omologie brevi (50 bp) 14 e, pertanto, le braccia di omologia (HA) possono essere comodamente incorporati in sequenze oligo sintetici. In un esperimento recombineering tipico, un oligo o un DNA a doppia elica (dsDNA) frammento contenente HA è elettroporate in recombineering competente E. coli contenenti il bersaglio sia situato sul cromosoma o su un plasmide 15. Il potenziale di ricombinazione è conferito dalla espressione inducibile della rec Redombination proteine del fago 16,17 o le proteine RecET del rac profago 18. Il exonuclease Red / Rece converte dsDNA lineare ad un singolo filamento di DNA (ssDNA) intermedio, che viene poi legato dal suo partner, Rosso / Rect, un singolo filamento di proteine ricottura (SSAP) 19. La ricottura di un lungo ssDNA o una breve oligo alla sua sequenza bersaglio complementare si verifica sul filamento in ritardo della forcella replica e conduce all'incorporazione della sequenza al sito bersaglio. Fanalino di filo ssDNA ricombinazione è la base della elevata efficienza di recombineering e può essere descritto dal 'beta' modello ricombinazione 20,21.
Un flusso di lavoro tipico recombineering per costruire un vettore gene targeting coinvolge uno dei due seguenti percorsi. Una via comporta subcloning regione genomica desiderata da un clone BAC topo in un plasmide seguita dall'inserimento sequenziale di siti di ricombinazione LoxP, un marcatore di selezione 22 ecc, o il percorso alternativo comporta mira il BAC locus genomico con i diversi elementi di targeting vettore di vari cicli di recombineering 10,23 e poi subcloning il locus modificato in un plasmide mediante riparazione gap clonazione 24,25. Variazioni su questo tema sono state utilizzate in diversi gasdotti ad alta produttività recombineering nell'ambito di programmi di produzione di grandi dimensioni del mouse 26,27. Tuttavia, queste procedure comportano fasi complicate e lunghe, richiedono l'uso di vettori specializzati e E. coli ceppi (ad esempio, cellule che esprimono Cre) e utilizzare uno o più stadi intermedi di purificazione del DNA vettoriale e ri-trasformazione (Tabella 1). Subcloning più inserimento (SPI) è una tecnica recombineering romanzo che combina beta ricombinazione e riparazione gap clonazione in un unico processo (Figura 1). Montaggio vettore SPI è semplice, veloce e flessibile e offre un miglioramento significativo recombi serieneering approcci (Tabella 1). Qui, dimostriamo la facilità e l'utilità di usare SPI per diverse applicazioni della costruzione del vettore con una particolare enfasi sulla costruzione di disegni vettoriali non standard e stimolanti. Casi di test inclusi la costruzione di una fluorescenza giornalista knockin vettore, una duplice espressione proteica tag Knockin vettore, un BAC fluorescenza giornalista vettoriale e un ko vettore di condizionale. Questi esaminate variamente il requisito di localizzare un recettore sulla superficie cellulare, purificare un complesso proteico nucleare o condizionatamente ablazione l'espressione di un gene.
Protocollo
1. Gene Targeting design
- Ordinare un adeguato clone BAC che copre la regione genomica di interesse. Assicurarsi che questo è isogenico al tipo di cellule ES da modificare ad esempio, cloni RPCI-23 e RPCI-24 BAC per gene targeting con C57BL / 6 ES cellule.
- Applicare gene convenzionale criteri di targeting durante la progettazione del vettore targeting. I parametri chiave includono se la modifica è richiesto di essere costitutiva o condizionale, definendo esoni critiche (CE) per la cancellazione in una strategia gene knockout e la spaziatura e la collocazione delle cassette intronici.
NOTA: Ognuno di questi è stato discusso in dettaglio altrove 10. - Scegli regioni genomiche ciascuno di 5-6 kb che fiancheggia il sito di modifica di destinazione.
NOTA: La dimensione dell'inserto subclonato è pertanto tipicamente 10-12 kb, anche se il limite superiore può essere alto come 80 kb con una copia bassa subcloning plasmide come p15A, ecc pBR322 e fino a 200 kb con BACvettoriale.
2. Multiplex recombineering Oligos
- Oligo di progettazione per la spina dorsale plasmide (subcloning plasmide) e il marcatore (cassette di inserimento). Progettare ogni oligo tal da contenere 180 bp HA fiancheggianti il sito di destinazione genomico e 20 bp di sequenza specifica priming della cassetta di inserimento o il plasmide subcloning (Figura 2).
NOTA: braccia omologia non dovrebbero contenere elementi ripetitivi, presenza di elementi ripetitivi comporterà il targeting errata e subcloning. Elementi ripetitivi possono essere rilevati utilizzando strumenti web based, come il '' Repeat Maschera ''. - Include un enzima di restrizione (RE) sito unico su una delle oligo vettoriali per linearizzare il vettore gene targeting per cellule ES di targeting (Figura 2).
- Controllare i parametri di oligo utilizzando un programma di analizzatore di oligo. Fare attenzione ad evitare strutture secondarie nella regione primings.
- Shorter HA (50 bp) della cassetta di inserimento è tollerato e produce minor numero di ricombinanti, ma assicurarsi che il plasmide subcloning HA è sempre più lungo (180 bp).
- Determinare l'orientamento dell'inserto di DNA genomico del particolare clone BAC utilizzando strumenti web based come '' cloneDB ''. Stabilire la direzione di replica da Oris controllando la mappa della backbone plasmide BAC utilizzati nella costruzione della libreria BAC.
NOTA: ORIS è generalmente opposta alla direzione trascrizionale del marcatore Cloramfenicolo (Chl) per tutti i plasmidi BAC comunemente usati. - Aggiungere due fosforotioato (PTO) legami terminali all'estremità 5 'del oligo che è opposta alla direzione di replica sul clone BAC. Aggiungere una modifica fosfato 5 'alla oligo inversa (Figura 2).
NOTA: Il asimmetrica phosphorothioated cassette PCR su digestione Red genera un ssDNA intermedio che può innescare il filo in ritardodella forcella replica.
NOTA: massima frequenza di ricombinazione multiplex si osserva con le cassette protette filo in ritardo. Leading filo protetta o cassette protette doppio non può produrre efficienza di ricombinazione equivalenti ad una loci. - Ordinare oligo con PAGE o HPLC purificazione.
3. BAC Trasformazione Clone con pSC101 BADgbaA recombineering plasmidi.
- Per rendere la E. coli recante la recombineering BAC abile, trasformarlo con il plasmide pSC101 BADgbaA contenente i geni rossi 16.
NOTA: Il plasmide pSC101 BADgbaA contiene i geni rossi e RecA sotto il controllo del Arabinose inducibile araC-P promoter BAD, un marcatore di selezione tetraciclina e la temperatura sensibile pSC101 replicone.- Pugnalare un puntale sterile cultura BAC agar e inoculare 5,0 ml di brodo lisogenia (LB) pH 8,0 contenente 12.556; g ml -1 Chl. Grow a 37 ° C per 5 ore in agitazione a 200 rpm.
- Raffreddare 10% (v / v) soluzione di glicerolo, microprovette e cuvette elettroporazione su ghiaccio. Raffreddare una grande centrifuga refrigerata e microcentrifuga a 4 ° C.
- Determinare la densità ottica (OD) della coltura utilizzando uno spettrofotometro e misurare l'assorbanza a 600 nm. Preparare cellule electrocompetent (come descritto sotto) quando viene raggiunto un OD 600 lettura da 0.3 a 0.8.
- Spin le cellule in una provetta da centrifuga da 50 ml in un grande centrifugare a 1.216 xg per 5 minuti a 4 ° C.
- Lavare le cellule con 1 ml di acqua refrigerata 10% glicerolo e spin giù cellule a 17.949 xg per 20 sec a 4 ° C. Eseguire il lavaggio passo per un totale di 3 volte.
- Risospendere le cellule in un volume totale di 50 ml di 10% glicerolo e aggiungere 10-200 ng del plasmide recombineering pSC101 BADgbaA. Ottenere una singola sospensione cellulare pipettandosu e giù parecchie volte e poi trasferire le cellule ad una pre-refrigerati 1 mm di aria elettroporazione cuvetta.
- Elettroporazione le cellule con una cornice di 1,8 kv, 25 uF e 200 Ω.
NOTA: Controllare le impostazioni corrette per ogni marca di elettroporatore. Una costante di tempo di elettroporazione inferiore 4 indica la presenza di sale e altre impurità. - Immediatamente recuperare le cellule in 1 ml di LB e trasferire le cellule in una provetta da centrifuga da 50 ml.
- Far crescere la cultura BAC gbaA a 30 ° C per 2 ore agitazione a 200 rpm.
NOTA: Il plasmide pSC101 BADgbaA si perde quando le cellule sono coltivate a 37 ° C a causa di inattivazione del fattore di replica rEPE sensibili alla temperatura. Crescere cellule gbaA a 30 ° C per mantenere le funzioni recombineering. - Aggiungere 9 ml di LB contenente 12,5 mg ml -1 Chl e 4 mg ml -1 tetraciclina (Tet) alla cultura BAC gbaA recuperato. Far crescere laBAC gbaA cultura O / N a 30 ° C agitazione a 200 rpm.
NOTA: L'efficienza della trasformazione electroporating del gbaA plasmide supercoiled è sufficientemente alta da permettere la crescita saturazione O / N in mezzi liquidi.
4. Preparazione di inserimento cassette e Subclonaggio Plasmidi
- Per incorporare HA nel cassetto di inserimento (s) e il plasmide subcloning, effettuare la reazione a catena della polimerasi (PCR) utilizzando oligonucleotidi lunghi modificati come descritto di seguito.
- Optional: per evitare plasmide riporto nella reazione recombineering, utilizzare un'origine R6K o simile modello ristretta gamma di ospiti plasmide per amplificare la cassetta di inserimento.
- In alternativa, linearizzare il modello plasmide con un RE digest (Tabella 2). Scegliere un RE che taglia fuori la regione di amplificazione PCR ed è il calore inattivato. Il calore inattivare la RE come raccomandato dal manifacturer.
- Impostare la reazione a catena della polimerasi (PCR) utilizzando un sistema di polimerasi hotstart DNA ad alta fedeltà. Preparare una master mix PCR come indicato (tabella 3). Eseguire cicli termici come mostrato (Tabella 3).
- Analizzare PCR prodotti di gel elettroforesi. Carico 1-5 microlitri di ogni PCR su un 1% (w / v) gel di agarosio contenente 0,5 mg ml -1 etidio bromuro (EtBr).
NOTA: La presenza di prodotti non specifici amplificazione non interferisce nella reazione recombineering. In alcuni casi, tuttavia, di primer-dimeri possono diminuire l'efficienza recombineering. - Purificare prodotti PCR utilizzando un kit di purificazione PCR.
- Optional: rimuovere il modello plasmide da PCR per trattamento con DpnI seguita da PCR clean-up. DNA Eluire in un volume minimo di acqua deionizzata sterile (come raccomandato dal costruttore).
NOTA: L'aggiunta di DpnI per non purificata PCR reazioni risultati in ridotta efficienza di scissione di the template metilato DNA plasmide.
- Optional: rimuovere il modello plasmide da PCR per trattamento con DpnI seguita da PCR clean-up. DNA Eluire in un volume minimo di acqua deionizzata sterile (come raccomandato dal costruttore).
- Quantificare PCR DNA amplificato mediante analisi gel contro una serie nota di standard di DNA ad esempio, λ HindIII digest o utilizzando uno spettrofotometro NanoDrop.
5. Subclonaggio più Inserimento
- Diluire la O / N BAC gbaA cultura 50 volte con l'aggiunta di 200 ml in 10 ml LB + Chl + Tet. Coltivare a 30 ° C agitazione a 200 rpm per 1 ora 50 min. Include un campione da utilizzare come controllo negativo.
- Raffreddare tutti i materiali e le attrezzature recombineering a 4 ° C, come descritto al punto 3.1.2.
- Versare LB agar pH 8 piastre contenenti la corretta concentrazione di antibiotico selettiva appropriata (s) che permetterà la selezione del frammento di DNA che viene inserito e per la selezione del vettore subcloning (Tabella 4).
NOTA: Alcuni antibiotici sono pH sensibili. Usa LBagar pH 8 come una regola. - Preparare 10% (w / v) di L-arabinosio. Filtrare sterilizzare attraverso un filtro a siringa da 0,2 micron.
NOTA: arabinosio induce l'espressione delle proteine recombineering dal plasmide gbaA. - Controllare il diametro esterno della cultura BAC gbaA utilizzando uno spettrofotometro. Una volta che un OD 600 di 0,25-0,3 viene raggiunto, indurre le proteine recombineering come descritto nel passaggio seguente.
- Aggiungere 200 microlitri della soluzione di arabinosio 10% a 10 ml della coltura BAC per ottenere una concentrazione finale di arabinosio del 0,2%. Includere una cultura non indotte (senza Arabinose) da utilizzare come controllo negativo.
- Trasferire la cultura BAC a 37 ° C incubatore agitazione e indurre l'espressione rosso per 45 minuti agitando a 230 rpm.
NOTA: L'espressione di proteine rosse è inefficiente a 30 ° C. - Spin le cellule e lavare con il 10% glicerolo 3 volte come descritto al punto 3.1.5.
- Aggiungi 600-1,000 ng del plasmide ciascuna subcloning e la cassetta di inserimento (s) per la reazione recombineering. Includere un vettore solo e vettoriale, più controlli singoli inserti di verificare la competenza ricombinazione e l'integrità del vettore e le cassette.
- Ottenere una sospensione di cellule singole pipettando su e giù. Eseguire elettroporazione come descritto al punto 3.1.7 e successivo recupero in 1 ml LB a 37 ° C per 1 ora per plasmidi multi-copia o in 10 ml di LB a 37 ° C per 3 ore per i vettori BAC.
- Piatto diverse diluizioni della coltura recuperato esempio, 90%, 10%, 1% sulle piastre doppi selezione agar e crescere a 37 ° C per 16 ore.
6. Analisi dei ricombinanti
- Optional: Scegliere da 6 a 12 colonie ed eseguire colonia PCR con un accompagnamento di fondo HA e un primer specifico inserto. Includere il plasmide subcloning e la cas inserimentosette plasmide così come il clone BAC genitore come controlli negativi.
- Eseguire l'analisi gel delle PCR colonia. Identificare cloni positivi per la presenza di una banda luminosa alla dimensione attesa.
NOTA: L'alta efficienza del processo di clonazione SPI genera ricombinanti principalmente corrette.
- Eseguire l'analisi gel delle PCR colonia. Identificare cloni positivi per la presenza di una banda luminosa alla dimensione attesa.
- Selezionare colonie in 5 ml LB pH 8 contenente l'antibiotico selettiva e crescere O / N a 37 ° C.
- Preparare minipreps DNA utilizzando un kit di purificazione colonna come da istruzioni del produttore.
- Preparare BAC miniprep DNA utilizzando standard di isolamento fenolo-cloroformio o un protocollo simile.
- Eseguire digerisce RE sul DNA miniprep. DNA separato mediante elettroforesi su gel di agarosio. Analizzare i modelli RE per identificare i cloni contenenti i frammenti formati attesi di targeting vettore di corretta. Scegliere un RE che discrimina chiaramente tra il vettore carente inserto (s) e il vettore contenente l'inserto (s).
- Optionale: per ottenere cellule che sono privi della BAC, che è ancora presente nella E. coli dopo recombineering, utilizzare il DNA miniprep plasmide trasformare DH5alpha o DH10B E. coli.
- Eseguire il sequenziamento del DNA attraverso l'HA e cassette di inserimento per verificare gli errori di sintesi oligo.
Risultati
Vettori Knockin
Vettori mirati Knockin riflettono la necessità di introdurre una caratteristica nuova sequenza nel genoma compresi singola coppia di basi sostituzione in una regione codificante della proteina, la fusione di un marcatore fluorescente o un tag di affinità per una proteina o l'integrazione di una cassetta di espressione genica. Per testare l'applicazione di SPI in strategie di costruzione Knockin vettore, sono stati esaminati due casi di test differenti. Dnttip1 codifica l'deoxynucleotidyltransferase, terminale, interagendo proteina 1A (TDIF1) che insieme alla classe I istone deacetilasi (HDAC) formano un complesso deacetilasi mitotico (Midac) 28. Per studiare il ruolo di Dnttip1 nella divisione cellulare, un approccio codifica tandem-affinità è stata presa per isolare il TDIF1 proteine interagenti. I precedenti tentativi di subclone una porzione del gene Dnttip1 utilizzando un vettore p15A contenente regioni di omologia lunghi portato a partire gap efficienza riparazione unnd frequenti prodotti ricombinazione aberranti (dati non riportati). Pertanto, la costruzione di un vettore knockin al locus Dnttip1 disponibile un esercizio recombineering impegnativo. Una strategia SPI è stato progettato per subclone una sezione 12 kb del gene Dnttip1 attraversa l'ultimo esone (esone 13) in una copia a basso p15A vettoriale e di inserire contemporaneamente un tag affinità doppia cassetta selezione in esone 13, sostituendo il codone di stop (Figura 3A ). La 2X proteina legante FLAG-calmodulina (CBP) legata FRT-PGK-EM7-neomicina (Neo) -BGhpA-FRT cassetta (2.0 kb) è stato amplificato da un RE linearizzato plasmide utilizzando dual PTO oligos modificati che conteneva 120 bp HA fiancheggia la Dnttip1 interrompere codone. Un p15A ZEO Dnttip1 subcloning vettore (1,7 kb) è stato costruito, che contenevano 200 bp regioni omologhe alle estremità della sequenza Dnttip1 da subclonate. Il plasmide subcloning Dnttip1 stato RE linearizzato e PCR amplificato utilizzando 20 bp modificato oligosche ha generato un importante filone di vettore protetta. I prodotti di PCR sono stati DpnI trattati, purificata e co-elettroporate in recombineering competente Dnttip1 BAC E. coli e cellule di controllo non indotte. Le reazioni sono state seminate su SPI Zeocin (Zeo) e kanamicina (Kan) contenente agar (Figura 3B). SPI ha prodotto il correttamente modificato Dnttip1 Knockin vettore in tutti i 12 ricombinanti analizzati (Figura 3C). Sequenziamento del DNA ha verificato l'assenza di errori derivanti dalla sintesi oligo o amplificazione PCR.
Un altro esempio di SPI coinvolto l'inserimento in-frame di una proteina fluorescente gialla migliorato (EYFP) collegato cassette selezione al gene P2rx1. Il gene codifica per un recettore P2rx1 G-protein-coupled che funziona come un canale ionico ATP-dipendenti 29. Linkage a YFP fluorescenza permette la tracciabilità del recettore P2X1 sulla superficie cellulare in vasaggi funzionali rious. P2rx1 è un altro luogo difficile, che ha presentato problemi significativi con metodologie recombineering convenzionali (dati non riportati). Utilizzando SPI, un segmento 12 kb del gene P2rx1 comprende l'esone terminale (esone 12) è stato subclonato in un vettore p15A zeo e modificato con l'inserimento di una cassetta concertata Neo EYFP -LoxP affiancato sostituendo il codone di stop nell'esone 12 per costruire il P2rx1-EYFP Knockin vettore (Figura 3D). In questo esempio, il filamento in ritardo protetta vettoriale p15A (1.7 kb) conteneva 230 bp HA e la cassetta EYFP conteneva 50 bp HA ed era anche lasciato non modificato. La cassetta inserimento EYFP (2.7 kb) è stato assemblato da splicing sovrapposizione PCR del gene EYFP, PCR amplificato da pEYFP-C1, e la cassetta LoxP- PGK-EM7-Neo-BGhpA-loxP, PCR amplificato da pL452 (NCI, Frederick) . La reazione SPI prodotto centinaia di colonie (dati non mostrati). RE analisi del DNAminipreps preparati con 11 cloni hanno mostrato la maggior conteneva il corretto montaggio P2rx1-EYFP vettore Knockin (Figura 3E). Convalida addizionale dal sequenziamento del DNA ha mostrato errori inserimento della cassetta EYFP. Tuttavia, alcuni cloni hanno mostrato profili del gap senza tag riparato e il divario riparato plasmidi tag nella stessa cella (corsie 6-9). Alcuni campioni contenevano anche erroneamente gap riparati (corsia 2) o plasmidi mistargeted (corsie 4 e 5). Il fallimento di subcloning concomitante e il targeting in alcuni ricombinanti SPI mette in evidenza i limiti della clonazione efficiente SPI quando si utilizzano grandi cassette (> 3 kb), che derivano dai vincoli processivity rosso di lunghi frammenti di DNA 30, o se si utilizza brevi HA. Infatti, aumentando la HA della cassetta EYFP a 200 bp maggiore efficienza SPI e la corretta destinazione della cassetta (dati non mostrati). Gene targeting con il P2rx1 - EYFP Knockin vettoriale in JM8.N4cellule di topo ES (C57BL / 6 ceppo) generati 6 cloni positivi su 96, che conteneva il bersaglio corretto sequenza P2rx1-EYFP (Figura 3F).
BAC Reporter Vettori
Un clone BAC del gene bersaglio spesso contiene tutti gli elementi necessari a monte ea valle di regolamentazione ad esempio, esaltatori, UTRs ecc così il promotore endogeno di guidare l'espressione genica a livelli naturali 31. Un vettore BAC giornalista è quindi il veicolo preferito per ricapitolare il pattern di espressione di un gene endogeno 32. Tuttavia, le grandi dimensioni di un plasmide BAC (fino a 200 kb) presenta notevoli problemi pratici con un BAC trasfezione in cellule intatte 33. La riduzione delle dimensioni del BAC dell'inserto genomico attraverso BAC rifilatura 34,35, pur mantenendo gli elementi regolatori necessari di un gene, consente una più facile gestione del BAC e provoca transfecti più efficienteon. Attuale tecnologia di ingegneria BAC coinvolge vari cicli di recombineering per raggiungere questo obiettivo 35. Per dimostrare l'utilità di SPI in BAC rifilatura, un vettore pBeloBAC11 BAC è stato usato per subclone una sequenza genomica 30 kb compreso il pieno gene lunghezza P2rx1 dal 168 kb P2rx1 BAC insieme con l'inserimento simultaneo di una cassetta EYFP nel gene P2rx1 (Figura 4A). Il ZEO vettore backbone pBeloBAC11 (6.5 kb) contenente 180 bp HA è stato amplificato con PCR oligonucleotidi modificati che hanno generato un vettore protetto filamento in ritardo. Lo stesso cassette EYFP Neo (3 kb), utilizzati nel precedente P2rx1 knockin vettore costruzione, era PCR amplificato utilizzando oligonucleotidi protette filamento in ritardo contenenti 180 bp HA e mirato il P2rx1 esone 12 sostituendo il codone di stop. Dopo elettroporazione combinato della cassetta EYFP e il vettore di zeo pBeloBAC11 in P2rx1 BAC cellule espressing le proteine recombineering gbaA, la coltura è stato recuperato in 10 ml LB pH 8 per 3 ore a 37 ° C per separare i plasmidi BAC. Pure ricombinanti sono stati selezionati su Zeo e Kan agar piastre e sono stati osservati con una frequenza di 2 x 10 -6. Analisi genotipizzazione Colony PCR ha rivelato BAC successo taglio nei 3 su 6 cloni che sono stati analizzati (Figura 4B). Lungo raggio amplificazione PCR attraverso il sito di inserzione EYFP confermato l'incorporazione cassette corretto nelle tre cloni positivi (Figura 4B). I tre cloni privi dell'inserto EYFP erano anche gap erroneamente riparato a 5 ', se la causa della mistargeting della cassetta EYFP in questi cloni può essere separato per la corretta chiusura del 5' estremità BAC. I tre cloni EYFP positivo BAC sono stati ulteriormente analizzati con digerisce RE e ha mostrato l'evoluzione prevista del ritagliata correttamente EYFP ricombinante BAC (Figura 4C </ Strong>). L'analisi di sequenza ha rivelato l'assenza di errori nella cassetta EYFP solo 1 clone dei 3 positivi.
Knockout condizionale (CKO) Vettori
Ablazione condizionale dell'espressione genica è uno strumento importante per studiare i processi di sviluppo o per studiare sistemi biologici in un determinato punto del tempo. Una strategia knockout gene condizionale in genere comporta il posizionamento di siti di ricombinazione LoxP circondano un esone critico (CE). La cancellazione di un CE sulla espressione Cre o l'attivazione produce un frameshift e un codone di stop prematuro, con conseguente degrado del mRNA causa di nonsense decadimento mediato (NMD). La costruzione di un vettore gene targeting condizionale è un compito complesso e coinvolge diversi passaggi di subcloning, il targeting e la trasformazione 22. SPI offre un percorso conveniente per semplificare questo processo. Come caso di test, un allele condizionale del gene Zrsr2 stato costruitoutilizzando la metodologia SPI (Figura 5A). Il gene codifica per un fattore Zrsr2 splicing e una singola copia si trova sul cromosoma X. Lo stato di delezione condizionale gene è particolarmente importante in questo caso per controllare la possibilità di adattare cellule della mancanza di Zrsr2 durante la selezione di cellule ES in una strategia delezione gene targeting costitutiva. SPI è stata eseguita per un subclone porzione 10 kb del gene ZrSr2 con inserimento simultaneo di due diverse cassette di selezione loxP affiancate. La FRT-PGK-EM7-Neo-FRT-loxP cassetta (2 kb) è stato amplificato da PCR RE pL451 con ritardo filo protetta oligonucleotidi che conteneva 180 bp HA targeting ZrSr2 introne 2. A seconda cassetta contenente Rox-PGK-em7- digerito Blasticidin (Bsd) -Rox-loxP (2 kb) è stato PCR amplificato da un plasmide R6K con oligonucleotidi protette filamento in ritardo contenenti 180 bp HA identica a una regione a valle in introne 3. I due siti loxP affiancati esone 3, la CE la cui dELIMINAZIONE su condizionali Cre risultati di attivazione in un frameshift e introduce un codone di stop prematuro. Il plasmide subcloning Zrsr2 (1.6 kb) è stato PCR amplificato da un RE linearizzato p15A zeo plasmide usando ritardo filo protetta oligo contenenti 180 bp HA corrispondere le estremità del 10 kb sequenza Zrsr2. Reazioni SPI sono state eseguite come descritto prima e placcato su piastre Zeo + Neo + BSD. Il ZrSr2 Targeting condizionale vettoriale è stato completato con successo nella maggior parte dei 12 ricombinanti esaminati (Figura 5B). Ulteriori analisi di sequenziamento del DNA ha dimostrato corretto inserimento di entrambi i marcatori di selezione nel vettore ZrSr2 CKO.

Figura 1. SPI clonazione. Sommario del processo di clonazione SPI combinando inserimento della cassetta e subcloning in un unico passaggio. (A) Il clone BAC è transformed con il plasmide pSC101 BADgbaA e cresciuto a 30 ° C. (B) A seguito di induzione arabinose per esprimere le proteine rosse, l'asimmetrica cassette inserimento modificato e plasmide subcloning vengono introdotti nel clone BAC e selezionato sia con l'inserimento e subcloning marcatori per generare il vettore finale. Clicca qui per vedere una versione più grande questa figura.
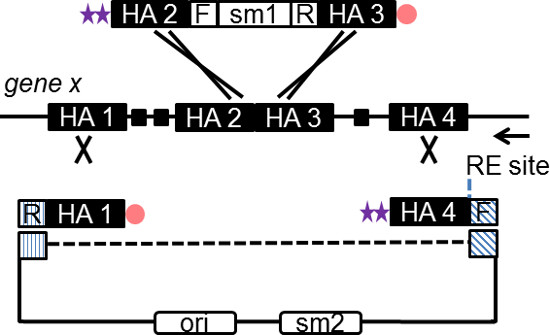
Figura 2. schematica illustrante la progettazione di oligonucleotidi recombineering SPI. Il plasmide subcloning e cassette di inserimento sono entrambi generati mediante PCR utilizzando una combinazione di PTO terminale e fosfato di oligonucleotidi modificati. Il frammento di PCR su digestione rosso in vivo, produce una ssDNA intermedio, che annealing al filamento in ritardo della forcella di replica. Gene specifica HA di 50-180 bp è incorporato in ciascun oligo come mostrato. La freccia indica la direzione di replicazione del DNA in un gene candidato. Linea tratteggiata rappresenta sequenza plasmide subcloning non incorporata nel prodotto della PCR. Sito RE, sito enzima di restrizione di linearizzazione di vettore di finale; F, la sequenza in avanti (20 bp) specifici per il plasmide subcloning o cassette di inserimento; R, sequenza di complemento inverso (20 bp) del plasmide o cassetta; sm, marcatore di selezione. Cliccate qui per vedere una versione più grande di questa figura.

Figura 3. SPI consente la costruzione di vettori knockin difficili. (A) Schema della strategia SPI utilizzato nella costruzione del Dnttip1 dual etichettato vettoriale. Arrow indiCates la direzione della replica sul clone BAC. risultati (B) placcatura dei campioni non indotte e indotte dell'esperimento Dnttip1 SPI. (C) EcoRI digest di cloni Dnttip1 SPI. M, 1 kb scala (NEB); C, gap p15A Dnttip1 riparato plasmide manca la cassetta dual tag. Dimensioni Frammento sono: Tagged, 9.2 + 4.4 + 2.2 kb; controllo; 9.2 + 4.6 kb. (D) SPI basate P2rx1-EYFP costruzione del vettore Knockin. (E) EcoRI digest di cloni P2rx1-EYFP SPI. M, 1 kb + scala (Invitrogen); C, gap p15A P2rx1 riparato plasmide manca la cassetta EYFP. Dimensioni Frammento sono: Tagged, 8.3 + 4.4 + 3.1 + 0,03 kb; controllo; 8.3 + 3.1 + 1.8 + 0.03 kb (F) analisi Southern blot di P2rx1 -. Gene EYFP targeting cellula cellule JM8.N4 ES. Pannello superiore, Southern blot con la sonda 3 'con PshAI digest. Viene mostrato il risultato proiezione di 5 cloni. Pannello inferiore, Southern blot utilizzandola 'sonda di fine e SpeI digest dei positivi individuati dal 3' 5 di screening fine. Sito PshAI RE; S, sito SpeI RE. La linea tratteggiata rappresenta la fine del HA vettoriale. Scatola nera indica sonda sud. Frammenti di restrizione attesi sono, PshAI: WT, 8.9 kb; EYFP-neo, 11,5 kb; SpeI:. WT, 6,9 kb; EYFP-neo, 7.6 kb Clicca qui per vedere una versione più grande di questa figura.
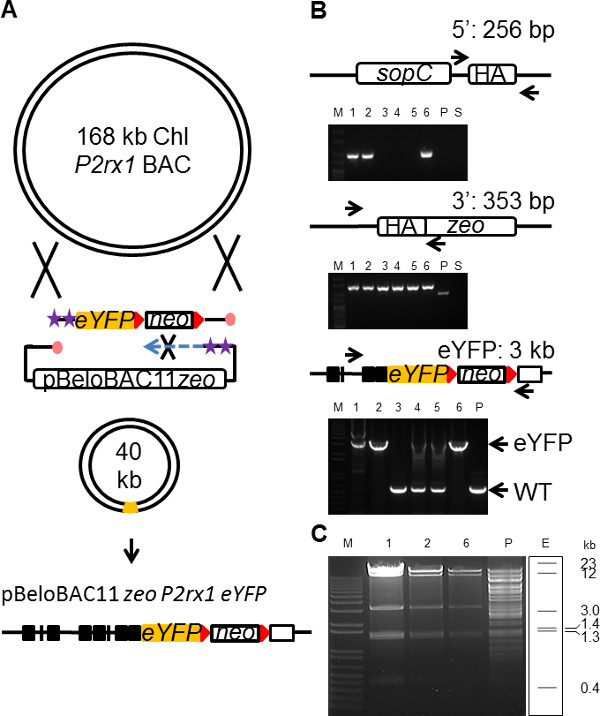
Figura 4. Semplificata BAC taglio utilizzando SPI. (A) Schema che illustra il concetto di BAC taglio utilizzando SPI. Legenda simboli è descritto in Figura 1. Amplificazione (B) attraverso il PCR 5 'e 3' estremità dell'inserto subclonato e tutti i insertio P2rx1-EYFPn sito. La strategia di screening è indicata per ogni tipo di PCR. M (pannello superiore), Hyperladder 25 bp, (pannelli inferiori), 1kb +; P, P2rx1 BAC; . S, pBeloBAC11 ZEO subcloning plasmide (C) HindIII digerire i tre EYFP positivi cloni SPI BAC da (B); E, atteso HindIII modello restrizione tagliato EYFP BAC. Cliccate qui per vedere una versione più grande di questa figura.
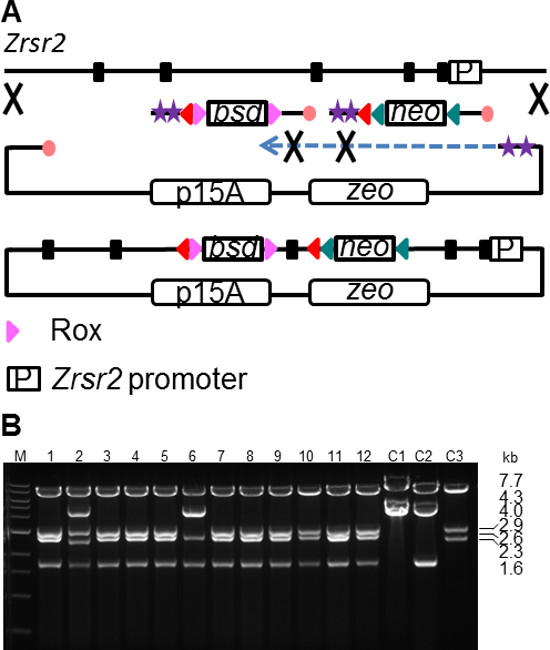
Figura 5. condizionale generazione knockout vettore utilizzando la clonazione SPI. (A) Schema di inserimento simultaneo di due diversi LoxP affiancato cassette di selezione durante subcloning dell'allele Zrsr2. Legenda simboli è descritto in Figura 1. (B) </ Digest strong> EcoRI di cloni Zrsr2 SPI. L, 1 kb scala (NEB); C1, p15A ZrSr2 gap riparato plasmide, C2, gap p15A ZrSr2 riparato plasmide contenente l'inserto neo; C3, gap p15A ZrSr2 plasmide contenente l'inserto bsd riparato. Dimensioni Frammento sono: CKO, 7.7 + 2.9 + 2.6 + 1.6 kb; C1, 7.7 + 4.0 kb; C2, 7.7 + 4.3 + 1.6 kb; C3, 7,7 + 2,9 + 2,3 kb. Cliccate qui per vedere una versione più grande di questa figura.
| No di passi | Recombineering convenzionale condotta una | Multiplex recombineering b |
| Fase 1 | Trasformazione di recombineering plasmide in serie BAC | Trasformazione di recombineering plasmide in serie BAC. Preparazione dei rivolte cassette e subcloning vettori. |
| Passo2 | Inserimento di cassette R1 / R2 Gateway | Gap Multiplex riparazione clonazione |
| Fase 3 | Inserimento di cassette Kan floxed | O / N cultura da singole colonie |
| Fase 4 | Gap riparazione in R3 / R4 plasmide | Miniprep e verifica |
| Passo 5 | Trasformazione in Cre + E. coli | |
| Passo 6 | Miniprep e verifica | |
| Step 7 | O / N a tre vie reazione Gateway | |
| Step 8 | Trasformazione del tre vie Gateway reazione in DH10B E. coli | |
| Step 9 | Cultura Overnight da singole colonie | |
| Step 10 | Miniprep e verifica la sequenza | |
| b Tempo medio di 4 giorni per clone verificato | ||
Tabella 1: Confronto tra recombineering convenzionale con SPI nella costruzione di vettori knockout condizionali.
| Buffer RE | 5 ml |
| DNA | 1 mcg plasmide o prodotti di PCR purificati |
| RE | 1 ml (5 o più unità) |
| TE | fino a 50 microlitri |
| Incubare a 37 ° C per almeno 1 ora. Riscaldare inacitvate secondo le istruzioni del produttore | |
Tabella 2: RE digest.
| Materiali PCR | Concentrazione finale |
| PCR buffer | 1x |
| dNTP | 200 Nm |
| MgSO4 | 1.5 mM |
| Betaina | 1.3 M |
| DMSO | 1% |
| Primer Forward | 200 Nm |
| Primer Reverse | 200 Nm |
| DNA polimerasi | 1 U |
| Sagoma | 10 ng di plasmidi multicopia o 2,5 ml di miniprep DNA per PCR genotipizzazione |
| Acqua | fino a 50 microlitri di una |
| a Per uno standard di 50 microlitri PCR con plasmidi multicopia. Lungo raggio PCR genotipizzazione sono stati istituiti in 25 microlitri reazioni PCR. | |
| Condizioni di PCR | |
| 95 ° C | 2 min |
| 92 ° C | 10 sec |
| 55 ° C | 30 sec |
| 72 ° C | 30 sec |
| 30 cicli b | |
| b ciclo non può essere esteso a 35 per BAC PCR genotipizzazione | |
Tabella 3: PCR Set-up e Condizioni.
| Antibiotici | Concentrazione un (mg ml -1) |
| Ampicllin | 50 |
| Blasticidin b | 40 |
| Cloramfenicolo | 12.5 |
| Gentamicina | 2 |
| Hygromycin c | 30 |
| Kanamicina b | 15 |
| Tetracycline | 4 |
| Trimetoprim c | 10 |
| Zeocin | 5 |
| raccomandabile per l'utilizzo con BAC e plasmidi multicopia quando usato in combinazione a recombineering multiplex | |
| b Blasticidin (35 mg ml -1) e kanamicina (6 mg ml -1) quando usate insieme in combinazione | |
| c Hygromycin e trimetoprim non sono raccomandati per la selezione con BAC copia singola. | |
Tabella 4. Consigliato concentrazioni di antibiotici per uso negli esperimenti SPI.
Discussione
Costruzione di linee di cellule ES e modelli di topo è storicamente coinvolto gene targeting utilizzando costrutti plasmide che contenevano l'allele 4 modificato. Tuttavia, la costruzione di questi complessi vettori gene targeting ha dimostrato di essere un collo di bottiglia significativo nella produzione tempestiva di tali modelli. Lo sviluppo di strategie di costruzione vettore recombineering base ha permesso di disegni vettoriali migliorate e assemblaggio vettore più efficiente. Tuttavia, protocolli recombineering attuali coinvolgono ancora più passaggi, richiedono intermedio di purificazione plasmide e utilizzano diversi ceppi batterici. Subcloning oltre inserimento offre un nuovo approccio alla costruzione del vettore che può essere eseguita in un evento elettroporazione nel ceppo ospite BAC residente. L'utilità di SPI in gene targeting stato qui testato in una varietà di applicazioni edili vettoriale. In tutti i casi esaminati qui, SPI ha dimostrato di essere efficiente e il plasmide ricombinante corretto è stato prodotto.Nella maggioranza dei casi, le molteplici cassette diversi stati correttamente inseriti nel vettore targeting. Grandi cassette di DNA e vettori erano facilmente alloggiati nel protocollo SPI e dimostrato la flessibilità del sistema.
SPI si basa sull'utilizzo di lunghe sequenze di omologia e fosforotioato (PTO) Protezione delle cassette di DNA lineari. Modifica PTO conferisce protezione da esonucleasi al DNA lineare 20,21 e la lunga HA aumenta l'efficienza di ricombinazione per consentire multiplexing. Tuttavia, la sintesi di sequenze oligo lunghi aumenta le possibilità di accumulare errori soprattutto le eliminazioni. Le mutazioni nel oligo può essere particolarmente dannoso se coprono le regioni codificanti proteine. Sequenziamento del DNA attraverso il HA e copre l'intera lunghezza della cassetta inserita è altamente raccomandato per eliminare cloni con alterazioni sequenza. L'uso di un sistema di DNA polimerasi ad alta fedeltà viene anche suggerito di evitare l'introduzione di unerrori y PCR. La lunghezza e la composizione del HA del plasmide subcloning è più critica rispetto a quella della cassetta di inserimento (dati non mostrati). Per applicazioni particolarmente sensibili come la costruzione di vettori Knockin, dove eventuali mutazioni in regioni esone non sono tollerati, l'HA della cassetta di inserimento può essere ridotto (50-120 bp) per evitare problemi connessi con lunghe oligo. La cassetta di inserimento può anche essere lasciata phosphorothioated non modificato o doppia (dove la conoscenza della direzione di replica non è disponibile). Ma multiplexing in questi casi richiede ancora molto protetto HA plasmidi subcloning. Un avvertimento di questa particolare strategia è l'abbassamento di efficienza multiplexing che possono potenzialmente influire clonazione SPI a diversi loci.
La lunghezza della cassetta inserimento e il plasmide subcloning è un altro parametro importante nella clonazione SPI. Molecole di DNA più grandi elettroporazione meno efficiente e l'effetto è cumulativo, dato il requirement introdurre tutte le cassette nella stessa cella (dati non mostrati). Multiplexing è più efficiente con cassette di inserimento più piccoli. DNA frammenti maggiore di 3 kb anche porre un limite elaborazione ssDNA Red mediata, che è più efficace fino a 3 kb 30. Cassette di inserimento superiori a 3 kb sono dual resecato e ricombinano meno efficiente attraverso una beta percorso indipendente 20. Pertanto, lo screening di colonie sufficienti a identificare il clone corretta diventa importante in questi casi. Una durata più lunga di ricombinazione dopo elettroporazione aumenta anche le possibilità di recupero del corretto clone in esercizi SPI difficili. Fino a quattro cassette piccole (<1,5 kb) possono essere inseriti simultaneamente con il processo di SPI, se l'efficienza multiplexing diminuisce con ciascuna cassetta aggiuntiva (dati non mostrati). Tuttavia, questo riflette il risultato di un esperimento ottimale SPI ed è opportuno considerare i limiti di multiplexing utilizzando molte grandi cassette.
Lo sviluppo di strumenti di editing del genoma come cluster brevi ripetizioni palindromiche regolarmente intervallati (CRISPR) sistema endonuclease -cas9 ha permesso la creazione di nuove modifiche del genoma e ha portato a più efficiente di ingegneria del genoma 36. Tuttavia, queste nuove tecnologie sono integrate vettori gene targeting piuttosto che soppiantato. Si prevede che il sistema CRISPR-cas potrebbe sostituire la selezione antibiotica e ulteriormente affinare il protocollo recombineering multiplex.
Divulgazioni
T.R.R is the inventor of a British patent application on multiplex recombineering technology.
Riconoscimenti
T.R.R conceived the project, designed and performed experiments and prepared the manuscript. E.J.K and S.E.M performed experiments. All authors contributed intellectually to the final manuscript. This work was funded by a University of Leicester Innovation Fellowship to SEM and by a UK Medical Research Council (MRC) Senior Research Fellowship to S.M.C (MR/J009202/1). DNA sequencing was performed by Protein and Nucleic Acid Laboratories (PNACL), Centre for Core Biotechnology Services, University of Leicester. We wish to thank A. Francis Stewart for providing the Rox and Dre plasmids. pL451 and pL452 plasmids were obtained from National Cancer Institute (NCI), Frederick, USA.
Materiali
| Name | Company | Catalog Number | Comments |
| Antibiotics except Zeocin | Sigma: http://www.sigmaaldrich.com | Ampicillin, A9518-5G; Chloramphenicol , C1919-5G; Blasticidin, 15205-25MG; Gentamicin, G1264-50MG; Hygromycin, H3274-50MG; Kanamycin,: K1876-1G; Tetracycline hydrochloride, T7660-5G; Trimethoprim, 92131-1G | |
| Zeocin | Invivogen: | ant-zn-1 | Zeocin is also available from Life technologies/Fisher |
| C57BL/6 BAC clones | CHORI: https://bacpac.chori.org/ | RPCI-23 or RPCI-24 BAC libraries | 129/Sv BAC clones can be obtained from Invivogen or Life technologies/Fisher |
| KOD hotstart DNA polymerase system | Millipore: https://www.merckmillipore.com | 71086-3 | |
| L-Arabinose | Sigma: http://www.sigmaaldrich.com | A3256-25G | |
| Long oligos | IDT: https://www.idtdna.com; Gene link: https://www.genelink.com | IDT Ultramers or Gene Link long oligos | PTO and Phosphosphate available as custom modifications. PAGE purification strongly recommended for long oligos. |
| MinElute PCR purification kit | Qiagen: http://www.qiagen.com | 28004 | Minimum elution PCR purifications kits are preferred. |
| QIAPrep Spin Mini Prep kit | Qiagen: http://www.qiagen.com | 27104 | Silica column or similar matrix kits are preferred. |
| Restriction enzymes | NEB: https://www.neb.com | DpnI: R0176L | See NEB website for a list of RE. |
Riferimenti
- Dovey, O. M., Foster, C. T., Cowley, S. M. Histone deacetylase 1 (HDAC1), but not HDAC2, controls embryonic stem cell differentiation. Proc. Natl. Acad. Sci. USA. 107, 8242-8247 (2010).
- Harvey, M., et al. Spontaneous and carcinogen-induced tumorigenesis in p53-deficient mice. Nat. Genet. 5, 225-229 (1993).
- Waterhouse, P., et al. Lymphoproliferative disorders with early lethality in mice deficient in Ctla-4. Science. 270, 985-988 (1995).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51, 503-512 (1987).
- Hasty, P., Rivera-Pérez, J., Bradley, A. The length of homology required for gene targeting in embryonic stem cells. Mol. Cell. Biol. 11, 5586-5591 (1991).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. Nature. 317, 230-234 (1985).
- Hirata, R., Chamberlain, J., Dong, R., Russell, D. W. Targeted transgene insertion into human chromosomes by adeno-associated virus vectors. Nat. Biotechnol. 20, 735-738 (2002).
- Szostak, J. W., Orr-Weaver, T. L., Rothstein, R. J., Stahl, F. W. The double-strand-break repair model for recombination. Cell. 33, 25-35 (1983).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309, 255-256 (1984).
- Fu, J., Teucher, M., Anastassiadis, K., Skarnes, W., Stewart, A. F. A recombineering pipeline to make conditional targeting constructs. Methods Enzymol. Soriano, P. M., Wassarman, P. M. 477, Academic Press. 125-144 (2010).
- Zhang, Y., Buchholz, F., Muyrers, J., Stewart, A. A new logic for DNA engineering using recombination in Escherichia coli. Nat. Genet. 20, 123-128 (1998).
- Copeland, N., Jenkins, N., Court, D. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Muyrers, J., Zhang, Y., Testa, G., Stewart, A. Rapid modification of bacterial artificial chromosomes by ET-recombination. Nucleic Acids Res. 27, 1555-1557 (1999).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. USA. 97, 5978-5983 (2000).
- Sharan, S. K., Thomason, L. C., Kuznetsov, S. G., Court, D. L. Recombineering: a homologous recombination-based method of genetic engineering. Nature Protoc. 4, 206-223 (2009).
- Wang, J., et al. An improved recombineering approach by adding RecA to λ red recombination. Mol. Biotechnol. 32, 43-53 (2006).
- Datta, S., Costantino, N., Court, D. L. A set of recombineering plasmids for gram-negative bacteria. Gene. 379, 109-115 (2006).
- Fu, J., et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting. Nat. Biotechnol. 30, 440-446 (2012).
- Muyrers, J., Zhang, Y., Buchholz, F., Stewart, A. RecE/RecT and Redalpha/Redbeta initiate double-stranded break repair by specifically interacting with their respective partners. Genes Develop. 14, 1971-1982 (2000).
- Maresca, M., et al. Single-stranded heteroduplex intermediates in lambda Red homologous recombination. BMC Mol. Biol. 11, 54(2010).
- Mosberg, J. A., Lajoie, M. J., Church, G. M. Lambda Red recombineering in Escherichia coli occurs through a fully single-stranded intermediate. Genetics. 186, 791-799 (2010).
- Liu, P., Jenkins, N. A., Copeland, N. G. A highly efficient recombineering-based method for generating conditional knockout mutations. Genome Res. 13, 476-484 (2003).
- Chan, W., et al. A recombineering based approach for high-throughput conditional knockout targeting vector construction. Nucleic Acids Res. 35, e64(2007).
- Zhang, Y., Muyrers, J. P. P., Testa, G., Stewart, A. F. DNA cloning by homologous recombination in Escherichia coli. Nat. Biotechnol. 18, 1314-1317 (2000).
- Lee, E. C., et al. A highly efficient Escherichia coli-based chromosome engineering system adapted for recombinogenic targeting and subcloning of BAC DNA. Genomics. 73, 56-65 (2001).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Valenzuela, D. M., et al. High-throughput engineering of the mouse genome coupled with high-resolution expression analysis. Nat. Biotechnol. 21, 652-659 (2003).
- Bantscheff, M., et al. Chemoproteomics profiling of HDAC inhibitors reveals selective targeting of HDAC complexes. Nat. Biotechnol. 29, 255-265 (2011).
- Mulryan, K., et al. Reduced vas deferens contraction and male infertility in mice lacking P2X1 receptors. Nature. 403, 86-89 (2000).
- Subramanian, K., Rutvisuttinunt, W., Scott, W., Myers, R. The enzymatic basis of processivity in lambda exonuclease. Nucleic Acids Res. 31, 1585-1596 (2003).
- Antoch, M. P., et al. Functional identification of the mouse circadian Clock gene by transgenic BAC rescue. Cell. 89, 655-667 (1997).
- Rostovskaya, M., et al. Transposon-mediated BAC transgenesis in human ES cells. Nucleic Acids Res. 40, e150(2012).
- Giraldo, P., Montoliu, L. Size matters: use of YACs, BACs and PACs in transgenic animals. Transgenic Res. 10, 83-103 (2001).
- Hill, F., et al. BAC trimming: minimizing clone overlaps. Genomics. 64, 111-113 (2000).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36(2005).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339, 819-823 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon