Method Article
Subclonación Plus Inserción (SPI) - Un nuevo método recombinería para la rápida construcción de la orientación de genes Vectores
En este artículo
Resumen
Gene targeting methodologies can be used to generate transgenic mice with knockout, knock-in and tagged alleles. Here, we describe an improved method of recombineering in E. coli, that we term ‘subcloning plus insertion’, which can be used to generate custom gene targeting vectors rapidly.
Resumen
Gene targeting refers to the precise modification of a genetic locus using homologous recombination. The generation of novel cell lines and transgenic mouse models using this method necessitates the construction of a ‘targeting’ vector, which contains homologous DNA sequences to the target gene, and has for many years been a limiting step in the process. Vector construction can be performed in vivo in Escherichia coli cells using homologous recombination mediated by phage recombinases using a technique termed recombineering. Recombineering is the preferred technique to subclone the long homology sequences (>4kb) and various targeting elements including selection markers that are required to mediate efficient allelic exchange between a targeting vector and its cognate genomic locus. Typical recombineering protocols follow an iterative scheme of step-wise integration of the targeting elements and require intermediate purification and transformation steps. Here, we present a novel recombineering methodology of vector assembly using a multiplex approach. Plasmid gap repair is performed by the simultaneous capture of genomic sequence from mouse Bacterial Artificial Chromosome libraries and the insertion of dual bacterial and mammalian selection markers. This subcloning plus insertion method is highly efficient and yields a majority of correct recombinants. We present data for the construction of different types of conditional gene knockout, or knock-in, vectors and BAC reporter vectors that have been constructed using this method. SPI vector construction greatly extends the repertoire of the recombineering toolbox and provides a simple, rapid and cost-effective method of constructing these highly complex vectors.
Introducción
El desarrollo de tecnologías de segmentación de genes ha permitido la construcción de líneas celulares y modelos de ratón para investigar diferentes sistemas biológicos 1-3. Una primera etapa clave en la modificación de una secuencia genómica es el diseño y la construcción de un gen vector dirigido 4. Vectores de abordaje son construcciones de plásmidos que llevan el alelo de interés que contiene las modificaciones deseado (s) flanqueados por un marcador de selección (por ejemplo, neomicina), y las regiones genómicas largos requeridos para la recombinación homóloga eficiente en células de mamífero 5. Modificación del gen preciso se consigue mediante la introducción del vector de orientación en células madre embrionarias (ES) 6 o células somáticas 7, con lo cual la recombinación homóloga entre tramos idénticos de secuencia de ADN en el vector de direccionamiento y los resultados locus genómico en la transferencia de la modificación prevista al genoma mediante conversión génica 8. Tal modified células ES pueden ser inyectadas en blastocistos de ratón para producir descendencia (quimeras) que luego pueden transmitir estos alelos modificados a través de la línea germinal 9. Una ruta alternativa para producir ratones transgénicos implica la microinyección de un vector de expresión de genes en cigotos de ratón unicelulares, que conduce a la integración aleatoria del vector en el genoma del ratón 10. Los métodos tradicionales de la construcción del vector se han basado en convencional 'cortar y pegar' clonación usando enzimas de restricción y ligasas de ADN para clonar el marcador de selección diferente y fragmentos genómicos en un esqueleto del vector. Sin embargo, un factor limitante inherente de clonación tradicional es el posicionamiento y la elección de los sitios de restricción, especialmente con secuencias de ADN más largos. Esto a menudo requiere múltiples etapas de subclonación y también introduce las secuencias de ADN extrañas en el vector, que a menudo conduce a una menor eficiencia de la orientación de genes.
Recombinería (eng recombinogenicineering) es una tecnología de la ingeniería de ADN que supera estas limitaciones mediante el uso de recombinación homóloga (HR) mediada por las proteínas de recombinación del fago en E. coli células 11,12. Dado que cualquier región de una secuencia homóloga puede servir como un sustrato de recombineering, se eliminan las limitaciones de disponibilidad de sitios de restricción. Las secuencias de ADN de gran tamaño pueden ser modificados sin problemas directamente in vivo, por tanto, también la preservación de su integridad estructural 13. Recombinería es muy eficiente con homologías cortas (50 pb) 14 y por lo tanto brazos de homología (HA) pueden ser convenientemente incorporados en las secuencias de oligo sintéticos. En un experimento típico recombinería, un oligo o un fragmento de ADN bicatenario (dsDNA) contiene HA se electroporación en recombinería E. competente células de E. coli que contienen el objetivo situado ya sea en el cromosoma o en un plásmido 15. El potencial de recombinación es conferida por la expresión inducible de la rec Redombination proteínas del fago 16,17 o las proteínas RECET del profago rac 18. La exonucleasa Red / RecE convierte dsDNA lineales a un ADN monocatenario (ssDNA) intermedio, que luego se une por su socio, Red / RecT, una proteína de recocido de una sola hebra (SSAP) 19. El recocido de una larga ssDNA o un corto oligo a su secuencia diana complementaria se produce en el capítulo menos del tenedor de replicación y conduce a la incorporación de la secuencia en el sitio diana. Retardamien hebra ssDNA recombinación es la base de la alta eficiencia de recombineering y puede ser descrita por el "beta" modelo de recombinación 20,21.
Un flujo de trabajo típico recombinería para construir un vector de orientación de genes involucra cualquiera de las dos siguientes rutas. Una ruta implica la subclonación de la región genómica deseada de un clon BAC de ratón en un plásmido seguido de la inserción secuencial de sitios de recombinación LoxP, un marcador de selección 22 etc., o la ruta alternativa implica la orientación del BAC locus genómico con los diferentes elementos de la orientación del vector de múltiples rondas de recombinería 10,23 y después subclonando el locus modificado en un plásmido mediante reparación brecha clonación 24,25. Variaciones sobre este tema se han utilizado en diferentes tuberías recombinería de alto rendimiento como parte de los programas de producción de gran ratón 26,27. Sin embargo, estos procedimientos implican etapas complicadas y largas, requieren el uso de vectores especializados y E. coli cepas (por ejemplo, células que expresan Cre) y utilizar una o más etapas intermedias de purificación de ADN vector y re-transformación (Tabla 1). Subclonación de inserción más (SPI) es una técnica recombineering novela que combina beta recombinación y reparación brecha de la clonación en un único proceso (Figura 1). Montaje vector SPI es simple, rápida y flexible y ofrece una mejora significativa en recombi estándaringe- se acerca (Tabla 1). Aquí, nos demuestran la facilidad y utilidad de usar SPI para diferentes aplicaciones en la construcción del vector con un énfasis particular en la construcción de diseños de vectores no estándar y desafiantes. Los casos de prueba incluyen la construcción de un vector knockin indicador fluorescente, una expresión de la proteína etiquetada vector knockin dual, un fluorescente reportero vector BAC y un vector knockout condicional. Estos examinaron diversamente el requisito de localizar un receptor de superficie celular, purificar un complejo de proteína nuclear o condicionalmente ablación de la expresión de un gen.
Protocolo
1. gene targeting Diseño
- Ordene un clon BAC apropiado que cubre la región genómica de interés. Asegúrese de que este es isogénica con el tipo de células madre embrionarias que ser modificado, por ejemplo, los clones RPCI-23 y RPCI-24 BAC para la orientación de genes con C57BL / 6 ES células.
- Aplicar gen convencional criterios de orientación en el diseño de los ataques de vectores. Los parámetros clave incluyen si se requiere la modificación a ser constitutivo o condicional, definiendo exones críticos (CE) para la supresión en una estrategia gen knockout y el espaciamiento y la colocación de cintas intrónicos.
NOTA: Cada uno de ellos ha sido discutido en detalle en otra parte 10. - Elija regiones genómicas cada uno de 5-6 kb que flanquean el sitio de modificación de destino.
NOTA: Por consiguiente, el tamaño del inserto subclonado es típicamente 10-12 kb, aunque el límite superior puede ser tan alto como 80 kb con un bajo número de copias del plásmido como subclonación p15A, etc. pBR322 y hasta 200 kb con un BACvector.
2. Multiplex recombinería Oligos
- Oligos de diseño para el esqueleto del plásmido (plásmido subclonación) y el marcador seleccionable (casete de inserción). Diseñar cada oligo tal que contiene 180 pb HA que flanquea el sitio diana genómico y 20 pb de la secuencia de cebado específico de la casete de inserción o el plásmido subclonación (Figura 2).
NOTA: brazos de homología no deben contener elementos repetitivos, la presencia de elementos repetitivos se traducirá en la orientación incorrecta y subclonación. Elementos repetitivos pueden ser detectados mediante el uso de herramientas basadas en la web, como el '' Repite Enmascarador ''. - Incluir un sitio único de enzima de restricción (RE) sobre uno de los oligos de vectores para linealizar el vector genes de la orientación para la orientación de células ES (Figura 2).
- Compruebe los parámetros oligo utilizando un programa analizador de oligo. Tenga cuidado de evitar estructuras secundarias en la región de cebados.
- Shorter HA (50 pb) del casete de inserción es tolerada y produce menor número de recombinantes, pero asegúrese de que el plásmido subcloning HA es siempre más larga (180 pb).
- Determinar la orientación del inserto de ADN genómico de lo particular clon BAC utilizando herramientas basadas en web como '' cloneDB ''. Establecer la dirección de replicación del oriS comprobando el mapa de la columna vertebral plásmido BAC utilizados en la construcción de la biblioteca BAC.
NOTA: oris es generalmente opuesta a la dirección de la transcripción del marcador de cloranfenicol (CHL) para todos los plásmidos BAC utilizados comúnmente. - Añadir dos enlaces terminales de fosforotioato (PTO) al extremo 5 'del oligo que es opuesta a la dirección de la replicación en el clon BAC. Añadir una modificación 5 'fosfato a la oligo inverso (Figura 2).
NOTA: La asimétrica phosphorothioated casete PCR tras la digestión Red genera un ssDNA intermedio que puede cebar la cadena retrasadadel tenedor de replicación.
NOTA: Maximal multiplex frecuencia de recombinación se observa con los cassettes protegidas filamento de revestimiento. Principal capítulo protegida o casetes protegidas duales no puede producir eficiencias de recombinación equivalentes en una loci. - Ordene oligos con la página o la purificación HPLC.
3. Transformación BAC clon con pSC101 BADgbaA recombinería plásmido.
- Para hacer que la E. coli cepa que lleva el recombinería BAC competentes, transformada con el plásmido pSC101 BADgbaA que contiene los genes rojos 16.
NOTA: El plásmido pSC101 BADgbaA contiene los genes rojos y RecA bajo el control de la inducible araC-P BAD promotor de arabinosa, un marcador de selección de tetraciclina y el replicón pSC101 sensible a la temperatura.- Apuñalar a una punta de pipeta estéril en el cultivo de agar BAC e inocular 5,0 ml de caldo lisogenia (LB) pH 8,0 que contenía 12,556; g ml -1 Chl. Crece a 37 ° C durante 5 horas con agitación a 200 rpm.
- Chill 10% (v / v) de solución de glicerol, tubos de microcentrífuga y cubetas de electroporación sobre hielo. Enfriar una gran centrifugadora y microcentrífuga refrigerada a 4 ° C.
- Determinar la densidad óptica (DO) del cultivo usando un espectrofotómetro y medir la absorbancia a 600 nm. Preparar células electrocompetentes (como se describe a continuación) cuando se alcanza una lectura OD 600 de 0,3 a 0,8.
- Centrifugar las células en un tubo de centrífuga de 50 ml en un gran centrifugar a 1216 xg durante 5 min a 4 ° C.
- Lavar las células con 1 ml de frío 10% de glicerol y centrifugar las células a 17.949 xg durante 20 segundos a 4 ° C. Realice el lavado de paso un total de 3 veces.
- Resuspender las células en un volumen total de 50 l de 10% de glicerol y añadir 10-200 ng del plásmido pSC101 recombineering BADgbaA. Obtener una única suspensión celular mediante pipeteoarriba y abajo varias veces y luego transferir las células a un pre-refrigerada 1 mm cubeta de electroporación de brecha.
- Electroporar las células con un ajuste de 1,8 kv, 25 mF y 200 Ω.
NOTA: Compruebe los ajustes correctos para cada marca de electroporador. Una constante de tiempo de la electroporación menos de 4 indica la presencia de sal y otras impurezas. - Inmediatamente recuperar las células en 1 ml de LB y transferir las células a un tubo de centrífuga de 50 ml.
- Mantener el cultivo GBAA BAC a 30 ° C durante 2 horas con agitación a 200 rpm.
NOTA: El plásmido pSC101 BADgbaA se pierde cuando las células se cultivaron a 37 ° C debido a la inactivación del factor de replicación sensible a la temperatura repe. Se cultivan las células GBAA a 30 ° C para mantener las funciones recombinería. - Añadir 9 ml de LB que contienen 12,5 mg ml -1 Chl y 4 mg ml -1 tetraciclina (Tet) a la cultura GBAA BAC recuperado. Crecer elBAC GBAA cultivo O / N a 30 ° C con agitación a 200 rpm.
NOTA: La eficiencia de transformación de la electroporación el plásmido superenrollado GBAA es suficientemente alta para permitir el crecimiento de saturación de O / N en medios líquidos.
4. Preparación de Inserción Casetes y Subclonación plásmidos
- Para incorporar HA en el casete (s) de inserción y el plásmido subclonación, lleve a cabo la reacción en cadena de la polimerasa (PCR) utilizando los oligos largo modificados como se describe a continuación.
- Opcional: para evitar plásmido arrastre en la reacción recombinería, utilice un origen R6K o plantilla plásmido estrecho rango de huéspedes similar a amplificar el casete de inserción.
- Alternativamente, linealizar el plásmido plantilla usando un compendio RE (Tabla 2). Elegir un RE que corta fuera de la región amplificación por PCR y es inactivado por calor. El calor inactiva la RE como recomienda la manufacturer.
- Configurar la reacción en cadena de la polimerasa (PCR) utilizando un sistema de ADN hotstart polimerasa de alta fidelidad. Preparar una mezcla maestra de PCR como se detalla (Tabla 3). Realizar ciclos térmicos como se muestra (Tabla 3).
- Analizar los productos de PCR mediante electroforesis en gel de agarosa. Cargar 1-5 l de cada PCR en un 1% (w / v) en gel de agarosa que contiene 0,5 mg ml -1 bromuro de etidio (EtBr).
NOTA: La presencia de productos de amplificación no específicos no interfiere en la reacción recombinería. En algunos casos, sin embargo, dímeros de cebadores pueden disminuir la eficiencia recombinería. - Purificar los productos de PCR utilizando un kit de purificación de PCR.
- Opcional: eliminar la plantilla plásmido de PCR mediante tratamiento con DpnI seguido por PCR de limpieza. Eluir el ADN en un volumen mínimo de agua desionizada estéril (según lo recomendado por el fabricante).
NOTA: La adición de DpnI para no purificado de PCR reacciones resultados en reducción de la eficacia de la escisión del XXe plantilla de ADN plásmido metilado.
- Opcional: eliminar la plantilla plásmido de PCR mediante tratamiento con DpnI seguido por PCR de limpieza. Eluir el ADN en un volumen mínimo de agua desionizada estéril (según lo recomendado por el fabricante).
- Cuantificar PCR de ADN amplificado mediante análisis en gel de agarosa en contra de un conjunto conocido de patrones de ADN, por ejemplo, λ digestión con HindIII o mediante el uso de un espectrofotómetro NanoDrop.
5. Subcloning Plus Inserción
- Diluir la O / N BAC GBAA cultivo de 50 veces mediante la adición de 200 l en 10 ml de LB + Chl + Tet. Crecer en 30 ° C con agitación a 200 rpm durante 1 hr 50 min. Incluir una muestra a ser utilizada como control negativo.
- Enfríe todos los materiales y equipos recombinería a 4 ° C como se describe en el paso 3.1.2.
- Verter LB agar pH 8 placas que contienen la concentración correcta del antibiótico selectivo apropiado (s) que permita la selección del fragmento de ADN que se inserta, así como para la selección del vector de subclonación (Tabla 4).
NOTA: Algunos antibióticos son sensibles al pH. Uso LBagar pH 8 como una regla. - Preparar una solución al 10% de L-arabinosa (v w /). Esterilizar por filtración a través de un filtro de jeringa de 0,2 micras.
NOTA: arabinosa induce la expresión de las proteínas recombinería a partir del plásmido GBAA. - Compruebe el diámetro exterior de la cultura GBAA BAC utilizando un espectrofotómetro. Una vez que un OD 600 de 0,25-0,3 se alcanza, inducir a las proteínas recombinería como se describe en el siguiente paso.
- Añadir 200 l de la solución de arabinosa 10% a 10 ml del cultivo de BAC para lograr una concentración final de 0,2% de arabinosa. Incluir un cultivo no inducido (sin arabinosa) para ser utilizado como control negativo.
- Transferir la cultura BAC a un 37 ° C incubadora de agitación e inducir la expresión Roja durante 45 minutos con agitación a 230 rpm.
NOTA: La expresión de proteínas rojas es ineficiente a 30 ° C. - Centrifugar las células y lavar con 10% de glicerol 3 veces tal como se describe en el paso 3.1.5.
- Añadir 600-1,000 ng de cada uno del plásmido subclonación y el casete (s) de inserción a la reacción recombineering. Incluir un único vector y vector más Insertar controles individuales para comprobar el dominio de la recombinación y la integridad del vector y los cassettes.
- Obtener una única suspensión celular pipeteando arriba y abajo. Realizar electroporación como se describe en el paso 3.1.7 y posterior recuperación en 1 ml de LB a 37 ° C durante 1 hora para los plásmidos multicopia o en 10 ml de LB a 37 ° C durante 3 horas para los vectores del BAC.
- Placa de diferentes diluciones de la cultura por ejemplo, recuperado, 90%, 10%, 1% en las placas de agar de doble selección y crecer a 37 ° C durante 16 horas.
6. Análisis de recombinantes
- Opcional: Pick 6 a 12 colonias y realizar colonia PCR utilizando un cebador flanqueante HA y un cebador específico de inserción. Incluya el plásmido subclonación y el cas de inserciónplásmido Sette, así como el clon BAC padre como controles negativos.
- Realizar análisis en gel de agarosa de los PCR de colonias. Identificar los clones positivos por la presencia de una banda brillante en el tamaño esperado.
NOTA: La alta eficiencia del proceso de clonación recombinantes SPI genera principalmente correctos.
- Realizar análisis en gel de agarosa de los PCR de colonias. Identificar los clones positivos por la presencia de una banda brillante en el tamaño esperado.
- Escoja colonias en 5 ml de LB pH 8 que contiene el antibiótico selectivo y crecer O / N a 37 ° C.
- Preparar minipreparaciones de ADN utilizando un kit de purificación de la columna según las instrucciones del fabricante.
- Preparar ADN miniprep BAC utilizando el aislamiento de fenol-cloroformo estándar o un protocolo similar.
- Realizar digiere RE en el ADN miniprep. ADN separadas por electroforesis en gel de agarosa. Analizar los patrones de RE para identificar los clones que contienen los fragmentos de tamaños esperados del vector de orientación correcta. Elija un RE que discrimina claramente entre el inserto (s) vector que carece y el vector que contiene el inserto (s).
- Optional: para obtener células que carecen de la BAC, que todavía está presente en la E. coli después recombinería, utilice el ADN plásmido miniprep transformar DH5alfa o DH10B E. células de E. coli.
- Realizar la secuenciación de ADN a través de la HA y el casete de inserción para comprobar errores de síntesis de oligo.
Resultados
Vectores Knockin
Vectores de abordaje Knockin reflejan la necesidad de introducir una característica novedosa secuencia en el genoma incluyendo única sustitución de pares de bases en una región de codificación de la proteína, la fusión de un marcador fluorescente o una etiqueta de afinidad a una proteína o la integración de un casete de expresión génica. Para probar la aplicación de SPI en las estrategias de construcción knockin vector, se examinaron dos casos de prueba diferentes. Dnttip1 codifica la deoxynucleotidyltransferase, terminal, interactuando 1A proteína (TDIF1) que, junto con la clase I histona deacetilasa (HDAC) formar un complejo histona mitótico (Midac) 28. Para investigar el papel de Dnttip1 en la división celular, un enfoque de marcado tándem afinidad fue tomada para aislar el TDIF1 proteínas que interactúan. Los intentos anteriores para subclonar una porción del gen Dnttip1 utilizando un vector p15A que contiene regiones de homología largas resultaron en una baja brecha de eficiencia de reparación de unand productos de recombinación aberrantes frecuentes (datos no mostrados). Por lo tanto, la construcción de un vector knockin en el locus Dnttip1 proporcionado un ejercicio recombineering desafiante. Una estrategia SPI fue diseñado para subclonar una sección de 12 kb del gen Dnttip1 que abarca el último exón (exón 13) en un vector de copia bajo p15A y para insertar al mismo tiempo una doble etiqueta de afinidad selección casete en el exón 13, sustituyendo el codón de parada (Figura 3A ). La proteína de unión de FLAG-calmodulina 2X (CBP) vinculado FRT-PGK-Em7-neomicina (Neo) -BGhpA-FRT de casete (2,0 kb) se amplificó a partir de un RE linealizado plásmido utilizando oligonucleótidos modificados dual PTO que contenía 120 pb HA flanqueando la Dnttip1 el codón de parada. Un Dnttip1 vector subcloning Zeo p15A (1,7 kb) fue construido que contenían 200 bp regiones homólogas a los extremos de la secuencia Dnttip1 a subclonarse. El plásmido subcloning Dnttip1 fue re linealizado y amplificado por PCR utilizando 20 pb modificado oligosque genera un vector protegida principal capítulo. Los productos de PCR fueron tratados DpnI, purificado y co-electroporación en recombinería competente Dnttip1 BAC E. células de E. coli, así como las células de control no inducidas. Las reacciones SPI se sembraron en zeocina (Zeo) y kanamicina (Kan) que contiene placas de agar (Figura 3B). SPI produjo el vector knockin Dnttip1 modificado correctamente en todos los 12 recombinantes analizados (Figura 3C). La secuenciación de ADN verificó la ausencia de cualquier error resultantes de la síntesis de oligo o amplificación por PCR.
Otro ejemplo de SPI implicó la inserción en el marco de una proteína fluorescente de color amarillo mejorado (eYFP) vinculado casete de selección en el gen P2rx1. El gen P2rx1 codifica un receptor G-acoplado a la proteína que funciona como un canal de iones 29 gated-ATP. Relación con YFP fluorescencia permite el seguimiento del receptor P2X1 en la superficie celular en VAensayos funcionales rias. P2rx1 es otro locus difícil que ha presentado problemas significativos con las metodologías recombinería convencionales (datos no mostrados). El uso de SPI, un segmento de 12 kb del gen P2rx1 que abarca la terminal de exón (exón 12) se subclonó en un vector zeo p15A y modificado con la inserción concertada de un casete Neo eYFP -LoxP flanqueado reemplazando el codón de parada en el exón 12 para construir el P2rx1-eYFP vector knockin (Figura 3D). En este ejemplo, la cadena protegida vector p15A retraso (1,7 kb) contenía 230 pb HA y el casete eYFP contenía 50 pb HA y también se dejó sin modificar. El casete de inserción eYFP (2,7 kb) fue montado por solapamiento de empalme PCR del gen eYFP, PCR amplificado a partir de pEYFP-C1, y el casete PGK-LoxP- Mim7-Neo-BGHpA-LoxP, PCR amplificado a partir de pL452 (NCI, Frederick) . La reacción SPI producido cientos de colonias (datos no mostrados). RE análisis de DNAminipreparados preparados a partir de 11 clones mostraron la mayoría contenía el P2rx1-eYFP vector knockin montado correctamente (Figura 3E). Validación adicional mediante secuenciación del ADN mostró error libre inserción del casete eYFP. Sin embargo, algunos de los clones mostraron perfiles de la brecha sin etiquetar reparado y los plásmidos brecha reparado etiquetados en la misma célula (carriles 6-9). Unas pocas muestras también contenían incorrectamente brecha reparado (carril 2) o plásmidos mistargeted (carriles 4 y 5). El fracaso de la subclonación concurrente y la focalización en algunos recombinantes SPI pone de relieve los límites de la clonación eficiente SPI cuando se utilizan grandes casetes (> 3 kb), que surgen de las limitaciones de capacidad de procesamiento Red de fragmentos de ADN largos 30, o si se utiliza corto HA. De hecho, el aumento de la HA de la casete eYFP a 200 pb aumenta la eficiencia SPI y la orientación correcta de la casete (datos no mostrados). La orientación de genes con el P2rx1 - knockin vector eYFP en JM8.N4Las células madre embrionarias de ratón (C57BL / 6 cepa) generaron 6 clones positivos de 96, que contenían la secuencia P2rx1-eYFP correctamente orientada (Figura 3F).
BAC Reportero Vectores
Un clon BAC del gen diana a menudo contiene todos los elementos necesarios aguas arriba y aguas abajo de regulación por ejemplo, potenciadores, etc., así UTRs el promotor endógeno para conducir la expresión génica en los niveles naturales 31. Por tanto, un vector reportero de BAC es el vehículo preferido para recapitular el patrón de expresión endógena de un gen 32. Sin embargo, el gran tamaño de un plásmido BAC (hasta 200 kb) presenta importantes problemas prácticos con la transfección de un BAC intacto en las células 33. La reducción del tamaño del inserto genómico BAC BAC a través de recorte de 34,35, mientras que conserva los elementos reguladores necesarios de un gen, permite un manejo más fácil de la BAC y los resultados en un transfecti más eficienteen. Tecnología de la ingeniería BAC actual involucra múltiples rondas de recombinería para lograr este objetivo 35. Para demostrar la utilidad de SPI en BAC recorte, se utilizó un vector pBeloBAC11 BAC para subclonar una secuencia genómica 30 kb que incluye el gen de longitud completa de la P2rx1 168 kb P2rx1 BAC junto con la inserción simultánea de un casete de eYFP en el gen P2rx1 (Figura 4A). El Zeo vector columna vertebral pBeloBAC11 (6,5 kb) que contiene 180 pb HA fue amplificado por PCR con oligonucleótidos modificados que generaron un vector protegida cadena retrasada. El mismo cartucho de eYFP Neo (3 kb), utilizado en la construcción del vector knockin P2rx1 anterior, se amplificó por PCR usando oligos protegidos filamento de revestimiento que contienen 180 pb HA y dirigido el exón P2rx1 12 sustituyendo el codón de parada. Después de la electroporación combinado de la casete eYFP y el Zeo vector pBeloBAC11 en P2rx1 BAC células expressing las proteínas recombinería GBAA, el cultivo se recuperó en 10 ml de LB pH 8 durante 3 horas a 37ºC para separar los plásmidos BAC. Puros recombinantes se seleccionaron en Zeo y Kan placas de agar y se observaron a una frecuencia de 2 x 10 -6. Análisis de genotipificación colonia PCR reveló BAC éxito recorte en los 3 de los 6 clones analizados (Figura 4B). De largo alcance amplificación por PCR a través del sitio de inserción eYFP confirmó la incorporación de cassette correcta en los tres clones positivos (Figura 4B). Los tres clones que carecen de la inserción eYFP también fueron incorrectamente brecha reparado en el extremo 5 ', aunque la causa de la mala focalización de la casete eYFP en estos clones puede ser separado para el cierre correcto de el extremo 5' de BAC. Los tres clones eYFP BAC positivo fueron analizados con digiere RE y mostraron el patrón esperado de la eYFP BAC recombinante recortado correctamente (Figura 4C </ Strong>). El análisis de secuencia reveló ausencia de errores en el casete eYFP en sólo 1 clon de los 3 positivos.
Knockout Condicional (CKO) Vectores
Ablación condicional de la expresión génica es una herramienta importante para investigar los procesos de desarrollo o para estudiar sistemas biológicos en un momento particular. Una estrategia de gen knockout condicional implica típicamente la colocación de sitios de recombinación LoxP que rodean un exón crítico (CE). La supresión de un CE sobre Cre expresión o activación produce un desplazamiento del marco y un codón de parada prematuro, lo que resulta en la degradación del ARNm debido a tonterías mediada la decadencia (NMD). La construcción de un vector de orientación de genes condicional es una tarea compleja y abarca varias etapas de subclonación, la orientación y la transformación 22. SPI ofrece una ruta conveniente para simplificar este proceso. Como caso de prueba, se construyó un alelo condicional del gen Zrsr2utilizando la metodología SPI (Figura 5A). El gen Zrsr2 codifica un factor de empalme y una sola copia se encuentra en el cromosoma X. El estado condicional de deleción del gen es particularmente importante en este caso para controlar la posibilidad de adaptación de las células a la falta de Zrsr2 durante la selección de células ES en una estrategia de deleción génica dirigida constitutiva. SPI se realizó para subclonar una porción 10 kb del gen ZrSr2 con la inserción simultánea de dos casetes de selección LoxP flanqueadas diferentes. El FRT-PGK-Em7-Neo-FRT-LoxP casete (2 kb) fue amplificado de PCR RE digerido pL451 utilizando quedando oligos hebra protegida que contenía 180 pb HA dirigida al ZrSr2 intrón 2. Un segundo casete que contiene Rox-PGK-em7- blasticidina (BSD) -Rox-LoxP (2 kb) se amplificó por PCR a partir de un plásmido R6K con oligos protegidos capítulo menos que contienen 180 pb HA idéntica a una región aguas abajo en el intrón 3. Los dos sitios LoxP flanqueadas exón 3, la CE cuyo deletion en los resultados condicionales de activación Cre en un marco de lectura e introduce un codón de parada prematuro. El plásmido subcloning Zrsr2 (1,6 kb) se amplificó por PCR a partir de un plásmido Zeo p15A RE linealizado usando retraso hebra protegida oligos que contienen 180 pb HA coincidir los extremos de la secuencia Zrsr2 10 kb. Reacciones SPI se realizaron como se ha descrito antes y se sembraron en placas Zeo + Neo + BSD. La focalización condicional vector ZrSr2 fue montado con éxito en la mayoría de los 12 recombinantes examinados (Figura 5B). Además el análisis de secuenciación de ADN mostró la inserción correcta de ambos los marcadores de selección en el vector ZrSr2 cko.

Figura 1. SPI clonación. Descripción general del proceso de clonación SPI combinando la inserción de cassette y subclonación en un solo paso. (A) El clon BAC es transformed con el plásmido pSC101 BADgbaA y se cultivaron a 30ºC. (B) Después de la inducción arabinosa para expresar las proteínas rojas, el casete de inserción modificado asimétrica y el plásmido de subclonación se introducen en el clon BAC y seleccionado tanto con las marcas de inserción y de subclonación para generar el vector final. Por favor, haga clic aquí para ver una versión más grande de esta cifra.
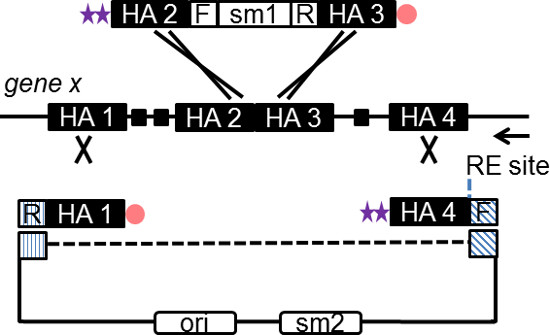
Figura 2. Esquema que ilustra el diseño de oligos recombinería SPI. El plásmido subclonación y el casete de inserción son ambos generados por PCR utilizando una combinación de PTO terminal y fosfato de oligos modificados. El fragmento de PCR tras la digestión Red in vivo, produce un intermedio ssDNA, que hibrida con el filamento de revestimiento del tenedor de replicación. Gene específico de HA de 50-180 pb se incorpora en cada oligo como se muestra. La flecha indica la dirección de replicación del ADN a través de un gen candidato. La línea representa la secuencia de plásmido subcloning no incorporado en el producto de PCR. RE sitio, sitio de enzimas de restricción de linealización del vector final; F, la secuencia hacia delante (20 pb) específica para el plásmido subclonación o casete de inserción; R, secuencia de complemento inverso (20 pb) del plásmido o casete; sm, marcador de selección. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. SPI permite la construcción de vectores knockin difíciles. (A) Esquema de la estrategia de SPI se utiliza en la construcción de la Dnttip1 doble etiquetado vector. Arrow indiCates la dirección de replicación en el clon BAC. resultados (B) Planchas de las muestras no inducidas e inducidas del experimento Dnttip1 SPI. (C) EcoRI compendio de clones Dnttip1 SPI. M, 1 escalera kb (NEB); C, brecha p15A Dnttip1 reparado plásmido que carece de la casete de etiquetas dual. Fragmento tamaños son: etiquetado, 9,2 + 4,4 + 2,2 kb; control; 9,2 + 4,6 kb. (D) SPI P2rx1-eYFP construcción vector knockin basado. (E) EcoRI Recopilación de clones P2rx1-eYFP SPI. M, 1 kb + escalera (Invitrogen); C, brecha p15A P2rx1 reparado plásmido que carece de la casete de eYFP. Fragmento tamaños son: etiquetado, 8,3 + 4,4 + 3,1 + 0,03 kb; control; 8,3 + 3,1 + 1,8 + 0,03 kb (F) el análisis de transferencia de Southern de P2rx1 -. Gen eYFP focalización en las células celulares JM8.N4 ES. El panel superior, transferencia de Southern con la sonda extremo 3 'usando PshAI resumen. Se muestra es el resultado de detección de 5 clones. Panel inferior, Southern blot usandola "sonda final y SpeI compendio de los aspectos positivos identificados a partir de los 3 '5 cribado final. Sitio PshAI RE; S, Spel RE sitio. La línea de puntos representa el final del vector de HA. Cuadro negro denota sonda sur. Fragmentos de restricción esperados son, PshAI: WT, 8,9 kb; eYFP-neo, 11,5 kb; Spel:. WT, 6,9 kb; eYFP-neo, 7,6 kb Haga clic aquí para ver una versión más grande de esta figura.
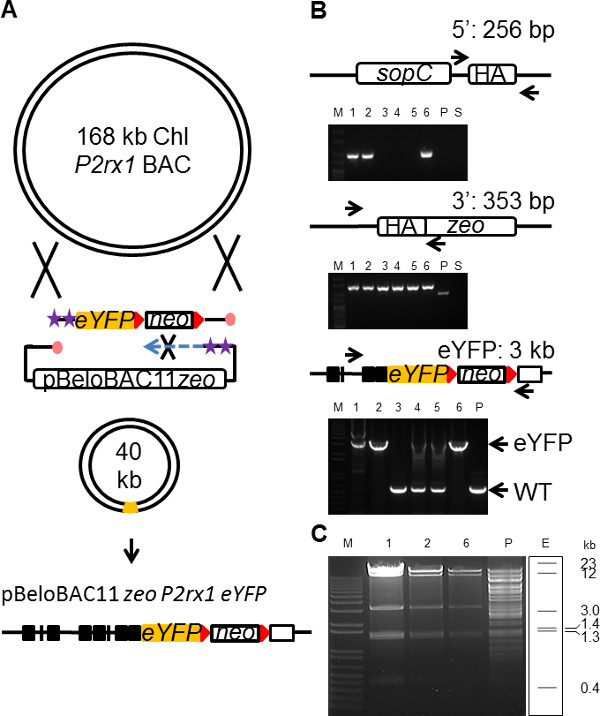
La Figura 4. Simplificado BAC de recorte con SPI. (A) Representación esquemática que ilustra el concepto de BAC de recorte con SPI. Explicación de los símbolos se describe en la Figura 1. Amplificación (B) a través de la PCR 5 'y 3' del inserto subclonado y en todo el insertio P2rx1-eYFPsitio n. La estrategia de cribado se muestra para cada tipo de PCR. M (panel superior), Hyperladder 25 pb, (paneles inferiores), 1kb +; P, P2rx1 BAC; . S, plásmido pBeloBAC11 Zeo subcloning (C) HindIII digerir de los tres eYFP positivos clones BAC de SPI (B); E, que se espera patrón de restricción HindIII del recortado eYFP BAC. Haga clic aquí para ver una versión más grande de esta figura.
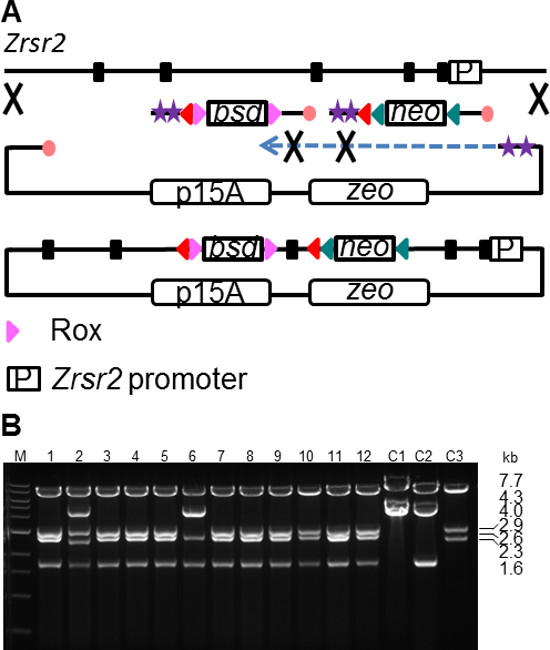
Figura 5. Condicional generación knockout vector usando SPI clonación. (A) Representación esquemática de la inserción simultánea de dos LoxP flanqueado casetes de selección diferentes durante la subclonación del alelo Zrsr2. Explicación de los símbolos se describe en la Figura 1. (B) </ Digiere strong> EcoRI de los clones Zrsr2 SPI. L, 1 escalera kb (NEB); C1, p15A ZrSr2 brecha reparado plásmido, C2, brecha p15A ZrSr2 reparado plásmido que contenía el inserto neo; C3, brecha p15A ZrSr2 reparado plásmido que contienen la inserción bsd. Fragmento tamaños son: cko, 7,7 + 2,9 + 2,6 + 1,6 kb; C1, 7,7 + 4,0 kb; C2, 7,7 + 4,3 + 1,6 kb; C3, 7,7 + 2,9 + 2,3 kb. Haga clic aquí para ver una versión más grande de esta figura.
| No de pasos | Recombinería convencional tubería una | Multiplex recombinería b |
| Paso 1 | Transformación del plásmido recombinería en anfitrión BAC | Transformación del plásmido recombinería en anfitrión BAC. Preparación de los vectores de abordaje casetes y subclonación. |
| Paso2 | La inserción de cassette de R1 / R2 de puerta de enlace | Brecha Multiplex clonación reparación |
| Paso 3 | La inserción del casete Kan floxed | O / N cultura a partir de colonias individuales |
| Paso 4 | Reparación Gap en el plásmido R3 / R4 | Preparación de plásmido y verificación |
| Paso 5 | Transformación en Cre + E. coli | |
| Paso 6 | Preparación de plásmido y verificación | |
| Paso 7 | O / N de tres vías reacción de Gateway | |
| Paso 8 | Transformación de la reacción de tres vías de puerta de enlace en DH10B E. coli células | |
| Paso 9 | Cultivo de la noche a partir de colonias individuales | |
| Paso 10 | Preparación de plásmido y verificación de la secuencia | |
| b Tiempo promedio de 4 días para clon verificado | ||
Tabla 1: Comparación de recombineering convencional con SPI en la construcción de vectores knock-out condicionales.
| Tampón RE | 5 l |
| ADN | 1 mg de plásmido o de los productos de PCR purificados |
| RE | 1 l (5 unidades o más) |
| TE | hasta 50 l |
| Incubar a 37 ° C durante al menos 1 hr. Calentar inacitvate de acuerdo con las instrucciones del fabricante | |
Tabla 2: RE digestión.
| Materiales de PCR | La concentración final |
| Tampón de PCR | 1x |
| dNTP | 200 nM |
| MgSO 4 | 1,5 mM |
| La betaína | 1,3 M |
| DMSO | 1% |
| Cebador directo | 200 nM |
| Cebador inverso | 200 nM |
| ADN polimerasa | 1 U |
| Plantilla | 10 ng de plásmidos de múltiples copias o 2,5 l de ADN miniprep para PCRs de genotipado |
| Agua | hasta 50 l una |
| a Para una reacción de PCR de 50 l de serie con plásmidos de múltiples copias. PCR genotipificación de largo alcance se establecieron en 25 reacciones de PCR mL. | |
| Las condiciones de PCR | |
| 95 ° C | 2 min |
| 92 ° C | 10 sec |
| 55 ° C | 30 sec |
| 72 ° C | 30 sec |
| 30 ciclos b | |
| b Ciclo no se puede extender a 35 para el genotipado BAC PCR | |
Tabla 3: PCR Set-up y Condiciones.
| Antibióticos | Concentración un (ml -1 g) |
| Ampicllin | 50 |
| Blasticidina b | 40 |
| El cloranfenicol | 12.5 |
| Gentamicina | 2 |
| Higromicina c | 30 |
| Kanamicina b | 15 |
| Tetraciclina | 4 |
| Trimetoprim c | 10 |
| Zeocina | 5 |
| Recomendado para un uso con BAC y plásmidos multicopia cuando se usa en combinaciones en recombinería multiplex | |
| b blasticidina (35 mg ml -1) y kanamicina (6 mg ml -1) cuando se usan juntos en combinación | |
| c higromicina y Trimetoprima no se recomiendan para la selección con BAC de copia única. | |
Tabla 4. Recomendado concentraciones de antibióticos para su uso en experimentos SPI.
Discusión
Construcción de líneas de células madre embrionarias y modelos de ratón históricamente ha incluido la orientación de genes utilizando construcciones de plásmidos que contenían el alelo modificado 4. Sin embargo, la construcción de estos vectores complejos orientación de genes ha demostrado ser un cuello de botella significativo en la producción oportuna de tales modelos. El desarrollo de estrategias de construcción vector recombinería basada ha permitido mejorar los diseños vectoriales y montaje vector más eficiente. Sin embargo, los protocolos recombinería actuales todavía implican varios pasos, requieren de purificación de plásmido intermedio y utilizan diferentes cepas bacterianas. Subclonación de inserción plus ofrece un nuevo enfoque para la construcción de vectores que se pueden realizar en un solo evento electroporación en la cepa huésped BAC residente. La utilidad de SPI en la dirección de genes fue probado aquí en una variedad de aplicaciones de construcción del vector. En todos los casos examinados aquí, SPI demostró ser eficiente y se produjo el plásmido recombinante correcta.En la mayoría de los casos, las múltiples cintas de diferentes fueron insertados correctamente en el vector de direccionamiento. Grandes casetes de ADN y vectores fueron alojados fácilmente en el protocolo SPI y demostraron la flexibilidad de este sistema.
SPI se basa en el uso de secuencias de homología largas y fosforotioato (PTO) la protección de los casetes de ADN lineales. Modificación PTO confiere protección contra exonucleasas al ADN lineal 20,21 y la larga HA aumenta la eficiencia de recombinación para permitir la multiplexación. Sin embargo, la síntesis de secuencias de oligo largos aumenta las posibilidades de acumulación de errores especialmente supresiones. Las mutaciones en el oligo pueden ser particularmente perjudicial si cubren regiones codificantes de proteínas. La secuenciación del ADN a través de la HA y cubre la longitud completa de la cinta insertada es muy recomendable para eliminar clones con las alteraciones de la secuencia. También se sugiere el uso de un sistema de ADN polimerasa de alta fidelidad para evitar la introducción de unaerrores y PCR. La longitud y la composición de la HA del plásmido subclonación es más crítica respecto a la de la casete de inserción (datos no mostrados). Para aplicaciones especialmente sensibles como la construcción de vectores knockin, donde no se toleran las mutaciones en las regiones de exón, la HA del casete de inserción se puede acortar (50-120 pb) para evitar problemas asociados con oligos largos. El casete de inserción también se puede dejar phosphorothioated no modificada o dual (donde el conocimiento de la dirección de la replicación no está disponible). Pero multiplexación en estos casos todavía requiere mucho protegidos HA plásmidos subclonación. Una advertencia de esta estrategia particular es la disminución de la eficiencia de multiplexación que potencialmente pueden afectar SPI clonación en diferentes loci.
La longitud de la casete de inserción y el plásmido subclonación es otro parámetro importante en el SPI clonación. Moléculas de ADN más grandes electroporar menos eficiencia y el efecto es acumulativo, dada la rerequisito de introducir todos los casetes en la misma célula (datos no mostrados). La multiplexación es más eficiente con casetes de inserción más pequeños. Fragmentos de ADN mayores de 3 kb también poner un límite en el procesamiento de ssDNA Roja mediada, que es más eficiente hasta 3 kb 30. Casetes Inserción de más de 3 kb son duales resecado y recombinan menos eficiente a través de una vía independiente beta 20. Por lo tanto, la detección de colonias suficientes para identificar el clon correcto se vuelve importante en estos casos. Una mayor duración de la post electroporación recombinación también aumenta las posibilidades de recuperación del clon correcto en ejercicios difíciles SPI. Hasta cuatro cassettes pequeños (<1,5 kb) pueden ser insertados simultáneamente con el proceso de SPI, aunque la eficiencia de multiplexación disminuye con cada casete adicional (datos no mostrados). Sin embargo, esto refleja el resultado de un experimento óptima SPI y es recomendable tener en cuenta los límites de multiplexación cuando se utiliza muchas grandes cassettes.
El desarrollo de herramientas de edición del genoma como agrupados interspaced regularmente repeticiones palindrómicas cortas (CRISPR) Sistema endonucleasa -cas9 ha permitido la creación de nuevas modificaciones del genoma y ha llevado a la ingeniería del genoma más eficiente 36. Sin embargo, estas nuevas tecnologías han complementado vectores de genes objetivo en lugar de ellos suplantado. Se prevé que el sistema de CRISPR-cas podría sustituir a la selección de antibióticos y perfeccionar el protocolo recombineering multiplex.
Divulgaciones
T.R.R is the inventor of a British patent application on multiplex recombineering technology.
Agradecimientos
T.R.R conceived the project, designed and performed experiments and prepared the manuscript. E.J.K and S.E.M performed experiments. All authors contributed intellectually to the final manuscript. This work was funded by a University of Leicester Innovation Fellowship to SEM and by a UK Medical Research Council (MRC) Senior Research Fellowship to S.M.C (MR/J009202/1). DNA sequencing was performed by Protein and Nucleic Acid Laboratories (PNACL), Centre for Core Biotechnology Services, University of Leicester. We wish to thank A. Francis Stewart for providing the Rox and Dre plasmids. pL451 and pL452 plasmids were obtained from National Cancer Institute (NCI), Frederick, USA.
Materiales
| Name | Company | Catalog Number | Comments |
| Antibiotics except Zeocin | Sigma: http://www.sigmaaldrich.com | Ampicillin, A9518-5G; Chloramphenicol , C1919-5G; Blasticidin, 15205-25MG; Gentamicin, G1264-50MG; Hygromycin, H3274-50MG; Kanamycin,: K1876-1G; Tetracycline hydrochloride, T7660-5G; Trimethoprim, 92131-1G | |
| Zeocin | Invivogen: | ant-zn-1 | Zeocin is also available from Life technologies/Fisher |
| C57BL/6 BAC clones | CHORI: https://bacpac.chori.org/ | RPCI-23 or RPCI-24 BAC libraries | 129/Sv BAC clones can be obtained from Invivogen or Life technologies/Fisher |
| KOD hotstart DNA polymerase system | Millipore: https://www.merckmillipore.com | 71086-3 | |
| L-Arabinose | Sigma: http://www.sigmaaldrich.com | A3256-25G | |
| Long oligos | IDT: https://www.idtdna.com; Gene link: https://www.genelink.com | IDT Ultramers or Gene Link long oligos | PTO and Phosphosphate available as custom modifications. PAGE purification strongly recommended for long oligos. |
| MinElute PCR purification kit | Qiagen: http://www.qiagen.com | 28004 | Minimum elution PCR purifications kits are preferred. |
| QIAPrep Spin Mini Prep kit | Qiagen: http://www.qiagen.com | 27104 | Silica column or similar matrix kits are preferred. |
| Restriction enzymes | NEB: https://www.neb.com | DpnI: R0176L | See NEB website for a list of RE. |
Referencias
- Dovey, O. M., Foster, C. T., Cowley, S. M. Histone deacetylase 1 (HDAC1), but not HDAC2, controls embryonic stem cell differentiation. Proc. Natl. Acad. Sci. USA. 107, 8242-8247 (2010).
- Harvey, M., et al. Spontaneous and carcinogen-induced tumorigenesis in p53-deficient mice. Nat. Genet. 5, 225-229 (1993).
- Waterhouse, P., et al. Lymphoproliferative disorders with early lethality in mice deficient in Ctla-4. Science. 270, 985-988 (1995).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51, 503-512 (1987).
- Hasty, P., Rivera-Pérez, J., Bradley, A. The length of homology required for gene targeting in embryonic stem cells. Mol. Cell. Biol. 11, 5586-5591 (1991).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. Nature. 317, 230-234 (1985).
- Hirata, R., Chamberlain, J., Dong, R., Russell, D. W. Targeted transgene insertion into human chromosomes by adeno-associated virus vectors. Nat. Biotechnol. 20, 735-738 (2002).
- Szostak, J. W., Orr-Weaver, T. L., Rothstein, R. J., Stahl, F. W. The double-strand-break repair model for recombination. Cell. 33, 25-35 (1983).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309, 255-256 (1984).
- Fu, J., Teucher, M., Anastassiadis, K., Skarnes, W., Stewart, A. F. A recombineering pipeline to make conditional targeting constructs. Methods Enzymol. Soriano, P. M., Wassarman, P. M. 477, Academic Press. 125-144 (2010).
- Zhang, Y., Buchholz, F., Muyrers, J., Stewart, A. A new logic for DNA engineering using recombination in Escherichia coli. Nat. Genet. 20, 123-128 (1998).
- Copeland, N., Jenkins, N., Court, D. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Muyrers, J., Zhang, Y., Testa, G., Stewart, A. Rapid modification of bacterial artificial chromosomes by ET-recombination. Nucleic Acids Res. 27, 1555-1557 (1999).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. USA. 97, 5978-5983 (2000).
- Sharan, S. K., Thomason, L. C., Kuznetsov, S. G., Court, D. L. Recombineering: a homologous recombination-based method of genetic engineering. Nature Protoc. 4, 206-223 (2009).
- Wang, J., et al. An improved recombineering approach by adding RecA to λ red recombination. Mol. Biotechnol. 32, 43-53 (2006).
- Datta, S., Costantino, N., Court, D. L. A set of recombineering plasmids for gram-negative bacteria. Gene. 379, 109-115 (2006).
- Fu, J., et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting. Nat. Biotechnol. 30, 440-446 (2012).
- Muyrers, J., Zhang, Y., Buchholz, F., Stewart, A. RecE/RecT and Redalpha/Redbeta initiate double-stranded break repair by specifically interacting with their respective partners. Genes Develop. 14, 1971-1982 (2000).
- Maresca, M., et al. Single-stranded heteroduplex intermediates in lambda Red homologous recombination. BMC Mol. Biol. 11, 54(2010).
- Mosberg, J. A., Lajoie, M. J., Church, G. M. Lambda Red recombineering in Escherichia coli occurs through a fully single-stranded intermediate. Genetics. 186, 791-799 (2010).
- Liu, P., Jenkins, N. A., Copeland, N. G. A highly efficient recombineering-based method for generating conditional knockout mutations. Genome Res. 13, 476-484 (2003).
- Chan, W., et al. A recombineering based approach for high-throughput conditional knockout targeting vector construction. Nucleic Acids Res. 35, e64(2007).
- Zhang, Y., Muyrers, J. P. P., Testa, G., Stewart, A. F. DNA cloning by homologous recombination in Escherichia coli. Nat. Biotechnol. 18, 1314-1317 (2000).
- Lee, E. C., et al. A highly efficient Escherichia coli-based chromosome engineering system adapted for recombinogenic targeting and subcloning of BAC DNA. Genomics. 73, 56-65 (2001).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Valenzuela, D. M., et al. High-throughput engineering of the mouse genome coupled with high-resolution expression analysis. Nat. Biotechnol. 21, 652-659 (2003).
- Bantscheff, M., et al. Chemoproteomics profiling of HDAC inhibitors reveals selective targeting of HDAC complexes. Nat. Biotechnol. 29, 255-265 (2011).
- Mulryan, K., et al. Reduced vas deferens contraction and male infertility in mice lacking P2X1 receptors. Nature. 403, 86-89 (2000).
- Subramanian, K., Rutvisuttinunt, W., Scott, W., Myers, R. The enzymatic basis of processivity in lambda exonuclease. Nucleic Acids Res. 31, 1585-1596 (2003).
- Antoch, M. P., et al. Functional identification of the mouse circadian Clock gene by transgenic BAC rescue. Cell. 89, 655-667 (1997).
- Rostovskaya, M., et al. Transposon-mediated BAC transgenesis in human ES cells. Nucleic Acids Res. 40, e150(2012).
- Giraldo, P., Montoliu, L. Size matters: use of YACs, BACs and PACs in transgenic animals. Transgenic Res. 10, 83-103 (2001).
- Hill, F., et al. BAC trimming: minimizing clone overlaps. Genomics. 64, 111-113 (2000).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36(2005).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339, 819-823 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados