Method Article
서브 클로닝 플러스 삽입 (SPI) - 유전자 타겟팅 벡터의 신속한 건설 소설 Recombineering 방법
요약
Gene targeting methodologies can be used to generate transgenic mice with knockout, knock-in and tagged alleles. Here, we describe an improved method of recombineering in E. coli, that we term ‘subcloning plus insertion’, which can be used to generate custom gene targeting vectors rapidly.
초록
Gene targeting refers to the precise modification of a genetic locus using homologous recombination. The generation of novel cell lines and transgenic mouse models using this method necessitates the construction of a ‘targeting’ vector, which contains homologous DNA sequences to the target gene, and has for many years been a limiting step in the process. Vector construction can be performed in vivo in Escherichia coli cells using homologous recombination mediated by phage recombinases using a technique termed recombineering. Recombineering is the preferred technique to subclone the long homology sequences (>4kb) and various targeting elements including selection markers that are required to mediate efficient allelic exchange between a targeting vector and its cognate genomic locus. Typical recombineering protocols follow an iterative scheme of step-wise integration of the targeting elements and require intermediate purification and transformation steps. Here, we present a novel recombineering methodology of vector assembly using a multiplex approach. Plasmid gap repair is performed by the simultaneous capture of genomic sequence from mouse Bacterial Artificial Chromosome libraries and the insertion of dual bacterial and mammalian selection markers. This subcloning plus insertion method is highly efficient and yields a majority of correct recombinants. We present data for the construction of different types of conditional gene knockout, or knock-in, vectors and BAC reporter vectors that have been constructed using this method. SPI vector construction greatly extends the repertoire of the recombineering toolbox and provides a simple, rapid and cost-effective method of constructing these highly complex vectors.
서문
유전자 타겟팅 기술들의 개발은 다른 생물학적 시스템 1-3 조사 세포주 및 마우스 모델의 구성을 가능하게했다. 게놈 서열의 변형 키를 제 4 단계는 벡터를 표적 유전자의 설계 및 시공된다. 타겟팅 벡터는 선별 마커 (예를 들면, 네오 마이신)에 의해 측면 원하는 변형 (들)을 포함하는 관심있는 대립 유전자를 운반하는 플라스미드 구조체, 및 포유 동물 세포에서 5 효율적인 상동 재조합에 필요한 긴 게놈 영역이다. 정확한 유전자 변형은 배아 줄기로 타겟팅 벡터의 도입 (ES) 세포를 6 또는 체세포 7에 의해 달성된다 타겟팅 벡터의 DNA 서열과 동일 뻗어 간의 상동 재조합과 의도 변형 예의 전사의 유전자좌 결과 유전자 변환 (8)에 의해 게놈. 그런 후 변형ED ES 세포는 배아 9 통해 이러한 대립 유전자 변형을 전송할 수있는 자손 (키메라)를 생성하기 위하여 마우스 포배 내로 주입 될 수있다. 트랜스 제닉 마우스를 제조하는 다른 경로는 마우스 게놈에서의 열 벡터의 임의의 통합에 이르게 단세포 마우스 접합체로 유전자 발현 벡터의 마이크로 인젝션을 포함한다. 벡터 건설의 전통적인 방법은 기존에 의존 한 벡터 백본에 다른 선택 마커 및 유전자 조각을 복제하는 제한 효소와 DNA 리가 제를 사용하여 복제 '잘라 내기 및 붙여 넣기'. 그러나, 기존의 복제 제한 요소는 고유 포지셔닝 및 제한 부위의 선택은 특히 긴 DNA 서열로이다. 이것은 종종 다수의 서브 클로닝 단계를 필요로하고 또한 종종 표적 유전자의 낮은 효율에 이르게 벡터 내로 외래 DNA 서열을 도입한다.
Recombineering (재조합 성 ENGineering) E. 파아지 재조합 단백질에 의해 매개 된 상 동성 재조합 (HR)를 사용하여 이러한 한계를 극복 DNA 공학적이며 대장균 세포 (11, 12). 상동 서열의 어느 지역이 recombineering의 기판 역할을 할 수 있기 때문에, 제한 사이트의 가용성의 제약이 제거됩니다. 큰 DNA 서열은 따라서 원활 또한 구조적 일체 (13)을 보존, 생체 내에서 직접 변형 될 수있다. Recombineering 짧은 동성 (50 BP) (14), 따라서 동성 암 (HA)을 편리하게 합성 올리고 시퀀스에 통합 할 수 있습니다 매우 효율적입니다. 전형적인 recombineering 실험, 올리고 또는 HA를 포함하는 이중 가닥 DNA (dsDNA) 조각에서 recombineering 관할 E.에 electroporation됩니다 염색체 나 플라스미드 (15)에 하나있는 대상을 포함하는 대장균 세포. 재결합 가능성은 레드 녹화의 유도 식으로 수여ombination 파지 16, 17의 단백질 또는 RAC의 프로 파지 (18)의 RecET 단백질. 레드 / RECE의 엑소 뉴 클레아 제는 파트너, 레드 / RECT, 단일 가닥 어닐링 단백질 (SSAP) (19)에 의해 결합되어 중간 단일 가닥 DNA (ssDNA를), 선형의 dsDNA를 변환합니다. ssDNA를 길게하거나 상보 표적 서열에 짧은 올리고의 어닐링 복제 포크의 보온 가닥 발생 및 표적 부위에서 서열의 혼입에 이르게. 스트랜드 ssDNA를 재결합하는 래깅 recombineering의 높은 효율의 기초와 '베타'재결합 모델 (20, 21)에 의해 설명 될 수있다.
일반적인 recombineering 워크 플로우는 유전자 표적 벡터는 다음과 같은 두 가지 경로 중 하나를 포함 빌드합니다. 하나의 경로에 loxP 재조합 사이트 순차 삽입, 선택 마커이어서 플라스미드로 마우스 BAC 클론에서 원하는 게놈 영역을 서브 클로닝 포함 (22) 등, 또는 대체 경로가 10,23을 recombineering 후 24, 25 복제 갭 수리하여 플라스미드로 수정 된 궤적을 서브 클로닝의 여러 라운드로 다른 타겟팅 벡터 요소 BAC 유전자좌를 대상으로 포함한다. 이 테마에 큰 변형 마우스 제작 프로그램 (26, 27)의 일부로서 다른 높은 처리량 recombineering 파이프 라인에서 사용되어왔다. 그러나, 이들 절차가 복잡하고 긴 스테이지들을 포함 특수 벡터와 E.의 사용을 필요 대장균 균주 (예, Cre 호텔은 발현 세포) 및 벡터 DNA를 정제하고 다시 변환 (표 1)의 하나 이상의 중간 단계를 이용한다. 플러스 삽입 (SPI)를 서브 클로닝하는 베타 재조합 단일 프로세스로 복제 갭 수리 (그림 1)를 결합하는 새로운 recombineering 기술이다. SPI 벡터 조립, 단순 신속하고 유연하며 표준 recombi에 상당한 개선을 제공합니다어링 (표 1)에 접근한다. 여기서는 용이성 및 비표준와 도전 벡터 디자인 구성에 특정 중점을 다른 벡터 건설 애플리케이션에 SPI를 사용하는 유틸리티를 보여준다. 테스트 케이스는 형광 리포터 올림피아 벡터의 구조, 듀얼 태그 올림피아 단백질 발현 벡터, BAC 형광 리포터 벡터 및 조건부 녹아웃 벡터를 포함. 이들은 각종 핵 단백질 복합체를 세포 표면 수용체 지역화 정화 요구 조건을 조사하거나 유전자의 발현을 브레이션.
프로토콜
디자인을 대상으로 1 유전자
- 관심의 게놈 영역을 다루는 적절한 BAC 클론을 주문. 이 예를 수정해야 ES 세포의 종류에 동질 있는지 확인, 유전자 RPCI-23 및 RPCI-24 BAC 클론 C57BL / 6 ES 세포를 대상으로.
- 대상 벡터를 설계 할 때 기준을 대상으로 기존의 유전자를 적용합니다. 주요 매개 변수는 수정이 인트론 카세트의 유전자 녹아웃 전략 및 간격과 배치를 삭제 중요한 엑손 (CE)을 정의, 구성 적 또는 조건부로 할 필요가 있는지 여부를 포함한다.
주 : 이들 각각은 다른 곳에서 열 상세히 논의되었다. - 대상 변형 사이트를 측면에서는 5~6킬로바이트 각각의 게놈 영역을 선택합니다.
주 : 상한 같은 플라스미드 p15A 서브 클로닝 적은 카피와 80킬로바이트만큼 높을 수 있지만 서브 클로닝 인서트의 크기는, 따라서, 전형적 10-12킬로바이트이다 인 pBR322 등과 BAC와 최대 200킬로바이트벡터.
2. 멀티 플렉스 Recombineering 올리고
- 플라스미드 백본 (서브 클로닝 플라스미드) 및 선별 마커 (삽입 카세트)에 대한 설계 올리고. 그것은 180 염기쌍의 HA의 측면에 게놈 대상 사이트와 삽입 카세트 또는 서브 클로닝 플라스미드 (그림 2)의 특정 프라이밍 서열의 20 bp의를 포함하도록 각 올리고 디자인.
참고 : 동성 암은 어떤 반복적 인 요소를 포함 할 수 없습니다, 반복적 인 요소의 존재가 잘못된 타겟팅 및 서브 클로닝 발생합니다. 반복 요소는 예컨대 '' '가리개를 반복'와 같은 웹 기반 툴을 이용하여 검출 할 수있다. - ES 세포 (그림 2)을 대상으로 유전자 대상으로 선형화 된 벡터를 벡터 올리고 중 하나에 고유 제한 효소 (RE) 사이트를 포함합니다.
- 올리고 분석기 프로그램을 사용하여 올리고 매개 변수를 확인합니다. 프라이밍 지역에서 차 구조 않도록주의하십시오의.
- 삽입 카세트의 짧은 HA (50 BP)은 허용과 재조합의 낮은 숫자를 산출하지만 서브 클로닝 플라스미드 HA가 이상 (180 BP) 항상 있는지 확인한다.
- 'cloneDB'추천 웹 기반 툴을 사용하여 특정 BAC 클론의 게놈 DNA 인서트의 방향을 결정한다. BAC 라이브러리의 구축에 사용 된 BAC 플라스미드 골격의 맵을 확인하여 오리스에서 복제의 방향을 설정.
참고 : 오리스 모든 일반적으로 사용되는 BAC 플라스미드의 클로람페니콜 (엽록소) 마커의 전사 방향과 반대 보통이다. - BAC 클론에 복제의 방향과 반대이다 올리고의 5 '말단에 두 개의 터미널 포스 (PTO) 채권을 추가합니다. 역 올리고 (그림 2)에 5 '인산 수정을 추가합니다.
참고 : 레드 소화에 따라 비대칭 phosphorothioated PCR 카세트는 수 소수 보온재 가닥 ssDNA를 중간 생성복제 포크의.
주 : 극대 다중화 재조합 빈도는 보온 스트랜드 보호 카세트로 관찰된다. 선도 가닥 보호 또는 이중 보호 카세트는 어떤 궤적에 상응하는 재조합 효율성을 생산하지 않을 수 있습니다. - PAGE 또는 HPLC 정제에 올리고 주문.
pSC101 BADgbaA Recombineering 플라스미드 3. BAC 클론 변환.
- E.을 만들려면 실력 BAC의 recombineering 베어링 대장균은 레드 유전자 (16)를 포함하는 pSC101 BADgbaA 플라스미드로 변환.
참고 : pSC101 BADgbaA 플라스미드 아라비 노스 유도 ARAÇ-P BAD 프로모터, 테트라 사이클린 선택 마커 및 온도에 민감한 pSC101 플리의 제어 아래에있는 빨간색과 RECA 유전자가 포함되어 있습니다.- BAC 한천 문화 멸균 피펫 팁을 찔러 원성 국물의 5.0 ml에 접종 (LB) 12.5을 포함하는 pH가 8.056; g ml의 -1 엽록소. 5 시간 200 rpm으로 흔들어 37 ° C에서 성장.
- 10 % (v / v) 글리세롤 용액, 마이크로 원심 분리 튜브 및 얼음에 전기 천공 큐벳 진정. 4 ° C에 냉장 대형 원심 분리기 및 마이크로 원심 쿨.
- 분광 광도계를 사용하여 배양 물의 광학 밀도 (OD)를 결정하고 600 nm에서 흡광도를 측정한다. 0.3 내지 0.8의 OD 판독이 600에 도달 할 때 (후술) electrocompetent 세포를 준비한다.
- 4 ° C에서 5 분 동안 1,216 XG에서 큰 원심 분리기 50 ㎖ 원심 분리 튜브에 세포를 스핀 다운.
- 냉장 10 % 글리세롤의 1 ml의 세포를 씻으 4 ° C에서 20 초 동안 17,949 XG에서 세포를 스핀 다운. 세탁 3 회 총 단계 수행합니다.
- 10 % 글리세롤 50 μl의 총 부피에 재현 탁하고 세포를 pSC101 BADgbaA의 recombineering 플라스미드 10-200 ng의 추가. 피펫에 의해 단일 세포 현탁액을 수득최대 여러 번 다운 후 미리 냉장 1mm 간격 전기 큐벳에 세포를 전송합니다.
- 1.8 KV, 25 μF 200 Ω의 설정을 사용하여 세포를 Electroporate.
참고 : electroporator의 각 브랜드에 대한 올바른 설정을 확인합니다. 보다 작은 4 일렉트로 시정 염 및 기타 불순물의 존재를 나타낸다. - 즉시 LB 1 ㎖에 균체를 회수하고 50 ㎖ 원심 분리 튜브에 세포를 옮긴다.
- 2 시간 200 rpm으로 흔들어 30 ° C에서 BAC의 gbaA 문화를 성장.
참고 : 세포 인해 온도에 민감한 REPE 복제 인자의 불 활성화에 37 ° C에서 성장하는 경우 pSC101 BADgbaA 플라스미드가 손실됩니다. recombineering 기능을 유지하기 위해 30 ° C에서 gbaA 세포 성장. - 12.5 μg의 ml의 -1 엽록소 4 μg의 ml의 -1 테트라 사이클린 (구정) 복구 된 BAC의 gbaA의 문화를 포함하는 LB의 9 ML을 추가합니다. 성장BAC gbaA 문화 O / N (30)에서 ° C 200 rpm으로 흔들어.
참고 :, 수퍼 gbaA 플라스미드를 electroporating의 변환 효율은 액체 미디어에 포화 성장 O / N을 허용하기에 충분히 높다.
삽입 카세트 및 서브 클로닝 플라스미드 4. 준비
- 후술하는 바와 같이 변형 된 긴 올리고 사용 (PCR)를, 카세트 삽입 (들) 및 서브 클로닝 플라스미드로 HA를 통합 중합 효소 연쇄 반응을 수행한다.
- 옵션 : recombineering 반응에 플라스미드 이월을 방지하기 위해 삽입 카세트를 증폭 R6K 원산지 또는 유사한 좁은 숙주 범위 플라스미드 템플릿을 사용합니다.
- 대안 적으로, RE 다이제스트 (표 2)를 이용하여 플라스미드를 선형화 템플릿. PCR 증폭 영역 이외의 절단 및 열 불 활성화 RE를 선택합니다. 증명할에서 권장하는 열은 RE를 비활성화acturer.
- 고 충실도 핫 스타트 DNA 중합 효소 시스템을 사용하여 중합 효소 연쇄 반응 (PCR)을 설정한다. (표 3)를 자세히 설명 PCR 마스터 믹스를 준비합니다. (표 3)과 같이 열 순환을 수행합니다.
- 아가로 오스 겔 전기 영동에 의해 PCR 생성물을 분석한다. 로드 1-5 1 % 상 각 PCR ㎕의 0.5 MG ml의 -1 에티 디움 브로마이드 (EtBr이)를 포함하는 아가로 오스 겔 (/ V w).
주 : 비 - 특이 적 증폭 생성물의 존재 recombineering 반응에 간섭하지 않는다. 그러나 일부 경우, 프라이머 다이머는 recombineering 효율을 감소시킬 수있다. - PCR 정제 키트를 사용하여 PCR 산물을 정제.
- 옵션 : PCR 청소 다음 DpnI으로 처리하여 PCR들에서 플라스미드 템플릿을 제거합니다. (제조업체에서 권장) 멸균 탈 이온수의 최소 볼륨에서 용출 DNA.
주 : DpnI 첨가 번째 개열의 저감 효율 PCR 반응 결과를하기 미정전자 메틸화 된 플라스미드 DNA 템플릿입니다.
- 옵션 : PCR 청소 다음 DpnI으로 처리하여 PCR들에서 플라스미드 템플릿을 제거합니다. (제조업체에서 권장) 멸균 탈 이온수의 최소 볼륨에서 용출 DNA.
- DNA 기준 예, λ를 HindIII 분해물 또는 집합에 대해 공지 nanodrop 분광 광도계를 이용하여 아가 로스 겔 분석에 의한 PCR 증폭 된 DNA 정량화.
5. 서브 클로닝 플러스 삽입
- 10 ㎖의 LB + 엽록소 + 구정에 200 μl를 추가하여 O / N BAC gbaA 문화 50 배 희석. 30 성장 ° C는 1 시간 50 분 동안 200 rpm으로 진탕. 샘플을 포함하는 음성 대조군으로 사용할 수있다.
- 단계 3.1.2에 설명 된대로 4 ° C에 모든 recombineering 용 재료 및 장비를 진정.
- LB 한천의 pH를 서브 클로닝 벡터 (표 4)를 선택할뿐만 아니라, 삽입 된 DNA 단편의 선택을 허용 할 것이다 적절한 항생제 선택 (들)의 정확한 농도를 함유하는 8 개의 플레이트를 붓는다.
참고 : 일부 항생제의 pH에 민감하다. 사용 LB원칙적으로 한천의 pH 8. - L 아라비 노스의 솔루션 (V / W) 10 %를 준비합니다. 0.2 μm의 주사기 필터를 통해 살균 여과.
주 : 아라비 노스가 gbaA 플라스미드로부터 recombineering 단백질의 발현을 유도한다. - 분광 광도계를 사용하여 BAC의 gbaA 문화의 OD를 확인합니다. 다음 단계에서 설명한 바와 같이 0.25 내지 0.3 600 도달 OD되면 recombineering 단백질을 유도한다.
- 0.2 % 아라비 노스의 최종 농도를 달성하기 BAC 배양을 10mL의 10 % 아라비 노스 용액 200 μL를 추가한다. (아라비 노스)없이 유도되지 않은 배양을 포함하는 음성 대조군으로 사용할 수있다.
- 37 BAC 문화를 전송 C 진탕 배양기를 °와 45 분 230 rpm에서 진탕 레드 발현을 유도.
참고 : 레드 단백질의 발현이 30 ° C에서 비효율적이다. - 세포를 스핀 다운 단계 3.1.5에 설명 된대로 10 % 글리세롤로 3 회 세척한다.
- , 600-1 추가000 개의 서브 클로닝 플라스미드 recombineering 반응에 삽입 카세트 (S)의 각각 겨. 벡터와 카세트의 재조합 능력과 무결성을 확인하는 유일한 벡터 및 벡터 플러스 단일 삽입 컨트롤을 포함합니다.
- 위아래로 pipetting하여 단일 세포 현탁액을 얻습니다. BAC 벡터를 3 시간 동안 37 ° C에서 다중 카피 플라스미드 또는 LB 10ml에 1 시간 동안 37 ° C에서 1 ml의 LB 스텝 3.1.7 이후 회복에 기재된 전기 천공을 수행한다.
- 회수 된 배양 예, 90 %, 10 %, 1 듀얼 선택 아가 플레이트에서 37 %의 성장 접시 다른 희석액 16 시간 동안 C를 °.
재조합 6. 분석
- 옵션 : 6 ~ 12 식민지를 선택하고 HA 플 랭킹 프라이머 및 삽입 특정 프라이머를 사용하여 식민지 PCR을 수행합니다. 서브 클로닝 플라스미드와 삽입 캐스 포함SETTE 플라스미드뿐만 아니라 부정적인 컨트롤 등의 부모 BAC 클론.
- 식민지의 PCR의 아가 로스 젤 분석을 수행합니다. 예상 크기에서 밝은 밴드의 존재에 의해 긍정적 인 클론을 확인합니다.
주 : SPI 클로닝 과정의 높은 효율을 거의 정확한 재조합 체를 생성한다.
- 식민지의 PCR의 아가 로스 젤 분석을 수행합니다. 예상 크기에서 밝은 밴드의 존재에 의해 긍정적 인 클론을 확인합니다.
- 선택적 항생제가 포함 된 5 ㎖의 LB의 pH를 8로 식민지를 선택하고 37 ° C에서 O / N 성장.
- 제조업체의 지침에 따라 열 정제 키트를 사용하여 DNA의 minipreps를 준비합니다.
- 표준 페놀 - 클로로포름 격리 또는 유사한 프로토콜을 사용하여 BAC 미니 프렙 DNA를 준비합니다.
- 미니 프렙 DNA에 RE 다이제스트를 수행합니다. 아가로 오스 겔 전기 영동에 의해 분리 DNA. 정확한 표적화 벡터 단편의 예상 크기를 포함하는 클론을 확인하는 RE 패턴을 분석. 분명히 벡터 부족 삽입 (들) 및 삽입물을 포함하는 벡터를 구별 RE를 선택합니다.
- 옵티오NAL : E. 여전히 존재한다 BAC, 결여되어 세포를 수득 대장균 recombineering 후 DH5alpha 또는 DH10B E.를 변환하는 플라스미드 미니 프렙 DNA를 사용하여 대장균 세포.
- 올리고 합성의 오류를 확인하기 위해 HA 및 삽입 카세트를 통해 DNA 시퀀싱을 수행합니다.
결과
올림피아 벡터
올림피아 타겟팅 벡터는 단일 염기쌍 단백질 코딩 영역의 치환, 형광 마커의 융합 또는 단백질에 친 화성 태그 또는 유전자 발현 카세트의 통합을 포함한 게놈에서 신규 한 시퀀스 기능을 도입 할 필요성을 반영한다. 올림피아 벡터 건설 전략 SPI의 응용 프로그램을 테스트하기 위해, 두 개의 서로 다른 테스트 케이스를 조사 하였다. Dnttip1 단백질 1A (TDIF1) 함께 클래스와 내가 탈 아세틸 화 효소 (HDAC) 히스톤 유사 분열 아세틸 라제 복합체를 형성 상호 작용, deoxynucleotidyltransferase, 터미널을 인코딩 (MIDAC) (28). 세포 분열에 Dnttip1의 역할을 조사하기 위해, 탠덤 친 화성 태그 방식은 단백질 상호 작용 TDIF1을 분리 찍은. 긴 상 동성 영역을 함유하는 p15A 벡터를 이용 Dnttip1 유전자의 일부분을 서브 클로닝하기 이전 시도가 낮은 갭 보수 효율 초래ND 빈번한 탈선 재조합 제품 (데이터는 보이지 않음). 따라서, Dnttip1의 궤적에서 쫓고 벡터의 구조는 도전 recombineering 운동을 제공했다. SPI 전략 (도 3a 종결 코돈을 치환, 적은 카피 p15A 벡터에 Dnttip1 유전자 마지막 엑손 스패닝 (엑손 13)의 12킬로바이트 부 서브 클로닝하고 동시에 엑손 (13)에 이중 친 화성 태그 선택 카세트를 삽입하도록 디자인되었다 ). 2X FLAG-칼 모듈 린 결합 단백질 (CBP)은 FRT-PGK-EM7 - 네오 마이신 (네오) -BGhpA-FRT 카세트 (2.0 킬로바이트)이 120 염기쌍의 HA가 Dnttip1의 측면에 포함 된 듀얼 PTO 수정 올리고를 사용하여 플라스미드 선형화 RE에서 증폭 연결 코돈 중지합니다. p15A의 ZEO Dnttip1의 서브 클로닝 벡터 (1.7 킬로바이트)는 Dnttip1 시퀀스의 끝 상동 200 염기쌍의 영역이 서브 클로닝 할 수록된 건설되었다. Dnttip1 서브 클로닝 플라스미드를 선형화하고, RE는 20 bp의 PCR 변성 올리고 증폭에 사용그 선두 가닥 보호 벡터를 생성합니다. PCR 제품은 DpnI 정제, 처리 및 recombineering 유능한 Dnttip1 BAC E.에 공동-일렉트로했다 콜라이 세포뿐만 아니라 유도되지 않은 대조군 세포. SPI 반응은 한천 플레이트 (그림 3B)를 포함 오신 (ZEO) 및 카나마이신 (칸)에 도금했다. SPI 분석 (12) 재조합 (그림 3C) 모두에서 올바르게 수정 Dnttip1의 쫓고 벡터를 생산했다. DNA 염기 서열은 올리고 합성 또는 PCR 증폭으로 인한 오류가 없음을 확인했습니다.
SPI의 또 다른 예는 P2rx1에서 선택 유전자 카세트를 연결 향상된 옐로우 형광 단백질 (EYFP)의 프레임 내 삽입을 포함했다. P2rx1 유전자는 G 단백질 결합 수용체를 코딩하는 ATP 관문 이온 채널 (29)로서 기능한다. YFP 형광에 링크가 VA의 세포 표면 수용체 P2x1의 추적을 허용rious 기능 분석. P2rx1 기존 recombineering 방법론에 심각한 문제를 제시하고 다른 어려운 궤적이다 (데이터가 표시되지 않음). SPI, 단말 엑손을 포괄 P2rx1 유전자 (엑손 12)의 12킬로바이트 세그먼트를 사용하는 것은 p15A의 ZEO 벡터에 서브 클로닝을 구성하는 엑손 (12)에 종결 코돈을 대체 EYFP -LoxP 형벌 네오 카세트의 공동 삽입 변성시켰다 P2rx1 - EYFP 쫓고 벡터 (그림 3D). 이 예에서, 후행 가닥 보호 p15A 벡터 (1.7 KB)는 BP (230)를 포함하고 HA EYFP 카세트 50 BP HA를 함유하고 또한 미 변성 방치 하였다. EYFP 삽입 카세트 (2.7 KB)을 접합 중첩 pEYFP-C1에서 증폭 된 EYFP 유전자의 PCR, PCR 및 LoxP- PGK-EM7 - 신 BGhpA-에 loxP 카세트에 의해 조립 된, PCR은 pL452 (NCI, 프레드릭)에서 증폭 . SPI 반응 콜로니 수백 생성 (데이터는 보이지 않음). DNA 분석 RE(11) 클론으로부터 제조 minipreps은 대부분이 제대로 조립 P2rx1-EYFP 쫓고 벡터 (그림 3E)을 포함했다. DNA 염기 서열에 의해 추가 검증이 EYFP 카세트의 오류없이 삽입을 보였다. 그러나, 클론의 일부는 수리 태그가없는 틈과 같은 셀 (차선 6-9)에서 태그 격차 수리 플라스미드의 프로필을 보여 주었다. 몇 가지 샘플도 잘못 포함 갭 수리 (레인 2) 또는 mistargeted 플라스미드 (레인 4, 5). 짧은 HA를 사용하는 경우, 또는 긴 DNA 절편 (30)의 레드 processivity에 제약으로부터 발생 대형 카세트 (> 3킬로바이트)를 사용하는 경우 동시 서브 클로닝 및 일부 SPI 재조합 체 타겟팅의 실패는 효율적인 SPI 복제의 제한을 강조한다. 실제로, BP 200에 EYFP 카세트의 HA 증가는 SPI 효율 및 카세트의 정확한 타겟팅 (데이터는 도시되지 않음) 증가 하였다. JM8.N4에서 EYFP의 쫓고 벡터 - P2rx1 타겟팅 유전자마우스 ES 세포 (C57BL / 6 변형) 제대로 대상 P2rx1 - EYFP 순서 (그림 3 층)를 포함 96 중 6 개의 긍정적 인 클론을 생성.
BAC 리포터 벡터
표적 유전자의 BAC 클론은 종종뿐만 아니라 모든 UTRs 등 필요한 업스트림 및 다운 스트림 규제 요소 예를 들어, 인핸서, 자연 레벨 31에서 유전자 발현을 구동하는 내인성 프로모터가 포함되어 있습니다. BAC 기자 벡터는 따라서 유전자 (32)의 내생 발현 패턴을 요점을 되풀이하는 선호하는 차량이다. 그러나, (200킬로바이트까지) BAC 플라스미드의 대형 셀 (33)에 그대로 BAC를 형질 감염에 중요한 실질적인 문제를 제공합니다. 유전자의 필요한 조절 요소를 유지하면서 BAC가 34, 35를 통해 트리밍 BAC 게놈 삽입 체의 크기를 감소,보다 효율적으로 transfecti BAC 및 결과의 취급이 용이있게에. 현재 BAC 공학 기술은 이러한 목적을 달성하기 위해 35 recombineering 여러 발사 속도를 포함한다. (도 BAC가 pBeloBAC11 BAC 벡터 P2rx1 유전자 EYFP 카세트의 동시 삽입 함께 1백68킬로바이트 P2rx1 BAC로부터 전체 길이 P2rx1 유전자를 포함 30킬로바이트 게놈 서열을 서브 클로닝하는 데 사용 된 트리밍에 SPI의 유용성을 입증 4A). 180 bp의 HA를 포함하는 pBeloBAC11 ZEO 벡터 백본 (6.5 킬로바이트)는 PCR 후행 가닥 보호 벡터를 생성 수정 올리고 증폭되었다. 이전 P2rx1 올림피아 벡터 건설에 사용되는 동일한 EYFP 네오 카세트 (3킬로바이트)는, PCR은 180 bp의 HA를 함유하는 보온 가닥 보호 된 올리고를 사용하여 증폭하고 종결 코돈을 대체 P2rx1 엑손 (12)을 대상으로 하였다. 결합 EYFP 카세트의 전기 및 P2rx1 BAC 세포 EXPR에 pBeloBAC11의 ZEO 벡터 다음gbaA의 recombineering 단백질 싱, 문화 BAC 플라스미드를 분리하는 37C에서 3 시간 동안 10 ml의 LB의 pH를 8로 회수 하였다. 순수한 재조합이 한천 플레이트와 ZEO 칸에서 선택하고, 2 × 10-6의 주파수에서 관찰되었다. 식민지 PCR의 유전자형 분석은 분석 하였다 3 6 중 클론 (그림 4B)에 트리밍 성공적으로 BAC를 한 것으로 밝혀졌습니다. EYFP 삽입 사이트에서 장거리 PCR 증폭은 세 가지 긍정적 인 클론 (그림 4B)에서 올바른 카세트 결합을 확인했다. BAC 단부 '이들 클론에서 EYFP 카세트 mistargeting의 원인 (5)의 올바른 폐쇄 분리 될 수도 있지만,'말단 EYFP 인서트가없는, 세 클론도 5에서 수리 된 갭 잘못되었다. 세 EYFP 긍정적 인 BAC 클론 더 RE 다이제스트 분석과 제대로 손질 EYFP 재조합 BAC (그림 4C <예상되는 패턴을 보였다/ strong>을). 서열 분석은 3 탐지에서 단지 1 복제에 EYFP 카세트에 오류가 없음을 밝혔다.
조건부 녹아웃 (CKO) 벡터
유전자 발현의 조건부 삭마 발달 과정을 조사하기 위해 또는 특정 시점에서 생물학적 시스템을 연구하기 위해 중요한 도구이다. 조건부 유전자 녹아웃 전략은 일반적으로 임계 엑손 (CE)를 주변에 loxP 재조합 부위의 위치를 포함한다. Cre 호텔 표현 또는 활성화시 CE의 삭제로 인해 말도 매개 붕괴 (NMD)에 mRNA를 분해 결과 프레임 이동 및 조기 종결 코돈을 생산하고 있습니다. 조건부 유전자를 대상 벡터의 구조는 복잡한 작업이며 여러 서브 클로닝의 단계, 대상 및 변환 (22)을 포함한다. SPI는이 프로세스를 단순화 할 수있는 편리한 경로를 제공한다. 테스트 케이스로서 Zrsr2 유전자의 대립 유전자가 조건부졌습니다SPI 방법 (도 5a)를 사용. Zrsr2 유전자는 스 플라이 싱 인자를 인코딩하여 하나의 사본은 X 염색체 상에 위치한다. 유전자 결실 조건부 상태는 구성 적 유전자 결실 표적 전략에 ES 세포의 선택시 Zrsr2 부족에 적응 세포의 가능성을 제어하기 위해 이러한 경우에 특히 중요하다. SPI는 두 개의 다른 선택은 loxP 형벌 카세트의 삽입과 동시에 ZrSr2 유전자 10킬로바이트 부를 서브 클로닝을 행 하였다. FRT-PGK-EM7 - 네오-FRT-에 loxP 카세트 (2킬로바이트가) PCR은 RE에서 증폭 된 180 bp의 HA 2. 인트론 록스 - PGK - em7-를 포함하는 두 번째 카세트를 ZrSr2을 대상으로 포함 가닥 보호 올리고 보온재 사용 pL451을 소화 블라스트 (BSD) -Rox-에 loxP (2킬로바이트)는 PCR 인트론 3에서 하류 지역과 동일 측면에 두에 loxP 사이트를 180 bp의 HA를 포함하는 지체 가닥 보호 올리고 엑손 3, D CE와 R6K 플라스미드에서 증폭틀 이동 조건부 Cre 호텔 활성화 결과에 eletion 및 조기 종결 코돈을 소개합니다. Zrsr2 서브 클로닝 플라스미드 (1.6 KB) PCR은 10킬로바이트 Zrsr2 시퀀스의 끝을 일치하는 180 bp의 HA를 포함 가닥 보호 올리고 보온재 사용 RE 선형화 (linearization) p15A의 ZEO 플라스미드에서 증폭되었다. ZEO + 네오 + BSD 판에 전에 설명과 도금으로 SPI 반응을 실시 하였다. ZrSr2 조건부 표적화 벡터 성공적 조사 12 재조합 체 (도 5b)의 대부분에서 조립 하였다. 또한 DNA 염기 서열 분석은 ZrSr2 CKO 벡터의 선택 마커 모두의 올바른 삽입을 보였다.

도 1. SPI 클로닝. 단일 단계 카세트 삽입 및 서브 클로닝을 조합 SPI 클로닝 과정의 개요. (A) BAC 클론은 트란pSC101 BADgbaA 플라스미드 sformed과 30C에서 성장. 레드 단백질을 표현하는 아라비 노스 유도 후 (B)는 비대칭 수정 삽입 카세트 및 서브 클로닝 플라스미드는 BAC 클론에 도입하고 최종 벡터를 생성 할 수있는 삽입 및 서브 클로닝 마커 모두 선택됩니다. 더 큰 버전을 보려면 여기를 클릭하십시오 이 그림.
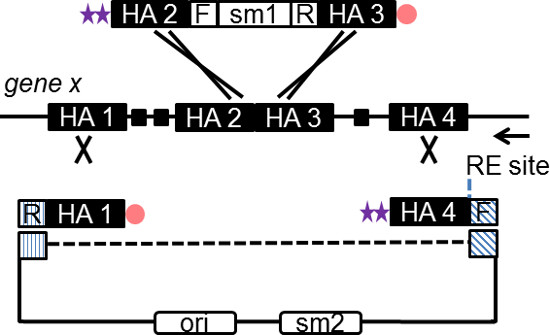
도 2의 도식은 SPI recombineering 올리고의 디자인을 도시. 서브 클로닝 플라스미드와 카세트 삽입 양단 PTO 인산 변성 올리고의 조합을 이용하여 PCR에 의해 생성된다. 생체 내에서 레드 소화에 따라 PCR 단편 복제 포크의 지체 가닥에 결합하고있는 ssDNA를 중간체를 생산하고 있습니다. 창그림과 같이 50 내지 180 BP의 전자 특정 HA는 각 올리고 통합됩니다. 화살표는 후보 유전자 걸쳐 DNA 복제의 방향을 나타낸다. 점선 PCR 생성물에 혼입되지 서브 클로닝 플라스미드 서열을 나타낸다. RE 사이트, 최종 벡터의 선형화의 제한 효소 사이트; F, 서브 클로닝 플라스미드 또는 삽입 카세트에 특정 앞으로 순서 (20 BP); R, 플라스미드 또는 카세트의 역 보완 시퀀스 (20 BP); SM, 선택 마커. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3. SPI는 어려운 쫓고 벡터의 건설을 가능하게한다. Dnttip1 이중의 건설에 사용되는 SPI 전략 (A) 도식 벡터 태그. 그러다가 화살표BAC 클론에 복제의 방향을 케이 츠. Dnttip1 SPI 실험의 유도되지 않은 및 유도 샘플 (B) 도금 결과. Dnttip1 SPI 클론 (C)를 EcoRI 다이제스트. M, 1킬로바이트 사다리 (NEB); C는 p15A Dnttip1 격차는 플라스미드 듀얼 태그 카세트가없는 수리. 조각 크기는 다음과 같습니다 태그, 9.2 + 4.4 + 2.2 킬로바이트; 제어; 9.2 + 4.6 킬로바이트 P2rx1 - EYFP SPI 클론의 (D) SPI 기반 P2rx1-EYFP 쫓고 벡터 건설. (E)를 EcoRI 다이제스트. M, 1킬로바이트 + 사다리 (인비 트 로젠); C는, p15A P2rx1 갭 플라스미드 EYFP 카세트 결여 수리. 조각 크기는 다음과 같습니다 태그, 8.3 + 4.4 + 3.1 + 0.03 KB; 제어; 8.3 + 3.1 + 1.8 + 0.03 KB (F) P2rx1 남부 오점 분석 -. JM8.N4 ES 세포의 세포에서 대상 EYFP 유전자. 상단 패널, PshAI 다이제스트를 사용하여 3 '말단 프로브 서던 블랏. 표시는 5 클론의 심사 결과입니다. 바닥 패널, 남부 사용하여 오최종 심사 (5) 3 '에서 확인 된 양성의 최종 프로브를 SpeI 다이제스트'. PshAI RE 사이트; S,를 SpeI RE 사이트. 점선 HA 벡터의 끝을 나타낸다. 블랙 박스 남부 프로브를 의미한다. 예상 제한 조각, PshAI 있습니다; EYFP - 신, 11.5 KB WT, 8.9 킬로바이트; 를 SpeI :. (WT), 6.9 킬로바이트, EYFP - 신, 7.6 KB 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
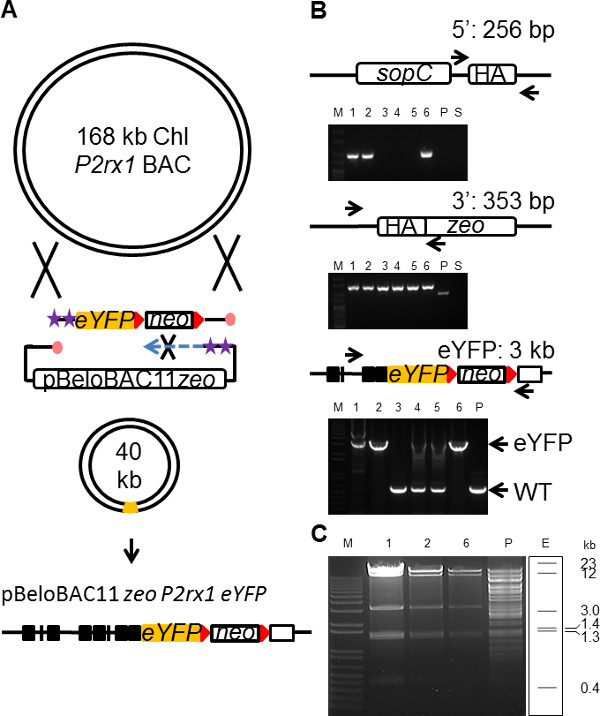
도 4. 간체 BAC는 SPI를 사용하여 BAC 트리밍의 개념을 도시 SPI. (A)를 사용하여 트리밍 회로도. 기호의 핵심은도 1에 기재되어있다. 5 '및 3'에 걸쳐 (B) PCR 증폭 클로닝 인서트 끝나고 P2rx1-EYFP의 insertio 걸쳐N 사이트. 스크리닝 전략은 PCR의 각 유형에 대해 도시된다. M (상단 패널), Hyperladder 25 BP (하단 패널), 1킬로바이트 +; P, P2rx1 BAC; . S, pBeloBAC11의 ZEO 서브 클로닝 플라스미드 (C)는 (B)에서 세 EYFP 긍정적 인 SPI BAC 클론의 소화를 HindIII; E, 트리밍 EYFP BAC의 HindIII로 제한 패턴을 예상했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
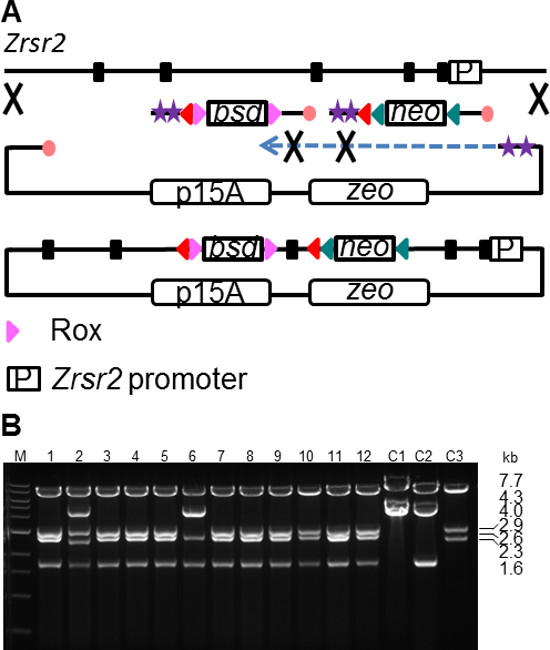
SPI 복제를 사용하여 그림 5. 조건부 녹아웃 벡터 생성. Zrsr2 대립 유전자의 서브 클로닝 동안 서로 다른 두 가지에 loxP 측면에 선택 카세트의 동시 삽입의 (A) 도식. 기호의 핵심은도 1에 기재되어있다. (B) </ Zrsr2 SPI 클론의 강한>를 EcoRI 다이제스트. L, 1킬로바이트 사다리 (NEB); C1은, p15A ZrSr2 격차 수리 플라스미드, C2는 p15A ZrSr2 격차는 플라스미드 신자유 삽입을 포함하는 수리; C3는 p15A ZrSr2 격차는 BSD 삽입을 포함하는 플라스미드를 수리. 조각 크기는 다음과 같습니다 CKO, 7.7 + 2.9 + 2.6 + 1.6 킬로바이트; C1, 7.7 + 4.0 킬로바이트; C2, 7.7 + 4.3 + 1.6 킬로바이트; C3, 7.7 + 2.9 + 2.3 킬로바이트 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 단계 없음 | 기존의 recombineering 파이프 라인 | 멀티 플렉스 recombineering의 B |
| 1 단계 | BAC 호스트에 recombineering 플라스미드의 변환 | BAC 호스트에 recombineering 플라스미드의 변환. 카세트를 대상으로하고 서브 클로닝 벡터의 준비. |
| 단계이 | R1 / R2 게이트웨이 카세트의 삽입 | 멀티 플렉스 갭 수리 복제 |
| 3 단계 | floxed 칸 카세트의 삽입 | 하나의 식민지에서 O / N 문화 |
| 4 단계 | R3 / R4 플라스미드에 갭 수리 | 플라스미드 제제 및 검증 |
| 5 단계 | Cre 호텔 + E.로 변환 대장균 | |
| 6 단계 | 플라스미드 제제 및 검증 | |
| 7 단계 | O / N은 세 방향 게이트웨이 반응 | |
| 8 단계 | E. DH10B로 삼방 게이트웨이의 변환 반응 대장균 세포 | |
| 9 단계 | 하나의 식민지에서 하룻밤 문화 | |
| 10 단계 | 플라스미드 제조 및 서열 확인 | |
| 확인 클론 4 일 B 평균 시간 | ||
표 1 : 조건부 녹아웃 벡터의 건설 SPI와 기존의 recombineering의 비교.
| RE 버퍼 | 5 μL |
| DNA | 플라스미드 또는 1 μg의 정제 된 PCR 제품 |
| RE | 1 μL (5 개 이상) |
| TE | 최대 50 μL |
| 적어도 1 시간 동안 37 ℃에서 배양한다. 제조업체의 지침에 따라 inacitvate 가열 | |
표 2 : 다이제스트 RE.
| PCR 자료 | 최종 농도 |
| PCR 버퍼 | 1 배 |
| 의 dNTP | 200 nm의 |
| 황산 | 1.5 mM의 |
| 베타 | 1.3 M |
| DMSO | 1 % |
| 앞으로 프라이머 | 200 nm의 |
| 역방향 프라이머 | 200 nm의 |
| DNA 중합 효소 | 1 U |
| 주형 | 다중 복제 플라스미드의 10 NG 또는 유전자형 PCR을위한 미니 프렙 DNA의 2.5 μL |
| 물 | 최대 50 μL |
| 다중 복제 플라스미드 표준 50 μL PCR 반응. 장거리 유전자형 PCR을 25 μl의 PCR 반응에 설치되었다. | |
| PCR 조건 | |
| 95 ° C | 2 분 |
| 92 ° C | 10 초 |
| 55 ° C | 30 초 |
| 72 ° C | 30 초 |
| 30 사이클 (B) | |
| B주기가 더 BAC PCR 유전형 35로 확장 할 수 없습니다 | |
표 3 : PCR 설정 및 조건.
| 항생제 | 농도 (μg의 ml의-1) |
| Ampicllin | (50) |
| 블라스트의 B | (40) |
| 클로람페니콜 | 12.5 |
| 겐타 마이신 | 이 |
| 하이 그로 마이신 C | (30) |
| 카나마이신의 B | (15) |
| 테트라 사이클린 | 4 |
| 트리 메소 프림의 C | (10) |
| 오신 | (5) |
| 멀티 플렉스 recombineering의 조합으로 사용되는 경우는 BAC에와 다중 복제 플라스미드와 함께 사용하도록 권장 | |
| B 블라스트 (35 μg의 ml의 1) 및 카나마이신 (6 μg의 ml의 1) 조합에서 함께 사용하는 경우 | |
| C 하이 그로 마이신 및 트리 메소 프림은 단일 복사본의 BAC와 선택하지 않는 것이 좋습니다. | |
SPI 실험에 사용하기 위해 표 4. 권장 항생제 농도.
토론
ES 세포주 및 마우스 모델의 제작은 역사적 변성 대립 유전자를 포함하는 플라스미드 (4) 구조를 이용하여 표적 유전자를 포함하고있다. 그러나, 이러한 복잡한 유전자 타겟팅 벡터의 구성은 이러한 모델 적시 생산 상당한 병목 것으로 입증되었다. recombineering 기반 벡터 건설 전략의 개발은 개선 된 벡터 설계 및보다 효율적인 벡터 조립체를 허용했다. 그럼에도 불구하고, 현재의 recombineering 프로토콜은 여전히 여러 단계를 포함하는 중간 플라스미드 정화를 필요로 다른 균주를 사용합니다. 플러스 삽입을 서브 클로닝하면 상주 BAC 호스트 변형 한 전기 이벤트에서 수행 할 수 벡터 건설에 대한 새로운 접근 방식을 제공합니다. 유전자 타겟팅에서 SPI의 유틸리티는 벡터 건설 다양한 응용 프로그램을 여기에 테스트되었습니다. 여기에 검사를 모든 경우에, SPI 효율적인 것으로 입증하고 올바른 재조합 플라스미드를 제조 하였다.대부분의 경우, 다른 복수의 카세트가 제대로 타겟팅 벡터에 삽입 하였다. 큰 DNA 카세트 및 벡터에 용이 SPI 프로토콜에 수용하고이 시스템의 융통성을 증명 하였다.
SPI는 긴 상 동성 서열과 선형 DNA 카세트 포스 (PTO) 보호의 사용에 의존한다. PTO 수정은 선형 DNA (20, 21)에 exonucleases에 대한 보호를 부여하고 긴 HA 다중화를 허용하는 재조합 효율을 증가시킨다. 그러나, 더 긴 서열의 올리고 합성 에러에게 특히 결실 축적 가능성을 증가시킨다. 그들은 단백질 코딩 영역을 포함하는 경우 올리고 돌연변이 특히 유해 할 수 있습니다. DNA의 HA를 통해 시퀀싱 및 삽입 된 카세트의 전체 길이를 커버는 매우 어떤 순서 변경과 클론을 제거하는 것이 좋습니다. 고 충실도 DNA 폴리머 라제 시스템의 사용은 또한 도입을 피하기 위해 제안Y PCR 오류가 발생합니다. 서브 클로닝 플라스미드의 HA의 길이 및 조성이 삽입 카세트의 그것보다 상대적으로 중요하다 (데이터는 보이지 않음). 엑손 영역에 어떤 돌연변이가 용납되지 올림피아 벡터, 건설 등의 특히 중요한 애플리케이션의 경우, 삽입의 카세트 HA 긴 올리고와 연관된 문제를 피하기 위해 (50~120 BP)을 단축 할 수있다. 삽입은 카세트 (복제의 방향에 대한 지식을 사용할 수없는) 변성 또는 이중 phosphorothioated 남게 될 수있다. 그러나이 경우에 다중 여전히 긴 HA 서브 클로닝 플라스미드 보호가 필요합니다. 이 특정 전략의주의 할 점은 잠재적으로 다른 궤적에서 SPI 복제에 영향을 줄 수있는 다중 효율의 저하이다.
카세트 삽입 및 서브 클로닝 플라스미드의 길이는 SPI 클로닝 또 다른 중요한 파라미터이다. 큰 DNA 분자는 덜 효율적 electroporate 및 효과는 다시 주어진 누적동일한 셀의 모든 카세트를 도입하는 조건을 가지 (데이터는 보이지 않음). 다중 작은 삽입 카세트와 가장 효율적입니다. DNA보다 큰 3킬로바이트도 3킬로바이트 30까지 가장 효율적인 레드 매개 ssDNA의 처리에 제한을 두지 파편입니다. 3킬로바이트를 초과 삽입 카세트 절제된 듀얼하고 베타 독립적 인 통로 (20)를 통해 덜 효율적으로 재결합. 따라서, 정확한 복제를 식별 할 수있는 충분한 식민지의 심사는 이러한 경우에 중요합니다. 재조합 포스트 전기의 긴 기간은 어려운 SPI 운동의 올바른 클론의 회복의 가능성이 높아집니다. 4 개의 소형 카세트 (<1.5 KB) 다중화 효율이 각각의 추가적인 카세트와 함께 감소하지만, SPI 프로세스와 동시에 삽입 될 수있는 최대 (데이타 미기재). 그러나 이것은 최적의 SPI 실험의 결과를 반영하며 많은 대형 카세트를 사용하는 경우 다중의 한계를 고려하는 것이 바람직하다.
클러스터 정기적으로 interspaced 짧은 회문 반복 같은 게놈 편집 도구의 개발은 (CRISPR) -cas9 효소 시스템은 새로운 게놈 수정의 생성을 활성화하고보다 효율적으로 게놈 엔지니어링 (36)하게되었다. 그러나 이러한 새로운 기술은 유전자 대상 벡터를 보완하기보다는 그들을 대신하게했다. 그것은 CRISPR-CAS 시스템은 항생제 선택을 교체하고 추가 멀티 플렉스 recombineering 프로토콜을 구체화 할 수 있다는 예상된다.
공개
T.R.R is the inventor of a British patent application on multiplex recombineering technology.
감사의 말
T.R.R conceived the project, designed and performed experiments and prepared the manuscript. E.J.K and S.E.M performed experiments. All authors contributed intellectually to the final manuscript. This work was funded by a University of Leicester Innovation Fellowship to SEM and by a UK Medical Research Council (MRC) Senior Research Fellowship to S.M.C (MR/J009202/1). DNA sequencing was performed by Protein and Nucleic Acid Laboratories (PNACL), Centre for Core Biotechnology Services, University of Leicester. We wish to thank A. Francis Stewart for providing the Rox and Dre plasmids. pL451 and pL452 plasmids were obtained from National Cancer Institute (NCI), Frederick, USA.
자료
| Name | Company | Catalog Number | Comments |
| Antibiotics except Zeocin | Sigma: http://www.sigmaaldrich.com | Ampicillin, A9518-5G; Chloramphenicol , C1919-5G; Blasticidin, 15205-25MG; Gentamicin, G1264-50MG; Hygromycin, H3274-50MG; Kanamycin,: K1876-1G; Tetracycline hydrochloride, T7660-5G; Trimethoprim, 92131-1G | |
| Zeocin | Invivogen: | ant-zn-1 | Zeocin is also available from Life technologies/Fisher |
| C57BL/6 BAC clones | CHORI: https://bacpac.chori.org/ | RPCI-23 or RPCI-24 BAC libraries | 129/Sv BAC clones can be obtained from Invivogen or Life technologies/Fisher |
| KOD hotstart DNA polymerase system | Millipore: https://www.merckmillipore.com | 71086-3 | |
| L-Arabinose | Sigma: http://www.sigmaaldrich.com | A3256-25G | |
| Long oligos | IDT: https://www.idtdna.com; Gene link: https://www.genelink.com | IDT Ultramers or Gene Link long oligos | PTO and Phosphosphate available as custom modifications. PAGE purification strongly recommended for long oligos. |
| MinElute PCR purification kit | Qiagen: http://www.qiagen.com | 28004 | Minimum elution PCR purifications kits are preferred. |
| QIAPrep Spin Mini Prep kit | Qiagen: http://www.qiagen.com | 27104 | Silica column or similar matrix kits are preferred. |
| Restriction enzymes | NEB: https://www.neb.com | DpnI: R0176L | See NEB website for a list of RE. |
참고문헌
- Dovey, O. M., Foster, C. T., Cowley, S. M. Histone deacetylase 1 (HDAC1), but not HDAC2, controls embryonic stem cell differentiation. Proc. Natl. Acad. Sci. USA. 107, 8242-8247 (2010).
- Harvey, M., et al. Spontaneous and carcinogen-induced tumorigenesis in p53-deficient mice. Nat. Genet. 5, 225-229 (1993).
- Waterhouse, P., et al. Lymphoproliferative disorders with early lethality in mice deficient in Ctla-4. Science. 270, 985-988 (1995).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51, 503-512 (1987).
- Hasty, P., Rivera-Pérez, J., Bradley, A. The length of homology required for gene targeting in embryonic stem cells. Mol. Cell. Biol. 11, 5586-5591 (1991).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. Nature. 317, 230-234 (1985).
- Hirata, R., Chamberlain, J., Dong, R., Russell, D. W. Targeted transgene insertion into human chromosomes by adeno-associated virus vectors. Nat. Biotechnol. 20, 735-738 (2002).
- Szostak, J. W., Orr-Weaver, T. L., Rothstein, R. J., Stahl, F. W. The double-strand-break repair model for recombination. Cell. 33, 25-35 (1983).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309, 255-256 (1984).
- Fu, J., Teucher, M., Anastassiadis, K., Skarnes, W., Stewart, A. F. A recombineering pipeline to make conditional targeting constructs. Methods Enzymol. Soriano, P. M., Wassarman, P. M. 477, Academic Press. 125-144 (2010).
- Zhang, Y., Buchholz, F., Muyrers, J., Stewart, A. A new logic for DNA engineering using recombination in Escherichia coli. Nat. Genet. 20, 123-128 (1998).
- Copeland, N., Jenkins, N., Court, D. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Muyrers, J., Zhang, Y., Testa, G., Stewart, A. Rapid modification of bacterial artificial chromosomes by ET-recombination. Nucleic Acids Res. 27, 1555-1557 (1999).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. USA. 97, 5978-5983 (2000).
- Sharan, S. K., Thomason, L. C., Kuznetsov, S. G., Court, D. L. Recombineering: a homologous recombination-based method of genetic engineering. Nature Protoc. 4, 206-223 (2009).
- Wang, J., et al. An improved recombineering approach by adding RecA to λ red recombination. Mol. Biotechnol. 32, 43-53 (2006).
- Datta, S., Costantino, N., Court, D. L. A set of recombineering plasmids for gram-negative bacteria. Gene. 379, 109-115 (2006).
- Fu, J., et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting. Nat. Biotechnol. 30, 440-446 (2012).
- Muyrers, J., Zhang, Y., Buchholz, F., Stewart, A. RecE/RecT and Redalpha/Redbeta initiate double-stranded break repair by specifically interacting with their respective partners. Genes Develop. 14, 1971-1982 (2000).
- Maresca, M., et al. Single-stranded heteroduplex intermediates in lambda Red homologous recombination. BMC Mol. Biol. 11, 54(2010).
- Mosberg, J. A., Lajoie, M. J., Church, G. M. Lambda Red recombineering in Escherichia coli occurs through a fully single-stranded intermediate. Genetics. 186, 791-799 (2010).
- Liu, P., Jenkins, N. A., Copeland, N. G. A highly efficient recombineering-based method for generating conditional knockout mutations. Genome Res. 13, 476-484 (2003).
- Chan, W., et al. A recombineering based approach for high-throughput conditional knockout targeting vector construction. Nucleic Acids Res. 35, e64(2007).
- Zhang, Y., Muyrers, J. P. P., Testa, G., Stewart, A. F. DNA cloning by homologous recombination in Escherichia coli. Nat. Biotechnol. 18, 1314-1317 (2000).
- Lee, E. C., et al. A highly efficient Escherichia coli-based chromosome engineering system adapted for recombinogenic targeting and subcloning of BAC DNA. Genomics. 73, 56-65 (2001).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Valenzuela, D. M., et al. High-throughput engineering of the mouse genome coupled with high-resolution expression analysis. Nat. Biotechnol. 21, 652-659 (2003).
- Bantscheff, M., et al. Chemoproteomics profiling of HDAC inhibitors reveals selective targeting of HDAC complexes. Nat. Biotechnol. 29, 255-265 (2011).
- Mulryan, K., et al. Reduced vas deferens contraction and male infertility in mice lacking P2X1 receptors. Nature. 403, 86-89 (2000).
- Subramanian, K., Rutvisuttinunt, W., Scott, W., Myers, R. The enzymatic basis of processivity in lambda exonuclease. Nucleic Acids Res. 31, 1585-1596 (2003).
- Antoch, M. P., et al. Functional identification of the mouse circadian Clock gene by transgenic BAC rescue. Cell. 89, 655-667 (1997).
- Rostovskaya, M., et al. Transposon-mediated BAC transgenesis in human ES cells. Nucleic Acids Res. 40, e150(2012).
- Giraldo, P., Montoliu, L. Size matters: use of YACs, BACs and PACs in transgenic animals. Transgenic Res. 10, 83-103 (2001).
- Hill, F., et al. BAC trimming: minimizing clone overlaps. Genomics. 64, 111-113 (2000).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36(2005).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339, 819-823 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유