Method Article
Monitorando a competição de subespécies em uma população de células bacterianas por cocultivação de cepas fluorescentes rotuladas
Neste Artigo
Resumo
As bactérias podem acumular mutações prejudiciais ou benéficas durante a vida. Em uma população de células indivíduos que acumularam mutações benéficas podem rapidamente superar seus companheiros. Aqui apresentamos um procedimento simples para visualizar a competição de intraespécies em uma população de células bacterianas ao longo do tempo usando indivíduos rotulados fluorescentes.
Resumo
Muitos microrganismos, como bactérias, proliferam extremamente rápido e as populações podem atingir altas densidades celulares. Pequenas frações de células em uma população sempre têm mutações acumuladas que são prejudiciais ou benéficas para a célula. Se o efeito fitness de uma mutação fornece a subpopulação com uma forte vantagem de crescimento seletivo, os indivíduos desta subpopulação podem rapidamente superar e até eliminar completamente seus companheiros imediatos. Assim, pequenas alterações genéticas e acúmulo de células que adquiriram mutações benéficas podem levar a uma mudança completa do genótipo de uma população celular. Aqui apresentamos um procedimento para monitorar a rápida expansão clonal e a eliminação de mutações benéficas e prejudiciais, respectivamente, em uma população celular bacteriana ao longo do tempo por cocultivação de indivíduos fluorescentes rotulados da bactéria modelo Gram-positivo Bacillus subtilis. O método é fácil de executar e muito ilustrativo para exibir competição intraespécie entre os indivíduos em uma população celular bacteriana.
Introdução
As bactérias do solo são geralmente dotadas de redes regulatórias flexíveis e amplas capacidades metabólicas. Ambas as características permitem que as células ajustem suas vias catabólicas e anabólicas para competir com seus companheiros e outros microrganismos pelos nutrientes, que estão disponíveis em um determinado nicho ecológico1. No entanto, se as bactérias não conseguem se adaptar ao seu ambiente, outros mecanismos podem explicar a sobrevivência de uma espécie. De fato, como muitas bactérias se proliferam rapidamente e as populações podem atingir altas densidades celulares, subpopulações podem ter mutações benéficas acumuladas espontaneamente que fornecem às células uma vantagem de crescimento seletiva e, portanto, aumentam sua aptidão. Além disso, hotspots mutacionais e mutagênese adaptativa induzida pelo estresse podem facilitar a evolução de uma bactéria mal adaptada2,3. Assim, o acúmulo de mutações e crescimento sob seleção contínua é a origem da enorme diversidade microbiana, mesmo dentro do mesmo gênero4,5. Como na natureza, a modelagem de genomas bacterianos também ocorre em laboratório devido ao cultivo contínuo em seleção. Isso é exemplificado pela domesticação da bactéria Gram-positive B. subtilis, que é usada mundialmente em pesquisa básica e na indústria. Na década de 1940, a subtilis foi tratada com raios-X prejudiciais ao DNA, seguido pelo cultivo sob uma condição específica de crescimento6. As mutações que se acumularam nas bactérias durante sua domesticação são responsáveis pela perda de muitas características de crescimento, ou seja, a cepa de laboratório B. subtilis 168 perdeu a capacidade de formar colônias complexas7,8.
Hoje em dia, para as bactérias modelo mais bem estudadas Escherichia coli e B. subtilis,uma variedade de ferramentas poderosas está disponível para manipular geneticamente seus genomas a fim de abordar questões científicas específicas. Às vezes, a inativação de um gene de interesse causa um grave defeito de crescimento, que é então claramente visível no meio de crescimento padrão9. Em contraste, mutações que causam um fraco defeito de crescimento e, portanto, apenas afetam ligeiramente o condicionamento físico da cepa são muitas vezes ignoradas. No entanto, em ambos os casos a incubação prolongada e a passagem das cepas mutantes por várias gerações geralmente resultam no acúmulo de mutantes supressores que restauraram o fenótipo da cepa dos pais2,9. A caracterização dos mutantes supressores e a identificação das mutações que restauraram o defeito de crescimento da cepa mutante dos pais é uma abordagem muito útil que permite a elucidação de processos celulares importantes e muitas vezes novos10,11.
Estamos interessados no controle da homeostase glutamato em B. subtilis12. Semelhante a E. coli, B. subtilis responde à perturbação da homeostase glutamato (ou seja,bloco na degradação do glutamato2) pelo acúmulo de mutantes supressores. As alterações genômicas nesses mutantes supressores que foram adquiridos por mutação espontânea foram demonstradas para restaurar rapidamente a homeostase glutamato9,13. Portanto, não é de surpreender que a adaptação de B. subtilis a uma condição específica de crescimento durante a domesticação da bactéria seja espelhada na síntese enzimática e nas atividades enzimáticas evoluídas, que estão envolvidas no metabolismo do glutamato12. Tem sido sugerido que a falta de glutamato exógeno no meio de crescimento durante o processo de domesticação foi a força motriz para o surgimento e fixação do gene dehidroase de glutamato enigmático (GDH) gudBCR na cepa laboratorial 1682,14. Esta hipótese é apoiada pela nossa observação de que a quantidade reduzida de atividade de GDH na cepa laboratorial fornece às bactérias uma vantagem de crescimento seletiva quando o glutamato exógeno é escasso2. Além disso, o cultivo de uma cepa B. subtilis, sintetizando o GDH GudB, na ausência de glutamato exógeno resulta no acúmulo de mutantes supressores que inativaram o gene gudB 2. Obviamente, a presença de um GDH catabolicamente ativo é desvantajosa para a célula porque o glutamato produzido endógenamente que poderia ser usado para o anabbolismo é degradado para amônio e 2-oxoglutarate(Figura 1). Em contraste, quando o glutamato é fornecido pelo meio, uma cepa B. subtilis equipada com atividade GDH de alto nível tem uma vantagem de crescimento seletiva sobre uma cepa que sintetiza apenas um GDH funcional. É razoável supor que a atividade gdh de alto nível permite que as bactérias utilizem o glutamato como segunda fonte de carbono, além de outras fontes de carbono fornecidas pelo meio2 (ver Figura 1). Assim, a atividade GDH afeta fortemente o condicionamento físico das bactérias, dependendo da disponibilidade de glutamato exógeno.
Aqui apresentamos um método muito ilustrativo para monitorar e visualizar a competição intraespécie entre duas cepas de B. subtilis que diferem em um único lócus no cromossomo(Figura 2). As duas cepas foram rotuladas com os genes YFP e CFP codificando os fluoroforos YFP e CFP, e cocultivadas em diferentes condições nutricionais. Através da amostragem ao longo do tempo e emplacando diluições apropriadas em placas de ágar, os sobreviventes em cada uma das culturas poderiam ser facilmente monitorados usando um microscópio de fluorescência estéreo comum. O procedimento descrito neste artigo é fácil de realizar e adequado para visualizar a rápida expansão clonal e eliminação de mutações benéficas e prejudiciais, respectivamente, em uma população celular ao longo do tempo.
Protocolo
1. Preparação de Placas de Ágar, Mídia de Cultura, Criostocks e Pré-Culturas
- Prepare a mídia de crescimento e os reagentes necessários (ver tabela de materiais e reagentes).
- Listrar as cepas B. subtilis (por exemplo. BP40 (rocG+ gudBCR amyE::PgudB-yfp) e BP52 (rocG+ gudB+ ame::PgudB-cfp) expressando um e dois GDHs ativos,respectivamente) 2 que serão usados no experimento de competição em placas de ágar médio sp para obter colônias únicas. Incubar as placas durante a noite a 37 °C.
- Pegue colônias simples e tubos de cultura estéril inoculados contendo meio líquido de 4 ml LB. Cresça as bactérias durante a noite a 28 °C e 220 rpm.
- Cresça culturas durante a noite a 28 °C para evitar que as células lise ou esporulam.
- Meça a densidade óptica das culturas em um comprimento de onda de 600 nm (OD600) usando um espectrofotômetro padrão.
- Se as culturas atingiram um OD600 de 2.0, misture 0,75 ml das culturas com 0,75 ml de uma solução estéril de 50% de glicerol em tubos de reação de 1,5 ml. O OD600 final deve ser 1.0 para obter criostocks contendo cerca de 108 células/ml.
- Armazene os tubos a -80 °C.
- Faça três pré-culturas de cada cepa rotulada com os genes de fluoróforo de codificação cfp e yfp. Inocular as pré-culturas (tubos de cultura estéril contendo meio líquido de 4 ml LB) com células de 1 μl a partir de -80 °C criostocks. Incubar as culturas durante a noite a 28 °C e 220 rpm.
2. Cocultivação de Bactérias, Coleta de Amostras e Armazenamento de Amostras
- Prepare recentemente 100 ml C-Glc e CE-Glc meio mínimo (ver tabela de reagentes e materiais), e transfira 20 ml de cada meio em frascos de shake estéreis de 100 ml.
- Pegue 0,1 ml das préculturas que foram cultivadas durante a noite, dilui-as com 0,9 ml de lb médio em um cuvette de 1,5 ml, e determine o OD600.
- Para o experimento de competição, pegue essas pré-culturas das diferentes cepas que têm um OD600 semelhante entre 1,0-1,5.
- Para obter populações de células mistas, diluir as células das préculturas que tinham o OD600 adequado para um OD600 de 0,05 em 20 ml C-Glc e CE-GLC meio mínimo suplementado em frascos de shake de 100 ml. Para o experimento de competição, as duas cepas devem ser misturadas em uma proporção de 1:1.
- Pegue 10 ml de amostras dos frascos, transfira-as para tubos plásticos de 15 ml, colmeia as células por centrifugação por centrifugação por 10 minutos a 4.000 x g em uma centrífuga superior de mesa padrão e descarte o supernascedor.
- Resuspenque as células em 0,5 ml de meio LB fresco, e transfira as células para um copo de reação estéril de 1,5 ml. Adicione 0,5 ml de glicerol 50% estéril, misture a suspensão por um rigoroso vórtice e armazene as amostras em um congelador de -80 °C até um novo tratamento.
- Incubar as células que são deixadas nos frascos de shake por até 24 horas a 37 °C e 220 rpm. Mantenha os frascos de agitação no escuro para evitar o branqueamento fotográfico dos fluoroforos.
- Pegue mais amostras de 0,1 ml após 7 horas e 24 horas de crescimento. Meça e observe o OD600 de diluições de 1:10 (em C-Glc ou CE-Glc médio mínimo) das amostras.
- Pegue a quantidade apropriada de células de cada cultura para fazer criostocks que têm um OD600 de 1.0. Armazene as amostras a -80 °C até um novo tratamento.
3. Tratamento amostral, chapeamento e incubação para análises quantitativas
- Depois de coletar todas as amostras, descongele os criostocks e dilua as células em uma solução salina de 0,9% (ver tabela de reagentes e materiais) até 10-3.
- Placa 0,1 ml das diluições de 10-3 em placas de ágar médio sp e distribuir as células usando pipetas de vidro estéreis.
- Incubar as placas durante a noite a 37 °C no escuro até que colônias únicas tenham aparecido.
4. Contando os Sobreviventes por Microscopia de Fluorescência Estéreo para Análise Quantitativa
- Divida a parte inferior da placa de ágar com uma caneta preta em quatro partes para uma melhor orientação enquanto conta os sobreviventes sob o microscópio de fluorescência estéreo.
- Coloque a placa de cabeça para baixo sob o microscópio e coloque as colônias em foco usando a fonte de luz fria.
- Uma vez que as colônias estejam em foco, mude para o conjunto de filtros apropriado para CFP para visualizar as células sobreviventes da cepa rotulada cfp. Conte os sobreviventes desta cepa rotulando as colônias com uma caneta e note o número.
- Remova as etiquetas com etanol e mude para o conjunto de filtros YFP para visualizar as células sobreviventes da cepa rotulada por yfp. Conte os sobreviventes novamente rotulando as colônias com uma caneta e note o número.
5. Tratamento amostral e Microscopia para Análises Semiquantitativas
- Para figuras ilustrativas, local 10 μl da diluição de 10-4 (aproximadamente 100 células) do passo 3.1 em uma placa de ágar SP (ver Figura 3).
- Incubar as placas durante a noite a 37 °C no escuro até que colônias únicas tenham aparecido.
- Coloque a placa de Petri sem tampa sob o microscópio e coloque o local em foco usando a fonte de luz fria.
- Tire fotos dos pontos para figuras ilustrativas. Escolha um tempo de exposição adequado para tirar as fotos das colônias com a fonte de luz fria.
- Sem mover a placa, mude para o conjunto do filtro CFP, ajuste o tempo de exposição e tire uma foto. Faça o mesmo com o conjunto de filtros YFP e guarde as imagens para análises posteriores.
6. Análise de dados
- Use softwares como o Excel para análises quantitativas de dados. Com base nas colônias contadas, calcule a porcentagem de colônias amarelas e azuis em relação ao número total de colônias, que estão fixadas em 100%.
- Use os números calculados para criar um diagrama de barra empilhado (ver Figura 4 e Figura 5). Use um programa de processamento de imagens como o Adobe Photoshop para construir fotos mescladas das fotos tiradas dos diferentes pontos. Alternativamente, o software ImageJ disponível livremente, pode ser usado http://rsbweb.nih.gov/ij/ para processamento de imagens.
- Abra as imagens de um ponto que foram tiradas com o conjunto de filtros CFP e o conjunto de filtros YFP. Otimize o contraste e o brilho para reduzir qualquer fluorescência de fundo da mídia.
- Vá a uma das fotos e selecione todas. Copie a foto e cole-a na outra foto.
- Use a função "esquiva de cor" para mesclar as imagens ou sobrepor as imagens fluorescentes usando a guia canais. Imagens mescladas das colônias de diferentes mídias e diferentes pontos de tempo representam o crescimento das diferentes cepas dentro da cultura líquida.
7. Dicas específicas: Experimento do interruptor de corante e cocultivação de cepas isogênicas rotuladas com cfp e yfp
A expressão de qualquer um dos dois genes codificadores de fluoróforos em B. subtilis pode influenciar o condicionamento físico e, portanto, a taxa de crescimento das bactérias. Portanto, recomenda-se realizar os seguintes experimentos, a fim de excluir que a eliminação de uma cepa concorrente da população celular durante o cultivo é simplesmente devido a um efeito negativo do fluorohore:
- Repita todo o experimento e cocultivamente rotulado cepas (por exemplo, a cepa anteriormente rotulada cfpé agora rotulada com o gene yfp e vice-versa). Embora inversos, os resultados obtidos devem ser comparáveis à observação anterior de que uma cepa tem uma vantagem de crescimento seletiva sobre a outra cepa.
- Repita todo o experimento e cocultivato cepas isogênicas rotuladas com yfp e cfp (por exemplo, BP40 (rocG+ gudBCR amyE::PgudB-yfp) e BP41(rocG+ gudBCR amyE::PgudB-cfp)). Estimar o efeito negativo de qualquer um dos dois fluoroforos na aptidão das bactérias seguindo a composição da população celular ao longo do tempo.
Resultados
O método aqui descrito foi aplicado com sucesso para visualizar a competição de intraespécies em uma população celular constituída por cepas de B. subtilis que foram rotuladas com os genes CFP e YFP codificando os fluoroforos CFP e YFP, respectivamente. Como mostrado na Figura 3,o método pode ser usado para visualizar a competição de intraespécies de forma muito ilustrativa. Ao detectar as amostras em pequenas áreas, a composição clonal da população celular tornou-se visível rapidamente. Embora não seja apropriada para análises quantitativas, essa abordagem é útil para estimar aproximadamente o efeito de diferentes parâmetros de crescimento (ou seja,fonte de nitrogênio) sobre o desenvolvimento de uma população celular que inicialmente continha ambas as cepas em quantidades iguais(Figura 3). Além disso, em uma abordagem em pequena escala, a aptidão de diferentes cepas de B. subtilis que foram cultivadas sob a mesma condição de crescimento pode ser testada usando uma única placa de ágar. Para análises quantitativas recomenda-se propagar as amostras sobre toda a superfície de uma placa de ágar. Isso evitará a sobreposição das colônias e, assim, permitirá a identificação e contagem distintas de colônias que emergiram de células únicas. Ao emplacar diluições apropriadas em placas de ágar, a composição clonal de uma população celular ao longo do tempo pode ser precisamente determinada simplesmente contando as colônias fluorescentes amarelas e azuis (ver Figura 4). Como já relatamos anteriormente, a atividade GDH afeta fortemente a aptidão de B. subtilis dependendo da disponibilidade de glutamato exógeno2. Obviamente, na ausência de glutamato exógeno a atividade GDH de alto nível é desvantajosa para as bactérias, pois as enzimas RocG e GudB degradam o glutamato que é necessário no anabolismo (ver Figura 1 e Figura 4A). Em contrapartida, se fornecido à bactéria, o glutamato pode servir como um doador do grupo amino em reações de transaminação. Além disso, o glutamato pode ser alimentado no metabolismo de carbono e usado como fonte de energia devido à presença dos GDHs catabolicamente ativos RocG e GudB (Figura 1 e Figura 4B). Como mostrado nas Figuras 4C e 4D,resultados semelhantes foram obtidos em um experimento de interruptor de corante. Mais uma vez, as bactérias equipadas com atividade GDH de alto nível foram superadas por células com atividade GDH reduzida em meios de crescimento sem glutamato. Em contrapartida, as bactérias sintetizando apenas um GDH ativo foram eliminadas da cultura quando o meio foi suplementado com glutamato. Como mostrado nas Figuras 5A e 5B, a composição inicial da população de células mistas permaneceu quase constante ao longo do tempo. Assim, no experimento de competição, a eliminação de qualquer uma das duas cepas que estavam equipadas com diferentes quantidades de atividade de GDH não foi devido a um defeito de crescimento causado pelos fluoroforos (ver Figura 4). Em conjunto, o uso de fluoroforos é uma poderosa ferramenta para monitorar a competição de intraespécies em uma população de células bacterianas.

Figura 1. A ligação entre o metabolismo de carbono e nitrogênio em B. subtilis. Quando o glutamato não é fornecido pelo meio, o principal doador de amino que é necessário para o anabolismo é sintetizado a partir de amônio e 2-oxoglutarate pela ação combinada da sitese glutamina (SG) e da sinthase glutamato (GOGAT). Em contraste, na presença de glutamato exógeno os GDHs catabolicamente ativos RocG e/ou GudB podem degradar o glutamato ao amônio e 2-oxoglutarate, que então serve como fonte de carbono.
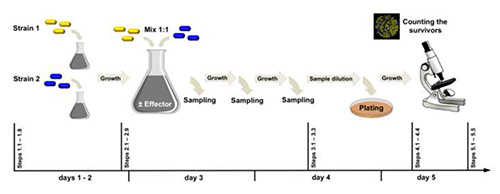
Figura 2. Fluxo de trabalho experimental. A cepa 1 (rotulada com yfp) e a tensão 2 (rotulada com cfp) diferem em um lócus um do outro. No exemplo aqui apresentado, comparamos o efeito do glutamato exógeno (effector) na mudança genotipada da população celular que inicialmente continha 50% das células rocG+ gudB+ (codificação de dois GDHs ativos) e 50% das células rocG+ gudBCR (codificando um GDH ativo). Clique aqui para ver imagem maior.

Figura 3. Abordagem semiquantitativa para visualizar a competição de subespécies de forma descritiva (ver seção 5). Antes da cocultivação (0h), e após 7 horas e 24 horas de diluições de crescimento (10-4) de células foram avistadas em placas de ágar sp. As células sobreviventes que formaram colônias após 12 horas de incubação a 37 °C foram identificadas por microscopia de fluorescência estéreo. Tempo de exposição, 0,6 seg; barra de escala, 1 mm. Esta figura foi modificada de Gunka et al. 20132.

Figura 4. Quantificação da competição de intraespécies. Após diluição amostral e propagação das células (ver passos 3.1-3.3) no meio de SP as placas foram incubadas durante a noite a 37 °C. Colônias amarelas e azuis foram quantificadas como descrito nos Protocolos 4 e 6. As barras de erro pretas representam desvios padrão para pelo menos quatro experimentos repetidos independentemente. Cada placa de ágar continha pelo menos 100 colônias contáveis. (A) Na ausência de glutamato exógeno, a cepa B. subtilis BP40 (amarela) equipada com apenas uma cepa funcional de GDH BP54 (azul), que sintetiza ambas as enzimas degradantes do glutamato, RocG e GudB. (B) Em contraste, a síntese de dois GDHs funcionais é vantajosa para as bactérias quando o glutamato exógeno está disponível porque, além da glicose, o glutamato é usado como fonte de carbono. Como mostrado em (C) e (D), os resultados comparáveis foram obtidos em um experimento de interruptor de corante. Esta figura foi modificada de Gunka et al. 20132. Clique aqui para ver imagem maior.

Figura 5. Teste de controle para avaliar o efeito dos genes cfp e yfp de codificação fluoroforeiro e yfp sobre a aptidão das bactérias. Populações mistas das cepas isogênicas BP40 (rocG+ gudBCR amyE:::yfp) e BP41 (rocG+ gudBCR amyE:::cfp) ou BP52(rocG+ gu aiE +amiE::cfp ) e BP156 (rocG+ gudB+ ame::yfp) foram cultivados na ausência (A) e na presença (B) de glutamato exógeno. As células sobreviventes foram contadas como descrito nos Protocolos 1-4 e 6, respectivamente. As barras representam desvios padrão para pelo menos quatro experimentos repetidos independentemente. Esta figura foi modificada de Gunka et al. 20132. Clique aqui para ver imagem maior.
Discussão
Vários métodos foram desenvolvidos para analisar a aptidão competitiva das bactérias16. Em muitos casos, as bactérias foram rotuladas com diferentes fitas de resistência a antibióticos17. Semelhante à nossa abordagem, a rotulagem das células com fitas de resistência a antibióticos permite a avaliação da aptidão competitiva das bactérias durante a cocultivação em condições de crescimento definidas. Além disso, este método pode ser usado para determinar a aptidão competitiva das células que diferem umas das outras em um lócus específico no cromossomo17. No entanto, existem algumas desvantagens usando fitas de resistência a antibióticos para monitorar o condicionamento físico competitivo. Como a expressão dos genes de resistência é principalmente impulsionada por promotores com força desigual, as enzimas que conferem resistência aos antibióticos são provavelmente produzidas em diferentes níveis. Portanto, efeitos fracos de aptidão podem não ser detectáveis com essa abordagem. Em nossa abordagem, ambos os genes fluoróforos foram integrados com o mesmo marcador de seleção e sua expressão é impulsionada pelos mesmos promotores2. Outra desvantagem para monitorar a aptidão competitiva com fitas de resistência a antibióticos pode ser que a abordagem seja mais trabalhosa, pois dois tipos de placas de ágar suplementadas com os antibióticos apropriados são necessárias para a contagem da colônia. Alternativamente, a aptidão das bactérias pode ser determinada simplesmente monitorando a taxa de crescimento e pelo cálculo do chamado índice de aptidão16. Obviamente, esta é a abordagem mais precisa porque as bactérias são cultivadas individualmente e compostos tóxicos que podem ser produzidos por uma cepa com um certo genótipo não afetarão o crescimento da cepa concorrente. Além disso, não há necessidade de usar antibióticos que possam afetar o crescimento das células. No entanto, ambas as abordagens não são muito ilustrativas, pois os números que descrevem a aptidão competitiva só podem ser apresentados de forma bastante neutra.
O uso de genes de codificação fluoróforo para monitorar e quantificar a concorrência de intraespécies tem várias vantagens sobre outros métodos. Se ambas as cepas integraram os genes de codificação fluoróforo por dupla recombinação homólogona no cromossomo, não há necessidade de usar antibióticos em nenhuma das etapas de cultivo. Portanto, amostras que são retiradas da cultura durante o cultivo podem ser analisadas no mesmo meio de crescimento e ambas as cepas são capazes de crescer. Essa abordagem permite a visualização da competição intraespécie de forma muito ilustrativa. Além disso, usando essa abordagem semiquantitativa várias condições de crescimento podem ser testadas ao mesmo tempo e muitas cepas diferentes podem ser comparadas em paralelo. Finalmente, não há necessidade de fixação das amostras em slides de microscópio porque as amostras que foram retiradas da cultura bacteriana durante a cocultivação podem ser armazenadas em um freezer18. Assim, todas as amostras e réplicas podem ser analisadas ao mesmo tempo.
Dois passos críticos em nosso protocolo devem ser mencionados. É importante notar que os criostocks devem conter quantidades iguais de células na mesma fase de crescimento de cada cepa concorrente. Uma desproporção inicial das cepas nos criostocks e, consequentemente, nos frascos de shake antes de iniciar o experimento terá um forte impacto no resultado do experimento de competição. Portanto, é prudente verificar a composição dos criostocks antes do experimento. Além disso, uma quantidade adequada de células deve ser propagada uniformemente sobre a placa. Caso contrário, as colônias emergiam estão muito próximas umas das outras e uma determinação precisa das células sobreviventes se tornará difícil.
Há também algumas limitações e desvantagens da abordagem baseada em fluorforeiro. A cocultivação em um leitor de placas multi-bem e a detecção simultânea dos sinais CFP e YFP não é possível, pois os espectros de excitação e emissão dos fluoroforos estão muito próximos um do outro. No entanto, esse problema técnico pode ser contornado usando diferentes proteínas fluorescentes, como aquelas que emitem luz verde e vermelha, baseadas idealmente no mesmo andaime proteico. Outra desvantagem da abordagem baseada em fluoróforos pode ser que algumas mutações causam apenas um fraco defeito de crescimento da bactéria. Assim, se o defeito de crescimento que pode ser causado por qualquer um dos genes codificadores de fluoroforeiro é mais forte do que o defeito de crescimento causado por uma certa mutação, a abordagem baseada em fluoróforo não é apropriada para analisar a concorrência de intraespécies. Portanto, antes de criar um conjunto inteiro de cepas recomenda-se primeiro rotular apenas a cepa pai com ambos, cfp e yfp, e cocultivar as cepas. Os experimentos de crescimento revelarão o quão forte os fluoroforos afetam o condicionamento físico das bactérias (ver Figuras 5A e 5B).
No futuro, será interessante testar se a abordagem baseada em fluoroforeiro para monitorar a concorrência de intraespécies será mais precisa e menos trabalhosa se as células sobreviventes forem contadas usando citometria de fluxo. Recentemente, a citometria de fluxo tem se mostrado uma ferramenta poderosa para analisar a composição dos biofilmes B. subtilis 19. Além disso, pode ser mais apropriado analisar efeitos fracos de aptidão que afetam a aptidão competitiva das bactérias por cocultivação contínua das bactérias rotuladas por fluoróforo em um fermentador. Em contraste com os frascos de agitação, essa abordagem permite manter as condições de crescimento constantes e, assim, monitorar a concorrência intraespécie por um longo período de tempo.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
O trabalho no laboratório dos autores foi apoiado pela Deutsche Forschungsgemeinschaft (http://www.dfg.de; CO 1139/1-1), a Fonds der Chemischen Industrie (http://www.vci.de/fonds) e o Centro de Biologia Molecular de Göttingen (GZMB). Os autores gostariam de reconhecer Jörg Stülke por comentários úteis e leitura crítica do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| (NH4)2SO4 | Roth, Germany | 3746 | |
| Agar | Difco, USA | 214010 | |
| Ammonium ferric citrate (CAF) | Sigma-Aldrich, Germany | 9714 | |
| CaCl2 | Roth, Germany | 5239 | |
| Glucose | Applichem, Germany | A3617 | |
| Glycerol | Roth, Germany | 4043 | |
| K2HPO4•3H2O | Roth, Germany | 6878 | |
| KCl | Applichem, Germany | A3582 | |
| KH2PO4 | Roth, Germany | 3904 | |
| KOH | Roth, Germany | 6751 | |
| MgSO4•7H2O | Roth, Germany | P027 | |
| MnCl2 | Roth, Germany | T881 | |
| MnSO4•4H2O | Merck Millipore, Germany | 102786 | |
| NaCl | Roth, Germany | 9265 | |
| Nutrient broth | Roth, Germany | X929 | |
| Potassium glutamate | Applichem, Germany | A3712 | |
| Tryptone | Roth, Germany | 8952 | |
| Tryptophan | Applichem, Germany | A3445 | |
| Yeast extract | Roth, Germany | 2363 | |
| 1.5 ml Reaction tubes | Sarstedt, Germany | 72,690,001 | |
| 2.0 ml Reaction tubes | Sarstedt, Germany | 72,691 | |
| 15 ml Plastic tubes with screw cap | Sarstedt, Germany | 62,554,001 | |
| Petri dishes | Sarstedt, Germany | 82.1473 | |
| 1.5 ml Polystyrene cuvettes | Sarstedt, Germany | 67,742 | |
| 15 ml Glass culture tubes | Brand, Germany | 7790 22 | |
| 100 ml Shake flasks with aluminium caps | Brand, Germany | 928 24 | |
| Sterile 10 ml glass pipettes | Brand, Germany | 278 23 | |
| Incubator (28 and 37 °C) | New Brunswick | M1282-0012 | |
| Standard pipette set (2-20 μl, 10-100 μl, 100-1,000 μl) | Eppendorf, Germany | 4910 000.034, 4910 000.042, | |
| Table top centrifuge for 1.5 and 2 ml reaction tubes | Thermo Scientific, Heraeus Fresco 21, Germany | 75002425 | |
| Table top centrifuge for 15 ml plastic tubes | Heraeus Biofuge Primo R, Germany | 75005440 | |
| Standard spectrophotometer | Amersham Biosciences Ultrospec 2100 pro, Germany | 80-2112-21 | |
| Stereofluorescence microscope | Zeiss SteREO Lumar V12, Germany | 495008-0009-000 | |
| Freezer (-20 and -80 °C) | - | - | |
| Fridge (4 °C) | - | - | |
| Autoclave | Zirbus, LTA 2x3x4, Germany | - | |
| pH meter | pH-meter 766, Calimatic, Knick, Germany | 766 | |
| Vortex | Vortex 3, IKA, Germany | 3340000 | |
| Balance | CP2202S, Sartorius, Germany | replaced by | |
| Black pen (permanent marker) | Staedler, Germany | 317-9 | |
| Powerpoint program | Microsoft, USA | - | |
| Office Excel program | Microsoft, USA | - | Program for data processing |
| Adobe Photoshop CS5 | Adobe, USA | replaced by CS6, download | Computer program for image processing |
| Computer | PC or Mac | - | |
| ZEN pro 2011 software for the stereofluorescence microscope | Zeiss, Germany | 410135 1002 110 | AxioCam MRc Rev. Obtained through Zeiss |
| Specific solution recipes | |||
| SP Medium | |||
| 8 g Nutrient broth | |||
| 0.25 mg MgSO4•7H2O | |||
| 1 g KCl | |||
| 15 g agar for solid SP medium | if required | ||
| add 1 L with H2O | autoclave for 20 min at 121 °C | ||
| 1 ml CaCl2 (0.5 M), sterilized by filtration | |||
| 1 ml MnCl2 (10 mM), sterilized by filtration | |||
| 2 ml ammonium ferric citrate (CAF, 2.2 mg/ml), sterilized by filtration | |||
| LB Medium | |||
| 10 g Tryptone | |||
| 5 g Yeast extract | |||
| 10 g NaCl | |||
| 15 g agar for solid LB medium | if required | ||
| add 1 L with H2O | autoclave for 20 min at 121 °C | ||
| C-Glc Minimal Medium | |||
| 200 ml 5 x C salts | |||
| 10 ml L-Tryptophan (5 mg/ml), sterilized by filtration | |||
| 10 ml ammonium ferric citrate (CAF, 2.2 mg/ml), sterilized by filtration | |||
| 10 ml III’ salts | |||
| 25 ml Glucose (20%) | autoclaved for 20 min at 121 °C | ||
| add 1 L with sterile H2O | |||
| CE-Glc Minimal Medium | |||
| 200 ml 5 x C salts | |||
| 10 ml L-Tryptophan (5 mg/ml), sterilized by filtration | |||
| 10 ml ammonium ferric citrate (CAF, 2.2 mg/ml), sterilized by filtration | |||
| 10 ml III’ salts | |||
| 20 ml Glutamate (40%) | |||
| 25 ml Glucose (20%), autoclaved for 20 min at 121 °C | |||
| add 1 L with sterile H2O | |||
| 5 x C salts | |||
| 20 g KH2PO4 | |||
| 80 g K2HPO4•3H2O | |||
| 16.5 g (NH4)2SO4 | |||
| add 1 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| III’ salts | |||
| 0.232 g MnSO4•4H2O | |||
| 12.3 g MgSO4•7H2O | |||
| add 1 L with sterile H2O, autoclave for 20 min at 121 °C | |||
| 40% Glutamate solution | |||
| 200 g L-Glutamic acid | |||
| adjust the pH to 7.0 by adding approximately 80 g KOH | |||
| add 0.5 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| 0.9% Saline (NaCl) Solution | |||
| add 1 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| 50% Glycerol solution | |||
| 295 ml Glycerol (87%) | |||
| add 0.5 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| Bacteria (All strains are based on the Bacillus subtilis strain 168) | |||
| Bacillus subtilis BP40 (rocG+ gudBCR amyE::PgudB-yfp) | Laboratory strain collection | ||
| Bacillus subtilis BP41 (rocG+ gudBCR amyE::PgudB-cfp) | |||
| Bacillus subtilis BP52 (rocG+ gudB+ amyE::PgudB-cfp) | |||
| Bacillus subtilis BP156 (rocG+ gudB+ amyE::PgudB-yfp) | |||
Referências
- Buescher, F. I., et al. Global network reorganization during dynamic adaptations of Bacillus subtilis metabolism. Science. 335, 1099-1103 (2012).
- Gunka, K., Stannek, L., Care, R. A., Commichau, F. M. Selection-driven accumulation of suppressor mutants in Bacillus subtilis: the apparent high mutation frequency of the cryptic gudB gene and the rapid clonal expansion of gudB+ suppressors are due to growth under selection. PLoS One. 8, (2013).
- Al Mamum, A. A. M., et al. Identity and function of a large gene network underlying mutagenic repair of DNA breaks. Science. 338, 1344-1348 (2012).
- Koeppel, A. F., Wertheim, J. O., Barone, L., Gentile, N., Krizanc, D., Cohan, F. M. Speedy speciation in a bacterial microcosm: new species can arise as frequently as adaptations within a species. ISME J. 7, 1080-1091 (2013).
- Maughan, H., Nicholson, W. L. Increased fitness and alteration of metabolic pathways during Bacillus subtilis evolution in the laboratory. Appl. Environ. Microbiol. 77, 4105-4118 (2011).
- Burkholder, P. R., Giles, N. H. Induced biochemical mutations in Bacillus subtilis. Am. J. Bot. 34, 345-348 (1947).
- McLoon, A. L., Guttenplan, S. B., Kearns, D. B., Kolter, R., Losick, R. Tracing the domestication of a biofilm-forming bacterium. J. Bacteriol. 193, 2027-2034 (2011).
- Zeigler, D. R., et al. The origins of 168, W23, and other Bacillus subtilis legacy strains. J. Bacteriol. 190, 6983-6995 (2008).
- Gunka, K., Tholen, S., Gerwig, J., Herzberg, C., Stülke, J., Commichau, F. M. A high-frequency mutation in Bacillus subtilis: requirements for the decryptification of the gudB glutamate dehydrogenase. 194, 1036-1044 (2012).
- Commichau, F. M., et al. Characterization of Bacillus subtilis mutants with carbon source-independent glutamate biosynthesis. J. Mol. Microbiol. Biotechnol. 12, 106-113 (2007).
- Beckwith, J. Genetic suppressors and recovery of repressed biochemical memory. J. Biol. Chem. 284, 12585-12592 (2009).
- Gunka, K., Commichau, F. M. Control of glutamate homeostasis in Bacillus subtilis: a complex interplay between ammonium assimilation, glutamate biosynthesis and degradation. Mol. Microbiol. 85, 213-224 (2012).
- Yan, D. Protection of the glutamate pool concentrations in enteric bacteria. Proc. Natl. Acad. Sci. U.S.A. 104, 9475-9480 (2007).
- Belitsky, B. R., Sonenshein, A. L. Role and regulation of Bacillus subtilis glutamate dehydrogenase genes. J. Bacteriol. 180, 6298-6305 (1998).
- Commichau, F. M., Herzberg, C., Tripal, P., Valerius, O., Stülke, J. A regulatory protein-protein interaction governs glutamate biosynthesis in Bacillus subtilis: the glutamate dehydrogenase RocG moonlights in controlling the transcription factor GltC. Mol. Microbiol. 65, 642-654 (2007).
- Gordo, I., Perfeito, L., Sousa, A. Fitness effects of mutations in bacteria. J. Mol. Microbiol. Biotechnol. 21, 20-35 (2011).
- Rabatinová, A., et al. The δ subunit of RNA polymerase is required for rapid changes in gene expression and competitive fitness of the cell. J. Bacteriol. 195, 2603-2611 (2013).
- Capra, E. J., Perchuk, B. S., Skerker, J. M., Laub, M. T. Adaptive mutations that prevent crosstalk enable the expansion of paralogous signalling protein families. Cell. 150, 222-232 (2012).
- García-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J. Vis. Exp. (15), (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados