Method Article
Monitoraggio della concorrenza intraspecie in una popolazione di cellule batteriche mediante cocultivazione di ceppi marcati fluorescentmente
In questo articolo
Riepilogo
I batteri possono accumulare mutazioni dannose o benefiche durante la loro vita. In una popolazione di cellule individui che hanno accumulato mutazioni benefiche possono rapidamente sovracompetire i loro simili. Qui presentiamo una procedura semplice per visualizzare la competizione intraspecie in una popolazione di cellule batteriche nel tempo utilizzando individui etichettati fluorescentmente.
Abstract
Molti microrganismi come i batteri proliferano estremamente velocemente e le popolazioni possono raggiungere alte densità cellulari. Piccole frazioni di cellule in una popolazione hanno sempre mutazioni accumulate che sono dannose o benefiche per la cellula. Se l'effetto di forma fisica di una mutazione fornisce alla sottopopolazione un forte vantaggio di crescita selettiva, gli individui di questa sottopopolazione possono rapidamente sovracompetarsi e persino eliminare completamente i loro compagni immediati. Pertanto, piccoli cambiamenti genetici e accumulo di cellule basate sulla selezione che hanno acquisito mutazioni benefiche possono portare a uno spostamento completo del genotipo di una popolazione cellulare. Qui presentiamo una procedura per monitorare la rapida espansione clonale e l'eliminazione delle mutazioni benefiche e dannose, rispettivamente, in una popolazione di cellule batteriche nel tempo per cocultivazione di individui etichettati fluorescentmente del batterio modello Gram-positivo Bacillus subtilis. Il metodo è facile da eseguire e molto illustrativo per mostrare la competizione intraspecie tra gli individui in una popolazione di cellule batteriche.
Introduzione
I batteri del suolo sono solitamente dotati di reti normative flessibili e ampie capacità metaboliche. Entrambe le caratteristiche consentono alle cellule di regolare i loro percorsi catabolici e anabolizzanti per competere con i loro simili e altri microrganismi per i nutrienti, che sono disponibili in una data nicchia ecologica1. Tuttavia, se i batteri non sono in grado di adattarsi al loro ambiente, altri meccanismi possono causare la sopravvivenza di una specie. Infatti, poiché molti batteri proliferano rapidamente e le popolazioni possono raggiungere sottopopolazioni ad alta densità cellulare possono avere mutazioni benefiche accumulate spontaneamente che forniscono alle cellule un vantaggio di crescita selettivo e quindi aumentano la loro forma fisica. Inoltre, gli hotspot mutazionali e la mutagenesi adattiva indotta dallo stress possono facilitare l'evoluzione di un batteriomaladotto 2,3. Pertanto, l'accumulo di mutazioni e la crescita sotto selezione continua è l'origine dell'enorme diversità microbica, anche all'interno dellostesso genere 4,5. Come in natura, la formatura dei genomi batterici si verifica anche in laboratorio a causa della coltivazione continua in fase di selezione. Ciò è esemplificato dall'addomesticamento del batterio Gram-positivo B. subtilis, che viene utilizzato in tutto il mondo nella ricerca di base e nell'industria. Negli anni '40 B. subtilis è stato trattato con raggi X dannosi per il DNA seguiti da coltivazioni in una specifica condizione di crescita6. Le mutazioni che si sono accumulate nei batteri durante la loro addomesticamento causano la perdita di molte caratteristiche di crescita, cioè il ceppo di laboratorio B. subtilis 168 ha perso la capacità di formare colonie complesse7,8.
Al giorno d'oggi, per i batteri modello meglio studiati Escherichia coli e B. subtilis, è disponibile una varietà di potenti strumenti per manipolare geneticamente i loro genomi al fine di affrontare specifiche questioni scientifiche. A volte l'inattivazione di un gene di interesse causa un grave difetto di crescita, che è quindi chiaramente visibile sul mezzo di crescita standard9. Al contrario, le mutazioni che causano un difetto di crescita debole e quindi influenzano solo leggermente la forma fisica del ceppo vengono spesso ignorate. Tuttavia, in entrambi i casi l'incubazione prolungata e la passatura dei ceppi mutanti per diverse generazioni di solito si traducono nell'accumulo di mutanti soppressori che hanno ripristinato il fenotipo del ceppogenitore 2,9. La caratterizzazione dei mutanti soppressori e l'identificazione delle mutazioni che hanno ripristinato il difetto di crescita del ceppo mutante genitore è un approccio molto utile che consente la spiegazione di processi cellulari importanti e spessonuovi 10,11.
Siamo interessati al controllo dell'omeostasi glutammato in B. subtilis12. Simile a E. coli, B. subtilis risponde alla perturbazione dell'omeostasi del glutammato ( cioè blocco nella degradazione del glutammato2)dall'accumulodi mutanti soppressori. Le alterazioni genomiche in questi mutanti soppressori che sono state acquisite da mutazioni spontanee hanno dimostrato di ripristinare rapidamente l'omeostasi del glutammato9,13. Pertanto, non sorprende che l'adattamento di B. subtilis a una condizione di crescita specifica durante l'addomesticamento del batterio si rispecchi nella sintesi enzimatica e nelle attività enzimatiche evolute, che sono coinvolte nel metabolismo del glutammato12. È stato suggerito che la mancanza di glutammato esogeno nel mezzo di crescita durante il processo di addomesticamento sia stata la forza trainante per l'emergere e la fissazione del gene criptico glutammato deidrogenasi (GDH) gudBCR nel ceppo di laboratorio 1682,14. Questa ipotesi è supportata dalla nostra osservazione che la ridotta quantità di attività GDH nel ceppo di laboratorio fornisce ai batteri un vantaggio di crescita selettivo quando il glutammato esogeno èscarso 2. Inoltre, la coltivazione di un ceppo di B. subtilis, sintetizzando il GDH GudB, in assenza di glutammato esogeno provoca l'accumulo di mutanti soppressori che hanno inattivato il gene gudB 2. Ovviamente, la presenza di un GDH catabolicamente attivo è svantaggiosa per la cellula perché il glutammato prodotto endogenamente che potrebbe altrimenti essere utilizzato per l'anabolismo è degradato all'ammonio e al 2-ossoglutarato (Figura 1). Al contrario, quando il glutammato è fornito dal mezzo, un ceppo B. subtilis dotato di attività GDH di alto livello ha un vantaggio di crescita selettivo rispetto a un ceppo che sintetizza un solo GDH funzionale. È ragionevole supporre che l'attività gdh di alto livello consenta ai batteri di utilizzare il glutammato come seconda fonte di carbonio oltre ad altre fonti di carbonio fornite dalmezzo 2 (vedi figura 1). Pertanto, l'attività GDH influisce fortemente sulla forma fisica dei batteri, a seconda della disponibilità di glutammato esogeno.
Qui presentiamo un metodo molto illustrativo per monitorare e visualizzare la competizione intraspecie tra due ceppi di B. subtilis che differiscono in un singolo locus sul cromosoma (Figura 2). I due ceppi sono stati etichettati con i geni yfp e cfp che codificano i fluorofori YFP e CFP e cocultivati in diverse condizioni nutrizionali. Campionando nel tempo e placcando adeguate diluizioni su piastre di agar, i sopravvissuti in ciascuna delle colture potrebbero essere facilmente monitorati utilizzando un comune microscopio a fluorescenza stereo. La procedura descritta in questo articolo è facile da eseguire e adatta a visualizzare la rapida espansione clonale e l'eliminazione delle mutazioni benefiche e dannose, rispettivamente, in una popolazione cellulare nel tempo.
Protocollo
1. Preparazione di piatti Agar, media culturali, criostock e preculture
- Preparare i mezzi di crescita e i reagenti richiesti (vedi tabella dei materiali e dei reagenti).
- Striature i ceppi di B. subtilis (ad es. BP40 (rocG+ gudBCR amyE::PgudB-yfp) e BP52 (rocG+ gudB+ amyE::PgudB-cfp) esprimendo rispettivamente uno e due GDH attivi)2 che saranno utilizzati nell'esperimento di competizione su piastre di agar medie SP per ottenere singole colonie. Incubare le piastre durante la notte a 37 °C.
- Assumere singole colonie e inoculare tubi di coltura sterile contenenti 4 ml di mezzo liquido LB. Far crescere i batteri durante la notte a 28 °C e 220 giri/min.
- Coltivare colture notturne a 28 °C per evitare che le cellule si lecivano o spopolano.
- Misurare la densità ottica delle colture ad una lunghezza d'onda di 600 nm (OD600) utilizzando uno spettrofotometro standard.
- Se le colture hanno raggiunto un OD600 di 2.0, mescolare 0,75 ml delle colture con 0,75 ml di una soluzione sterile di glicerolo al 50% in tubi di reazione da 1,5 ml. L'OD600 finale deve essere 1.0 per ottenere criostock contenenti circa 108 cellule/ml.
- Conservare i tubi a -80 °C.
- Creare tre preculture di ogni ceppo etichettate con i geni cfp e yfp che codificano i geni del fluoroforo. Inoculare le precolture (tubi di coltura sterile contenenti 4 ml di mezzo liquido LB) con 1 μl di cellule da criostock a -80 °C. Incubare le colture durante la notte a 28 °C e 220 giri/min.
2. Cocultivazione di batteri, raccolta di campioni e stoccaggio dei campioni
- Preparare al minimo 100 ml di C-Glc e CE-Glc (vedi tabella dei reagenti e dei materiali) e trasferire 20 ml di ogni mezzo in contenitori sterili da 100 ml di frullato.
- Assumere 0,1 ml delle preculture coltivate durante la notte, diluirle con un mezzo LB da 0,9 ml in una cuvetta da 1,5 ml e determinare l'OD600.
- Per l'esperimento di competizione, prendere quelle preculture dei diversi ceppi che hanno un OD600 simile tra 1.0-1.5.
- Per ottenere popolazioni di cellule miste, diluire le cellule delle precolture che avevano l'OD600 appropriato a un OD600 di 0,05 in 20 ml di C-Glc e ce-Glc minimo mezzo integrato in flaconi di frullato da 100 ml. Per l'esperimento di competizione i due ceppi dovrebbero essere mescolati in un rapporto 1:1.
- Prelevare campioni da 10 ml dai contenitori, trasferirli in tubi di plastica da 15 ml, raccogliere le cellule per centrifugazione per 10 min a 4.000 x g in una centrifuga da tavolo standard e scartare il supernatante.
- Rimorsi le cellule in un mezzo LB fresco da 0,5 ml e trasferire le cellule in una tazza di reazione sterile da 1,5 ml. Aggiungere 0,5 ml di glicerolo sterile al 50%, mescolare la sospensione con un vortice rigoroso e conservare i campioni in un congelatore a -80 °C fino a un ulteriore trattamento.
- Incubare le cellule che vengono lasciate nei contenitori di scuotimento per un massimo di 24 ore a 37 °C e 220 giri/min. Tenere i mastri di scuotimento al buio per evitare lo sbiancamento fotografico dei fluorofori.
- Prelevare ulteriori campioni da 0,1 ml dopo 7 ore e 24 ore di crescita. Misurare e notare l'OD600 di diluizioni 1:10 (in C-Glc o CE-Glc mezzo minimo) dei campioni.
- Prendi la quantità appropriata di cellule da ogni coltura per creare criostock che hanno un OD600 di 1,0. Conservare i campioni a -80 °C fino a un ulteriore trattamento.
3. Trattamento del campione, placcatura e incubazione per analisi quantitative
- Dopo aver raccolto tutti i campioni, scongelare i criostock e diluire le cellule in una soluzione salina allo 0,9% (vedi tabella dei reagenti e dei materiali) fino a10-3.
- Piastra 0,1 ml delle10-3 diluizioni su piastre di agar medie SP e distribuire le celle utilizzando pipette di vetro sterili.
- Incubare le piastre durante la notte a 37 °C al buio fino a quando non sono apparse singole colonie.
4. Conteggio dei sopravvissuti mediante microscopia a fluorescenza stereo per l'analisi quantitativa
- Dividi il fondo della piastra dell'agar con una penna nera in quattro parti per un migliore orientamento mentre conti i sopravvissuti sotto il microscopio a fluorescenza stereo.
- Posizionare la piastra capovolta al microscopio e mettere a fuoco le colonie utilizzando la fonte di luce fredda.
- Una volta che le colonie sono a fuoco, passare al set di filtri appropriato per la PCP per visualizzare le cellule sopravvissute del ceppoetichettato cfp. Conta i sopravvissuti di questo ceppo etichettando le colonie con una penna e annota il numero.
- Rimuovere le etichette con etanolo e passare al filtro YFP impostato per visualizzare le cellule sopravvissute del ceppo etichettato yfp. Conta di nuovo i sopravvissuti etichettando le colonie con una penna e annota il numero.
5. Trattamento del campione e microscopia per analisi semiquantitative
- Per cifre illustrative, individuare 10 μl della diluizione10-4 (circa 100 celle) dal passaggio 3.1 su una piastra di agar SP (vedere figura 3).
- Incubare le piastre durante la notte a 37 °C al buio fino a quando non sono apparse singole colonie.
- Posizionare la piastra di Petri senza coperchio al microscopio e mettere a fuoco il punto utilizzando la fonte di luce fredda.
- Scatta foto dei punti per figure illustrative. Scegli un tempo di esposizione appropriato per scattare le foto delle colonie con la fonte di luce fredda.
- Senza spostare la piastra, passare al set di filtri CFP, regolare il tempo di esposizione e scattare una foto. Fai lo stesso con il set di filtri YFP e salva le immagini per ulteriori analisi.
6. Analisi dei dati
- Utilizzare software come Excel per le analisi quantitative dei dati. In base alle colonie contate, calcola la percentuale di colonie gialle e blu rispetto all'intero numero di colonie, che sono impostate al 100%.
- Utilizzare i numeri calcolati per creare un diagramma a barre in pila (vedere figura 4 e figura 5). Utilizzate un programma di elaborazione delle immagini come Adobe Photoshop per costruire immagini unite delle immagini scattate dai diversi punti. In alternativa, il software liberamente disponibile ImageJ, scaricabile da http://rsbweb.nih.gov/ij/ può essere utilizzato per l'elaborazione delle immagini.
- Aprire le immagini da un punto scattate con il set di filtri CFP e il set di filtri YFP. Ottimizza il contrasto e la luminosità per ridurre la fluorescenza di fondo dal supporto.
- Passare a una delle immagini e selezionare tutte. Copiare l'immagine e incollarla nell'altra immagine.
- Utilizzare la funzione "schivare il colore" per unire le immagini o sovrapporre le immagini fluorescenti utilizzando la scheda canali. Immagini unite delle colonie da diversi media e diversi punti di tempo rappresentano la crescita dei diversi ceppi all'interno della coltura liquida.
7. Suggerimenti specifici: esperimento di commutazione di colorante e cocultivazione di ceppi isogenici etichettati con cfp e yfp
L'espressione di uno dei due geni codificanti al fluoroforo in B. subtilis potrebbe influenzare la forma fisica e quindi il tasso di crescita dei batteri. Pertanto, si raccomanda di eseguire i seguenti esperimenti al fine di escludere che l'eliminazione di un ceppo concorrente dalla popolazione cellulare durante la coltivazione sia semplicemente dovuta a un effetto negativo del fluoroforo:
- Ripetere l'intero esperimento e cocultivare ceppi etichettati inversamente (ad esempio, il ceppo precedente etichettato cfpè ora etichettato con il gene yfp e viceversa). Sebbene inversi, i risultati ottenuti dovrebbero essere paragonabili all'osservazione precedente secondo cui un ceppo ha un vantaggio di crescita selettivo rispetto all'altro ceppo.
- Ripetere l'intero esperimento e cocultivare i ceppi isogenicietichettati con yfp e cfp ( ad esempio BP40 (rocG+ gudBCR amyE::PgudB-yfp) e BP41 (rocG+ gudBCR amyE::PgudB-cfp)). Stimare l'effetto negativo di uno dei due fluorofori sulla forma fisica dei batteri seguendo la composizione della popolazione cellulare nel tempo.
Risultati
Il metodo qui descritto è stato applicato con successo per visualizzare la concorrenza intraspecie in una popolazione cellulare costituita da ceppi di B. subtilis etichettati rispettivamente con i geni cfp e yfp che codificano i fluorofori CFP e YFP. Come illustrato nella figura 3, il metodo può essere utilizzato per visualizzare la concorrenza intraspecie in modo molto illustrativo. Individuando i campioni su piccole aree, la composizione clonale della popolazione cellulare è stata resa visibile a colpo d'occhio. Sebbene non sia appropriato per le analisi quantitative, questo approccio è utile per stimare approssimativamentel'effetto dei diversi parametri di crescita (cioè la fonte diazoto) sullo sviluppo di una popolazione cellulare che inizialmente conteneva entrambi i ceppi in quantità uguali(figura 3). Inoltre, in un approccio su piccola scala l'idoneità di diversi ceppi di B. subtilis coltivati nelle stesse condizioni di crescita può essere testata utilizzando un'unica piastra di agar. Per le analisi quantitative si raccomanda di propagare i campioni su tutta la superficie di una piastra di agar. Ciò impedirà la sovrapposizione delle colonie e quindi consentirà l'identificazione distinta e il conteggio delle colonie emerse da singole cellule. Placcando adeguate diluizioni sulle placche di agar, la composizione clonale di una popolazione cellulare nel tempo può essere determinata con precisione semplicemente contando le colonie fluorescenti gialle e blu (vedi figura 4). Come abbiamo precedentemente riferito, l'attività GDH influisce fortemente sulla forma fisica di B. subtilis a seconda della disponibilità di glutammato esogeno2. Ovviamente, in assenza di glutammato esogeno, l'attività gdh di alto livello è svantaggiosa per i batteri in quanto gli enzimi RocG e GudB degradano il glutammato necessario nell'anabolismo (vedi figura 1 e figura 4A). Al contrario, se fornito ai batteri, il glutammato può servire come donatore di amminogruppo nelle reazioni di transaminazione. Inoltre, il glutammato può essere immesso nel metabolismo del carbonio e utilizzato come fonte di energia a causa della presenza dei GDHs RocG e GudB catabolicamente attivi(figura 1 e figura 4B). Come mostrato nelle figure 4C e 4D, risultati simili sono stati ottenuti in un esperimento di interruttore di tintura. Ancora una volta, i batteri dotati di attività GDH di alto livello sono stati sovracompetati da cellule con ridotta attività GDH nei mezzi di crescita privi di glutammato. Al contrario, i batteri che sintetizzano un solo GDH attivo sono stati eliminati dalla coltura quando il mezzo è stato integrato con glutammato. Come mostrato nelle figure 5A e 5B, la composizione iniziale della popolazione di cellule miste è rimasta quasi costante nel tempo. Pertanto, nell'esperimento di concorrenza l'eliminazione di uno dei due ceppi che erano dotati di diverse quantità di attività GDH non era dovuta a un difetto di crescita causato dai fluorofori (cfr. figura 4). Nel complesso, l'uso di fluorofori è un potente strumento per monitorare la concorrenza intraspecie in una popolazione di cellule batteriche.

Figura 1. Il legame tra il metabolismo del carbonio e quello dell'azoto in B. subtilis. Quando il glutammato non è fornito dal mezzo, il principale amminodonante necessario per l'anabolismo viene sintetizzato dall'ammonio e 2-ossoglutarato dall'azione combinata della glutammina sintetasi (GS) e del glutammato sintasi (GOGAT). Al contrario, in presenza di glutammato esogeno, i GDHs catabolicamente attivi RocG e/o GudB possono degradare il glutammato in ammonio e 2-ossoglutarato, che poi funge da fonte di carbonio.
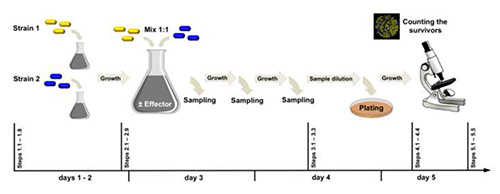
Figura 2. Flusso di lavoro sperimentale. Il ceppo 1 (etichettato con yfp) e il ceppo 2 (etichettato con cfp)differiscono in un locus l'uno dall'altro. Nell'esempio qui presentato, abbiamo confrontato l'effetto del glutammato esogeno (effettore) sullo spostamento genotipico della popolazione cellulare che inizialmente conteneva il 50% di rocG+ gudB+ (codificando due GDH attivi) e il 50% delle cellule rocG+ gudBCR (codificando un GDH attivo). Clicca qui per visualizzare l'immagine più grande.

Figura 3. Approccio semiquantitativo per visualizzare la concorrenza intraspecie in modo descrittivo (cfr. sezione 5). Prima della cocultivazione (0 ore) e dopo 7 ore e 24 ore di diluizioni di crescita(10-4)di cellule sono state avvistate su piastre di agar SP. Le cellule sopravvissute che hanno formato colonie dopo 12 ore di incubazione a 37 °C sono state identificate dalla microscopia a fluorescenza stereo. Tempo di esposizione, 0,6 sec; barra di scala, 1 mm. Questa cifra è stata modificata da Gunka et al. 20132.

Figura 4. Quantificazione della concorrenza intraspecie. Dopo la diluizione del campione e la propagazione delle cellule (vedere i passaggi 3.1-3.3) su SP medium le piastre sono state incubate durante la notte a 37 °C. Le colonie gialle e blu sono state quantificate come descritto nei protocolli 4 e 6. Le barre di errore nere rappresentano deviazioni standard per almeno quattro esperimenti ripetuti indipendentemente. Ogni piatto di agar conteneva almeno 100 colonie contate. (A) In assenza di glutammato esogeno, il ceppo B. subtilis BP40 (giallo) dotato di un solo GDH funzionale sovracompeti il ceppo BP54 (blu), che sintetizza sia gli enzimi degradanti al glutammato, RocG e GudB. (B) Al contrario, la sintesi di due GDH funzionali è vantaggiosa per i batteri quando è disponibile glutammato esogeno perché, oltre al glucosio, il glutammato viene utilizzato come fonte di carbonio. Come mostrato nelle ( C )e( D ),risultaticomparabili sono stati ottenuti in un esperimento di interruttore di colorante. Questa cifra è stata modificata da Gunka et al. 20132. Clicca qui per visualizzare l'immagine più grande.

Figura 5. Esperimento di controllo per valutare l'effetto dei geni cfp e yfp di codifica fluoroforica sulla forma fisica dei batteri. Popolazioni miste dei ceppi isogenici BP40 (rocG+ gudBCR amyE::yfp) e BP41 (rocG+ gudBCR amyE::cfp) o BP52 (rocG+ gu dB+ amyE::cfp) e BP156 (rocG+ gudB+ amyE::yfp) sono stati coltivati in assenza (A) e in presenza (B) di glutammato esogeno. Le cellule sopravvissute sono state contate come descritto rispettivamente nei protocolli 1-4 e 6. Le barre rappresentano deviazioni standard per almeno quattro esperimenti ripetuti indipendentemente. Questa cifra è stata modificata da Gunka et al. 20132. Clicca qui per visualizzare l'immagine più grande.
Discussione
Diversi metodi sono stati sviluppati per analizzare la forma fisica competitiva deibatteri 16. In molti casi i batteri sono stati etichettati con diverse cassette di resistenza agli antibiotici17. Simile al nostro approccio, l'etichettatura delle cellule con cassette di resistenza agli antibiotici consente la valutazione della forma fisica competitiva dei batteri durante la cocultivazione in condizioni di crescita definite. Inoltre, questo metodo può essere utilizzato per determinare la forma fisica competitiva delle cellule che differiscono l'una dall'altra in un locus specifico sulcromosoma 17. Tuttavia, ci sono alcuni svantaggi che utilizzano cassette di resistenza agli antibiotici per monitorare la forma fisica competitiva. Poiché l'espressione dei geni di resistenza è per lo più guidata da promotori con una forza iniqua, gli enzimi che conferiscono resistenza agli antibiotici sono probabilmente prodotti a diversi livelli. Pertanto, gli effetti deboli della forma fisica potrebbero non essere rilevabili con questo approccio. Nel nostro approccio, entrambi i geni fluorofori sono stati integrati con lo stesso marcatore di selezione e la loro espressione è guidata dagli stessi promotori2. Un altro svantaggio per monitorare l'idoneità competitiva con cassette di resistenza agli antibiotici potrebbe essere che l'approccio è più laborioso in quanto sono necessari due tipi di piastre di agar integrate con gli antibiotici appropriati per il conteggio della colonia. In alternativa, l'idoneità dei batteri può essere determinata semplicemente monitorando il tasso di crescita e il calcolo di un cosiddetto indice di forma fisica16. Ovviamente, questo è l'approccio più preciso perché i batteri sono coltivati individualmente e i composti tossici che potrebbero essere prodotti da un ceppo con un certo genotipo non influenzeranno la crescita del ceppo concorrente. Inoltre, non è necessario utilizzare antibiotici che potrebbero influire sulla crescita delle cellule. Tuttavia, entrambi gli approcci non sono molto illustrativi in quanto i numeri che descrivono l'idoneità competitiva possono essere presentati solo in modo piuttosto neutro.
L'uso di geni codificanti a fluoroforo per monitorare e quantificare la concorrenza intraspecie presenta diversi vantaggi rispetto ad altri metodi. Se entrambi i ceppi hanno integrato i geni codificanti del fluoroforo mediante doppia ricombinazione omologa nel cromosoma, non è necessario utilizzare antibiotici in nessuna delle fasi di coltivazione. Pertanto, i campioni prelevati dalla coltura durante la coltivazione possono essere analizzati sullo stesso mezzo di crescita ed entrambi i ceppi sono in grado di crescere. Questo approccio consente la visualizzazione della competizione intraspecie in modo molto illustrativo. Inoltre, utilizzando questo approccio semiquantitativo, è possibile testare diverse condizioni di crescita contemporaneamente e molti ceppi diversi possono essere confrontati in parallelo. Infine, non è necessario fissare i campioni su vetrini al microscopio perché i campioni prelevati dalla coltura batterica durante la cocultivazione possono essere conservati in un congelatore18. Pertanto, tutti i campioni e le repliche possono essere analizzati contemporaneamente.
Occorre fare due passi fondamentali nel nostro protocollo. È importante notare che i criostock dovrebbero contenere quantità uguali di cellule nella stessa fase di crescita di ogni ceppo concorrente. Una sproporzione iniziale dei ceppi nei criostock e di conseguenza nei contenitori di scuotimento prima di iniziare l'esperimento avrà un forte impatto sull'esito dell'esperimento di competizione. Pertanto, è consigliabile controllare la composizione dei criostock prima dell'esperimento. Inoltre, una quantità appropriata di cellule dovrebbe essere propagata uniformemente sulla piastra. Altrimenti le colonie emerse sono troppo vicine l'una all'altra e una determinazione precisa delle cellule sopravvissute diventerà difficile.
Ci sono anche alcune limitazioni e svantaggi dell'approccio basato sul fluoroforo. La cocultivazione in un lettore di lastre multi-pozzo e l'individuazione simultanea dei segnali CFP e YFP non è possibile in quanto gli spettri di eccitazione ed emissione dei fluorofori sono troppo vicini l'uno all'altro. Tuttavia, questo problema tecnico potrebbe essere aggirato utilizzando diverse proteine fluorescenti, come quelle che emettono luce verde e rossa, in modo ottimale sulla base della stessa impalcatura proteica. Un altro inconveniente dell'approccio basato sul fluoroforo potrebbe essere che alcune mutazioni causano solo un debole difetto di crescita dei batteri. Pertanto, se il difetto di crescita che potrebbe essere causato da uno dei geni codificanti al fluoroforo è più forte del difetto di crescita causato da una certa mutazione, l'approccio basato sul fluoroforo non è appropriato per analizzare la competizione intraspecie. Pertanto, prima di creare un intero set di ceppi si consiglia di etichettare prima solo il ceppo genitore con entrambi, cfp e yfp, e di cocultivare i ceppi. Gli esperimenti di crescita riveleranno quanto forti siano i fluorofori che influenzano la forma fisica dei batteri (vedi figure 5A e 5B).
In futuro sarà interessante verificare se l'approccio basato sul fluoroforo per monitorare la concorrenza intraspecie sarà più accurato e meno laborioso se le cellule sopravvissute saranno conteggiate utilizzando la citometria a flusso. Recentemente, la citometria a flusso ha dimostrato di essere un potente strumento per analizzare la composizione dei biofilm B. subtilis 19. Inoltre, potrebbe essere più appropriato analizzare i deboli effetti di forma fisica che influenzano la forma fisica competitiva dei batteri mediante cocultivazione continua dei batteri etichettati al fluoroforo in un fermentatore. A differenza dei fusti di scuotizione, questo approccio consente di mantenere costanti le condizioni di crescita e quindi di monitorare la concorrenza intraspecie per un lungo periodo di tempo.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Il lavoro nel laboratorio degli autori è stato sostenuto dalla Deutsche Forschungsgemeinschaft (http://www.dfg.de; CO 1139/1-1), il Fonds der Chemischen Industrie (http://www.vci.de/fonds) e il Centro di Biologia Molecolare di Gottinga (GZMB). Gli autori vorrebbero riconoscere Jörg Stülke per i commenti utili e la lettura critica del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| (NH4)2SO4 | Roth, Germany | 3746 | |
| Agar | Difco, USA | 214010 | |
| Ammonium ferric citrate (CAF) | Sigma-Aldrich, Germany | 9714 | |
| CaCl2 | Roth, Germany | 5239 | |
| Glucose | Applichem, Germany | A3617 | |
| Glycerol | Roth, Germany | 4043 | |
| K2HPO4•3H2O | Roth, Germany | 6878 | |
| KCl | Applichem, Germany | A3582 | |
| KH2PO4 | Roth, Germany | 3904 | |
| KOH | Roth, Germany | 6751 | |
| MgSO4•7H2O | Roth, Germany | P027 | |
| MnCl2 | Roth, Germany | T881 | |
| MnSO4•4H2O | Merck Millipore, Germany | 102786 | |
| NaCl | Roth, Germany | 9265 | |
| Nutrient broth | Roth, Germany | X929 | |
| Potassium glutamate | Applichem, Germany | A3712 | |
| Tryptone | Roth, Germany | 8952 | |
| Tryptophan | Applichem, Germany | A3445 | |
| Yeast extract | Roth, Germany | 2363 | |
| 1.5 ml Reaction tubes | Sarstedt, Germany | 72,690,001 | |
| 2.0 ml Reaction tubes | Sarstedt, Germany | 72,691 | |
| 15 ml Plastic tubes with screw cap | Sarstedt, Germany | 62,554,001 | |
| Petri dishes | Sarstedt, Germany | 82.1473 | |
| 1.5 ml Polystyrene cuvettes | Sarstedt, Germany | 67,742 | |
| 15 ml Glass culture tubes | Brand, Germany | 7790 22 | |
| 100 ml Shake flasks with aluminium caps | Brand, Germany | 928 24 | |
| Sterile 10 ml glass pipettes | Brand, Germany | 278 23 | |
| Incubator (28 and 37 °C) | New Brunswick | M1282-0012 | |
| Standard pipette set (2-20 μl, 10-100 μl, 100-1,000 μl) | Eppendorf, Germany | 4910 000.034, 4910 000.042, | |
| Table top centrifuge for 1.5 and 2 ml reaction tubes | Thermo Scientific, Heraeus Fresco 21, Germany | 75002425 | |
| Table top centrifuge for 15 ml plastic tubes | Heraeus Biofuge Primo R, Germany | 75005440 | |
| Standard spectrophotometer | Amersham Biosciences Ultrospec 2100 pro, Germany | 80-2112-21 | |
| Stereofluorescence microscope | Zeiss SteREO Lumar V12, Germany | 495008-0009-000 | |
| Freezer (-20 and -80 °C) | - | - | |
| Fridge (4 °C) | - | - | |
| Autoclave | Zirbus, LTA 2x3x4, Germany | - | |
| pH meter | pH-meter 766, Calimatic, Knick, Germany | 766 | |
| Vortex | Vortex 3, IKA, Germany | 3340000 | |
| Balance | CP2202S, Sartorius, Germany | replaced by | |
| Black pen (permanent marker) | Staedler, Germany | 317-9 | |
| Powerpoint program | Microsoft, USA | - | |
| Office Excel program | Microsoft, USA | - | Program for data processing |
| Adobe Photoshop CS5 | Adobe, USA | replaced by CS6, download | Computer program for image processing |
| Computer | PC or Mac | - | |
| ZEN pro 2011 software for the stereofluorescence microscope | Zeiss, Germany | 410135 1002 110 | AxioCam MRc Rev. Obtained through Zeiss |
| Specific solution recipes | |||
| SP Medium | |||
| 8 g Nutrient broth | |||
| 0.25 mg MgSO4•7H2O | |||
| 1 g KCl | |||
| 15 g agar for solid SP medium | if required | ||
| add 1 L with H2O | autoclave for 20 min at 121 °C | ||
| 1 ml CaCl2 (0.5 M), sterilized by filtration | |||
| 1 ml MnCl2 (10 mM), sterilized by filtration | |||
| 2 ml ammonium ferric citrate (CAF, 2.2 mg/ml), sterilized by filtration | |||
| LB Medium | |||
| 10 g Tryptone | |||
| 5 g Yeast extract | |||
| 10 g NaCl | |||
| 15 g agar for solid LB medium | if required | ||
| add 1 L with H2O | autoclave for 20 min at 121 °C | ||
| C-Glc Minimal Medium | |||
| 200 ml 5 x C salts | |||
| 10 ml L-Tryptophan (5 mg/ml), sterilized by filtration | |||
| 10 ml ammonium ferric citrate (CAF, 2.2 mg/ml), sterilized by filtration | |||
| 10 ml III’ salts | |||
| 25 ml Glucose (20%) | autoclaved for 20 min at 121 °C | ||
| add 1 L with sterile H2O | |||
| CE-Glc Minimal Medium | |||
| 200 ml 5 x C salts | |||
| 10 ml L-Tryptophan (5 mg/ml), sterilized by filtration | |||
| 10 ml ammonium ferric citrate (CAF, 2.2 mg/ml), sterilized by filtration | |||
| 10 ml III’ salts | |||
| 20 ml Glutamate (40%) | |||
| 25 ml Glucose (20%), autoclaved for 20 min at 121 °C | |||
| add 1 L with sterile H2O | |||
| 5 x C salts | |||
| 20 g KH2PO4 | |||
| 80 g K2HPO4•3H2O | |||
| 16.5 g (NH4)2SO4 | |||
| add 1 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| III’ salts | |||
| 0.232 g MnSO4•4H2O | |||
| 12.3 g MgSO4•7H2O | |||
| add 1 L with sterile H2O, autoclave for 20 min at 121 °C | |||
| 40% Glutamate solution | |||
| 200 g L-Glutamic acid | |||
| adjust the pH to 7.0 by adding approximately 80 g KOH | |||
| add 0.5 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| 0.9% Saline (NaCl) Solution | |||
| add 1 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| 50% Glycerol solution | |||
| 295 ml Glycerol (87%) | |||
| add 0.5 L with sterile H2O | autoclave for 20 min at 121 °C | ||
| Bacteria (All strains are based on the Bacillus subtilis strain 168) | |||
| Bacillus subtilis BP40 (rocG+ gudBCR amyE::PgudB-yfp) | Laboratory strain collection | ||
| Bacillus subtilis BP41 (rocG+ gudBCR amyE::PgudB-cfp) | |||
| Bacillus subtilis BP52 (rocG+ gudB+ amyE::PgudB-cfp) | |||
| Bacillus subtilis BP156 (rocG+ gudB+ amyE::PgudB-yfp) | |||
Riferimenti
- Buescher, F. I., et al. Global network reorganization during dynamic adaptations of Bacillus subtilis metabolism. Science. 335, 1099-1103 (2012).
- Gunka, K., Stannek, L., Care, R. A., Commichau, F. M. Selection-driven accumulation of suppressor mutants in Bacillus subtilis: the apparent high mutation frequency of the cryptic gudB gene and the rapid clonal expansion of gudB+ suppressors are due to growth under selection. PLoS One. 8, (2013).
- Al Mamum, A. A. M., et al. Identity and function of a large gene network underlying mutagenic repair of DNA breaks. Science. 338, 1344-1348 (2012).
- Koeppel, A. F., Wertheim, J. O., Barone, L., Gentile, N., Krizanc, D., Cohan, F. M. Speedy speciation in a bacterial microcosm: new species can arise as frequently as adaptations within a species. ISME J. 7, 1080-1091 (2013).
- Maughan, H., Nicholson, W. L. Increased fitness and alteration of metabolic pathways during Bacillus subtilis evolution in the laboratory. Appl. Environ. Microbiol. 77, 4105-4118 (2011).
- Burkholder, P. R., Giles, N. H. Induced biochemical mutations in Bacillus subtilis. Am. J. Bot. 34, 345-348 (1947).
- McLoon, A. L., Guttenplan, S. B., Kearns, D. B., Kolter, R., Losick, R. Tracing the domestication of a biofilm-forming bacterium. J. Bacteriol. 193, 2027-2034 (2011).
- Zeigler, D. R., et al. The origins of 168, W23, and other Bacillus subtilis legacy strains. J. Bacteriol. 190, 6983-6995 (2008).
- Gunka, K., Tholen, S., Gerwig, J., Herzberg, C., Stülke, J., Commichau, F. M. A high-frequency mutation in Bacillus subtilis: requirements for the decryptification of the gudB glutamate dehydrogenase. 194, 1036-1044 (2012).
- Commichau, F. M., et al. Characterization of Bacillus subtilis mutants with carbon source-independent glutamate biosynthesis. J. Mol. Microbiol. Biotechnol. 12, 106-113 (2007).
- Beckwith, J. Genetic suppressors and recovery of repressed biochemical memory. J. Biol. Chem. 284, 12585-12592 (2009).
- Gunka, K., Commichau, F. M. Control of glutamate homeostasis in Bacillus subtilis: a complex interplay between ammonium assimilation, glutamate biosynthesis and degradation. Mol. Microbiol. 85, 213-224 (2012).
- Yan, D. Protection of the glutamate pool concentrations in enteric bacteria. Proc. Natl. Acad. Sci. U.S.A. 104, 9475-9480 (2007).
- Belitsky, B. R., Sonenshein, A. L. Role and regulation of Bacillus subtilis glutamate dehydrogenase genes. J. Bacteriol. 180, 6298-6305 (1998).
- Commichau, F. M., Herzberg, C., Tripal, P., Valerius, O., Stülke, J. A regulatory protein-protein interaction governs glutamate biosynthesis in Bacillus subtilis: the glutamate dehydrogenase RocG moonlights in controlling the transcription factor GltC. Mol. Microbiol. 65, 642-654 (2007).
- Gordo, I., Perfeito, L., Sousa, A. Fitness effects of mutations in bacteria. J. Mol. Microbiol. Biotechnol. 21, 20-35 (2011).
- Rabatinová, A., et al. The δ subunit of RNA polymerase is required for rapid changes in gene expression and competitive fitness of the cell. J. Bacteriol. 195, 2603-2611 (2013).
- Capra, E. J., Perchuk, B. S., Skerker, J. M., Laub, M. T. Adaptive mutations that prevent crosstalk enable the expansion of paralogous signalling protein families. Cell. 150, 222-232 (2012).
- García-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J. Vis. Exp. (15), (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon