Method Article
Um
Neste Artigo
Resumo
Nós desenvolvemos uma In vitro Malária-HIV-1 modelo de co-infecção para estudar o impacto da Plasmodium falciparum Sobre o ciclo replicativo do HIV-1 em humanos primários macrófagos derivados de monócitos. Este sistema versátil pode ser facilmente adaptado a outros tipos de células primárias sensíveis à infecção HIV-1.
Resumo
Plasmodium falciparum, o agente causador da forma mais letal de malária, e vírus da imunodeficiência humana tipo 1 (HIV-1) estão entre os problemas de saúde mais importantes a nível mundial, sendo responsável por um total de 4 milhões de mortes anualmente 1. Devido às suas extensas sobreposição nas regiões em desenvolvimento, especialmente da África Subsariana, co-infecções com malária e HIV-1 são comuns, mas a interação entre as duas doenças é mal compreendido. Relatórios epidemiológicos sugerem que a infecção por malária transientemente aumenta a replicação do HIV-1 e aumenta a carga de HIV-1 viral em indivíduos co-infectados 2,3. Porque este viremia permanece elevada durante várias semanas após o tratamento com anti-maláricos, este fenómeno pode ter um impacto na progressão da doença e de transmissão.
Os mecanismos imunológicos celulares por trás dessas observações foram estudadas apenas mal. Os poucos estudos in vitro Investigção de impacto da malária em HIV-1 demonstraram que a exposição a antigénios solúveis malária pode aumentar infecção HIV-1 e reactivação em células imunes. No entanto, estes estudos utilizaram extratos de células inteiras de P. falciparum esquizonte parasitas palco e células mononucleares do sangue periférico (PBMC), tornando-o difícil de decifrar qual componente da malária (s) foi responsável pelos efeitos observados e que as células hospedeiras alvo eram 4,5. Um trabalho recente demonstrou que a exposição de células imaturas derivados de monócitos dendríticas para a malária pigmento hemozoin aumentaram a sua capacidade de transferir o HIV-1 a células T CD4 + 6,7, mas que diminuiu o HIV-1 infecção de macrófagos 8. Para lançar luz sobre este processo complexo, uma análise sistemática das interações entre o parasita da malária e HIV-1 em diferentes populações de células humanas relevantes primário é extremamente necessárias.
Várias técnicas para investigar o impacto de HIV-1 sobre a fagocitose de microrganismos eo efeito de agentes patogénicos tais sobre a replicação do HIV-1 têm sido descritos. Nós aqui apresentar um método para investigar os efeitos de P. falciparum infectadas eritrócitos sobre a replicação do HIV-1 em humanos primários macrófagos derivados de monócitos. O impacto da exposição parasita sobre o HIV-1 transcricionais / translacional eventos é monitorizada usando vírus de ciclo único pseudotyped em que um gene repórter da luciferase substituiu o gene Env enquanto que o efeito sobre a quantidade de vírus libertado pelos macrófagos infectados é determinada através da medição do HIV-1 de proteína da cápside p24 por ELISA em sobrenadantes de células.
Protocolo
Nota: Experimentos com HIV-1 e Plasmodium falciparum deve ser realizada no próprio nível de biossegurança para laboratórios (BSL2 de parasitas, e BSL3 para HIV-1 e co-infecções) e as precauções especiais devem ser tomados ao usar potencialmente infectados sangue humano.
1. As células mononucleares do sangue periférico Purificação (PBMC) (baseado em 8 e 9)
- Começar com sangue humano fresco a partir de dadores saudáveis (500 ml) tratados com anticoagulantes (heparina, citrato, citrato ácido dextrose ou citrato de fosfato de dextrose). Use-endotoxina livres reagentes quando purificação e isolamento de PBMC e em todo o resto do protocolo.
- Desinfectar o saco de sangue, incluindo o tubo, com etanol a 70%, cortar o tubo e transferir cuidadosamente o sangue para um balão T150.
- Prepare 16 tubos com 15 ml de meio Linfócitos Separação (Ficoll) por 50 tubo ml (certifique-se que Ficoll atingiu a temperatura ambiente).Cuidadosamente ml camada 30 de sangue fresco em cima.
- Centrifugar a 400 xg durante 30 min a 20 ° C com intervalos de folga.
- Durante a centrifugação, preparar 8 x tubos de 50 ml cónicos com 25 ml de solução à temperatura ambiente Salina Equilibrada de Hank (HBSS) por tubo.
- Transferir cuidadosamente o anel de célula turvo na interface (contém PBMC) para o tubo de 50 ml contendo o HBSS (anéis de piscina 2 celulares por tubo). Completar volumes de até 50 ml com HBSS.
- Centrifugar a 150 xg durante 15 min a 20 ° C com quebras na.
- Durante a centrifugação do anel célula nublado, recolher a camada superior amarela a partir do passo Ficoll, o pool de plasma para encher um tubo cónico de 50 ml. Inactivar o complemento no plasma por meio de aquecimento do tubo de 50 ml a 56 ° C durante 30 min e girar 10 min a 1.800 x g. Manter o sobrenadante (contém plasma autólogo diluída que pode ser utilizado como suplemento de meio em passos posteriores).
- Cuidadosamente remover o sobrenadante e resuspend o pellet celular a partir do passo 1.7 em 25 ml de HBSS e piscina 2 pelotas por 50 ml de tubo. Girar a 300 xg durante 8 minutos a 20 ° C.
- Remover o sobrenadante cuidadosamente e ressuspender as pelotas em 10 ml de HBSS e piscina em um tubo de 50 ml.
- Tome uma alíquota, executar trypan exclusão azul para avaliar a mortalidade e, em seguida, contar PBMC.
- Volume completo a 50 ml com HBSS e min de centrifugação 10 a 300 x g. Remover o sobrenadante e ressuspender as células em meio RPMI 1640 a 5x10 6 células / ml (pode-se obter cerca de 400-500 x 10 6 PBMC de 500 ml de sangue).
2. Monócitos-macrófagos derivados de Diferenciação (MDM) (baseado em 8 e 9)
- Semente de aproximadamente 1,25 x 10 8 PBMC em 15 pratos cm com RPMI 1640 (25 ml prato /).
- Deixe células aderir durante 1-2 horas a 37 ° C com 5% de atmosfera de CO2.
- Transferência não aderidas células num balão de novo e lavar a placa com 15 ml de endotoxina livre de PBS duas vezes (5min, 300 xg). As células aderentes são monócitos isolados.
- Para permitir que os monócitos isolados de fresco para se diferenciarem em MDM, adicionar 20 ml de meio RPMI 1640 suplementado com 5% de plasma autólogo (do passo 1.8). Adicionar o factor Colony humana recombinante Macrófago Estimulante (M-CSF, 25 ng / ml) e penicilina / estreptomicina solução (PS, 100 U / ml de penicilina, 100 ug / ml de estreptomicina). Incubar durante 2 dias, troca de meio e incubar 2-3 dias adicionais a 37 ° C em 5% de CO 2 atmosfera.
- Remover meio velho e lave com endotoxina livre PBS vez, adicione o PBS suavemente e aspirar imediatamente. Para separar MDM totalmente diferenciados, tratar as células com ~ 7 ml Accutase (Accutase, uma mistura comercialmente disponível de proteases e da actividade colagenolítica que permitem a separação de células a partir do prato de plástico) para 15-20 min a 37 ° C em um 5% de CO 2 atmosfera, balançar suavemente no meio-tempo.
- Adicionar 3-5 ml de plasma autólogo (do passo 1,8) em cada placa (para ajudar a proteger as células, enquantoraspagem).
- Raspe células certificando-se que o meio abrange as células em todos os tempos e de transferência / piscina em um tubo de 50 ml.
- Lavar pratos com endotoxina livre de PBS para recuperar tantas células quanto possível.
- Girar 5 min a 200 xg, elimine o sobrenadante.
- Ressuspender o sedimento em 5 ml de MDM meio de cultura (RPMI 1640 suplementado com 10% de complemento de soro fetal de bovino inactivado (OIF), PS, HEPES 25 mM) e contagem de células (MDM produzir geralmente 2-8% de PBMC total). Nota: uma alíquota de MDM podem ser tomadas este passo para avaliar a pureza população de células por citometria de fluxo, geralmente 99% do obtido MDM expressa CD14.
- Semente 1x10 5 MDM em 600 uL de meio de cultura MDM por poço em placas de 24 poços.
- Incubar durante a noite a 37 ° C em uma atmosfera de 5% de CO 2 antes de infecções.
3. Produção e determinação quantitativa de HIV-1 reservas virais
- Produzir stocks virais utilizando transfecção com fosfato de cálcio em células HEK293T seguinprotocolo a asa da Fortin et al. 10.
- A fim de obter os vírus de ciclo único contendo o gene repórter da luciferase de codificação, células co-transfectar HEK293T quer com ug de 20 NL4.3 Luc + Env - R + e 10 ug de pHCMV-G, ou 15 ug de NL4.3 Luc + Env - R + e 15 mg de pJRFL env. Use 30 mg de NL4.3 Luc + Env - R + para gerar controle glicoproteína deficiente vírus. Para o vírus replicativa totalmente, utilize 30 ug de NL4.3Bal env. Os vectores pHCMV-G e pJRFL env codifica as glicoproteínas do envelope do vírus da estomatite vesicular (VSV-G) e de um CXCR5-trópico HIV-1 (diferente do que NL4.3Bal env), respectivamente. Informação adicional sobre os plasmídeos podem ser obtidos a partir de 8. Os plasmídeos podem ser obtidos através do AIDS Research NIH e Programa de Referência (NIAID, NIH).
- Quantificar todas as reservas virais usando ELISA para a proteína HIV-1 p24 da cápside 11, E verificar o seu potencial infeccioso antes de usar. Nós avaliar a infectividade dos estoques de vírus nossos através da utilização de células TZM-bl indicador 12.
4. Cultura de parasitas Plasmodium falciparum (Baseado em 13)
4,1 cultura geral e manutenção de parasitas
- Manter P. falciparum 3D7 assexuadas parasitas fase em eritrócitos humanos (o grupo sanguíneo O +) em um hematócrito de 4% em meio RPMI 1640 (tamponado com HEPES 25 mM e 25 mM de NaHCO 3) suplementado com 0,5% (w / v) Albumax II, 25 ug / ml de gentamicina e 370μM Hipoxantina a 37 ° C em uma mistura gasosa de oxigénio de 1% / dióxido de carbono a 5% em azoto.
- Parasitemia pode ser monitorizado através de um esfregaço fino da cultura e da coloração com uma solução Giemsa a 15%.
- Sempre manter uma não infectadas células vermelhas do sangue (uRBC) prato de cultura, nas mesmas condições como parasitas para usar como um controlo.
Para obter Parasit fase tardiaes (trofozoítos / esquizontes primeiros), sincronizar os parasitas, como segue:
4,2 sincronização Parasite
- Na manhã seguinte, colher a cultura parasita, min de centrifugação a 300 xg 5 e descartar o sobrenadante.
- Ressuspender o sedimento em 5 volumes de células embaladas com 5% (w / v) de D-sorbitol e incubar 5 minutos a 37 ° C.
- Gire 5 min a 300 x g, elimine o sobrenadante, ressuspender o pellet em 30 ml meio de cultura e colocar de volta na incubadora.
- Cerca de 8 horas mais tarde, as etapas de repetição 4.2.1 a 4.2.3, a fim de obter parasitas firmemente sincronizados. Parasitas fase tardia (trofozoítos / esquizontes precoces) pode então ser colhida na manhã seguinte para realizar a experiência. Antes da purificação, realização da coloração Giemsa para confirmar fase do ciclo de vida.
4.3 Plasmodium falciparum infectados glóbulos vermelhos (iRBC) purificação
- Esterilizar o separador VarioMacs (stand e imã) com etanol 70% e prendas que sob a capa biológica.
- Fixe a torneira de três vias para CS parte inferior da coluna.
- Cortar o final do protector de plástico da agulha com o cortador Miltenyi especial e fixar a agulha para o fundo torneira.
- Bloquear a coluna no ímã com cuidado.
- Remover todas as bolhas de ar na coluna por injecção lentamente 10 ml de MDM meio de cultura com a seringa kit fechado aparafusado no lado esquerdo da torneira.
- Lavar a coluna com 20 ~ meio de cultura ml MDM.
- Colheita RBC da placa de cultura, lavar uma vez com 10 ml médio MDM (300 xg, 5 min) e ressuspender o pellet em 12 RBC meio de cultura MDM ml. Lentamente, adicionar à coluna e permitir o fluxo de suspensão através de (apenas infectadas células vermelhas do sangue contendo parasitas no trofozoíto ou estágio esquizonte irá ser retido pelo campo magnético devido à presença de cristais de hemozoin).
- Lavar uma vez com 20 ml de meio e parar o fluxo de líquido quando ele atinge o disco branco no topo da coluna.
- Remover a coluna do íman e iRBC eluir em um tubo limpo estéril com 12 ml de MDM meio de cultura.
- Alíquota Smear um do iRBC recuperado e realizar Giemsa coloração para confirmar etapa do ciclo de vida.
- Contar iRBC recuperado usando um hemocitómetro (isolado RBC são iRBC 100%).
- As células da pelota (300 xg, 5 min), elimine o sobrenadante e tratar as células com 500 uL AB frescos + soro humano (passo opsonisation 14).
- Incubar durante 30 min a 37 ° C em uma atmosfera de 5% de CO 2.
- Lavar uma vez com 10 ml de MDM meio de cultura (300 x g, 5 min), e ressuspender iRBC a concentração desejada. Geralmente, um 10% de parasitemia 15 prato de cultura centímetros aproximadamente dá ~ 0,5-1x10 8 iRBC firmemente sincronizado.
5. Exposição do MDM para Plasmodium falciparum
- Para expor o MDM para uRBC ou iRBC, as células devem ser ressuspensas em meio de cultura MDM a fim de obter uma proporção de uRBC / iRBC: MDM de 75:1. Em anteriormentepreparadas placas de 24 poços contendo MDM, adicionar volume de suspensão adequado RBC a cada poço. Realizar todos os experimentos em triplicata.
- Incubar co-culturas durante 4 horas a 37 ° C em uma atmosfera de 5% de CO 2.
- Lavar as células com 600 ul de PBS isento de endotoxina-3 vezes, adicionar o PBS suavemente e aspirar imediatamente.
- Para lisar o RBC restante, lavar os poços por adição de 200 ul de água gelada estéril para 20 seg e aspirado.
- Adicionar 600 uL meio de cultura MDM e incubar 24 hr a 37 ° C em uma atmosfera de 5% de CO 2.
6. A infecção de MDM com um replicativa totalmente HIV-1
- A fim de avaliar o efeito de P. falciparum em replicação do HIV-1 em MDM, adicionar, de acordo com o esquema delineado na Figura 1A, 10 ng de p24 (em 300 ul de MDM media) de vírus NL4.3Bal env nos poços adequados; adicionar apenas com meio de MDM em poços de controlo .
- Incubar 2 horas a 37 ° C em um 5% De CO 2 atmosfera.
- Lavar as células com PBS isento de endotoxina-3 vezes, adicionar o PBS suavemente e aspirar imediatamente. Adicionar 600 ul de meio MDM fresco por poço.
- Incubar a 37 ° C em uma atmosfera de 5% de CO 2.
- No dia 3, 6, 9 e 12 após o início da infecção viral, a colheita 200 uL de cada sobrenadante, a adição de 200 ul de meio fresco MDM em seguida. Manter os sobrenadantes colhidos a -20 ° C para a quantificação dos principais HIV-1 de proteína da cápside p24 por ELISA após Fortin et al. 10.
7. Infecção do MDM com único luciferase Encoding Ciclo Virus
- A fim de determinar o impacto do parasita em HIV-1 a expressão do gene em MDM, adicionar, de acordo com o esquema delineado na Figura 1B, 10 ng de p24 (em 300 ul de meio de MDM) de qualquer NL4.3 Luc + Env - R + ( VSV-G), NL4.3 Luc + Env - R + (JRFL env) ou NL4.3 Luc +Env - R + vírus nos poços correspondentes.
- Incubar 2 horas a 37 ° C em uma atmosfera de 5% de CO 2.
- Lavar as células com 600 ul de PBS isento de endotoxina-3 vezes, adicionar o PBS suavemente e aspirar imediatamente. Adicionar 600 ul de meio MDM fresco por poço.
- Incubar a 37 ° C em uma atmosfera de 5% de CO 2.
- Remover a infecção hr médio 72 seguinte e adicionar 200 ul de tampão de lise 1X (fornecido com o kit de ensaio de luciferase). Permitir que as células lisar à temperatura ambiente com agitação suave. Armazenar a -20 ° C.
- Descongelar a placa e transferir 40 ul do lisado para uma placa de luminómetro de 96 poços; adicionar 100 uL de substrato de luciferase (fornecido com o kit de ensaio de luciferase) e medir a actividade (luciferase do pirilampo) luciferase num luminómetro seguindo as instruções do fabricante.
8. Os resultados representativos
Usando nosso modelo de co-infecção, mostramos que exposure de P. falciparum a MDM diminui a sua susceptibilidade à infecção HIV-1. Com efeito, uma diminuição significativa (p <0,05; 2 ANOVA, dia 12) na libertação de partículas virais, tal como medido por HIV-1 de proteína da cápside p24 no sobrenadante, é observada em MDM pré-tratados com parasitas (Figura 2A). Esta observação é confirmada em células infectadas por vírus que codificam um gene repórter da luciferase. MDM infecção com vírus tais albergando quer exógena de VSV-G ou de HIV-1 conduz a glicoproteínas significativamente a produção de luciferase (p <0,05, teste t de Student-) menos em células expostas a P. falciparum (Figura 2B). É de notar que VSV-G-pseudotyped vírus produziu muito maior actividade de luciferase que os seus homólogos JRFLenv, isto devido à maior eficiência de infecção de VSV-G partículas pseudotyped 15. Dado que a exposição ao parasita impactos MDM ambos os tipos de vírus, isso sugere que ele influencia em alguma etapa ex gene viralpressão (Figura 2B). É também importante mencionar que a viabilidade celular não foi afectada pela exposição a MDM iRBC (dados não mostrados), indicando que a inibição observada é específica e não devido a mortalidade das células.
Figura 1. A) O regime de Placa para a infecção com um vírus MDM replicativa totalmente Mock:. Células não infectadas. uRBC: células expostas a não infectadas células vermelhas do sangue. iRBC: células expostas ao Plasmodium falciparum infectadas células vermelhas do sangue. Bal: células infectadas com NL4.3Bal env Bal / uRBC:. Células expostas ao uRBC e infectados com NL4.3Bal env Bal / iRBC:.. Células expostas ao iRBC e infectados com NL4.3Bal env B) O regime de Placa para a infecção com o MDM único ciclo do vírus luciferase codificação de Mock:. células não infectadas. uRBC: células expostas a não infectadas células vermelhas do sangue. iRBC: células expostas a Plasmodium falciparum-infected células vermelhas do sangue. Δenv: células infectadas com NL4.3 Luc + Env - R + JRFL:. Células infectadas com NL4.3 Luc + Env - R + (JRFL env). VSV-G: células infectadas com NL4.3 Luc + Env - R + (VSV-G) Δenv / uRBC:.. células expostas a uRBC e infectados com NL4.3 Luc + Env-R + Δenv / iRBC: células expostas a iRBC e infectados com NL4.3 Luc + Env - R + JRFL / uRBC: células expostas ao uRBC e infectados com NL4.3 Luc + Env - R + (JRFL env).. JRFL / iRBC: células expostas ao iRBC e infectados com NL4.3 Luc + Env - R + (JRFL env). vsv-g/uRBC: células expostas a uRBC e infectados com NL4.3 Luc + Env - R + (VSV-G): vsv-g/iRBC. células expostas a iRBC e infectados com NL4.3 Luc + Env - R + (VSV-G).
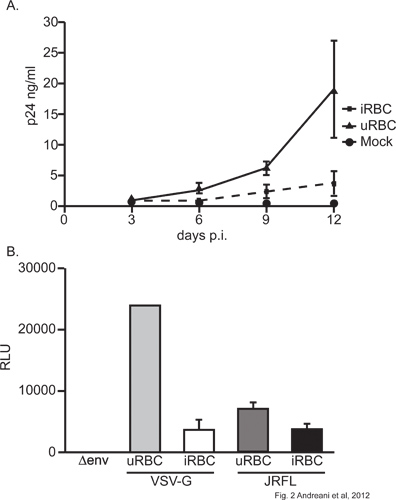
Figura 2. . A) Efeito do Plasmodium falciparum em HIV-1 de produção virai no MDM MDM foram expostos a uRBC ou iRBC a uma razão de 75:1 (uRBC / iRBC: MDM) durante 4 horas e lavou-se extensivamente. As células foram infectadas com 10 ng de p24 NL4.3Bal env durante 2 horas. Produção virai foi monitorada por ELISA para o HIV-1 p24 em células livres sobrenadante em pontos de tempo diferentes após a infecção virai inicial. Uma experiência representativa é mostrado B) Efeito do Plasmodium falciparum em HIV-1 de transcrição virais no MDM MDM foram infectadas com uRBC ou iRBC a uma razão de 75:1 (uRBC / iRBC:.. MDM). As células foram então infectadas com 10 ng de p24 do vírus de um único ciclo (ou NL4.3 Luc + Env - R +, NL4.3 Luc + Env - R + (JRFL env) ou NL4.3 Luc + Env - R + (VSV- g)). A expressão da luciferase foi avaliada em células lisados 72 hr após a infecção pelo vírus inicial. Um experimento representativo é mostrado.
Discussão
Temos ilustrado aqui duas abordagens diferentes para analisar o impacto do parasita da malária no HIV-1 ciclo viral, ou seja, por meio da análise, quer a expressão do gene virai ou a produção de progenitura do vírus e replicação em macrófagos derivados de monócitos. Abordagens semelhantes têm sido utilizados para a outra HIV-1-parasita co-infecções 16. No entanto, estes novos dados são um passo em frente na investigação da malária-HIV-1 co-infecções. . Na verdade, Diou et al 8 estudaram o efeito da hemozoin, parasitas não vivos, sobre o HIV-1 de replicação; de acordo com nossos resultados, eles observaram que hemozoin era por si só suficiente para inibir a produção viral por MDM, e não MDM.
Usando o esquema descrito experimental, observou-se que P. falciparum exerce um efeito claro prejudicial sobre o ciclo replicativo do HIV-1 em macrófagos: uma inibição significativa da produção virai é observada em macrófagos pré-expostos a parasitas (Figure 2A) e um impacto específico do parasita na transcrição viral é ilustrado na Figura 2B. No entanto, não podemos deixar de fora quaisquer efeitos adicionais do parasita na entrada viral ou fusão (decapsidation), ou sobre pós-integração de mecanismos, tais como a síntese de proteínas ou montagem de partículas virais e brotação. Além disso, é possível que um impacto diferente sobre a replicação do HIV-1 seria obtida se o parasita foram adicionados quer ao mesmo tempo ou infecção MDM seguinte com o vírus.
O nosso modelo in vitro de co-infecção fornece uma ferramenta poderosa para realizar investigações detalhadas de HIV-1 / p interacções falciparum na célula hospedeira. Por exemplo, a combinação desta disposição experimental com outras técnicas tais como quantitativa PCR em tempo real, o que pode ter como alvo e quantificar os passos específicos em retrotranscription viral e integração do genoma virai no genoma da célula hospedeira, são bastante viável e deve produzir INSI maisghts dos mecanismos envolvidos na co-infecções. Além disso, os ensaios específicos para avaliar os passos iniciais do ciclo viral (viral de fusão, decapsidation quantificação de entrada,) pode ser aplicado a este protocolo básico para analisar ainda mais o efeito sobre a replicação virai. Estas modificações mostram como flexível este protocolo é relativo HIV-1 quantificação e detecção: de facto, mesmo padrão ensaios de quantificação do HIV-1 usando transcriptase reversa marcado com trítio nucleótidos pode conseguir substituir o ELISA para o HIV-1 p24. Além MDM, esperamos nosso sistema para ser adaptável a outros tipos de células relevantes para o HIV-1-malária interacções, tais como monócitos e células dendríticas. O fato de que o MDM são células aderentes facilita a sua manipulação, permitindo fácil lavagem de iRBC que não tenham sido tomadas, ou que estão em contato com a MDM, sem eliminar os macrófagos. Essa vantagem não se aplica às células dendríticas e monócitos, que são cultivadas em suspensão, dificultando a siparação de iRBC uRBC e livre com essas células e atualmente estamos abordando este problema. O nosso sistema poderia também ser útil para estudar as interacções mais complexas, tais como os efeitos de Plasmodium-expostas derivados de monócitos células no ciclo de HIV-1 viral em outras células imunitárias, tais como CD4-células T positivas em co-cultura experimentos. Finalmente, o nosso protocolo poderia ser adequado para experimentos olhando para o efeito de P. iRBCs falciparum sobre o HIV-1 reativação em PBMC de HIV-1 indivíduos infectados.
Declaração de ética
Human PBMC foram obtidas do sangue de doadores saudáveis, em conformidade com as diretrizes do Comitê de Bioética do Centro Hospitalar de l'Université Laval Centro de Pesquisa. O consentimento escrito foi obtido de todos os doadores de sangue.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado pelos Institutos Canadenses de Pesquisa em Saúde através de um catalisador Co-infecções e co-morbidades subvenção. Eritrócitos humanos foram fornecidos pelo Centro Hospitalar de l'Université Laval banco de sangue.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| RPMI 1640 | Multicélula | 350-000-CL | |
| PBS-endotoxina livre | Sigma | D8662 | |
| FBS | Wisent | 080-150 | Inactivado pelo calor a 56 ° C durante 30 min |
| Accutase | eBiosciences | 00-4555-56 | |
| Albumax II | Gibco | 11021-037 | Dissolvido em meio RPMI 1640, esterilizada por filtração |
| Meio de separação de linfócitos | Multicélula | 305-010-CL | |
| Brancos placas luminómetro | Promega | Z3291 | |
| CS Macs separação columms | Miltenyi Biotech | 130-041-305 | |
| Separador VarioMacs | Miltenyi Biotech | 130-090-282 | |
| Penicilina / estreptomicina solução | Wisent | 450-201-EL | |
| D-sorbitol | Sigma-Aldrich | S1876 | |
| Raspador de células (25 cm) | Sarstedt | 83,1830 | |
| De 24 poços de cultura placas da célula | BD Falcon | 353047 | |
| 150 x 20 milímetros prato de cultura | Sarstedt | 83.1803.003 | |
| M-CSF | GenScript | Z02001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| De soro humano AB + | Vale Biomédica | HP1022 | |
| HBSS | Wisent | 311-505-CL | |
| Gentamicina | Sigma-Aldrich | G1397 | |
| Hipoxantina | Sigma-Aldrich | H9636 | |
| De NaHCO3 | Sigma-Aldrich | S5761 | |
| Sistema de ensaio de luciferase | Promega | E1500 | |
| Glóbulos vermelhos humanos | Obtido purificada a partir de banco de sangue CHUL. |
Referências
- WHO. World Malaria Report. , (2005).
- Kublin, J. G., Patnaik, P., Jere, C. S. Effect of Plasmodium falciparum malaria on concentration of HIV-1-RNA in the blood of adults in rural Malawi: a prospective cohort study. Lancet. 365, 233-240 (2005).
- Cuadros, D. F., Branscum, A. J., Crowley, P. H. HIV-malaria co-infection: effects of malaria on the prevalence of HIV in East sub-Saharan Africa. Int. J. Epidemiol. 40, 931-939 (2011).
- Xiao, L., Owen, S. M., Rudolph, D. L., Lal, R. B., Lal, A. A. Plasmodium falciparum antigen-induced human immunodeficiency virus type 1 replication is mediated through induction of tumor necrosis factor-alpha. J. Infect. Dis. 177, 437-445 (1998).
- Froebel, K., Howard, W., Schafer, J. R. Activation by malaria antigens renders mononuclear cells susceptible to HIV infection and re-activates replication of endogenous HIV in cells from HIV-infected adults. Parasite Immunol. 26, 213-217 (2004).
- Diou, J., Tardif, M. R., Barat, C., Tremblay, M. J. Dendritic cells derived from hemozoin-loaded monocytes display a partial maturation phenotype that promotes HIV-1 trans-infection of CD4+ T cells and virus replication. J. Immunol. 184, 2899-2907 (2010).
- Diou, J., Tardif, M. R., Barat, C., Tremblay, M. J. Malaria hemozoin modulates susceptibility of immature monocyte-derived dendritic cells to HIV-1 infection by inducing a mature-like phenotype. Cell Microbiol. 12, 615-625 (2010).
- Diou, J., Gauthier, S., Tardif, M. R. Ingestion of the malaria pigment hemozoin renders human macrophages less permissive to HIV-1 infection. Virology. 395, 56-66 (2009).
- Davies, J. Q., Gordon, S. Isolation and culture of human macrophages. Methods Mol. Biol. 290, 105-116 (2005).
- Fortin, J. F., Cantin, R., Lamontagne, G., Tremblay, M. Host-derived ICAM-1 glycoproteins incorporated on human immunodeficiency virus type 1 are biologically active and enhance viral infectivity. J. Virol. 71, 3588-3596 (1997).
- Bounou, S., Leclerc, J. E., Tremblay, M. J. Presence of host ICAM-1 in laboratory and clinical strains of human immunodeficiency virus type 1 increases virus infectivity and CD4(+)-T-cell depletion in human lymphoid tissue, a major site of replication in vivo. J. Virol. 76, 1004-1014 (2002).
- Wei, X., Decker, J. M., Liu, H. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob. Agents Chemother. 46, 1896-1905 (2002).
- Trager, W., Jensen, J. B. Human malaria parasites in continuous culture. Science. 193, 673-675 (1976).
- Turrini, F., Ginsburg, H., Bussolino, F., Pescarmona, G. P., Serra, M. V., Arese, P. Phagocytosis of Plasmodium falciparum-infected human red blood cells by human monocytes: involvement of immune and nonimmune determinants and dependence on parasite developmental stage. Blood. 80, 801-808 (1992).
- Akkina, R. K., Walton, R. M., Chen, M. L., Li, Q. X., Planelles, V., Chen, I. S. High-efficiency gene transfer into CD34+ cells with a human immunodeficiency virus type 1-based retroviral vector pseudotyped with vesicular stomatitis virus envelope glycoprotein G. J. Virol. 70, 2581-2585 (1996).
- Zhao, C., Papadopoulou, B., Tremblay, M. J. Leishmania infantum enhances human immunodeficiency virus type-1 replication in primary human macrophages through a complex cytokine network. Clinical Immunol. 113, 81-88 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados