Method Article
Single-Molecule Dwell-Time Analysis of restriction Endonuclease-mediated DNA cleavage(제한 엔도뉴클레아제 매개 DNA 절단의 단일 분자 체류 시간 분석)
요약
quantum-dot-labeled DNA와 total internal reflection fluorescence microcopy를 사용하여 unlabeled protein을 사용하면서 restriction endonucleases의 반응 메커니즘을 조사할 수 있습니다. 이 단일 분자 기법을 사용하면 개별 단백질-DNA 상호 작용을 대규모로 다중화하여 관찰할 수 있으며, 데이터를 풀링하여 잘 채워진 체류 시간 분포를 생성할 수 있습니다.
초록
이 새로운 전반사 형광 현미경 기반 분석법은 한 번의 실험에서 수백 개의 개별 제한 엔도뉴클레아제(REase) 분자에 대한 촉매 주기의 길이를 동시에 측정할 수 있도록 합니다. 이 분석은 단백질 라벨링이 필요하지 않으며 단일 이미징 채널로 수행할 수 있습니다. 또한 여러 개별 실험의 결과를 취합하여 잘 채워진 체류 시간 분포를 생성할 수 있습니다. 결과 체류 시간 분포를 분석하면 직접 관찰할 수 없는 운동 단계의 존재를 밝혀 DNA 절단 메커니즘을 설명하는 데 도움이 될 수 있습니다. 잘 연구된 REase, EcoRV(회문 서열 GAT↓ATC(여기서 ↓는 절단 부위)를 절단하는 이합체 유형 IIP 제한 엔도뉴클레아제와 함께 이 분석을 사용하여 수집된 예제 데이터는 이전 연구와 일치합니다. 이러한 결과는 EcoRV가 DNA가 없을 때 마그네슘이 결합한 후 마그네슘을 도입하여 시작되는 DNA 절단 경로에 최소 3단계가 있음을 시사하며, 각 단계에 대해 평균 0.17 s-1 의 비율이 있습니다.
서문
제한 엔도뉴클레아제(REase)는 DNA의 염기서열 특이적 이중 가닥 절단에 영향을 미치는 효소입니다. 1970년대에 REases의 발견은 재조합 DNA 기술의 개발로 이어졌고, 이러한 효소는 이제 유전자 변형 및 조작에 없어서는 안 될 실험실 도구가 되었습니다1. Type II REase는 인식 서열 내 또는 근처의 고정된 위치에서 DNA를 절단하기 때문에 이 등급에서 가장 널리 사용되는 효소입니다. 그러나 Type II REase 사이에는 많은 변이가 있으며, 진화 관계에 따라 분류되기보다는 특정 효소 특성에 따라 여러 하위 유형으로 나뉩니다. 각 아형(subtype) 중에는 분류 체계에 대한 빈번한 예외가 있으며, 많은 효소가 여러 아형(subtype)에 속한다2. 수천 개의 Type II REase가 확인되었으며 그 중 수백 개가 상업적으로 이용 가능합니다.
그러나 Type II REase 간의 다양성에도 불구하고 자세히 연구된 REase는 거의 없습니다. 1975년 리처드 로버츠 경(Sir Richard Roberts)에 의해 확립된 제한효소 데이터베이스인 REBASE에따르면3, 발표된 동역학 데이터는 이러한 효소 중 20개 미만에 대해 사용할 수 있습니다. 게다가, 몇몇 REases는 그들의 인식 순서 4,5,6,7에 만나서 결합하기 전에 DNA를 따라서 확산하는 동안 단 하나 분자 수준에서 직접 관찰되는 동안, 그들의 절단 반응 역학에 대한 단 하나 분자 학문은 거의 없습니다. 기존 연구들은 단일 절단사건이 발생하는 시간의 변동을 상세히 분석하기에 충분한 통계를 보고하지 못하거나8,9,10 분할 횟수11의 전체 분포를 포착할 수 없다. 이러한 유형의 분석은 상대적으로 수명이 긴 kinetic 중간체의 존재를 밝힐 수 있으며 REase 매개 DNA 절단의 메커니즘을 더 잘 이해할 수 있습니다.
단일 분자 수준에서, 생화학적 과정은 확률적이며, 과정의 단일 인스턴스가 발생할 때까지 기다리는 시간( τ)은 가변적입니다. 그러나 τ 의 많은 측정값은 발생하는 프로세스 유형을 나타내는 확률 분포 p(τ)를 따를 것으로 예상할 수 있습니다. 예를 들어, 효소에서 제품 분자를 방출하는 것과 같은 단일 단계 프로세스는 Poisson 통계를 따르고 p(τ)는 음의 지수 분포의 형태를 취합니다.
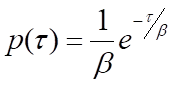
여기서 β은 평균 대기 시간입니다. 프로세스의 속도 k는 평균 대기 시간의 역수인 1/β와 같습니다. 둘 이상의 단계가 필요한 공정의 경우 p(τ)는 각 개별 단계에 대한 단일 지수 분포의 컨볼루션이 됩니다. 평균 대기 시간 β이 동일한 N 단일 지수 감쇠 함수의 컨볼루션에 대한 일반적인 솔루션은 감마 확률 분포입니다.

여기서 Γ(N)은 감마 함수로, N-1의 계승을 N의 정수가 아닌 값으로의 보간을 설명합니다. 이 일반적인 솔루션은 개별 단계의 평균 대기 시간이 유사한 경우 근사치로 사용할 수 있지만, 상대적으로 빠른 단계의 존재는 대기 시간이 훨씬 긴 단계에 의해 가려진다는 점을 이해해야 합니다. 즉, N 값은단계 12 수에 대한 하한을 나타냅니다. 적절한 수의 대기 시간 측정을 통해 이벤트를 범주화하고 감마 분포를 결과 히스토그램에 맞추거나 최대우도 추정 방법을 사용하여 매개변수 β 및 N을 추정할 수 있습니다. 따라서 이러한 유형의 분석은 앙상블 분석에서 쉽게 해결할 수 없는 역학 단계의 존재를 드러낼 수 있으며 매개변수를 정확하게 추정하기 위해 많은 수의 관찰이 필요합니다12,13.
이 논문은 양자 점 표지 DNA 및 전반사 형광(TIRF) 현미경을 사용하여 수백 개의 개별 REase 매개 DNA 절단 이벤트를 병렬로 관찰하는 방법을 설명합니다. 분석의 설계를 통해 여러 실험의 결과를 통합할 수 있으며 수천 개의 이벤트를 포함하는 체류 시간 분포를 생성할 수 있습니다. 양자점의 높은 광안정성과 밝기는 실험 시작 후 몇 분 후에도 발생하는 절단 이벤트를 관찰할 수 있는 능력을 희생하지 않고 10Hz 시간 분해능을 허용합니다. 우수한 시간 분해능과 넓은 동적 범위, 대규모 데이터 세트를 수집할 수 있는 기능이 결합되어 체류 시간 분포의 정확한 특성 분석을 통해 회전율이 1min-1 범위인 REase의 절단 경로에 여러 역학 단계가 존재하는지 확인할 수 있습니다. EcoRV의 경우 세 가지 역학 단계를 해결할 수 있으며, 모두 다른 수단을 통해 식별되어 분석이 이러한 단계의 존재에 민감하다는 것을 확인합니다.
관심 인식 서열을 포함하는 Duplex DNA 기질은 비오틴화된 올리고뉴클레오티드를 단일 공유 결합 반도체 나노결정 양자점으로 표지된 상보적인 가닥에 어닐링하여 생산됩니다. 이 기판은 고분자량 폴리에틸렌 글리콜(PEG) 분자가 표면에 공유 결합되어 있는 잔디가 있는 유리 커버슬립 위에 구축된 유동 채널로 도입됩니다. DNA 기질은 자유 말단에 비오틴이 있는 PEG 분자의 일부에 의해 비오틴-스트렙타비딘-비오틴 결합을 통해 포획됩니다. TIRF 현미경에서는 유리-액체 계면으로부터의 거리에 따라 기하급수적으로 감소하는 증발파가 조명을 제공합니다. 침투 깊이는 사용되는 빛의 파장 순서에 있습니다. 이러한 조건에서는 기능성 유리 표면에 포착된 DNA 분자에 의해 표면에 연결된 양자점만 여기됩니다. 용액에서 자유로운 양자점은 조명 영역 내에서 제한되지 않으므로 발광하지 않습니다. 양자점을 표면에 묶고 있는 DNA가 절단되면 해당 양자점은 표면에서 자유롭게 확산될 수 있으며 형광 이미지에서 사라집니다.
많은 제2형 REase가 마그네슘14가 없을 때 DNA와 결합하는 것으로 알려져 있지만, 모든 REase는 DNA 절단15을 매개하기 위해 마그네슘을 필요로 한다. 이러한 REase는 마그네슘이 없을 때 표면 고정화된 DNA와 결합할 수 있습니다. 마그네슘 함유 완충액이 REase가 DNA에 사전 결합되어 있는 채널을 통해 흐르면 양자점이 사라지는 것처럼 절단이 즉시 시작됩니다. REase 분자를 사전 결합한 다음 마그네슘을 주입하여 DNA 절단을 시작함으로써 달성된 동기화는 실험에서 관찰된 효소 집단의 각 분자에 대해 독립적으로 DNA 절단 완료까지의 지연 시간 측정을 용이하게 합니다. 플루오레세인은 마그네슘 함유 완충액에 추적자 염료로 포함되어 마그네슘이 시야에 도착했음을 나타냅니다. 마그네슘 함유 완충액에는 효소가 포함되어 있지 않기 때문에 마그네슘 함유 완충액이 도착한 후 각 양자점이 소멸될 때까지의 지연 시간은 이미 DNA에 결합된 REase가 DNA를 절단하고 유리 표면에서 양자점을 방출하는 데 걸리는 시간을 나타냅니다. 양자점 소멸은 빠르게 발생하여 강도 궤적의 급격한 감소를 초래하여 주어진 DNA 분자가 절단되는 시간을 명확하게 표시합니다. 이벤트 시간의 결정은 강도 궤적의 수학적 분석을 통해 이루어지며, 일반적인 실험에서는 수백 개의 식별 가능한 절단 이벤트가 발생합니다. 여러 실험의 결과를 취합하여 비선형 최소제곱 또는 최대우도 분석을 통해 매개변수 N 및 β를 추정할 수 있는 적절한 통계를 제공할 수 있습니다.
프로토콜
1. 일반 정보
- 올리고뉴클레오티드 설계
참고: 60 염기쌍(bp) 길이의 DNA 기질은 100mM NaCl에서 이중 용융 온도가 75°C인 한 쌍의 상보적 올리고뉴클레오티드로 형성됩니다.- 단일 5' 비오틴 변형으로 합성된 올리고뉴클레오티드 하나와 5' 티올 변형(6탄소 스페이서 사용)으로 합성된 다른 올리고뉴클레오티드를 주문하십시오. 인식 사이트를 이중 영역의 가운데에 배치합니다.
참고: EcoRV와 함께 사용하기 위한 올리고뉴클레오티드 염기서열은 아래와 같습니다(인식 부위는 굵은 글씨체).
5' 비오틴 - AAA, ACC, GAC, ATG, TG, TTG, ATTC, TGA, TGA, AAC, AAC, GA, G, ATA, TC, ACA, AAG, CCA, TGA, ACA, AAG, CAG, CCG-3'
5' 티올 - CGG, CTG, CTT, TGT, TCA, TGG, CTT,TGA, TAT, C, CC, GTT, TCA, GGA, AAT, CAA, CAT, GTC, GGT, TTT-3'
- 단일 5' 비오틴 변형으로 합성된 올리고뉴클레오티드 하나와 5' 티올 변형(6탄소 스페이서 사용)으로 합성된 다른 올리고뉴클레오티드를 주문하십시오. 인식 사이트를 이중 영역의 가운데에 배치합니다.
- 모든 단계에 18MOhm 저항률의 초순수를 사용하십시오.
- 광퇴색을 방지하기 위해 양자점을 포함하는 모든 용액을 빛으로부터 보호합니다.
- 압축 공기 소스를 사용하여 이 프로토콜을 완료합니다.
2. quantum-dot-labeled DNA 기질 물질의 제조
참고: 위에서 설명한 올리고뉴클레오티드 외에도 다른 재료에 대해서는 재료 표를 참조하고, 양자 점 표지 DNA 기질의 제조에 필요한 완충액에 대해서는 표 1 을 참조하십시오.
- 양자점에 결합할 oligonucleotide의 5' 티올 그룹을 줄입니다.
- 각 티올화 올리고뉴클레오티드를 100μM 농도의 물에 재현탁시킵니다.
- 각 올리고뉴클레오티드 샘플에 대해 650μL의 물을 크기 배제 스핀 컬럼에 피펫하고 ~15초 동안 소용돌이를 일으킵니다. 컬럼을 30분 동안 포장하도록 두십시오.
- DTT는 용액에서 빠르게 분해되므로 매번 사용하기 직전에 새로운 100mM 디티오트레이톨(DTT) 용액을 준비하십시오. 7.7mg의 DTT가 들어있는 바이알 하나를 조심스럽게 열고 500μL의 인산나트륨 완충액을 바이알에 피펫으로 넣습니다. 소용돌이를 철저히 섞습니다.
- 각 재현탁 올리고뉴클레오티드에 대해 새 튜브를 준비하고 50μL의 올리고뉴클레오티드와 50μL의 DTT 용액을 튜브에 추가합니다. 피펫을 위아래로 피펫하여 혼합합니다. 올리고뉴클레오티드의 5' 말단에서 산화된 티올기 사이의 이황화물 결합을 줄이기 위해 실온에서 30분 동안 배양합니다.
- 스핀 컬럼 캡을 제거하고 각 컬럼을 수집 튜브에 넣습니다. 스핀 컬럼을 750× g 에서 2분 동안 원심분리하고 용리액을 버립니다.
- 스핀 컬럼을 새 원심분리기 튜브로 옮깁니다. DNA/DTT 혼합물 샘플 한 개의 전체 부피(100μL)를 준비된 각 컬럼에 부드럽게 피펫합니다. 750 × g 에서 2분 동안 원심분리하고 260nm에서 용리액의 흡광도를 측정하여 농도가 약 40μM인지 확인합니다.
- 즉시 사용하지 않을 샘플은 -20 °C에서 보관하여 티올 그룹의 산화와 이황화 결합의 형성을 방지합니다.
- DNA와 양자점의 결합
- 양자점 스톡 50μL의 부분 표본 1개를 투석 장치에 피펫으로 삽입하여 각 구조를 만들고 피펫 팁이 있는 멤브레인을 건드리지 않도록 합니다. 시료 부피의 1000배 이상인 N-cyclohexyl-2-aminoethanesulfonic acid(CHES) 완충액에 대해 ~100rpm으로 15분 동안 교반하여 투석합니다.
- 매번 사용 직전에 CHES 완충액에 설포숙시니미딜-4-(N-말레이미도메틸) 시클로헥산-1-카르복실레이트(설포-SMCC)의 새로운 6mM 용액을 준비합니다. 2mg의 sulfo-SMCC가 들어 있는 바이알 하나를 조심스럽게 열고 800μL의 CHES 완충액을 바이알에 피펫으로 넣습니다. 소용돌이를 철저히 섞습니다.
- 피펫터를 사용하여 투석 장치에서 부유 양자점을 조심스럽게 회수하고 동일한 부피의 sulfo-SMCC 용액이 들어 있는 새 튜브로 옮깁니다. 위아래로 피펫을 만들어 섞습니다. sulfo-SMCC가 양자점의 1차 아민과 반응할 수 있도록 1000rpm에서 진탕하면서 실온에서 1시간 동안 배양합니다.
- 피펫터를 사용하여 각 샘플을 새 투석 장치로 조심스럽게 옮깁니다. 과도한 sulfo-SMCC를 제거하려면 투석 장치에 포함된 부피의 1000배 이상인 CHES 완충액에 대해 15분 동안 교반하면서 투석합니다. 완충액을 2번 교환하고 새 CHES 완충액을 총 3번 사용하여 투석을 수행하여 각 완충액 교환 후 15분 후에 투석을 진행할 수 있습니다.
- 2차 반응을 준비하기 위해 완충액을 교환하려면 투석 장치를 장치에 포함된 부피의 1000배 이상인 인산염 완충 식염수(PBS)가 들어 있는 비커에 넣습니다. 15분 동안 교반하면서 투석하고, 완충액을 2번 교환하고, 신선한 PBS 3배로 총 3번 투석을 수행하여 각 완충액 교환 후 15분 동안 투석을 진행할 수 있습니다.
- 피펫터를 사용하여 투석 장치에서 양자점이 포함된 용액을 조심스럽게 회수하고 각 샘플을 PBS에 희석된 등몰량의 티올화 올리고뉴클레오티드가 들어 있는 새 튜브로 옮깁니다. 이 시점에서 양자점 농도는 ~4μM이므로 동일한 부피의 PBS와 환원 및 정제된 올리고뉴클레오티드 샘플의 부피(약 40μM 농도)의 1/10을 결합합니다. 1000rpm에서 진탕하면서 실온에서 2시간 동안 배양합니다.
- 각 샘플에 소 혈청 알부민(BSA, 10mg/mL)을 추가하여 최종 BSA 농도 0.5mg/mL를 얻습니다. 피펫터를 사용하여 각 샘플을 조심스럽게 새 투석 장치로 옮기고 투석 장치에 포함된 부피의 1000배 이상인 저장 완충액에 대해 투석합니다. 완충액을 2번 교환하고, 새 저장 완충액으로 총 3번 투석을 실시하여 완충액 교환 후 15분 동안 투석을 진행할 수 있습니다.
- 피펫터를 사용하여 각 샘플을 조심스럽게 회수하고 새 튜브에 넣고 4°C에서 보관합니다.
알림: -20 °C에서 보관하지 마십시오. 퀀텀닷은 동결에 의해 손상됩니다.
- quantum-dot-labeled oligonucleotide를 biotinylated oligonucleotide로 어닐링
- 각 비오틴화 올리고뉴클레오티드를 100μM 농도의 보관 버퍼에 재현탁시킵니다. 사용하지 않은 샘플은 -20°C에서 보관합니다.
- 양자점 표지 올리고뉴클레오티드 샘플과 10배 이상의 비오틴화 올리고뉴클레오티드 몰을 결합합니다. 퀀텀 도트 라벨링 샘플의 예상 DNA 농도는 2μM이므로 이 샘플의 10μL당 0.2μL의 비오틴화 올리고뉴클레오티드를 추가합니다.
- 열 블록의 혼합물을 75°C(상보 영역의 용융 온도)로 가열합니다. 그 온도에서 5분 동안 유지합니다. 히트 블록을 끄고 천천히 식히십시오. 완성 된 구조물을 4 ° C에서 보관하십시오. 얼리지 마십시오.
3. 커버슬립의 표면 기능화
참고: 이 프로세스는 이전에 다른 JoVE 비디오 프로토콜16,17에서 설명되었습니다. 이 프로토콜은 더 작은 유리 슬라이드를 수용하기 위해 약간의 변경이 있는 절차의 수정된 버전을 설명합니다. 커버슬립의 표면 기능화에 필요한 다른 재료에 대해서는 재료 표를 참조하십시오.
- 각 커버슬립 홀더에 5개의 커버슬립을 넣습니다. 서로 달라붙지 않도록 각 커버슬립 사이에 한 칸씩 건너뛰십시오. 홀더의 커버 슬립을 커버가 있는 병에 넣고 커버슬립이 덮일 때까지 병에 에탄올을 넣고 상단을 단단히 조입니다. 항아리 전체를 욕조 초음파 발생기의 물에 넣고 완전히 담그지 않고 30 분 동안 초음파 처리합니다.
- 깨끗한 비커나 항아리에 초순수를 채웁니다. 금속 핀셋을 사용하여 에탄올에서 홀더의 커버슬립을 제거하고 물에 담가 헹굽니다. 그런 다음 커버슬립과 홀더를 1M 수산화칼륨(KOH) 용액이 들어 있는 병에 옮깁니다. 상단을 단단히 조이고 항아리를 초음파 발생기에 넣고 30 분 동안 초음파 처리합니다.
알림: KOH 용액은 부식성이며 자극성이 있으므로 취급할 때 주의하십시오. - 에탄올과 KOH 용액에서 초음파 처리를 반복하고 각 단계 사이에 위에서 설명한대로 커버 슬립을 물로 헹굽니다.
- 홀더의 커버슬립을 순수한 아세톤으로 채워진 깨끗한 병에 옮깁니다. 위에서 설명한대로 항아리를 30 분 동안 초음파 처리하여 청소를 마칩니다. 이 단계 후에는 물로 헹구지 마십시오.
- 금속 핀셋을 사용하여 홀더의 커버슬립을 80mL의 새 아세톤과 마이크로 스케일 젓는 막대가 들어 있는 깨끗한 100mL 비커에 옮깁니다. 최소 1,000rpm으로 설정된 자기 교반 플레이트에 비커를 놓습니다. 아세톤을 세게 저어주는 동안 1.6mL의 3-아미노프로필트리에톡시실란(APTES)을 비커에 피펫하여 2% v/v 용액을 만듭니다.
알림: APTES는 부식성이 있으므로 피펫팅할 때 주의하십시오. - 커버슬립을 용액에서 2분 동안 배양한 다음 금속 핀셋을 사용하여 홀더의 커버슬립을 물 비커에 옮겨 반응을 담금질합니다. 커버슬립을 두 번 더 헹구고 비커의 물을 교체합니다.
- 실란화 된 유리를 120 ° C의 오븐에서 75 분 동안 경화시킵니다. 다음 단계를 즉시 계속하지 않는 경우 커버슬립을 최대 며칠 동안 진공 상태에서 보관하십시오.
- N-하이드록시숙신이미드(NHS) 에스테르 유도체화 폴리에틸렌글리콜(PEG)을 100mM 중탄산나트륨(pH 8.2)에 용해시킵니다. ~100mg/mL의 메톡시 종단 PEG와 비오틴 종단 PEG의 10:1 비율을 사용합니다.
- PEG 용액 100μL를 건식 실란화 커버슬립의 절반에 피펫하고 각각을 두 번째 커버슬립으로 덮습니다. 커버슬립이 서로 달라붙는 것을 방지하기 위해 각 모서리에 배치된 작은 파라필름 조각을 스페이서로 사용하십시오.
- 커버슬립을 습한 환경에서 3.5시간 동안 배양합니다. 커버슬립 샌드위치를 분리합니다. 물병을 사용하여 각 커버슬립을 다량의 물로 세척하고 압축 공기로 건조시킵니다.
- 기능성 커버슬립은 진공 상태에서 보관하십시오. PEG 처리된 면은 다른 면이 DNA 기질을 포착하지 못하므로 위쪽을 유지해야 합니다.
4. Microfluidic 장치 건축
참고: 미세유체 장치의 구성에 필요한 다른 재료에 대해서는 재료 테이블을 참조하십시오.
- 테이퍼 다이아몬드 포인트 휠 비트가 장착된 휴대용 회전식 멀티툴을 사용하여 석영 슬라이드의 반대쪽 모서리에 두 개의 구멍을 뚫어 입구와 출구 역할을 합니다. 드릴링하는 동안 슬라이드를 제자리에 고정하고 비트에 윤활유를 바르고 물로 계속 미끄러지십시오. 각 실험이 끝나면 사용한 장치를 아세톤에 담가 접착제와 에폭시를 용해시켜 재사용할 수 있도록 준비된 석영 슬라이드를 회수합니다. 나머지 구성 요소를 폐기합니다.
알림: 드릴링은 수동으로 수행할 수 있지만 멀티툴용 프레스 스타일 홀더를 사용하면 프로세스가 더 쉬워집니다. - 25 μL의 스트렙타비딘 용액을 80 μL의 PBS 및 20 μL의 차단 완충액과 결합합니다. PEG 처리된 커버슬립에 이 혼합물을 코팅하고 습한 환경에서 30분 동안 배양합니다.
- 커버슬립을 배양하는 동안 이미징 스페이서를 준비하여 흐름 채널을 생성합니다. 이미징 스페이서 재료의 1인치 정사각형 조각을 자른 다음 석영 슬라이드에 뚫린 구멍 끝에 정렬된 2mm 너비의 채널을 표시합니다(그림 2). 메스로 스페이서 재료에서 채널을 잘라냅니다.
- 이미지 스페이서에서 뒷면의 한쪽을 떼어내고 입구와 출구 구멍을 막지 않도록 주의하면서 석영 슬라이드에 조심스럽게 놓습니다. 이전 실험에서 남아 있는 접착제를 제거하기 위해 석영 슬라이드를 아세톤으로 철저히 청소해야 합니다.
- 커버슬립을 물로 씻고 압축 공기로 건조시킵니다. 이미징 스페이서에서 뒷면의 다른 쪽을 제거하고 석영 슬라이드와 기능화된 스트렙타비딘 코팅된 커버슬립 사이에 끼우고 플라스틱 핀셋을 사용하여 어셈블리를 함께 누르고 접착제에서 기포를 제거합니다.
- 30cm 길이의 폴리에틸렌 튜브 하나를 석영 슬라이드의 각 구멍에 삽입합니다. 용액이 자유롭게 흐를 수 있도록 튜브의 끝을 비스듬히 절단해야 합니다. 튜브 랙 또는 기타 지지대를 사용하여 튜브를 제자리에 고정하고 튜브를 제자리에 밀봉하고 조립된 장치의 가장자리를 에폭시로 밀봉합니다.
알림: 에폭시가 굳었을 때 바닥에서 쉽게 벗겨질 수 있는 파라필름 조각에 장치를 만드는 것이 좋습니다. - 에폭시가 굳으면 빈 주사기의 뭉툭한 바늘을 장치의 출구 튜브에 삽입하고 입구 튜브의 끝을 차단 완충액으로 채워진 용기에 담그십시오. 주사기 플런저를 뒤로 당겨 장치에 차단 완충액을 채웁니다. 사용하기 최소 30분 전에 장치를 배양하도록 두십시오.
5. quantum-dot-labeled DNA 기질의 표면 테더링
참고: 위에서 설명한 미세유체 장치, DNA 기질 및 차단 완충액 외에도 다른 물질에 대한 재료 표 와 양자 점 표지된 DNA 기질의 표면 테더링에 필요한 완충액에 대한 표 1 을 참조하십시오.
- 미세유체 장치를 테이프로 현미경 스테이지 플레이트에 부착하고, 대물렌즈를 장치 바닥에 접촉시키고, 시야가 미세유체 채널 내에 있도록 대물렌즈를 배치합니다.
- 출구 튜브를 주사기 펌프에 연결한 후 주사기 플런저를 뒤로 당겨 새로운 차단 완충액으로 미세유체 채널을 세척합니다. 튜브나 채널에 기포가 끼어 있지 않은지 확인하십시오.
알림: 이 시점부터 기포가 장치에 유입되지 않도록 흡입 튜브가 액체 상태인지 확인하십시오. - 준비된 DNA 기질 1μL를 1mL의 차단 완충액으로 희석합니다. 입구 튜브를 희석된 DNA 기판에 넣고 200μL/분의 속도로 채널을 통해 800μL의 기판 용액을 흐르게 합니다. DNA 용액이 흐름이 멈춘 후 15분 동안 방해받지 않고 채널에서 배양하도록 합니다.
- 200 μL/min의 속도로 채널을 통해 최소 800 μL의 차단 완충액을 흘려 채널에서 결합되지 않은 DNA를 헹궈냅니다.
- 표면 테더링된 양자점이 명확하게 보이도록 레이저 출력, 현미경 초점 및 TIRF 각도를 조정합니다.
알림: 양자점은 빠르게 표백되지 않지만 깜박임을 최소화하기 위해 전력을 가능한 한 낮게 유지하는 것이 가장 좋습니다.
6. REase 매개 DNA 절단
참고: 재료에 대해서는 재료 표를 참조하고 REase 매개 DNA 절단에 필요한 완충액에 대해서는 표 1 을 참조하십시오.
- 카메라가 최적의 작동 온도로 냉각되고 0.10초의 노출 시간으로 고속 스트리밍을 위해 설정되어 있는지 확인하십시오. ~4분 동안 데이터를 수집할 준비를 합니다.
- 200 μL/min의 유속으로 마그네슘이 없는 800 μL의 실험 버퍼로 미세유체 장치를 플러시합니다.
- REase를 마그네슘이 없는 실험용 완충액의 부분 표본(1mL)에 넣고 피펫팅으로 부드럽게 혼합합니다. EcoRV 400개/mL에 해당하는 100,000개/mL 스톡 4μL를 사용합니다. 200 μL/min의 유속으로 채널을 통해 800 μL의 희석된 효소를 흐르게 합니다.
- 마그네슘과 플루오레세인이 포함된 실험용 완충액을 200μL/분의 유속으로 흐르게 하여 실험을 시작합니다. 주사기 펌프를 시작한 직후 데이터 캡처를 시작합니다. 800 μL의 버퍼를 흘린 후 데이터 수집을 중지합니다.
7. 데이터 분석
참고: 이 프로토콜에 사용되는 데이터 분석 소프트웨어에 대한 재료 표를 참조하고 다른 분석 플랫폼을 사용하는 경우 조정하십시오.
- 제로 타임 포인트 결정
- Image Processing Toolbox에 내장된 형태학적 top-hat 필터링 함수인 imtophat 함수를 사용하여 실험 영화의 각 프레임에서 배경 형광을 뺍니다. 반지름이 3픽셀인 디스크를 구조 요소로 선택합니다. 원본 영상에서 필터링된 영상을 빼서 배경 농도를 구합니다.
참고: 필터는 배경보다 밝고 구조 요소보다 작은 이미지 피처만 유지합니다. - 각 동영상 프레임에서 빼낸 배경의 평균을 구합니다. 이 값의 궤적을 사용하여 실험의 영점 시점을 결정합니다(그림 3A). 데드 볼륨 플로우 동안 증가율이 변화율의 표준 편차의 3배를 초과하는 첫 번째 프레임을 찾아 시작 시간을 결정합니다.
참고: 각 동영상 프레임에 대해 뺀 배경의 평균 값 변화율이 급격히 증가하면 트레이서 염료를 함유한 최종 실험 버퍼가 플로우 셀에 유입된 시간을 나타냅니다(그림 3B).
- Image Processing Toolbox에 내장된 형태학적 top-hat 필터링 함수인 imtophat 함수를 사용하여 실험 영화의 각 프레임에서 배경 형광을 뺍니다. 반지름이 3픽셀인 디스크를 구조 요소로 선택합니다. 원본 영상에서 필터링된 영상을 빼서 배경 농도를 구합니다.
- Quantum dot intensity trajectory 계산 및 분석
- 트레이서 염료가 도착하기 전에 기록된 배경 보정된 동영상 프레임 세트에 대한 최대 강도 투영을 계산합니다. pkfnd와 같은 피크 찾기 함수를 사용하여 이 투영 이미지에서 양자점의 위치를 1픽셀 정확도로 결정합니다.
- 피크 찾기 함수에 의해 반환된 위치를 둘러싼 한 변당 3픽셀의 정사각형 영역의 각 동영상 프레임에서 평균 강도를 계산하여 개별 양자점에 대한 강도 궤적을 생성합니다.
참고: 위에서 설명한 배경 보정은 추적자 염료에 의해 도입된 아티팩트를 제거합니다. 그 결과 발생하는 강도 궤적은 상대적으로 평평해야 하지만 여전히 자연적으로 발생하는 강도 변동을 포함해야 합니다(그림 4). - 강도 궤적의 통계적 분석을 통해 양자점 소멸 이벤트를 식별합니다. 각 양자점 강도 궤적에 대해 궤적에서 관찰된 노이즈에 따라 해당 궤적에 대한 최대값과 최소값 간의 차이의 적절한 비율만큼 최소값보다 높은 임계값 강도를 계산합니다.
참고: 양자점의 강도 변동은 일반적으로 배경 변동보다 훨씬 큽니다. 이러한 값을 비교하여 적절한 값을 결정할 수 있습니다: 선택한 임계값은 가장 높은 배경 값보다 높아야 하지만, 이상적으로는 양자점 형광의 가장 낮은 값보다 낮아야 합니다. 제시된 데이터의 경우, 사용된 임계값은 각 궤적의 최소값에 해당 궤적의 최대값과 최소값 간의 차이의 1/3을 더한 값보다 큰 값으로 계산되었습니다. 양자점은 해당 위치에서 관찰된 강도가 이 임계값을 초과한 마지막 시점에서 사라지는 것으로 간주될 수 있습니다. - 실종 추정 사건을 확인합니다. 관찰된 강도가 사라지기 전 영화 프레임의 절반 이상에 대한 궤적의 최소 및 최대 강도 값 사이의 중간점보다 높고 이벤트 후 강도 궤적의 표준 편차가 감소한 경우에만 이벤트를 최종 분석에 포함합니다.
참고: 유효한 실종 이벤트만 기록되도록 하기 위해 다른 통계 테스트를 사용할 수 있습니다.
결과
플로우 셀은 대물렌즈 투과 TIRF 이미징을 위한 레이저 조명이 장착된 도립 현미경에서 높은 개구수 오일 이멀젼 60x 배율 대물렌즈에 직접 연결됩니다(그림 5A). DNA 기질을 도입하고 과도한 DNA와 양자점을 씻어낸 후에는 일반적으로 시야에 수천 개의 개별 양자점이 있습니다(그림 5B). 이러한 양자점은 유리 표면에 안정적으로 부착되며 실험 시간 동안 눈에 띄는 디밍이나 심각한 표백을 겪지 않습니다. 그러나 마그네슘과 적절한 REase를 모두 포함하는 완충액이 유동 채널을 통해 흐르는 경우 일반적인 4분 관찰 시간이 끝날 때 실험 시작 시 존재하는 양자점의 최소 30%가 시야에서 사라집니다. 마그네슘 요구량을 확인하면, 마그네슘이 없을 때 REase가 채널을 통해 흐를 때 실험 시작 시 존재하는 양자점의 최소 95%는 관찰 기간이 끝날 때도 여전히 볼 수 있습니다(그림 5C). 그러나 마그네슘이 없는 상태에서 REase가 표면 결합 DNA에 결합하도록 한 후 마그네슘 함유 완충액을 흐르게 하면 관찰 기간이 끝날 때까지 양자점의 최대 절반이 사라지며(그림 5D), 이는 REase와 마그네슘이 채널을 통해 함께 흐를 때 관찰된 것과 유사합니다. 이벤트의 정확한 수율은 사용된 조건에서 효소의 효율성에 따라 달라집니다. 마그네슘 함유 완충액이 이전에 적절한 REase를 유동 채널에 도입하지 않고 유동되면 관찰 기간 동안 양자점의 5% 미만이 사라지고 이벤트의 히스토그램에 대해 식별할 수 있는 피크가 없습니다. 이 결과는 prebound REase 분자가 양자점을 유리 표면에 묶는 DNA의 절단을 매개하며, 이 DNA 절단이 이러한 실험에서 관찰된 양자점 소멸 사건의 대부분을 담당한다는 것을 나타냅니다.
양자점 소멸은 빠르게 발생하며 강도 궤적이 급격히 감소하여 주어진 DNA 분자가 절단되는 시간을 명확하게 표시합니다(그림 5E). 이벤트 시간의 결정은 강도 궤적의 수학적 분석을 통해 수행됩니다. 단일 양자점은 사용된 이미지 조건에서 배경보다 훨씬 더 높은 분산을 가진 강도 궤적을 생성하므로, 강도 궤적의 분산이 관찰된 강도 강하 후 배경의 분산과 유사한 수준으로 감소할 때 추정 이벤트가 확인됩니다. 또한, 추정 소멸 사건 이전에 많은 수준의 깜박임을 포함하는 궤적은 최종 분석에서 제외됩니다. 그러나 일반적인 실험에서는 이러한 기준을 충족하는 수백 개의 이벤트가 발생하며, 여러 실험의 결과를 취합하여 비선형 최소제곱 곡선 피팅 또는 최대우도 파라미터 추정을 통해 N 및 β 매개변수를 추정할 수 있는 적절한 통계를 제공할 수 있습니다.
제시된 대표 데이터는 잘 연구된 Type IIP REase, EcoRV(비디오 1)로 이 실험을 수행하여 수집되었습니다. 60bp 길이의 듀플렉스 DNA 기질은 듀플렉스 한 가닥의 5' 말단에 비오틴 분자가 있고 다른 가닥의 5' 말단에 공유 결합으로 부착된 양자점으로 구성됩니다. DNA 기질에는 하향 화살표(↓)로 표시된 것처럼 중간에서 EcoRV에 의해 절단되는 인식 서열 GAT↓ATC의 단일 복사본이 포함되어 있습니다. 마그네슘이 없는 상태에서 EcoRV를 DNA 기질에 미리 결합한 다음 마그네슘 함유 완충액을 흘려 DNA 절단을 시작했습니다. 대표 데이터에는 5개의 개별 실험의 통합 결과가 포함되어 있으며, 총 3451개의 관찰된 절단 이벤트가 산출됩니다. 0 시점 이전 또는 눈에 띄는 활동 피크 외부에서 발생한 이벤트를 제외한 후 2987개의 이벤트가 남았으며, 이는 히스토그램을 1초 빈으로 채우기에 충분했습니다. 비선형 최소제곱 곡선 피팅과 최대우도 파라미터 추정을 모두 사용하여 데이터에서 N과 β의 값을 추출했습니다. 비선형 최소제곱 피팅의 경우 N = 3.52(95% 신뢰 구간: 3.32-3.71) 및 β = 5.78초(95% 신뢰 구간: 5.41-6.15초), 최대 우도 추정의 경우 N = 3.41(95% 신뢰 구간: 3.25-3.58) 및 β = 6.06초(95% 신뢰 구간: 5.75-6.39초)로 두 매개변수 추정 방법이 일치했습니다(그림 6A).
EcoRV의 메커니즘에 대해 알려진 것을 감안할 때 최소 3개의 운동 단계를 추정하는 것이 합리적입니다. 이 효소는 마그네슘14가 없을 때 DNA를 비특이적으로 결합하는 것으로 알려져 있습니다. 정지 유동 조건에서 EcoRV에서 트립토판 잔기의 고유 형광 변화에 대한 벌크 용액 상 관찰은 마그네슘이 없는 상태에서 DNA에 결합된 EcoRV가 마그네슘이 활성 부위(18)에 진입하기 전에 구조적 변화를 겪어야 함을 시사했습니다. 이 관찰은 결정 구조 데이터19에 의해 확증됩니다. 트립토판 형광 연구의 결과는 또한 DNA 절단과 산물 방출이 유사한 속도로 발생하는 별개의 단계임을 나타낸다18. 따라서 이 실험에서 EcoRV 매개 DNA 절단에 대한 합리적인 반응 메커니즘은 다음과 같습니다.
ES → ES* → EP → E+P
이 메커니즘에서 초기 효소-기질 복합체인 ES는 마그네슘이 없을 때 EcoRV가 DNA에 결합될 때 형성됩니다. 마그네슘이 플로우 셀(flow cell)에 들어가면 효소-기질 복합체는 구조적 변화를 겪어 활성화된 효소-기질 복합체인 ES*가 됩니다. 이 활성화된 복합체는 DNA를 절단하지만 생성물 분자를 즉시 방출하지 않아 효소-제품 복합체(EP)가 됩니다. 마지막으로 제품 P가 마지막 단계에서 릴리스됩니다. 이 메커니즘에는 제시된 데이터와 일치하는 세 단계가 필요합니다. 각 단계의 평균 대기 시간에 대한 결과 추정치는 ~6초이며, 이는 각 단계에 대해 0.17초-1 의 속도에 해당합니다. 이 계산은 DNA 절단 및 산물 방출의 경우 0.3-0.4 s-1 정도로, 구조적 재배열을 위해 ~ 0.5 s-1 의 순서로 이러한 과정에 대한 속도의 이전 추정치와 일반적으로 일치합니다.

그림 1: 라벨링된 DNA 및 효소 반응 개략도. 양자 점 표지 DNA 분자는 비오틴-스트렙타비딘-비오틴 연결을 통해 기능화된 유리 표면에 연결되어 있으며 TIRF 현미경을 사용하여 관찰됩니다. DNA 분자에는 관심 REase에 대한 인식 부위가 포함되어 있습니다. DNA 분자가 REase에 의해 절단되면 양자점은 표면에서 조명 영역 밖으로 자유롭게 확산됩니다. 약어: REase = 제한 엔도뉴클레아제; PEG = 폴리에틸렌 글리콜; TIRF = 전반사 형광. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 미세유체 플로우 셀 장치. (A) 장치를 만드는 데 사용된 세 가지 레이어, 즉 하단의 기능성 유리 커버슬립, 상단에 입구 및 출구 구멍이 있는 석영 슬라이드, 중간에 채널이 절단된 양면 접착 이미징 스페이서를 보여주는 폭발도. (B) 폴리에틸렌 튜브가 제자리에 밀봉되어 있고 가장자리가 에폭시로 코팅된 완성된 장치. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
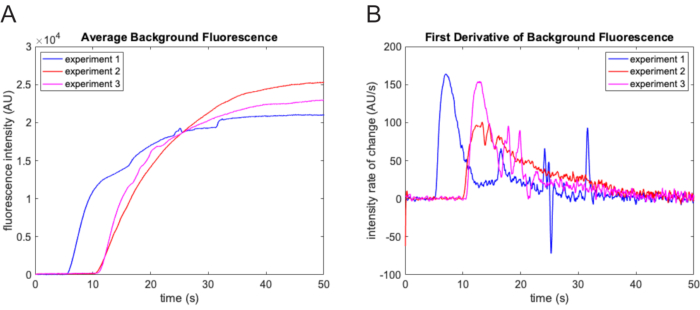
그림 3: 제로 시점 결정. (A) 동영상의 각 프레임에 대해 형태학적 top-hat 필터링 기능을 사용하여 결정된 평균 배경 강도. 배경 강도는 플루오레세인을 함유한 실험용 완충액이 시야에 들어올 때 현저하게 증가합니다. 세 가지 다른 실험의 결과가 다음과 같습니다. 실험마다 지연 시간에는 상당한 차이가 있습니다. (B) 배경 형광 강도 궤적의 변화율의 급격한 증가는 영점 점의 정확한 결정을 용이하게 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 배경 보정. 마그네슘의 도착을 나타내는 fluorescein 추적자 염료로 인한 배경 강도의 증가는 개별 양자점(회색 궤적)에 대한 원시 형광 강도 궤적에서 볼 수 있습니다. 형태학적 탑햇 필터링 기능을 사용하여 배경을 뺀 후, 트레이서 염료에 의해 도입된 아티팩트가 궤적(검은색 궤적)에서 제거되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: DNA 절단 이벤트의 병렬 관찰을 위한 단일 분자 TIRF 실험. (A) 양자 점 표지 DNA를 캡처하도록 설계된 기능성 유리 표면에 구성된 플로우 셀은 TIRF 이미징을 위한 높은 개구수 60x 오일 이멀젼 현미경 대물렌즈에 직접 연결됩니다. (B) 실험 시작 시점의 대표 이미지. 양자 점 표지 DNA가 플로우 셀에 로딩되었고, 과도하게 결합되지 않은 양자 점이 씻겨 나갔습니다. 개별 DNA 테더링 양자점은 서로 분리될 수 있습니다. (C) DNA 테더를 절단할 수 있는 REase를 포함하는 완충액이 마그네슘이 없는 상태에서 4분 동안 유동 채널을 통해 흐른 후 동일한 시야. DNA로 연결된 양자점의 유의미한 손실은 없었다. (D) 실험 종료 시 동일한 시야. 마그네슘 함유 완충액은 마그네슘이 없는 완충액에서 REase를 유동 한 직후 유동 채널을 통해 유동하였다. 이 이미지는 약 4분의 버퍼 흐름 후에 획득되었습니다. Prebound REase는 많은 DNA 테더를 절단하여 표면에서 양자점을 방출했습니다. 쉽게 볼 수 있도록 대물렌즈 시야의 중앙 사분면만 각 이미지에 표시됩니다. 양자점 점멸의 영향을 줄이기 위해 10개의 영화 프레임(관찰 시간 1초에 해당)을 평균화했습니다. 밝기 및 대비 설정은 세 이미지 모두에 대해 동일합니다. (E) 실험 시작 시 양자점이 존재했던 이미지 위치의 대표적인 형광 강도 궤적. 표면에서 방출되지 않은 양자점(회색)에 해당하는 이미지 위치에서 얻은 궤적은 낮은 강도 수준으로의 짧은 강하를 나타낼 수 있지만 높은 강도 수준에서 시작하고 끝납니다. 표면(검은색)에서 방출되는 양자점에 해당하는 이미지 위치에서 얻은 궤적은 실험의 시간 해상도(10Hz)와 관련하여 즉각적으로 낮은 배경 수준으로 강도가 급격히 감소하는 것을 나타냅니다. 방출된 양자점은 4분의 관찰 시간 내에 다시 나타나지 않습니다. 약어: REase = 제한 엔도뉴클레아제; TIRF = 전반사 형광. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 6: EcoRV 매개 DNA 절단의 체류 시간 분포 분석. (A) EcoRV를 사용한 5개의 통합 실험 세트에서 주 활동 피크 내에서 발생하는 2987개의 절단 이벤트에 대한 히스토그램 및 예측 엔벨로프. 예측된 두 곡선은 거의 동일하며, 피팅 잔차(히스토그램 아래)는 비선형 최소제곱 피팅의 체계적 오차를 나타내지 않습니다. (B) 0 시점 이후에 발생한 3393개의 분할 이벤트의 전체 세트에 대한 히스토그램. 감마 확률 분포가 히스토그램을 둘러싸지 못한다고 가정하는 매개변수(끊어지지 않은 선, MLE 곡선)의 최대 우도 추정에 의해 예측된 곡선입니다. 빈 높이(점선, NLS 곡선)에 대한 감마 확률 분포 공식의 비선형 최소제곱 피팅으로 예측된 곡선이 더 우수하지만 피팅의 잔차(히스토그램 아래)는 체계적인 오류를 나타냅니다. 약어: MLE: 최대우도 추정; NLS = 비선형 최소제곱. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 버퍼의 이름 | 구성 요소 | 농도 | 25 °C에서의 pH |
| 인산나트륨 완충액 | 인산나트륨 | 100 밀리미터 | 8.3 |
| CHES 버퍼 | N-시클로헥실-2-아미노에탄술폰산(CHES) | 10 밀리미터 | 9.0 |
| 증권 시세 표시기 | 인산나트륨 | 3의 mM | 7.2 - 7.6 |
| 소금 | 150 밀리미터 | ||
| 인산칼륨 | 1.05 밀리미터 | ||
| 스토리지 버퍼 | 소금 | 100 밀리미터 | 8.0 |
| 트리스-HCl | 50 mM | ||
| 소 혈청 알부민(BSA) | 0.5mg/mL | ||
| 중탄산나트륨 | 중탄산나트륨 | 100 밀리미터 | 8.2 |
| 블로킹 버퍼 | 트리스-HCl | 20의 mM | 7.5 |
| 에틸렌디아민테트라아세트산(EDTA) | 2의 mM | ||
| 소금 | 50 mM | ||
| Tween-20 (트윈-20) | 0.005% (v/v) | ||
| 소 혈청 알부민(BSA) | 0.2mg/mL | ||
| 마그네슘이 없는 실험용 완충액 | 소금 | 100 밀리미터 | 7.9 |
| 트리스-HCl | 50 mM | ||
| 디티오트레이톨(DTT) | 1 m엠 | ||
| 마그네슘이 함유된 실험용 완충액 | 소금 | 100 밀리미터 | 7.9 |
| 트리스-HCl | 50 mM | ||
| 염화 마그네슘 | 10 밀리미터 | ||
| 디티오트레이톨(DTT) | 1 m엠 | ||
| 플루오레세인 | 이미징 조건에 따라 조정 | ||
| DNase 완충액 | 염화 마그네슘 | 2.5 밀리미터 | 7.6 |
| 트리스-HCl | 10 밀리미터 | ||
| 염화칼슘 | 0.1 m엠 |
표 1: 버퍼 테이블.
비디오 1: 단일 분자 동영상의 예. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
토론
이 분석을 위한 DNA 기질은 sulfo-SMCC를 사용하는 2단계 반응 방식을 사용하여 양자점으로 표지됩니다. 이 이중작용 가교제는 일차 아민과 반응할 수 있는 NHS 에스테르 부분과 sulfhydryl기(20)와 반응할 수 있는 maleimide 부분으로 구성됩니다. 기판을 제조하는 데 사용되는 티올화 올리고뉴클레오티드는 산화된 형태로 배송됩니다. 커플링 절차를 진행하기 전에 설명된 대로 이들을 환원하고 정제하는 것이 중요하며, 그렇지 않으면 커플링 반응의 효율이 감소할 것입니다. 사용 직전에 새로운 DTT 및 sulfo-SMCC 용액을 준비하는 것이 중요합니다. 이 분자는 수용액에서 빠르게 가수분해됩니다. 용액을 투석 장치 안팎으로 옮길 때 주의하십시오 - 피펫 팁이 멤브레인에 닿으면 파열이 발생하고 샘플이 손실됩니다. 샘플 손실을 방지하기 위해 섹션 2의 모든 단계를 중단 없이 완료하는 것이 이상적입니다.
quantum-dot-labeled oligonucleotide를 생성하는 데 사용되는 커플링 반응은 비효율적입니다. 퀀텀-도트 라벨링된 올리고뉴클레오티드와 비오틴화된 올리고뉴클레오티드를 결합하고 어닐링한 후, 준비된 기질 샘플에 라벨링되지 않은 DNA가 과도하게 존재할 것입니다. 이러한 종은 기능화된 표면에 결합할 수 있지만 권장되는 이미징 조건에서는 보이지 않으며 실험 판독에 기여하지 않습니다. 완성된 구조체의 절단으로 인해 표면에서 양자점이 방출되는지 확인하려면 프로토콜의 섹션 5에 설명된 대로 표면 테더링을 수행하고 표면의 영역을 이미지화하여 얼마나 많은 양자점이 존재하는지 확인합니다. 그런 다음 DNase 완충액 (2.5mM MgCl 2; 10mM Tris-HCl, 0.1mM CaCl 2, pH 7.6)에서 DNase 완충액 (2.5mM MgCl2; 10mM Tris-HCl; 0.1mM CaCl2, pH 7.6)에서 800 μL의 DNase I을 유황시킵니다. DNase I을 채널에서 15분 동안 배양하고 800μL의 DNase 완충액(효소 제외)으로 세척한 다음 동일한 관심 영역을 이미징합니다. 양자점의 약 90%는 구조와 표면이 적절하게 준비된 경우 표면에서 방출되어야 합니다.
메톡시 종단 및 비오틴 종단 PEG 분자의 혼합물을 공유 결합하려면 유리 표면을 처리하여 이미징을 위해 표면의 양자 점 표지 DNA 기판을 캡처해야 합니다. 모든 청소 단계를 엄격하게 수행하는 것이 매우 중요합니다. 표면 불순물을 제거하고 유리 표면에 하이드록실기를 노출시켜 실란의 알콕시기와 반응하여 공유 결합을 형성하기 위해 여러 세척 단계가 필요합니다. 에탄올과 KOH 용액을 사용하여 세척 단계 사이에 초순수로 슬라이드를 헹구는 것이 중요하지만 아세톤으로 최종 세척한 후에는 물을 사용해서는 안 됩니다. 실란화 단계 동안 과도한 물의 존재는 표면 결합을 희생하는 대신 용액 내 실란의 다중화를 촉진합니다. 또한 실란 용액을 미리 준비해서는 안됩니다. 사용 수명을 연장하려면 실란 스톡을 건조기 캐비닛에 보관해야 합니다.
단일 분자 절단 분석은 미세유체 장치에서 수행되도록 설계되었습니다(그림 2). 각 실험 전에 새 장치를 제작하여 장치 상단을 형성하는 뚫린 석영 슬라이드를 재사용합니다. 이 절차는 이전에 설명되었으며 16,17 그 변형은 TIRF 현미경 검사에 널리 사용됩니다. 본원에 기술된 절차는 1인치 정사각형 슬라이드를 수용하도록 조정되었다. 기능화된 표면이 공기에 노출되는 동안 성능이 저하되지 않도록 장치를 신속하게 제작하는 것이 중요합니다. 에폭시는 형광성이므로 채널을 에폭시로 덮지 않도록 주의하십시오. 천공된 석영 슬라이드는 사용한 장치를 아세톤에 담그고 분해하여 재사용할 수 있습니다.
이 분석은 데이터 수집을 위해 EMCCD(electron-multiplying charge-coupled device) 카메라가 장착된 대물렌즈 전면 내부 반사 조명 기능이 있는 도립 현미경에서 수행하도록 설계되었습니다. 60x의 높은 개구수 대물렌즈와 488nm 레이저 여기 소스를 사용하고, 레이저에 적합한 single-bandpass emission filter와 single-edge dichroic이 포함된 Filter Cube를 사용하고, 양자점의 형광(655nm에서 방출 피크)과 적어도 일부 fluorescein 형광(512nm에서 방출 피크)이 모두 통과할 수 있는 방출 필터를 사용합니다. 예를 들어, 여기에 제시된 데이터는 482/18nm 단일 대역 통과 여기 필터, 488nm 단일 에지 이색성 및 675/67nm 단일 대역 통과 방출 필터가 장착된 필터 큐브로 수집되었습니다. 분석은 이 프로토콜에 설명된 대로 단일 스펙트럼 채널에서 이미징하여 수행할 수 있습니다. 원하는 경우, 형광 이미지를 두 개의 스펙트럼 채널로 분할하여 분석을 수행할 수도 있는데, 하나는 양자점용이고 다른 하나는 형광 방출용입니다. 640 nm single edge dichroic mirror가 장착된 image splitter가 이 목적에 적합합니다. 그러나 다중 채널 이미징은 필요하지 않습니다.
실험을 시작하기 전에 채널에서 연결되지 않은 DNA 기질을 씻어내는 것이 중요합니다. 플루오레세인은 최종 완충액에 50μM 농도로 포함되어 마그네슘이 시야에 도착했음을 알립니다. 현미경 설정에 따라 플루오레세인의 방출이 양자점을 가리지 않도록 플루오레세인의 농도를 조정해야 할 수도 있습니다. 마그네슘 함유 완충액의 흐름을 시작한 직후에 데이터 수집을 시작해야 합니다. 마그네슘 함유 완충액이 도착하기 전에 지연 시간 동안 데이터를 기록하여 배경 강도 변화율의 평균 및 표준 편차를 계산하여 제로 시점의 정확한 결정을 용이하게 할 수 있습니다. 가장 정확한 영점 시점은 배경 형광의 변화율이 이 측정의 자연적 변동을 초과하는 첫 번째 프레임입니다. 배경 형광 강도는 이 시점 이후에도 플루오레세인이 채널의 층류에 의해 설정된 경계층을 가로질러 확산됨에 따라 계속 증가합니다. 플루오레세인보다 훨씬 작은 마그네슘은 더 빨리 확산되고 그 농도는 훨씬 더 빨리 평형에 도달할 것으로 예상할 수 있습니다.
제시된 예제 데이터의 경우, 0 시점 이후에 발생한 모든 3393 이벤트의 히스토그램은 실험의 처음 60초 이내에 두드러진 피크를 표시합니다. 지연 시간이 더 긴 많은 bin에는 이벤트가 포함되지 않으며, events가 포함된 bin에는 활성 REase가 없을 때 관찰되는 것과 유사하게 몇 개만 포함됩니다. 모든 비닝된 데이터에 대한 감마 확률 분포의 비선형 최소제곱 곡선 피팅은 95% 신뢰 구간이 2.94-3.18이고 β = 7.04초이며 95% 신뢰 구간이 6.72-7.36초입니다. 그러나 매개변수에 대한 최대 우도 추정치는 N = 1.30(95% 신뢰 구간: 1.24-1.35) 및 β = 26.7초(95% 신뢰 구간: 25.3-28.1초)였으며 육안 검사에 의해 최대 우도 매개변수 추정치에 의해 예측된 곡선은 데이터와 잘 일치하지 않습니다(그림 6B). 또한, 비선형 최소제곱 피팅에 의해 추정된 매개변수에 의해 예측된 곡선은 관측된 이벤트 빈도의 히스토그램 모양을 비교적 잘 따르지만, 잔차(관측된 이벤트 빈도와 예측된 이벤트 빈도 간의 차이)는 약 60초 이후의 시점에 대해 일관되게 양수가 됩니다(그림 6B).
이후 시점에서의 사건 빈도는 활성 효소가 없을 때 관찰되는 양자점 소멸 빈도와 비슷하기 때문에, 이러한 후기 사건의 대부분은 REase 매개 DNA 절단에 의해 발생하지 않는 낮은 수준의 양자점 손실의 일부일 가능성이 높습니다. 따라서 이러한 빈을 피팅에서 제외하는 것이 합리적입니다. 각 실험의 제로 시점 이후 60초 이상 발생한 모든 이벤트를 제외했을 때 두 파라미터 추정 방법이 일치했습니다(그림 6A). 따라서 최상의 모수 추정치를 얻으려면 활동 피크 범위를 벗어난 이벤트를 제외하고 최대우도 모수 추정치와 비선형 최소제곱 곡선 적합 결과가 수렴했는지 확인하십시오.
이 논문에 설명된 실험적 접근 방식은 단일 분자 수준에서 REase 매개 DNA 절단을 실시간으로 관찰할 수 있습니다. 각 개별 실험에서 수백 개의 이벤트를 관찰할 수 있으며, 여러 실험의 결과를 취합하여 데이터를 감마 확률 분포에 적합시켜 대기 시간 분포를 특성화하는 데 필요한 많은 수의 관측치를 얻을 수 있습니다. 이 분석은 마그네슘 또는 다른 보조 인자가 없는 경우 DNA와 결합하지만 절단하지 않는 REase에 대해서만 설명된 대로 사용할 수 있지만 많은 REase가 이 범주에 속합니다. 이 분석으로 모든 중간 단계의 정확한 세부 사항이 드러나지 않는다는 점을 인식하는 것이 중요합니다. 속도 제한 단계보다 훨씬 빠른 단계는 체류 시간 분포에 영향을 미치지 않으며 데이터(12)에서 추출된 N의 값을 계산하지 않습니다. 마찬가지로, 데이터에서 추출할 수 있는 β의 값은 체류 시간 분포에 영향을 주는 단계에 대한 대기 시간의 대략적인 평균입니다. 각 단계의 실제 대기 시간은 β12의 값과 다를 수 있습니다. 그러나 이 논문에 설명된 단일 분자 절단 분석은 반응 중간체 자체를 직접 관찰할 수 없더라도 반응 경로를 따라 여러 중간 단계가 존재한다는 증거를 제공할 수 있습니다.
공개
저자는 경쟁하는 재정적 이익이나 기타 이해 상충이 없습니다
감사의 말
이 연구는 미국 국립종합의학연구소(National Institute of General Medical Sciences)로부터 CME에 수여된 Award Number K12GM074869의 지원을 받았습니다. 내용은 전적으로 저자의 책임이며 National Institute of General Medical Sciences 또는 National Institutes of Health의 공식 견해를 반드시 나타내는 것은 아닙니다.
자료
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltriethoxysilane (APTS) | Sigma Aldrich | 440140-100ML | Store in dessicator box |
| 5 Minute Epoxy | Devcon | 20845 | use for sealing the microfluidic device |
| acetone | Pharmco | 329000ACS | use for cleaning coverslips |
| bath sonicator | Fisher Scientific | CPXH Model 2800 | catalog number 15-337-410 |
| Beaker, glass, 100 mL | |||
| Benchtop centrifuge | |||
| Biotin-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | BIO-SVA-5K | Succinimidyl valerate has a longer half-life than succinimidyl carbonate |
| biotinylated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Biotin modification and HPLC purification |
| Bovine Serum Albumin (BSA) | VWR | 0903-5G | prepare a 10 mg/mL solution (aq) and heat to 95 °C before using |
| Centri-Spin-10 Size Exclusion Spin Columns | Princeton Separations | CS-100 or CS-101 | used to purify thiolated oligonucleotides after reducing the disulfide bond |
| Centrifuge tubes 1.5. mL | |||
| Coverslips, 1-inch square glass | |||
| coverslip holders | |||
| diamond point wheel | Dremel | 7134 | use for drilling holes in quartz flow cell topper |
| dithiothreitol (DTT) | Thermo Scientific | A39255 | No-Weigh Format, 7.7 mg/vial |
| drill press rotary tool workstation stand | Dremel | 220-01 | facilitates quartz drilling |
| EcoRV (REase used to generate example data) | New England Biolabs | R0195T or R0195M | Use 100,000 units/mL stock to avoid adding excess glycerol Check REBASE for suppliers of other REases |
| ethanol | various | CAS 64-17-5 | denatured or 95% are acceptable, use for cleaning coverslips |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | EDS | BioUltra, anhydrous, store in dessicator box |
| Flea Micro Spinbar | Fisherbrand | 14-513-65 | 3 mm x 10 mm size to fit beneath coverslip rack |
| fluorescein | Acros Organics | 17324 | use to make experimental buffers |
| gravity convection oven | Binder | 9010-0131 | |
| handheld rotary multitool | Dremel | 8220 | use for drilling holes in quartz flow cell topper |
| ImagEM X2 EM-CCD Camera | Hamamatsu | C9100-23B | air cooling is adequate for this experiment, use HCImage software or similar to control |
| Imaging spacer, double-sided, adhesive | |||
| Jar, glass with screw cap, (approximately 50 mm diameter by 50 mm high) | |||
| magnesium chloride hexahydrate | Fisher Bioreagents | BP214-500 | use to make experimental buffer with magnesium |
| MATLAB software | Data analysis | ||
| metal tweezers | Fisher Brand | 16-100-110 | |
| methoxy-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | M-SVA-5K | Both PEGs should have the same NHS ester so that the rate of reaction is consistent |
| microcentrifuge | Eppendorf | 5424 | |
| multiposition magnetic stirrer | VWR | 12621-022 | |
| N-cyclohexyl-2-aminoethanesulfonic acid (CHES) | Acros Organics | AC20818 | CAS 103-47-9, use to make CHES buffer |
| orbital shaker and heater for microcentrifuge tubes | Q Instruments | 1808-0506 | with 1808-1061 adaptor for 24 x 2.0 mL or 15 x 0.5 mL tubes |
| Parafilm | |||
| PE60 Polyethylene tubing (inner diameter 0.76 mm, outer diameter 1.22 mm) | Intramedic | 6258917 | 22 G blunt needles are a good fit for this tubing size |
| Phosphate-Buffered Saline (PBS) 10x | Sigma Aldrich | P7059 | Use at 1x strength |
| potassium hydroxide | VWR Chemicals BDH | BDH9262 | use a 1 M solution to clean coverslips |
| Qdot 655 ITK Amino (PEG) Quantum Dots | Invitrogen | Q21521MP | |
| Quartz Slide, 1 inch square, 1 mm thick | Electron Microscopy Sciences | 72250-10 | holes must be drilled in the corners for inlet and outlet tubing insertion |
| reinforced plastic tweezers | Rubis | K35a | use for handling coverslips and building microfluidic device |
| SecureSeal Adhesive Sheets | Grace Biolabs | SA-S-1L | cut to form spacer for microfluidic device |
| Single channel syringe pump for microfluidics | New Era Pump Systems | NE-1002X-US | fitted with a 50 mL syringe and a 22 G blunt needle |
| Slide-a-Lyzer MINI Dialysis Devices, 10 kDa MWCO, 0.1 mL | Thermo Scientific | 69570 or 69572 | used for buffer exchange during quantum dot coupling to DNA |
| sodium bicarbonate | EMD Millipore | SX0320 | use to make buffer for surface functionalization; 100 mM, pH 8 |
| sodium chloride | Macron | 7581-12 | use to make experimental buffers |
| Sodium phosphate dibasic solution (BioUltra, 0.5 M in water) | Sigma Aldrich | 94046 | use to make 100 mM sodium phosphate buffer |
| Sodium phosphate monobasic solution (BioUltra, 5M in water) | Sigma Aldrich | 74092 | use to adjust pH of 100 mM sodium phosphate buffer |
| Streptavidin from Streptomyces avidinii | Sigma Aldrich | S4762 | dissolve at 1 mg/mL and store 25 mL aliqouts at -20 ? |
| Sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexane-1-carboxylate (sulfo-SMCC) | Thermo Scientific | A39268 | No-Weigh Format, 2 mg/vial |
| Syringe fitted with blunt 21 G needle | |||
| Syringe pump | |||
| thiolated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Thiol Modifier C6 S-S and HPLC purificaiton |
| TIRF imaging system with 488 nm laser illumination | various | custom built | |
| Tris -HCl | Research Products International | T60050 | use to make experimental buffers |
| Tris base | JT Baker | 4101 | use to make experimental buffers |
| Tween-20 | Sigma | P7949 | use to make blocking buffer |
| Ultrapure water | |||
| vortex mixer | VWR | 10153-842 | |
| Wash-N-Dry Coverslip Rack | Electron Microscopy Sciences | 70366-16 | used for surface functionalization of coverslips |
참고문헌
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5905-5908 (2005).
- Loenen, W. A., Dryden, D. T., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Research. 42 (1), 3-19 (2014).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE--a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research. 43, Database issue 298-299 (2015).
- Bonnet, I., et al. Sliding and jumping of single EcoRV restriction enzymes on non-cognate DNA. Nucleic Acids Research. 36 (12), 4118-4127 (2008).
- Biebricher, A., Wende, W., Escude, C., Pingoud, A., Desbiolles, P. Tracking of single quantum dot labeled EcoRV sliding along DNA manipulated by double optical tweezers. Biophysical Journal. 96 (8), 50-52 (2009).
- Blainey, P. C., et al. Nonspecifically bound proteins spin while diffusing along DNA. Nature Structural & Molecular Biology. 16 (12), 1224-1229 (2009).
- Huang, C. -F., et al. Direct visualization of DNA recognition by restriction endonuclease EcoRI. Journal of Experimental & Clinical Medicine. 5 (1), 25-29 (2013).
- Palma, M., et al. Selective biomolecular nanoarrays for parallel single-molecule investigations. Journal of American Chemical Society. 133 (20), 7656-7659 (2011).
- Reinhard, B. M., Sheikholeslami, S., Mastroianni, A., Alivisatos, A. P., Liphardt, J. Use of plasmon coupling to reveal the dynamics of DNA bending and cleavage by single EcoRV restriction enzymes. Proceedings of the National Academy of Sciences of the United States of America. 104 (8), 2667-2672 (2007).
- van den Broek, B., Noom, M. C., Wuite, G. J. DNA-tension dependence of restriction enzyme activity reveals mechanochemical properties of the reaction pathway. Nucleic Acids Research. 33 (8), 2676-2684 (2005).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Loparo, J. J., van Oijen, A. Single-molecule enzymology. Handbook of Single-Molecule Biophysics. Hinterdorfer, P., Oijen, A. , Springer. New York, NY. 165-182 (2009).
- Taylor, J. D., Badcoe, I. G., Clarke, A. R., Halford, S. E. EcoRV restriction endonuclease binds all DNA sequences with equal affinity. Biochemistry. 30 (36), 8743-8753 (1991).
- Pingoud, A., Jeltsch, A. Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29 (18), 3705-3727 (2001).
- Tanner, N. A., Loparo, J. J., van Oijen, A. M. Visualizing single-molecule DNA replication with fluorescence microscopy. Journal of Visualized Experiments: JoVE. (32), e1529(2009).
- Kulczyk, A. W., Tanner, N. A., Loparo, J. J., Richardson, C. C., van Oijen, A. M. Direct observation of enzymes replicating DNA using a single-molecule DNA stretching assay. Journal of Visualized Experiments: JoVE. (37), e1689(2010).
- Baldwin, G. S., Vipond, I. B., Halford, S. E. Rapid reaction analysis of the catalytic cycle of the EcoRV restriction endonuclease. Biochemistry. 34 (2), 705-714 (1995).
- Winkler, F. K., et al. The crystal-structure of Ecorv endonuclease and of Its complexes with cognate and non-cognate DNA fragments. EMBO Journal. 12 (5), 1781-1795 (1993).
- Sioss, J. A., Stoermer, R. L., Sha, M. Y., Keating, C. D. Silica-coated, Au/Ag striped nanowires for bioanalysis. Langmuir. 23 (22), 11334-11341 (2007).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유