Method Article
Einzelmolekül-Verweilzeitanalyse der Restriktionsendonuklilease-vermittelten DNA-Spaltung
In diesem Artikel
Zusammenfassung
Mit Hilfe von Quantenpunkt-markierter DNA und Totalreflexionsfluoreszenzmikroskopie können wir den Reaktionsmechanismus von Restriktionsendonukleasen untersuchen, während wir unmarkierte Proteine verwenden. Diese Einzelmolekültechnik ermöglicht die massiv gemultiplexte Beobachtung einzelner Protein-DNA-Wechselwirkungen, und Daten können gepoolt werden, um gut bevölkerte Verweilzeitverteilungen zu erzeugen.
Zusammenfassung
Dieser neuartige, auf der Totalreflexionsfluoreszenzmikroskopie basierende Assay ermöglicht die gleichzeitige Messung der Länge des katalytischen Zyklus für Hunderte von einzelnen Restriktionsendonuklease (REase)-Molekülen in einem Experiment. Dieser Assay erfordert keine Proteinmarkierung und kann mit einem einzigen Bildgebungskanal durchgeführt werden. Darüber hinaus können die Ergebnisse mehrerer Einzelexperimente gepoolt werden, um gut gefüllte Verweilzeitverteilungen zu erzeugen. Die Analyse der resultierenden Verweilzeitverteilungen kann helfen, den DNA-Spaltungsmechanismus aufzuklären, indem sie das Vorhandensein von kinetischen Schritten aufdeckt, die nicht direkt beobachtet werden können. Beispieldaten, die mit diesem Assay mit der gut untersuchten REase EcoRV - einer dimeren Typ-IIP-Restriktionsendonuklease, die die palindromische Sequenz GAT↓ATC (wobei ↓ die Schnittstelle ist) - gesammelt wurden, stimmen mit früheren Studien überein. Diese Ergebnisse deuten darauf hin, dass es mindestens drei Schritte auf dem Weg zur DNA-Spaltung gibt, der durch die Einführung von Magnesium eingeleitet wird, nachdem EcoRV die DNA in deren Abwesenheit gebunden hat, mit einer durchschnittlichen Rate von 0,17 s-1 für jeden Schritt.
Einleitung
Restriktionsendonukleasen (REasen) sind Enzyme, die sequenzspezifische Doppelstrangbrüche in der DNA bewirken. Die Entdeckung von REasen in den 1970er Jahren führte zur Entwicklung der rekombinanten DNA-Technologie, und diese Enzyme sind heute unverzichtbare Laborwerkzeuge für die genetische Veränderung und Manipulation1. Typ-II-REasen sind die am weitesten verbreiteten Enzyme dieser Klasse, da sie die DNA an einer festen Stelle entweder innerhalb oder in der Nähe ihrer Erkennungssequenz spalten. Es gibt jedoch eine große Variation zwischen den Typ-II-REasen, und sie werden in mehrere Subtypen unterteilt, die auf bestimmten enzymatischen Eigenschaften basieren, anstatt nach ihren evolutionären Beziehungen klassifiziert zu werden. Bei jedem Subtyp gibt es häufige Ausnahmen vom Klassifikationsschema, und viele Enzyme gehören zu mehreren Subtypen2. Tausende von Typ-II-REasen wurden identifiziert, und Hunderte von ihnen sind kommerziell erhältlich.
Trotz der Vielfalt unter den Typ-II-REasen wurden jedoch nur sehr wenige REasen im Detail untersucht. Laut REBASE, der von Sir Richard Roberts 1975 gegründeten Datenbank für Restriktionsenzyme3, liegen für weniger als 20 dieser Enzyme veröffentlichte Kinetikdaten vor. Während einige REasen direkt auf Einzelmolekülebene beobachtet wurden, während sie entlang der DNA diffundierten, bevor sie auf ihre Erkennungssequenztrafen 4,5,6,7, gibt es nur sehr wenige Einzelmolekülstudien über ihre Spaltreaktionskinetik. Die vorhandenen Studien liefern entweder keine ausreichenden Statistiken, um eine detaillierte Analyse der Variation der Zeitpunkte durchzuführen, zu denen einzelne Spaltungsereignisse stattfinden 8,9,10, oder sie sind nicht in der Lage, die vollständige Verteilung der Spaltungszeitenzu erfassen 11. Diese Art der Analyse kann das Vorhandensein von relativ langlebigen kinetischen Zwischenprodukten aufdecken und könnte zu einem besseren Verständnis der Mechanismen der REase-vermittelten DNA-Spaltung führen.
Auf der Ebene der Einzelmoleküle sind biochemische Prozesse stochastisch - die Wartezeit auf das Auftreten einer einzelnen Instanz des Prozesses, τ, ist variabel. Bei vielen Messungen von τ kann jedoch erwartet werden, dass sie einer Wahrscheinlichkeitsverteilung (p(τ) folgen, die auf die Art des stattfindenden Prozesses hinweist. Zum Beispiel gehorcht ein einstufiger Prozess, wie z. B. die Freisetzung eines Produktmoleküls aus einem Enzym, der Poisson-Statistik, und p(τ) nimmt die Form einer negativen Exponentialverteilung an:
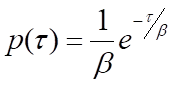
wobei β die mittlere Wartezeit ist. Beachten Sie, dass die Geschwindigkeit des Prozesses k gleich 1/β ist, dem Kehrwert der mittleren Wartezeit. Bei Prozessen, die mehr als einen Schritt erfordern, ist p(τ) die Faltung der einfachen Exponentialverteilungen für jeden der einzelnen Schritte. Eine allgemeine Lösung für die Faltung von N einfach-exponentiellen Zerfallsfunktionen mit identischen mittleren Wartezeiten, β, ist die Gamma-Wahrscheinlichkeitsverteilung:

Dabei ist Γ(N) die Gamma-Funktion, die die Interpolation der Fakultät von N-1 zu nicht-ganzzahligen Werten von N beschreibt. Obwohl diese allgemeine Lösung als Annäherung verwendet werden kann, wenn die mittleren Wartezeiten einzelner Schritte ähnlich sind, muss verstanden werden, dass das Vorhandensein relativ schneller Schritte durch Schritte mit deutlich längeren Wartezeiten maskiert wird. Mit anderen Worten, der Wert von N stellt eine untere Grenze für die Anzahl der Schritte12 dar. Bei einer ausreichenden Anzahl von Wartezeitmessungen können die Parameter β und N geschätzt werden, indem die Ereignisse klassifiziert und die Gammaverteilung an das resultierende Histogramm angepasst wird, oder indem ein Maximum-Likelihood-Schätzansatz verwendet wird. Diese Art der Analyse kann daher das Vorhandensein von kinetischen Schritten aufdecken, die in Ensemble-Assays nicht leicht aufgelöst werden können und eine große Anzahl von Beobachtungen erfordern, um die Parameter genau zu schätzen12,13.
In diesem Artikel wird eine Methode beschrieben, mit der mit Quantenpunkten markierte DNA und Totalreflexionsfluoreszenzmikroskopie (TIRF) Hunderte von einzelnen REase-vermittelten DNA-Spaltungsereignissen parallel beobachtet werden können. Das Design des Assays ermöglicht es, die Ergebnisse mehrerer Experimente zu bündeln und Verweilzeitverteilungen mit Tausenden von Ereignissen zu erstellen. Die hohe Photostabilität und Helligkeit der Quantenpunkte ermöglichen eine Zeitauflösung von 10 Hz, ohne die Fähigkeit zu beeinträchtigen, Spaltungsereignisse zu beobachten, die auch viele Minuten nach dem Start des Experiments auftreten. Die gute zeitliche Auflösung und ein breiter dynamischer Bereich, kombiniert mit der Möglichkeit, einen großen Datensatz zu sammeln, ermöglichen eine genaue Charakterisierung der Verweilzeitverteilungen, um das Vorhandensein mehrerer kinetischer Schritte in den Spaltungswegen von REasen aufzudecken, die Umsatzraten im Bereich von 1 min-1 aufweisen. Im Fall von EcoRV können drei kinetische Schritte aufgelöst werden, die alle auf andere Weise identifiziert wurden, was bestätigt, dass der Assay empfindlich auf das Vorhandensein solcher Schritte reagiert.
Duplex-DNA-Substrate, die die interessierende Erkennungssequenz enthalten, werden hergestellt, indem ein biotinyliertes Oligonukleotid an einen komplementären Strang geglüht wird, der mit einem einzelnen, kovalent gebundenen Halbleiter-Nanokristall-Quantenpunkt markiert ist. Diese Substrate werden in einen Strömungskanal eingebracht, der auf einem Glasdeckglas aufgebaut ist, an dessen Oberfläche ein Rasen aus hochmolekularen Polyethylenglykol (PEG)-Molekülen befestigt ist. Die DNA-Substrate werden über eine Biotin-Streptavidin-Biotin-Bindung von einem Teil der PEG-Moleküle eingefangen, die an ihrem freien Ende ein Biotin haben. In der TIRF-Mikroskopie sorgt eine evaneszente Welle, die exponentiell mit zunehmender Entfernung von der Glas-Flüssig-Grenzfläche abklingt, für Beleuchtung; Die Eindringtiefe liegt in der Größenordnung der Wellenlänge des verwendeten Lichts. Unter diesen Bedingungen werden nur Quantenpunkte angeregt, die durch ein DNA-Molekül an die Oberfläche gebunden sind, das auf der funktionalisierten Glasoberfläche eingefangen wurde. Quantenpunkte, die in Lösung frei sind, werden innerhalb des beleuchteten Bereichs nicht eingeschränkt und leuchten daher nicht. Wenn die DNA, die einen Quantenpunkt an die Oberfläche bindet, gespalten wird, kann dieser Quantenpunkt frei von der Oberfläche weg diffundieren und verschwindet aus dem Fluoreszenzbild.
Obwohl bekannt ist, dass viele Typ-II-REasen in Abwesenheit vonMagnesium 14 DNA binden, benötigen alle Magnesium, um die DNA-Spaltungzu vermitteln 15. Diese REasen können die oberflächenimmobilisierte DNA in Abwesenheit von Magnesium binden. Wenn magnesiumhaltiger Puffer durch einen Kanal fließt, in dem REase an die DNA vorgebunden ist, beginnt die Spaltung sofort, was durch das Verschwinden der Quantenpunkte angezeigt wird. Die Synchronisation, die durch die Vorbindung der REase-Moleküle und die anschließende Initiierung der DNA-Spaltung durch die Einführung von Magnesium erreicht wird, erleichtert die Messung der Verzögerungszeit bis zum Abschluss der DNA-Spaltung unabhängig für jedes Molekül in der Population der Enzyme, die in einem Experiment beobachtet wurden. Fluorescein ist als Tracerfarbstoff in dem magnesiumhaltigen Puffer enthalten, um die Ankunft von Magnesium im Sichtfeld anzuzeigen. Da kein Enzym in dem magnesiumhaltigen Puffer enthalten ist, gibt die Verzögerungszeit von der Ankunft des magnesiumhaltigen Puffers bis zum Verschwinden jedes Quantenpunkts die Zeit an, die eine bereits an die DNA gebundene REase benötigt, um die DNA zu spalten und den Quantenpunkt von der Glasoberfläche freizusetzen. Das Verschwinden von Quantenpunkten geschieht schnell und führt zu einer starken Abnahme der Intensitätskurve, was einen klaren Hinweis auf den Zeitpunkt gibt, zu dem ein bestimmtes DNA-Molekül gespalten wird. Die Bestimmung der Ereigniszeiten erfolgt durch die mathematische Analyse von Intensitätsverläufen, und ein typisches Experiment führt zu Hunderten von identifizierbaren Spaltungsereignissen. Die Ergebnisse mehrerer Experimente können zusammengefasst werden, um angemessene Statistiken zu liefern, die eine Schätzung der Parameter N und β entweder durch nichtlineare Analysen der kleinsten Quadrate oder durch Maximum-Likelihood-Analysen ermöglichen.
Protokoll
1. Allgemeine Informationen
- Oligonukleotid-Design
HINWEIS: Das 60 Basenpaare (bp) lange DNA-Substrat wird aus einem Paar komplementärer Oligonukleotide mit einer Duplex-Schmelztemperatur von 75 °C in 100 mM NaCl gebildet.- Bestellen Sie ein Oligonukleotid, das mit einer einzelnen 5'-Biotin-Modifikation synthetisiert wird, und das andere mit einer 5'-Thiol-Modifikation (mit einem Sechs-Kohlenstoff-Spacer). Platzieren Sie die Erkennungsstelle in der Mitte des Duplexbereichs.
HINWEIS: Die Oligonukleotidsequenzen zur Verwendung mit EcoRV sind unten dargestellt (Erkennungsstelle in Fett).
5' Biotin - AAA ACC GAC ATG TTG ATT TCC TGA AAC GGG ATA TCA TCA AAG CCA TGA ACA AAG CAG CCG - 3'
5' Thiol - CGG CTG CTT TGT TCA TGG CTT TGA TGA TAT CCC GTT TCA GGA AAT CAA CAT GTC GGT TTT - 3'
- Bestellen Sie ein Oligonukleotid, das mit einer einzelnen 5'-Biotin-Modifikation synthetisiert wird, und das andere mit einer 5'-Thiol-Modifikation (mit einem Sechs-Kohlenstoff-Spacer). Platzieren Sie die Erkennungsstelle in der Mitte des Duplexbereichs.
- Verwenden Sie für alle Schritte Reinstwasser mit einem spezifischen Widerstand von 18 MOhm.
- Schützen Sie alle Lösungen, die Quantenpunkte enthalten, vor Licht, um Photobleiche zu verhindern.
- Verwenden Sie eine Druckluftquelle, um dieses Protokoll abzuschließen.
2. Herstellung von Quantenpunkt-markierten DNA-Substratmaterialien
ANMERKUNG: Neben den oben beschriebenen Oligonukleotiden siehe Materialtabelle für andere Materialien und Tabelle 1 für Puffer, die für die Herstellung von Quantenpunkt-markierten DNA-Substraten erforderlich sind.
- Reduzieren Sie 5'-Thiolgruppen auf dem Oligonukleotid, das an Quantenpunkte gekoppelt werden soll.
- Jedes thiolierte Oligonukleotid wird in Wasser in einer Konzentration von 100 μM resuspendiert.
- Pipettieren Sie 650 μl Wasser in eine Spin-Säule mit Größenausschluss für jede Oligonukleotidprobe und wirbeln Sie sie ~15 s lang ein. Lassen Sie die Säule 30 Minuten lang packen.
- Bereiten Sie unmittelbar vor jedem Gebrauch frische 100 mM Dithiothreitol (DTT)-Lösung vor, da DTT in Lösung schnell abgebaut wird. Öffnen Sie vorsichtig eine Durchstechflasche mit 7,7 mg DVB-T und pipettieren Sie 500 μl Natriumphosphatpuffer in die Durchstechflasche. Vortex, um gründlich zu mischen.
- Für jedes resuspendierte Oligonukleotid wird ein frisches Röhrchen vorbereitet und 50 μl des Oligonukleotids und 50 μl DTT-Lösung in das Röhrchen gegeben. Zum Mischen nach oben und unten pipettieren. 30 min bei Raumtemperatur inkubieren, um die Disulfidbindungen zwischen den oxidierten Thiolgruppen am 5'-Ende der Oligonukleotide zu reduzieren.
- Entfernen Sie die Spin-Säulenkappen und setzen Sie jede Säule in das Auffangröhrchen ein. Die Spin-Säulen werden 2 Minuten lang bei 750 × g zentrifugiert und das Eluat verworfen.
- Übertragen Sie die Spin-Säulen in frische Zentrifugenröhrchen. Das gesamte Volumen (100 μl) einer Probe des DNA/DTT-Gemisches wird vorsichtig auf jede vorbereitete Säule pipetiert. Zentrifugieren Sie 2 Minuten lang bei 750 × g und messen Sie die Extinktion des Eluats bei 260 nm, um zu bestätigen, dass die Konzentration etwa 40 μM beträgt.
- Proben, die nicht sofort verwendet werden, sind bei -20 °C zu lagern, um die Oxidation der Thiolgruppen und die Bildung von Disulfidbindungen zu verhindern.
- Kopplung von DNA an Quantenpunkte
- Pipettieren Sie für jedes herzustellende Konstrukt ein Aliquot von 50 μl des Quantenpunktstocks in ein Dialysegerät, wobei Sie darauf achten müssen, die Membran nicht mit der Pipettenspitze zu berühren. Dialysieren Sie gegen ein Volumen von N-Cyclohexyl-2-aminoethansulfonsäure (CHES)-Puffer, das mindestens das 1000-fache des Probenvolumens beträgt, und rühren Sie 15 Minuten lang bei ~100 U/min.
- Bereiten Sie unmittelbar vor jedem Gebrauch eine frische 6 mM Lösung von Sulfosuccinimidyl-4-(N-maleimidomethyl)-cyclohexan-1-carboxylat (Sulfo-SMCC) in CHES-Puffer vor. Öffnen Sie vorsichtig eine Durchstechflasche mit 2 mg Sulfo-SMCC und pipettieren Sie 800 μl CHES-Puffer in die Durchstechflasche. Vortex, um gründlich zu mischen.
- Entnehmen Sie mit einer Pipette vorsichtig die suspendierten Quantenpunkte aus dem Dialysegerät und überführen Sie sie in ein frisches Röhrchen, das das gleiche Volumen der Sulfo-SMCC-Lösung enthält. Zum Mischen nach oben und unten pipettieren. 1 h bei Raumtemperatur unter Schütteln bei 1000 U/min inkubieren, damit das Sulfo-SMCC mit den primären Aminen auf den Quantenpunkten reagieren kann.
- Übertragen Sie jede Probe mit einer Pipette vorsichtig in ein frisches Dialysegerät. Um überschüssiges Sulfo-SMCC zu entfernen, dialysieren Sie 15 Minuten lang unter Rühren gegen ein Volumen des CHES-Puffers, das mindestens dem 1000-fachen des in dem/den Dialysegerät(en) enthaltenen Volumens entspricht. Tauschen Sie den Puffer 2x aus und führen Sie die Dialyse mit frischem CHES-Puffer insgesamt 3x durch, so dass die Dialyse 15 Minuten nach jedem Pufferwechsel fortgesetzt werden kann.
- Um den Puffer zur Vorbereitung der zweiten Reaktion auszutauschen, übertragen Sie die Dialysegeräte in ein Becherglas mit einem Volumen an phosphatgepufferter Kochsalzlösung (PBS), das mindestens dem 1000-fachen des in den Geräten enthaltenen Volumens entspricht. Dialysieren Sie 15 Minuten lang unter Rühren, tauschen Sie den Puffer 2x aus und führen Sie die Dialyse mit frischem PBS insgesamt 3x durch, so dass die Dialyse nach jedem Pufferwechsel 15 Minuten lang fortgesetzt werden kann.
- Gewinnen Sie mit einer Pipette vorsichtig die Lösung mit den Quantenpunkten aus dem/den Dialysegerät(en) und überführen Sie jede Probe in ein frisches Röhrchen, das eine äquimolare Menge an thioliertem Oligonukleotid enthält, das in PBS verdünnt ist. Kombinieren Sie ein gleiches Volumen PBS und ein1 /10 des Volumens der reduzierten und gereinigten Oligonukleotidprobe (ca. 40 μM Konzentration), da die Quantenpunktkonzentration zu diesem Zeitpunkt ~4 μM beträgt. 2 h bei Raumtemperatur inkubieren und bei 1000 U/min schütteln.
- Jede Probe wird mit Rinderserumalbumin (BSA, 10 mg/ml) versorgt, um eine BSA-Endkonzentration von 0,5 mg/ml zu erhalten. Übertragen Sie jede Probe vorsichtig mit einer Pipette in ein frisches Dialysegerät und dialysieren Sie gegen ein Volumen des Speicherpuffers, das mindestens dem 1000-fachen des in dem/den Dialysegerät(en) enthaltenen Volumens entspricht. Tauschen Sie den Puffer 2x aus und führen Sie die Dialyse mit frischem Lagerpuffer insgesamt 3x durch, so dass die Dialyse nach jedem Pufferwechsel 15 Minuten lang fortgesetzt werden kann.
- Entnehmen Sie jede Probe vorsichtig mit einer Pipette, geben Sie sie in ein frisches Röhrchen und lagern Sie sie bei 4 °C.
HINWEIS: Nicht bei -20 °C lagern, da Quantenpunkte durch Einfrieren beschädigt werden.
- Annealing von quantenpunktmarkiertem Oligonukleotid zu biotinyliertem Oligonukleotid
- Jedes biotinylierte Oligonukleotid wird in einem Lagerpuffer in einer Konzentration von 100 μM resuspendiert. Nicht verwendete Proben werden bei -20 °C gelagert.
- Kombinieren Sie eine Probe eines mit Quantenpunkten markierten Oligonukleotids mit einem 10-fachen molaren Überschuss an biotinyliertem Oligonukleotid. Da die geschätzte DNA-Konzentration in der mit Quantenpunkten markierten Probe 2 μM beträgt, fügen Sie 0,2 μl biotinyliertes Oligonukleotid pro 10 μl dieser Probe hinzu.
- Die Mischung in einem Heizblock auf 75 °C erhitzen (die Schmelztemperatur für den Komplementärbereich). Bei dieser Temperatur 5 Minuten halten. Schalten Sie den Heizblock aus und lassen Sie ihn langsam abkühlen. Lagern Sie das fertige Konstrukt bei 4 °C; Nicht einfrieren.
3. Oberflächenfunktionalisierung von Deckgläsern
HINWEIS: Dieser Vorgang wurde bereits in anderen JoVE-Videoprotokollen16,17 beschrieben. Dieses Protokoll beschreibt eine angepasste Version des Verfahrens mit geringfügigen Änderungen, um einen kleineren Objektträger aufzunehmen. In der Materialtabelle finden Sie weitere Materialien, die für die Oberflächenfunktionalisierung von Deckgläsern erforderlich sind.
- Legen Sie 5 Deckgläser in jeden Deckglashalter. Achten Sie darauf, zwischen jedem Deckglaspaar ein Leerzeichen zu überspringen, damit sie nicht aneinander kleben. Legen Sie die Deckgläser in ihren Halter in ein Glas mit Deckel, geben Sie Ethanol in das Glas, bis die Deckgläser bedeckt sind, und schrauben Sie den Deckel fest fest. Stellen Sie das gesamte Glas in das Wasser des Badbeschallungsgeräts, ohne es vollständig einzutauchen, und beschallen Sie es 30 Minuten lang.
- Füllen Sie ein sauberes Becherglas oder Glas mit Reinstwasser. Entferne die Deckgläser in ihrer Halterung mit einer Metallpinzette vom Ethanol und tauche sie zum Spülen in Wasser. Füllen Sie dann die Deckgläser und den Halter in ein Glas mit einer 1 M Kaliumhydroxid (KOH)-Lösung. Schrauben Sie den Deckel fest auf, stellen Sie das Glas in das Beschallungsbad und beschallen Sie es 30 Minuten lang.
HINWEIS: Seien Sie vorsichtig beim Umgang mit der KOH-Lösung, da sie ätzend und reizend ist. - Wiederholen Sie die Beschallung in Ethanol und KOH-Lösung und spülen Sie die Deckgläser zwischen den einzelnen Schritten wie oben beschrieben mit Wasser ab.
- Übertragen Sie die Deckgläser in ihrem Halter in ein sauberes Glas, das mit reinem Aceton gefüllt ist. Beenden Sie die Reinigung, indem Sie das Glas wie oben beschrieben 30 Minuten lang beschallen. Nach diesem Schritt nicht mit Wasser abspülen.
- Übertragen Sie die Deckgläser in ihrem Halter mit einer Metallpinzette in ein sauberes 100-ml-Becherglas, das 80 mL frisches Aceton und einen Mikrorührstab enthält. Stellen Sie den Becher auf eine magnetische Rührplatte, die auf mindestens 1.000 U/min eingestellt ist. Während das Aceton kräftig gerührt wird, pipettieren Sie 1,6 ml 3-Aminopropyltriethoxysilan (APTES) in das Becherglas, um eine 2%ige v/v-Lösung herzustellen.
HINWEIS: Seien Sie vorsichtig, wenn Sie APTES pipettieren, da es ätzend ist. - Lassen Sie die Deckgläser 2 Minuten lang in der Lösung inkubieren, dann verwenden Sie eine Metallzange, um die Deckgläser in ihrem Halter in ein Becherglas mit Wasser zu übertragen, um die Reaktion zu löschen. Spülen Sie die Deckgläser noch zwei Mal aus und ersetzen Sie das Wasser im Becherglas.
- Das silanisierte Glas im Backofen bei 120 °C 75 min härten. Wenn Sie nicht sofort mit dem nächsten Schritt fortfahren, lagern Sie die Deckgläser maximal einige Tage lang unter Vakuum.
- Löse N-Hydroxysuccinimid (NHS)-ester-derivatisierte Polyethylenglykole (PEGs) in 100 mM Natriumbicarbonat (pH 8,2). Verwenden Sie ein Verhältnis von 10:1 von methoxyterminiertem PEG zu Biotin-terminiertem PEG mit ~100 mg/ml methoxyterminiertem PEG.
- Pipettieren Sie 100 μl der PEG-Lösung auf die Hälfte der trockenen silanisierten Deckgläser und bedecken Sie jedes mit einem zweiten Deckglas. Verwenden Sie kleine Stücke Parafilm, die an jeder Ecke als Abstandshalter platziert werden, um zu verhindern, dass die Deckgläser aneinander kleben.
- Inkubieren Sie die Deckgläser 3,5 h lang in einer feuchten Umgebung. Trennen Sie die Deckglas-Sandwiches. Waschen Sie jedes Deckglas mit einer Spritzflasche mit reichlich Wasser und trocknen Sie es mit Druckluft.
- Lagern Sie funktionalisierte Deckgläser unter Vakuum. Achten Sie darauf, die PEG-behandelte Seite nach oben zu halten, da die andere Seite das DNA-Substrat nicht einfängt.
4. Bau mikrofluidischer Geräte
HINWEIS: In der Materialtabelle finden Sie weitere Materialien, die für die Konstruktion des mikrofluidischen Geräts erforderlich sind.
- Bohren Sie mit einem handgeführten Rotationsmultitool, das mit einem konischen Diamantschleifer ausgestattet ist, zwei Löcher in gegenüberliegende Ecken des Quarzschlittens, die als Ein- und Auslass dienen. Achten Sie darauf, den Schlitten an Ort und Stelle zu sichern, den Bohrer zu schmieren und während des Bohrens ständig mit Wasser zu gleiten. Nach jedem Experiment den vorbereiteten Quarzobjektträger zur Wiederverwendung zurückgewinnen, indem Sie ein gebrauchtes Gerät in Aceton einweichen, um den Klebstoff und das Epoxidharz aufzulösen. Verwerfen Sie die restlichen Komponenten.
HINWEIS: Das Bohren kann von Hand durchgeführt werden, aber die Verwendung eines Presshalters für das Multitool erleichtert den Vorgang. - Kombinieren Sie 25 μl Streptavidin-Lösung mit 80 μl PBS und 20 μl Blockierungspuffer. Bestreichen Sie ein mit PEG behandeltes Deckglas mit dieser Mischung und inkubieren Sie es 30 Minuten lang in einer feuchten Umgebung.
- Bereiten Sie während der Inkubation des Deckglases den Imaging-Spacer vor, um einen Strömungskanal zu erstellen. Schneiden Sie ein 1 Zoll großes quadratisches Stück Abstandshaltermaterial ab und markieren Sie dann einen 2 mm breiten Kanal, der an den Enden der Löcher ausgerichtet ist, die in den Quarzschlitten gebohrt wurden (Abbildung 2). Schneiden Sie den Kanal mit einem Skalpell aus dem Abstandsmaterial heraus.
- Ziehen Sie eine Seite der Rückseite vom Bildabstandshalter ab und legen Sie sie vorsichtig auf den Quarzdia, wobei Sie darauf achten, dass die Einlass- und Auslasslöcher nicht verdeckt werden. Achten Sie darauf, den Quarzobjektträger gründlich mit Aceton zu reinigen, um alle Klebstoffreste von früheren Experimenten zu entfernen.
- Waschen Sie den Deckglas mit Wasser und trocknen Sie ihn mit Druckluft. Entfernen Sie die andere Seite der Rückseite vom Abstandshalter und klemmen Sie sie zwischen den Quarzobjektträger und das funktionalisierte, mit Streptavidin beschichtete Deckglas, indem Sie die Baugruppe mit der Kunststoffpinzette zusammendrücken und Luftblasen aus dem Klebstoff entfernen.
- Führen Sie in jedes Loch des Quarzschiebers einen 30 cm langen Polyethylenschlauch ein. Achten Sie darauf, die Enden des Schlauchs schräg abzuschneiden, um einen freien Fluss der Lösung zu gewährleisten. Verwenden Sie ein Röhrchengestell oder eine andere Stütze, um die Röhrchen an Ort und Stelle zu halten, und versiegeln Sie die Röhrchen an Ort und Stelle und die Kanten des zusammengebauten Geräts mit Epoxidharz.
HINWEIS: Es funktioniert gut, das Gerät auf einem Stück Parafilm zu bauen, der sich leicht von der Unterseite abziehen lässt, wenn das Epoxidharz ausgehärtet ist. - Sobald das Epoxidharz ausgehärtet ist, führen Sie die stumpfe Nadel der leeren Spritze in das Auslassrohr des Geräts ein und tauchen Sie das Ende des Einlassrohrs in einen mit Blockierungspuffer gefüllten Behälter. Ziehen Sie den Spritzenkolben zurück, um das Gerät mit Blockierungspuffer zu füllen. Lassen Sie das Gerät vor der Verwendung mindestens 30 Minuten lang inkubieren.
5. Oberflächen-Tethering von Quantenpunkt-markiertem DNA-Substrat
HINWEIS: Neben dem oben beschriebenen mikrofluidischen Gerät, DNA-Substrat und Blockierungspuffer finden Sie in der Materialtabelle für andere Materialien und in Tabelle 1 für Puffer, die für die Oberflächenbindung von Quantenpunkt-markierten DNA-Substraten erforderlich sind.
- Befestigen Sie das mikrofluidische Gerät mit Klebeband an der Tischplatte des Mikroskops, bringen Sie das Objektiv in Kontakt mit der Unterseite des Geräts und positionieren Sie das Objektiv so, dass sich das Sichtfeld innerhalb des mikrofluidischen Kanals befindet.
- Spülen Sie den mikrofluidischen Kanal mit frischem Blockpuffer, indem Sie den Spritzenkolben zurückziehen, nachdem Sie den Auslassschlauch an die Spritzenpumpe angeschlossen haben. Stellen Sie sicher, dass keine Blasen im Schlauch oder im Kanal eingeschlossen sind.
HINWEIS: Achten Sie ab diesem Zeitpunkt darauf, dass sich das Einlaufrohr in Flüssigkeit befindet, damit keine Luftblasen in das Gerät gelangen. - 1 μl des vorbereiteten DNA-Substrats in 1 ml Blockierungspuffer verdünnen. Setzen Sie das Einlassröhrchen in das verdünnte DNA-Substrat ein und lassen Sie 800 μl der Substratlösung mit einer Geschwindigkeit von 200 μl/min durch den Kanal fließen. Lassen Sie die DNA-Lösung 15 Minuten lang ungestört im Kanal inkubieren, nachdem der Fluss gestoppt wurde.
- Mindestens 800 μl Blockierungspuffer werden mit einer Geschwindigkeit von 200 μl/min durch den Kanal geleitet, um die ungebundene DNA aus dem Kanal zu spülen.
- Passen Sie die Laserleistung, den Mikroskopfokus und den TIRF-Winkel so an, dass die oberflächengebundenen Quantenpunkte deutlich sichtbar sind.
HINWEIS: Obwohl Quantenpunkte nicht schnell ausbleichen, ist es am besten, die Leistung so gering wie möglich zu halten, um das Blinken zu minimieren.
6. REase-vermittelte DNA-Spaltung
HINWEIS: In der Materialtabelle finden Sie die Materialien und in Tabelle 1 die Puffer, die für die REase-vermittelte DNA-Spaltung erforderlich sind.
- Stellen Sie sicher, dass die Kamera auf die optimale Betriebstemperatur gekühlt und für Highspeed-Streaming mit einer Belichtungszeit von 0,10 s eingerichtet ist. Seien Sie darauf vorbereitet, ~4 Minuten lang Daten zu sammeln.
- Spülen Sie das mikrofluidische Gerät mit 800 μl experimentellem Puffer ohne Magnesium bei einer Durchflussrate von 200 μl/min.
- Die REase wird in ein Aliquot (1 ml) experimenteller Puffer ohne Magnesium gegeben und durch Pipettieren vorsichtig gemischt. Verwenden Sie 4 μl von 100.000 Einheiten/ml, was 400 Einheiten/ml EcoRV entspricht. 800 μl des verdünnten Enzyms mit einer Durchflussrate von 200 μl/min durch den Kanal fließen lassen.
- Beginnen Sie das Experiment, indem Sie einen experimentellen Puffer mit Magnesium und Fluorescein mit einer Durchflussrate von 200 μl/min fließen lassen. Beginnen Sie sofort nach dem Starten der Spritzenpumpe mit der Datenerfassung. Nachdem 800 μl Puffer geflossen sind, stoppen Sie die Datenerfassung.
7. Datenanalyse
HINWEIS: In der Materialtabelle finden Sie die für dieses Protokoll verwendete Datenanalysesoftware, und nehmen Sie Anpassungen vor, wenn Sie eine andere Analyseplattform verwenden.
- Nullpunktbestimmung
- Subtrahieren Sie die Hintergrundfluoreszenz von jedem Bild des Experimentalfilms mit der Imtophat-Funktion , einer integrierten morphologischen Top-Hat-Filterfunktion in der Image Processing Toolbox. Wählen Sie eine Scheibe mit einem Radius von drei Pixeln als Strukturierungselement aus. Ermitteln Sie die Hintergrundintensität, indem Sie das gefilterte Bild vom Originalbild subtrahieren.
HINWEIS: Der Filter behält nur Bildmerkmale bei, die heller als der Hintergrund und kleiner als das Strukturierungselement sind. - Mittelung des subtrahierten Hintergrunds über jedes Filmbild. Verwenden Sie eine Trajektorie dieses Werts, um den Nullzeitpunkt für das Experiment zu bestimmen (Abbildung 3A). Bestimmen Sie den Startzeitpunkt, indem Sie das erste Bild ermitteln, bei dem die Anstiegsrate das 3-fache der Standardabweichung der Änderungsrate während des Totvolumenstroms überschreitet.
HINWEIS: Ein starker Anstieg der Änderungsrate des Durchschnittswerts des subtrahierten Hintergrunds für jedes Filmbild zeigt den Zeitpunkt an, zu dem der letzte experimentelle Puffer mit dem Tracerfarbstoff in die Durchflusszelle eingetreten ist (Abbildung 3B).
- Subtrahieren Sie die Hintergrundfluoreszenz von jedem Bild des Experimentalfilms mit der Imtophat-Funktion , einer integrierten morphologischen Top-Hat-Filterfunktion in der Image Processing Toolbox. Wählen Sie eine Scheibe mit einem Radius von drei Pixeln als Strukturierungselement aus. Ermitteln Sie die Hintergrundintensität, indem Sie das gefilterte Bild vom Originalbild subtrahieren.
- Berechnung und Analyse der Quantenpunktintensität
- Berechnen Sie eine Projektion mit maximaler Intensität für den Satz von hintergrundkorrigierten Filmbildern, die vor dem Eintreffen des Tracerfarbstoffs aufgenommen wurden. Bestimmen Sie die Positionen von Quantenpunkten in diesem Projektionsbild mit einer Genauigkeit von einem Pixel mithilfe einer Peak-Finding-Funktion wie z. B. pkfnd.
- Generieren Sie Intensitätstrajektorien für einzelne Quantenpunkte, indem Sie die durchschnittliche Intensität in jedem Filmbild eines quadratischen Bereichs von drei Pixeln pro Seite berechnen, der die von der Peak-Finding-Funktion zurückgegebene Position umgibt.
HINWEIS: Die oben beschriebene Hintergrundkorrektur entfernt die Artefakte, die durch den Tracerfarbstoff eingeführt wurden. Die resultierenden Intensitätsverläufe sollten relativ flach sein, aber dennoch natürlich auftretende Intensitätsschwankungen einbeziehen (Abbildung 4). - Identifizieren Sie Ereignisse des Verschwindens von Quantenpunkten durch statistische Analyse der Intensitätsverläufe. Berechnen Sie für jede Quantenpunkt-Intensitätskurve eine Schwellenwertintensität, die je nach dem in den Trajektorien beobachteten Rauschen über dem Minimalwert liegt, und zwar um einen angemessenen Bruchteil der Differenz zwischen den Maximal- und Minimalwerten für diese Trajektorie.
HINWEIS: Die Intensitätsfluktuation für den Quantenpunkt ist in der Regel viel größer als die Hintergrundfluktuation. Durch den Vergleich dieser Werte kann ein geeigneter Wert ermittelt werden: Der gewählte Schwellenwert muss höher sein als der höchste Hintergrundwert, idealerweise aber niedriger als der niedrigste Wert für die Quantenpunktfluoreszenz. Für die vorgelegten Daten wurde der verwendete Schwellenwert so berechnet, dass er über dem Mindestwert für jede Trajektorie zuzüglich eines Drittels der Differenz zwischen den Höchst- und Minimalwerten für diese Trajektorie liegt. Man kann davon ausgehen, dass ein Quantenpunkt zu dem Zeitpunkt verschwindet, zu dem die an seinem Standort beobachtete Intensität zuletzt über dieser Schwelle lag. - Bestätigen Sie mutmaßliche Verschwindenereignisse. Ereignisse werden nur dann in die abschließende Analyse einbezogen, wenn die beobachtete Intensität über dem Mittelwert zwischen den minimalen und maximalen Intensitätswerten für die Trajektorie für mehr als die Hälfte der Filmbilder vor dem Zeitpunkt des Verschwindens liegt und die Standardabweichung der Intensitätstrajektorie nach dem Ereignis abgenommen hat.
HINWEIS: Es können auch andere statistische Tests durchgeführt werden, um sicherzustellen, dass nur gültige Verschwindenereignisse protokolliert werden.
Ergebnisse
Die Durchflusszelle ist direkt mit einem Ölimmersionsobjektiv mit 60-facher Vergrößerung mit hoher numerischer Apertur an einem inversen Mikroskop gekoppelt, das mit Laserbeleuchtung für die durchzielbare TIRF-Bildgebung ausgestattet ist (Abbildung 5A). Nach dem Einbringen des DNA-Substrats und dem Wegspülen von überschüssiger DNA und Quantenpunkten befinden sich typischerweise Tausende von einzelnen Quantenpunkten in einem Sichtfeld (Abbildung 5B). Diese Quantenpunkte sind stabil an der Glasoberfläche befestigt und erleiden im Laufe des Experiments keine merkliche Verdunkelung oder signifikante Bleichung. Fließt jedoch ein Puffer, der sowohl Magnesium als auch eine geeignete REase enthält, durch den Strömungskanal, so sind am Ende eines typischen vierminütigen Beobachtungszeitraums mindestens 30 % der zu Beginn eines Experiments vorhandenen Quantenpunkte aus dem Sichtfeld verschwunden. Um den Magnesiumbedarf zu bestätigen, sind mindestens 95 % der zu Beginn des Experiments vorhandenen Quantenpunkte am Ende des Beobachtungszeitraums noch zu sehen, wenn die REase in Abwesenheit von Magnesium durch den Kanal geströmt wird (Abbildung 5C). Wenn jedoch Magnesium-haltiger Puffer unmittelbar nach der Bindung der oberflächengebundenen DNA an die oberflächengebundene DNA durch die REase in Abwesenheit von Magnesium geflossen wird, sind bis zum Ende des Beobachtungszeitraums bis zur Hälfte der Quantenpunkte verschwunden (Abbildung 5D), ähnlich wie bei der gemeinsamen Strömung von REase und Magnesium durch den Kanal. Die genaue Ausbeute an Ereignissen hängt von der Effizienz des Enzyms unter den verwendeten Bedingungen ab. Wenn magnesiumhaltiger Puffer geströmt wird, ohne zuvor die entsprechende REase in den Strömungskanal eingebracht zu haben, verschwinden weniger als 5 % der Quantenpunkte während des Beobachtungszeitraums, und es gibt keinen erkennbaren Peak für ein Histogramm der Ereignisse. Dieses Ergebnis deutet darauf hin, dass vorgebundene REase-Moleküle die Spaltung der DNA vermitteln, die die Quantenpunkte an die Glasoberfläche bindet, und diese DNA-Spaltung ist für die überwiegende Mehrheit der in diesen Experimenten beobachteten Ereignisse des Verschwindens von Quantenpunkten verantwortlich.
Das Verschwinden von Quantenpunkten geschieht schnell und führt zu einer starken Abnahme der Intensitätskurve, was einen klaren Hinweis auf den Zeitpunkt gibt, zu dem ein bestimmtes DNA-Molekül gespalten wird (Abbildung 5E). Die Bestimmung der Ereigniszeiten erfolgt durch die mathematische Analyse von Intensitätsverläufen. Einzelne Quantenpunkte erzeugen Intensitätstrajektorien mit einer signifikant höheren Varianz als der Hintergrund unter den verwendeten Bildbedingungen, so dass mutmaßliche Ereignisse bestätigt werden, wenn die Varianz der Intensitätstrajektorie auf ein Niveau abnimmt, das mit der Varianz des Hintergrunds nach dem beobachteten Intensitätsabfall vergleichbar ist. Darüber hinaus werden Trajektorien, die ein hohes Maß an Blinzeln vor einem mutmaßlichen Verschwinden beinhalten, von der abschließenden Analyse ausgeschlossen. Ein typisches Experiment führt jedoch zu Hunderten von Ereignissen, die diese Kriterien erfüllen, und die Ergebnisse mehrerer Experimente können zusammengefasst werden, um angemessene Statistiken bereitzustellen, die eine Schätzung der Parameter N und β entweder durch nichtlineare Kurvenanpassung der kleinsten Quadrate oder durch Maximum-Likelihood-Parameterschätzung ermöglichen.
Die vorgestellten repräsentativen Daten wurden durch die Durchführung dieses Experiments mit der gut untersuchten Typ IIP REase, EcoRV, erhoben (Video 1). Das 60 bp lange Duplex-DNA-Substrat besteht aus einem Biotin-Molekül am 5'-Ende eines Strangs des Duplex und einem Quantenpunkt, der kovalent an das 5'-Ende des anderen Strangs gebunden ist. Das DNA-Substrat enthält eine einzelne Kopie der Erkennungssequenz, GAT↓ATC, die in der Mitte von EcoRV gespalten wird, wie durch den Pfeil nach unten (↓) angezeigt. EcoRV wurde in Abwesenheit von Magnesium an das DNA-Substrat vorgebunden, und dann wurde magnesiumhaltiger Puffer geflößt, um die DNA-Spaltung zu initiieren. Die repräsentativen Daten umfassen die gepoolten Ergebnisse von fünf separaten Experimenten, die insgesamt 3451 beobachtete Spaltungsereignisse ergeben. Nach dem Ausschluss von Ereignissen, die vor dem Nullpunkt oder außerhalb des ausgeprägten Aktivitätspeaks auftraten, blieben 2987 Ereignisse übrig, die ausreichten, um ein Histogramm mit Ein-Sekunden-Bins zu füllen. Sowohl die nichtlineare Kurvenanpassung der kleinsten Quadrate als auch die Maximum-Likelihood-Parameterschätzung wurden verwendet, um die Werte von N und β aus den Daten zu extrahieren. Die beiden Parameterschätzungsmethoden stimmten überein (Abbildung 6A), wobei N = 3,52 (95 %-Konfidenzintervall: 3,32-3,71) und β = 5,78 s (95 %-Konfidenzintervall: 5,41-6,15 s) für die nichtlineare Anpassung der kleinsten Quadrate und N = 3,41 (95 %-Konfidenzintervall: 3,25-3,58) und β = 6,06 s (95 %-Konfidenzintervall: 5,75-6,39 s) für die Maximum-Likelihood-Schätzung war.
Die Schätzung von mindestens drei kinetischen Schritten ist angesichts dessen, was über den Mechanismus von EcoRV bekannt ist, vernünftig. Es ist bekannt, dass dieses Enzym in Abwesenheit vonMagnesium 14 unspezifisch an DNA bindet. Beobachtungen der Änderungen der intrinsischen Fluoreszenz von Tryptophanresten in EcoRV unter gestoppten Strömungsbedingungen in der Bulk-Lösungsphase deuteten darauf hin, dass EcoRV, das in Abwesenheit von Magnesium an DNA gebunden hat, vor dem Eintritt von Magnesium in das aktive Zentrum eine Konformationsänderung durchlaufen muss18. Diese Beobachtung wird durch Kristallstrukturdaten19 bestätigt. Die Ergebnisse von Tryptophan-Fluoreszenzstudien deuten auch darauf hin, dass die DNA-Spaltung und die Produktfreisetzung getrennte Schritte sind, die mit ähnlichen Raten auftreten18. Daher ist ein vernünftiger Reaktionsmechanismus für die EcoRV-vermittelte DNA-Spaltung in diesem Experiment:
ES → ES* → EP → E+P
Bei diesem Mechanismus wird ES, der anfängliche Enzym-Substrat-Komplex, gebildet, wenn EcoRV in Abwesenheit von Magnesium an die DNA vorgebunden wird. Wenn Magnesium in die Durchflusszelle eintritt, erfährt der Enzym-Substrat-Komplex eine Konformationsänderung und wird zu ES*, dem aktivierten Enzym-Substrat-Komplex. Dieser aktivierte Komplex spaltet dann die DNA, setzt die Produktmoleküle aber nicht sofort frei und wird zu einem Enzym-Produkt-Komplex (EP). Schließlich wird im letzten Schritt das Produkt P freigegeben. Dieser Mechanismus erfordert drei Schritte, die mit den präsentierten Daten übereinstimmen. Die resultierende Schätzung für die durchschnittliche Wartezeit für jeden Schritt beträgt ~6 s, was einer Rate von 0,17 s-1 für jeden Schritt entspricht. Diese Berechnung stimmt im Allgemeinen mit früheren Schätzungen der Raten für diese Prozesse überein - in der Größenordnung von 0,3-0,4 s-1 für die DNA-Spaltung und Produktfreisetzung und ~0,5 s-1 für die Konformationsumlagerung.

Abbildung 1: Schema der markierten DNA- und Enzymreaktion. Quantenpunkt-markierte DNA-Moleküle werden über eine Biotin-Streptavidin-Biotin-Bindung an die funktionalisierte Glasoberfläche gebunden und mit Hilfe der TIRF-Mikroskopie beobachtet. Die DNA-Moleküle enthalten die Erkennungsstelle für die REase von Interesse. Wenn ein DNA-Molekül von der REase gespalten wird, kann der Quantenpunkt frei von der Oberfläche weg und aus der Beleuchtungszone diffundieren. Abkürzungen: REase = Restriktionsendonuklease; PEG = Polyethylenglykol; TIRF = Totalreflexionsfluoreszenz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Mikrofluidische Durchflusszellenvorrichtung. (A) Explosionszeichnung, die die drei Schichten zeigt, die zur Herstellung des Geräts verwendet wurden: das funktionalisierte Glasdeckglas auf der Unterseite, der Quarzschieber mit Einlass- und Auslasslöchern auf der Oberseite und der doppelseitig klebende Abstandshalter mit einem eingeschnittenen Kanal in der Mitte. (B) Ein fertiges Gerät mit versiegelten Polyethylenrohren und Kanten, die mit Epoxidharz beschichtet sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
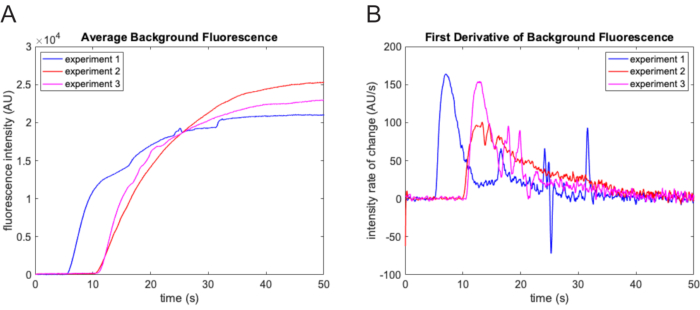
Abbildung 3: Bestimmung des Nullzeitpunkts. (A) Durchschnittliche Hintergrundintensität, bestimmt mit der morphologischen Top-Hat-Filterfunktion für jedes Bild des Films. Die Hintergrundintensität nimmt deutlich zu, wenn der experimentelle Puffer, der Fluorescein enthält, in das Sichtfeld eintritt. Die Ergebnisse von drei verschiedenen Experimenten werden hier gezeigt. Die Verzögerungszeit von Experiment zu Experiment ist sehr unterschiedlich. (B) Die starke Zunahme der Änderungsrate der Hintergrundfluoreszenzintensität erleichtert die genaue Bestimmung des Nullpunkts. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Korrektur im Hintergrund. Die Zunahme der Hintergrundintensität aufgrund des Fluorescein-Tracer-Farbstoffs, der auf die Ankunft von Magnesium hinweist, ist in den rohen Fluoreszenzintensitätstrajektorien für einzelne Quantenpunkte (graue Trajektorie) zu sehen. Nach Subtraktion des Hintergrunds mit der morphologischen Top-Hat-Filterfunktion wurden die durch den Tracerfarbstoff eingeführten Artefakte aus der Trajektorie entfernt (schwarze Trajektorie). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Einzelmolekül-TIRF-Experiment zur parallelisierten Beobachtung von DNA-Spaltungsereignissen. (A) Eine Flusszelle, die auf einer funktionalisierten Glasoberfläche aufgebaut ist, um quantenpunktmarkierte DNA einzufangen, ist direkt mit einem 60-fachen Ölimmersionsmikroskopobjektiv mit hoher numerischer Apertur für die TIRF-Bildgebung gekoppelt. (B) Repräsentatives Bild vom Beginn eines Experiments. Mit Quantenpunkten markierte DNA wurde in die Flusszelle geladen, und überschüssige ungebundene Quantenpunkte wurden weggespült. Einzelne DNA-gebundene Quantenpunkte können voneinander aufgelöst werden. (C) Das gleiche Sichtfeld, nachdem ein Puffer, der eine REase enthält, die in der Lage ist, die DNA-Fesseln zu spalten, vier Minuten lang in Abwesenheit von Magnesium durch den Strömungskanal geleitet wurde. Es gab keinen signifikanten Verlust von DNA-gebundenen Quantenpunkten. (D) Das gleiche Sichtfeld am Ende eines Versuchs. Magnesiumhaltiger Puffer wurde unmittelbar nach dem Einströmen von REase in einen magnesiumfreien Puffer durch den Strömungskanal geströmt. Dieses Bild wurde nach etwa vier Minuten Pufferfluss aufgenommen. Vorgebundene REasen haben viele der DNA-Fesseln gespalten und die Quantenpunkte von der Oberfläche freigesetzt. Zur besseren Betrachtung wird in jedem Bild nur der mittlere Quadrant des objektiven Sichtfelds angezeigt. Zehn Filmbilder (entspricht einer Sekunde Beobachtungszeit) wurden gemittelt, um die Auswirkungen des Blinzelns von Quantenpunkten zu verringern. Die Helligkeits- und Kontrasteinstellungen sind für alle drei Bilder identisch. (E) Repräsentative Fluoreszenzintensitätsverläufe von Bildpositionen, an denen zu Beginn des Experiments ein Quantenpunkt vorhanden war. Trajektorien, die von Bildpositionen erhalten wurden, die Quantenpunkten entsprechen, die nicht von der Oberfläche freigesetzt werden (grau), können kurze Einbrüche auf ein niedrigeres Intensitätsniveau aufweisen, aber sie beginnen und enden bei einem hohen Intensitätsniveau. Trajektorien, die aus Bildpositionen erhalten wurden, die Quantenpunkten entsprechen, die von der Oberfläche freigesetzt werden (schwarz), zeigen einen schnellen Abfall der Intensität auf ein niedriges Hintergrundniveau, das in Bezug auf die Zeitauflösung des Experiments (10 Hz) augenblicklich ist. Freigesetzte Quantenpunkte tauchen innerhalb des vierminütigen Beobachtungszeitraums nicht wieder auf. Abkürzungen: REase = Restriktionsendonuklease; TIRF = Totalreflexionsfluoreszenz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Analyse der Verweilzeitverteilung der EcoRV-vermittelten DNA-Spaltung. (A) Histogramm und vorhergesagte Hüllkurven für die 2987 Spaltungsereignisse, die innerhalb des Hauptaktivitätspeaks in einer Reihe von fünf gepoolten Experimenten mit EcoRV auftreten. Die beiden vorhergesagten Kurven sind nahezu identisch, und die Anpassungsresiduen (unterhalb des Histogramms) weisen nicht auf einen systematischen Fehler bei der nichtlinearen Anpassung der kleinsten Quadrate hin. (B) Histogramm des gesamten Satzes von 3393 Spaltungsereignissen, die nach dem Nullzeitpunkt eingetreten sind. Die Kurve, die durch die Maximum-Likelymi-Schätzung der Parameter (ununterbrochene Linie, MLE-Kurve) unter der Annahme einer Gamma-Wahrscheinlichkeitsverteilung vorhergesagt wird, schließt das Histogramm nicht ein. Die Kurve, die durch eine nichtlineare Anpassung der kleinsten Quadrate der Formel für die Gamma-Wahrscheinlichkeitsverteilung auf die Bin-Höhen (gestrichelte Linie, NLS-Kurve) vorhergesagt wird, ist überlegen, aber die Residuen der Anpassung (unterhalb des Histogramms) zeigen einen systematischen Fehler. Abkürzungen: MLE: Maximum-Likelihood-Schätzung; NLS = nichtlineare kleinste Quadrate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Name des Puffers | Bestandteil | Konzentration | pH-Wert bei 25 °C |
| Natriumphosphat-Puffer | Natriumphosphat | ca. 100 mM | 8.3 |
| CHES Puffer | N-Cyclohexyl-2-aminoethansulfonsäure (CHES) | ca. 10 mM | 9.0 |
| PBS | Natriumphospat | 3 mM | 7.2 - 7.6 |
| Natriumchlorid | ca. 150 mM | ||
| Kaliumphosphat | ca. 1,05 Mio. | ||
| Lager-Puffer | Natriumchlorid | ca. 100 mM | 8.0 |
| Tris-HCl | 50 mM | ||
| Rinderserumalbumin (BSA) | 0,5 mg/ml | ||
| Natriumbicarbonat | Natriumbicarbonat | ca. 100 mM | 8.2 |
| Blockierender Puffer | Tris-HCl | ca. 20 mM | 7.5 |
| Ethylendiamintetraessigsäure (EDTA) | 2 mM | ||
| Natriumchlorid | 50 mM | ||
| Tween-20 | 0,005 % (v/v) | ||
| Rinderserumalbumin (BSA) | 0,2 mg/ml | ||
| Experimenteller Puffer ohne Magnesium | Natriumchlorid | ca. 100 mM | 7.9 |
| Tris-HCl | 50 mM | ||
| Dithiothreitol (DTT) | 1 mM | ||
| Experimenteller Puffer mit Magnesium | Natriumchlorid | ca. 100 mM | 7.9 |
| Tris-HCl | 50 mM | ||
| Magnesiumchlorid | ca. 10 mM | ||
| Dithiothreitol (DTT) | 1 mM | ||
| Fluorescein | Anpassung an die Bildgebungsbedingungen | ||
| DNase-Puffer | Magnesiumchlorid | 2,5 Mio. M. | 7.6 |
| Tris-HCl | ca. 10 mM | ||
| Calciumchlorid | 0,1 mM |
Tabelle 1: Tabelle der Puffer.
Video 1: Beispiel für einen Einzelmolekül-Film. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
Das DNA-Substrat für diesen Assay wird unter Verwendung eines zweistufigen Reaktionsschemas unter Verwendung von Sulfo-SMCC mit einem Quantenpunkt markiert. Dieser bifunktionelle Vernetzer besteht aus einer NHS-Estergruppe, die mit einem primären Amin reagieren kann, und einer Maleimidgruppe, die mit einer Sulfhydrylgruppe20 reagieren kann. Die thiolierten Oligonukleotide, die zur Herstellung des Substrats verwendet werden, werden in ihrer oxidierten Form versandt. Es ist wichtig, sie wie beschrieben zu reduzieren und zu reinigen, bevor mit dem Kopplungsverfahren fortgefahren wird, da sonst die Effizienz der Kopplungsreaktion verringert wird. Es ist wichtig, frische DVB-T- und Sulfo-SMCC-Lösungen unmittelbar vor der Verwendung vorzubereiten. Diese Moleküle hydrolysieren in wässriger Lösung schnell. Seien Sie vorsichtig, wenn Sie Lösungen in und aus den Dialysegeräten übertragen – wenn die Pipettenspitze die Membran berührt, führt dies zu einem Bruch und die Probe geht verloren. Es ist ideal, alle Schritte in Abschnitt 2 ohne Unterbrechung durchzuführen, um Probenverluste zu vermeiden.
Die Kopplungsreaktion, die zur Erzeugung des Quantenpunkt-markierten Oligonukleotids verwendet wird, ist ineffizient. Nach dem Kombinieren und Glühen des Quantenpunkt-markierten Oligonukleotids mit dem biotinylierten Oligonukleotid ist in der vorbereiteten Substratprobe überschüssige unmarkierte DNA vorhanden. Obwohl diese Spezies an die funktionalisierte Oberfläche binden können, sind sie unter den empfohlenen Bildgebungsbedingungen nicht sichtbar und tragen nicht zum Auslesen des Experiments bei. Um zu bestätigen, dass die Spaltung des fertigen Konstrukts zur Freisetzung von Quantenpunkten von der Oberfläche führt, führen Sie das Oberflächen-Tethering durch, wie in Abschnitt 5 des Protokolls beschrieben, und bilden Sie einen Bereich der Oberfläche ab, um zu bestimmen, wie viele Quantenpunkte vorhanden sind. Dann fließen 800 μl eines 1 mL Aliquots von 10 U/mL DNase I in DNase-Puffer (2,5 mM MgCl2; 10 mM Tris-HCl; 0,1 mM CaCl2, pH 7,6) durch den mikrofluidischen Kanal. Lassen Sie die DNase I 15 Minuten lang im Kanal inkubieren, waschen Sie sie mit 800 μl DNase-Puffer (ohne Enzym) und bilden Sie die gleiche Region von Interesse ab. Etwa 90 % der Quantenpunkte sollten von der Oberfläche gelöst werden, wenn das Konstrukt und die Oberfläche richtig vorbereitet sind.
Um eine Mischung aus methoxyterminierten und Biotin-terminierten PEG-Molekülen kovalent zu binden, müssen Glasoberflächen so behandelt werden, dass das mit Quantenpunkten markierte DNA-Substrat auf der Oberfläche für die Bildgebung eingefangen wird. Es ist von entscheidender Bedeutung, alle Reinigungsschritte rigoros durchzuführen. Mehrere Reinigungsschritte sind erforderlich, um Oberflächenverunreinigungen zu entfernen und Hydroxylgruppen auf der Glasoberfläche freizulegen, die mit den Alkoxygruppen auf dem Silan reagieren, um kovalente Bindungen zu bilden. Während es wichtig ist, die Objektträger zwischen den Reinigungsschritten mit Reinstwasser mit Ethanol und KOH-Lösungen zu spülen, sollte nach der Endreinigung mit Aceton kein Wasser verwendet werden. Das Vorhandensein von überschüssigem Wasser während des Silanisierungsschritts fördert die Multimerisierung des Silans in Lösung auf Kosten der Oberflächenbindung. Außerdem darf die Silanlösung nicht im Voraus zubereitet werden. Um die Lebensdauer zu verlängern, sollte der Silanvorrat in einem Exsikkatorschrank gelagert werden.
Der Einzelmolekül-Spaltungsassay ist für die Durchführung in einem mikrofluidischen Gerät konzipiert (Abbildung 2). Stellen Sie vor jedem Experiment ein neues Gerät her, indem Sie den gebohrten Quarzobjektträger, der die Oberseite des Geräts bildet, wiederverwenden. Dieses Verfahren wurde bereits beschrieben16,17 und seine Variationen werden häufig für die TIRF-Mikroskopie verwendet. Das hierin beschriebene Verfahren wurde angepasst, um quadratische 1-Zoll-Folien aufzunehmen. Es ist wichtig, schnell an der Konstruktion des Geräts zu arbeiten, damit sich die funktionalisierte Oberfläche nicht verschlechtert, während sie der Luft ausgesetzt ist. Epoxidharz ist fluoreszierend, achten Sie also darauf, den Kanal nicht damit zu bedecken. Der gebohrte Quarzschlitten kann zur Wiederverwendung zurückgewonnen werden, indem das gebrauchte Gerät in Aceton getränkt und zerlegt wird.
Dieser Assay ist für die Durchführung an einem inversen Mikroskop konzipiert, das über eine objektive interne Totalreflexionsbeleuchtung verfügt und zur Datenerfassung mit einer EMCCD-Kamera (Electron-Multiplying Charge-Coupled Device) ausgestattet ist. Verwenden Sie ein 60-faches Objektiv mit hoher numerischer Apertur und eine 488-nm-Laseranregungsquelle und verwenden Sie einen Filterwürfel, der einen für den Laser geeigneten Single-Bandpass-Emissionsfilter und einen dichroitischen Einkantenfilter sowie einen Emissionsfilter enthält, der sowohl die Fluoreszenz der Quantenpunkte (Emissionspeak bei 655 nm) als auch zumindest einen Teil der Fluoreszenzfluoreszenz (Emissionspeak bei 512 nm) durchlässt. Zum Beispiel wurden die hier vorgestellten Daten mit einem Filterwürfel gesammelt, der mit einem 482/18 nm Einzelbandpass-Anregungsfilter, einem 488 nm Einkanten-Siegroitischen und einem 675/67 nm Einzelbandpass-Emissionsfilter ausgestattet war. Der Assay kann durch Bildgebung in einem einzelnen Spektralkanal durchgeführt werden, wie in diesem Protokoll beschrieben. Falls gewünscht, kann der Assay auch durchgeführt werden, indem das Fluoreszenzbild in zwei Spektralkanäle aufgeteilt wird, einen für die Quantenpunkte, einen für die Fluoreszeinemission. Zu diesem Zweck wäre ein Bildsplitter geeignet, der mit einem dichroitischen Spiegel mit einer einzigen Kante von 640 nm ausgestattet ist. Mehrkanal-Imaging ist jedoch nicht erforderlich.
Es ist wichtig, vor Beginn des Experiments alle ungebundenen DNA-Substrate aus dem Kanal auszuwaschen. Fluorescein ist in einer Konzentration von 50 μM im endgültigen Puffer enthalten, um die Ankunft des Magnesiums im Sichtfeld zu signalisieren. Je nach Mikroskopaufbau kann es notwendig sein, die Konzentration des Fluoresceins so einzustellen, dass seine Emission die Quantenpunkte nicht verdeckt. Stellen Sie sicher, dass Sie sofort nach dem Start des Flusses des magnesiumhaltigen Puffers mit der Datenerfassung beginnen. Aufzeichnung der Daten während der Verzögerungszeit vor dem Eintreffen des magnesiumhaltigen Puffers, so dass der Mittelwert und die Standardabweichung der Änderungsrate der Hintergrundintensität berechnet werden können, um die genaue Bestimmung des Nullpunkts zu erleichtern. Beachten Sie, dass der genaueste Nullzeitpunkt das erste Bild ist, bei dem die Änderungsrate der Hintergrundfluoreszenz die natürlichen Schwankungen bei dieser Messung übersteigt. Die Hintergrundfluoreszenzintensität nimmt nach diesem Punkt weiter zu, da Fluorescein über die Grenzschicht diffundiert, die durch die laminare Strömung im Kanal gebildet wird. Magnesium, das viel kleiner als Fluorescein ist, diffundiert schneller, und es ist zu erwarten, dass seine Konzentration viel schneller ins Gleichgewicht kommt.
Für die vorgestellten Beispieldaten zeigt ein Histogramm aller 3393 Ereignisse, die nach dem Nullzeitpunkt auftraten, einen markanten Peak innerhalb der ersten 60 s der Experimente. Viele Bins, die längeren Verzögerungszeiten entsprechen, enthalten keine Ereignisse, und die Bins, die Ereignisse enthalten, enthalten nur wenige, ähnlich wie bei der Aktivität, die beobachtet wird, wenn kein aktives REase vorhanden ist. Eine nichtlineare Kurvenanpassung der Gamma-Wahrscheinlichkeitsverteilung an alle klassifizierten Daten führte zu Parameterschätzungen von N = 3,06 mit einem 95 %-Konfidenzintervall von 2,94-3,18 und β = 7,04 s mit einem 95 %-Konfidenzintervall von 6,72-7,36 s. Die Maximum-Likelialy-Schätzungen für die Parameter betrugen jedoch N = 1,30 (95 %-Konfidenzintervall: 1,24-1,35) und β = 26,7 s (95 %-Konfidenzintervall: 25,3-28,1 s), und bei visueller Inspektion stimmt die von der Maximum-Likelihood-Parameterschätzung vorhergesagte Kurve nicht gut mit den Daten überein (Abbildung 6B). Obwohl die Kurve, die durch die durch die nichtlineare Anpassung der kleinsten Quadrate geschätzten Parameter vorhergesagt wird, der Form des Histogramms der beobachteten Ereignishäufigkeiten relativ gut folgt, werden die Residuen (Differenz zwischen beobachteten und vorhergesagten Ereignishäufigkeiten) für Zeitpunkte jenseits von etwa 60 s konsistent positiv (Abbildung 6B).
Da die Häufigkeit von Ereignissen in den späteren Zeitpunkten vergleichbar mit der Häufigkeit des Verschwindens von Quantenpunkten ist, die in Abwesenheit eines aktiven Enzyms beobachtet wird, sind die meisten dieser späteren Ereignisse wahrscheinlich Teil eines niedrigen Hintergrundniveaus des Quantenpunktverlusts, der nicht durch REase-vermittelte DNA-Spaltung verursacht wird. Es ist daher sinnvoll, diese Behälter vom Einbau auszunehmen. Wenn alle Ereignisse ausgeschlossen wurden, die mehr als 60 s nach dem Nullzeitpunkt jedes Experiments auftraten, stimmten die beiden Parameterschätzungsmethoden überein (Abbildung 6A). Um die besten Parameterschätzungen zu erzielen, schließen Sie daher Ereignisse außerhalb des Aktivitätspeaks aus, und bestätigen Sie, dass die Schätzung der Maximum-Likelihood-Parameter und die Ergebnisse der nichtlinearen Kurvenanpassung der kleinsten Quadrate konvergiert sind.
Der in dieser Arbeit skizzierte experimentelle Ansatz ermöglicht Echtzeitbeobachtungen der REase-vermittelten DNA-Spaltung auf Einzelmolekülebene. In jedem einzelnen Experiment können Hunderte von Ereignissen beobachtet werden, und die Ergebnisse mehrerer Experimente können zusammengefasst werden, um die große Anzahl von Beobachtungen zu erhalten, die erforderlich sind, um die Verteilung der Wartezeiten zu charakterisieren, indem die Daten an die Gamma-Wahrscheinlichkeitsverteilung angepasst werden. Während dieser Assay nur wie beschrieben mit REasen verwendet werden kann, die DNA binden, aber nicht spalten, wenn Magnesium oder ein anderer Cofaktor vorhanden ist, fallen viele REasen in diese Kategorie. Es ist wichtig zu wissen, dass die genauen Details aller Zwischenschritte durch diesen Assay nicht preisgegeben werden. Schritte, die deutlich schneller sind als der geschwindigkeitsbegrenzende Schritt, wirken sich nicht auf die Verweilzeitverteilung aus und fließen nicht in den Wert von N ein, der aus den Datenextrahiert wird 12. Ebenso ist der Wert der β, der aus den Daten extrahiert werden kann, ein ungefährer Durchschnitt der Wartezeit für die Schritte, die sich auf die Verweilzeitverteilung auswirken. Die tatsächliche Wartezeit für jeden Schritt kann vom Wert von β12 abweichen. Der in dieser Arbeit beschriebene Einzelmolekül-Spaltungsassay kann jedoch den Nachweis erbringen, dass mehrere Zwischenschritte entlang des Reaktionswegs vorhanden sind, selbst wenn die Reaktionszwischenprodukte selbst nicht direkt beobachtet werden können.
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen oder andere Interessenkonflikte
Danksagungen
Diese Arbeit wurde durch die Auszeichnung Nummer K12GM074869 an CME des National Institute of General Medical Sciences unterstützt. Der Inhalt liegt in der alleinigen Verantwortung der Autoren und gibt nicht unbedingt die offizielle Meinung des National Institute of General Medical Sciences oder der National Institutes of Health wieder.
Materialien
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltriethoxysilane (APTS) | Sigma Aldrich | 440140-100ML | Store in dessicator box |
| 5 Minute Epoxy | Devcon | 20845 | use for sealing the microfluidic device |
| acetone | Pharmco | 329000ACS | use for cleaning coverslips |
| bath sonicator | Fisher Scientific | CPXH Model 2800 | catalog number 15-337-410 |
| Beaker, glass, 100 mL | |||
| Benchtop centrifuge | |||
| Biotin-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | BIO-SVA-5K | Succinimidyl valerate has a longer half-life than succinimidyl carbonate |
| biotinylated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Biotin modification and HPLC purification |
| Bovine Serum Albumin (BSA) | VWR | 0903-5G | prepare a 10 mg/mL solution (aq) and heat to 95 °C before using |
| Centri-Spin-10 Size Exclusion Spin Columns | Princeton Separations | CS-100 or CS-101 | used to purify thiolated oligonucleotides after reducing the disulfide bond |
| Centrifuge tubes 1.5. mL | |||
| Coverslips, 1-inch square glass | |||
| coverslip holders | |||
| diamond point wheel | Dremel | 7134 | use for drilling holes in quartz flow cell topper |
| dithiothreitol (DTT) | Thermo Scientific | A39255 | No-Weigh Format, 7.7 mg/vial |
| drill press rotary tool workstation stand | Dremel | 220-01 | facilitates quartz drilling |
| EcoRV (REase used to generate example data) | New England Biolabs | R0195T or R0195M | Use 100,000 units/mL stock to avoid adding excess glycerol Check REBASE for suppliers of other REases |
| ethanol | various | CAS 64-17-5 | denatured or 95% are acceptable, use for cleaning coverslips |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | EDS | BioUltra, anhydrous, store in dessicator box |
| Flea Micro Spinbar | Fisherbrand | 14-513-65 | 3 mm x 10 mm size to fit beneath coverslip rack |
| fluorescein | Acros Organics | 17324 | use to make experimental buffers |
| gravity convection oven | Binder | 9010-0131 | |
| handheld rotary multitool | Dremel | 8220 | use for drilling holes in quartz flow cell topper |
| ImagEM X2 EM-CCD Camera | Hamamatsu | C9100-23B | air cooling is adequate for this experiment, use HCImage software or similar to control |
| Imaging spacer, double-sided, adhesive | |||
| Jar, glass with screw cap, (approximately 50 mm diameter by 50 mm high) | |||
| magnesium chloride hexahydrate | Fisher Bioreagents | BP214-500 | use to make experimental buffer with magnesium |
| MATLAB software | Data analysis | ||
| metal tweezers | Fisher Brand | 16-100-110 | |
| methoxy-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | M-SVA-5K | Both PEGs should have the same NHS ester so that the rate of reaction is consistent |
| microcentrifuge | Eppendorf | 5424 | |
| multiposition magnetic stirrer | VWR | 12621-022 | |
| N-cyclohexyl-2-aminoethanesulfonic acid (CHES) | Acros Organics | AC20818 | CAS 103-47-9, use to make CHES buffer |
| orbital shaker and heater for microcentrifuge tubes | Q Instruments | 1808-0506 | with 1808-1061 adaptor for 24 x 2.0 mL or 15 x 0.5 mL tubes |
| Parafilm | |||
| PE60 Polyethylene tubing (inner diameter 0.76 mm, outer diameter 1.22 mm) | Intramedic | 6258917 | 22 G blunt needles are a good fit for this tubing size |
| Phosphate-Buffered Saline (PBS) 10x | Sigma Aldrich | P7059 | Use at 1x strength |
| potassium hydroxide | VWR Chemicals BDH | BDH9262 | use a 1 M solution to clean coverslips |
| Qdot 655 ITK Amino (PEG) Quantum Dots | Invitrogen | Q21521MP | |
| Quartz Slide, 1 inch square, 1 mm thick | Electron Microscopy Sciences | 72250-10 | holes must be drilled in the corners for inlet and outlet tubing insertion |
| reinforced plastic tweezers | Rubis | K35a | use for handling coverslips and building microfluidic device |
| SecureSeal Adhesive Sheets | Grace Biolabs | SA-S-1L | cut to form spacer for microfluidic device |
| Single channel syringe pump for microfluidics | New Era Pump Systems | NE-1002X-US | fitted with a 50 mL syringe and a 22 G blunt needle |
| Slide-a-Lyzer MINI Dialysis Devices, 10 kDa MWCO, 0.1 mL | Thermo Scientific | 69570 or 69572 | used for buffer exchange during quantum dot coupling to DNA |
| sodium bicarbonate | EMD Millipore | SX0320 | use to make buffer for surface functionalization; 100 mM, pH 8 |
| sodium chloride | Macron | 7581-12 | use to make experimental buffers |
| Sodium phosphate dibasic solution (BioUltra, 0.5 M in water) | Sigma Aldrich | 94046 | use to make 100 mM sodium phosphate buffer |
| Sodium phosphate monobasic solution (BioUltra, 5M in water) | Sigma Aldrich | 74092 | use to adjust pH of 100 mM sodium phosphate buffer |
| Streptavidin from Streptomyces avidinii | Sigma Aldrich | S4762 | dissolve at 1 mg/mL and store 25 mL aliqouts at -20 ? |
| Sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexane-1-carboxylate (sulfo-SMCC) | Thermo Scientific | A39268 | No-Weigh Format, 2 mg/vial |
| Syringe fitted with blunt 21 G needle | |||
| Syringe pump | |||
| thiolated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Thiol Modifier C6 S-S and HPLC purificaiton |
| TIRF imaging system with 488 nm laser illumination | various | custom built | |
| Tris -HCl | Research Products International | T60050 | use to make experimental buffers |
| Tris base | JT Baker | 4101 | use to make experimental buffers |
| Tween-20 | Sigma | P7949 | use to make blocking buffer |
| Ultrapure water | |||
| vortex mixer | VWR | 10153-842 | |
| Wash-N-Dry Coverslip Rack | Electron Microscopy Sciences | 70366-16 | used for surface functionalization of coverslips |
Referenzen
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5905-5908 (2005).
- Loenen, W. A., Dryden, D. T., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Research. 42 (1), 3-19 (2014).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE--a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research. 43, Database issue 298-299 (2015).
- Bonnet, I., et al. Sliding and jumping of single EcoRV restriction enzymes on non-cognate DNA. Nucleic Acids Research. 36 (12), 4118-4127 (2008).
- Biebricher, A., Wende, W., Escude, C., Pingoud, A., Desbiolles, P. Tracking of single quantum dot labeled EcoRV sliding along DNA manipulated by double optical tweezers. Biophysical Journal. 96 (8), 50-52 (2009).
- Blainey, P. C., et al. Nonspecifically bound proteins spin while diffusing along DNA. Nature Structural & Molecular Biology. 16 (12), 1224-1229 (2009).
- Huang, C. -F., et al. Direct visualization of DNA recognition by restriction endonuclease EcoRI. Journal of Experimental & Clinical Medicine. 5 (1), 25-29 (2013).
- Palma, M., et al. Selective biomolecular nanoarrays for parallel single-molecule investigations. Journal of American Chemical Society. 133 (20), 7656-7659 (2011).
- Reinhard, B. M., Sheikholeslami, S., Mastroianni, A., Alivisatos, A. P., Liphardt, J. Use of plasmon coupling to reveal the dynamics of DNA bending and cleavage by single EcoRV restriction enzymes. Proceedings of the National Academy of Sciences of the United States of America. 104 (8), 2667-2672 (2007).
- van den Broek, B., Noom, M. C., Wuite, G. J. DNA-tension dependence of restriction enzyme activity reveals mechanochemical properties of the reaction pathway. Nucleic Acids Research. 33 (8), 2676-2684 (2005).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Loparo, J. J., van Oijen, A. Single-molecule enzymology. Handbook of Single-Molecule Biophysics. Hinterdorfer, P., Oijen, A. , Springer. New York, NY. 165-182 (2009).
- Taylor, J. D., Badcoe, I. G., Clarke, A. R., Halford, S. E. EcoRV restriction endonuclease binds all DNA sequences with equal affinity. Biochemistry. 30 (36), 8743-8753 (1991).
- Pingoud, A., Jeltsch, A. Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29 (18), 3705-3727 (2001).
- Tanner, N. A., Loparo, J. J., van Oijen, A. M. Visualizing single-molecule DNA replication with fluorescence microscopy. Journal of Visualized Experiments: JoVE. (32), e1529(2009).
- Kulczyk, A. W., Tanner, N. A., Loparo, J. J., Richardson, C. C., van Oijen, A. M. Direct observation of enzymes replicating DNA using a single-molecule DNA stretching assay. Journal of Visualized Experiments: JoVE. (37), e1689(2010).
- Baldwin, G. S., Vipond, I. B., Halford, S. E. Rapid reaction analysis of the catalytic cycle of the EcoRV restriction endonuclease. Biochemistry. 34 (2), 705-714 (1995).
- Winkler, F. K., et al. The crystal-structure of Ecorv endonuclease and of Its complexes with cognate and non-cognate DNA fragments. EMBO Journal. 12 (5), 1781-1795 (1993).
- Sioss, J. A., Stoermer, R. L., Sha, M. Y., Keating, C. D. Silica-coated, Au/Ag striped nanowires for bioanalysis. Langmuir. 23 (22), 11334-11341 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten