Method Article
Analisi del tempo di permanenza di una singola molecola della scissione del DNA mediata da endonucleasi di restrizione
In questo articolo
Riepilogo
Utilizzando il DNA marcato con punti quantici e la microscopia a fluorescenza a riflessione interna totale, possiamo studiare il meccanismo di reazione delle endonucleasi di restrizione utilizzando proteine non marcate. Questa tecnica a singola molecola consente l'osservazione massicciamente multiplexata delle singole interazioni proteina-DNA e i dati possono essere raggruppati per generare distribuzioni del tempo di permanenza ben popolate.
Abstract
Questo nuovo test basato sulla microscopia a fluorescenza a riflessione interna totale facilita la misurazione simultanea della lunghezza del ciclo catalitico per centinaia di singole molecole di endonucleasi di restrizione (REasi) in un unico esperimento. Questo test non richiede la marcatura delle proteine e può essere eseguito con un singolo canale di imaging. Inoltre, i risultati di più esperimenti individuali possono essere raggruppati per generare distribuzioni del tempo di permanenza ben popolate. L'analisi delle distribuzioni del tempo di permanenza risultanti può aiutare a chiarire il meccanismo di scissione del DNA rivelando la presenza di passaggi cinetici che non possono essere osservati direttamente. I dati di esempio raccolti utilizzando questo test con la ben studiata REasi, EcoRV - un'endonucleasi dimerica di restrizione di tipo IIP che slega la sequenza palindromica GAT↓ATC (dove ↓ è il sito di taglio) - sono in accordo con studi precedenti. Questi risultati suggeriscono che ci sono almeno tre passaggi nel percorso di scissione del DNA che viene avviato introducendo il magnesio dopo che EcoRV si lega al DNA in sua assenza, con un tasso medio di 0,17 s-1 per ogni passaggio.
Introduzione
Le endonucleasi di restrizione (REasi) sono enzimi che effettuano rotture del doppio filamento sequenzia-specifiche nel DNA. La scoperta delle REasi negli anni '70 ha portato allo sviluppo della tecnologia del DNA ricombinante, e questi enzimi sono ora strumenti di laboratorio indispensabili per la modificazione e la manipolazione genetica1. Le REasi di tipo II sono gli enzimi più utilizzati in questa classe in quanto scindono il DNA in una posizione fissa all'interno o vicino alla loro sequenza di riconoscimento. Tuttavia, c'è una grande quantità di variazioni tra le REasi di Tipo II, e sono divise in diversi sottotipi in base a particolari proprietà enzimatiche piuttosto che essere classificate in base alle loro relazioni evolutive. Tra ogni sottotipo, ci sono frequenti eccezioni allo schema di classificazione e molti enzimi appartengono a più sottotipi2. Sono state identificate migliaia di REasi di Tipo II e centinaia di esse sono disponibili in commercio.
Tuttavia, nonostante la diversità tra le SI di Tipo II, pochissime SI sono studiate in dettaglio. Secondo REBASE, il database degli enzimi di restrizione istituito da Sir Richard Roberts nel 19753, i dati cinetici pubblicati sono disponibili per meno di 20 di questi enzimi. Inoltre, mentre alcune REasi sono state osservate direttamente a livello di singola molecola mentre si diffondevano lungo il DNA prima di incontrare e legarsi alla loro sequenza di riconoscimento 4,5,6,7, ci sono pochissimi studi su singole molecole della loro cinetica di reazione di scissione. Gli studi esistenti non riportano statistiche adeguate per intraprendere un'analisi dettagliata della variazione dei tempi in cui si verificano singoli eventi di scissione 8,9,10 o non sono in grado di catturare l'intera distribuzione dei tempi di scissione11. Questo tipo di analisi può rivelare la presenza di intermedi cinetici relativamente longevi e potrebbe portare a una migliore comprensione dei meccanismi di scissione del DNA mediata da REasi.
A livello di singola molecola, i processi biochimici sono stocastici: il tempo di attesa per il verificarsi di una singola istanza del processo, τ, è variabile. Tuttavia, ci si può aspettare che molte misure di τ obbediscano a una distribuzione di probabilità, p(τ), che è indicativa del tipo di processo in corso. Ad esempio, un processo a passo singolo, come il rilascio di una molecola di prodotto da un enzima, obbedirà alla statistica di Poisson e p(τ) assumerà la forma di una distribuzione esponenziale negativa:
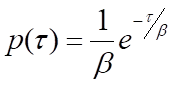
dove β è il tempo medio di attesa. Si noti che la velocità del processo, k, sarà pari a 1/β, l'inverso del tempo medio di attesa. Per i processi che richiedono più di un passo, p(τ) sarà la convoluzione delle distribuzioni a singolo esponenziale per ciascuno dei singoli passaggi. Una soluzione generale per la convoluzione di N funzioni di decadimento a singolo esponenziale con identico tempo medio di attesa, β, è la distribuzione di probabilità gamma:

dove Γ(N) è la funzione gamma, che descrive l'interpolazione del fattoriale di N-1 a valori non interi di N. Sebbene questa soluzione generale possa essere utilizzata come approssimazione quando i tempi medi di attesa dei singoli passaggi sono simili, si deve comprendere che la presenza di passaggi relativamente veloci sarà mascherata da passaggi con tempi di attesa significativamente più lunghi. In altre parole, il valore di N rappresenta un limite inferiore al numero di passi12. Con un numero adeguato di misurazioni del tempo di attesa, i parametri β e N possono essere stimati raggruppando gli eventi e adattando la distribuzione gamma all'istogramma risultante o utilizzando un approccio di stima della massima verosimiglianza. Questo tipo di analisi può quindi rivelare la presenza di passaggi cinetici che non possono essere facilmente risolti nei saggi d'insieme e richiede un gran numero di osservazioni per stimare accuratamente i parametri12,13.
Questo articolo descrive un metodo per utilizzare il DNA marcato con punti quantici e la microscopia a fluorescenza a riflessione interna totale (TIRF) per osservare centinaia di singoli eventi di scissione del DNA mediati da REasi in parallelo. La progettazione del test consente di riunire i risultati di diversi esperimenti e può creare distribuzioni del tempo di permanenza contenenti migliaia di eventi. L'elevata fotostabilità e luminosità dei punti quantici consente una risoluzione temporale di 10 Hz senza sacrificare la capacità di osservare gli eventi di clivaggio che si verificano anche molti minuti dopo l'inizio dell'esperimento. Una buona risoluzione temporale e un ampio intervallo dinamico, combinati con la capacità di raccogliere un ampio set di dati, consentono una caratterizzazione accurata delle distribuzioni del tempo di permanenza per scoprire la presenza di più passaggi cinetici nelle vie di scissione delle REasi, che hanno tassi di turnover nell'intervallo 1 min-1 . Nel caso di EcoRV, è possibile risolvere tre passaggi cinetici, tutti identificati con altri mezzi, confermando che il test è sensibile alla presenza di tali passaggi.
I substrati di DNA duplex contenenti la sequenza di riconoscimento di interesse sono prodotti mediante ricottura di un oligonucleotide biotinilato in un filamento complementare marcato con un singolo punto quantico di nanocristalli semiconduttori attaccato covalentemente. Questi substrati vengono introdotti in un canale di flusso costruito sopra un vetrino coprioggetti con un prato di molecole di polietilenglicole (PEG) ad alto peso molecolare attaccate in modo covalente alla sua superficie. I substrati del DNA vengono catturati tramite un legame biotina-streptavidina-biotina da una frazione delle molecole PEG che hanno una biotina all'estremità libera. Nella microscopia TIRF, un'onda evanescente che decade esponenzialmente con la distanza dall'interfaccia vetro-liquido fornisce illuminazione; La profondità di penetrazione è dell'ordine della lunghezza d'onda della luce utilizzata. In queste condizioni, saranno eccitati solo i punti quantici che sono legati alla superficie da una molecola di DNA che è stata catturata sulla superficie di vetro funzionalizzato. I punti quantici che sono liberi in soluzione non saranno vincolati all'interno della regione illuminata e quindi non si illumineranno. Se il DNA che lega un punto quantico alla superficie viene tagliato, quel punto quantico sarà libero di diffondersi lontano dalla superficie e scomparirà dall'immagine a fluorescenza.
Sebbene molte RACESI di Tipo II siano note per legare il DNA in assenza di magnesio14, tutte richiedono magnesio per mediare la scissione del DNA15. Queste REasi possono legare il DNA immobilizzato in superficie in assenza di magnesio. Quando il tampone contenente magnesio viene fatto scorrere attraverso un canale con REasi prelegata al DNA, la scissione inizia immediatamente, come indicato dalla scomparsa dei punti quantici. La sincronizzazione ottenuta prelegando le molecole di REasi e quindi avviando la scissione del DNA introducendo il magnesio, facilita la misurazione del tempo di ritardo al completamento della scissione del DNA indipendentemente per ciascuna molecola nella popolazione di enzimi osservata in un esperimento. La fluoresceina è inclusa come colorante tracciante nel tampone contenente magnesio per indicare l'arrivo del magnesio nel campo visivo. Poiché nessun enzima è incluso nel tampone contenente magnesio, il tempo di ritardo dall'arrivo del tampone contenente magnesio alla scomparsa di ciascun punto quantico indica il tempo necessario a una REasi che è già legata al DNA per scindere il DNA e rilasciare il punto quantico dalla superficie di vetro. La scomparsa dei punti quantici avviene rapidamente e si traduce in una forte diminuzione della traiettoria di intensità, fornendo una chiara indicazione del tempo in cui una data molecola di DNA viene scissa. La determinazione dei tempi degli eventi viene effettuata mediante analisi matematica delle traiettorie di intensità e un tipico esperimento produce centinaia di eventi di scissione identificabili. I risultati di più esperimenti possono essere raggruppati per fornire statistiche adeguate per consentire la stima dei parametri, N e β, mediante analisi dei minimi quadrati non lineari o di massima verosimiglianza.
Protocollo
1. Informazioni generali
- Progettazione di oligonucleotidi
NOTA: Il substrato di DNA lungo 60 coppie di basi (bp) è formato da una coppia di oligonucleotidi complementari con una temperatura di fusione duplex di 75 °C in 100 mM di NaCl.- Ordina un oligonucleotide sintetizzato con una singola modifica della biotina 5' e l'altro con una modifica tiolica 5' (con uno spaziatore a sei atomi di carbonio). Posizionare il sito di riconoscimento al centro dell'area fronte/retro.
NOTA: Le sequenze di oligonucleotidi per l'uso con EcoRV sono mostrate di seguito (sito di riconoscimento in grassetto).
5' biotina - AAA ACC GAC ATG TTG ATT TCC TGA AAC GGG ATA TCA TCA AAG CCA TGA ACA AAG CAG CCG - 3'
5' tiolo - CGG CTG CTT TGT TCA TGG CTT TGA TGA TAT CCC GTT TCA GGA AAT CAA CAT GTC GGT TTT - 3'
- Ordina un oligonucleotide sintetizzato con una singola modifica della biotina 5' e l'altro con una modifica tiolica 5' (con uno spaziatore a sei atomi di carbonio). Posizionare il sito di riconoscimento al centro dell'area fronte/retro.
- Utilizzare acqua ultrapura con resistività di 18 MOhm per tutti i passaggi.
- Proteggere tutte le soluzioni contenenti punti quantici dalla luce per prevenire il fotosbiancamento.
- Utilizzare una fonte di aria compressa per completare questo protocollo.
2. Preparazione di materiali di substrato di DNA marcati con punti quantici
NOTA: Oltre agli oligonucleotidi sopra descritti, vedere la Tabella dei materiali per altri materiali e la Tabella 1 per i tamponi necessari per la preparazione di substrati di DNA marcati con punti quantici.
- Ridurre i gruppi tiolici 5' sull'oligonucleotide da accoppiare ai punti quantici.
- Risospendere ciascun oligonucleotide tiolato in acqua a una concentrazione di 100 μM.
- Pipettare 650 μL di acqua in una colonna di spin ad esclusione dimensionale per ogni campione di oligonucleotide e vorticare per ~15 s. Lasciare impacchettare la colonna per 30 min.
- Preparare una soluzione fresca di ditiotreitolo (DTT) da 100 mM immediatamente prima di ogni utilizzo poiché il DTT si degrada rapidamente in soluzione. Aprire con cautela un flaconcino contenente 7,7 mg di DTT e pipettare 500 μL di tampone fosfato di sodio nel flaconcino; vortice per mescolare accuratamente.
- Per ogni oligonucleotide risospeso, preparare una provetta nuova e aggiungere 50 μl di oligonucleotide e 50 μl di soluzione DTT alla provetta. Pipet su e giù per mescolare. Incubare per 30 minuti a temperatura ambiente per ridurre i legami disolfuro tra i gruppi tiolici ossidati all'estremità 5' degli oligonucleotidi.
- Rimuovere i cappucci della colonna di centrifuga e posizionare ciascuna colonna nella provetta di raccolta. Centrifugare le colonne centrifughe a 750 × g per 2 minuti ed eliminare l'eluato.
- Trasferire le colonne di centrifuga in provette da centrifuga fresche. Pipetettare delicatamente l'intero volume (100 μl) di un campione di miscela DNA/DTT su ciascuna colonna preparata. Centrifugare a 750 × g per 2 minuti e misurare l'assorbanza dell'eluato a 260 nm per confermare che la concentrazione è di circa 40 μM.
- Conservare i campioni che non verranno utilizzati immediatamente a -20 °C per evitare l'ossidazione dei gruppi tiolici e la formazione di legami disolfuro.
- Accoppiamento del DNA ai punti quantici
- Inserire un'aliquota di 50 μl del quantum dot stock in un dispositivo di dialisi per ogni costrutto da realizzare, facendo attenzione a non toccare la membrana con il puntale della pipetta. Dializzare contro un volume di tampone dell'acido N-cicloesil-2-amminoetansolfonico (CHES) che sia almeno 1000 volte il volume del campione con agitazione a ~100 giri/min per 15 minuti.
- Preparare una soluzione fresca da 6 mM di sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexano-1-carbossilato (sulfo-SMCC) in tampone CHES immediatamente prima di ogni utilizzo. Aprire con cautela un flaconcino contenente 2 mg di sulfo-SMCC e versare 800 μL di tampone CHES nel flaconcino. Vortice per mescolare accuratamente.
- Utilizzando un pipettatore, recuperare con cura i punti quantici sospesi dal dispositivo di dialisi e trasferirli in una nuova provetta contenente un volume uguale della soluzione sulfo-SMCC; Pipettare su e giù per mescolare. Incubare per 1 ora a temperatura ambiente agitando a 1000 giri/min per consentire al sulfo-SMCC di reagire con le ammine primarie sui punti quantici.
- Utilizzando un pipettatore, trasferire con cura ogni campione in un nuovo dispositivo di dialisi. Per rimuovere l'eccesso di solfo-SMCC, dializzare con un volume di tampone CHES pari ad almeno 1000 volte il volume contenuto nella/e dialisi, agitando, per 15 minuti. Sostituire il tampone 2 volte ed eseguire la dialisi con un tampone CHES fresco 3 volte in totale, consentendo alla dialisi di procedere 15 minuti dopo ogni sostituzione del tampone.
- Per sostituire il tampone in preparazione della seconda reazione, trasferire i dispositivi di dialisi in un becher contenente un volume di soluzione salina tamponata con fosfato (PBS) che sia almeno 1000 volte il volume contenuto nel dispositivo o nei dispositivi. Dializzare agitando per 15 minuti, sostituire il tampone 2 volte ed eseguire la dialisi con PBS fresco 3 volte in totale, consentendo alla dialisi di procedere per 15 minuti dopo ogni sostituzione del tampone.
- Utilizzando un pipettatore, recuperare con cura la soluzione contenente i punti quantici dal dispositivo o dai dispositivi di dialisi e trasferire ciascun campione in una nuova provetta contenente una quantità equimolare di oligonucleotide tiolato diluito in PBS. Combinare un volume uguale di PBS e 1/10del volume del campione di oligonucleotidi ridotto e purificato (concentrazione di circa 40 μM) poiché la concentrazione di punti quantici a questo punto è ~4 μM. Incubare per 2 ore a temperatura ambiente, agitando a 1000 giri/min.
- Aggiungere albumina sierica bovina (BSA, 10 mg/mL) a ciascun campione per ottenere una concentrazione finale di BSA di 0,5 mg/mL. Utilizzando un pipettatore, trasferire con cura ogni campione in un nuovo dispositivo di dialisi e dializzare con un volume di tampone di conservazione almeno 1000 volte il volume contenuto nel dispositivo o nei dispositivi di dialisi. Sostituire il tampone 2 volte ed eseguire la dialisi con un tampone di conservazione fresco 3 volte in totale, consentendo alla dialisi di procedere per 15 minuti dopo ogni sostituzione del tampone.
- Utilizzando un pipettatore, recuperare con cura ogni campione, metterlo in una provetta nuova e conservarlo a 4 °C.
NOTA: Non conservare a -20 °C poiché i punti quantici vengono danneggiati dal congelamento.
- Ricottura di oligonucleotide marcato con punti quantici in oligonucleotide biotinilato
- Risospendere ciascun oligonucleotide biotinilato in un tampone di conservazione a una concentrazione di 100 μM. Conservare i campioni inutilizzati a -20 °C.
- Combinare un campione di oligonucleotide marcato con punti quantici con un eccesso molare di 10 volte di oligonucleotide biotinilato. Poiché la concentrazione stimata di DNA nel campione marcato con punti quantici è di 2 μM, aggiungere 0,2 μL di oligonucleotide biotinilato per ogni 10 μL di questo campione.
- Riscaldare la miscela in un blocco termico a 75 °C (la temperatura di fusione per la regione complementare). Mantieni quella temperatura per 5 minuti. Spegnere il blocco termico e lasciarlo raffreddare lentamente. Conservare il costrutto completato a 4 °C; Non congelare.
3. Funzionalizzazione superficiale dei vetrini coprioggetti
NOTA: Questo processo è stato descritto in precedenza in altri protocolli video JoVE 16,17. Questo protocollo descrive una versione adattata della procedura con piccole modifiche per ospitare un vetrino più piccolo. Vedere la Tabella dei Materiali per gli altri materiali necessari per la funzionalizzazione superficiale dei vetrini.
- Metti 5 vetrini coprioggetti in ogni portavetrini. Assicurati di saltare uno spazio tra ogni coppia di vetrini coprioggetti in modo che non si attacchino l'uno all'altro. Metti i vetrini coprioggetti nel loro supporto in un barattolo con coperchio, aggiungi etanolo al barattolo fino a coprire i vetrini coprioggetti e avvita saldamente la parte superiore. Mettere l'intero barattolo nell'acqua del sonicatore da bagno, senza immergerlo completamente, e sonicare per 30 minuti.
- Riempi un bicchiere o un barattolo pulito con acqua ultrapura. Usa una pinzetta di metallo per rimuovere i vetrini coprioggetti nel loro supporto dall'etanolo e immergili in acqua per risciacquarli. Quindi, trasferire i vetrini coprioggetti e il supporto in un barattolo contenente una soluzione di idrossido di potassio (KOH) 1 M. Avvitare saldamente la parte superiore, posizionare il barattolo nel bagno del sonicatore e sonicare per 30 minuti.
NOTA: Prestare attenzione quando si maneggia la soluzione KOH in quanto è corrosiva e irritante. - Ripetere la sonicazione in soluzione di etanolo e KOH, sciacquando i vetrini coprioggetti in acqua come descritto sopra tra ogni passaggio.
- Trasferisci i vetrini coprioggetti nel loro supporto in un barattolo pulito riempito di acetone puro. Terminare la pulizia sonicando il barattolo come descritto sopra per 30 minuti. Non risciacquare con acqua dopo questo passaggio.
- Utilizzando una pinzetta di metallo, trasferire i vetrini coprioggetti nel loro supporto in un becher pulito da 100 ml contenente 80 ml di acetone fresco e una barra di agitazione su microscala. Posizionare il becher su una piastra di agitazione magnetica impostata ad almeno 1.000 giri/min. Mentre l'acetone viene agitato energicamente, versare 1,6 mL di 3-amminopropiltrietossisilano (APTES) nel becher per ottenere una soluzione al 2% v/v.
NOTA: Prestare attenzione durante il pipettaggio di APTES in quanto è corrosivo. - Lasciare incubare i vetrini coprioggetti nella soluzione per 2 minuti, quindi utilizzare una pinzetta di metallo per trasferire i vetrini coprioggetti nel loro supporto in un bicchiere d'acqua per spegnere la reazione. Sciacquare i vetrini coprioggetti altre due volte, rimettendo l'acqua nel becher.
- Polimerizzare il vetro silanizzato in forno a 120 °C per 75 min. Se non si prosegue immediatamente con il passaggio successivo, conservare i vetrini coprioggetti sotto vuoto per un massimo di alcuni giorni.
- Sciogliere i polietilenglicoli (PEG) derivati dall'estere N-idrossisuccinimide (NHS) in 100 mM di bicarbonato di sodio (pH 8,2). Utilizzare un rapporto 10:1 tra PEG con metossi-terminazione e PEG con biotina, con ~100 mg/mL di PEG con metossi-terminazione.
- Versare 100 μL della soluzione PEG su metà dei vetrini coprioggetti silanizzati asciutti e coprire ciascuno di essi con un secondo vetrino coprioggetti. Usa piccoli pezzi di parafilm posizionati ad ogni angolo come distanziatori per evitare che i vetrini coprioggetti si attacchino l'uno all'altro.
- Incubare i vetrini in ambiente umido per 3,5 ore. Separare i panini coprioggetti. Utilizzare un flacone a spruzzo per lavare ogni vetrino coprioggetti con abbondante acqua e asciugare con aria compressa.
- Conservare i vetrini funzionalizzati, sotto vuoto. Assicurati di tenere il lato trattato con PEG rivolto verso l'alto perché l'altro lato non catturerà il substrato di DNA.
4. Costruzione di dispositivi microfluidici
NOTA: Vedere la Tabella dei materiali per gli altri materiali necessari per la costruzione del dispositivo microfluidico.
- Utilizzando un multiutensile rotante portatile dotato di una punta a punta di diamante conica, praticare due fori negli angoli opposti della slitta di quarzo per fungere da ingresso e uscita. Assicurarsi di fissare la slitta in posizione, lubrificare la punta e scorrere costantemente con acqua durante la perforazione. Dopo ogni esperimento, recuperare il vetrino di quarzo preparato per il riutilizzo immergendo un dispositivo usato nell'acetone per sciogliere l'adesivo e la resina epossidica; Eliminare i componenti rimanenti.
NOTA: La foratura può essere eseguita a mano, ma l'uso di un supporto a pressa per il multiutensile facilita il processo. - Combinare 25 μL di soluzione di streptavidina con 80 μL di PBS e 20 μL di tampone bloccante. Rivestire un vetrino coprioggetti trattato con PEG con questa miscela e incubare in un ambiente umido per 30 minuti.
- Durante l'incubazione del vetrino coprioggetti, preparare il distanziatore di imaging per creare un canale di flusso. Tagliare un pezzo quadrato di 1 pollice di materiale distanziatore per immagini, quindi segnare un canale largo 2 mm allineato alle estremità dei fori praticati nel vetrino di quarzo (Figura 2). Taglia il canale dal materiale distanziatore con un bisturi.
- Staccare un lato del supporto dal distanziatore dell'immagine e posizionarlo con cura sul vetrino di quarzo, facendo attenzione a non coprire i fori di ingresso e uscita. Assicurati di pulire accuratamente il vetrino di quarzo con acetone per rimuovere eventuali residui di adesivo da esperimenti precedenti.
- Lavare il vetrino coprioggetti con acqua e asciugarlo con aria compressa. Rimuovere l'altro lato del supporto dal distanziatore di imaging e inserirlo tra il vetrino al quarzo e il vetrino coprioggetti funzionalizzato rivestito di streptavidina, utilizzando le pinzette di plastica per premere insieme il gruppo e rimuovere le bolle d'aria dall'adesivo.
- Inserire un tubo in polietilene lungo 30 cm in ogni foro del vetrino di quarzo. Assicurarsi di tagliare le estremità del tubo ad angolo per garantire il libero flusso della soluzione. Utilizzare una rastrelliera per provette o un altro supporto per tenere le provette in posizione e sigillare le provette in posizione e i bordi del dispositivo assemblato con resina epossidica.
NOTA: Funziona bene costruire il dispositivo su un pezzo di parafilm, che può essere facilmente staccato dal fondo quando la resina epossidica si è solidificata. - Una volta che la resina epossidica si è solidificata, inserire l'ago smussato sulla siringa vuota nel tubo di uscita del dispositivo e immergere l'estremità del tubo di ingresso in un contenitore riempito con tampone bloccante. Tirare indietro lo stantuffo della siringa per riempire il dispositivo con il tampone di bloccaggio. Lasciare incubare il dispositivo per almeno 30 minuti prima dell'uso.
5. Tethering superficiale del substrato di DNA marcato con punti quantici
NOTA: Oltre al dispositivo microfluidico, al substrato di DNA e al tampone di blocco descritti sopra, vedere la Tabella dei materiali per altri materiali e la Tabella 1 per i tamponi necessari per il tethering superficiale di substrati di DNA marcati con punti quantici.
- Fissare il dispositivo microfluidico alla piastra del tavolino del microscopio con del nastro adesivo, portare l'obiettivo a contatto con la parte inferiore del dispositivo e posizionare l'obiettivo in modo che il campo visivo si trovi all'interno del canale microfluidico.
- Lavare il canale microfluidico con un nuovo tampone di blocco tirando indietro lo stantuffo della siringa dopo aver collegato il tubo di uscita alla pompa a siringa. Verificare che non vi siano bolle intrappolate nel tubo o nel canale.
NOTA: Da questo punto in poi, assicurarsi che il tubo di ingresso sia in un liquido in modo che non vengano introdotte bolle d'aria nel dispositivo. - Diluire 1 μL del substrato di DNA preparato in 1 mL di tampone bloccante. Posizionare il tubo di ingresso nel substrato di DNA diluito e far fluire 800 μl della soluzione di substrato attraverso il canale a una velocità di 200 μl/min. Lasciare che la soluzione di DNA incubi nel canale indisturbata per 15 minuti dopo l'arresto del flusso.
- Far fluire almeno 800 μL di tampone bloccante attraverso il canale a una velocità di 200 μL/min per risciacquare il DNA non legato fuori dal canale.
- Regola la potenza del laser, la messa a fuoco del microscopio e l'angolo TIRF in modo che i punti quantici legati alla superficie siano chiaramente visibili.
NOTA: Sebbene i punti quantici non sbiancano rapidamente, è meglio mantenere la potenza il più bassa possibile per ridurre al minimo il battito delle palpebre.
6. Scissione del DNA mediata da REasi
NOTA: Vedere la Tabella dei materiali per i materiali e la Tabella 1 per i tamponi necessari per la scissione del DNA mediata da REasi.
- Assicurarsi che la fotocamera sia raffreddata alla temperatura di esercizio ottimale e predisposta per lo streaming ad alta velocità con un tempo di esposizione di 0,10 s. Preparati a raccogliere dati per ~4 minuti.
- Lavare il dispositivo microfluidico con 800 μL di tampone sperimentale senza magnesio a una velocità di flusso di 200 μL/min.
- Aggiungere la REasi a un'aliquota (1 mL) di tampone sperimentale senza magnesio e mescolare delicatamente mediante pipettaggio. Utilizzare 4 μL di 100.000 unità/mL di stock, che corrispondono a 400 unità/mL di EcoRV. Flusso di 800 μl dell'enzima diluito attraverso il canale a una velocità di flusso di 200 μl/min.
- Iniziare l'esperimento facendo scorrere un tampone sperimentale contenente magnesio e fluoresceina a una velocità di flusso di 200 μL/min. Iniziare a raccogliere i dati immediatamente dopo aver avviato la pompa a siringa. Dopo aver fatto fluire 800 μL di tampone, interrompere l'acquisizione dei dati.
7. Analisi dei dati
NOTA: Consultare la Tabella dei materiali per il software di analisi dei dati utilizzato per questo protocollo e apportare modifiche se si utilizza una piattaforma di analisi diversa.
- Determinazione del punto di tempo zero
- Sottrai la fluorescenza di fondo da ogni fotogramma del filmato sperimentale utilizzando la funzione imtophat , una funzione di filtraggio morfologico a cappello superiore integrata nella casella degli strumenti di elaborazione delle immagini. Selezionate un disco con un raggio di tre pixel come elemento strutturante. Ottenere l'intensità dello sfondo sottraendo l'immagine filtrata dall'immagine originale.
NOTA: Il filtro mantiene solo le caratteristiche dell'immagine più luminose dello sfondo e più piccole dell'elemento strutturante. - Calcolate la media dello sfondo sottratto su ogni fotogramma del filmato. Utilizzare una traiettoria di questo valore per determinare il punto temporale zero per l'esperimento (Figura 3A). Determinare l'ora di inizio trovando il primo fotogramma in cui la velocità di aumento supera di 3 volte la deviazione standard della velocità di variazione durante il flusso di volume morto.
NOTA: Un forte aumento della velocità di variazione del valore medio dello sfondo sottratto per ogni fotogramma del filmato indica il momento in cui il tampone sperimentale finale contenente il colorante tracciante è entrato nella cella di flusso (Figura 3B).
- Sottrai la fluorescenza di fondo da ogni fotogramma del filmato sperimentale utilizzando la funzione imtophat , una funzione di filtraggio morfologico a cappello superiore integrata nella casella degli strumenti di elaborazione delle immagini. Selezionate un disco con un raggio di tre pixel come elemento strutturante. Ottenere l'intensità dello sfondo sottraendo l'immagine filtrata dall'immagine originale.
- Calcolo e analisi della traiettoria dell'intensità dei punti quantici
- Calcola una proiezione di intensità massima per l'insieme di fotogrammi di filmati corretti per lo sfondo registrati prima dell'arrivo del colorante tracciante. Determinare le posizioni dei punti quantici con una precisione di un pixel in questa immagine di proiezione utilizzando una funzione di rilevamento dei picchi, ad esempio pkfnd.
- Genera traiettorie di intensità per singoli punti quantici calcolando l'intensità media in ogni fotogramma del filmato di una regione quadrata di tre pixel per lato che circonda la posizione restituita dalla funzione di ricerca dei picchi.
NOTA: La correzione dello sfondo sopra descritta rimuove gli artefatti introdotti dal colorante tracciante. Le traiettorie di intensità risultanti dovrebbero essere relativamente piatte, ma includere comunque le fluttuazioni di intensità che si verificano naturalmente (Figura 4). - Identificare gli eventi di scomparsa dei punti quantici mediante l'analisi statistica delle traiettorie di intensità. Per ciascuna traiettoria di intensità del punto quantico, calcolare un'intensità di soglia superiore al valore minimo di una frazione appropriata della differenza tra i valori massimo e minimo per tale traiettoria, a seconda del rumore osservato nelle traiettorie.
NOTA: La fluttuazione dell'intensità per il punto quantico è in genere molto maggiore della fluttuazione di fondo. Confrontando questi valori è possibile determinare un valore appropriato: la soglia selezionata deve essere superiore al valore di fondo più alto, ma idealmente deve essere inferiore al valore più basso per la fluorescenza dei punti quantici. Per i dati presentati, la soglia utilizzata è stata calcolata in modo da essere superiore al valore minimo per ciascuna traiettoria più un terzo della differenza tra i valori massimo e minimo per tale traiettoria. Si può ritenere che un punto quantico scompaia nell'ultimo punto temporale in cui l'intensità osservata nella sua posizione era superiore a questa soglia. - Confermare gli eventi di presunta scomparsa. Includere gli eventi nell'analisi finale solo se l'intensità osservata è superiore al punto medio tra i valori di intensità minima e massima per la traiettoria per oltre la metà dei fotogrammi del filmato prima del momento della scomparsa e se la deviazione standard della traiettoria di intensità è diminuita dopo l'evento.
NOTA: Altri test statistici possono essere utilizzati per garantire che vengano registrati solo gli eventi di scomparsa validi.
Risultati
La cella di flusso è direttamente accoppiata a un obiettivo di ingrandimento 60x ad immersione in olio ad alta apertura numerica su un microscopio invertito dotato di illuminazione laser per l'imaging TIRF attraverso l'obiettivo (Figura 5A). Dopo aver introdotto il substrato di DNA e aver lavato via il DNA in eccesso e i punti quantici, ci sono in genere migliaia di singoli punti quantici in un campo visivo (Figura 5B). Questi punti quantici sono stabilmente attaccati alla superficie del vetro e non subiscono un notevole oscuramento o sbiancamento significativo nel corso del tempo dell'esperimento. Tuttavia, se un tampone contenente sia magnesio che una REasi appropriata viene fatto scorrere attraverso il canale di flusso, alla fine di un tipico periodo di osservazione di quattro minuti, almeno il 30% dei punti quantici presenti all'inizio di un esperimento sarà scomparso dal campo visivo. A conferma del fabbisogno di magnesio, quando la REasi viene fatta scorrere attraverso il canale in assenza di magnesio, almeno il 95% dei punti quantici presenti all'inizio dell'esperimento può ancora essere visto alla fine del periodo di osservazione (Figura 5C). Tuttavia, quando il tampone contenente magnesio viene fatto scorrere immediatamente dopo aver permesso alla REasi di legare il DNA legato alla superficie in assenza di magnesio, fino alla metà dei punti quantici sarà scomparsa entro la fine del periodo di osservazione (Figura 5D), in modo simile a quanto osservato quando REasi e magnesio sono stati fatti scorrere insieme attraverso il canale. L'esatta resa degli eventi dipenderà dall'efficienza dell'enzima nelle condizioni utilizzate. Quando il tampone contenente magnesio viene fatto scorrere senza aver precedentemente introdotto la REasi appropriata nel canale di flusso, meno del 5% dei punti quantici scompare durante il periodo di osservazione e non vi è alcun picco distinguibile per un istogramma degli eventi. Questo risultato indica che le molecole di REasi prelegate mediano la scissione del DNA che lega i punti quantici alla superficie del vetro, e questa scissione del DNA è responsabile della stragrande maggioranza degli eventi di scomparsa dei punti quantici osservati in questi esperimenti.
La scomparsa dei punti quantici avviene rapidamente e si traduce in una brusca diminuzione della traiettoria di intensità, fornendo una chiara indicazione del momento in cui una data molecola di DNA viene scissa (Figura 5E). La determinazione dei tempi degli eventi è realizzata mediante analisi matematica delle traiettorie di intensità. I singoli punti quantici generano traiettorie di intensità con una varianza significativamente più elevata rispetto allo sfondo nelle condizioni dell'immagine utilizzate, quindi gli eventi putativi vengono confermati quando la varianza della traiettoria di intensità diminuisce a un livello paragonabile alla varianza dello sfondo dopo il calo di intensità osservato. Inoltre, le traiettorie che includono un ampio grado di battito di ciglia prima di un presunto evento di scomparsa sono escluse dall'analisi finale. Tuttavia, un esperimento tipico produce centinaia di eventi che soddisfano questi criteri e i risultati di più esperimenti possono essere raggruppati per fornire statistiche adeguate per consentire la stima dei parametri N e β mediante l'adattamento della curva dei minimi quadrati non lineare o la stima dei parametri di massima verosimiglianza.
I dati rappresentativi presentati sono stati raccolti eseguendo questo esperimento con la ben studiata REase di tipo IIP, EcoRV (Video 1). Il substrato di DNA duplex lungo 60 bp è costruito con una molecola di biotina all'estremità 5' di un filamento del duplex e un punto quantico attaccato covalentemente all'estremità 5' dell'altro filamento. Il substrato di DNA contiene una singola copia della sequenza di riconoscimento, GAT↓ATC, che viene scissa da EcoRV al centro, come indicato dalla freccia verso il basso (↓). EcoRV è stato prelegato al substrato di DNA in assenza di magnesio, quindi è stato fatto scorrere un tampone contenente magnesio per avviare la scissione del DNA. I dati rappresentativi includono i risultati aggregati di cinque esperimenti separati, per un totale di 3451 eventi di scissione osservati. Dopo aver escluso gli eventi che si sono verificati prima del punto temporale zero o al di fuori del picco di attività prominente, sono rimasti 2987 eventi, sufficienti per popolare un istogramma con contenitori di un secondo. Per estrarre i valori di N e β dai dati sono stati utilizzati sia l'adattamento della curva dei minimi quadrati non lineare che la stima dei parametri di massima verosimiglianza. I due metodi di stima dei parametri erano in accordo (Figura 6A), con N = 3,52 (intervallo di confidenza al 95%: 3,32-3,71) e β = 5,78 s (intervallo di confidenza al 95%: 5,41-6,15 s) per l'adattamento dei minimi quadrati non lineari e N = 3,41 (intervallo di confidenza al 95%: 3,25-3,58) e β = 6,06 s (intervallo di confidenza al 95%: 5,75-6,39 s) per la stima di massima verosimiglianza.
La stima di almeno tre passaggi cinetici è ragionevole dato ciò che è noto sul meccanismo di EcoRV. Questo enzima è noto per legare il DNA in modo non specifico in assenza di magnesio14. Le osservazioni della fase di soluzione di massa dei cambiamenti nella fluorescenza intrinseca dei residui di triptofano in EcoRV in condizioni di flusso arrestato hanno suggerito che l'EcoRV che si è legato al DNA in assenza di magnesio deve subire un cambiamento conformazionale prima dell'ingresso del magnesio nel sito attivo18. Questa osservazione è corroborata dai dati sulla struttura cristallina19. I risultati degli studi di fluorescenza del triptofano indicano anche che la scissione del DNA e il rilascio del prodotto sono fasi separate che si verificano con tassi simili18. Pertanto, un meccanismo di reazione ragionevole per la scissione del DNA mediata da EcoRV in questo esperimento è:
ES → ES* → EP → E+P
In questo meccanismo, l'ES, il complesso enzima-substrato iniziale, si forma quando l'EcoRV è prelegato al DNA in assenza di magnesio. Quando il magnesio entra nella cella a flusso, il complesso enzima-substrato subisce un cambiamento conformazionale per diventare ES*, il complesso enzima-substrato attivato. Questo complesso attivato quindi scinde il DNA, ma non rilascia immediatamente le molecole del prodotto, diventando un complesso enzima-prodotto (EP). Infine, il prodotto, P, viene rilasciato nella fase finale. Questo meccanismo richiede tre passaggi, che sono in accordo con i dati presentati. La stima risultante per il tempo medio di attesa per ogni passo è di ~6 s, equivalente a un tasso di 0,17 s-1 per ogni passo. Questo calcolo è in generale in accordo con le precedenti stime dei tassi per questi processi, dell'ordine di 0,3-0,4 s-1 per la scissione del DNA e il rilascio del prodotto e ~0,5 s-1 per il riarrangiamento conformazionale.

Figura 1: Schema di reazione enzimatica e DNA marcato. Le molecole di DNA marcate con punti quantici sono legate alla superficie di vetro funzionalizzata tramite il legame biotina-streptavidina-biotina e osservate utilizzando la microscopia TIRF. Le molecole di DNA contengono il sito di riconoscimento per la REasi di interesse. Quando una molecola di DNA viene scissa dalla REasi, il punto quantico è libero di diffondersi lontano dalla superficie e fuori dalla zona di illuminazione. Abbreviazioni: REase = endonucleasi di restrizione; PEG = polietilenglicole; TIRF = fluorescenza a riflessione interna totale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Dispositivo a cella a flusso microfluidico. (A) Vista esplosa che mostra i tre strati utilizzati per creare il dispositivo: il vetrino in vetro funzionalizzato sul fondo, il vetrino in quarzo con fori di ingresso e uscita sulla parte superiore e il distanziatore di imaging biadesivo con un canale tagliato al centro. (B) Un dispositivo completo con tubi di polietilene sigillati in posizione e con bordi rivestiti di resina epossidica. Clicca qui per visualizzare una versione più grande di questa figura.
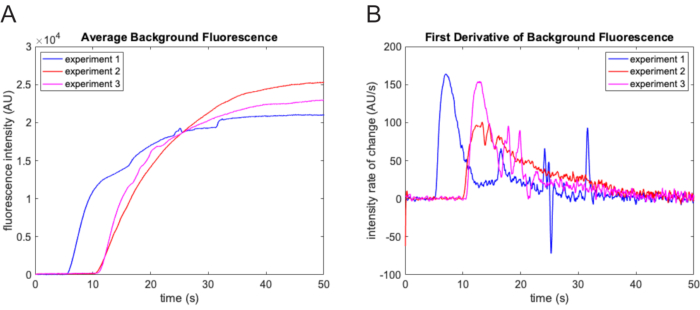
Figura 3: Determinazione del punto temporale zero. (A) Intensità media dello sfondo determinata utilizzando la funzione di filtraggio morfologico del cappello superiore su ogni fotogramma del filmato. L'intensità di fondo aumenta notevolmente quando il tampone sperimentale contenente fluoresceina entra nel campo visivo. I risultati di tre diversi esperimenti sono mostrati qui. C'è una variazione sostanziale nel tempo di ritardo da esperimento a esperimento. (B) Il forte aumento del tasso di variazione della traiettoria di intensità della fluorescenza di fondo facilita la determinazione accurata del punto di tempo zero. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Correzione dello sfondo. L'aumento dell'intensità di fondo dovuto al colorante tracciante di fluoresceina che indica l'arrivo del magnesio può essere visto nelle traiettorie di intensità della fluorescenza grezza per i singoli punti quantici (traiettoria grigia). Dopo aver sottratto lo sfondo utilizzando la funzione di filtraggio morfologico del cappello a cilindro, gli artefatti introdotti dal colorante tracciante sono stati rimossi dalla traiettoria (traiettoria nera). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Esperimento TIRF a singola molecola per osservazioni parallelizzate di eventi di scissione del DNA. (A) Una cella a flusso costruita su una superficie di vetro funzionalizzata progettata per catturare il DNA marcato con punti quantici è direttamente accoppiata a un obiettivo per microscopio ad immersione in olio 60x ad alta apertura numerica per l'imaging TIRF. (B) Immagine rappresentativa dall'inizio di un esperimento. Il DNA marcato con punti quantici è stato caricato nella cella di flusso e i punti quantici non legati in eccesso sono stati lavati via. I singoli punti quantici legati al DNA possono essere risolti l'uno dall'altro. (C) Lo stesso campo visivo dopo che il tampone contenente una REasi in grado di scindere i lacci del DNA è stato fatto scorrere attraverso il canale di flusso in assenza di magnesio per quattro minuti. Non c'è stata alcuna perdita significativa di punti quantici legati al DNA. (D) Lo stesso campo visivo alla conclusione di un esperimento. Il tampone contenente magnesio è stato fatto scorrere attraverso il canale di flusso immediatamente dopo aver fatto fluire REase in un tampone privo di magnesio. Questa immagine è stata acquisita dopo circa quattro minuti di flusso tampone. Le REasi prelegate hanno tagliato molti dei legami del DNA, rilasciando i punti quantici dalla superficie. Per facilitare la visualizzazione, in ogni immagine viene mostrato solo il quadrante centrale del campo visivo dell'obiettivo. Dieci fotogrammi di film (equivalenti a un secondo di tempo di osservazione) sono stati mediati per diminuire gli effetti del lampeggio dei punti quantici. Le impostazioni di luminosità e contrasto sono identiche per tutte e tre le immagini. (E) Traiettorie rappresentative dell'intensità della fluorescenza da posizioni di immagini in cui un punto quantico era presente all'inizio dell'esperimento. Le traiettorie ottenute dalle posizioni delle immagini corrispondenti a punti quantici che non vengono rilasciati dalla superficie (grigio) possono mostrare brevi cali a un livello di intensità inferiore, ma iniziano e terminano a un livello di intensità elevato. Le traiettorie ottenute dalle posizioni delle immagini, corrispondenti a punti quantici che vengono rilasciati dalla superficie (nero), mostrano una rapida diminuzione di intensità fino a un basso livello di fondo che è istantaneo rispetto alla risoluzione temporale dell'esperimento (10 Hz). I punti quantici rilasciati non riappaiono entro il periodo di osservazione di quattro minuti. Abbreviazioni: REase = endonucleasi di restrizione; TIRF = fluorescenza a riflessione interna totale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Analisi della distribuzione del tempo di permanenza della scissione del DNA mediata da EcoRV. (A) Istogramma e inviluppi previsti per i 2987 eventi di clivaggio che si verificano all'interno del picco di attività principale in una serie di cinque esperimenti aggregati con EcoRV. Le due curve previste sono quasi identiche e i residui di adattamento (sotto l'istogramma) non indicano un errore sistematico nell'adattamento dei minimi quadrati non lineare. (B) Istogramma dell'intera serie di 3393 eventi di scissione che si sono verificati dopo il punto di tempo zero. La curva prevista dalla stima di massima verosimiglianza dei parametri (linea ininterrotta, curva MLE) assumendo una distribuzione di probabilità gamma non riesce a racchiudere l'istogramma. La curva prevista da un adattamento dei minimi quadrati non lineare della formula per la distribuzione di probabilità gamma alle altezze dei contenitori (linea tratteggiata, curva NLS) è superiore, ma i residui dell'adattamento (sotto l'istogramma) rivelano un errore sistematico. Abbreviazioni: MLE: stima della massima verosimiglianza; NLS = minimi quadrati non lineari. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome del buffer | Componente | Concentrazione | pH a 25 °C |
| tampone fosfato di sodio | fosfato di sodio | 100 mM | 8.3 |
| Tampone CHES | Acido N-cicloesil-2-amminoetansolfonato (CHES) | 10 mM | 9.0 |
| PBS | fosfato di sodio | 3 mM | 7.2 - 7.6 |
| cloruro di sodio | 150 mM | ||
| fosfato di potassio | 1,05 mM | ||
| buffer di archiviazione | cloruro di sodio | 100 mM | 8.0 |
| Tris-HCl | 50 mM | ||
| albumina sierica bovina (BSA) | 0,5 mg/mL | ||
| bicarbonato di sodio | bicarbonato di sodio | 100 mM | 8.2 |
| buffer di blocco | Tris-HCl | 20 mM | 7.5 |
| Acido etilendiamminotetraacetico (EDTA) | 2 mM | ||
| cloruro di sodio | 50 mM | ||
| Interpolazione-20 | 0,005% (v/v) | ||
| albumina sierica bovina (BSA) | 0,2 mg/mL | ||
| tampone sperimentale senza magnesio | cloruro di sodio | 100 mM | 7.9 |
| Tris-HCl | 50 mM | ||
| ditiotreitolo (DTT) | 1 mM | ||
| tampone sperimentale con magnesio | cloruro di sodio | 100 mM | 7.9 |
| Tris-HCl | 50 mM | ||
| cloruro di magnesio | 10 mM | ||
| ditiotreitolo (DTT) | 1 mM | ||
| fluoresceina | Regola in base alle condizioni di imaging | ||
| Tampone DNasi | Cloruro di magnesio | 2,5 mM | 7.6 |
| Tris-HCl | 10 mM | ||
| cloruro di calcio | 0,1 mM |
Tabella 1: Tabella dei buffer.
Video 1: Esempio di filmato a singola molecola. Clicca qui per scaricare questo video.
Discussione
Il substrato di DNA per questo saggio è marcato con un punto quantico utilizzando uno schema di reazione in due fasi utilizzando sulfo-SMCC. Questo reticolante bifunzionale è costituito da una porzione estere NHS che può reagire con un'ammina primaria e da una porzione maleimmidica che può reagire con un gruppo sulfidrilico20. Gli oligonucleotidi tiolati utilizzati per preparare il substrato vengono spediti nella loro forma ossidata. È importante ridurli e purificarli, come descritto, prima di procedere con la procedura di accoppiamento, altrimenti l'efficienza della reazione di accoppiamento sarà ridotta. È fondamentale preparare soluzioni fresche di DTT e sulfo-SMCC immediatamente prima dell'uso. Queste molecole si idrolizzano rapidamente in soluzione acquosa. Prestare attenzione durante il trasferimento delle soluzioni all'interno e all'esterno dei dispositivi di dialisi: se la punta della pipetta tocca la membrana, si verifica una rottura e il campione va perso. L'ideale è completare tutti i passaggi della sezione 2 senza interruzioni per evitare la perdita di campione.
La reazione di accoppiamento utilizzata per generare l'oligonucleotide marcato con punti quantici è inefficiente. Dopo aver combinato e ricotto l'oligonucleotide marcato con punti quantici con l'oligonucleotide biotinilato, ci sarà un eccesso di DNA non marcato presente nel campione di substrato preparato. Sebbene queste specie possano legarsi alla superficie funzionalizzata, non sono visibili nelle condizioni di imaging raccomandate e non contribuiscono alla lettura dell'esperimento. Per confermare che la scissione del costrutto completato provoca il rilascio di punti quantici dalla superficie, eseguire il tethering della superficie come descritto nella sezione 5 del protocollo e visualizzare una regione della superficie per determinare quanti punti quantici sono presenti. Quindi, far fluire 800 μL di un'aliquota da 1 mL di 10 U/mL di DNasi I nel tampone DNasi (2,5 mM MgCl2; 10 mM Tris-HCl; 0,1 mM CaCl2, pH 7,6) attraverso il canale microfluidico. Lasciare incubare la DNasi I nel canale per 15 minuti, lavare con 800 μL di tampone DNasi (senza enzima) e visualizzare la stessa regione di interesse. Circa il 90% dei punti quantici dovrebbe essere rilasciato dalla superficie se il costrutto e la superficie sono adeguatamente preparati.
Per legare in modo covalente una miscela di molecole PEG con terminazione con metossi e biotina, le superfici in vetro devono essere trattate per catturare il substrato di DNA marcato con punti quantici sulla superficie per l'imaging. È di fondamentale importanza eseguire rigorosamente tutte le fasi di pulizia. Sono necessarie più fasi di pulizia per rimuovere le impurità superficiali e per esporre i gruppi ossidrilici sulla superficie del vetro che reagiranno con i gruppi alcossi sul silano per formare legami covalenti. Sebbene sia importante sciacquare i vetrini con acqua ultrapura tra le fasi di pulizia utilizzando soluzioni di etanolo e KOH, non utilizzare acqua dopo la pulizia finale con acetone. La presenza di acqua in eccesso durante la fase di silanizzazione favorirà la multimerizzazione del silano in soluzione a scapito del legame superficiale. Inoltre, la soluzione di silano non deve essere preparata in anticipo. Per prolungarne la vita utile, il materiale silano deve essere conservato in un armadio essiccatore.
Il saggio di scissione di una singola molecola è progettato per essere eseguito in un dispositivo microfluidico (Figura 2). Costruisci un nuovo dispositivo prima di ogni esperimento, riutilizzando la slitta di quarzo forata che forma la parte superiore del dispositivo. Questa procedura è stata descritta in precedenza16,17 e le sue variazioni sono ampiamente utilizzate per la microscopia TIRF. La procedura qui descritta è stata adattata per ospitare vetrini quadrati da 1 pollice. È importante lavorare rapidamente per costruire il dispositivo in modo che la superficie funzionalizzata non si degradi mentre è esposta all'aria. La resina epossidica è fluorescente, quindi fai attenzione a evitare di coprire il canale con essa. Il vetrino in quarzo forato può essere recuperato per il riutilizzo immergendo il dispositivo usato nell'acetone e smontandolo.
Questo test è progettato per essere eseguito su un microscopio invertito dotato di capacità di illuminazione a riflessione interna totale attraverso l'obiettivo, dotato di una fotocamera EMCCD (Electron-Multiplying Charge-Coupled Device) per la raccolta dei dati. Utilizzare un obiettivo ad alta apertura numerica 60x e una sorgente di eccitazione laser da 488 nm e utilizzare un cubo filtrante che includa un filtro di emissione passa-banda singolo e un dicroico a bordo singolo adatto per il laser e un filtro di emissione che consenta il passaggio sia della fluorescenza dei punti quantici (picco di emissione a 655 nm) che di almeno una parte della fluorescenza della fluoresceina (picco di emissione a 512 nm). Ad esempio, i dati qui presentati sono stati raccolti con un cubo di filtro dotato di un filtro di eccitazione passa-banda singolo da 482/18 nm, un dicroico a bordo singolo da 488 nm e un filtro di emissione passa-banda singolo da 675/67 nm. Il saggio può essere effettuato mediante imaging in un singolo canale spettrale, come descritto in questo protocollo. Se lo si desidera, il test può essere effettuato anche suddividendo l'immagine di fluorescenza in due canali spettrali, uno per i punti quantici, un altro per l'emissione di fluoresceina. A questo scopo sarebbe appropriato uno splitter di immagini dotato di uno specchio dicroico a bordo singolo da 640 nm. Tuttavia, l'imaging multicanale non è richiesto.
È importante lavare via tutti i substrati di DNA non legati dal canale prima di iniziare l'esperimento. La fluoresceina è inclusa a una concentrazione di 50 μM nel tampone finale per segnalare l'arrivo del magnesio nel campo visivo. A seconda della configurazione del microscopio, potrebbe essere necessario regolare la concentrazione di fluoresceina in modo che la sua emissione non oscuri i punti quantici. Assicurarsi di iniziare ad acquisire i dati subito dopo aver avviato il flusso del tampone contenente magnesio. Registrare i dati durante il tempo di ritardo prima dell'arrivo del tampone contenente magnesio in modo da poter calcolare la media e la deviazione standard del tasso di variazione dell'intensità di fondo per facilitare la determinazione accurata del punto temporale zero. Si noti che il punto temporale zero più accurato è il primo fotogramma in cui la velocità di variazione della fluorescenza di fondo supera le fluttuazioni naturali in questa misurazione. L'intensità della fluorescenza di fondo continua ad aumentare dopo questo punto man mano che la fluoresceina si diffonde attraverso lo strato limite stabilito dal flusso laminare nel canale. Il magnesio, che è molto più piccolo della fluoresceina, si diffonde più rapidamente e ci si può aspettare che la sua concentrazione raggiunga l'equilibrio molto più rapidamente.
Per i dati di esempio presentati, un istogramma di tutti i 3393 eventi che si sono verificati dopo il punto di tempo zero mostra un picco prominente entro i primi 60 s degli esperimenti. Molti contenitori che corrispondono a tempi di ritardo più lunghi non contengono eventi e quelli che contengono eventi ne contengono solo alcuni, in modo simile a quanto osservato quando non è presente alcuna REase attiva. Un adattamento non lineare della curva dei minimi quadrati della distribuzione di probabilità gamma a tutti i dati raggruppati ha portato a stime dei parametri di N = 3,06, con un intervallo di confidenza del 95% di 2,94-3,18, e β = 7,04 s, con un intervallo di confidenza del 95% di 6,72-7,36 s. Tuttavia, le stime di massima verosimiglianza per i parametri erano N = 1,30 (intervallo di confidenza al 95%: 1,24-1,35) e β = 26,7 s (intervallo di confidenza al 95%: 25,3-28,1 s) e, mediante ispezione visiva, la curva prevista dalla stima del parametro di massima verosimiglianza non corrisponde bene ai dati (Figura 6B). Inoltre, sebbene la curva prevista dai parametri stimati dall'adattamento dei minimi quadrati non lineari segua relativamente bene la forma dell'istogramma delle frequenze degli eventi osservati, i residui (differenza tra le frequenze degli eventi osservati e quelli previsti) diventano costantemente positivi per i punti temporali oltre i 60 s circa (Figura 6B).
Poiché la frequenza degli eventi nei punti temporali successivi è paragonabile alla frequenza di scomparsa dei punti quantici osservata in assenza di enzima attivo, è probabile che la maggior parte di questi eventi successivi faccia parte di un basso livello di fondo di perdita di punti quantici che non è causato dalla scissione del DNA mediata da REasi. È quindi ragionevole escludere tali contenitori dal montaggio. Quando tutti gli eventi che si sono verificati più di 60 secondi dopo il punto di tempo zero di ciascun esperimento sono stati esclusi, i due metodi di stima dei parametri erano in accordo (Figura 6A). Pertanto, per ottenere le migliori stime dei parametri, escludere gli eventi al di fuori del picco di attività e confermare che i risultati della stima del parametro di massima verosimiglianza e dell'adattamento della curva dei minimi quadrati non lineari siano convergenti.
L'approccio sperimentale delineato in questo articolo consente osservazioni in tempo reale della scissione del DNA mediata da REasi a livello di singola molecola. Centinaia di eventi possono essere osservati in ogni singolo esperimento, e i risultati di diversi esperimenti possono essere raggruppati per ottenere il gran numero di osservazioni necessarie per caratterizzare la distribuzione dei tempi di attesa adattando i dati alla distribuzione di probabilità gamma. Sebbene questo test possa essere utilizzato solo come descritto con le REasi che si legano ma non scindono il DNA in assenza di magnesio o di un altro cofattore, molte REasi rientrano in questa categoria. È importante riconoscere che i dettagli esatti di tutte le fasi intermedie non saranno rivelati da questo test. I passaggi che sono significativamente più veloci del passaggio di limitazione della velocità non influiranno sulla distribuzione del tempo di permanenza e non figureranno nel valore di N estratto dai dati12. Analogamente, il valore dei β che possono essere estratti dai dati è una media approssimativa del tempo di attesa per i passaggi che influiscono sulla distribuzione del tempo di permanenza. Il tempo di attesa effettivo per ogni passaggio può variare dal valore di β12. Tuttavia, il saggio di scissione di una singola molecola descritto in questo articolo può fornire la prova della presenza di più passaggi intermedi lungo il percorso di reazione, anche se gli stessi intermedi di reazione non possono essere osservati direttamente.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse
Riconoscimenti
Questo lavoro è stato supportato dal premio numero K12GM074869 all'ECM dall'Istituto Nazionale di Scienze Mediche Generali. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali dell'Istituto Nazionale di Scienze Mediche Generali o del National Institutes of Health.
Materiali
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltriethoxysilane (APTS) | Sigma Aldrich | 440140-100ML | Store in dessicator box |
| 5 Minute Epoxy | Devcon | 20845 | use for sealing the microfluidic device |
| acetone | Pharmco | 329000ACS | use for cleaning coverslips |
| bath sonicator | Fisher Scientific | CPXH Model 2800 | catalog number 15-337-410 |
| Beaker, glass, 100 mL | |||
| Benchtop centrifuge | |||
| Biotin-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | BIO-SVA-5K | Succinimidyl valerate has a longer half-life than succinimidyl carbonate |
| biotinylated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Biotin modification and HPLC purification |
| Bovine Serum Albumin (BSA) | VWR | 0903-5G | prepare a 10 mg/mL solution (aq) and heat to 95 °C before using |
| Centri-Spin-10 Size Exclusion Spin Columns | Princeton Separations | CS-100 or CS-101 | used to purify thiolated oligonucleotides after reducing the disulfide bond |
| Centrifuge tubes 1.5. mL | |||
| Coverslips, 1-inch square glass | |||
| coverslip holders | |||
| diamond point wheel | Dremel | 7134 | use for drilling holes in quartz flow cell topper |
| dithiothreitol (DTT) | Thermo Scientific | A39255 | No-Weigh Format, 7.7 mg/vial |
| drill press rotary tool workstation stand | Dremel | 220-01 | facilitates quartz drilling |
| EcoRV (REase used to generate example data) | New England Biolabs | R0195T or R0195M | Use 100,000 units/mL stock to avoid adding excess glycerol Check REBASE for suppliers of other REases |
| ethanol | various | CAS 64-17-5 | denatured or 95% are acceptable, use for cleaning coverslips |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | EDS | BioUltra, anhydrous, store in dessicator box |
| Flea Micro Spinbar | Fisherbrand | 14-513-65 | 3 mm x 10 mm size to fit beneath coverslip rack |
| fluorescein | Acros Organics | 17324 | use to make experimental buffers |
| gravity convection oven | Binder | 9010-0131 | |
| handheld rotary multitool | Dremel | 8220 | use for drilling holes in quartz flow cell topper |
| ImagEM X2 EM-CCD Camera | Hamamatsu | C9100-23B | air cooling is adequate for this experiment, use HCImage software or similar to control |
| Imaging spacer, double-sided, adhesive | |||
| Jar, glass with screw cap, (approximately 50 mm diameter by 50 mm high) | |||
| magnesium chloride hexahydrate | Fisher Bioreagents | BP214-500 | use to make experimental buffer with magnesium |
| MATLAB software | Data analysis | ||
| metal tweezers | Fisher Brand | 16-100-110 | |
| methoxy-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | M-SVA-5K | Both PEGs should have the same NHS ester so that the rate of reaction is consistent |
| microcentrifuge | Eppendorf | 5424 | |
| multiposition magnetic stirrer | VWR | 12621-022 | |
| N-cyclohexyl-2-aminoethanesulfonic acid (CHES) | Acros Organics | AC20818 | CAS 103-47-9, use to make CHES buffer |
| orbital shaker and heater for microcentrifuge tubes | Q Instruments | 1808-0506 | with 1808-1061 adaptor for 24 x 2.0 mL or 15 x 0.5 mL tubes |
| Parafilm | |||
| PE60 Polyethylene tubing (inner diameter 0.76 mm, outer diameter 1.22 mm) | Intramedic | 6258917 | 22 G blunt needles are a good fit for this tubing size |
| Phosphate-Buffered Saline (PBS) 10x | Sigma Aldrich | P7059 | Use at 1x strength |
| potassium hydroxide | VWR Chemicals BDH | BDH9262 | use a 1 M solution to clean coverslips |
| Qdot 655 ITK Amino (PEG) Quantum Dots | Invitrogen | Q21521MP | |
| Quartz Slide, 1 inch square, 1 mm thick | Electron Microscopy Sciences | 72250-10 | holes must be drilled in the corners for inlet and outlet tubing insertion |
| reinforced plastic tweezers | Rubis | K35a | use for handling coverslips and building microfluidic device |
| SecureSeal Adhesive Sheets | Grace Biolabs | SA-S-1L | cut to form spacer for microfluidic device |
| Single channel syringe pump for microfluidics | New Era Pump Systems | NE-1002X-US | fitted with a 50 mL syringe and a 22 G blunt needle |
| Slide-a-Lyzer MINI Dialysis Devices, 10 kDa MWCO, 0.1 mL | Thermo Scientific | 69570 or 69572 | used for buffer exchange during quantum dot coupling to DNA |
| sodium bicarbonate | EMD Millipore | SX0320 | use to make buffer for surface functionalization; 100 mM, pH 8 |
| sodium chloride | Macron | 7581-12 | use to make experimental buffers |
| Sodium phosphate dibasic solution (BioUltra, 0.5 M in water) | Sigma Aldrich | 94046 | use to make 100 mM sodium phosphate buffer |
| Sodium phosphate monobasic solution (BioUltra, 5M in water) | Sigma Aldrich | 74092 | use to adjust pH of 100 mM sodium phosphate buffer |
| Streptavidin from Streptomyces avidinii | Sigma Aldrich | S4762 | dissolve at 1 mg/mL and store 25 mL aliqouts at -20 ? |
| Sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexane-1-carboxylate (sulfo-SMCC) | Thermo Scientific | A39268 | No-Weigh Format, 2 mg/vial |
| Syringe fitted with blunt 21 G needle | |||
| Syringe pump | |||
| thiolated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Thiol Modifier C6 S-S and HPLC purificaiton |
| TIRF imaging system with 488 nm laser illumination | various | custom built | |
| Tris -HCl | Research Products International | T60050 | use to make experimental buffers |
| Tris base | JT Baker | 4101 | use to make experimental buffers |
| Tween-20 | Sigma | P7949 | use to make blocking buffer |
| Ultrapure water | |||
| vortex mixer | VWR | 10153-842 | |
| Wash-N-Dry Coverslip Rack | Electron Microscopy Sciences | 70366-16 | used for surface functionalization of coverslips |
Riferimenti
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5905-5908 (2005).
- Loenen, W. A., Dryden, D. T., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Research. 42 (1), 3-19 (2014).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE--a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research. 43, Database issue 298-299 (2015).
- Bonnet, I., et al. Sliding and jumping of single EcoRV restriction enzymes on non-cognate DNA. Nucleic Acids Research. 36 (12), 4118-4127 (2008).
- Biebricher, A., Wende, W., Escude, C., Pingoud, A., Desbiolles, P. Tracking of single quantum dot labeled EcoRV sliding along DNA manipulated by double optical tweezers. Biophysical Journal. 96 (8), 50-52 (2009).
- Blainey, P. C., et al. Nonspecifically bound proteins spin while diffusing along DNA. Nature Structural & Molecular Biology. 16 (12), 1224-1229 (2009).
- Huang, C. -F., et al. Direct visualization of DNA recognition by restriction endonuclease EcoRI. Journal of Experimental & Clinical Medicine. 5 (1), 25-29 (2013).
- Palma, M., et al. Selective biomolecular nanoarrays for parallel single-molecule investigations. Journal of American Chemical Society. 133 (20), 7656-7659 (2011).
- Reinhard, B. M., Sheikholeslami, S., Mastroianni, A., Alivisatos, A. P., Liphardt, J. Use of plasmon coupling to reveal the dynamics of DNA bending and cleavage by single EcoRV restriction enzymes. Proceedings of the National Academy of Sciences of the United States of America. 104 (8), 2667-2672 (2007).
- van den Broek, B., Noom, M. C., Wuite, G. J. DNA-tension dependence of restriction enzyme activity reveals mechanochemical properties of the reaction pathway. Nucleic Acids Research. 33 (8), 2676-2684 (2005).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Loparo, J. J., van Oijen, A. Single-molecule enzymology. Handbook of Single-Molecule Biophysics. Hinterdorfer, P., Oijen, A. , Springer. New York, NY. 165-182 (2009).
- Taylor, J. D., Badcoe, I. G., Clarke, A. R., Halford, S. E. EcoRV restriction endonuclease binds all DNA sequences with equal affinity. Biochemistry. 30 (36), 8743-8753 (1991).
- Pingoud, A., Jeltsch, A. Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29 (18), 3705-3727 (2001).
- Tanner, N. A., Loparo, J. J., van Oijen, A. M. Visualizing single-molecule DNA replication with fluorescence microscopy. Journal of Visualized Experiments: JoVE. (32), e1529(2009).
- Kulczyk, A. W., Tanner, N. A., Loparo, J. J., Richardson, C. C., van Oijen, A. M. Direct observation of enzymes replicating DNA using a single-molecule DNA stretching assay. Journal of Visualized Experiments: JoVE. (37), e1689(2010).
- Baldwin, G. S., Vipond, I. B., Halford, S. E. Rapid reaction analysis of the catalytic cycle of the EcoRV restriction endonuclease. Biochemistry. 34 (2), 705-714 (1995).
- Winkler, F. K., et al. The crystal-structure of Ecorv endonuclease and of Its complexes with cognate and non-cognate DNA fragments. EMBO Journal. 12 (5), 1781-1795 (1993).
- Sioss, J. A., Stoermer, R. L., Sha, M. Y., Keating, C. D. Silica-coated, Au/Ag striped nanowires for bioanalysis. Langmuir. 23 (22), 11334-11341 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon