Method Article
限制性核酸内切酶介导的 DNA 切割的单分子停留时间分析
摘要
利用量子点标记的DNA和全内反射荧光显微镜,我们可以研究使用未标记蛋白质时限制性核酸内切酶的反应机制。这种单分子技术允许对单个蛋白质-DNA相互作用进行大规模多路复用观察,并且可以合并数据以生成填充良好的停留时间分布。
摘要
这种基于全内反射荧光显微镜的新型检测方法有助于在一次实验中同时测量数百个单独的限制性核酸内切酶 (REase) 分子的催化循环长度。该测定不需要蛋白质标记,可以使用单个成像通道进行。此外,可以将多个单独实验的结果合并在一起,以生成填充良好的停留时间分布。对所得停留时间分布的分析可以通过揭示无法直接观察到的动力学步骤的存在来帮助阐明 DNA 切割机制。使用该测定法与经过充分研究的 REase EcoRV(一种二聚体 IIP 型限制性核酸内切酶)收集的示例数据,可切割回文序列 GAT↓ATC(其中 ↓ 是切割位点) - 与先前的研究一致。这些结果表明,在 EcoRV 在没有 DNA 的情况下结合 DNA 后,通过引入镁来启动 DNA 切割的途径中至少有三个步骤,每个步骤的平均速率为 0.17 s-1 。
引言
限制性核酸内切酶 (REase) 是影响 DNA 中序列特异性双链断裂的酶。1970 年代 REase 的发现导致了重组 DNA 技术的发展,这些酶现在是基因改造和操作不可或缺的实验室工具1。II 型 REase 是这类中使用最广泛的酶,因为它们在其识别序列内或附近的固定位置切割 DNA。然而,II 型 REase 之间存在很大差异,它们根据特定的酶特性分为几种亚型,而不是根据它们的进化关系进行分类。在每种亚型中,分类方案经常出现异常,许多酶属于多个亚型2。已经鉴定出数以千计的II型REase,其中数百种是市售的。
然而,尽管 II 型 REase 之间存在多样性,但很少有 REases 得到详细研究。根据理查德·罗伯茨爵士(Sir Richard Roberts)于1975年建立的限制性内切酶数据库REBASE的数据3,这些酶中只有不到20种的已发表的动力学数据可用。此外,虽然在遇到并与其识别序列结合之前,在单分子水平上直接观察到一些 REase 沿 DNA 扩散 4,5,6,7,但对其裂解反应动力学的单分子研究很少。现有的研究要么没有报告足够的统计数据来详细分析单次裂解事件发生时间的变化8,9,10,要么无法捕捉裂解时间的全部分布11。这种类型的分析可以揭示相对长寿命的动力学中间体的存在,并可能导致对REase介导的DNA切割机制的更好理解。
在单分子水平上,生化过程是随机的——该过程发生的单个实例的等待时间 τ是可变的。然而,可以预期 τ 的许多测量值都服从概率分布 p(τ),该分布表示正在发生的过程类型。例如,单步过程(例如从酶中释放产物分子)将遵循泊松统计,而 p(τ) 将采用负指数分布的形式:
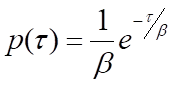
其中 β 是平均等待时间。请注意,该过程的速率 k 将等于 1/β,即平均等待时间的倒数。对于需要多个步骤的过程,p(τ) 将是每个单独步骤的单指数分布的卷积。β,在相同平均等待时间下对 N 个单指数衰减函数进行卷积的一般解是伽玛概率分布:

其中 Γ(N) 是 gamma 函数,它描述了 N-1 的阶乘到 N 的非整数值的插值。尽管当各个步骤的平均等待时间相似时,这种通用解决方案可以用作近似值,但必须理解的是,相对快速的步骤的存在将被等待时间明显更长的步骤所掩盖。换言之,N 的值表示步数12 的下限。在足够数量的等待时间测量的情况下,可以通过对事件进行分箱并将伽马分布拟合到生成的直方图或使用最大似然估计方法来估计参数 β 和 N。因此,这种类型的分析可以揭示动力学步骤的存在,这些步骤在集成测定中无法轻松解决,并且需要大量的观察才能准确估计参数12,13。
本文介绍了一种使用量子点标记 DNA 和全内反射荧光 (TIRF) 显微镜并行观察数百个 REase 介导的 DNA 切割事件的方法。该测定的设计使得汇集多个实验的结果成为可能,并可以创建包含数千个事件的停留时间分布。量子点的高光稳定性和亮度允许 10 Hz 的时间分辨率,而不会牺牲观察即使在实验开始后几分钟内发生的裂解事件的能力。良好的时间分辨率和宽动态范围,结合收集大型数据集的能力,可以准确表征停留时间分布,从而揭示 REase 裂解途径中存在的多个动力学步骤,这些 REase 的周转率在 1 min-1 范围内。在EcoRV的情况下,可以解决三个动力学步骤,所有这些步骤都已通过其他方法鉴定,证实了该测定对这些步骤的存在是敏感的。
包含目标识别序列的双链 DNA 底物是通过将生物素化的寡核苷酸退火到用单个共价连接的半导体纳米晶体量子点标记的互补链来产生的。这些基质被引入构建在玻璃盖玻片顶部的流道中,该流道具有共价附着在其表面的高分子量聚乙二醇 (PEG) 分子。DNA 底物通过生物素-链霉亲和素-生物素键被游离端具有生物素的 PEG 分子的一部分捕获。在TIRF显微镜中,随着玻璃-液体界面的距离呈指数衰减的倏逝波提供照明;穿透深度与所用光的波长成量级。在这些条件下,只有被捕获在功能化玻璃表面上的DNA分子拴在表面的量子点才会被激发。在溶液中游离的量子点不会受到限制在被照射区域内,因此不会发光。如果将量子点拴在表面的DNA被切割,则该量子点将可以自由地从表面扩散开来,并且将从荧光图像中消失。
尽管已知许多 II 型 REase 在不存在镁14 的情况下结合 DNA,但所有 REase 都需要镁来介导 DNA 切割15。这些 REase 可以在没有镁的情况下结合表面固定的 DNA。当含镁缓冲液流过预结合到DNA的REase通道时,裂解立即开始,如量子点的消失所示。通过预结合 REase 分子,然后通过引入镁来启动 DNA 切割来实现的同步,有助于测量实验中观察到的酶群中每个分子独立完成 DNA 切割的滞后时间。荧光素作为示踪染料包含在含镁缓冲液中,以指示镁进入视野。由于含镁缓冲液中不含酶,因此从含镁缓冲液到达到每个量子点消失的滞后时间表明,已经与DNA结合的REase切割DNA并从玻璃表面释放量子点所需的时间。量子点消失发生得很快,并导致强度轨迹急剧下降,从而清楚地表明了给定DNA分子被切割的时间。事件时间的确定是通过对强度轨迹的数学分析来完成的,一个典型的实验会产生数百个可识别的分裂事件。可以将多个实验的结果合并在一起,以提供足够的统计数据,从而可以通过非线性最小二乘法或最大似然法分析来估计参数 N 和 β。
研究方案
1. 一般信息
- 寡核苷酸设计
注:60 个碱基对 (bp) 长的 DNA 底物由一对互补寡核苷酸形成,在 100 mM NaCl 中的双链体熔解温度为 75 °C。- 订购一种寡核苷酸,该寡核苷酸由单个 5' 生物素修饰合成,另一种采用 5' 硫醇修饰(带有六碳间隔物)。将识别位点放置在双工区域的中心。
注:用于 EcoRV 的寡核苷酸序列如下所示(识别位点以粗体显示)。
5' 生物素 - AAA ACC GAC ATG TTG ATT TCC TGA AAC GGG ATA TCA TCA AAG CCA TGA ACA AAG CAG CCG - 3'
5' 硫醇 - CGG CTG CTT TGT TCA TGG CTGA TGA TAT CCC GTT TCA GGA AAT CAA CAT GTC GGT TTT - 3'
- 订购一种寡核苷酸,该寡核苷酸由单个 5' 生物素修饰合成,另一种采用 5' 硫醇修饰(带有六碳间隔物)。将识别位点放置在双工区域的中心。
- 所有步骤均使用电阻率为 18 MOhm 的超纯水。
- 保护所有含有量子点的溶液免受光照射,以防止光漂白。
- 使用压缩空气源完成此协议。
2. 量子点标记DNA底物材料的制备
注:除上述寡核苷酸外,其他材料参见 材料表 ,制备量子点标记的DNA底物所需的缓冲液见 表1 。
- 减少寡核苷酸上的5'硫醇基团,以与量子点偶联。
- 将每种硫醇化的寡核苷酸重悬于水中,浓度为100μM。
- 将 650 μL 水移入每个寡核苷酸样品的体积排阻离心柱中,涡旋 ~15 秒。将色谱柱放置 30 分钟。
- 每次使用前立即制备新鲜的 100 mM 二硫苏糖醇 (DTT) 溶液,因为 DTT 在溶液中会迅速降解。小心地打开一个装有 7.7 mg DTT 的小瓶,并将 500 μL 磷酸钠缓冲液吸取到小瓶中;涡旋充分混合。
- 对于每种重悬的寡核苷酸,准备一个新管,并向管中加入 50 μL 寡核苷酸和 50 μL DTT 溶液。上下移液混合。在室温下孵育 30 分钟以减少寡核苷酸 5' 端氧化硫醇基团之间的二硫键。
- 取下离心柱盖,将每根柱放入收集管中。将离心柱以 750 × g 离心 2 分钟,并丢弃洗脱液。
- 将离心柱转移到新鲜的离心管中。将整个体积 (100 μL) 的 DNA/DTT 混合物样品轻轻移液到每个制备的柱上。以750 ×g 离心2分钟,并测量洗脱液在260nm处的吸光度,以确认浓度约为40μM。
- 将任何不会立即使用的样品储存在-20°C,以防止硫醇基团的氧化和二硫键的形成。
- DNA与量子点的偶联
- 将 50 μL 量子点原液的等分试样移液到透析装置中,用于制备每种构建体,确保不要用移液器吸头接触膜。在~100rpm下搅拌15分钟,对体积至少为样品体积1000倍的 N-环己基-2-氨基乙磺酸(CHES)缓冲液进行透析。
- 每次使用前立即在 CHES 缓冲液中制备新鲜的 6 mM 磺基琥珀酰亚胺基-4-(N-马来酰亚胺甲基)环己烷-1-羧酸酯 (sulfo-SMCC) 溶液。小心地打开一个装有 2 mg sulfo-SMCC 的小瓶,并将 800 μL CHES 缓冲液移入小瓶中。涡旋使充分混合。
- 使用移液器,小心地从透析装置中取出悬浮的量子点,然后转移到含有等体积的磺基-SMCC溶液的新鲜管中;上下移液混合。在室温下以1000rpm振荡孵育1小时,以使sulfo-SMCC与量子点上的伯胺反应。
- 使用移液器小心地将每个样品转移到新鲜的透析装置中。为了去除多余的sulfo-SMCC,在搅拌下对至少为透析装置中所含体积1000倍的CHES缓冲液进行透析15分钟。交换缓冲液 2 次,并用新鲜的 CHES 缓冲液进行透析 3 次,每次缓冲液交换后 15 分钟进行透析。
- 为了交换缓冲液以准备第二次反应,将透析装置转移到含有磷酸盐缓冲盐水 (PBS) 体积的烧杯中,该体积至少是装置中所含体积的 1000 倍。搅拌透析 15 分钟,交换缓冲液 2 次,并用新鲜 PBS 进行透析 3 次,每次缓冲液交换后透析进行 15 分钟。
- 使用移液器,小心地从透析装置中回收含有量子点的溶液,并将每个样品转移到含有等摩尔量的硫醇化寡核苷酸的新鲜管中,该管在PBS中稀释。将等体积的PBS和还原和纯化的寡核苷酸样品体积的1/10(浓度约为40μM浓度)混合,因为此时的量子点浓度为~4μM。 在室温下孵育2小时,以1000rpm振荡。
- 向每个样品中加入牛血清白蛋白(BSA,10 mg/mL),以获得 0.5 mg/mL 的最终 BSA 浓度。使用移液器小心地将每个样品转移到新鲜的透析装置中,并用至少为透析装置所含体积 1000 倍的储存缓冲液进行透析。交换缓冲液 2 次,并用新鲜储存缓冲液进行透析 3 次,每次交换缓冲液后允许透析进行 15 分钟。
- 使用移液器,小心地回收每个样品,将其放入新管中,并储存在4°C。
注意:不要在 -20 °C 下储存,因为量子点会因冻结而损坏。
- 量子点标记寡核苷酸退火为生物素化寡核苷酸
- 将每种生物素化的寡核苷酸重悬于浓度为100μM的储存缓冲液中。 将未使用的样品储存在-20°C。
- 将量子点标记的寡核苷酸样品与摩尔过量的生物素化寡核苷酸混合 10 倍。由于量子点标记样品中 DNA 的估计浓度为 2 μM,因此每 10 μL 该样品添加 0.2 μL 生物素化寡核苷酸。
- 将加热块中的混合物加热至 75 °C(互补区域的熔化温度)。在该温度下保持 5 分钟。关闭加热块,让其慢慢冷却。将完成的构建体储存在4°C;不要冻结。
3. 盖玻片的表面功能化
注意:此过程之前已在其他JoVE视频协议16,17中描述过。该协议描述了该程序的改编版本,并进行了微小的更改以适应较小的载玻片。有关盖玻片表面功能化所需的其他材料,请参阅材料表。
- 将 5 个盖玻片放入每个盖玻片架中。确保在每对盖玻片之间跳过一个空间,以免它们相互粘连。将盖玻片的支架放入带盖的罐子中,向罐子中加入乙醇,直到盖玻片被盖住,然后拧紧顶部。将整个罐子放入浴超声仪的水中,不要将其完全浸入水中,并超声处理 30 分钟。
- 用超纯水填充干净的烧杯或罐子。使用金属镊子从乙醇中取出支架中的盖玻片,然后将它们浸入水中冲洗。然后,将盖玻片和支架转移到装有 1 M 氢氧化钾 (KOH) 溶液的罐子中。拧紧顶部,将罐子放入超声仪浴中,并超声处理 30 分钟。
注意:处理 KOH 溶液时要小心,因为它具有腐蚀性和刺激性。 - 在乙醇和KOH溶液中重复超声处理,如上所述在每个步骤之间用水冲洗盖玻片。
- 将支架中的盖玻片转移到装满纯丙酮的干净罐子中。如上所述对罐子进行超声处理 30 分钟,完成清洁。此步骤后请勿用水冲洗。
- 使用金属镊子将支架中的盖玻片转移到含有 80 mL 新鲜丙酮和微量搅拌棒的干净 100 mL 烧杯中。将烧杯放在设置为至少 1,000 rpm 的磁力搅拌板上。在剧烈搅拌丙酮的同时,将 1.6 mL 3-氨丙基三乙氧基硅烷 (APTES) 移入烧杯中,制成 2% v/v 溶液。
注意:移液 APTES 时要小心,因为它具有腐蚀性。 - 让盖玻片在溶液中孵育 2 分钟,然后使用金属镊子将盖玻片在其支架中转移到水烧杯中以淬灭反应。再冲洗两次盖玻片,更换烧杯中的水。
- 在120°C的烤箱中固化硅烷化玻璃75分钟。如果不立即继续下一步,请将盖玻片存放在真空下最多几天。
- 将 N-羟基琥珀酰亚胺(NHS)酯衍生化聚乙二醇(PEG)溶解在100mM碳酸氢钠(pH 8.2)中。使用 10:1 比例的甲氧基封端 PEG 与生物素封端 PEG,含 ~100 mg/mL 甲氧基封端 PEG。
- 将 100 μL PEG 溶液移液到一半的干燥硅烷化盖玻片上,并用第二个盖玻片覆盖每个盖玻片。使用放置在每个角落的小块封口膜作为垫片,以防止盖玻片相互粘附。
- 将盖玻片在潮湿环境中孵育3.5小时。将盖玻片三明治分开。使用喷瓶用大量水清洗每个盖玻片,并用压缩空气干燥。
- 将功能化的盖玻片储存在真空下。确保将PEG处理的一面朝上,因为另一侧不会捕获DNA底物。
4. 微流控器件构建
注:有关构建微流体装置所需的其他材料,请参阅 材料表 。
- 使用装有锥形金刚石尖轮钻头的手持式旋转多功能工具,在石英滑轨的相对角上钻两个孔,用作入口和出口。务必将滑轨固定到位,润滑钻头,并在钻孔时不断用水滑动。每次实验后,通过将用过的设备浸泡在丙酮中以溶解粘合剂和环氧树脂来回收准备好的石英载玻片以供重复使用;丢弃剩余的组件。
注意:钻孔可以手动进行,但使用多功能工具的压机式支架可以使该过程更容易。 - 将 25 μL 链霉亲和素溶液与 80 μL PBS 和 20 μL 封闭缓冲液混合。用该混合物涂覆PEG处理的盖玻片,并在潮湿环境中孵育30分钟。
- 在盖玻片孵育期间,准备成像垫片以创建流道。切割一块 1 英寸见方的成像垫片材料,然后标记一个 2 毫米宽的通道,对齐于石英滑轨中钻孔的末端(图 2)。用手术刀从间隔材料中切出通道。
- 从图像垫片上撕下背衬的一侧,然后小心地将其放在石英载玻片上,注意不要覆盖入口和出口孔。务必用丙酮彻底清洁石英载玻片,以去除先前实验中残留的粘合剂。
- 用水清洗盖玻片,并用压缩空气干燥。从成像垫片上取下背衬的另一侧,将其夹在石英载玻片和功能化的链霉亲和素包被的盖玻片之间,使用塑料镊子将组件压在一起并去除粘合剂上的气泡。
- 将一根 30 厘米长的聚乙烯管插入石英滑轨的每个孔中。确保将管道的末端切成一定角度,以确保溶液自由流动。使用管架或其他支撑物将管子固定到位,并用环氧树脂将管子密封到位并密封组装设备的边缘。
注意:将设备构建在一块石蜡膜上效果很好,当环氧树脂凝固时,该石蜡膜可以很容易地从底部剥落。 - 环氧树脂凝固后,将空注射器上的钝针插入设备的出口管中,然后将入口管的末端浸入装有阻塞缓冲液的容器中。向后拉注射器柱塞,用阻塞缓冲液填充设备。使用前让设备孵育至少 30 分钟。
5. 量子点标记DNA底物的表面拴系
注:除了上述微流控装置、DNA 底物和封闭缓冲液外,其他材料参见 材料表 ,量子点标记 DNA 底物表面拴系所需的缓冲液见 表 1 。
- 用胶带将微流控装置固定在显微镜载物台板上,使物镜与设备底部接触,并放置物镜,使视场在微流控通道内。
- 将出口管连接到注射泵后,拉回注射器柱塞,用新鲜的阻塞缓冲液冲洗微流体通道。检查以确保管道或通道中没有气泡滞留。
注意: 从现在开始,请确保入口管处于液体中,以免气泡进入设备。 - 将 1 μL 制备的 DNA 底物稀释到 1 mL 封闭缓冲液中。将入口管放入稀释的 DNA 底物中,以 200 μL/min 的速率流过通道 800 μL 底物溶液。流动停止后,让 DNA 溶液在通道中不受干扰地孵育 15 分钟。
- 以 200 μL/min 的速率流过通道,将至少 800 μL 封闭缓冲液流过通道,以将未结合的 DNA 从通道中冲洗出来。
- 调整激光功率、显微镜焦距和 TIRF 角度,使表面拴系的量子点清晰可见。
注意:虽然量子点不会快速漂白,但最好将功率保持在尽可能低的水平,以尽量减少闪烁。
6. REase介导的DNA切割
注:有关材料,请参阅 材料表 ,有关REase介导的DNA切割所需的缓冲液,请参见 表1 。
- 确保摄像机冷却到最佳工作温度,并设置为曝光时间为 0.10 秒的高速流媒体。准备好收集 ~4 分钟的数据。
- 用 800 μL 不含镁的实验缓冲液以 200 μL/min 的流速冲洗微流体装置。
- 将 REase 加入不含镁的实验缓冲液的等分试样 (1 mL) 中,并通过移液轻轻混合。使用 4 μL 100,000 单位/mL 原液,相当于 400 单位/mL EcoRV。将 800 μL 稀释的酶以 200 μL/min 的流速流过通道。
- 通过以 200 μL/min 的流速流出含有镁和荧光素的实验缓冲液开始实验。启动注射泵后立即开始捕获数据。流出 800 μL 缓冲液后,停止数据采集。
7. 数据分析
注意:请参阅用于此协议的数据分析软件 的材料表 ,如果使用不同的分析平台,请进行调整。
- 零时间点确定
- 使用 imtophat 函数从实验电影的每一帧中减去背景荧光,imtophat 函数是 Image Processing Toolbox 中内置的形态学顶帽滤波函数。选择半 径为 3 个像素的磁盘 作为结构元素。通过从原始图像中减去滤后的图像来获得背景强度。
注意:滤镜仅保留比背景亮且比结构元素小的图像特征。 - 平均每个电影帧上减去的背景。使用此值的轨迹来确定实验的零时间点(图3A)。通过查找第一帧来确定开始时间,在该帧时,增加率超过死体积流动期间变化率标准差的 3 倍。
注:每个电影帧减去背景平均值的变化率急剧增加,表明含有示踪染料的最终实验缓冲液进入流通池的时间(图3B)。
- 使用 imtophat 函数从实验电影的每一帧中减去背景荧光,imtophat 函数是 Image Processing Toolbox 中内置的形态学顶帽滤波函数。选择半 径为 3 个像素的磁盘 作为结构元素。通过从原始图像中减去滤后的图像来获得背景强度。
- 量子点强度轨迹计算与分析
- 计算在示踪染料到达之前记录的一组背景校正电影帧的最大强度投影。使用峰值查找函数(如 pkfnd)确定此投影图像中量子点的位置,精度为一像素。
- 通过计算峰值查找函数返回的位置周围每侧三个像素的正方形区域的每个电影帧的平均强度,为单个量子点生成强度轨迹。
注意:上述背景校正可去除示踪染料引入的伪影。由此产生的强度轨迹应该相对平坦,但仍包括自然发生的强度波动(图4)。 - 通过对强度轨迹的统计分析来识别量子点消失事件。对于每个量子点强度轨迹,根据轨迹中观察到的噪声,计算出高于最小值的阈值强度的阈值强度,该阈值是该轨迹的最大值和最小值之差的适当分数。
注意:量子点的强度波动通常远大于背景波动。通过比较这些值,可以确定适当的值:所选阈值必须高于最高背景值,但理想情况下应低于量子点荧光的最低值。对于提供的数据,所用阈值的计算值高于每条轨迹的最小值加上该轨迹的最大值和最小值之差的三分之一。量子点在其位置观察到的强度高于此阈值的最后一个时间点可以被视为消失。 - 确认推定的消失事件。仅当观察到的强度高于消失时间之前超过一半的电影帧的轨迹最小强度值和最大强度值之间的中点,并且强度轨迹的标准偏差在事件发生后减小时,才在最终分析中包括事件。
注意:可以使用其他统计测试来确保仅记录有效的消失事件。
结果
流通池直接连接到配备激光照明的倒置显微镜上的高数值孔径油浸式 60 倍放大物镜,用于穿透物镜 TIRF 成像(图 5A)。在引入DNA底物并洗去多余的DNA和量子点后,视野中通常有数千个单独的量子点(图5B)。这些量子点稳定地附着在玻璃表面,并且在实验的时间尺度上它们不会经历明显的变暗或显着的漂白。然而,如果含有镁和适当REase的缓冲液流过流道,在典型的四分钟观察期结束时,至少30%的实验开始时存在的量子点将从视野中消失。确认镁的需求,当REase在没有镁的情况过通道时,在观察期结束时仍可以看到实验开始时存在的至少95%的量子点(图5C)。然而,当含镁的缓冲液在允许REase在没有镁的情况下结合表面结合的DNA后立即流动,到观察期结束时,多达一半的量子点将消失(图5D),类似于当REase和镁一起流过通道时观察到的情况。事件的确切产量将取决于酶在所用条件下的效率。当含镁缓冲液在未事先将适当的REase引入流道的情况动时,在观察期间,只有不到5%的量子点消失,并且事件的直方图没有可辨别的峰值。这一结果表明,预结合的REase分子介导了将量子点拴在玻璃表面的DNA裂解,而这种DNA裂解是这些实验中观察到的绝大多数量子点消失事件的原因。
量子点消失发生得很快,并导致强度轨迹急剧下降,从而清楚地表明了给定DNA分子被切割的时间(图5E)。事件时间的确定是通过对强度轨迹的数学分析来完成的。在所使用的图像条件下,单个量子点产生的强度轨迹的方差明显高于背景,因此,当强度轨迹的方差减小到与观察到的强度下降后背景方差相当的水平时,推定事件得到了确认。此外,在推定的消失事件发生之前包括大量闪烁的轨迹被排除在最终分析之外。然而,一个典型的实验会产生数百个满足这些条件的事件,并且可以将多个实验的结果合并以提供足够的统计数据,从而允许通过非线性最小二乘曲线拟合或最大似然参数估计来估计参数 N 和 β 。
所呈现的代表性数据是通过使用经过充分研究的 IIP 型 REase EcoRV 进行该实验收集的(视频 1)。60 bp 长的双链体 DNA 底物由一条链的 5' 端的生物素分子和共价连接到另一链的 5' 端的量子点构成。DNA 底物包含识别序列的单个拷贝 GAT↓ATC,该序列在中间被 EcoRV 切割,如向下箭头 (↓) 所示。在没有镁的情况下,将 EcoRV 预结合到 DNA 底物上,然后流入含镁的缓冲液以启动 DNA 切割。代表性数据包括五个独立实验的汇总结果,总共产生了 3451 个观察到的切割事件。在排除在零时间点之前或突出活动峰值之外发生的事件后,还剩下 2987 个事件,这些事件足以用一秒条柱填充直方图。采用非线性最小二乘曲线拟合和最大似然参数估计方法从数据中提取 N 值和 β 值。两种参数估计方法一致(图6A),非线性最小二乘拟合为 N = 3.52(95%置信区间:3.32-3.71)和 β = 5.78 s(95%置信区间:5.41-6.15 s),最大似然估计为 N = 3.41(95%置信区间:3.25-3.58)和β = 6.06 s(95%置信区间:5.75-6.39 s)。
鉴于对 EcoRV 机制的了解,至少三个动力学步骤的估计是合理的。已知这种酶在没有镁14 的情况下会非特异性地结合 DNA。在停止流动条件下,对 EcoRV 中色氨酸残基特性荧光变化的本体溶液相观察表明,在没有镁的情况下与 DNA 结合的 EcoRV 必须在镁进入活性位点之前发生构象变化18。这一观察结果得到了晶体结构数据19的证实。色氨酸荧光研究的结果还表明,DNA 切割和产物释放是以相似的速率发生的独立步骤18。因此,本实验中EcoRV介导的DNA切割的合理反应机理为:
ES → ES* → EP → E+P
在这种机制中,当 EcoRV 在没有镁的情况下预结合到 DNA 时,会形成 ES,即初始酶-底物复合物。当镁进入流通池时,酶-底物复合物发生构象变化,成为 ES*,即活化的酶-底物复合物。然后,这种活化的复合物切割DNA,但不会立即释放产物分子,成为酶-产物复合物(EP)。最后,在最后一步中释放产品 P。这种机制需要三个步骤,这与所提供的数据是一致的。每个步骤的平均等待时间的估计值为 ~6 秒,相当于每个步骤的比率为 0.17 s-1 。该计算结果与之前对这些过程的速率估计值基本一致——DNA 切割和产物释放的估计值约为 0.3-0.4 s-1 ,构象重排的估计值约为 ~0.5 s-1 。

图 1:标记的 DNA 和酶反应示意图。 量子点标记的 DNA 分子通过生物素-链霉亲和素-生物素键拴在功能化的玻璃表面,并使用 TIRF 显微镜观察。DNA 分子包含目标 REase 的识别位点。当DNA分子被REase切割时,量子点可以自由地从表面扩散到照明区之外。缩写:REase=限制性核酸内切酶;PEG = 聚乙二醇;TIRF = 全内反射荧光。 请点击这里查看此图的较大版本.

图 2:微流控流通池装置。 (A) 分解图 显示了用于创建该设备的三层:底部的功能化玻璃盖玻片,顶部带有入口和出口孔的石英载玻片,以及中间夹有通道的双面胶成像垫片。(B) 一个完整的装置,其聚乙烯管密封到位,边缘涂有环氧树脂。 请点击这里查看此图的较大版本.
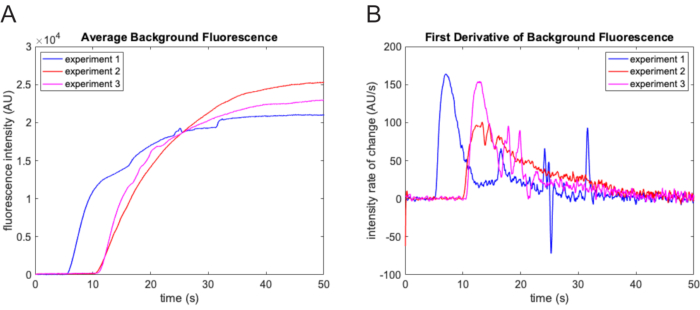
图 3:零时间点确定。 (A) 使用形态学顶帽滤波功能在电影的每一帧上确定的平均背景强度。当含有荧光素的实验缓冲液进入视野时,背景强度显着增加。此处显示了三个不同实验的结果。不同实验的滞后时间存在很大差异。(B)背景荧光强度轨迹变化率的急剧增加有利于准确确定零时间点。 请点击这里查看此图的较大版本.

图 4:背景校正。 由于荧光素示踪染料表明镁的到来,背景强度的增加可以在单个量子点的原始荧光强度轨迹(灰色轨迹)中看到。在使用形态顶帽过滤功能减去背景后,示踪染料引入的伪影已从轨迹(黑色轨迹)中去除。 请点击这里查看此图的较大版本.

图 5:用于并行观察 DNA 切割事件的单分子 TIRF 实验。 (A) 构建在功能化玻璃表面上的流通池,设计用于捕获量子点标记的 DNA,直接耦合到用于 TIRF 成像的高数值孔径 60 倍油浸式显微镜物镜。(B) 实验开始时的代表性图像。量子点标记的DNA已被加载到流通池中,多余的未结合量子点已被洗去。单个DNA拴系的量子点可以相互分辨。(C) 在没有镁的情况下,将含有能够切割 DNA 系绳的 REase 的缓冲液流过流道 4 分钟后,相同的视野。DNA拴系的量子点没有显着损失。(D) 实验结束时的相同视野。在无镁缓冲液中流动REase后,立即将含镁缓冲液流过流道。该图像是在缓冲液流动大约四分钟后获得的。预结合的 REase 已经切割了许多 DNA 系绳,从表面释放出量子点。为了便于查看,每张图像中仅显示客观视野的中心象限。对十个电影帧(相当于一秒的观察时间)进行平均,以减少量子点闪烁的影响。所有三张图像的亮度和对比度设置都相同。(E) 在实验开始时存在量子点的图像位置的代表性荧光强度轨迹。从与未从表面释放的量子点(灰色)相对应的图像位置获得的轨迹可能会短暂下降到较低强度水平,但它们在高强度水平开始和结束。从图像位置获得的轨迹,对应于从表面释放的量子点(黑色),显示强度迅速降低到相对于实验时间分辨率(10 Hz)而言是瞬时的低背景水平。释放的量子点在四分钟的观察期内不会再次出现。缩写:REase=限制性核酸内切酶;TIRF = 全内反射荧光。 请点击这里查看此图的较大版本.

图 6:EcoRV 介导的 DNA 切割的停留时间分布分析。 (A) 在一组 5 个 EcoRV 合并实验中,在主活动峰内发生的 2987 个切割事件的直方图和预测包络图。两条预测曲线几乎相同,拟合残差(直方图下方)并不表示非线性最小二乘拟合中的系统误差。(B) 在零时间点之后发生的整组 3393 个裂解事件的直方图。假设 gamma 概率分布,通过参数的最大似然估计(不间断线、MLE 曲线)预测的曲线无法包围直方图。通过伽玛概率分布公式的非线性最小二乘拟合预测到条柱高度的曲线(虚线,NLS 曲线)是更好的,但拟合的残差(直方图下方)揭示了系统误差。缩写:MLE:最大似然估计;NLS = 非线性最小二乘法。 请点击这里查看此图的较大版本.
| 缓冲区名称 | 元件 | 浓度 | pH 值为 25 °C |
| 磷酸钠缓冲液 | 磷酸钠 | 100毫米 | 8.3 |
| CHES缓冲液 | N-环己基-2-氨基乙磺酸(CHES) | 10毫米 | 9.0 |
| 美国公共广播公司(PBS) | 磷酸钠 | 3毫米 | 7.2 - 7.6 |
| 氯化钠 | 150毫米 | ||
| 磷酸钾 | 1.05毫米 | ||
| 存储缓冲区 | 氯化钠 | 100毫米 | 8.0 |
| Tris-HCl(三盐酸) | 50毫米 | ||
| 牛血清白蛋白 (BSA) | 0.5毫克/毫升 | ||
| 碳酸氢钠 | 碳酸氢钠 | 100毫米 | 8.2 |
| 阻塞缓冲区 | Tris-HCl(三盐酸) | 20毫米 | 7.5 |
| 乙二胺四乙酸(EDTA) | 2毫米 | ||
| 氯化钠 | 50毫米 | ||
| 吐温-20 | 0.005% (伏/伏) | ||
| 牛血清白蛋白 (BSA) | 0.2毫克/毫升 | ||
| 不含镁的实验缓冲液 | 氯化钠 | 100毫米 | 7.9 |
| Tris-HCl(三盐酸) | 50毫米 | ||
| 二硫苏糖醇 (DTT) | 1毫米 | ||
| 含镁的实验缓冲液 | 氯化钠 | 100毫米 | 7.9 |
| Tris-HCl(三盐酸) | 50毫米 | ||
| 氯化镁 | 10毫米 | ||
| 二硫苏糖醇 (DTT) | 1毫米 | ||
| 荧光 素 | 根据成像条件进行调整 | ||
| DNase缓冲液 | 氯化镁 | 2.5毫米 | 7.6 |
| Tris-HCl(三盐酸) | 10毫米 | ||
| 氯化钙 | 0.1毫米 |
表 1:缓冲区表。
视频 1:单分子电影示例。请按此下载此影片。
讨论
该测定的 DNA 底物使用使用 sulfo-SMCC 的两步反应方案用量子点标记。这种双功能交联剂由一个可以与伯胺反应的 NHS 酯部分和一个可以与巯基反应的马来酰亚胺部分组成20。用于制备底物的硫醇化寡核苷酸以氧化形式运输。如前所述,在进行偶联程序之前减少和纯化它们非常重要,否则偶联反应的效率将降低。在使用前立即制备新鲜的DTT和sulfo-SMCC溶液至关重要。这些分子在水溶液中迅速水解。将溶液移入和移出透析设备时要小心 - 如果移液器吸头接触膜,将导致破裂,样品将丢失。理想的做法是不间断地完成第 2 部分中的所有步骤,以避免样品损失。
用于生成量子点标记寡核苷酸的偶联反应效率低下。在将量子点标记的寡核苷酸与生物素化的寡核苷酸结合并退火后,制备的底物样品中将存在过量的未标记 DNA。尽管这些物质可以与功能化表面结合,但在推荐的成像条件下它们不可见,并且对实验的读数没有贡献。为了确认已完成构造的裂解导致量子点从表面释放,请按照协议第 5 节中的描述执行表面束缚,并对表面区域进行成像以确定存在多少量子点。然后,在 DNase 缓冲液(2.5 mM MgCl2;10 mM Tris-HCl;0.1 mM CaCl2,pH 7.6)中流出 800 μL 10 U/mL DNase I 等分试样,通过微流体通道。让 DNase I 在通道中孵育 15 分钟,用 800 μL DNase 缓冲液(不含酶)洗涤,并对相同的感兴趣区域进行成像。如果结构和表面准备得当,大约 90% 的量子点应该从表面释放出来。
为了共价连接甲氧基封端和生物素封端的 PEG 分子的混合物,必须对玻璃表面进行处理以捕获表面上的量子点标记的 DNA 底物以进行成像。严格执行所有清洁步骤至关重要。需要多个清洁步骤来去除表面杂质并暴露玻璃表面的羟基,这些羟基将与硅烷上的烷氧基反应形成共价键。虽然在使用乙醇和 KOH 溶液的清洁步骤之间用超纯水冲洗载玻片很重要,但在用丙酮进行最终清洁后不应使用水。在硅烷化步骤中存在过量的水将促进溶液中硅烷的多聚化,而牺牲了表面结合。此外,不得提前准备硅烷溶液。为了延长其使用寿命,硅烷原料应储存在干燥器柜中。
单分子裂解测定设计为在微流控装置中进行(图2)。在每次实验之前制造一个新设备,重复使用形成设备顶部的钻孔石英滑轨。该程序在前面已经描述过16,17,其变体广泛用于 TIRF 显微镜检查。本文中描述的程序已经过调整以适应 1 英寸的方形载玻片。重要的是要快速构建设备,以便功能化表面在暴露于空气时不会降解。环氧树脂是荧光的,所以要注意避免用它覆盖通道。通过将用过的设备浸泡在丙酮中并拆卸,可以将钻孔的石英滑块回收以供重复使用。
该测定设计为在倒置显微镜上进行,该显微镜具有穿透物镜全内反射照明功能,并配有电子倍增电荷耦合器件 (EMCCD) 相机用于数据收集。使用 60 倍高数值孔径物镜和 488 nm 激光激发源,并使用包括适用于激光的单带通发射滤光片和单边二向色性的滤光片立方体,以及允许量子点的荧光(发射峰在 655 nm 处)和至少部分荧光素荧光(发射峰在 512 nm 处)通过的发射滤光片。例如,此处提供的数据是使用装有 482/18 nm 单带通激发滤光片、488 nm 单边二向色滤光片和 675/67 nm 单带通发射滤光片的滤光片立方体收集的。如本协议所述,可以通过在单个光谱通道中成像来进行测定。如果需要,也可以通过将荧光图像分成两个光谱通道来进行测定,一个用于量子点,另一个用于荧光素发射。装有 640 nm 单边二向色镜的图像分割器适用于此目的。但是,不需要多通道成像。
在开始实验之前,从通道中洗掉任何未束缚的 DNA 底物非常重要。在最终缓冲液中加入浓度为 50 μM 的荧光素,以指示镁进入视野。根据显微镜设置,可能需要调整荧光素的浓度,使其发射不会遮挡量子点。确保在开始含镁缓冲液的流动后立即开始采集数据。在含镁缓冲液到达之前的滞后时间内记录数据,以便计算背景强度变化率的平均值和标准差,以便于准确确定零时间点。请注意,最准确的零时间点是背景荧光变化率超过此测量中自然波动的第一帧。在此点之后,背景荧光强度继续增加,因为荧光素扩散到通道中层流建立的边界层。镁比荧光素小得多,扩散速度更快,其浓度可以预期更快地达到平衡。
对于所呈现的示例数据,在零时间点之后发生的所有 3393 个事件的直方图在实验的前 60 秒内显示了一个突出的峰值。许多对应于较长滞后时间的箱子不包含任何事件,而那些包含事件的箱子只包含几个事件,类似于在没有活性 REase 时观察到的情况。伽马概率分布与所有分箱数据的非线性最小二乘曲线拟合导致参数估计值为 N = 3.06,95% 置信区间为 2.94-3.18,β = 7.04 秒,95% 置信区间为 6.72-7.36 秒。然而,参数的最大似然估计值为N = 1.30(95%置信区间:1.24-1.35)和β = 26.7 s(95%置信区间:25.3-28.1 s),通过目视检查,最大似然参数估计值预测的曲线与数据不匹配(图6B)。此外,尽管通过非线性最小二乘拟合估计的参数预测的曲线相对很好地遵循观测事件频率的直方图形状,但在大约60 s的时间点内,残差(观测到的事件频率和预测的事件频率之间的差异)始终变为正值(图6B)。
由于后期时间点的事件频率与在没有活性酶的情况下观察到的量子点消失的频率相当,因此这些后期事件中的大多数可能是量子点损失的低背景水平的一部分,而量子点损失不是由REase介导的DNA切割引起的。因此,将这些垃圾桶排除在安装之外是合理的。当排除每个实验的零时间点后超过60秒发生的所有事件时,两种参数估计方法一致(图6A)。因此,要获得最佳参数估计值,应排除活动峰值之外的事件,并确认最大似然参数估计值和非线性最小二乘曲线拟合结果已收敛。
本文概述的实验方法允许在单分子水平上实时观察 REase 介导的 DNA 切割。在每个单独的实验中可以观察到数百个事件,并且可以将几个实验的结果合并在一起,以获得通过将数据拟合到 gamma 概率分布来表征等待时间分布所需的大量观测值。虽然该测定只能与在没有镁或其他辅因子的情况下结合但不切割 DNA 的 REase 一起使用,但许多 REase 属于这一类。重要的是要认识到,该测定不会揭示所有中间步骤的确切细节。明显快于速率限制步长的步长不会影响停留时间分布,也不会计入从数据中提取的 N 值12。同样,可以从数据中提取的β值是影响停留时间分布的步骤的等待时间的近似平均值。每个步骤的实际等待时间可能与β12 的值不同。然而,本文中描述的单分子裂解测定可以提供证据,证明反应路径中存在多个中间步骤,即使不能直接观察反应中间体本身。
披露声明
作者没有相互竞争的经济利益或其他利益冲突
致谢
这项工作得到了美国国家普通医学科学研究所对CME的K12GM074869号奖的支持。内容完全由作者负责,并不一定代表美国国家普通医学科学研究所或美国国立卫生研究院的官方观点。
材料
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltriethoxysilane (APTS) | Sigma Aldrich | 440140-100ML | Store in dessicator box |
| 5 Minute Epoxy | Devcon | 20845 | use for sealing the microfluidic device |
| acetone | Pharmco | 329000ACS | use for cleaning coverslips |
| bath sonicator | Fisher Scientific | CPXH Model 2800 | catalog number 15-337-410 |
| Beaker, glass, 100 mL | |||
| Benchtop centrifuge | |||
| Biotin-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | BIO-SVA-5K | Succinimidyl valerate has a longer half-life than succinimidyl carbonate |
| biotinylated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Biotin modification and HPLC purification |
| Bovine Serum Albumin (BSA) | VWR | 0903-5G | prepare a 10 mg/mL solution (aq) and heat to 95 °C before using |
| Centri-Spin-10 Size Exclusion Spin Columns | Princeton Separations | CS-100 or CS-101 | used to purify thiolated oligonucleotides after reducing the disulfide bond |
| Centrifuge tubes 1.5. mL | |||
| Coverslips, 1-inch square glass | |||
| coverslip holders | |||
| diamond point wheel | Dremel | 7134 | use for drilling holes in quartz flow cell topper |
| dithiothreitol (DTT) | Thermo Scientific | A39255 | No-Weigh Format, 7.7 mg/vial |
| drill press rotary tool workstation stand | Dremel | 220-01 | facilitates quartz drilling |
| EcoRV (REase used to generate example data) | New England Biolabs | R0195T or R0195M | Use 100,000 units/mL stock to avoid adding excess glycerol Check REBASE for suppliers of other REases |
| ethanol | various | CAS 64-17-5 | denatured or 95% are acceptable, use for cleaning coverslips |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | EDS | BioUltra, anhydrous, store in dessicator box |
| Flea Micro Spinbar | Fisherbrand | 14-513-65 | 3 mm x 10 mm size to fit beneath coverslip rack |
| fluorescein | Acros Organics | 17324 | use to make experimental buffers |
| gravity convection oven | Binder | 9010-0131 | |
| handheld rotary multitool | Dremel | 8220 | use for drilling holes in quartz flow cell topper |
| ImagEM X2 EM-CCD Camera | Hamamatsu | C9100-23B | air cooling is adequate for this experiment, use HCImage software or similar to control |
| Imaging spacer, double-sided, adhesive | |||
| Jar, glass with screw cap, (approximately 50 mm diameter by 50 mm high) | |||
| magnesium chloride hexahydrate | Fisher Bioreagents | BP214-500 | use to make experimental buffer with magnesium |
| MATLAB software | Data analysis | ||
| metal tweezers | Fisher Brand | 16-100-110 | |
| methoxy-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | M-SVA-5K | Both PEGs should have the same NHS ester so that the rate of reaction is consistent |
| microcentrifuge | Eppendorf | 5424 | |
| multiposition magnetic stirrer | VWR | 12621-022 | |
| N-cyclohexyl-2-aminoethanesulfonic acid (CHES) | Acros Organics | AC20818 | CAS 103-47-9, use to make CHES buffer |
| orbital shaker and heater for microcentrifuge tubes | Q Instruments | 1808-0506 | with 1808-1061 adaptor for 24 x 2.0 mL or 15 x 0.5 mL tubes |
| Parafilm | |||
| PE60 Polyethylene tubing (inner diameter 0.76 mm, outer diameter 1.22 mm) | Intramedic | 6258917 | 22 G blunt needles are a good fit for this tubing size |
| Phosphate-Buffered Saline (PBS) 10x | Sigma Aldrich | P7059 | Use at 1x strength |
| potassium hydroxide | VWR Chemicals BDH | BDH9262 | use a 1 M solution to clean coverslips |
| Qdot 655 ITK Amino (PEG) Quantum Dots | Invitrogen | Q21521MP | |
| Quartz Slide, 1 inch square, 1 mm thick | Electron Microscopy Sciences | 72250-10 | holes must be drilled in the corners for inlet and outlet tubing insertion |
| reinforced plastic tweezers | Rubis | K35a | use for handling coverslips and building microfluidic device |
| SecureSeal Adhesive Sheets | Grace Biolabs | SA-S-1L | cut to form spacer for microfluidic device |
| Single channel syringe pump for microfluidics | New Era Pump Systems | NE-1002X-US | fitted with a 50 mL syringe and a 22 G blunt needle |
| Slide-a-Lyzer MINI Dialysis Devices, 10 kDa MWCO, 0.1 mL | Thermo Scientific | 69570 or 69572 | used for buffer exchange during quantum dot coupling to DNA |
| sodium bicarbonate | EMD Millipore | SX0320 | use to make buffer for surface functionalization; 100 mM, pH 8 |
| sodium chloride | Macron | 7581-12 | use to make experimental buffers |
| Sodium phosphate dibasic solution (BioUltra, 0.5 M in water) | Sigma Aldrich | 94046 | use to make 100 mM sodium phosphate buffer |
| Sodium phosphate monobasic solution (BioUltra, 5M in water) | Sigma Aldrich | 74092 | use to adjust pH of 100 mM sodium phosphate buffer |
| Streptavidin from Streptomyces avidinii | Sigma Aldrich | S4762 | dissolve at 1 mg/mL and store 25 mL aliqouts at -20 ? |
| Sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexane-1-carboxylate (sulfo-SMCC) | Thermo Scientific | A39268 | No-Weigh Format, 2 mg/vial |
| Syringe fitted with blunt 21 G needle | |||
| Syringe pump | |||
| thiolated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Thiol Modifier C6 S-S and HPLC purificaiton |
| TIRF imaging system with 488 nm laser illumination | various | custom built | |
| Tris -HCl | Research Products International | T60050 | use to make experimental buffers |
| Tris base | JT Baker | 4101 | use to make experimental buffers |
| Tween-20 | Sigma | P7949 | use to make blocking buffer |
| Ultrapure water | |||
| vortex mixer | VWR | 10153-842 | |
| Wash-N-Dry Coverslip Rack | Electron Microscopy Sciences | 70366-16 | used for surface functionalization of coverslips |
参考文献
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5905-5908 (2005).
- Loenen, W. A., Dryden, D. T., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Research. 42 (1), 3-19 (2014).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE--a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research. 43, Database issue 298-299 (2015).
- Bonnet, I., et al. Sliding and jumping of single EcoRV restriction enzymes on non-cognate DNA. Nucleic Acids Research. 36 (12), 4118-4127 (2008).
- Biebricher, A., Wende, W., Escude, C., Pingoud, A., Desbiolles, P. Tracking of single quantum dot labeled EcoRV sliding along DNA manipulated by double optical tweezers. Biophysical Journal. 96 (8), 50-52 (2009).
- Blainey, P. C., et al. Nonspecifically bound proteins spin while diffusing along DNA. Nature Structural & Molecular Biology. 16 (12), 1224-1229 (2009).
- Huang, C. -F., et al. Direct visualization of DNA recognition by restriction endonuclease EcoRI. Journal of Experimental & Clinical Medicine. 5 (1), 25-29 (2013).
- Palma, M., et al. Selective biomolecular nanoarrays for parallel single-molecule investigations. Journal of American Chemical Society. 133 (20), 7656-7659 (2011).
- Reinhard, B. M., Sheikholeslami, S., Mastroianni, A., Alivisatos, A. P., Liphardt, J. Use of plasmon coupling to reveal the dynamics of DNA bending and cleavage by single EcoRV restriction enzymes. Proceedings of the National Academy of Sciences of the United States of America. 104 (8), 2667-2672 (2007).
- van den Broek, B., Noom, M. C., Wuite, G. J. DNA-tension dependence of restriction enzyme activity reveals mechanochemical properties of the reaction pathway. Nucleic Acids Research. 33 (8), 2676-2684 (2005).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Loparo, J. J., van Oijen, A. Single-molecule enzymology. Handbook of Single-Molecule Biophysics. Hinterdorfer, P., Oijen, A. , Springer. New York, NY. 165-182 (2009).
- Taylor, J. D., Badcoe, I. G., Clarke, A. R., Halford, S. E. EcoRV restriction endonuclease binds all DNA sequences with equal affinity. Biochemistry. 30 (36), 8743-8753 (1991).
- Pingoud, A., Jeltsch, A. Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29 (18), 3705-3727 (2001).
- Tanner, N. A., Loparo, J. J., van Oijen, A. M. Visualizing single-molecule DNA replication with fluorescence microscopy. Journal of Visualized Experiments: JoVE. (32), e1529(2009).
- Kulczyk, A. W., Tanner, N. A., Loparo, J. J., Richardson, C. C., van Oijen, A. M. Direct observation of enzymes replicating DNA using a single-molecule DNA stretching assay. Journal of Visualized Experiments: JoVE. (37), e1689(2010).
- Baldwin, G. S., Vipond, I. B., Halford, S. E. Rapid reaction analysis of the catalytic cycle of the EcoRV restriction endonuclease. Biochemistry. 34 (2), 705-714 (1995).
- Winkler, F. K., et al. The crystal-structure of Ecorv endonuclease and of Its complexes with cognate and non-cognate DNA fragments. EMBO Journal. 12 (5), 1781-1795 (1993).
- Sioss, J. A., Stoermer, R. L., Sha, M. Y., Keating, C. D. Silica-coated, Au/Ag striped nanowires for bioanalysis. Langmuir. 23 (22), 11334-11341 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。