Method Article
마우스 췌 장 내 분 비 세포의 단일 셀 Transcriptomic 분석
요약
단일 셀 RNA 시퀀싱 뒤 미 발달, 신생아 및 산 후 췌 내 분 비 세포의 격리 하는 방법을 설명 합니다. 이 방법을 사용 하면 분석 췌 장 내 분 비 계보 개발의 셀이 transcriptomic 역학.
초록
독도에서 클러스터는 췌 장 내 분 비 세포는 혈액 포도 당 안정성과 에너지 대사 조절. 인슐린 분 비 하는 β 세포를 포함 하 여 독도에 뚜렷한 세포 유형 배아 단계에서 일반적인 내 분 비 창시자에서 구분 됩니다. 미 숙 내 분 비 세포는 세포 증식을 통해 확장 하 고 긴 출생 후 발달 기간 동안 성숙. 그러나, 이러한 프로세스를 기본 메커니즘 명확 하 게 정의 되지 않습니다. 단일 셀 RNA 시퀀싱은 뚜렷한 세포 인구와 추적 세포 계보 차별화 경로 특성에 대 한 유망한 접근 이다. 여기, 우리는 배아, 신생아 및 산 후 췌에서 고립 된 췌 장 β 세포의 단일 셀 RNA 시퀀싱에 대 한 메서드를 설명합니다.
서문
췌는 포유동물에 중요 한 변화 기관 이다. 췌 장 내 분 비와 외 분 비 구획 구성 됩니다. 췌 장 내 분 비 세포, 인슐린을 생산 하는 β 세포와 글 루카 곤 생성 α 세포를 포함 하 여 독도 Langerhans에 함께 클러스터링 하 고 전할 조직의 포도 당 항상성 조절. 내 분 비 세포의 기능 장애 결과 당뇨병 mellitus, 전세계 주요 공중 보건 문제가 되 고 있습니다.
췌 장 내 분 비 세포는 Ngn3에서 파생 된+ embryogenesis1동안 창시자. 나중에, perinatal 기간 동안 내 분 비 세포 증식 양식 미 성숙한 독도를. 이 미 성숙한 세포 개발 하 고 점차 성숙한 독도, 성인2혈액 포도 당 항상성 조절에 나가도록 풍부한 되는 계속.
비록 transcriptional 요인의 그룹 β 세포 분화를 조절 하는 발견 되었습니다, β 세포의 정확한 성숙 통로 아직 명확 하지 않습니다. 또한, β 세포 성숙 프로세스는 또한 셀 번호 확장3,4 의 규정 및 세포질이5,6세대 포함 한다. 그러나, 이러한 프로세스의 규제 메커니즘 하지 되었습니다 잘 공부 했습니다.
단일 셀 RNA 시퀀싱은 셀 부분 모집단을 프로 파일링 하 고 세포 계보 발달 경로7추적 수 있는 강력한 방법입니다. 이 기술, 단일 셀 레벨8에서 췌 장 섬 개발 중 발생 하는 이벤트를 해독 수 있습니다 키를 활용. 단일 셀 RNA 시퀀싱 프로토콜 중 스마트 seq2 향상 된 감도 정확도, 그리고 낮은 비용9표준 시 약의 사용 전장 cDNA의 생성을 허용 한다. 스마트 seq2 시퀀싱10cDNA 라이브러리를 만드는 데 약 2 일 소요 됩니다.
여기, 우리는 형광 활성화 된 세포 분류 (FACS)를 사용 하 여 성인 Ins1-RFP 유전자 변형 마우스11, 태아의 췌에서 형광 표시 된 β 세포의 격리 하는 방법을 제안 하 고 transcriptomic의 성능 분석에 스마트 seq2 기술 (그림 1)를 사용 하 여 단일 셀 수준입니다. 이 프로토콜은 일반, 병리학 및 노화 상태에서 각종 췌 장 내 분 비 세포의 transcriptomes 분석을 확장할 수 있습니다.
프로토콜
여기에 설명 된 모든 메서드는 기관 동물 관리 및 사용 위원회 (IACUC) 북경 대학에 의해 승인 되었습니다.
1. 췌 장 격리
-
E17.5 (배아 일 17.5) 배아에 대 한:
- 질 플러그 나타나면 시간 포인트에 따라 배아 하루 0.5 예상.
- CO2 관리에 의해 임신 쥐를 희생. 스프레이 70% 알코올로 복 부 모피입니다.
- 갈비뼈에 확장 하는 생식 기 지역에서가 위로 V 자형 절 개를 확인 합니다. 이 프로세스는 완전히 복 부 구멍을 열립니다 합니다.
- 복 부 구멍에서 자 궁 해 부를 포함 하는 차가운 PBS 10 cm 접시에 넣습니다.
- 다른 조직, 태 반 및 탯 줄 등을 제거 하는 stereomicroscope 아래 얇은 밀고 집게와 자 궁에서 태아를 해 부. 차가운 PBS를 포함 하는 10 cm 접시에 모든 배아를 배치 합니다.
- 태아의 복 부 구멍을 열고 눈물 고 팔꿈치 핀셋으로 내장 조직에 밖으로 파고. 내장 조직 차가운 PBS를 포함 하는 6 cm 블랙 하단 접시에 전송 합니다.
- 췌 장 부분 복 부의 왼쪽 상단에 있으며, 위, 비장, 십이지 장 ( 그림 2A에서 노란색 점선)12에. 집게를 사용 하 여 내장 조직에서 췌를 분리 하 고 0.5 mg/mL 차가운 콜라 솔루션의 5 mL를 포함 하는 20 mL 유리병으로 췌 장 조직 함께 수영장: 0.5 mg/mL 콜라 P 절연 버퍼 (HBSS 10 mM HEPES, 1 mM MgCl2 를 포함 하 5 mM 포도 당, pH 7.4).
-
P0-P15 (출생 후 하루 0-15) 마우스:
- 희생 하 고 벤치탑 보호 테이프의 조각에 사지를 준수 하 여 마우스를 해결. 스프레이 70% 알코올로 복 부 모피입니다.
- 1.1.3 단계에서 설명한 대로 완전히 복 부 구멍을 엽니다. 마우스의 왼쪽을 밖으로 창 자를 당겨. 이 절차는 췌 십이지 장, 위, 및 비장 돌출부를 노출 합니다. 신중 하 게 해 부 췌 (그림 2B)12 의 모든 엽 고 0.5 mg/mL 차가운 콜라 솔루션의 5 mL를 포함 하는 20 mL 유리병으로 췌 장 조직 함께 수영장.
-
P18-P60 (출생 후 하루 18-60) 마우스:
- 콜라 솔루션을 준비 합니다. 사용할 준비까지 얼음에 계속.
- 마우스를 희생 하 고 (단계 1.1.2와 1.1.3) 위에서 언급 한 복 부 구멍을 엽니다.
참고: 간 다치게 하지 않아, 간 어떤 상처에 담 즙 덕트 흐름 압력을 감소 다음 단계의 관류 효율성을 줄일 것 이다. - 칼을 제거 합니다. 마우스의 오른쪽을 창 밖으로 당겨 그리고 쓸 개 ( 그림 2C에서 흰색 화살표) 및 일반적인 담 관을 노출 하는 각 측에의 왼쪽 및 오른쪽 중간 엽 밀어.
- 측면에 서는 담 관 십이지 장 (그림 2D)를 입력 하는 사이트는 위와 더 낮은 위치에서 작은 선박 클램프의 쌍으로 십이지 장을 클램프.
참고: 췌 장의 위 또는 비장 돌출부 클램프 하지 마십시오. 콜라 솔루션 하지 고정 하는 경우 다음 단계에서 췌의 위장과 비장 엽으로 흐르게 됩니다. - 0.5 mg/mL 차가운 콜라 솔루션의 5 mL을 주사기를 쓸 개에 30g 바늘을 삽입. 신중 하 고 원활 하 게, 일반적인 담 관을 통해 바늘을 조작 합니다.
- 마우스의 크기에 따라 0.5 mg/mL 차가운 콜라 솔루션의 1-5 mL를 주입 하 여 췌 장 perfuse. 주사는 느리고 지속적인 덕트에서 미 끄 러에서 바늘을 방지 하 고 높은 액체 압력 파열에서 소장을 방지 해야 합니다. 주사는 췌 완전히 확장 완료 됩니다.
- 재 관류 후 즉시 집게를 사용 하 여 복 부 구멍에서 췌 장 (노란 점선 그림2d에서)를 해 부. 장소 20 mL 유리병 0.5 mg/mL 차가운 콜라 솔루션의 5 mL를 포함 하는 조직. 한 번에 둘 이상의 마우스를 조작, 마우스 하나를 perfuse 하 고 모든 마우스 해 부 될 때까지 20 mL 유리병에 차가운 콜라 솔루션으로 조직 수영장.
2. 콜라 소화와 섬 고립
- 37 ° C 물 목욕으로 췌 장 조직을 포함 하는 20 mL 유리병 장소와 온도 equilibrate를 3 분 동안 품 어.
- 부드럽게 다른 3 ~ 5 분에 대 한 튜브를 흔들. 경우 췌 장 조직을 완벽 하 게 비정상적으로 증가, 그것은 마지막으로 균일 하 게 분산 때까지 작은 조직 조각으로 점차적으로 해리 됩니다. 소화 시간은 췌 크기와 관류 효율에 따라 달라 집니다.
- 새로운 50 mL 원심 분리기 관으로 0.25 m m 나일론 여과기를 통해 소화 제품을 필터링 합니다. 철저 하 게 얼음 처럼 차가운 PBS를 포함 20 mL 주사기를 사용 하 여 여과기 세척.
- E17.5 P15 췌 위해 3.1 단계로 건너뜁니다.
- P18-P60 췌, 1 분 동안 200 x g 에서 원심 및 삭제는 상쾌한. 다시 차가운 PBS와 조직을 일시 중단 합니다.
- 6 cm 블랙 하단 접시에 조직 서 스 펜 션의 5 mL를 붓으십시오. 췌 장 독도 작은, 소형, 유백색 백색 구조 이며 포상 조직을 느슨하게 하 고 반투명 화이트. 독도와 200 μ 피 펫을 선택 하 고 차가운 PBS의 작은 금액을 포함 하는 1.5 mL 튜브에 그들을 전송.
3. 췌 장 조직이 나 독도의 트립 신 소화
- 원심 관 췌 장 조직이 나 200 x g 4 ° C에서 1 분 동안에 독도 포함 하 고는 펠 렛을 방해 하지 않고는 상쾌한 삭제.
- 다시 0.25 %trypsin-EDTA와 펠 릿을 일시 중단 하 고 37 ° C 물 욕조에 품 어. 외피의 4 분 후 부드럽게 및 가끔 1 분 200 μ 팁을 사용 하 여 (피 펫)을 발음.
참고: E17.5 P3 췌, 1 mL 트립 신-EDTA 추가. P4-P15 췌 3 mL 트립 신-EDTA를 추가 합니다. 100-300 독도 대 한 트립 신-EDTA 1 mL를 추가 합니다. 트립 신-EDTA 조직의 양에 따라의 볼륨을 조정 합니다. - 0.4 x 양의 찬 태아 둔감 한 혈 청 (FBS)를 추가 하 여 소화를 중지 하 고 부드러운 소용돌이 의해 혼합.
- 4 ° c.에 3 분 동안 250 x g 에서 원심 분리기 펠 릿을 방해 하지 않고는 상쾌한을 버리십시오.
- 다시 200 μ 차가운 FACS 버퍼와 셀을 일시 중단 (HBSS 포함 하는 1 %FBS, pH 7.4). 5 mL FACS 튜브에 세포를 전송.
- 신속 하 게 세포 현 탁 액 소화 되지 않은 큰 조직 파편을 제거 하는 필터를 통해 전달할 수 있도록 FACS 튜브를 회전 합니다. 단일 셀 서 스 펜 션 튜브에서는 이제 FACS 정렬에 대 한 준비가 되었습니다.
4. 단일 세포 세포의 용 해
-
세포 세포의 용 해 버퍼를 준비 합니다.
참고: 층 류와 UV 소독 후드 아래 모든 실험을 수행 합니다. 모든 튜브, 접시와 피 펫 팁 해야 RNase-DNase 무료 /. 후드와 사용 하기 전에 RNase 멀리 솔루션 펫 오염을.- 얼음에 시 약을 녹여: dNTP (10 m m), 올리고 dT 프라이 머 (10 μ M), 및 ERCC 재고 솔루션 (1:20).
- 1:5 x 10 ERCC 희석 nuclease 무료 물5 .
- 필요한 세포 세포의 용 해 버퍼의 볼륨을 계산 합니다. RNase 억제제 (40 U/μ), 0.2% (vol/vol) 트라이 톤 X-100, dNTP, 올리고 dT 뇌관의 1 μ의 각 셀에 대 한 4.05 μ의 최종 볼륨을 희석된 ERCC의 0.05 μ 1 μ의 1.9 μ의 0.1 μ를 추가 합니다.
- 약 0.2 mL 얇은 8-스트라이프 PCR 튜브 또는 96 잘 접시에 세포 세포의 용 해 버퍼 수. 원심 관 또는 30 접시 4 ° c.에 s
참고: x g 및 다음 단계에서 800 x g에서 96 잘 접시 7500에서 0.2 mL 얇은 8-스트라이프 PCR 튜브 원심.
-
단일 셀 따기 그리고 세포입니다.
- 수동으로 선택 FACS 8 스트립 PCR 튜브는 30-40 µ m 모 세관 피 펫를 사용 하 여 Ins1-RFP+ FACS 버퍼에 단일 셀을 정렬 하거나 직접 96 잘 접시로 Ins1-RFP+ 단일 셀을 정렬 합니다. 단일 셀을 포함 하는 볼륨 미만 0.3 μ 간주 됩니다.
참고: 사용 하 여 앞으로 살포 높이 (FSC-H) vs 분산형 영역 (FSC-A) 남자 용 상의 차별에 대 한 제어 전략, FSC-H 대 측 분산형 지역 파편 및 형광 Ins1-RFP+ 셀 (그림 3A-3C) 정렬을 위한 게이팅 (SSC A). 채집 방법에 대 한 300 μ FACS 버퍼를 포함 하는 1.5 mL 튜브에 대상 셀을 정렬 합니다. 적절 한 최종 농도 5-10 셀/μ. 따라 버퍼 볼륨을 조정 합니다. 플레이트 수집 방법에 대 한 악기의 설명서에 따라 96 잘 접시의 각 음에 단일 셀을 정렬 합니다. 7 , 13 - 소용돌이 관 또는 세포를 lyse RNA를 분리 하는 접시. 원심 관 또는 30 접시 s 4 ° C에서 그리고 즉시 얼음에 그들을 배치.
참고: 셀 저장할 수 있습니다-80 ° C에 1 주일에 대 한.
- 수동으로 선택 FACS 8 스트립 PCR 튜브는 30-40 µ m 모 세관 피 펫를 사용 하 여 Ins1-RFP+ FACS 버퍼에 단일 셀을 정렬 하거나 직접 96 잘 접시로 Ins1-RFP+ 단일 셀을 정렬 합니다. 단일 셀을 포함 하는 볼륨 미만 0.3 μ 간주 됩니다.
5. 단일 셀 cDNA 증폭
-
반전 녹음 방송 (실시간)
- RT 시 약 (표 1) 얼음에 녹여
- 3 분 동안 72 ° C에서 샘플을 품 어 및 즉시 넣어 튜브 또는 적어도 1 분에 대 한 얼음에 플레이트 짧게 원심 튜브 또는 30 접시 4 ° c.에 s
참고: 사용 하 여 105 ° C가 열 뚜껑 열 cycler 모든 외피. - 표 1에 설명 된 대로 모든 반응에 대 한 실시간 믹스를 준비 합니다.
- 10 μ의 총 볼륨을가지고 각 샘플에 실시간 믹스의 5.7 μ 분배. 부드럽게 소용돌이 믹스와 30에 대 한 원심 분리기 4 ° c.에 s
- 샘플 열 cycler에 놓고 다음과 같은 실시간 프로그램을 시작: 90 분, 10 주기 (2 분, 2 분 동안 42 ° C 50 ° C), 42 ° C 15 분 및 4 ° c.에 보류에 대 한 70 ° C
-
PCR의 사전 증폭
- PCR 시 약 (표 2) 얼음에 녹여
- 표 2에 설명 된 대로 모든 반응에 대 한 PCR 믹스를 준비 합니다.
- 첫 번째 가닥 반응을 포함 하는 각 샘플을 PCR 혼합의 15 μ 분배. 부드럽게 소용돌이 믹스와 30에 대 한 원심 분리기 4 ° c.에 s
- 샘플 열 cycler에 놓고 다음 PCR 프로그램 시작: 3 분, 18 주기 98 ° C (20 98 ° C s, 67 ° C 15에 대 한 s, 6 분 동안 72 ° C), 72 ° C 5 분 및 4 ° c.에서 개최
참고: PCR 제품 1 주일 미만 4 ° C에 또는에서-20 ° C /-80 ° C 최대 6 개월까지 저장할 수 있습니다.
-
PCR 정화입니다.
- 소용돌이 의해 잘 혼합 하 고 이전 단계에서 각 샘플을 다시 일시 중단 된 DNA 정화 구슬 (1x)의 25 μ를 추가 합니다. 다음, 신속 하 게 튜브 또는 액체를 수집 하지만 구슬의 타협을 방지를 위해 상 온에서 판 스핀.
참고: 15 분을 사용 하기 전에 철저 하 게 소용돌이 실내 온도에 DNA 정화 구슬 Equilibrate. - 실 온에서 5 분 동안 품 어.
- 튜브 또는 자기 접시에 적절 한 해결책은 때까지 서 서 다음 신중 하 게 제거 하 고 삭제는 상쾌한.
- 마그네틱 스탠드에 있는 동안 구슬 씻어 갓된 80% 에탄올의 200 μ 추가, 30 품 어 s, 다음 신중 하 게 제거 하 고 에탄올 솔루션 삭제.
참고: 80% (vol/vol) 에탄올 솔루션 준비 되어야 한다 갓 각 시간. - 2 세척 총 5.3.4 단계를 반복 합니다.
- 조심 스럽게 제거 하 고 나머지 삭제 또는 플레이트 마그네틱 스탠드에는 에탄올 솔루션 및 공기 건조 튜브 동안 구슬.
참고: 최대 차입 효율성을 지키는 구슬 지나치게 건조 하지 마십시오. - 구슬에서 DNA 대상 elute 하는 소용돌이 의해 잘 혼합 nuclease 무료 물 11 μ를 추가 합니다. 다음, 신속 하 게 튜브 또는 격판덮개 회전 고 마그네틱 스탠드에 배치 하는 때까지 솔루션은 분명 하다. 새로운 PCR 튜브에 샘플의 10 μ를 전송.
참고: 경우 이합체 cDNA 크기 분포 감지에 따라 정화의 단일 라운드는 이합체를 완전히 제거 하려면 다시 정화. 샘플에 남아 허용 하는 경우는 이합체 cDNA 수율 계산을 좌우할 수 있다.
- 소용돌이 의해 잘 혼합 하 고 이전 단계에서 각 샘플을 다시 일시 중단 된 DNA 정화 구슬 (1x)의 25 μ를 추가 합니다. 다음, 신속 하 게 튜브 또는 액체를 수집 하지만 구슬의 타협을 방지를 위해 상 온에서 판 스핀.
-
CDNA의 품질 확인 합니다.
- 임의로 샘플 cDNA 총 수확량은 fluorometer를 사용 하 여 검색을 선택 합니다.
- 실시간 PCR (정량) (그림 4E)에 의해 마커 유전자 표현 레벨을 평가 합니다. 40 배 희석 하 여 샘플을 정량 384-잘 접시를 사용 하 여 수행의 1 μ를 제거 합니다. 표 3에 설명 된 대로 정량 믹스를 준비 합니다. 사이클링 조건: 10 분, 45 주기 동안 95 ° C (95 ° C 10 s, 15에 대 한 60 ° C s, 72 ° C 15 s).
- 임의로 샘플 악기를 병렬 모 세관 전기 이동 법을 사용 하 여 크기 분포를 검색을 선택 합니다.
6입니다. cDNA 도서관 건축
-
Tagmentation Tn5 transposase에 의해 반응입니다.
- 검색 단계는 fluorometer를 사용 하 여 5.4.2에서 정량-선택한 셀의 cDNA 총 수익률. 시작 물자로 cDNA의 사용 2 ng.
- Tagmentation 반응 시 약 (표 4) 얼음에 녹여
- 0.2 mL 얇은 8-스트라이프 PCR 튜브, 표 4에 설명 된 대로 tagmentation 반응 준비 하 고 신중 하 게 소용돌이 의해 혼합. 다음, 신속 하 게 상 온에서 솔루션 아래로 회전 합니다.
- 10 분 동안 55 ° C에서 샘플을 품 어와 4 ° c.에서 개최
- 즉시 tagmented 반응을 중지 하는 DNA를 포함 하는 각 샘플을 5 x TS의 2 μ를 추가 합니다. 소용돌이에 의해 신중 하 게 혼합 하 고 스핀 실 온에서 솔루션 다운 신속 하 게.
- 실 온에서 5 분을 위한 혼합물을 품 어. DNA는 최종 농축 PCR 즉시 처리 해야 합니다.
-
어댑터 출혈 조각 증폭
- PCR 시 약 (표 5) 얼음에 녹여
- 표 5에 설명 된 대로, 농축 PCR 믹스를 준비 하 고 소용돌이 의해 신중 하 게 혼합. 다음, 신속 하 게 상 온에서 솔루션 아래로 회전 합니다.
- 다음 프로그램을 사용 하 여 PCR을 수행: 72 ° C 10 분, 30 대 98 ° C에 대 한 s, 8 사이클 (15 98 ° C s, 60 ° C 30에 대 한 s, 3 분 동안 72 ° C), 72 ° C 5 분 및 4 ° c.에서 개최
참고: 주기 수 예상된 라이브러리 DNA 금액에 따라 달라 집니다.
-
크기 선택과 PCR 정화입니다.
- 소용돌이 의해 잘 혼합 하 고 이전 단계에서 각 샘플을 다시 일시 중단 된 DNA 정화 구슬 (0.7 x)의 14 μ를 추가 합니다. 다음, 신속 하 게 액체를 수집 하지만 구슬의 타협을 방지를 위해 상 온에서 관 회전.
참고: 15 분을 사용 하기 전에 철저 하 게 소용돌이 실내 온도에 DNA 정화 구슬 Equilibrate. - 실 온에서 5 분 동안 품 어.
- 튜브 적절 한 마그네틱 스탠드에 솔루션은 분명 때까지, 신중 하 게 새로운 튜브 스트립에는 상쾌한 전송 놓고 이전 튜브 스트라이프를 삭제 합니다.
- 다시 일시 중단 된 DNA 정화 구슬의 3 μ 추가 (0.15 x) 각 샘플 튜브 스트라이프에 소용돌이 의해 잘 혼합. 다음, 신속 하 게 상 온에서 관 회전.
- 실 온에서 5 분 동안 품 어.
- 튜브 또는 자기 접시에 적절 한 해결책은 때까지 서 서 다음 신중 하 게 제거 하 고 삭제는 상쾌한.
- 마그네틱 스탠드에 있는 동안 구슬 씻어 갓된 80% 에탄올의 200 μ 추가, 30 품 어 s, 다음 신중 하 게 제거 하 고 에탄올 솔루션 삭제.
참고: 80% (vol/vol) 에탄올 솔루션 준비 되어야 한다 갓 각 시간. - 2 세척 총 6.3.7 단계를 반복 합니다.
- 조심 스럽게 제거 하 고 나머지 삭제 또는 플레이트 마그네틱 스탠드에는 에탄올 솔루션 및 공기 건조 튜브 동안 구슬.
참고: 최대 차입 효율성을 지키는 구슬 지나치게 건조 하지 마십시오. - 구슬에서 DNA 대상 elute 하는 소용돌이 의해 잘 혼합 nuclease 무료 물 11 μ를 추가 합니다. 다음, 신속 하 게 튜브 또는 격판덮개 회전 고 마그네틱 스탠드에 배치 하는 때까지 솔루션은 분명 하다. 새로운 튜브 스트라이프에 샘플의 10 μ를 전송.
- 소용돌이 의해 잘 혼합 하 고 이전 단계에서 각 샘플을 다시 일시 중단 된 DNA 정화 구슬 (0.7 x)의 14 μ를 추가 합니다. 다음, 신속 하 게 액체를 수집 하지만 구슬의 타협을 방지를 위해 상 온에서 관 회전.
-
최종 cDNA 라이브러리의 품질 확인 합니다.
- fluorometer을 사용 하 여 각 라이브러리의 농도 측정 하 고 병렬 모 세관 전기 이동 법 악기를 사용 하 여 크기 분포를 확인 합니다.
참고: DNA 수율은 일반적으로 각 라이브러리에 대 한 15-25 ng 사이. 250에서 배열 하는 조각 450에 혈압 혈압 관찰 될 것 이다. 이합체 정화, 후 그대로 크기 분포 검사에 의해 확인, 만약 한 번 더 DNA 정화 구슬 x 1와 정화.
- fluorometer을 사용 하 여 각 라이브러리의 농도 측정 하 고 병렬 모 세관 전기 이동 법 악기를 사용 하 여 크기 분포를 확인 합니다.
-
라이브러리 풀링
- 대략 조각 크기, 그들 중 누구도 같은 조합의 N6XX 및 N8XX 어댑터를 포함 하는 보장 하는 각 샘플에서 DNA의 수영장 동일 금액을 기반으로 합니다.
7. DNA 시퀀싱
- 높은 처리량 시퀀싱 시스템을 사용 하 여 51 bp 일자형 연속을 주제 바코드 라이브러리. 제조 업체의 프로토콜에 따라 시퀀싱을 수행 합니다. 각 셀의 시퀀싱 깊이 약 1 백만 읽고 평균8, 적어도 0.5 백만 셀14당 읽습니다.
8. 생물 정보학 분석
-
시퀀싱 품질 평가 및 정렬.
- 다음 매개 변수 FastQC (v0.11.3)15 를 사용 하 여 시퀀스 된 읽기의 품질 평가: "fastqc-추출-o output_dir input_fastq".
- ERCC 시퀀스는 명령을 사용 하 여 마우스 게놈 병합 "mm10.fa ERCC.fa 고양이 > mm10_ERCC.fa".
- 빌드 bowtie2 (v2.2.5)16 인덱스 다음 매개 변수: "bowtie2-빌드 mm10_ERCC.fa mm10_ERCC".
- Tophat2를 사용 하 여 읽기 정렬 (v2.1.0)17 다음 매개 변수: "tophat2-o output_dir-G gene.gtf-transcriptome 색인 trans_index mm10_ERCC input_fastq".
-
유전자 표현 레벨을 계량.
- 수 매핑된 매개 변수가 HTSeq (v 0.6.0)18 를 사용 하 여 각 유전자에 대 한 읽기: ' htseq-카운트-f bam-r pos-s-a 30 accepted_hits.bam gene.gtf > read_count.txt '.
- 진 식 레벨 백만 (TPM)19당 성적을 정상화.
-
셀의 품질 관리
- 미만 0.5 백만 매핑된 읽기 또는 미만 4000 유전자를 가진 세포를 제외 (TPM > 1).
참고: 제외 기준 셀 유형 및 시퀀스 깊이에 따라 달라 집니다. - 내 분 비 마커 ( Gcg α 세포, β 세포에 대 한예를 들어, Ins1 )에 게, 표현 하는 세포를 유지 하 고 비 내 분 비 마커 (백혈구에 대 한예를 들어, Spi1 )를 표현 하는 셀을 제외 합니다.
- 미만 0.5 백만 매핑된 읽기 또는 미만 4000 유전자를 가진 세포를 제외 (TPM > 1).
-
주성분 분석 (PCA)
- 앞에서 설명한20ERCC 스파이크 기능에 따라 매우 변수 유전자를 식별 합니다.
- PCA "PCA" r에서와 FactoMineR (v1.31.4)21, 패키지 기능을 사용 하 여 수행 log2(TPM + 0.1) 높은 변수 유전자의.
- Ggplot2와 PCA 결과 시각화 (v2.0.0)22.
-
계층적 클러스터링입니다.
- FactoMineR (v1.31.4)21"dimdesc" 기능을 사용 하 여 가장 높은 주성분 (PC) 선적으로 유전자를 식별 합니다.
- Log2 함수 R 패키지 gplots (v3.0.1)23, "heatmap.2"를 사용 하 여 계층적 클러스터링 수행 (TPM + 1) 유전자를 로드 하는 높은 PC의 상대 값.
결과
췌 미 발달, 신생아 및 산 후 마우스에서 해 부 했다 (그림 2A 및 2B). 쥐 출생 후 하루 18 보다 더 오래 된, 대 한 소화 효과 관류;의 정도에 따라 달라 집니다. 따라서, 주사 섬 격리에 대 한 가장 중요 한 단계는 (그림 2C-2E 및 표 6). 이 단계 동안 췌를 채울 수 있어서 많은 콜라 주입 했다. 완전히 비정상적된 췌 2D 그림에에서 표시 됩니다. 경우는 관류 성공 (그림 2E), 하지만 샘플은 소중한 췌 나중에 충분 한 소화를 위한 작은 조각으로 찢 겨 수 있습니다.
관류, 후 췌 장 조직 독도 (그림 2F) 출시를 작은 조각으로 소화 했다. FACS 정렬 시간 단축, 우리는 독도 미리 선택 하 여 내 분 비 세포를 농축. 독도의 크기는 마우스 연령과 소화 강도 따라 달라질 수 있습니다. 때때로,는 독도 모양에서 라운드 되지 않습니다. 독도 색상과 소형 상태 (그림 2G 및 표 6)에 따라 선택 되어야 합니다. 유전자 변형 마우스 스트레인은 GFP 등 내 분 비 세포에 대 한 RFP 기자 진 경우는 독도 수 또한 형광 현미경 선택 됩니다.
Ins1-RFP+ 세포 (그림 3A-3 C) 분류, FACS에 의해 순화 했다 하 고 단일 셀 모 세관 피 펫을 사용 하 여 단일 셀 RNA-seq (그림 3D)에 대 한 선택 했다. 성공적으로 증폭 된 cDNA 전장 500 위에 있어야 혈압 1.5 kb에서 2 kb로 농축 되 고. 또한, 일반적으로 인슐린 성적표 (그림 4A) 나타내는 수 있습니다 Ins1-RFP+ 세포에서 관찰 된 cDNA의 500-600 bp 농축이입니다. 그러나, 일부 비정상적인 상황 관찰된10 (표 6) 되었습니다. 예를 들어 cDNA 조각 100에 가까운 혈압은 뇌관 이합체 (그림 4B)는 일반적으로 DNA 정화 단계를 반복 하 여 제거 해야 하는 과도 한 뇌관에 의해 발생. 뇌관 이합체의 존재 다음 도서관 건축을 위해 사용 되는 전체 cDNA 수익률의 계산에 영향을 미칠 수 있습니다. 100 사이 cDNA 조각 bp 및 500 bp 보통 대표 저하 RNase 오염 등 나쁜 세포 상태 또는 시 약 문제에 의해 발생 하는 cDNA (그림 4C). 이 조건 하에서 DNA 저하의 원인을 파악 하 고 소요를 제외 해야 합니다. 예를 들어, 좋은 셀 상태를 보장 하기 위해, 조직을 소화 해야 하 고 신속 하 고 부드럽게, 셀 정렬 되어야 작업을 모든 종류의 오염 물질을 피하기 위해 신중 하 게 수행 되어야 합니다.
시퀀싱에 대 한 cDNA 라이브러리의 정화 후 우리는 샘플을 DNA 정화 구슬의 다른 비율을 추가 하기 위한 지침에 따라 다양 한 크기의 cDNA 조각을 얻었다. 예를 들어 우리가 250에서 배열 하는 cDNA를 얻을 수 있습니다 혈압 450 추가 0.7 x와 첫 번째 DNA 정화 구슬 x 0.15 bp와 두 번째 라운드 정화, 각각 (그림 4D). 이 단계는 높은 성공 비율이 있다. 그러나, 약 100의 조각이 있다면 혈압, 이합체 (표 6) 제거 하려면 DNA 정화 구슬 x 1으로 다시 라이브러리를 순화 하는 것이 좋습니다. Unremoved 이합체 DNA 정량화를 기울일 것 이다 고 각 샘플에서 고르지 못한 데이터 수집에는 샘플 풀링 결과 좌우할 것 이다.
우리는 생물 정보학 접근법 (그림 5A)과 시퀀싱 데이터를 분석. 시퀀싱 품질 평가 점수는 품질 평가 (그림 5B)을 시퀀싱 하는 동안 30 보다 큰 해야 합니다. 맞춤 후 읽기의 80-90% 참조 게놈에 매핑될 것 예상 된다. 다운스트림 분석에 대 한 고품질의 세포를, 우리 미만 0.5 백만 매핑된 읽기로 셀 제외 또는 4000 미만으로 검출 유전자 (그림 5C 및 5d). PCA와 계층적 클러스터링, 후 우리와 다른 그룹8heterogeneously 표현 했다 유전자 확인 셀의 다른 그룹 특징.

그림 1: 단일 셀 마우스 췌 장 내 분 비 세포의 RNA-seq 도식 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2: 췌 장 해 부, 관류 및 섬 따기. (A) E17.5 배아 (위)에서 (더 낮은) 배아 췌 장 조직의 해 부. 노란 점선 정하는 췌 장 조직. 눈금 막대 = 1000 µ m. (B) P10 마우스 (왼쪽)에서 (오른쪽) 산 후 췌 장 조직의 해 부. 눈금 막대 = 1000 µ m. (C D) (C) 전에 P60 마우스의 췌 장 조직 및 (D) 재 관류 후. 노란 점선 정하는 췌 장 조직. 흰색 화살표 쓸 개를 나타냅니다. (E) P60 마우스의 부분적으로 perfused 췌 장 조직. 빨간색 화살표는 췌 장의 잘 perfused 영역을 나타냅니다. 파란색 화살표는 췌의 저조한 perfused 영역을 나타냅니다. (F)는 췌 장 조직 (위)에 전에 전후 (낮은) 콜라 소화. (G) 화살표 콜라 소화 췌 장 조직에서 발표 되었던 독도 가리킵니다. 눈금 막대 = 200 µ m. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3: 30-40 µ m 모 세관 피펫은 현미경으로 세포를 수동으로 따기. (A C) 단계별 FACS 게이팅 Ins1-RFP+ 셀을 정렬. (D)는 밝은 동그라미 표시 셀 이며 튜브 모 세관 피 펫. 더 나은 형태 (화살표) 셀을 선택 하 고 클러스터 셀 (화살촉)를 무시 한다. 눈금 막대 = 200 µ m. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 4: 품질 탐지 악기를 병렬 모 세관 전기 이동 법에 의해 Ins1-RFP+ 세포의 cDNA 및 라이브러리 크기 분포의. 성공적으로 미리 증폭 된 cDNA의 결과 (A) 대표. 일반적으로, cDNA 프로필 500 이상 이어야 한다 ~1.5–2 kb 피크와 혈압. (B) 뇌관 이합체 피크 미리 증폭 된 cDNA의 대표적인 결과. 뇌관 이합체 피크는 약 100 100 사이 bp. (C) 파편 중 증폭 된 cDNA의 프로필 bp 및 500 bp cDNA 저하의 가능성을 나타냅니다. (D) cDNA의 크기 분포 라이브러리 250 사이 혈압과 450 bp 다음이이 프로토콜에서 언급 된 정화 단계. (E) 식 정량 (왼쪽)와 상대 cDNA 라이브러리에서 Ins2 의 수준을 시퀀싱 데이터 (오른쪽). x-axes 고유 8 단일 셀 샘플을 나타냅니다. Y 축 (왼쪽) Gapdh (CtGapdh-CtIns2 + 6)를 기준으로 정규화 ΔCt을 나타내고 y 축 (오른쪽) Ins2 Gapdh (로그2(TPM 기준의 표준화 된 식 수준 Ins2 / TPMGapdh)). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
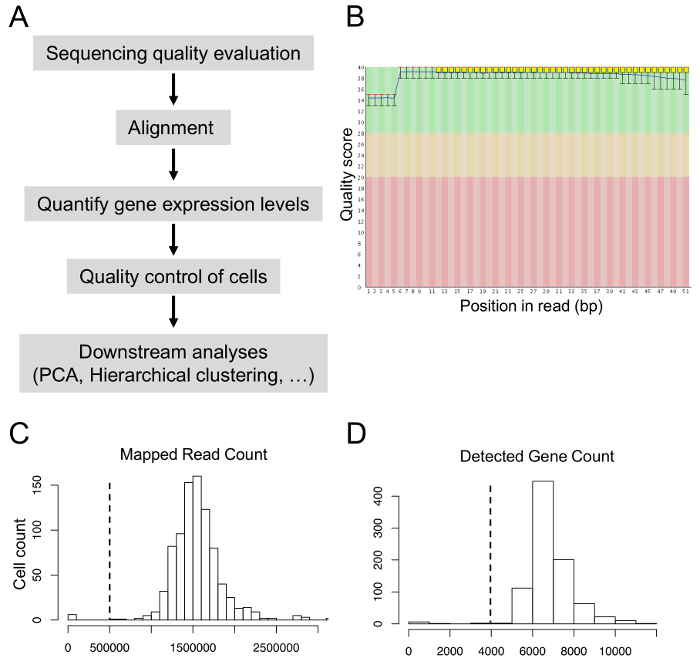
그림 5: 단일 셀 transcriptome 데이터의 생물 정보학 분석. (A)의 생물 정보학 분석 파이프라인. (B) 읽기의 모든 기지를 통해 품질 평가 점수를 시퀀싱. (C) 매핑된 읽기 수의 분포. (D) 검색 된 유전자 수의 분포. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 구성 요소 | 볼륨 (µ L) |
| 역전사 (200 U / µ L) | 0.5 |
| RNase 억제제 | 0.25 |
| 첫 번째 가닥 버퍼 (5 배) | 2 |
| DTT (100 mM) | 0.5 |
| Betaine (5m) | 2 |
| MgCl2 (1m) | 0.06 |
| TSO (100 Μ M) | 0.1 |
| Nuclease 무료 물 | 0.29 |
| 총 | 5.7 |
표 1: RT 시 약 5.7 µ L 반응 각 샘플에 대 한 구성 요소를 믹스.
| 구성 요소 | 볼륨 (µ L) |
| 첫번째 물가 반응 | 10 |
| DNA 중 합 효소 (2 x ReadyMix) | 12.5 |
| PCR 뇌관 (10 µ M)은 | 0.25 |
| Nuclease 무료 물 | 2 월 25 일 |
| 총 | 25 |
표 2: PCR 사전 증폭 시 약 혼합 25 µ L 반응 각 샘플에 대 한 구성 요소
| 구성 요소 | 볼륨 (µ L) |
| SYBR 녹색 마스터 믹스 | 5 |
| 뇌관 (5 µ M) | 0.5 |
| Nuclease 무료 물 | 2.5 |
| cDNA | 2 |
| 총 | 10 |
표 3: 정량 Pcr 시 약 10 µ L 반응 표준 384-잘 접시를 사용 하 여 구성 요소를 믹스.
| 구성 요소 | 볼륨 (µ L) |
| 5 x TTBL | 1.6 |
| 2 ng cDNA | 변수 |
| ddH2O | 변수 |
| 마주 믹스 V5 | 2 |
| 총 | 8 |
표 4: Tagmentation 시 약 각 샘플에 대 한 8 µ L 반응에서 구성 요소를 믹스.
| 구성 요소 | 볼륨 (µ L) |
| ddH2O | 1.6 |
| 이전 단계의 제품 | 10 |
| 5 탭 x | 4 |
| N6XX | 2 |
| N8XX | 2 |
| 태 | 0.4 |
| 총 | 20 |
표 5: 농축 PCR 시 약 믹스 구성 요소 20 µ L 각 샘플에 대 한 반응에.
| 단계 | 문제 | 가능성 | 솔루션 |
| 1.3.4 | 클램프의 미 끄 러 | 내장의 외부 표면은 젖은 작은 양의 중간 collegenase 및 leakness의 존재에 의해 발생 | 부드럽게 십이지 장 벽과 면봉과 복 부 구멍에 있는 다른 기관 벽을 교환 |
| 1.3.6 | 내장 파열 | 소장 액체 압력 너무 높습니다. | 사출 속도 느리게 |
| 2.6 | 독도 따기 때 독도 외 분 비 조직에 충실 | Insufficent 소화 | 소화 기간과 강도 떨고 연장 |
| 5.3 | PCR 확대 후 cDNA 항복이 낮습니다. | 나쁜 상태에 있는 셀 | 신선 하 고 좋은 상태에서 세포를 유지 |
| 5.4 또는 6.4 | 뇌관 이합체를 볼 수 있다 | 과도 한 프라이 머 | 정화는 cDNA DNA 정화 구슬 (0.8:1 또는 1:1 비율)을 한 번 더 |
| 5.4 | PCR 확대 후 저하 된 cDNA | 셀 이나 오염 된 시 약의 품질 | 좋은 조건에서 세포를 유지 또는 시 약 변경 |
| 6.3 | DNA의 양이 낮은 도서관 건설 후 | 너무 몇 PCR 주기 또는 cDNA의 품질은 좋지 않다 | 사이클의 수를 증가 또는 cDNA 도서관 건축 하기 전에 품질을 보장 |
표 6: 문제 해결.
토론
이 프로토콜에서 우리는 췌 장 β 세포의 단일 셀 식 프로필 공부에 대 한 효과적이 고 사용 하기 쉬운 방법 시연. 배아, 신생아 및 산 후 췌 내 분 비 세포를 분리 하 고 단일 셀 transcriptomic 분석을 수행 하려면이 메서드를 사용할 수 있습니다.
가장 중요 한 단계 좋은 조건에서 단일 β 세포의 고립입니다. 완전히 끼얹는다 췌 후속 소화에 더 나은 응답합니다. 일반적으로 등 쪽 췌 장에서 발생, 부족 한 관류 낮은 섬 항복 발생 합니다. 관류, 후 소화 시간과 강도 떨고 특별 한 주의 필요로 합니다. -소화, 긴 외피 시간에서 그리고 활기찬 떨고, 조각으로는 독도 끊을 수 있다. 부족 한 소화는 완전히 인접 조직에서는 독도 분리 하지 것입니다. 췌 장의 소화, 후에 독도 손 따기 보다는 조밀도 기온 변화도 원심 분리에 의해 정화 되었다. 밀도 그라데이션 원심 독도 풍요롭게 수 있습니다, 비록 많은 작은 독도 또는 포상 조직으로 연결 하는 독도 실수로 삭제 됩니다. 따기 포상 조직, 비효율적인 트립 신 소화를 발생할 수 있습니다 고 순도 β 세포의 감소를 방지 하려고 합니다.
독립적인 생물 복제는 생물학적 다양성 및 일괄 처리 효과 구별을 위한 필요 합니다. 2 생물 복제 하는 경우 PCA에는 비슷한 패턴을가지고 결과 클러스터링에 비슷한 하위 그룹을 포함,이 샘플 신뢰할 수 있는 생물 학적 변화를 공개 수 있습니다. 그렇지 않으면, 추가 생물 복제 결과 확인 해야 합니다. 췌 장 같은 대사 기관에 대 한 circadian 시계24 와 관련 된 셀 상태 일괄 차이 대 한 계정 수 있습니다. 이 문제를 해결 하려면 하루 중 같은 시간에 모든 샘플을 수집 하는 것이 좋습니다.
이 방법의 제한 때문에 우리는 각 셀에 대 한 라이브러리를 생성 하는 낮은 처리량입니다. 반응에 과도 한 뇌관으로 cDNA 도서관 건설 전후 일반적으로 해야 될 정화 뇌관 이합체를 완전히 제거 하려면 두 번. 이 프로세스는 시간이 많이 소요 됩니다. 최근에, 수정 된 프로토콜25 어느 정도이 문제를 해결할 수 있는 보고 되었다. 이 프로토콜에서 셀 전용 바코드 반전 녹음 방송 단계 cDNA 파편을 라벨. 따라서, 각 셀의 cDNA 함께 풀링된 수 있습니다 및 다음 정화, 도서관 건축에 의해 따라. 이 수정 상당히 도서관 건축의 처리량을 증가 시킵니다. 그러나,이 수정된 방법은 덜 민감한 이며 셀 당 적은 유전자를 감지. 또한,이 방식은 3' 감소 읽기 범위와 메서드를 계산 합니다. 따라서, 스마트 seq2 여전히 췌 장 개발에 대 한 가장 적합 한 방법입니다.
다른 종에서 췌 장의 준비 다를 수 있습니다. 인간의 췌에서 독도 이전 프로토콜26,27다음 격리 될 수 있습니다. 그리고 고립 된 독도 단일 셀28 으로 해리 될 수 있습니다이 메서드를 사용 하 여 단일 세포 분석을 수행 합니다. 이 메서드는 포유류 췌 장의 개발, 질병 및 중생의 연구에 널리 적용할 수 있습니다.
공개
저자는 공개 없다.
감사의 말
우리는 생명 과학 컴퓨팅 플랫폼에 대 한 단백질 과학, 베이징 (북경 대학) 및 북경 칭화 센터에 대 한 국립 센터를 감사합니다. 이 작품의 과학 및 기술 (2015CB942800), 국립 자연 과학 재단의 중국 (31521004, 31471358, 및 31522036), 중국과의 북경 칭화 센터에서 C. R.X. 생명 과학에 대 한 자금에 의해 지원 되었다
자료
| Name | Company | Catalog Number | Comments |
| Collagenase P | Roche | 11213873001 | |
| Trypsin-EDTA (0.25 %), phenol red | Thermo Fisher Scientific | 25200114 | |
| Fetal bovine serum (FBS) | Hyclone | SH30071.03 | |
| Dumont #4 Forceps | Roboz | RS-4904 | |
| Dumont #5 Forceps | Roboz | RS-5058 | |
| 30 G BD Needle 1/2" Length | BD | 305106 | |
| Stereo Microscope | Zeiss | Stemi DV4 | |
| Stereo Fluorescence microscope | Zeiss | Stereo Lumar V12 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424R | |
| Polystyrene Round-Bottom Tube with Cell-Strainer Cap | BD-Falcon | 352235 | |
| 96-Well PCR Microplate | Axygen | PCR-96-C | |
| Silicone Sealing Mat | Axygen | AM-96-PCR-RD | |
| Thin Well PCR Tube | Extragene | P-02X8-CF | |
| Cell sorter | BD Biosciences | BD FACSAria | |
| Capillary pipette | Sutter | B100-58-10 | |
| RNaseZap | Ambion | AM9780 | |
| ERCC RNA Spike-In Mix | Life Technologies | 4456740 | |
| Distilled water | Gibco | 10977 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| dNTP mix | New England Biolabs | N0447 | |
| Recombinant RNase Inhibitor | Takara | 2313 | |
| Superscript II reverse transcriptase | Invitrogen | 18064-014 | |
| First-strand buffer (5x) | Invitrogen | 18064-014 | |
| DTT | Invitrogen | 18064-014 | |
| Betaine | Sigma-Aldrich | 107-43-7 | |
| MgCl2 | Sigma-Aldrich | 7786-30-3 | |
| Nuclease-free water | Invitrogen | AM9932 | |
| KAPA HiFi HotStart ReadyMix (2x) | KAPA Biosystems | KK2601 | |
| VAHTS DNA Clean Beads XP beads | Vazyme | N411-03 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| AceQ qPCR SYBR Green Master Mix | Vazyme | Q121-02 | |
| TruePrep DNA Library Prep Kit V2 for Illumina | Vazyme | TD502 | Include 5x TTBL, 5x TTE, 5x TS, 5x TAB, TAE |
| TruePrep Index Kit V3 for Illumina | Vazyme | TD203 | Include 16 N6XX and 24 N8XX |
| High Sensitivity NGS Fragment Analysis Kit | Advanced Analytical Technologies | DNF-474 | |
| 1x HBSS without Ca2+ and Mg2+ | 138 mM NaCl; 5.34 mM KCl 4.17 mM NaHCO3; 0.34 mM Na2HPO4 0.44 mM KH2PO4 | ||
| Isolation buffer | 1 × HBSS containing 10 mM HEPES, 1 mM MgCl2, 5 mM Glucose, pH 7.4 | ||
| FACS buffer | 1 × HBSS containing 15 mM HEPES, 5.6 mM Glucose, 1% FBS, pH 7.4 | ||
| NaCl | Sigma-Aldrich | S5886 | |
| KCl | Sigma-Aldrich | P9541 | |
| NaHCO3 | Sigma-Aldrich | S6297 | |
| Na2HPO4 | Sigma-Aldrich | S5136 | |
| KH2PO4 | Sigma-Aldrich | P5655 | |
| D-(+)-Glucose | Sigma-Aldrich | G5767 | |
| HEPES | Sigma-Aldrich | H4034 | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Oligo-dT30VN primer | 5'-AAGCAGTGGTATCAA CGCAGAGTACT30VN-3' | ||
| TSO | 5'-AAGCAGTGGTATCAAC GCAGAGTACATrGrG+G-3' | ||
| ISPCR primers | 5'-AAGCAGTGGTAT CAACGCAGAGT-3' | ||
| Gapdh Forward primer | 5'-ATGGTGAAGGTC GGTGTGAAC-3' | ||
| Gapdh Reverse primer | 5'-GCCTTGACT GTGCCGTTGAAT-3' | ||
| Ins2 Forward primer | 5'-TGGCTTCTTC TACACACCCA-3' | ||
| Ins2 Reverse primer | 5'-TCTAGTTGCA GTAGTTCTCCA-3' |
참고문헌
- Gu, G., Dubauskaite, J., Melton, D. A. Direct evidence for the pancreatic lineage: NGN3+ cells are islet progenitors and are distinct from duct progenitors. Development. 129 (10), 2447-2457 (2002).
- Oliver-Krasinski, J. M., Stoffers, D. A. On the origin of the beta cell. Genes & Development. 22 (15), 1998-2021 (1998).
- Dor, Y., Brown, J., Martinez, O. I., Melton, D. A. Adult pancreatic beta-cells are formed by self-duplication rather than stem-cell differentiation. Nature. 429 (6987), 41-46 (2004).
- Smukler, S. R., et al. The adult mouse and human pancreas contain rare multipotent stem cells that express insulin. Cell Stem Cell. 8 (3), 281-293 (2011).
- Dorrell, C., et al. Human islets contain four distinct subtypes of beta cells. Nature Communications. 7, 11756 (2016).
- Bader, E., et al. Identification of proliferative and mature beta-cells in the islets of Langerhans. Nature. 535 (7612), 430-434 (2016).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Qiu, W. L., et al. Deciphering pancreatic islet beta cell and alpha cell maturation pathways and characteristic features at the single-cell level. Cell Metabolism. 25 (5), 1194-1205 (2017).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Piccand, J., et al. Pak3 promotes cell cycle exit and differentiation of beta-cells in the embryonic pancreas and is necessary to maintain glucose homeostasis in adult mice. Diabetes. 63 (1), 203-215 (2014).
- Veite-Schmahl, M. J., Regan, D. P., Rivers, A. C., Nowatzke, J. F., Kennedy, M. A. Dissection of the mouse pancreas for histological analysis and metabolic profiling. Journal of Visualized Experiments. (126), (2017).
- Hu, P., Zhang, W., Xin, H., Deng, G. Single cell isolation and analysis. Frontiers in Cell and Developmental Biology. 4, 116 (2016).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), 75 (2017).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), (2013).
- Anders, S., Pyl, P. T., Huber, W. HTSeq--a Python framework to work with high-throughput sequencing data. Bioinformatics. 31 (2), 166-169 (2015).
- Wagner, G. P., Kin, K., Lynch, V. J. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples. Theory in Biosciences. 131 (4), 281-285 (2012).
- Brennecke, P., et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature Methods. 10 (11), 1093-1095 (2013).
- Le, S., Josse, J., Husson, F. FactoMineR: An R package for multivariate analysis. Journal of Statistical Software. 25 (1), (2008).
- Hadley, W. . ggplot2: Elegant graphics for data analysis. , (2009).
- . gplots: Various R Programming Tools for Plotting Data Available from: https://cran.r-project.org/package=gplots (2016)
- Marcheva, B., et al. Disruption of the clock components CLOCK and BMAL1 leads to hypoinsulinaemia and diabetes. Nature. 466 (7306), 627-631 (2010).
- Li, L., et al. Single-cell RNA-seq analysis maps development of human germline cells and gonadal niche interactions. Cell Stem Cell. , (2017).
- Qi, M., et al. Human pancreatic islet isolation: Part I: Digestion and collection of pancreatic tissue. Journal of Visualized Experiments. (27), (2009).
- Qi, M., et al. Human pancreatic islet isolation: Part II: Purification and culture of human islets. Journal of Visualized Experiments. (27), (2009).
- Teo, A. K. K., et al. Single-cell analyses of human islet cells reveal de-differentiation signatures. Cell Death Discovery. 4 (14), (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유