Method Article
Analisi trascrittomica unicellulare delle cellule Endocrine del pancreas del Mouse
In questo articolo
Riepilogo
Descriviamo un metodo per l'isolamento di cellule endocrine da pancreas embrionale, neonatale e postnatale, seguite dal sequenziamento di RNA di cella singola. Questo metodo consente analisi dello sviluppo del pancreas endocrino lignaggio, eterogeneità e trascrittomica dinamiche di cella.
Abstract
Le cellule endocrine del pancreas, che sono raggruppate in isolotti, regolano la stabilità di glucosio nel sangue e il metabolismo energetico. I tipi distinti delle cellule in isolotti, tra cui insulina-secrezione delle cellule β, sono differenziati da progenitori endocrini comuni durante la fase embrionale. Le cellule endocrine immature espandere tramite proliferazione delle cellule e maturare durante un periodo di sviluppo lungo postnatale. Tuttavia, i meccanismi alla base di questi processi non sono chiaramente definiti. Cella singola RNA-sequenziamento è un approccio promettente per la caratterizzazione di popolazioni distinte delle cellule e l'analisi delle cellule lignaggio differenziazione pathways. Qui, descriviamo un metodo per il cella singola RNA-sequenziamento delle cellule β del pancreas isolato dal pancreas embrionale, neonatale e postnatale.
Introduzione
Il pancreas è un organo metabolico vitale nei mammiferi. Il pancreas è costituito da scomparti endocrini ed esocrini. Le cellule endocrine del pancreas, tra cui cellule β che producono insulina e glucagone-producendo le cellule α, raggruppare in cluster degli isolotti di Langerhans e coordinatamente regolano l'omeostasi del glucosio sistemico. Disfunzione delle cellule endocrine si traduce in diabete mellito, che è diventato un problema importante di sanità pubblica in tutto il mondo.
Le cellule endocrine del pancreas sono derivate da Ngn3+ progenitori durante l'embriogenesi1. Più tardi, durante il periodo perinatale, le cellule endocrine proliferano negli isolotti immaturo di forma. Queste cellule immature continuano a sviluppare e gradualmente diventare maturi isolotti, che riccamente vascolarizzati per regolare l'omeostasi del glucosio di anima in adulti2.
Anche se è stato identificato un gruppo di fattori trascrizionali che regolano la differenziazione delle cellule β, la via precisa maturazione delle cellule β è ancora poco chiara. Inoltre, il processo di maturazione delle cellule β coinvolge anche il regolamento di cella numero espansione3,4 e la generazione di eterogeneità cellulare5,6. Tuttavia, i meccanismi di regolamentazione di questi processi non sono stati studiati bene.
Cella singola RNA-sequenziamento è un approccio potente che può profile sottopopolazioni delle cellule e traccia cella lignaggio vie dello sviluppo7. Approfittando di questa tecnologia, la chiave di eventi che si verificano durante lo sviluppo dell'isolotto pancreatico possono essere decifrati al livello unicellulare8. Tra i protocolli di RNA-sequenziamento unicellulare, Smart-seq2 consente la generazione di cDNA integrale con una migliore sensibilità e precisione e l'uso di reagenti normalizzati a più basso costo9. Smart-seq2 richiede circa due giorni per costruire una libreria di cDNA per sequenziamento10.
Qui, vi proponiamo un metodo per l'isolamento delle cellule β fluorescenza-etichettati dal pancreas del feto per adulto Ins1-RFP topi transgenici11, usando la fluorescenza-attivato delle cellule ordinano (FACS), e le prestazioni di trascrittomica analizza presso il livello di singola cellula, utilizzando la tecnologia Smart-seq2 (Figura 1). Questo protocollo può essere esteso per analizzare i trascrittomi di tutti i tipi di cellula endocrina pancreatica negli stati normali, patologici e invecchiamento.
Protocollo
Tutti i metodi descritti qui sono stati approvati dal istituzionale Animal Care e uso Committee (IACUC) dell'Università di Pechino.
1. pancreas isolamento
-
Per gli embrioni E17.5 (giorno embrionale 17,5):
- Stimare il giorno embrionale 0,5 in base al punto di tempo quando viene visualizzato il plug vaginale.
- Sacrificare i topi incinto di somministrazione di CO2 . Spruzzare la pelliccia addominale con alcool al 70%.
- Praticare un'incisione a forma di V con le forbici dalla zona genitale che si estende alle costole. Questo processo viene aperto completamente la cavità addominale.
- Sezionare l'utero fuori della cavità addominale e metterlo in un piatto di 10 cm che contiene PBS freddo.
- Sezionare gli embrioni dall'utero con una pinza con punta sottile sotto un microscopio stereoscopico, rimozione di altri tessuti, quali la placenta e cordone ombelicale. Mettere tutti gli embrioni in un piatto di 10 cm contenente PBS freddo.
- Apertura a strappo cavità addominale degli embrioni e scavare il tessuto viscerale con le pinzette di gomito. Trasferire il tessuto viscerale in un piatto di nero-Fondo di 6 cm contenente PBS freddo.
- Il pancreas si trova in alto a sinistra dell'addome e si attacca al stomaco, milza e duodeno (linea gialla tratteggiata in Figura 2A)12. Staccare i pancreas dal tessuto viscerale usando il forcipe e riunire il tessuto pancreatico in un flaconcino da 20 mL contenente 5 mL di soluzione fredda collagenosi 0,5 mg/mL: P della collagenosi di 0,5 mg/mL in tampone di isolamento (HBSS contenente 10 mM HEPES, 1 mM MgCl2 5 mM glucosio, pH 7.4).
-
Per topi P0-P15 (postnatale giorno 0-15):
- Sacrificio e difficoltà del mouse aderendo le membra di un pezzo di benchtop protezione con nastro adesivo. Spruzzare la pelliccia addominale con alcool al 70%.
- Aprire completamente la cavità addominale come descritto al punto 1.1.3. Estrarre le viscere e sul lato sinistro del mouse. Questa procedura esporrà i lobi splenici, gastrici e duodenali del pancreas. Accuratamente sezionare tutti i lobi del pancreas (Figura 2B)12 e riunire il tessuto pancreatico in un flaconcino da 20 mL contenente 5 mL di soluzione fredda collagenosi 0,5 mg/mL.
-
Per topi P18-P60 (postnatale giorno 18-60):
- Preparare la soluzione di collagenasi. Tenere il ghiaccio fino a che pronto per l'uso.
- Sacrificare il mouse e aprire la cavità addominale, come accennato in precedenza (punto 1.1.2 e 1.1.3).
Nota: Non fanno male al fegato, qualsiasi ferita al fegato ridurrà la pressione di flusso nel dotto biliare e ridurre l'efficienza di aspersione il passaggio seguente. - Rimuovere il xifoideo. Estrarre le viscere e al lato destro del mouse e spingere i lobi mediali di destra e di sinistro del fegato da ogni lato per esporre la cistifellea (freccia bianca nella Figura 2) e il dotto biliare comune.
- Morsetto del duodeno con una coppia di morsetti dei piccoli vasi nella posizione superiore e inferiore, che fiancheggiano il sito dove il dotto biliare entra nel duodeno (Figura 2D).
Nota: Non stringere i lobi gastrici o splenici del pancreas. La soluzione di collagenasi non scorre nel lobo gastrico e splenico del pancreas nel passaggio successivo se bloccato. - Riempire una siringa con 5 mL di soluzione fredda collagenosi 0,5 mg/mL e inserire un ago 30g nella cistifellea. Con attenzione e senza intoppi, manipolare l'ago attraverso il dotto biliare comune.
- Irrorare il pancreas iniettando 1 – 5 mL di soluzione di collagenasi freddo di 0,5 mg/mL, a seconda delle dimensioni del mouse. L'iniezione deve essere lenta e costante per evitare che l'ago scivoli fuori del condotto e per impedire la rottura sotto alta pressione liquido l'intestino. L'iniezione è completa quando il pancreas è espanso completamente.
- Sezionare il pancreas (la linea gialla tratteggiata in Figura 2D) fuori della cavità addominale con forcipe subito dopo aspersione. Posizionare il tessuto in un flaconcino da 20 mL contenente 5 mL di soluzione fredda collagenosi 0,5 mg/mL. Se manipolare più di un mouse a un tempo, irrorare i topi uno per uno e la piscina i tessuti nella soluzione della collagenosi freddo in un flaconcino da 20 mL fino a quando tutti i topi sono stati sezionati.
2. collagenosi digestione e isolamento dell'isolotto
- Mettere il flaconcino da 20 mL contenente il tessuto pancreatico in bagnomaria a 37 ° C ed incubare per 3 min equilibrare la temperatura.
- Agitare delicatamente il tubo per un altro 3-5 min. Se il tessuto pancreatico è completamente gonfio, gradualmente dissocierà pezzetti di tessuto piccolo fino a quando finalmente è disperso in modo uniforme. Il tempo di digestione varia a seconda delle dimensioni del pancreas e l'efficienza di aspersione.
- Filtrare il prodotto digerito attraverso un filtro di nylon 0,25 mm in una nuova provetta da centrifuga 50 mL. Lavare accuratamente il filtro utilizzando una siringa da 20 mL contenente PBS ghiacciata.
- Per E17.5-P15 pancreas, passare al punto 3.1.
- Per pancreas P18-P60, centrifugare a 200 x g per 1 min e scartare il surnatante. Risospendere il tessuto con PBS freddo.
- Versare 5 mL della sospensione del tessuto in un piatto fondo nero di 6 cm. Gli isolotti pancreatici sono strutture bianche piccole, compatte, latte e tessuto acinoso è sciolto e traslucido bianco. Scegli isolotti con una pipetta di 200 μL e trasferirli in una provetta da 1,5 mL contenente una piccola quantità di PBS freddo.
3. tripsina digestione del tessuto pancreatico o isolotti
- Centrifugare la provetta contenente tessuto pancreatico o isolotti a 200 x g per 1 min a 4 ° C e scartare il sopranatante senza disturbare il pellet.
- Risospendere il pellet con 0,25% tripsina-EDTA e incubare a bagnomaria a 37 ° C. Dopo 4 min di incubazione, delicatamente e, occasionalmente, aspirare (pipetta) per 1 min utilizzando 200 μL suggerimenti.
Nota: Per E17.5-P3 pancreas, aggiungere 1 mL di tripsina-EDTA. Per pancreas P4-P15, aggiungere 3 mL di tripsina-EDTA. Per 100-300 isolotti, aggiungere 1 mL di tripsina-EDTA. Regolare il volume di tripsina-EDTA secondo la quantità di tessuto. - Interrompere la digestione aggiungendo 0,4 x volume di freddo siero bovino fetale (FBS) e mescolare dal vortice delicato.
- Centrifuga a 250 x g per 3 min a 4 ° C. Scartare il sopranatante senza disturbare il pellet.
- Risospendere le cellule con buffer di FACS freddo di 200 μL (HBSS contenente 1% FBS, pH 7.4). Trasferire le cellule in una provetta 5 mL FACS.
- Ruota velocemente il tubo di FACS per consentire la sospensione cellulare passare attraverso il filtro per rimuovere i residui di tessuto grande non digerito. La sospensione singola cella nel tubo è ora pronta per l'ordinamento di FACS.
4. single-cell Lysis
-
Preparare il tampone di lisi delle cellule.
Nota: Eseguire tutti gli esperimenti sotto una cappa UV-sterilizzato con flusso laminare. Tutti i tubi, piastre e puntali per pipette dovrebbero essere RNAsi- / dnasi-free. Decontaminare la cappa e le pipette con soluzione via RNasi prima dell'uso.- Scongelare i reagenti sul ghiaccio: dNTP (10 mM), primer oligo-dT (10 μM) ed ERCC stock soluzione (01:20).
- Diluire l'ERCC da 1:5 x 105 con acqua priva di nucleasi.
- Calcolare il volume di tampone di lisi delle cellule necessarie. Aggiungere 0,1 μL di inibitore di RNAsi (40 U/μL), 1,9 μL dello 0,2% (vol/vol) Triton X-100, 1 μL di dNTP, 1 μL di primer oligo-dT e 0,05 μL di ERCC diluito ad un volume finale di 4.05 μL per ogni cella.
- Aliquotare il buffer di lisi delle cellule in provette per PCR 0,2 mL a parete sottile 8-striscia o in piastre da 96 pozzetti. Centrifugare i tubi o piastre per 30 s a 4 ° C.
Nota: Centrifugare le provette per PCR 0,2 mL a parete sottile 8-striscia a 7500 x g e piastre da 96 pozzetti a 800 x g nei passaggi seguenti.
-
Cella singola pennata e Lisi.
- Manualmente pick FACS ordinati celluli Ins1-RFP+ nel buffer di FACS in strip 8 provette per PCR usando una pipetta capillare di 30 – 40 µm o ordinare direttamente le singole cellule Ins1-RFP+ in piastre da 96 pozzetti. Il volume che contiene una singola cella è considerato meno di 0,3 µ l.
Nota: Uso forward scatter altezza (FSC-H) vs trasmettere la zona di dispersione (FSC-A) strategia di gating per discriminazione doppietto, FSC-H vs laterale zona di dispersione (SSC-A) per detriti e fluorescenza gating per l'ordinamento delle cellule (Figura 3A-3C) Ins1-RFP+ . Per il metodo di prelievo, è necessario ordinare le celle di destinazione in una provetta da 1,5 mL contenente 300 μL FACS tampone. La concentrazione finale appropriata è di 5 – 10 cellule/μL. Regolare di conseguenza il volume di accumulo. Per il metodo di raccolta di piastra, ordinare una cella singola in ciascun pozzetto di una piastra a 96 pozzetti seguendo il manuale dello strumento. 7 , 13 - Vortice i tubi o piastre per lisare le cellule e rilasciare il RNA. Centrifugare i tubi o piastre per 30 s a 4 ° C e immediatamente metterli sul ghiaccio.
Nota: Le cellule possono essere conservate a-80 ° C per una settimana.
- Manualmente pick FACS ordinati celluli Ins1-RFP+ nel buffer di FACS in strip 8 provette per PCR usando una pipetta capillare di 30 – 40 µm o ordinare direttamente le singole cellule Ins1-RFP+ in piastre da 96 pozzetti. Il volume che contiene una singola cella è considerato meno di 0,3 µ l.
5. cella singola cDNA amplificazione
-
Trascrizione inversa (RT).
- Scongelare i reagenti RT (tabella 1) sul ghiaccio.
- Incubare i campioni a 72 ° C per 3 min e subito messo i tubi o piastre in ghiaccio per almeno 1 min brevemente Centrifugare i tubi o piastre per 30 s a 4 ° C.
Nota: Utilizzare un termociclatore con un coperchio riscaldata a 105 ° C per tutte le incubazioni. - Preparare la miscela di RT per tutte le reazioni, come descritto nella tabella 1.
- Dispensare 5,7 μL di miscela di RT ad ogni campione da portare il volume a un totale di 10 μL. Delicatamente il mix di vortex e centrifugare per 30 s a 4 ° C.
- Inserire i campioni in un termociclatore e avviare il programma di RT come segue: 42 ° C per 90 min, 10 cicli (50 ° C per 2 min, 42 ° C per 2 min), 70 ° C per 15 min e permanenza a 4 ° C.
-
Pre-amplificazione di PCR.
- Scongelare i reagenti PCR (tabella 2) sul ghiaccio.
- Preparare il mix PCR per tutte le reazioni, come descritto nella tabella 2.
- Dispensare 15 μL di miscela di PCR per ogni campione, che contiene le reazioni del primo-filo. Delicatamente il mix di vortex e centrifugare per 30 s a 4 ° C.
- Inserire i campioni in un termociclatore e avviare il seguente programma PCR: 98 ° C per 3 min, 18 cicli (98 ° C per 20 s, 67 ° C per 15 s, 72 ° C per 6 min), 72 ° C per 5 min e permanenza a 4 ° C.
Nota: Il prodotto PCR può essere conservato a 4 ° C per meno di una settimana o a-20 ° C /-80 ° C fino a 6 mesi.
-
Purificazione di PCR.
- Aggiungere 25 μL di perline per la purificazione di DNA ri-sospensione (1x) per ogni campione dal passaggio precedente e mescolare bene dal vortice. Quindi, ruota velocemente il tubo o piastre a temperatura ambiente per raccogliere il liquido, ma evitare l'insediamento di perline.
Nota: Portare i branelli di purificazione di DNA a temperatura ambiente per 15 min e vortex accuratamente prima dell'uso. - Incubare per 5 min a temperatura ambiente.
- Posto il tubo o la piastra su un appropriato magnetico riposare fino a quando la soluzione è limpida, poi rimuovere delicatamente e scartare il surnatante.
- Aggiungere 200 µ l di etanolo di 80% preparata al momento di lavare le perle mentre nello stand magnetico, incubare per 30 s, quindi rimuovere con cautela e scartare la soluzione di etanolo.
Nota: La soluzione di etanolo di 80% (vol/vol) deve essere preparata ogni volta. - Ripetere il passaggio 5.3.4 per un totale di due lavaggi.
- Con attenzione rimuovere ed eliminare il rimanente soluzione etanolica e asciugare all'aria le perline mentre il tubo o piatto è sul supporto magnetico.
Nota: Evitare sovra le perline per garantire l'efficienza massima di eluizione. - Aggiungere 11 μL di acqua priva di nucleasi per eluire il DNA bersaglio dalle perline e mescolare bene dal vortice. Poi, rapidamente girare il tubo o la piastra e metterlo su un supporto magnetico fino a quando la soluzione è limpida. Trasferire 10 μL del campione in una provetta PCR nuova.
Nota: In presenza di dimeri dopo un singolo ciclo di purificazione, basata sulla rilevazione del cDNA dimensioni distribuzione, purificare nuovamente per rimuovere completamente i dimeri. I dimeri possono influenzare il calcolo della resa del cDNA, se autorizzati a rimanere nel campione.
- Aggiungere 25 μL di perline per la purificazione di DNA ri-sospensione (1x) per ogni campione dal passaggio precedente e mescolare bene dal vortice. Quindi, ruota velocemente il tubo o piastre a temperatura ambiente per raccogliere il liquido, ma evitare l'insediamento di perline.
-
Controllo di qualità dei cDNA.
- Scegliere casualmente campioni per rilevare la resa totale di cDNA utilizzando un fluorimetro.
- Valutare i livelli di espressione del gene marcatore di PCR in tempo reale (qPCR) (Figura 4E). Rimuovere 1 μL del campione per diluire 40 volte ed eseguire qPCR utilizzando una piastra 384 pozzetti. Preparare la miscela di qPCR come descritto nella tabella 3. Condizioni di ciclismo: 95 ° C per 10 min, 45 cicli (95 ° C per 10 s, 60 ° C per 15 s, 72 ° C per 15 s).
- Scegliere casualmente campioni per rilevare la distribuzione delle dimensioni utilizzando uno strumento parallelo elettroforesi capillare.
6. costruzione della libreria di cDNA
-
Reazione di Tagmentation della transposase Tn5.
- Rilevare la resa totale di cDNA di cellule qPCR-selezionato dal punto 5.4.2 utilizzando un fluorimetro. Uso 2 ng di cDNA come materiale di partenza.
- Scongelare i reagenti di reazione di tagmentation (tabella 4) sul ghiaccio.
- Preparare la reazione di tagmentation in una provetta PCR 0,2 mL a parete sottile 8-striscia, come descritto nella tabella 4e mescolate con cura dal vortice. Poi, rapidamente girare giù la soluzione a temperatura ambiente.
- Incubare i campioni a 55 ° C per 10 min e tenere a 4 ° C.
- Immediatamente aggiungere 2 μL di 5 x TS ad ogni campione contenente il DNA per arrestare la reazione di tagmented. Mescolare con cura dal vortice e quindi rallentare rapidamente la soluzione a temperatura ambiente.
- Incubare la miscela per 5 min a temperatura ambiente. Il DNA deve essere elaborato per l'arricchimento finale PCR immediatamente.
-
Amplificazione dei frammenti adattatore-legati.
- Scongelare i reagenti PCR (tabella 5) sul ghiaccio.
- Preparare la miscela PCR di arricchimento, come descritto nella tabella 5e mescolate con cura dal vortice. Poi, rapidamente girare giù la soluzione a temperatura ambiente.
- Eseguire la PCR utilizzando il seguente programma: 72 ° C per 10 min, 98 ° C per 30 s, 8 cicli (98 ° C per 15 s, 60 ° C per 30 s, 72 ° C per 3 min), 72 ° C per 5 min e permanenza a 4 ° C.
Nota: Il numero di cicli dipende la quantità di DNA biblioteca previsto.
-
Purificazione di PCR con selezione dimensione.
- Aggiungere 14 μL di perline per la purificazione di DNA ri-sospensione (0,7 x) per ogni campione dal passaggio precedente e mescolare bene dal vortice. Poi, rapidamente girare il tubo a temperatura ambiente per raccogliere il liquido, ma evitare l'insediamento di perline.
Nota: Portare i branelli di purificazione di DNA a temperatura ambiente per 15 min e vortex accuratamente prima dell'uso. - Incubare per 5 min a temperatura ambiente.
- Collocare la provetta su un appropriato supporto magnetico fino a quando la soluzione è limpida, attentamente trasferire il surnatante per una nuova striscia di tubo e scartare la striscia tubo precedente.
- Aggiungere 3 μL di ri-sospensione perline di purificazione di DNA (0.15 x) per ogni campione nella striscia tubo e mescolare bene dal vortice. Poi, rapidamente girare il tubo a temperatura ambiente.
- Incubare per 5 min a temperatura ambiente.
- Posto il tubo o la piastra su un appropriato magnetico riposare fino a quando la soluzione è limpida, poi rimuovere delicatamente e scartare il surnatante.
- Aggiungere 200 µ l di etanolo di 80% preparata al momento di lavare le perle mentre nello stand magnetico, incubare per 30 s, quindi rimuovere con cautela e scartare la soluzione di etanolo.
Nota: La soluzione di etanolo di 80% (vol/vol) deve essere preparata ogni volta. - Ripetere il passaggio 6.3.7 per un totale di due lavaggi.
- Con attenzione rimuovere ed eliminare il rimanente soluzione etanolica e asciugare all'aria le perline mentre il tubo o piatto è sul supporto magnetico.
Nota: Evitare sovra le perline per garantire l'efficienza massima di eluizione. - Aggiungere 11 μL di acqua priva di nucleasi per eluire il DNA bersaglio dalle perline e mescolare bene dal vortice. Poi, rapidamente girare il tubo o la piastra e metterlo su un supporto magnetico fino a quando la soluzione è limpida. Trasferire 10 μL del campione per una nuova striscia di tubo.
- Aggiungere 14 μL di perline per la purificazione di DNA ri-sospensione (0,7 x) per ogni campione dal passaggio precedente e mescolare bene dal vortice. Poi, rapidamente girare il tubo a temperatura ambiente per raccogliere il liquido, ma evitare l'insediamento di perline.
-
Controllo di qualità della libreria di cDNA finale.
- Misurare la concentrazione di ogni libreria utilizzando un fluorimetro e controllare la distribuzione delle dimensioni utilizzando uno strumento parallelo elettroforesi capillare.
Nota: Il rendimento del DNA è in genere tra 15-25 ng per ogni libreria. I frammenti che vanno da 250 bp a 450 bp sarà osservato. Se dimeri rimangono dopo la purificazione, come confermato dal controllo della distribuzione di dimensione, purificare con 1 x perle di purificazione del DNA una volta di più.
- Misurare la concentrazione di ogni libreria utilizzando un fluorimetro e controllare la distribuzione delle dimensioni utilizzando uno strumento parallelo elettroforesi capillare.
-
Pool di biblioteca
- Base alla dimensione del frammento approssimativo, piscina uguali quantità di DNA da ogni campione, assicurando che nessuno di loro contengono le stesse combinazioni di schede N6XX e N8XX.
7. DNA Sequencing
- Librerie di codice a barre in base alla sequenziazione di single-end bp 51 utilizzando un sistema di sequenziamento ad alte prestazioni. Eseguire il sequenziamento seguendo il protocollo del produttore. La profondità di sequenziamento di ogni cella è circa 1 milione si legge in media8, almeno 0,5 milioni letture al cellulare14.
8. analisi bioinformatica
-
Valutazione della qualità di sequenziamento e allineamento.
- Valutare la qualità di letture in sequenza utilizzando FastQC (v0.11.3)15 con i seguenti parametri: "fastqc - estratto -o output_dir input_fastq".
- Unire il genoma del topo con le sequenze ERCC utilizzando il comando "cat mm10.fa ERCC.fa > mm10_ERCC.fa".
- Costruire bowtie2 indice16 (v 2.2.5) con i seguenti parametri: "bowtie2-build mm10_ERCC.fa mm10_ERCC".
- Allineare le letture utilizzando tophat2 (v. 2.1.0)17 con i seguenti parametri: "tophat2 -o output_dir -G gene.gtf - trascrittoma-Indice trans_index mm10_ERCC input_fastq".
-
Quantificare i livelli di espressione genica.
- Conteggio mappato legge per ogni gene utilizzando HTSeq (v 0.6.0)18 con i seguenti parametri: cm htseq-Conte - f bam - r pos -s no - un 30 gene.gtf accepted_hits.bam > read_count.txt '.
- Normalizzare i livelli di espressione genica di trascrizioni per milione (TPM)19.
-
Controllo di qualità delle cellule.
- Escludere le celle con meno di 0,5 milioni di letture mappate o meno di 4.000 geni (TPM > 1).
Nota: il criterio di esclusione dipende da tipi di cellule e profondità di sequenza. - Conservare le cellule che esprimono gli indicatori endocrini (ad es., Ins1 per le cellule β, Gcg per le cellule α) ed escludere le cellule che esprimono gli indicatori non endocrini (ad es., Spi1 per leucociti).
- Escludere le celle con meno di 0,5 milioni di letture mappate o meno di 4.000 geni (TPM > 1).
-
Analisi delle componenti principali (PCA)
- Identificare i geni altamente variabile secondo ERCC spike-ins, come descritto in precedenza20.
- Eseguire PCA utilizzando la funzione "APC" nel R pacchetto FactoMineR (v1.31.4)21, con log2(TPM + 0.1) di geni altamente variabile.
- Visualizzare i risultati PCA con ggplot2 (v. 2.0.0)22.
-
Hierarchical clustering.
- Identificare i geni con i più alti carichi di componente principale (PC) utilizzando la funzione "dimdesc" FactoMineR (v1.31.4)21.
- Eseguire il clustering gerarchico utilizzando la funzione "heatmap.2" nel R pacchetto gplots (v 3.0.1)23, con log2 (TPM + 1) valori relativi di PC di elevato carico di geni.
Risultati
Pancreas sono stati sezionati da topi embrionali, neonatali e postnatali (Figura 2A e 2B). Per topi oltre postnatale giorno 18 anni di età, l'effetto digestivo dipende dal grado di aspersione; di conseguenza, l'iniezione è il passo più importante per l'isolamento dell'isolotto (Figura 2-2E e tabella 6). Come era possibile riempire il pancreas durante questo passaggio, è stata iniettata tanto collagenosi. Il pancreas completamente gonfio è mostrato in Figura 2D. Se la perfusione non è successo (Figura 2E), ma il campione è prezioso, il pancreas può essere strappato a pezzettini per sufficiente digestione più tardi.
Dopo aspersione, il tessuto pancreatico è stato digerito in piccoli pezzi per rilasciare isolotti (Figura 2F). Per accorciare i tempi di smistamento di FACS, abbiamo arricchito le cellule endocrine scegliendo gli isolotti in anticipo. La dimensione degli isolotti può variare a seconda dell'età del mouse e l'intensità di digestione. Occasionalmente, gli isolotti non sono di forma arrotondata. Isolotti dovrebbero essere selezionati secondo colore e stato compatto (Figura 2 e tabella 6). Se il ceppo di topi transgenici ha un gene reporter, come GFP o RFP per cellule endocrine, gli isolotti potrebbero inoltre essere raccolti sotto un microscopio a fluorescenza.
Le cellule Ins1-RFP+ sono state purificate dal FACS ordinamento (Figura 3A-3C), e quindi singole cellule sono state raccolte utilizzando una pipetta capillare per cella singola RNA-seq (Figura 3D). CDNA amplificato con successo deve avere una lunghezza superiore a 500 bp e arricchita da 1,5 kb a 2 kb. Inoltre, c'è solitamente un arricchimento di bp di 500-600 di cDNA osservato nelle cellule Ins1-RFP+ , che possono rappresentare le trascrizioni di insulina (Figura 4A). Tuttavia, ci sono stati alcune situazioni anormali osservati10 (tabella 6). Ad esempio, i frammenti di cDNA near 100 bp sono dimeri dell'iniettore (fig. 4B), che sono di solito causati dai primer del eccessivo, che deve essere rimosso ripetendo il passaggio di purificazione del DNA. L'esistenza di dimeri può influenzare i calcoli dei rendimenti di cDNA totale, che sono usati per la costruzione della libreria seguente. I frammenti di cDNA tra 100 bp e 500 bp di solito rappresentano degradata cDNA (Figura 4), che è causato da problemi di stato o reagente di cattiva cella, come contaminazione RNasi. In questa condizione, dobbiamo identificare la causa della degradazione del DNA ed escludere il disturbo. Per esempio, per garantire lo stato buon cellulare, tessuti dovrebbero essere digeriti e cellule devono essere ordinate rapidamente e delicatamente e operazioni devono essere eseguite con cautela per evitare qualsiasi tipo di inquinanti.
Dopo la purificazione delle librerie di cDNA per il sequenziamento, abbiamo ottenuto frammenti di cDNA di diverse dimensioni seguendo le istruzioni per l'aggiunta di diversi rapporti di perline di purificazione di DNA al campione. Ad esempio, possiamo ottenere cDNA che vanno da 250 bp 450 bp da aggiungendo x 0,7 e 0,15 x perline di purificazione di DNA al primo e secondo round di purificazione, rispettivamente (Figura 4). Questo passaggio ha un alto tasso di successo. Tuttavia, se ci sono frammenti di circa 100 bp, è suggerito per purificare le librerie nuovo con 1 x perline di purificazione di DNA per rimuovere dimeri (tabella 6). Unremoved dimeri si inclina la quantificazione di DNA e influenzeranno il campione pool di risultati, che si traduce nell'acquisizione dati irregolari da ciascun campione.
Abbiamo analizzato i dati di sequenziamento con approcci di bioinformatica (Figura 5A). I punteggi di qualità di sequenziamento dovrebbero essere maggiori di 30 durante la sequenziazione di valutazione della qualità (figura 5B). Dopo l'allineamento, 80-90% di letture dovrebbe essere mappato il genoma di riferimento. Per ottenere le cellule di alta qualità per analisi successive, abbiamo escluso cellule con meno di 0,5 milioni di letture mappate o con meno di 4.000 rilevato geni (Figura 5 e 5D). Dopo PCA e clustering gerarchico, abbiamo caratterizzato i diversi gruppi di cellule e identificato i geni che sono stati espressi eterogeneo in diversi gruppi8.

Figura 1: schema generale per cella singola RNA-seq del pancreas endocrino delle cellule di topo. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Pancreas dissezione, perfusione e isolotto picking. (A) dissezione del tessuto pancreatico embrionale (inferiore) da un embrione E17.5 (superiore). La linea gialla tratteggiata delimita il tessuto pancreatico. Barra della scala = 1.000 µm. (B) dissezione del tessuto pancreatico postnatale (a destra) da un mouse di P10 (a sinistra). Barra della scala = 1.000 µm. (C-D), il tessuto pancreatico di un mouse P60 prima (C) e dopo (D) aspersione. La linea gialla tratteggiata delimita il tessuto pancreatico. Freccia bianca indica la cistifellea. (E) il tessuto pancreatico parzialmente irrorato del mouse P60. La freccia rossa indica la zona ben irrorata del pancreas. Le frecce blu indica le aree scarsamente irrorate del pancreas. (F) il pancreas tessuto prima (in alto) e dopo digestione della collagenosi (inferiore). (G) frecce puntano agli isolotti che sono stati rilasciati dal tessuto pancreatico collagenosi digerito. Barra della scala = 200 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: raccogliere manualmente le celle con una pipetta capillare di 30 – 40 µm sotto un microscopio. (A-C) Graduale FACS gating per ordinamento celle Ins1-RFP+ . (D) la brillante cerchi indicano le cellule e il tubo è una pipetta capillare. Scegli le cellule con morfologia migliore (freccia) e ignorare le cellule cluster (punta di freccia). Barra della scala = 200 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Rilevamento di qualità di cDNA e libreria di distribuzione di dimensione delle cellule Ins1-RFP+ da uno strumento parallelo elettroforesi capillare. (A), il rappresentante risultato di cDNA con successo pre-amplificato. Normalmente, il profilo di cDNA dovrebbe essere superiore a 500 bp con un picco di ~1.5–2 kb. (B) il risultato rappresentativo del cDNA pre-amplificato con un picco di dimero di primer. Il picco di dimero di primer è di circa 100 BP. (C), il profilo di cDNA pre-amplificato con frammenti tra 100 bp e 500 bp indica la possibilità di degradazione di cDNA. (D) la distribuzione delle dimensioni del cDNA biblioteca comprese tra 250 bp e 450 bp seguenti, la procedura di purificazione citata in questo protocollo. Livelli (E) l'espressione di Ins2 in librerie di cDNA di qPCR (a sinistra) e relativa dati di sequenziamento (a destra). Il x-axes rappresentano distinte 8 campioni di singola cellula. Rappresenta l'asse y (a sinistra) ΔCt normalizzato rispetto Gapdh (CtGapdh- CtIns2 + 6) e l'asse y (a destra) rappresenta il livello di espressione normalizzato di Ins2 relativo Gapdh (registro2(TPM Ins2 / TPMGapdh)). Clicca qui per visualizzare una versione più grande di questa figura.
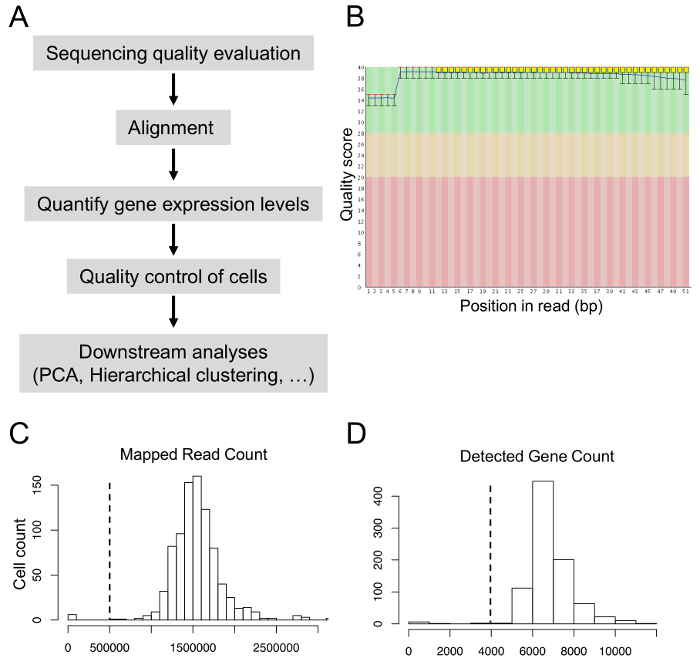
Figura 5: analisi bioinformatica dei dati di cella singola transcriptome. (A) Pipeline di analisi bioinformatica. (B) sequenziamento punteggi di qualità attraverso tutte le basi di letture. (C) distribuzione del conteggio lettura mappata. (D) distribuzione del conteggio di gene riconosciuto. Clicca qui per visualizzare una versione più grande di questa figura.
| Componente | Volume (µ l) |
| Trascrittasi inversa (200 U / µ l) | 0,5 |
| Inibitore di RNAsi | 0.25 |
| Buffer di primo-filo (5x) | 2 |
| DTT (100 mM) | 0,5 |
| Betaina (5 M) | 2 |
| MgCl2 (1m) | 0,06 |
| TSO (100 ΜM) | 0.1 |
| Acqua priva di nucleasi | 0,29 |
| Totale | 5.7 |
Tabella 1: Reagente RT miscelare i componenti in una reazione di 5,7 µ l per ogni campione.
| Componente | Volume (µ l) |
| Reazione del primo-filo | 10 |
| DNA polimerasi (2x ReadyMix) | 12.5 |
| È gli iniettori di PCR (10 µM) | 0.25 |
| Acqua priva di nucleasi | 2.25 |
| Totale | 25 |
Tabella 2: Reagente di pre-amplificazione di PCR miscelare i componenti in una reazione di 25 µ l per ogni campione
| Componente | Volume (µ l) |
| SYBR Green Master Mix | 5 |
| Primer (5 µM) | 0,5 |
| Acqua priva di nucleasi | 2.5 |
| cDNA | 2 |
| Totale | 10 |
Tabella 3: qPCR reagente miscelare i componenti in una reazione di µ l 10 utilizzando una piastra 384 pozzetti standard.
| Componente | Volume (µ l) |
| 5 x TTBL | 1.6 |
| 2 ng cDNA | Variabile |
| ddH2O | Variabile |
| TTE Mix V5 | 2 |
| Totale | 8 |
Tabella 4: Tagmentation reagente miscelare i componenti in una reazione di 8 µ l per ogni campione.
| Componente | Volume (µ l) |
| ddH2O | 1.6 |
| Prodotto del passaggio precedente | 10 |
| 5 scheda x | 4 |
| N6XX | 2 |
| N8XX | 2 |
| TAE | 0.4 |
| Totale | 20 |
Tabella 5: Arricchimento PCR componenti della miscela reagente in una reazione di 20 µ l per ogni campione.
| Passo | Problema | Possibilità | Soluzione |
| 1.3.4 | Scivolamento dei morsetti | La superficie esterna dell'intestino è bagnata causato dall'esistenza della piccola quantità di leakness di collegenase e del liquido interstiziale | Swap delicatamente la parete del duodeno e altre pareti dell'organo nella cavità addominale con tamponi di cotone |
| 1.3.6 | Scoppio dell'intestino | Pressione del liquido è troppo alto nell'intestino | Rallentare la velocità di iniezione |
| 2.6 | Isolotti di aderire al tessuto exocrine quando picking isolotti | Digestione insufficent | Prolungare la durata della digestione e scuotendo la forza |
| 5.3 | Il rendimento di cDNA è basso dopo amplificazione mediante PCR | Le cellule sono in cattive condizioni | Mantenere le cellule fresche e in buone condizioni |
| 5.4 o 6.4 | Dimeri dell'iniettore può essere visto | Iniettori eccessiva | Purificare il cDNA ancora una volta con i branelli di purificazione del DNA (0.8:1 o rapporto 1:1) |
| 5.4 | CDNA degradato dopo amplificazione mediante PCR | Scarsa qualità delle cellule o reagenti contaminati | Mantenere le cellule in buone condizioni o cambiare i reagenti |
| 6.3 | La quantità di DNA è bassa dopo la costruzione della libreria | Troppo pochi cicli di PCR o la qualità di cDNA non è buona | Aumentare il numero di cicli o garantire qualità di cDNA prima della costruzione della libreria |
Tabella 6: risoluzione dei problemi.
Discussione
In questo protocollo, abbiamo dimostrato un metodo efficace e facile da usare per studiare i profili di espressione di singole cellule delle cellule β del pancreas. Questo metodo potrebbe essere utilizzato per isolare le cellule endocrine da pancreas embrionale, neonatale e postnatale e per eseguire analisi trascrittomica unicellulare.
Il punto più critico è l'isolamento di cellule β singolo in buone condizioni. Pancreas completamente irrorato rispondono meglio alla successiva digestione. Insufficiente perfusione, che si presenta solitamente nel pancreas dorsale, si tradurrà in un rendimento basso isolotto. Dopo l'aspersione, il tempo di digestione e scuotendo intensità richiedono particolare attenzione. Sovra-digestione, risultante dai tempi di incubazione lunga e vigorosa agitazione, può rompere le isolette in pezzi. Digestione insufficiente non separerà completamente gli isolotti dai tessuti adiacenti. Dopo la digestione del pancreas, le isolette sono state purificate tramite brucatura a mano anziché tramite centrifugazione in gradiente di densità. Anche se la centrifugazione in gradiente di densità possa arricchire gli isolotti, molti piccoli isolotti o isolotti collegati con tessuto acinoso erroneamente vengono scartati. Cercare di evitare di raccogliere tessuto acinoso, che può causare la digestione della tripsina inefficiente e ridurre la purezza delle cellule β.
Replicati biologici indipendenti sono necessari per distinguere gli effetti biologici di variabilità e batch. Se viene replicato due biologico hanno un andamento simile nell'APC e includono sottogruppi simili in clustering di risultati, questi campioni potrebbero rivelare variabilità biologica affidabile. In caso contrario, ulteriori repliche biologiche sono richiesti per confermare i risultati. Per un organo metabolico, come il pancreas, lo stato delle cellule connesso con l' orologio circadiano24 può rappresentare le differenze batch. Per risolvere questo problema, si consiglia di raccogliere tutti i campioni allo stesso tempo del giorno.
La limitazione di questo approccio è bassa velocità effettiva perché dobbiamo costruire una libreria per ogni cella. A causa di eccessiva iniettori nella reazione, il cDNA prima e dopo la costruzione della libreria generalmente dovrebbe essere purificato due volte per rimuovere completamente i dimeri di primer. Questi processi sono che richiede tempo. Recentemente, un protocollo modificato25 è stato segnalato che potrebbe risolvere questo problema in una certa misura. In questo protocollo, il cellula-specifico codice a barre etichette frammenti di cDNA durante la fase di trascrizione inversa. Di conseguenza, il cDNA di ogni cella possono essere raggruppato e poi essere purificato, seguita dalla costruzione della libreria. Questa modifica aumenta significativamente la velocità effettiva della costruzione della libreria. Tuttavia, questo metodo modificato è meno sensibile e rileva meno geni per cella. Inoltre, questo metodo è una 3' metodo con ridotta copertura saperne di conteggio. Pertanto, Smart-seq2 è ancora il metodo più adatto per lo sviluppo del pancreas.
La preparazione del pancreas da altre specie potrebbe essere diversa. Per pancreas umano, gli isolotti possono essere isolati seguendo precedenti protocolli26,27. Quindi, gli isolotti isolati possono essere dissociati in cellule singole28 ed eseguire analisi unicellulare usando questo metodo. Questo metodo può essere ampiamente applicato alla ricerca di mammiferi sviluppo pancreatico, malattie e rigenerazione.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo il centro nazionale per Protein Sciences, Beijing (Peking University) e il centro di Tsinghua di Pechino per la piattaforma di Computing di Life Science. Questo lavoro è stato supportato dal Ministero della scienza e tecnologia della Cina (2015CB942800), National Foundation Natural Science of China (31521004, 31471358 e 31522036) e finanziamenti da Tsinghua di Pechino centro per le scienze della vita a C.-chrysantha
Materiali
| Name | Company | Catalog Number | Comments |
| Collagenase P | Roche | 11213873001 | |
| Trypsin-EDTA (0.25 %), phenol red | Thermo Fisher Scientific | 25200114 | |
| Fetal bovine serum (FBS) | Hyclone | SH30071.03 | |
| Dumont #4 Forceps | Roboz | RS-4904 | |
| Dumont #5 Forceps | Roboz | RS-5058 | |
| 30 G BD Needle 1/2" Length | BD | 305106 | |
| Stereo Microscope | Zeiss | Stemi DV4 | |
| Stereo Fluorescence microscope | Zeiss | Stereo Lumar V12 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424R | |
| Polystyrene Round-Bottom Tube with Cell-Strainer Cap | BD-Falcon | 352235 | |
| 96-Well PCR Microplate | Axygen | PCR-96-C | |
| Silicone Sealing Mat | Axygen | AM-96-PCR-RD | |
| Thin Well PCR Tube | Extragene | P-02X8-CF | |
| Cell sorter | BD Biosciences | BD FACSAria | |
| Capillary pipette | Sutter | B100-58-10 | |
| RNaseZap | Ambion | AM9780 | |
| ERCC RNA Spike-In Mix | Life Technologies | 4456740 | |
| Distilled water | Gibco | 10977 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| dNTP mix | New England Biolabs | N0447 | |
| Recombinant RNase Inhibitor | Takara | 2313 | |
| Superscript II reverse transcriptase | Invitrogen | 18064-014 | |
| First-strand buffer (5x) | Invitrogen | 18064-014 | |
| DTT | Invitrogen | 18064-014 | |
| Betaine | Sigma-Aldrich | 107-43-7 | |
| MgCl2 | Sigma-Aldrich | 7786-30-3 | |
| Nuclease-free water | Invitrogen | AM9932 | |
| KAPA HiFi HotStart ReadyMix (2x) | KAPA Biosystems | KK2601 | |
| VAHTS DNA Clean Beads XP beads | Vazyme | N411-03 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| AceQ qPCR SYBR Green Master Mix | Vazyme | Q121-02 | |
| TruePrep DNA Library Prep Kit V2 for Illumina | Vazyme | TD502 | Include 5x TTBL, 5x TTE, 5x TS, 5x TAB, TAE |
| TruePrep Index Kit V3 for Illumina | Vazyme | TD203 | Include 16 N6XX and 24 N8XX |
| High Sensitivity NGS Fragment Analysis Kit | Advanced Analytical Technologies | DNF-474 | |
| 1x HBSS without Ca2+ and Mg2+ | 138 mM NaCl; 5.34 mM KCl 4.17 mM NaHCO3; 0.34 mM Na2HPO4 0.44 mM KH2PO4 | ||
| Isolation buffer | 1 × HBSS containing 10 mM HEPES, 1 mM MgCl2, 5 mM Glucose, pH 7.4 | ||
| FACS buffer | 1 × HBSS containing 15 mM HEPES, 5.6 mM Glucose, 1% FBS, pH 7.4 | ||
| NaCl | Sigma-Aldrich | S5886 | |
| KCl | Sigma-Aldrich | P9541 | |
| NaHCO3 | Sigma-Aldrich | S6297 | |
| Na2HPO4 | Sigma-Aldrich | S5136 | |
| KH2PO4 | Sigma-Aldrich | P5655 | |
| D-(+)-Glucose | Sigma-Aldrich | G5767 | |
| HEPES | Sigma-Aldrich | H4034 | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Oligo-dT30VN primer | 5'-AAGCAGTGGTATCAA CGCAGAGTACT30VN-3' | ||
| TSO | 5'-AAGCAGTGGTATCAAC GCAGAGTACATrGrG+G-3' | ||
| ISPCR primers | 5'-AAGCAGTGGTAT CAACGCAGAGT-3' | ||
| Gapdh Forward primer | 5'-ATGGTGAAGGTC GGTGTGAAC-3' | ||
| Gapdh Reverse primer | 5'-GCCTTGACT GTGCCGTTGAAT-3' | ||
| Ins2 Forward primer | 5'-TGGCTTCTTC TACACACCCA-3' | ||
| Ins2 Reverse primer | 5'-TCTAGTTGCA GTAGTTCTCCA-3' |
Riferimenti
- Gu, G., Dubauskaite, J., Melton, D. A. Direct evidence for the pancreatic lineage: NGN3+ cells are islet progenitors and are distinct from duct progenitors. Development. 129 (10), 2447-2457 (2002).
- Oliver-Krasinski, J. M., Stoffers, D. A. On the origin of the beta cell. Genes & Development. 22 (15), 1998-2021 (1998).
- Dor, Y., Brown, J., Martinez, O. I., Melton, D. A. Adult pancreatic beta-cells are formed by self-duplication rather than stem-cell differentiation. Nature. 429 (6987), 41-46 (2004).
- Smukler, S. R., et al. The adult mouse and human pancreas contain rare multipotent stem cells that express insulin. Cell Stem Cell. 8 (3), 281-293 (2011).
- Dorrell, C., et al. Human islets contain four distinct subtypes of beta cells. Nature Communications. 7, 11756(2016).
- Bader, E., et al. Identification of proliferative and mature beta-cells in the islets of Langerhans. Nature. 535 (7612), 430-434 (2016).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Qiu, W. L., et al. Deciphering pancreatic islet beta cell and alpha cell maturation pathways and characteristic features at the single-cell level. Cell Metabolism. 25 (5), 1194-1205 (2017).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Piccand, J., et al. Pak3 promotes cell cycle exit and differentiation of beta-cells in the embryonic pancreas and is necessary to maintain glucose homeostasis in adult mice. Diabetes. 63 (1), 203-215 (2014).
- Veite-Schmahl, M. J., Regan, D. P., Rivers, A. C., Nowatzke, J. F., Kennedy, M. A. Dissection of the mouse pancreas for histological analysis and metabolic profiling. Journal of Visualized Experiments. (126), (2017).
- Hu, P., Zhang, W., Xin, H., Deng, G. Single cell isolation and analysis. Frontiers in Cell and Developmental Biology. 4, 116(2016).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), 75(2017).
- Andrews, S. FastQC: A quality control tool for high throughput sequence data. , Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), (2013).
- Anders, S., Pyl, P. T., Huber, W. HTSeq--a Python framework to work with high-throughput sequencing data. Bioinformatics. 31 (2), Oxford, England. 166-169 (2015).
- Wagner, G. P., Kin, K., Lynch, V. J. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples. Theory in Biosciences. 131 (4), 281-285 (2012).
- Brennecke, P., et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature Methods. 10 (11), 1093-1095 (2013).
- Le, S., Josse, J., Husson, F. FactoMineR: An R package for multivariate analysis. Journal of Statistical Software. 25 (1), (2008).
- Hadley, W. ggplot2: Elegant graphics for data analysis. , Springer Science & Business Media. (2009).
- Warnes, G., Bolker, B., Bonebakker, L., Gentleman, R., Huber, W., Liaw, A., Lumley, T., Mächler, M., Magnusson, A., Möller, S. gplots: Various R Programming Tools for Plotting Data. , Available from: https://cran.r-project.org/package=gplots (2016).
- Marcheva, B., et al. Disruption of the clock components CLOCK and BMAL1 leads to hypoinsulinaemia and diabetes. Nature. 466 (7306), 627-631 (2010).
- Li, L., et al. Single-cell RNA-seq analysis maps development of human germline cells and gonadal niche interactions. Cell Stem Cell. , (2017).
- Qi, M., et al. Human pancreatic islet isolation: Part I: Digestion and collection of pancreatic tissue. Journal of Visualized Experiments. (27), (2009).
- Qi, M., et al. Human pancreatic islet isolation: Part II: Purification and culture of human islets. Journal of Visualized Experiments. (27), (2009).
- Teo, A. K. K., et al. Single-cell analyses of human islet cells reveal de-differentiation signatures. Cell Death Discovery. 4 (14), (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Tutti i diritti riservati
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.