Method Article
Analyse transcriptomique unicellulaire de cellules endocrines pancréatiques de souris
Dans cet article
Résumé
Nous décrivons une méthode pour isoler des cellules endocrines du pancréas embryonnaires, néonatals et postnatals suivies par séquençage de RNA unicellulaires. Cette méthode permet des analyses du développement de la lignée endocrine du pancréas, de la cellule hétérogénéité et transcriptomique dynamique.
Résumé
Les cellules endocrines du pancréas, qui sont regroupées en îlots, réglementent la stabilité de glucose de sang et le métabolisme énergétique. Les types cellulaires distincts dans les îlots, y compris les cellules sécrétrices d’insuline β, sont distinguent des progéniteurs endocriniens communes durant le stade embryonnaire. Cellules endocrines immatures étendre via la prolifération cellulaire et mûrissent durant une période de développement longtemps après la naissance. Cependant, les mécanismes qui sous-tendent ces processus ne sont pas clairement définis. Unicellulaire RNA-sequencing est une approche prometteuse pour la caractérisation de populations cellulaires distincts et voies de différenciation lignée suivi cellulaire. Nous décrivons ici une méthode pour la cellule unique RNA-séquençage des cellules β du pancréas isolé du pancréas embryonnaires, néonatals et postnatals.
Introduction
Le pancréas est un organe vital métabolique chez les mammifères. Le pancréas est composé de compartiments endocrines et exocrines. Les cellules endocrines du pancréas, y compris les cellules productrices d’insuline β et cellules productrices de glucagon α, cluster ensemble dans les îlots de Langerhans et coordonnée de réguler l’homéostasie du glucose systémique. La dysfonction des cellules endocrines entraîne dans mellitus de diabète, qui est devenue un enjeu majeur de santé publique dans le monde entier.
Les cellules endocrines du pancréas sont dérivés de Ngn3+ progéniteurs au cours de l’embryogenèse1. Plus tard, durant la période périnatale, les cellules endocrines prolifèrent aux îlots immatures de forme. Ces cellules immatures continuent de développer et de devenir progressivement des îlots matures, qui deviennent richement vascularisés pour réguler l’homéostasie du glucose de sang dans les adultes,2.
Bien qu’un groupe de facteurs de transcriptionnelles a été identifié qui régulent la différenciation des cellules β, la voie précise de la maturation des cellules β est encore incertaine. En outre, le processus de maturation des cellules β comporte également la régulation de la cellule numéro expansion3,4 et la génération de l’hétérogénéité cellulaire5,6. Cependant, les mécanismes de régulation de ces processus n’ont pas été bien étudiées.
Unicellulaire RNA-sequencing est une approche puissante qui peut profil sous-populations de cellules et tracer cell lineage voies développementales7. Profitant de cette technologie, la clé des événements qui se produisent au cours du développement des îlots pancréatiques peuvent être déchiffrés au niveau unicellulaire8. Parmi les protocoles de RNA-sequencing unicellulaires, Smart-seq2 permet la génération d’ADNc pleine longueur avec une sensibilité améliorée et précision et l’utilisation des réactifs types à bas coût9. Puce-seq2 prend environ deux jours pour construire une banque d’ADNc pour séquençage10.
Ici, nous proposons une méthode pour isoler des cellules marquées fluorescence β du pancréas de foetus pour adultes de souris transgéniques Ins1-DP11, à l’aide de la cellule activée par fluorescence triant (FACS) et analyse de la performance de transcriptomique à la niveau unicellulaire, utilisant la technologie Smart-seq2 (Figure 1). Ce protocole peut être étendu pour analyser les transcriptions de tous les types de cellules endocrines pancréatiques dans les États normales, pathologiques et vieillissement.
Protocole
Toutes les méthodes décrites ici ont été approuvés par l’animalier institutionnel et utilisation Comité (IACUC) de l’Université de Pékin.
1. pancréas isolement
-
Pour les embryons E17.5 (embryonnaire jour 17,5) :
- Estimer embryonnaire jour 0,5 d’après le point dans le temps lorsque la fiche vaginale apparaît.
- Sacrifier les souris enceintes en CO2 administration. Vaporiser la fourrure abdominale avec 70 % d’alcool.
- Faire une incision en forme de V avec des ciseaux de la région génitale, s’étendant aux côtes. Ce processus s’ouvre complètement la cavité abdominale.
- Disséquer l’utérus hors de la cavité abdominale et le placer dans un plat de 10 cm contenant PBS froid.
- Disséquer les embryons de l’utérus avec une pince à pointe fine sous un stéréomicroscope, enlever les autres tissus, tels que le placenta et le cordon ombilical. Placer tous les embryons dans un plat de 10 cm contenant PBS froid.
- Déchirer la cavité abdominale des embryons et déterrer les tissus viscéraux avec des pincettes de coude. Transférer les tissus viscéraux dans une boite de fond noir 6 cm contenant PBS froid.
- Le pancréas est situé dans le coin supérieur gauche de l’abdomen et l’attache à l’estomac, la rate et du duodénum (jaune pointillé dans la Figure 2 a)12. Détacher les pancréas du tissu viscéral à l’aide de pinces et de regrouper le tissu pancréatique dans un flacon de 20 mL contenant 5 mL de solution de collagènase froid de 0,5 mg/mL : la collagénase P de 0,5 mg/mL dans le tampon de l’isolement (HBSS contenant 10 mM HEPES, 1 mM MgCl2 5 mM de Glucose, pH 7,4).
-
Pour les souris P0-P15 (jour après la naissance 0-15) :
- Sacrifier et fixer la souris en respectant les branches d’un morceau de paillasse protecteur avec du ruban adhésif. Vaporiser la fourrure abdominale avec 70 % d’alcool.
- Ouvrir complètement la cavité abdominale comme indiqué au point 1.1.3. Ainsi qu’à la gauche de la souris, tirez sur l’intestin. Cette procédure va exposer les lobes duodénales, gastriques et de la rate, du pancréas. Soigneusement disséquer tous les lobes du pancréas (Figure 2 b)12 et regrouper le tissu pancréatique dans un flacon de 20 mL contenant 5 mL de solution de collagènase froid de 0,5 mg/mL.
-
Pour les souris P18-P60 (jour après la naissance de 18-60) :
- Préparer la solution de la collagénase. Rester sur la glace jusqu'à prêt à l’emploi.
- Sacrifier la souris et ouvrir la cavité abdominale, comme mentionné ci-dessus (étape 1.1.2 et 1.1.3).).
Remarque : Ne pas blesser le foie, toute blessure au foie réduira la pression d’écoulement dans le canal cholédoque et réduire l’efficacité de la perfusion de l’étape suivante. - Retirez la xiphoïde. Tirez sur l’intestin, ainsi qu’à la droite de la souris et poussez le lobe gauche et droit médial du foie de chaque côté pour exposer la vésicule biliaire (flèche blanche dans la Figure 2) et le canal cholédoque.
- Serrez le duodénum avec une paire de pinces de petit bateau à la position supérieure et inférieure, flanquant le site où le canal cholédoque pénètre dans le duodénum (Figure 2D).
Remarque : Ne pas clamper les lobes gastriques ou spléniques du pancréas. La solution de collagénase ne coulera pas dans le lobe gastrique et de la rate, du pancréas à l’étape suivante si serré. - Remplir une seringue de 5 mL de solution de collagènase froid de 0,5 mg/mL et insérer une aiguille 30 G dans la vésicule biliaire. Soigneusement et en douceur, manipuler l’aiguille dans le canal cholédoque.
- Perfuse le pancréas par l’injection de 1 à 5 mL de solution de collagènase froid de 0,5 mg/mL, selon la taille de la souris. L’injection doit être lent et constant pour que l’aiguille ne glisse pas hors de la gaine et empêcher l’intestin contre l’éclatement sous haute pression liquide. L’injection est terminée lorsque le pancréas est pleinement développé.
- Disséquer le pancréas (le jaune pointillé dans la Figure 2D) hors de la cavité abdominale à l’aide de forceps immédiatement après la perfusion. Placer le tissu dans un flacon de 20 mL contenant 5 mL de solution de collagènase froid de 0,5 mg/mL. Si manipuler plus d’une souris à la fois, perfuse les souris un par un et centraliser les tissus dans solution de collagènase froid dans un flacon de 20 mL, jusqu'à ce que toutes les souris ont été disséqués.
2. Digestion de collagénase et l’isolement de l’Islet
- Le flacon de 20 mL contenant le tissu pancréatique dans un bain-marie à 37 ° C et incuber pendant 3 min équilibrer la température.
- Agiter doucement le tube pendant 3 à 5 min. Si le tissu pancréatique est bien gonflé, elle dissociera graduellement en morceaux petit tissu jusqu'à ce qu’il est finalement dispersée uniformément. Le temps de digestion varie selon la taille du pancréas et de l’efficacité de la perfusion.
- Filtrer le produit digéré à travers un tamis en nylon de 0,25 mm dans un tube à centrifuger de nouveau 50 mL. Bien laver le filtre à l’aide d’une seringue de 20 mL contenant PBS glacée.
- Pour E17.5-P15 pancréas, passez à l’étape 3.1.
- Pour pancréas P18-P60, centrifuger à 200 x g pendant 1 min et éliminer le surnageant. Remettre en suspension le tissu avec du PBS froid.
- Verser 5 mL de la suspension des tissus dans un plat de fond noir 6 cm. Les îlots pancréatiques sont des structures de blancs laiteux, petits, compacts et tissu acinaire est lâche et translucide blanc. Choisir des îlots avec une pipette μL 200 et transférez-les dans un tube de 1,5 mL contenant une petite quantité de froid PBS.
3. la trypsine Digestion du tissu pancréatique ou îlots
- Centrifuger le tube contenant du tissu pancréatique ou îlots à 200 x g pendant 1 min à 4 ° C et éliminer le liquide surnageant sans déranger le culot.
- Resuspendre le culot avec 0,25 % de trypsine-EDTA et incuber dans un bain-marie à 37 ° C. Après 4 min d’incubation, doucement et parfois aspirer (pipette) pendant 1 min à l’aide de 200 conseils μL.
Remarque : Pour E17.5-P3 pancréas, ajouter 1 mL de trypsine-EDTA. Pour pancréas P4-P15, ajouter 3 mL trypsine-EDTA. Pour les 100-300 îlots, ajouter 1 mL de trypsine-EDTA. Réglez le volume de la trypsine-EDTA selon la quantité de tissu. - Arrêter la digestion en ajoutant 0,4 x volume de sérum froid fœtal (SVF) et mélanger en douce vortex.
- Centrifuger à 250 x g pendant 3 min à 4 ° C. Éliminer le liquide surnageant sans déranger le culot.
- Remettre en suspension les cellules avec tampon de 200 μL froid FACS (HBSS contenant 1 % FBS, pH 7,4). Transférer les cellules dans un tube de 5 mL FACS.
- Rapidement tourner le tube de FACS pour permettre la suspension cellulaire de passer par le filtre pour enlever les débris non digérés grand tissu. La suspension monocellulaire dans le tube est maintenant prête pour le tri des FACS.
4. single-cell lyse
-
Préparer le tampon de lyse cellulaire.
Remarque : Effectuez toutes les expériences sous une hotte de stérilisation UV avec hotte à flux laminaire. Tous les tubes, les plaques et les pointes de pipette devraient être RNase - DNase- libre. Décontaminer le capot et les pipettes avec solution away RNase avant utilisation.- Décongeler les réactifs sur la glace : dNTP (10 mM), primer oligo-dT (10 μM) et SCGDE stock solution (01:20).
- Diluer la SCGDE à 1:5 x 105 avec de l’eau exempte de nucléase.
- Calculer le volume de tampon de lyse cellulaire nécessaire. Ajouter 0,1 μL d’inhibiteur de RNase (40 U/μl), 1,9 μL de 0,2 % (vol/vol) X-100 Triton, 1 μL de dNTP et 1 μL de primer oligo-dT 0,05 μL de SCGDE dilué pour un volume final de 4,05 μL de chaque cellule.
- Aliquoter le tampon de lyse cellulaire en tubes PCR de 0,2 mL mince-mur 8 bandes ou plaques 96 puits. Centrifuger les tubes ou les planches pendant 30 s à 4 ° C.
Remarque : Centrifuger les tubes PCR de 0,2 mL mince-mur 8 bandes à 7500 x g et plaques 96 puits à 800 x g dans les étapes suivantes.
-
Cueillette de cellule unique et lyse.
- Manuellement pic FACS Trier monocellules Ins1-DP+ dans un tampon FACS dans 8-bande des tubes PCR à l’aide d’une pipette capillaire de 30 à 40 µm ou directement Trier monocellules Ins1-DP+ dans des plaques à 96 puits. Le volume contenant une seule cellule est considérée être inférieure à 0,3 μL.
Remarque : Utilisation des nuages de points avant hauteur (FSC-H) vs avant zone de dispersion (FSC-A) stratégie de blocage pour discrimination doublet, FSC-H vs côté zone de dispersion (SSC-A) pour les débris et fluorescence gating Ins1-DP+ dans le tri cellulaire (Figure 3 a-3C). Pour la méthode de cueillette, trier les cellules cibles dans un tube de 1,5 mL contenant 300 μL FACS tampon. La concentration finale appropriée est de 5 à 10 cellules/μl. Ajuster le volume tampon en conséquence. Pour la méthode collection, trier une seule cellule dans chaque puits d’une plaque de 96 puits suivant le manuel de l’instrument. 7 , 13 - Vortex tubes ou plaques à lyser les cellules et libérer l’ARN. Centrifuger les tubes ou les planches pendant 30 s à 4 ° C et immédiatement les placer sur la glace.
Remarque : Les cellules peuvent être stockées à-80 ° C pendant une semaine.
- Manuellement pic FACS Trier monocellules Ins1-DP+ dans un tampon FACS dans 8-bande des tubes PCR à l’aide d’une pipette capillaire de 30 à 40 µm ou directement Trier monocellules Ins1-DP+ dans des plaques à 96 puits. Le volume contenant une seule cellule est considérée être inférieure à 0,3 μL.
5. cellule unique cDNA Amplification
-
Transcription inverse (RT).
- Décongeler les réactifs de RT (tableau 1) sur la glace.
- Incuber les échantillons à 72 ° C pendant 3 min et mettre immédiatement les tubes ou des plaques sur la glace pendant au moins 1 min. Centrifuger brièvement les tubes ou les planches pendant 30 s à 4 ° C.
Remarque : Utilisez un thermocycleur avec un couvercle de 105 ° C chauffé pour tous les incubations. - Préparer le mélange de RT pour toutes les réactions, comme décrit dans le tableau 1.
- Ajouter 5,7 μL de mélange RT à chaque échantillon pour porter le volume à un total de 10 μL. Doucement le mélange de vortex et centrifuger pendant 30 s à 4 ° C.
- Placer les échantillons dans un thermocycleur et démarrez le programme RT comme suit : 42 ° C pendant 90 min, 10 cycles (50 ° C pendant 2 min, 42 ° C pendant 2 min), 70 ° C pendant 15 min et maintenez à 4 ° C.
-
Avant l’amplification par PCR.
- Décongeler les réactifs PCR (tableau 2) sur la glace.
- Préparer le mélange PCR pour toutes les réactions, comme décrit dans le tableau 2.
- Ajouter 15 μL de mélange PCR pour chaque échantillon, qui contient les réactions du premier-brin. Doucement le mélange de vortex et centrifuger pendant 30 s à 4 ° C.
- Placer les échantillons dans un thermocycleur et démarrer le programme PCR suivant : 98 ° C pendant 3 min, 18 cycles (98 ° C, 20 s, 67 ° C pendant 15 s, 72 ° C pendant 6 min), 72 ° C pendant 5 min et maintenez à 4 ° C.
Remarque : Le produit de la PCR peut être stocké à 4 ° C pendant moins d’une semaine ou à-20 ° C /-80 ° C pendant 6 mois.
-
Purification de la PCR.
- Ajouter 25 μL de remises en suspension perles de purification de l’ADN (1 x) pour chaque échantillon de l’étape précédente et mélangez bien en vortex. Puis, faites tourner rapidement le tube ou les plaques à température ambiante pour recueillir le liquide, mais éviter la colonisation de perles.
NOTE : Equilibrer les perles de purification de l’ADN à température ambiante pendant 15 minutes et vortex avant utilisation. - Incuber 5 min à température ambiante.
- Place le tube ou plaque sur un approprié magnétique reposer jusqu'à ce que la solution est claire, puis soigneusement retirer et jeter le surnageant.
- Ajouter 200 μL d’éthanol à 80 % fraîchement préparés pour laver les perles tandis que dans le support magnétique, incuber pendant 30 s, puis soigneusement retirer et jeter la solution d’éthanol.
Remarque : La solution d’éthanol à 80 % (vol/vol) doit être fraîchement préparée chaque fois. - Répétez l’étape 5.3.4 pour un total de deux lavages.
- Avec précaution, retirer et jeter le reste de solution d’éthanol et de l’air sec les perles tout en tube ou plaque est sur le support magnétique.
Remarque : Éviter de trop sécher les perles pour assurer l’efficacité maximale d’élution. - Ajouter 11 μL d’eau exempte de nucléase pour Éluer la cible de l’ADN de la perle et bien mélanger en vortex. Puis, rapidement tourner le tube ou la plaque et placer sur un support magnétique jusqu'à ce que la solution est claire. Transférer 10 μl de l’échantillon dans un nouveau tube PCR.
Remarque : Si dimères existent après un rond simple de purification, basée sur la taille de la cDNA distribution détection, purifier à nouveau pour enlever les dimères complètement. Les dimères peuvent influer sur le calcul de rendement d’ADNc si autorisé à rester dans l’échantillon.
- Ajouter 25 μL de remises en suspension perles de purification de l’ADN (1 x) pour chaque échantillon de l’étape précédente et mélangez bien en vortex. Puis, faites tourner rapidement le tube ou les plaques à température ambiante pour recueillir le liquide, mais éviter la colonisation de perles.
-
Contrôle de la qualité applicables à l’ARNC.
- Choisir au hasard les échantillons pour détecter le rendement total de cDNA en utilisant un fluorimètre.
- Évaluer les niveaux d’expression de gène marqueur par PCR en temps réel (qPCR) (Figure 4E). Retirer 1 μL d’échantillon à diluer 40 fois et effectuer à l’aide d’une plaque 384 puits de qPCR. Préparer le mélange de qPCR tel que décrit dans le tableau 3. Conditions de cyclisme : 95 ° C pendant 10 min, 45 cycles (95 ° C pendant 10 s, 60 ° C pendant 15 s, 72 ° C pendant 15 s).
- Choisir au hasard les échantillons pour détecter la distribution de taille en utilisant un instrument de l’électrophorèse capillaire parallèle.
6. cDNA Library Construction
-
Réaction de Tagmentation par la transposase Tn5.
- Détecter le rendement total de l’ADNc des cellules qPCR-sélectionné à l’étape 5.4.2 utilisant un fluorimètre. Utilisation 2 ng de l’ADNc comme produit de départ.
- Décongeler les réactifs de réaction tagmentation (tableau 4) sur la glace.
- Préparer la réaction de tagmentation dans un tube de PCR de 0,2 mL mince-mur 8 bandes, tel que décrit dans le tableau 4et mélanger soigneusement en vortex. Puis, faites tourner rapidement vers le bas de la solution à température ambiante.
- Incuber les échantillons à 55 ° C pendant 10 min et de tenir à 4 ° C.
- Immédiatement ajouter 2 μL de 5 x TS dans chaque échantillon contenant la tagmented ADN pour arrêter la réaction. Mélanger soigneusement par vortex et ensuite faites tourner rapidement vers le bas de la solution à température ambiante.
- Incuber le mélange pendant 5 min à température ambiante. L’ADN doit être traité immédiatement pour l’enrichissement final PCR.
-
Amplification de fragments adaptateur-ligaturé.
- Décongeler les réactifs PCR (tableau 5) sur la glace.
- Préparer le mélange PCR de l’enrichissement, comme décrit dans le tableau 5et mélanger soigneusement en vortex. Puis, faites tourner rapidement vers le bas de la solution à température ambiante.
- Effectuer la PCR en utilisant le programme suivant : 72 ° C pendant 10 min, 98 ° C pendant 30 s, 8 cycles (98 ° C pendant 15 s, 60 ° C pendant 30 s, 72 ° C pendant 3 min), 72 ° C pendant 5 min et maintenez à 4 ° C.
Remarque : Le nombre de cycles dépend de la quantité d’ADN de bibliothèque attendue.
-
Purification de la PCR avec sélection de la taille.
- Ajouter 14 μL de remises en suspension perles de purification de l’ADN (0,7 x) pour chaque échantillon de l’étape précédente et mélangez bien en vortex. Puis, rapidement tourner le tube à température ambiante pour recueillir le liquide, mais éviter la colonisation de perles.
NOTE : Equilibrer les perles de purification de l’ADN à température ambiante pendant 15 minutes et vortex avant utilisation. - Incuber 5 min à température ambiante.
- Placer le tube sur un support magnétique approprié jusqu'à ce que la solution est claire, soigneusement le surnageant de transfert à une nouvelle bande de tube et jeter la bande précédente de tube.
- Ajouter 3 μL de remises en suspension perles de purification de l’ADN (0,15 x) à chaque échantillon dans la bande de tube et mélangez bien en vortex. Puis, rapidement tourner le tube à la température ambiante.
- Incuber 5 min à température ambiante.
- Place le tube ou plaque sur un approprié magnétique reposer jusqu'à ce que la solution est claire, puis soigneusement retirer et jeter le surnageant.
- Ajouter 200 μL d’éthanol à 80 % fraîchement préparés pour laver les perles tandis que dans le support magnétique, incuber pendant 30 s, puis soigneusement retirer et jeter la solution d’éthanol.
Remarque : La solution d’éthanol à 80 % (vol/vol) doit être fraîchement préparée chaque fois. - Répétez l’étape 6.3.7 pour un total de deux lavages.
- Avec précaution, retirer et jeter le reste de solution d’éthanol et de l’air sec les perles tout en tube ou plaque est sur le support magnétique.
Remarque : Éviter de trop sécher les perles pour assurer l’efficacité maximale d’élution. - Ajouter 11 μL d’eau exempte de nucléase pour Éluer la cible de l’ADN de la perle et bien mélanger en vortex. Puis, rapidement tourner le tube ou la plaque et placer sur un support magnétique jusqu'à ce que la solution est claire. Transférer 10 μl de l’échantillon à une nouvelle bande de tube.
- Ajouter 14 μL de remises en suspension perles de purification de l’ADN (0,7 x) pour chaque échantillon de l’étape précédente et mélangez bien en vortex. Puis, rapidement tourner le tube à température ambiante pour recueillir le liquide, mais éviter la colonisation de perles.
-
Contrôle de la qualité d’ADNc final.
- Mesurer la concentration de chaque bibliothèque à l’aide d’un fluorimètre et vérifier la granulométrie en utilisant un instrument de l’électrophorèse capillaire parallèle.
Remarque : Le rendement de l’ADN est généralement entre 15 et 25 ng pour chaque bibliothèque. Les fragments allant de 250 bp 450 bp est observé. Si les dimères restent après purification, comme l’a confirmé le contrôle de distribution de taille, purifier avec 1 x perle de purification de l’ADN, une fois de plus.
- Mesurer la concentration de chaque bibliothèque à l’aide d’un fluorimètre et vérifier la granulométrie en utilisant un instrument de l’électrophorèse capillaire parallèle.
-
Mise en commun de bibliothèque
- Selon la taille de fragment approximative, quantités égales de pool d’ADN de chaque échantillon, en veillant à ce qu’aucun d'entre eux contiennent les mêmes combinaisons de cartes N6XX et N8XX.
7. séquençage de l’ADN
- Bibliothèques du sujet avec code à barres au séquençage de single-end bp 51 en utilisant un système de séquençage haut-débit. Effectuer le séquençage suivant le protocole du fabricant. La profondeur de séquençage de chaque cellule est environ 1 million se lit en moyenne8, au moins 0,5 millions lit par cellule14.
8. Analyses bioinformatiques
-
Séquençage de l’évaluation de la qualité et l’alignement.
- Évaluer la qualité des lectures séquencées à l’aide de FastQC (v0.11.3)15 avec les paramètres suivants : « fastqc--extrait -o output_dir input_fastq ».
- Fusionner le génome de la souris avec les séquences SCGDE en utilisant la commande « cat mm10.fa ERCC.fa > mm10_ERCC.fa ».
- Construire bowtie2 indice16 (v2.2.5) avec les paramètres suivants : « bowtie2-build mm10_ERCC.fa mm10_ERCC ».
- Aligner les lectures à l’aide de tophat2 (v2.1.0)17 avec les paramètres suivants : « tophat2 -o output_dir -G gene.gtf--transcriptome-index trans_index mm10_ERCC input_fastq ».
-
Quantifier les niveaux d’expression de gène.
- Comte mappé lit pour chaque gène à l’aide de HTSeq (v 0.6.0)18 avec les paramètres suivants : '' htseq-comte, bam -f - r pos -s pas - une 30 gene.gtf accepted_hits.bam > read_count.txt''.
- Normaliser les niveaux d’expression de gène aux transcriptions par million (TPM)19.
-
Contrôle de la qualité des cellules.
- Exclure les cellules avec moins de 0,5 millions de lectures mappés ou moins de 4 000 gènes (TPM > 1).
NOTE : le critère d’exclusion dépend sur les types de cellules et de la profondeur de la séquence. - Conserver les cellules expriment des marqueurs endocrines (p. ex., Ins1 pour les cellules β, Gcg pour cellules α) et exclure les cellules expriment des marqueurs non endocrines (p. ex., Spi1 de leucocytes).
- Exclure les cellules avec moins de 0,5 millions de lectures mappés ou moins de 4 000 gènes (TPM > 1).
-
Analyse en composantes principales (PCA)
- Identifier les gènes très variables selon SCGDE spike-ins, comme précédemment décrit20.
- Effectuer des PCA à l’aide de la fonction « APC » dans le R package FactoMineR (v1.31.4)21, avec log2(TPM + 0.1) de gènes très variables.
- Visualiser les résultats de l’APC avec ggplot2 (v2.0.0)22.
-
Hiérarchique de clustering.
- Identifier les gènes avec les plus hautes charges composantes principales (PC) en utilisant la fonction « dimdesc » FactoMineR (v1.31.4)21.
- Effectuer un regroupement hiérarchique à l’aide de la fonction « heatmap.2 » dans le R paquet gplots (v3.0.1)23, avec log2 (TPM + 1) les valeurs relatives de haute PC chargement des gènes.
Résultats
Pancréas ont été disséqués souris embryonnaires, néonatals et postnatals (Figure 2 a et 2 b). Pour plus de jours après la naissance 18 souris, l’effet digestif dépend du degré de perfusion ; par conséquent, l’injection est l’étape la plus importante pour l’isolement des îlots pancréatiques (Figure 2-2E et le tableau 6). Autant collagénase a été injectée comme c’était possible de remplir le pancréas au cours de cette étape. Le pancréas entièrement gonflé est illustré à la Figure 2D. Si la perfusion n’est pas réussie (Figure 2E), mais l’échantillon est précieux, le pancréas peut être déchiré en petits morceaux pour la digestion suffisante plus tard.
Après la perfusion, le tissu pancréatique a été digéré en petits morceaux pour libérer des îlots (Figure 2F). Pour raccourcir le temps de tri de FACS, nous avons enrichi les cellules endocrines en choisissant les îlots à l’avance. La taille des îlots peut-être varier selon l’âge de la souris et l’intensité de la digestion. Occasionnellement, les îlots ne sont pas forme rondes. Îlots devraient être choisis selon la couleur et état compact (Figure 2 et tableau 6). Si la souche de souris transgéniques a un gène rapporteur, comme la GFP ou DP pour les cellules endocrines, les îlots pourraient également être pris sous un microscope à fluorescence.
Les cellules INS1-DP+ ont été purifiées par FACS Trier (Figure 3 a-3 C), et puis cellules individuelles ont été recueillis à l’aide d’une pipette capillaire pour monocellulaires RNA-seq (Figure 3D). ADNc amplifié avec succès doit avoir une longueur totale supérieure à 500 bp et s’enrichir de 1,5 Ko à 2 kb. En outre, il y a généralement un enrichissement de bp 500-600 du cDNA observée dans les cellules Ins1-DP+ , ce qui peuvent représenter les transcriptions de l’insuline (Figure 4 a). Cependant, il y a certaines situations anormales observées10 (tableau 6). Par exemple, les fragments d’ADNc près de 100 bp sont des dimères d’amorce (Figure 4 b), qui sont généralement causées par les amorces excessives, qui doivent être supprimés en répétant l’étape de purification de l’ADN. L’existence de dimères d’amorce peut influer sur les calculs de cDNA total des rendements, qui sont utilisés pour la construction de la bibliothèque suivante. Les fragments d’ADNc entre 100 bp et 500 bp habituellement dégradés représentent cDNA (Figure 4), qui est causée par des problèmes de statut ou réactif mauvaise cellule, tels que la contamination de la RNase. Dans ces conditions, nous devrions identifier la cause de la dégradation de l’ADN et exclure la perturbation. Par exemple, afin d’assurer le statut de bonnes cellules, tissus doivent être digérés et cellules doivent être triés, rapidement et en douceur, et opérations doivent être effectuées avec précaution pour éviter tout type de polluants.
Après la purification des banques d’ADNc pour le séquençage, nous avons obtenu des fragments d’ADNc de différentes tailles en suivant les instructions pour l’ajout de divers rapports de perles de purification de l’ADN à l’échantillon. Par exemple, nous pouvons obtenir des ADNc variant de 250 bp 450 bp par ajout de 0,7 x et 0,15 x perles de purification de l’ADN au premier et deuxième tours de purification, respectivement (Figure 4). Cette étape a un taux de réussite élevé. Toutefois, s’il y a des fragments d’environ 100 bp, il est conseillé de purifier les bibliothèques avec 1 x perle de purification de l’ADN pour enlever des dimères (tableau 6). Dimères laissées mettra en travers la quantification de l’ADN et influeront sur l’exemple de mise en commun des résultats, qui se traduit par l’acquisition de données inégales de chaque échantillon.
Nous avons analysé les données de séquençage avec approches bioinformatiques (Figure 5 a). Les scores de qualité de séquençage devraient être supérieurs à 30 au cours de la séquence d’évaluation de la qualité (Figure 5 b). Après mise en conformité, 80 à 90 % de lectures sont censés être mappé sur le génome de référence. Pour obtenir des cellules de haute qualité pour des analyses en aval, nous exclus des cellules avec moins de 0,5 millions de lectures mappés ou avec moins de 4 000 gènes (Figure 5 et 5D) a détecté. Après APC et clustering hiérarchique, nous caractérise les différents groupes de cellules et identifié les gènes qui ont été exprimées hétérogène dans différents groupes8.

Figure 1 : vue d’ensemble schématique pour monocellulaires RNA-seq de cellules endocrines pancréatiques de souris. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : dissection pancréas, de perfusion et de cueillette de l’îlot. (A) la Dissection des tissus pancréatiques embryonnaires (en bas) d’un embryon E17.5 (en haut). La ligne pointillée jaune délimite le tissu pancréatique. Echelle = 1 000 µm. (B) la Dissection des tissus pancréatiques postnatal (à droite) d’une souris de P10 (à gauche). Echelle = 1 000 µm. (C-D), le tissu du pancréas de souris P60 avant (C) et après la perfusion (D). La ligne pointillée jaune délimite le tissu pancréatique. Flèche blanche indique la vésicule biliaire. (E) le tissu pancréatique partiellement perfusé de souris P60. Flèche rouge indique la zone bien perfusée du pancréas. Flèches bleues indiquent les zones mal perfusées du pancréas. (F) le pancréas tissus avant (en haut) et après digestion de collagénase (en bas). (G) flèches pointent vers les îlots qui ont été libérés des tissus pancréatiques collagénase digéré. Echelle = 200 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : cueillette manuelle des cellules avec une pipette capillaire de 30 à 40 µm sous un microscope. (A-C) Par étapes FACS Gate pour le tri des cellules Ins1-DP+ . (D) la brillante cercles indiquent les cellules et le tube est une pipette capillaire. Sélectionnez les cellules à la morphologie bien mieux (flèche) et ignorer les cellules en cluster (pointe de flèche). Echelle = 200 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : Détection de qualité d’ADNc et bibliothèque distribution de taille des cellules Ins1-DP+ par un instrument de l’électrophorèse capillaire parallèle. Résultat (A), le représentant de l’ADNc avec succès pré amplifié. Normalement, le profil de l’ARNC devrait être supérieure à 500 bp avec un pic de Ko ~1.5–2. (B) le résultat représentatif du cDNA pré amplifié avec un pic de dimère d’amorce. Le pic de dimère d’amorce est d’environ 100 BP. (C) le profil de l’ADNc pré amplifié avec des fragments entre 100 bp et 500 bp indique la possibilité de dégradation de l’ADNc. (D) la distribution granulométrique des ADNc bibliothèque variant entre 250 bp et 450 bp suivants, les étapes de purification mentionnés dans le présent protocole. Niveaux (E), l’expression Ins2 dans banques d’ADNc par qPCR (à gauche) et parent (à droite) les données du séquençage. Les x-axes représentent distinctes 8 échantillons de cellule unique. L’axe des ordonnées (à gauche) représentant ΔCt normalisée par rapport à Gapdh (CtGapdh- CtIns2 + 6) et l’axe des y (à droite) représentant le niveau d’expression normalisée Ins2 par rapport à Gapdh (journal2(TPM Ins2 / TPMGapdh)). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
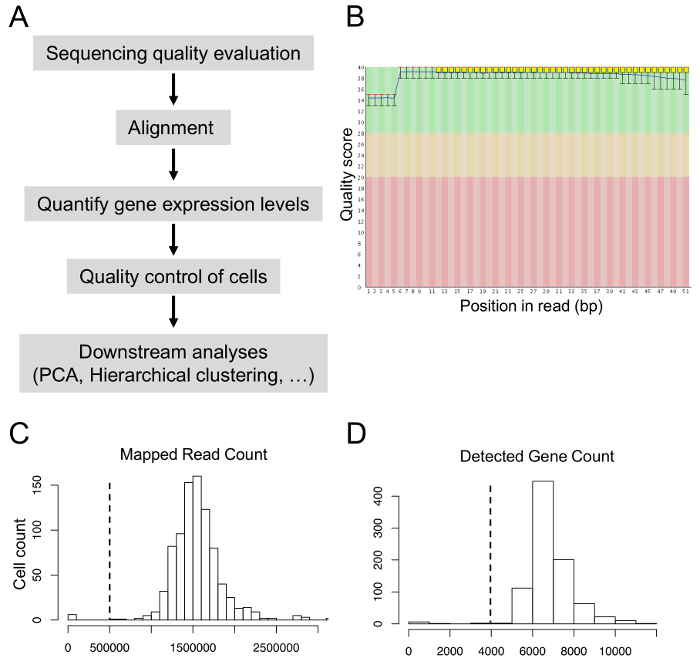
Figure 5 : analyses bioinformatiques de données single-cell transcriptome. (A) Pipeline d’analyses bioinformatiques. (B) séquençage des scores de qualité dans l’ensemble de toutes les bases de lectures. (C) Distribution du comte lecture mappé. (D) répartition du nombre de gènes détectés. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Composant | Volume (µL) |
| Transcriptase inverse (200 U/µL) | 0,5 |
| Inhibiteur de RNase | 0.25 |
| Tampon de premier-brin (x 5) | 2 |
| TNT (100 mM) | 0,5 |
| Bétaïne (5 M) | 2 |
| MgCl2 (1 M) | 0,06 |
| TSO (100 ΜM) | 0,1 |
| Eau exempte de nucléase | 0,29 |
| Total | 5.7 |
Tableau 1 : Réactif RT mélanger des composants dans une réaction µL 5,7 pour chaque échantillon.
| Composant | Volume (µL) |
| Réaction de premier-brin | 10 |
| ADN polymérase (2 x ReadyMix) | 12,5 |
| EST des amorces de PCR (10 µM) | 0.25 |
| Eau exempte de nucléase | 2.25 |
| Total | 25 |
Tableau 2 : Réactif de pré amplification PCR mélanger des composants dans une réaction de 25 µL de chaque échantillon
| Composant | Volume (µL) |
| SYBR Green Master Mix | 5 |
| Amorces (5 µM) | 0,5 |
| Eau exempte de nucléase | 2.5 |
| ADNc | 2 |
| Total | 10 |
Tableau 3 : réactif de qPCR mélanger des composants dans une réaction µL 10 à l’aide d’une plaque 384 puits standard.
| Composant | Volume (µL) |
| 5 x TTBL | 1.6 |
| 2 ng cDNA | Variable |
| ddH2O | Variable |
| TTE Mix V5 | 2 |
| Total | 8 |
Tableau 4 : Tagmentation réactif mélanger des composants dans une réaction de 8 µL de chaque échantillon.
| Composant | Volume (µL) |
| ddH2O | 1.6 |
| Produit de l’étape précédente | 10 |
| 5 x onglet | 4 |
| N6XX | 2 |
| N8XX | 2 |
| TAE | 0,4 |
| Total | 20 |
Tableau 5 : Enrichissement PCR composants mélange réactif dans une réaction de 20 µL de chaque échantillon.
| Étape | Problème | Possibilité | Solution |
| 1.3.4 | Glissement des brides | La surface externe de l’intestin est humide provoquée par l’existence de la petite quantité d’interstitielle leakness fluide et collagénase | Swap doucement la paroi du duodénum et autres murs d’orgue dans la cavité abdominale avec des cotons-tiges |
| 1.3.6 | Intestin rafale | Liquide est trop élevée dans l’intestin | Ralentir la vitesse d’injection |
| 2.6 | Îlots collent au tissu exocrine lors du prélèvement des îlots | Insuffisant pour la digestion | Prolonger la durée de la digestion et en agitant force |
| 5.3 | Le rendement de l’ADNc est faible après amplification par PCR | Les cellules sont en mauvais état | Garder les cellules fraîches et en bon état |
| 5.4 ou 6,4 | Dimères d’amorce peuvent être vu | Amorces excessives | Purifier l’ADNc codant pour une fois de plus avec des perles de purification de l’ADN (0,8 pour 1 ou 1:1 ratio) |
| 5.4 | ADNc dégradée après amplification par PCR | Mauvaise qualité des cellules ou des réactifs contaminés | Maintenir les cellules en bon état ou modifier des réactifs |
| 6.3 | La quantité d’ADN est faible après la construction de la bibliothèque | Trop peu de cycles PCR ou la qualité de l’ADNc n’est pas bonne | Augmenter le nombre de cycles ou d’assurer la qualité des ADNc avant la construction de la bibliothèque |
Tableau 6 : dépannage.
Discussion
Dans ce protocole, nous avons démontré une méthode efficace et facile à utiliser pour étudier les profils d’expression unicellulaire des cellules β pancréatiques. Cette méthode pourrait être utilisée pour isoler les cellules endocrines du pancréas embryonnaires, néonatals et postnatals et d’effectuer des analyses transcriptomiques unicellulaires.
L’étape la plus critique est l’isolement des cellules β unique en bon état. Pancréas complètement perfusés répondent mieux à la digestion ultérieure. Perfusion insuffisante, qui survient généralement dans le pancréas dorsal, se traduira par un rendement faible îlot. Après la perfusion, le temps de digestion et de secousses d’intensité nécessitent une attention particulière. Digestion excessive, résultante de périodes d’incubation longue et vigoureuse agitation, peut briser les îlots en morceaux. Une digestion insuffisante se séparera pas complètement les îlots de tissus adjacents. Après la digestion du pancréas, les îlots ont été purifiés par la cueillette de main plutôt que par centrifugation en gradient de densité. Bien que la centrifugation en gradient de densité peut enrichir des îlots, de nombreux petits îlots ou îlots reliés avec tissu acinaire sont ignorées par erreur. Essayez d’éviter de choisir le tissu acinaire, qui risque de provoquer la digestion trypsique inefficace et la pureté des cellules β.
Réplicats biologiques indépendants sont nécessaires pour distinguer les effets biologiques de la variabilité et le lot. Si deux biologiques réplique ont un profil similaire au PCA et comprennent des sous-groupes similaires au regroupement des résultats, ces échantillons pourraient révéler la variabilité biologique fiable. Dans le cas contraire, des réplicats biologiques supplémentaires sont nécessaires pour confirmer les résultats. Pour un organe métabolique comme le pancréas, l’état de cellule associée à l' horloge circadienne24 pourrait expliquer les différences de traitement par lots. Pour résoudre ce problème, il est recommandé de recueillir tous les échantillons à la fois de la journée.
La limitation de cette approche est faible débit parce que nous devons construire une bibliothèque pour chaque cellule. En raison de l’excessives amorces dans la réaction, l’ADNc avant et après la construction de la bibliothèque doit généralement être purifié deux fois afin d’éliminer complètement les dimères d’amorce. Ces processus sont coûteuses en temps. Un protocole modifié25 a été rapporté récemment, qui pourrait résoudre ce problème dans une certaine mesure. Dans ce protocole, le code à barres spécifique des cellules étiquettes de fragments d’ADNc pendant l’étape de transcription inverse. Par conséquent, l’ADNc de chaque cellule peuvent être regroupé ensemble et ensuite être purifié, suivie de la construction de la bibliothèque. Cette modification augmente considérablement le débit de la construction de la bibliothèque. Toutefois, cette méthode modifiée est moins sensible et détecte moins de gènes par cellule. En outre, cette méthode est une méthode avec une couverture réduite lecture de comptage 3'. Smart-seq2 est donc toujours la méthode la plus appropriée pour le développement du pancréas.
La préparation du pancréas d’autres espèces peut-être être différente. Pour pancréas humain, les îlots peuvent être isolés à la suite de précédents protocoles26,27. Ensuite, les îlots isolés peuvent être dissociés en cellules individuelles28 et effectuent des analyses de cellule unique à l’aide de cette méthode. Cette méthode peut être largement appliquée à la recherche de développement du pancréas chez les mammifères, de maladies et de régénération.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions le Centre National pour les Sciences de la protéine, Pékin (Peking University) et le centre de Pékin-Tsinghua le Life Science Computing Platform. Ce travail a été soutenu par le ministère de la Science et technologie de la Chine (2015CB942800), la Fondation National sciences naturelles de Chine (31521004, 31471358 et 31522036) et le financement du centre de Pékin-Tsinghua pour les Sciences de la vie à C.-R.X.
matériels
| Name | Company | Catalog Number | Comments |
| Collagenase P | Roche | 11213873001 | |
| Trypsin-EDTA (0.25 %), phenol red | Thermo Fisher Scientific | 25200114 | |
| Fetal bovine serum (FBS) | Hyclone | SH30071.03 | |
| Dumont #4 Forceps | Roboz | RS-4904 | |
| Dumont #5 Forceps | Roboz | RS-5058 | |
| 30 G BD Needle 1/2" Length | BD | 305106 | |
| Stereo Microscope | Zeiss | Stemi DV4 | |
| Stereo Fluorescence microscope | Zeiss | Stereo Lumar V12 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424R | |
| Polystyrene Round-Bottom Tube with Cell-Strainer Cap | BD-Falcon | 352235 | |
| 96-Well PCR Microplate | Axygen | PCR-96-C | |
| Silicone Sealing Mat | Axygen | AM-96-PCR-RD | |
| Thin Well PCR Tube | Extragene | P-02X8-CF | |
| Cell sorter | BD Biosciences | BD FACSAria | |
| Capillary pipette | Sutter | B100-58-10 | |
| RNaseZap | Ambion | AM9780 | |
| ERCC RNA Spike-In Mix | Life Technologies | 4456740 | |
| Distilled water | Gibco | 10977 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| dNTP mix | New England Biolabs | N0447 | |
| Recombinant RNase Inhibitor | Takara | 2313 | |
| Superscript II reverse transcriptase | Invitrogen | 18064-014 | |
| First-strand buffer (5x) | Invitrogen | 18064-014 | |
| DTT | Invitrogen | 18064-014 | |
| Betaine | Sigma-Aldrich | 107-43-7 | |
| MgCl2 | Sigma-Aldrich | 7786-30-3 | |
| Nuclease-free water | Invitrogen | AM9932 | |
| KAPA HiFi HotStart ReadyMix (2x) | KAPA Biosystems | KK2601 | |
| VAHTS DNA Clean Beads XP beads | Vazyme | N411-03 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| AceQ qPCR SYBR Green Master Mix | Vazyme | Q121-02 | |
| TruePrep DNA Library Prep Kit V2 for Illumina | Vazyme | TD502 | Include 5x TTBL, 5x TTE, 5x TS, 5x TAB, TAE |
| TruePrep Index Kit V3 for Illumina | Vazyme | TD203 | Include 16 N6XX and 24 N8XX |
| High Sensitivity NGS Fragment Analysis Kit | Advanced Analytical Technologies | DNF-474 | |
| 1x HBSS without Ca2+ and Mg2+ | 138 mM NaCl; 5.34 mM KCl 4.17 mM NaHCO3; 0.34 mM Na2HPO4 0.44 mM KH2PO4 | ||
| Isolation buffer | 1 × HBSS containing 10 mM HEPES, 1 mM MgCl2, 5 mM Glucose, pH 7.4 | ||
| FACS buffer | 1 × HBSS containing 15 mM HEPES, 5.6 mM Glucose, 1% FBS, pH 7.4 | ||
| NaCl | Sigma-Aldrich | S5886 | |
| KCl | Sigma-Aldrich | P9541 | |
| NaHCO3 | Sigma-Aldrich | S6297 | |
| Na2HPO4 | Sigma-Aldrich | S5136 | |
| KH2PO4 | Sigma-Aldrich | P5655 | |
| D-(+)-Glucose | Sigma-Aldrich | G5767 | |
| HEPES | Sigma-Aldrich | H4034 | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Oligo-dT30VN primer | 5'-AAGCAGTGGTATCAA CGCAGAGTACT30VN-3' | ||
| TSO | 5'-AAGCAGTGGTATCAAC GCAGAGTACATrGrG+G-3' | ||
| ISPCR primers | 5'-AAGCAGTGGTAT CAACGCAGAGT-3' | ||
| Gapdh Forward primer | 5'-ATGGTGAAGGTC GGTGTGAAC-3' | ||
| Gapdh Reverse primer | 5'-GCCTTGACT GTGCCGTTGAAT-3' | ||
| Ins2 Forward primer | 5'-TGGCTTCTTC TACACACCCA-3' | ||
| Ins2 Reverse primer | 5'-TCTAGTTGCA GTAGTTCTCCA-3' |
Références
- Gu, G., Dubauskaite, J., Melton, D. A. Direct evidence for the pancreatic lineage: NGN3+ cells are islet progenitors and are distinct from duct progenitors. Development. 129 (10), 2447-2457 (2002).
- Oliver-Krasinski, J. M., Stoffers, D. A. On the origin of the beta cell. Genes & Development. 22 (15), 1998-2021 (1998).
- Dor, Y., Brown, J., Martinez, O. I., Melton, D. A. Adult pancreatic beta-cells are formed by self-duplication rather than stem-cell differentiation. Nature. 429 (6987), 41-46 (2004).
- Smukler, S. R., et al. The adult mouse and human pancreas contain rare multipotent stem cells that express insulin. Cell Stem Cell. 8 (3), 281-293 (2011).
- Dorrell, C., et al. Human islets contain four distinct subtypes of beta cells. Nature Communications. 7, 11756 (2016).
- Bader, E., et al. Identification of proliferative and mature beta-cells in the islets of Langerhans. Nature. 535 (7612), 430-434 (2016).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Qiu, W. L., et al. Deciphering pancreatic islet beta cell and alpha cell maturation pathways and characteristic features at the single-cell level. Cell Metabolism. 25 (5), 1194-1205 (2017).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Piccand, J., et al. Pak3 promotes cell cycle exit and differentiation of beta-cells in the embryonic pancreas and is necessary to maintain glucose homeostasis in adult mice. Diabetes. 63 (1), 203-215 (2014).
- Veite-Schmahl, M. J., Regan, D. P., Rivers, A. C., Nowatzke, J. F., Kennedy, M. A. Dissection of the mouse pancreas for histological analysis and metabolic profiling. Journal of Visualized Experiments. (126), (2017).
- Hu, P., Zhang, W., Xin, H., Deng, G. Single cell isolation and analysis. Frontiers in Cell and Developmental Biology. 4, 116 (2016).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), 75 (2017).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), (2013).
- Anders, S., Pyl, P. T., Huber, W. HTSeq--a Python framework to work with high-throughput sequencing data. Bioinformatics. 31 (2), 166-169 (2015).
- Wagner, G. P., Kin, K., Lynch, V. J. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples. Theory in Biosciences. 131 (4), 281-285 (2012).
- Brennecke, P., et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature Methods. 10 (11), 1093-1095 (2013).
- Le, S., Josse, J., Husson, F. FactoMineR: An R package for multivariate analysis. Journal of Statistical Software. 25 (1), (2008).
- Hadley, W. . ggplot2: Elegant graphics for data analysis. , (2009).
- . gplots: Various R Programming Tools for Plotting Data Available from: https://cran.r-project.org/package=gplots (2016)
- Marcheva, B., et al. Disruption of the clock components CLOCK and BMAL1 leads to hypoinsulinaemia and diabetes. Nature. 466 (7306), 627-631 (2010).
- Li, L., et al. Single-cell RNA-seq analysis maps development of human germline cells and gonadal niche interactions. Cell Stem Cell. , (2017).
- Qi, M., et al. Human pancreatic islet isolation: Part I: Digestion and collection of pancreatic tissue. Journal of Visualized Experiments. (27), (2009).
- Qi, M., et al. Human pancreatic islet isolation: Part II: Purification and culture of human islets. Journal of Visualized Experiments. (27), (2009).
- Teo, A. K. K., et al. Single-cell analyses of human islet cells reveal de-differentiation signatures. Cell Death Discovery. 4 (14), (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon