Method Article
希少コドンリッチマーカーを用いてアミノ酸過剰生産者を同定
要約
本研究では、希少コドンリッチマーカーを用いてアミノ酸過剰生産者を同定する従来の有毒アナログベースの方法に代わる戦略を提示し、精度、感度、高スループットを同時に達成する。
要約
増え続けるアミノ酸市場を満たすためには、高性能な生産株が必要です。アミノ酸過剰生産者は、従来、アミノ酸とその類似体との競争を利用して同定される。しかし、このアナログベースの方法は精度が低く、特定のアミノ酸に対する適切なアナログは限られています。ここでは、希少コドンリッチマーカーを用いたアミノ酸オーバープロデューサーの正確で敏感でハイスループットのスクリーニングを可能にする代替戦略を紹介する。この戦略は、コードDNAにおける発生頻度に基づいてコドンが一般的または希少なものに分類されるタンパク質翻訳におけるコドン使用バイアスの現象に触発されています。希少コドンの翻訳は、飢餓下の歯車アミノ酸では完全に充電できない、対応する希少転写RNA(tRNA)に依存します。理論的には、同義の一般的な同化性等化剤を充電した後にアミノ酸の余剰がある場合、希少なtRNAを充電することができます。したがって、希少なコドンによって引き起こされる遅滞翻訳は、対応するアミノ酸の摂食または細胞内過剰産生によって回復することができる。この仮定の下で、標的アミノ酸の一般的なコドンを抗生物質耐性遺伝子または遺伝子の同義的希少な代替物に置き換えることによって、アミノ酸過剰生産者を同定するための選択またはスクリーニングシステムが確立される。蛍光または発色性タンパク質をコードする。我々は、タンパク質発現が希少なコドンの取り込みによって大きく妨げられ、タンパク質のレベルがアミノ酸濃度と正に相関することを示す。このシステムを使用して、複数のアミノ酸の過剰生産者は、変異ライブラリーから容易にスクリーニングすることができる。この希少コドンベースの戦略は、単一の改変遺伝子のみを必要とし、宿主は他の方法よりも選択を逃れる可能性が低い。これは、アミノ酸オーバープロデューサーを得るための代替アプローチを提供しています.
概要
アミノ酸の現在の生産は、発酵に大きく依存しています。しかし、ほとんどのアミノ酸産生株の力価および収量は、数十億ドルの価値がある世界的なアミノ酸市場の需要の高まりを下回っています1,2.アミノ酸産業のアップグレードには、高性能アミノ酸過剰生産者の獲得が重要です。
アミノ酸過剰生産者を同定する従来の戦略は、タンパク質合成におけるアミノ酸とその類似体との競争を利用する3,4.これらの類似体は、対応するアミノ酸を認識するtRNAを充電し、したがってペプチド鎖の伸びを阻害し、逮捕された増殖または細胞死5につながる。アナログストレスに抵抗する 1 つの方法は、細胞内アミノ酸の濃度を増加させます。.濃縮アミノ酸は、有限tRNAのアナログを上回り、機能性タンパク質の正しい合成を保証します。したがって、類似体を生き残る株は選択することができ、対応するアミノ酸の過剰生産者である可能性が高い。
L-ロイシン6などのアミノ酸の過剰生産者の選択に成功したが、アナログベースの戦略は深刻な欠点に苦しんでいる。主な懸念の1つは、変異形成の過程または自発的な突然変異によって生じたアナログ抵抗である。抵抗を持つ株は、アナログ5を遮断、エクスポート、または劣化することによって選択を逃れることができます。もう一つの懸念は、他の細胞プロセス7にアナログの有毒な副作用です。その結果、アナログ選択を生き残る株はアミノ酸過剰生産者ではないかもしれませんが、所望の過剰生産者は負の副作用のために誤って根絶される可能性があります。
ここでは、アミノ酸過剰生産者の正確かつ迅速な同定を達成するために、コドンバイアスの法則に基づく新しい戦略を提示する。ほとんどのアミノ酸は、宿主生物8、9によって異なって好まれる複数のヌクレオチドトリプレットによってコードされる。一部のコドンはコーディングシーケンスではほとんど使用されず、希少なコドンと呼ばれています。アミノ酸への彼らの翻訳は、対応するアミノ酸を運ぶ認知tRNAに依存しています.しかし、希少なコドンを認識するtRNAは、通常、一般的なコドン10、11のtRNAよりもはるかに低い存在量を持っています。その結果、これらの希少なtRNAは、他のアイソセプターとの競争で遊びのアミノ酸を捕捉する可能性が低く、希少コドンリッチ配列の翻訳は、アミノ酸の量が限られているときに減速し始めるか、あるいは終了する10.翻訳は、理論的には、対応するアミノ酸12の過剰産生または余分な供給に起因する同義の一般的なtRNAを充電した後にアミノ酸の余剰がある場合に復元することができる。希少コドンリッチ遺伝子が選択マーカーまたはスクリーニングマーカーをコードする場合、対応するフェノタイプを示す株を容易に同定することができ、標的アミノ酸の過剰生産者である可能性が高い。
上記の戦略は、アミノ酸過剰生産者の同定のための選択およびスクリーニングシステムを確立するために適用される。選択システムは、抗生物質耐性遺伝子(例えば、カンR)をマーカーとして使用し、スクリーニングシステムは蛍光をコードする遺伝子(例えば、緑色蛍光タンパク質[GFP])または発色性(例えば、PrancerPurple)タンパク質を使用する。両方のシステムのマーカー遺伝子は、標的アミノ酸の共通コドンの定義された数をその代名詞希少代替に置き換えることによって修飾される。希少コドンリッチマーカー遺伝子を保有する変異ライブラリー内の株は、適切な条件下で選択またはスクリーニングされ、標的アミノ酸の過剰生産者を容易に同定することができる。ワークフローは、希少コドンリッチマーカー遺伝子システムの構築から始まり、その後、労働条件の最適化、次にアミノ酸オーバープロデューサーの同定と検証が行われます。このアナログ非依存戦略は、タンパク質翻訳の教義に基づいており、アミノ酸オーバープロデューサーの正確かつ迅速な同定を可能にするために実質的に検証されています。理論的には、希少なコドンを含むアミノ酸やすべての微生物に直接使用することができます。全体として、希少コドンベースの戦略は、特定のアミノ酸に対する適切なアナログが利用できない場合、または高い偽陽性率が主な懸念事項である場合に、従来のアナログベースのアプローチに代わる効率的な代替手段として機能します。以下のプロトコルは、ロイシンレアコドンを使用して、エシェリヒア大腸菌L-ロイシンオーバープロデューサーを同定する際にこの戦略を実証します。
プロトコル
1. 希少コドン豊富なマーカー遺伝子を発現するプラスミドの構築
- 標的アミノ酸に対する一般的なコドンの適切な数を含むマーカー遺伝子を選択します。
注:L-ロイシンの場合、29個のロイシンコドンを含むカナマイシン耐性遺伝子カンRは、そのうち27個が一般的なコドンであり、選択システム13の構築に使用される。19個のロイシンコドンのうち17個の一般的なコドンを含むgfp遺伝子、または14個のロイシン共通コドンを収容する紫色のタンパク質コード遺伝子プランサーパープル(ppg)がスクリーニングシステムに使用されます(補足表1)). - マーカー遺伝子の一般的なコドンを同義の希少コドンに置き換えます。L-ロイシンの場合は、カンR、gfp、またはppgのコドンを希少なコドンCTAに置き換え、カンR-RC、gfp-RC、またはppg-RCを生成し、それぞれ13 ( 補足表1)
注:マーカー遺伝子の希少なコドンの頻度は、選択またはスクリーニングシステムのストリンジェンシーに影響を与えます。一般に、希少なコドンの数を増やすと、選択またはスクリーニングシステムのストリンジェンシーが増加します。適切な選択またはスクリーニング強度を達成するために、希少なコドンの異なる数を収容する一連のマーカー遺伝子を設計し、その効果を比較します。 - 遺伝子合成のためのGeneDesign 14(http://54.235.254.95/gd/)などのツールを使用して、希少なコドン豊富なマーカー遺伝子のビルディングブロックを生成します。あるいは、市販の遺伝子合成サービスからマーカー遺伝子を注文する。
- [GeneDesign] ページで、[ビルディング ブロックの設計(一定の長さの重なり)]を選択します。
- シーケンスボックスに希少なコドンが豊富なマーカー遺伝子の配列を貼り付けます。
- アセンブリオリゴ間のオーバーラップ長を定義します。デフォルトの40 bpは、ほとんどのシーケンスで正常に動作することを念頭に置いてください。
注: 他のパラメータの設定の詳細については、オンラインマニュアルを参照してください。 - [デザイン]ビルディングブロックボタンをクリックし、ページに記載されているオリゴヌクレオチドを並べ替えます。
- ポリメラーゼ連鎖反応(PCR)ベースの正確な合成15により希少コドン修飾遺伝子を合成する。
- 希少なコドンが豊富なカンR-RCをベクトルpET-28a、gfp-RCをpSB1C3に、ppg-RCをCPB-37-441 16にリゲートする。
注: pET-28a および pSB1C3 プラスミド マップは、SnapGene オンラインプラスミド データベース (補足表1) で入手できます。CPB-37-441プラスミドマップはATUM発色タンパク質ウェブサイトで入手できます。- 氷上で、組み立てミックスのモル比1:1~7.5 μLのベクトルとマーカーフラグメントを合計体積10μLに加えます(材料の表を参照)。サンプルを50°Cで1時間インキュベートします。
- 組立製品の5μLを50μLの有能な細胞(材料の表を参照)に30sの42 °Cで変換します。
- SOC(カタボライト抑圧付き超最適なスープ)培地で1時間37°Cの培地で細胞を回収し、LB(リソジェニーブロス)寒天培地にプレートし、一晩37°Cでインキュベートします。
- コロニーをLB培地に接種し、37°Cで8時間インキュベートします。
- 好ましい市販キットを用いてプラスミドを単離する。
2. 選択条件の最適化
- 有能な細胞17に変異形成に使用される親株を作る。
- 野生型カンRを運ぶプラスミドの1μLを有する有能な細胞の50 μLを変換し、希少なすべてのロイシンコドンを置き換えたすべてのロイシンコドンを含むプラスミドを有するプラスミドで別の有能な細胞のセットを変換するコドンCTA。
- SOC培地の950 μLを加え、1時間37°Cで250rpmでシェーカーでサンプルをインキュベートします。
- 50μg·mL-1カナマイシンを含むLB寒天培地上の細胞培養のプレート100μLを、コロニーが現れるまで約8時間37°Cでインキュベートする。
- 野生型カンRとロイシン希少コドンリッチカンR-RC29を入れたコロニーを選び、50μg mL-1カナマイシンを含む5倍希釈LB培地(0.2x LB)の5mLを接種します。37°Cで250 rpmでシェーカーでサンプルをインキュベートします。
注: メディアは、選択システムにとって非常に重要です。希少なコドン修飾誘導体からではなく、野生型遺伝子からの抗生物質耐性タンパク質の発現を可能にするのに十分な炭素と窒素を含む必要があります。この場合、1x LB培地中のL-ロイシン濃度は高すぎて、希少コドンが豊富なカンRからのタンパク質発現を完全に阻害する。したがって、5などの希釈因子を用いた希釈LB培地は、野生型と希少コドンが豊富な遺伝子からタンパク質発現の明確な違いを生成するために使用される。 - 各細胞培養物の200 μLを、定義された時点(例えば、8時間、16時間、24時間)で三分線状の96ウェルプレートに移す。プレートリーダーを使用してOD 600(600nmの光学密度)を測定します。
注:野生型マーカー遺伝子を保有する株のOD600と比較して希少コドンが豊富なマーカー遺伝子を持つ株に対して細胞OD600の減少が検出できない場合は、マーカー遺伝子またはより希釈された媒体を使用する。 - アミノ酸供給アッセイを行い、希少コドン豊富なマーカー遺伝子(例えば、カンR−RC29)の発現が標的アミノ酸の濃度を増加させることによって回復できるかどうかを試験する。
注:L-ロイシンオーバープロデューサーの選択のために、L-ロイシンは供給に使用されます。- カンR-RC29マーカー遺伝子を1gの供給の有無にかかわらず0.2x LB(50 μg·mL-1カナマイシンを含む)の5mLに接種する株を接種する·L-1 L-ロイシンコントロールとして野生型カンRを抱える株で0.2x LBの別の5 mLを接種します。37°Cで250 rpmでシェーカーでサンプルをインキュベートします。
- 定義された時点(例えば、15時間、17時間、19時間、22時間)で各培養のOD600を測定します。
3. 審査条件の最適化
- 野生型gfpまたは野生型ppgを運ぶプラスミドの1 μL(〜50 ng·μL-1)で変異形成に使用される親株の50 μLを変換する。また、マーカー遺伝子の希少コドン豊富な誘導体で親株を変換します。
- SOC培地の950 μLを加え、1時間37°Cで250rpmでシェーカーでサンプルをインキュベートします。
- 適切な抗生物質を含むLB寒天培地上の細胞培養物のプレート100μL(cm Rマーカーを運ぶgfpプラスミド用25μg·mL-1クロラムフェニコール、またはppgに対して50μg·mL-1カナマイシン) カンRマーカーを運ぶプラスミド)と37°Cで一晩インキュベートする。
- 野生型のgfpまたはppgを保有するコロニーと、対応する希少コドンリッチ誘導体を持つコロニーを1つ選び、適切に希釈されたLB培地の5mLに個別に移します。37°Cで250 rpmでシェーカーでサンプルをインキュベートします。
注:ロイシン希少コドンリッチgfp-RCまたはppg-RCを収容する大腸菌株の場合、希釈されていないLB培地(1x LB)を使用して、野生型および希少コドンリッチマーカーの表現に大きな違いを生み出すことができます。遺伝子。また、誘導性プロモーターは、スクリーニングマーカー遺伝子の発現を駆動するために使用される。細胞がより良い判分を達成するために指数相に入ったときに誘導を開始します。 - 蛍光マーカーの場合、各細胞培養物の200 μLを、定義された時点(例えば、2時間、4時間、6時間)で三分線状の96ウェル透明底部の黒板に移します。OD600および蛍光を測定し、蛍光強度(OD600に対する蛍光の比率)を計算する。発色マーカーの場合、細胞培養物の色の発達を測定する。
注:野生型gfpを保有する株と比較してgfp-RCを保有する株に対して低い蛍光強度が検出できない場合、または紫色のタンパク質を発現する細胞に対して明るい色が検出されない場合野生型遺伝子よりもppg-RCから、マーカー遺伝子上の希少なコドンの数を増やすか、より希釈された培地を使用してみてください。 - 給餌アッセイを実行します(手順 2.7.1 および 2.7.2 を参照)。定義された時点(例えば、12時間、18時間、24時間)における蛍光強度または色の発達を測定する。
4. アミノ酸過剰生産者の同定
- 変異体の培養物の100 μLをLB培地の5mL(2%v/v)に接種し、OD 600の値が0.4に達するまで37°Cで250rpmでシェーカーでインキュベートします。
- 有能な細胞に変異体を作る17.
- プラスミドの1μLを有する変異細胞の50μLを形質転換する(〜50ng·μL-1)選択マーカーkan R-RC29またはスクリーニングマーカーgfp-RCまたはppg-RCを運ぶ。SOC培地の950 μLを追加し、1時間37°Cシェーカーでサンプルを回転させます。
- アミノ酸オーバープロデューサーを選択します。.
- 5分間4,000xgで細胞培養を遠心分離し、上清を廃棄し、50μg·mL-1カナマイシンを含む0.2x LB培地の5 mLを添加する。一晩37°Cシェーカーでサンプルをインキュベートします。
- 一晩培養をプレート(例えば、100μL、培養の細胞密度に依存)を50μg·mL-1カナマイシンを含む0.2x LB寒天培地上に、12時間37°Cでインキュベートする。
注:開発されたコロニーは、標的アミノ酸過剰生産者および、この場合、L-ロイシン過剰生産者の候補である。
- アミノ酸オーバープロデューサーをスクリーニングします。
- 適切な数の細胞(例えば、100 μL)を、適切な抗生物質を含むLB寒天培地(25 μg·mL-1クロランフェニコール)にスクリーニングマーカーを収容し、gfp-RCおよび50 μg mLをスクリーニングする。-1 ppg-RC)でスクリーニングし、37°Cで8時間インキュベートする。
- 適切な抗生物質を含むLB培地を、プレートから各単一コロニーで接種する(ステップ4.5.1参照)。37°Cで250 rpmでシェーカーでサンプルをインキュベートします。
- OD600と蛍光を測定し、gfp-RCを使用する場合の蛍光強度を計算します。ppg-RCを使用する場合の細胞培養の色の発達を測定します。なお、親株よりも高い蛍光強度または深い色を示す株は、候補アミノ酸過剰生産者および、この場合、L-ロイシン過剰生産者である。
注:蛍光活性化細胞選別は、希少コドンが豊富な蛍光タンパク質遺伝子をスクリーニングに使用する場合、単細胞高生成物の同定にも適しています。
- 候補株のアミノ酸の産物性を確認する。
- 候補株のそれぞれでLB培地の5 mLを接種し、細胞を37°Cで250rpmでシェーカーで一晩成長させる。
- 細胞培養の1mLから4,000 x gで2分間遠心分離により細胞を収穫し、上清を捨て、滅菌水の1mLでペレットを再懸濁する。
- 細胞懸濁液の200μLを含むM9培地の20mLを接種し、250 mLシェーカーで250mLシェーカーで250°C、24時間24時間インキュベートする。
- 培養培地の遠心分離機1 mLを4,000 x gで5分間5分間、上清の200μLをきれいな1.5mLチューブに移す。0.01、0.05、0.1、0.5、および1 gのL-ロイシン溶液(HPLC(高性能液体クロマトグラフィー)グレード)を調製する標準としてL-1。
- 上清および標準に1mMトリエチルアミンの100 μLと1Mフェニルイソチオシアネートの100 μLを加え、それらを穏やかに混合し、1h18の室温でインキュベートする。
注意: トリエチルアミンとフェニルイソチオシアネートは、重度の皮膚火傷や眼の損傷を引き起こす可能性があり、吸入した場合に有害です.手袋とマスクを着用し、可能であれば、ヒュームフードでこのステップを実行します。 - 同じチューブに400 μLのn-ヘキサンを加え、10sのために渦を塗り、0.2μmのポリテトラフルオロエチレン膜を通してアミノ酸誘導体を含む下相を濾過する。
注意: n-ヘキサンは皮膚刺激を引き起こす可能性があります。手袋と防護服を着用してください。皮膚に触れたら、たっぷりの水で皮をすいでください。 - 0.1Mアセテート(pH 6.5)とアセトニトリルを99.3:0.7の体積比で混合して移動相Aを調作する。移動相Bとしてアセトニトリル(80%v/v)を調製し、0.2μmポリテトラフルオロエチレン膜を通してすべての移動相を濾過します。
注意:アセトニトリルは吸入すると有害であり、皮膚や目の炎症を引き起こす可能性があります。手袋と防護服を着用し、ヒュームフードでこのステップを実行します。 - 表1の溶出プログラムに従ってC18カラムを搭載した超HPLC上でサンプルの1 μLを実行し、流量は0.42 mL·min-1、カラム温度は40°Cです。ダイオードアレイ検出器で254nmの標的アミノ酸を検出し、ピーク領域を標準曲線にマッピングして濃度を計算します。
| 時間 (分) | モバイル フェーズ A (%) | モバイル フェーズ B (%) |
| 0 | 98歳 | 2 |
| 3.5年 | 70歳 | 30歳 |
| 7 | 43歳 | 57歳 |
| 7.1年 | 0 | 100人 |
| 11歳 | 98歳 | 2 |
表1:アミノ酸の定量のための溶出プログラム。
結果
選択システムの場合、希少コドンが豊富な抗生物質耐性遺伝子を保有する菌株に対するOD600の急激な減少は、適当に培養した場合に野生型抗生物質耐性遺伝子を保有する株と比較して観察されるべきである。ミディアム(図1a)。同じ条件下で、抗生物質耐性遺伝子の希少なコドンの数が増加するにつれて、細胞OD600の減少がより明らかになる(図1a)。タンパク質発現に対する希少なコドンの阻害は、主に飢餓状態下で起こる。従って、LB培地が適切に希釈されていない場合、野生型遺伝子を保有する株と比較して希少コドンリッチマーカー遺伝子を保有する株に対して細胞OD600の有意な減少は認められない(図1b)。対応するアミノ酸の余分な摂食後、希少コドン豊富な抗生物質耐性遺伝子を保有する菌株に対するOD600が有意に増加し、野生型遺伝子を保有する株のそれに近づく(図1c)。
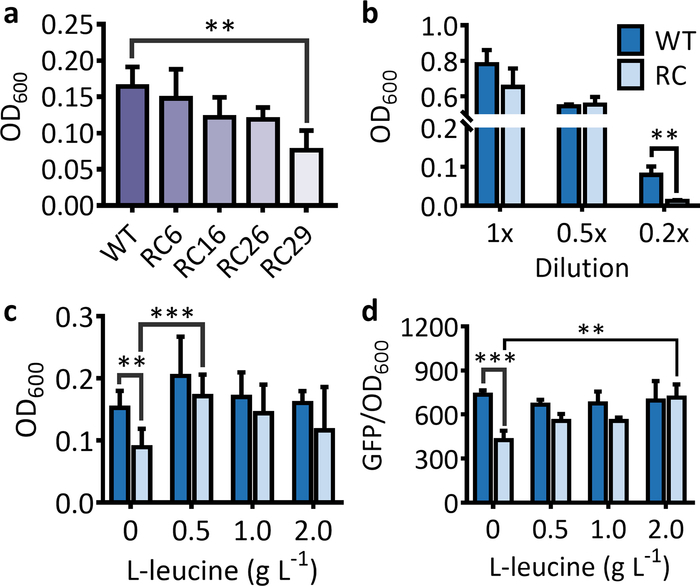
図1:希少コドンが選択およびスクリーニングシステムに用いるマーカー遺伝子の発現に及ぼす影響(a) 6、16、26、および29ロイシン希少コドン(RC6、RC16、RC26、およびRC29)を持つ抗生物質耐性遺伝子(kanR)を持つ大腸菌株に対する細胞OD600は、インキュベーションの5時間後に置換する。(b)5時間後に野生型(WT)および希少コドンリッチカンR(RC)を1x、0.5x、および0.2x LB培養後に保有する大腸菌株の細胞OD600。(c) インキュベーションの5時間後にロイシン希少コドンリッチカンR遺伝子を保有する大腸菌株の細胞増殖に対するL-ロイシンの摂食の効果。値と誤差余数は平均値と SD(n = 6) を表します。L-ロイシンの摂食は、希少なコドンが豊富なカンRを収容する細胞のOD600を有意に増加させた。唯一の例外は、2 g·摂食処理のためのOD600の高いSDによるL-1 L-ロイシン。(d) 16時間後のインキュベーション後の野生型(WT)およびロイシン希少コドンリッチ(RC)遺伝子からのGFP表現に対する希少コドンおよびL-ロイシン摂食の効果。0.5-2L-1 L-ロイシンの摂食は、希少なコドンが豊富なgfpを収容する細胞の蛍光強度を有意に増加させた。値と誤差余数は平均値と SD(n = 3) を表します。**P < 0.01, ***P < 0.001 は、両尾t-test によって決定され、最も有意な結果のみが示された。 この図のより大きなバージョンを表示するには、ここをクリックしてください。
スクリーニングシステムの場合、希少コドン豊富な遺伝子から蛍光タンパク質を発現する株に対して蛍光強度と蛍光細胞数が野生型遺伝子よりも有意に低くなる(図1dおよび図2)紫色のタンパク質を使用する場合、希少なコドンが豊富なppgから開発された色は、同じ潜伏期間に同じ条件下で発現した場合、野生型遺伝子の色よりも軽くすべきである(図3)。対応するアミノ酸を摂食すると、希少なコドンが豊富な遺伝子からタンパク質発現を回復します。希少コドンが豊富なgfpを持つ株の場合、蛍光強度(図1d)と蛍光細胞数(図2)が大幅に増加し、野生型gfpを含む株の方に近づく必要があります。 .希釈されていないLBを使用すると、培地中のアミノ酸は、余分なL-ロイシン供給なしでも希少コドン豊富なppgの発現を遅くするのに十分であり、発現した紫色のタンパク質は、細胞がペレット化されると見えてくる(図3)しかし、これは、希少なコドン豊富なppgからの遺伝子発現が2gにL-ロイシンを供給することによって劇的に増強されたという事実を隠すものではありません·L-1、特に液体培養で観察された場合(図3)。したがって、液体培養は発色タンパク質に基づくスクリーニングのためのより良い選択であり、希釈されたLB培地を使用すると、野生型と希少なコドンが豊富な遺伝子によって誘発される表現型の間により大きな違いをもたらすであろう。

図 2:L-ロイシンを添加した後に野生型gfpまたはロイシン希少コドンリッチgfp(gfp-RC)を保有する蛍光大腸菌細胞の数。細胞を1xLB培地で培養した。スケールバー = 20 μm.この図のより大きなバージョンを表示するには、ここをクリックしてください。

図3:1xLB培地(左パネル)で紫色タンパク質をコードする野生型(WT)とロイシン希少コドンリッチ(RC)ppg遺伝子を含む細胞の色開発と、L-ロイシン摂食が細胞培養色に及ぼす影響(右パネル)ppg遺伝子は、細胞が指数相に入り、画像が誘導後3時間で捕捉されたときに誘導された。L-ロイシンを摂食アッセイ中の誘導者と共に培地に添加した。色付きの円は、細胞培養物と細胞ペレットの色を選ぶことによって生成された。この図のより大きなバージョンを表示するには、ここをクリックしてください。
希少コドンベースの戦略は、変異ライブラリーから標的アミノ酸の過剰産生を同定することができ、これらの変異体は親株よりも高い量の標的アミノ酸を産生する必要があります(図4)。

図4:希少コドンベースの戦略によって同定された野生型および変異株によって産生されるアミノ酸。(a)カンR-RC29(EL-1からEL-5)および29および19ルーシン希少コドン(EL-6からEL-10)を保有するgfp-RCによって変異ライブラリーから同定された大腸菌株のL-ロイシン産生。(b) 8つのアルギニンレアコドン(AGG)を含む希少コドンリッチカンRによって選択されたコリネバクテリウムグルタミカム株のL-アルギニン産生。マーカー遺伝子は、野生型株ATCC13032に由来するC.グルタミカム変異ライブラリーに導入された。選択媒体は0.3倍CGIIIを25μg·mL-1カナマイシンで供給した。この図のより大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
マーカー遺伝子および選択またはスクリーニング媒体中の希少コドンの数は、希少コドン修飾マーカー遺伝子からのタンパク質発現を阻害するために重要である。野生型マーカー遺伝子とその誘導体からのタンパク質発現に有意な差が検出されない場合、希少なコドンの数を増やすか、または栄養制限培地を使用すると、その違いを増幅する可能性があります。しかしながら、阻害効果が強すぎると、対応するアミノ酸の余分な摂食によってもタンパク質発式が回復しない場合がある。この場合、マーカー遺伝子中の希少なコドンの数を減らしてストレスの一部を緩和する必要があります。選択またはスクリーニングのストリンジェンシーを微調整するもう 1 つの方法は、希少なコドンが豊富なマーカー遺伝子のコピー番号と発現レベルを調整することです。マーカー遺伝子のコピー数と発現レベルを減少させることは、通常、アミノ酸過剰生産者と初期株との間のより強い区別につながる。したがって、p15AやpSC101などの低コピー数複製起源を含むベクターと弱いプロモーターを使用すべきである。マーカー遺伝子が誘導性プロモーターによって駆動される場合、低誘導が推奨される。
アミノ酸過剰生産者の選択またはスクリーニングのための希少コドンベースの戦略は、外因性タンパク質の発式を促進することを目的とする「コドン最適化」の一般的に使用される戦略の逆適応である。コドン最適化では、標的遺伝子上の希少なコドンは、宿主に対する同義の一般的なものに置き換えられます。したがって、他の生物からの遺伝子は、希少なコドン19の割合が高い外因性遺伝子よりもタンパク質にはるかに迅速に翻訳することができる。したがって、共通のコドンを同義の希少なコドンに切り替える「逆最適化」は、遺伝子発現を阻害すべきであると考えるのが妥当である。しかし、標的アミノ酸が細胞内に蓄積すると、対応する希少tRNAの強化充電によって遺伝子発現を回復する必要があります。希少コドンの組み込みは、タンパク質発現におけるアミノ酸濃度の閾値を増加させ、適切なマーカー遺伝子と組み合わせるとアミノ酸オーバープロデューサーを選択またはスクリーニングする潜在的な戦略を提供する。
抗生物質耐性遺伝子、蛍光タンパク質遺伝子、およびプロトコルで使用される発色タンパク質遺伝子に加えて、様々なマーカー遺伝子を用いて、希少コドンベースの選択またはスクリーニングシステムを確立することができる。例えば、tolC 20およびsacB21のような致死的な遺伝子は、アミノ酸過剰生産者を選択するために使用することができる。この場合、解毒剤系に属する遺伝子上の一般的なコドンは、標的アミノ酸の同義的な希少コドンに置き換えられるべきである。標的アミノ酸を過剰産生する菌株は解毒剤系を起動し、したがって、致死的な遺伝子によって引き起こされる毒性効果を生き残ることができる。
摂食アッセイに多量のアミノ酸を使用すると副作用が生じることに留意すべきである。これは、一部のアミノ酸が微生物に有毒であるためです。例えば、約100mgの濃度·L-セリンのL-1は、大腸菌22の増殖を阻害することができる。しかし、野生型遺伝子よりも低いが、最大2gの摂食が見つかった。L-1 L-セリンは、セリン希少コドン13を豊富に含む抗生物質耐性遺伝子の発現を回復させることができる。したがって、アミノ酸毒性は、少なくともL-セリンに対して、摂食アッセイの信頼性を損なうものではない。標的株の生成性に対するアミノ酸毒性の潜在的な負の影響を克服するために、ランダム突然変異およびアミノ酸輸出の増強などの戦略23を適用することができる。実際には、希少コドンベースの方法は、毒性レベルを超えるアミノ酸に耐えたり過剰に産生したりすることができる耐性株を同定するのに適しています。アミノ酸耐性を付与する主要な突然変異を同定し、標的株に導入することができ、これはアミノ酸過剰生産者の構築のための理想的な宿主となるであろう。
希少コドンベースの選択またはスクリーニングシステムは高い忠実度を保障する。言い換えれば、システムによって同定された株は、標的アミノ酸の過剰生産者であると考えられる。しかし、場合によっては、抗生物質選択を生き残る候補は、親株よりも高い量の標的アミノ酸を産生することができない。これは、変異発生を介して株によって獲得された抗生物質耐性に起因し、次いで選択プラスミド24の損失に起因し得る。その結果、強化されたアミノ酸の産物性のない株は、抗生物質のストレスを生き残り、選択を逃れることができました。これらの偽陽性株は、別の抗生物質に対する耐性を付与する野生型遺伝子のような選択プラスミドに別の選択マーカーを挿入することによって排除することができる。選択プラスミドを失った株は、2つの抗生物質に対する二重耐性を得る可能性が低く、選択中に排除される。
希少コドンベースのシステムによって同定された変異体は、初期株と比較して標的アミノ酸を過剰産生することができるはずです。しかしながら、選択された株に対するアミノ酸の製品性は、依然として工業的要件よりも低い場合がある。これは、歪みの性能が選択またはスクリーニングプロセスから独立しているが、初期株の特性、変異形成のアプローチ、サイズなどの要因に依存するとして、希少コドンベースの戦略の失敗を示唆するものではありません。変異ライブラリ、および発酵条件。高産生株を得るためには、ランダムな変異やアミノ酸生合成経路の合理的な設計など、歪み工学の戦略に注意を払う必要があります。適応実験室の進化と希少なコドンベースの戦略を組み合わせることで、アミノ酸の過剰生産者の獲得が容易になります。
メチオニンとトリプトファンは、20 タンパク質アミノ酸の中で代替コドンを持っていません。.したがって、この戦略は、これらのアミノ酸に直接採用することはできません。可能な解決策の 1 つは、これらのアミノ酸を運ぶためにストップコドンを認識できるエンジニアリング tRNA を使用することです。したがって、対応するストップコドンは、これらのアミノ酸25、26の人工希少コドンとして採用することができる。
アミノ酸過剰生産者の選択のための従来のアナログベースの戦略に関する最大の欠点の一つは、高い偽陽性率5、27です。変異発生を通過する株は、毒性アミノ酸類似体に対する耐性を容易に獲得することができ、また、耐性は、変異原27の助けを借りずに獲得され得る。これらの株は、アミノ酸類似体からの選択圧力を容易に逃れることができ、その結果、選択された株は、通常、選択プロセスの効率を大きく犠牲にする真のアミノ酸過剰生産者ではない。
対照的に、希少コドンベースの戦略は、アミノ酸オーバープロデューサーの正確かつ迅速な同定を可能にすることにより、従来のアナログベースの方法を上回ります。私たちの知るところでは、これはコドンバイアスの自然法則を採用する最初の戦略です。それは単一の希少なコドン豊富なマーカー遺伝子に依存し、したがって、有毒なアナログの使用を排除します。マーカー遺伝子は一般に宿主株に対して無毒であり、希少コドン豊富な遺伝子からのタンパク質発現は、コドンバイアスの普遍的かつ厳格な法則のために、主に対応するアミノ酸の細胞内濃度に依存する。すべての種にわたって。これにより、株が選択圧力をエスケープするのを防ぐことができます。また、マーカー遺伝子の多様性が大きいため、希少コドンベースの戦略は、アミノ酸オーバープロデューサーの選択とスクリーニングの両方に様々な選択肢を提供することができます。
すべての生物におけるコドンバイアスの普遍的現象28により、希少コドンベースの選択またはスクリーニング戦略は、理論的には大腸菌以外の微生物、特に工業用微生物に用いられる可能性がある。電位。別の宿主に変更する場合、マーカー遺伝子の設計に使用される希少なコドンの選択は、特定の宿主に対するコドン使用頻度および対応するtRNAの豊富さに基づくものでなければならない。選択またはスクリーニングに使用される媒体もそれに応じて最適化する必要があります。一例は、アミノ酸発酵において一般的に使用されるC.グルタミカムである。8つのアルギニン希少コドン(AGG)を含む希少コドン修飾カンR遺伝子は、以前の研究13(図4b)によってC.グルタミカムL-アルギニン過剰生産者を選択する際に有効であることが示されている。希少コドンベースの戦略の探求は、アミノ酸過剰生産者の構造と理解を容易にする必要があります。アミノ酸に加えて、希少コドンベースの戦略は、イソブタノール、3-メチル-1-ブタノール、2-メチル-1-ブタノール、および特定のアミノ酸と同じ生合成経路を共有する他の製品を使用することができます29。これらのアミノ酸の希少なコドンを保有するマーカー遺伝子によって同定された株は、前駆体化合物を過剰に産生することができ、これはアミノ酸誘導体の合成に導くことができる。したがって、希少コドンベースの戦略は、これらの化学物質を細胞内または細胞外に蓄積する上で株の可能性を反映する間接的かつ迅速な方法として役立つ可能性があります。様々な過剰生産者からのアミノ酸の産物性を増大させる主要な突然変異は、深いシーケンシングによって同定され、さらにアミノ酸産生を改善するために、個別または同時に工業株に導入することができる。
開示事項
著者は何も開示していない。
謝辞
この研究は、中国国家自然科学財団(助成金第21676026号)、中国国家鍵研究開発プログラム(助成金第2017YFD0201400)、中国ポストドクター科学財団(助成第2017M620643号)が共同で支援した。UCLA進歩研究所(蘇州)の作品は、江蘇省と蘇州工業団地からの内部助成金によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Thermo | 51101 | |
| EasyPure HiPure Plasmid MiniPrep Kit | Transgen | EM111-01 | |
| EasyPure Quick Gel Extraction Kit | Transgen | EG101-01 | |
| Gibson assembly master mix | NEB | E2611S | |
| Isopropyl β-D-1-thiogalactopyranoside | Solarbio | I8070 | |
| L-leucine | Sigma | L8000 | |
| Microplate reader | Biotek | Synergy 2 | |
| n-hexane | Thermo | H3061 | |
| Phenyl isothiocyanate | Sigma | P1034 | |
| PrancerPurple CPB-37-441 | ATUM | CPB-37-441 | |
| TransStar FastPfu Fly DNA polymerase | Transgen | AP231-01 | |
| Triethylamine | Sigma | T0886 | |
| Ultra-high performance liquid chromatography | Agilent | 1290 Infinity II | |
| Wild type C. glutamicum | ATCC | 13032 | |
| XL10-Gold E. coli competent cell | Agilent | 200314 | |
| ZORBAX RRHD Eclipse Plus C18 column | Agilent | 959759-902K |
参考文献
- Tatsumi, N., Inui, M. . Corynebacterium glutamicum: biology and biotechnology. , (2012).
- Tonouchi, N., Ito, H., Yokota, A., Ikeda, M. Present global situation of amino acids in industry. Amino Acid Fermentation. , 3-14 (2017).
- Gusyatiner, M., Lunts, M., Kozlov, Y., Ivanovskaya, L., Voroshilova, E. . DNA coding for mutant isopropylmalate synthase, L-leucine-producing microorganism and method for producing L-leucine. , (2005).
- Park, J. H., Lee, S. Y. Towards systems metabolic engineering of microorganisms for amino acid production. Current Opinion in Biotechnology. 19 (5), 454-460 (2008).
- Norris, R., Lea, P. The use of amino acid analogues in biological studies. Science Progress. , 65-85 (1976).
- Park, J. H., Lee, S. Y. Fermentative production of branched chain amino acids: a focus on metabolic engineering. Applied Microbiology and Biotechnology. 85 (3), 491-506 (2010).
- Bach, T. M., Takagi, H. Properties, metabolisms, and applications of L-proline analogues. Applied Microbiology and Biotechnology. 97 (15), 6623-6634 (2013).
- Crick, F. H. C. On the genetic code. Science. 139 (3554), 461-464 (1963).
- Plotkin, J. B., Kudla, G. Synonymous but not the same: the causes and consequences of codon bias. Nature Reviews Genetics. 12 (1), 32-42 (2011).
- Dittmar, K. A., Sørensen, M. A., Elf, J., Ehrenberg, M., Pan, T. Selective charging of tRNA isoacceptors induced by amino‐acid starvation. EMBO Reports. 6 (2), 151-157 (2005).
- Elf, J., Nilsson, D., Tenson, T., Ehrenberg, M. Selective charging of tRNA isoacceptors explains patterns of codon usage. Science. 300 (5626), 1718-1722 (2003).
- Sørensen, M. A. Charging levels of four tRNA species in Escherichia coli Rel+ and Rel− strains during amino acid starvation: a simple model for the effect of ppGpp on translational accuracy. Journal of Molecular Biology. 307 (3), 785-798 (2001).
- Zheng, B., et al. Utilization of rare codon-rich markers for screening amino acid overproducers. Nature Communications. 9 (1), 3616 (2018).
- Richardson, S. M., Wheelan, S. J., Yarrington, R. M., Boeke, J. D. GeneDesign: rapid, automated design of multikilobase synthetic genes. Genome Research. 16 (4), 550-556 (2006).
- Xiong, A. -. S., et al. PCR-based accurate synthesis of long DNA sequences. Nature Protocols. 1 (2), 791-797 (2006).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Green, M. R., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Cohen, S. A., Bidlingmeyer, B. A., Tarvin, T. L. PITC derivatives in amino acid analysis. Nature. 320 (6064), 769-770 (1986).
- Zhou, J., Liu, W. J., Peng, S. W., Sun, X. Y., Frazer, I. Papillomavirus capsid protein expression level depends on the match between codon usage and tRNA availability. Journal of Virology. 73 (6), 4972-4982 (1999).
- Gregg, C. J., et al. Rational optimization of tolC as a powerful dual selectable marker for genome engineering. Nucleic Acids Research. 42 (7), 4779-4790 (2014).
- Pelicic, V., Reyrat, J. M., Gicquel, B. Expression of the Bacillus subtilis sacB gene confers sucrose sensitivity on mycobacteria. Journal of Bacteriology. 178 (4), 1197-1199 (1996).
- Avcilar-Kucukgoze, I., et al. Discharging tRNAs: a tug of war between translation and detoxification in Escherichia coli. Nucleic Acids Research. 44 (17), 8324-8334 (2016).
- Mundhada, H., Schneider, K., Christensen, H. B., Nielsen, A. T. Engineering of high yield production of L-serine in Escherichia coli. Biotechnology and Bioengineering. 113 (4), 807-816 (2016).
- Makosky, P. C., Dahlberg, A. E. Spectinomycin resistance at site 1192 in 16S ribosomal RNA of E. coli: an analysis of three mutants. Biochimie. 69 (8), 885-889 (1987).
- Feng, L., Tumbula-Hansen, D., Toogood, H., Söll, D. Expanding tRNA recognition of a tRNA synthetase by a single amino acid change. Proceedings of the National Academy of Sciences of the United States of America. 100 (10), 5676-5681 (2003).
- Naganuma, M., et al. The selective tRNA aminoacylation mechanism based on a single G• U pair. Nature. 510 (7506), 507 (2014).
- Hoesl, M. G., et al. Chemical evolution of a bacterial proteome. Angewandte Chemie International Edition. 54 (34), 10030-10034 (2015).
- Hershberg, R., Petrov, D. A. Selection on codon bias. Annual Review of Genetics. 42, 287-299 (2008).
- Huo, Y. -. X., et al. Conversion of proteins into biofuels by engineering nitrogen flux. Nature Biotechnology. 29, 346 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved