Method Article
Identificare gli amminoacidi sovraproduttori utilizzando marcatori rari-codoni-ricchi
In questo articolo
Riepilogo
Questo studio presenta una strategia alternativa al metodo tradizionale basato sull'analogico tossico nell'identificazione di sovraproduttori di amminoacidi utilizzando marcatori rari ricchi di codoni per ottenere contemporaneamente precisione, sensibilità e alta produttività.
Abstract
Per soddisfare il mercato in continua crescita per gli amminoacidi, sono necessari ceppi di produzione ad alte prestazioni. I prodotti non amnopesonoari sono convenzionalmente identificati sfruttando i concorsi tra gli amminoacidi e i loro analoghi. Tuttavia, questo metodo basato su analogico è di bassa precisione e gli analoghi appropriati per gli amminoacidi specifici sono limitati. Qui, presentiamo una strategia alternativa che consente uno screening accurato, sensibile e ad alta velocità di produzione di amminoacidi utilizzando marcatori rari ricchi di codoni. Questa strategia è ispirata al fenomeno della distorsione dell'uso del codone nella traduzione delle proteine, per la quale i codoni sono classificati in comuni o rari in base alle loro frequenze di occorrenza nel DNA codificante. La traduzione di codoni rari dipende dai corrispondenti RNA di trasferimento rari (tRNA), che non possono essere completamente addebitati dagli amminoacidi cognati in condizioni di fame. Teoricamente, i tRNA rari possono essere addebitati se c'è un surplus di aminoacidi dopo aver caricato i sinonimi isoacceptors comuni. Pertanto, le traduzioni ritardate causate da codoni rari potrebbero essere ripristinate alimentando o sovrapproduzione intracellulari degli amminoacidi corrispondenti. In base a questa ipotesi, viene istituito un sistema di selezione o screening per l'identificazione dei sovraproduttori di amminoacidi sostituendo i codoni comuni degli amminoacidi mirati con le loro alternative rare sinonimi nei geni di resistenza agli antibiotici o nei geni codificare proteine fluorescenti o cromogene. Dimostriamo che le espressioni proteiche possono essere notevolmente ostacolate dall'incorporazione di codoni rari e che i livelli di proteine sono correlati positivamente con le concentrazioni di amminoacidi. Utilizzando questo sistema, gli sovraproduttori di aminoacidi multipli possono essere facilmente esaminati dalle librerie di mutazioni. Questa strategia basata su codone rara richiede solo un singolo gene modificato, e l'ospite è meno probabile che sfuggirà alla selezione rispetto ad altri metodi. Offre un approccio alternativo per ottenere i non amminoacidi sovraproduttori.
Introduzione
L'attuale produzione di amminoacidi dipende fortemente dalla fermentazione. Tuttavia, i titoli e le rese per la maggior parte dei ceppi di produzione di amminoacidi sono al di sotto delle crescenti richieste del mercato globale degli amminoacidi che vale miliardi di dollari1,2. Ottenere prodotti aminoacidi ad alte prestazioni sono fondamentali per l'aggiornamento dell'industria degli amminoacidi.
La strategia tradizionale per identificare i sovraproduttori di amminoacidi sfrutta i concorsi tra aminoacidi e i loro analoghi nella sintesi proteica3,4. Questi analoghi sono in grado di caricare i tRNA che riconoscono gli aminoacidi corrispondenti e quindi inibiscono gli allungamenti delle catene di peptidi, portando alla crescita arrestata o alla morte cellulare5. Un modo per resistere alle sollecitazioni analogiche è quello di aumentare le concentrazioni di aminoacidi intracellulari. Gli amminoacidi arricchiti supereranno gli analoghi per i tRNA finiti e garantiranno la corretta sintesi delle proteine funzionali. Pertanto, i ceppi che sopravvivono agli analoghi possono essere selezionati e sono probabilmente i sovraproduttori degli amminoacidi corrispondenti.
Sebbene si sia dimostrata efficace nella selezione di sovraproduttori di amminoacidi come L-leucina6, la strategia analogica soffre di gravi inconvenienti. Una delle principali preoccupazioni è la resistenza analogica originatadal processo di mutagenesi o attraverso mutazioni spontanee. I ceppi di resistenza possono sfuggire alla selezione bloccando, esportando o degradando gli analoghi5. Un'altra preoccupazione sono gli effetti collaterali tossici degli analoghi su altri processi cellulari7. Di conseguenza, i ceppi che sopravvivono alla selezione analogica potrebbero non essere i sovraproduttori di amminoacidi, mentre gli overproduttori desiderati potrebbero essere falsamente sterminati a causa degli effetti collaterali negativi.
Qui viene presentata una nuova strategia basata sulla legge del pregiudizio del codone al fine di ottenere identificazioni accurate e rapide dei sovraproduttori di amminoacidi. La maggior parte degli amminoacidi sono codificati da più di una tripletta nucleotide che è favorita in modo diverso dagli organismi ospiti8,9. Alcuni codoni sono raramente utilizzati nelle sequenze di codifica e sono indicati come i codoni rari. Le loro traduzioni in amminoacidi si basano sui tRNA cognati che trasportano gli aminoacidi corrispondenti. Tuttavia, i tRNA che riconoscono codoni rari di solito hanno abbondanza molto più basse rispetto ai tRNA dei codoni comuni10,11. Di conseguenza, questi rari tRNA hanno meno probabilità di catturare gli amminoacidi liberi nei concorsi con altri isoaccettatori, e le traduzioni delle sequenze rare ricche di codoni iniziano a rallentare o addirittura vengono terminate quando le quantità di aminoacidi sono limitate 10. Le traduzioni potrebbero, teoricamente, essere ripristinate se c'è un surplus di amminoacidi dopo aver caricato i sinonimi tRNA comuni a causa di sovraproduzioni o alimentamenti supplementari degli amminoacidi corrispondenti12. Se il gene raro ricco di codone codifica un marcatore di selezione o di screening, i ceppi che esibiscono i fenotipi corrispondenti possono essere facilmente identificati e sono probabilmente i sovraproduttori degli amminoacidi mirati.
La strategia di cui sopra viene applicata per stabilire una selezione e un sistema di screening per l'identificazione dei sovraproduttori di amminoacidi. Il sistema di selezione utilizza come marcatori i geni di resistenza agli antibiotici (ad esempio, kanR),mentre il sistema di screening utilizza le proteine fluorescenti di codifica dei geni (ad esempio, proteine fluorescenti verdi [GFP]) o cromogeniche (ad esempio, PrancerPurple). I geni marcatori in entrambi i sistemi vengono modificati sostituendo il numero definito dei codoni comuni per l'amminoacido mirato con la sua rara alternativa sinonimo. I ceppi nella libreria di mutazioni che ospitano il gene marcatore raro-codon-ricco sono selezionati o sottoposti a screening in condizioni adeguate, e gli overproduttori degli amminoacidi mirati possono essere facilmente identificati. Il flusso di lavoro inizia con la costruzione del sistema genetico marcatore raro-codon-ricco, seguita dall'ottimizzazione delle condizioni di lavoro, e quindi con l'identificazione e la verifica degli amminoacidi sovraproduttori. Questa strategia analogico-indipendente si basa sul dogma nella traduzione delle proteine ed è stata praticamente verificata per consentire identificazioni accurate e rapide dei sovraproduttori di amminoacidi. Teoricamente, potrebbe essere impiegato direttamente agli amminoacidi con codoni rari e a tutti i microrganismi. In tutto, la strategia basata su codone raro servirà come un'alternativa efficiente all'approccio analogico convenzionale quando non sono disponibili analoghi appropriati per specifici amminoacidi, o quando un alto tasso di falsi positivi è la preoccupazione principale. Il protocollo seguente utilizza codone raro leucina per dimostrare questa strategia nell'identificazione di Escherichia coli L-leucine iperproduttori.
Protocollo
1. Costruzione dei plasmidi che esprimono i geni marcatori rari-codoni-ricchi
- Selezionare un gene marcatore contenente un numero appropriato di codoni comuni per l'amminoacido mirato.
NOTA: Per L-leucine, il gene di resistenza alla kanamicina kanR, che contiene 29 codoni di leucina, di cui 27 sono codoni comuni, viene utilizzato per la costruzione del sistema di selezione13. Il gene gfp, che contiene 17 codoni comuni su 19 codoni leule, o il gene di codifica delle proteine viola prancerpurple (ppg), che ospita 14 codoni comuni di leucina, viene utilizzato per il sistema di screening (supplementari tabella 1 ). - Sostituire i codoni comuni nei geni marcatori con il codone raro sinonimo. Per L-leucine, sostituire i suoi codoni in kanR, gfpo ppg con il raro codon CTA, generando kan R-R-RCs, gfp-RCo ppg-RC, rispettivamente13 ( Tabella supplementare 1).
NOTA: La frequenza del codone raro nei geni marcatori influenzerà la severità del sistema di selezione o screening. In generale, l'aumento del numero di codoni rari aumenterà la severità del sistema di selezione o screening. Per ottenere la giusta forza di selezione o screening, progettare una serie di geni marcatori che ospitano diversi numeri di codoni rari e confrontarne gli effetti. - Genera elementi costitutivi dei geni marcatori rari ricchi di codoni utilizzando strumenti come GeneDesign14 (http://54.235.254.95/gd/) per la sintesi genica. In alternativa, ordinare i geni marcatori dai servizi di sintesi genica commerciale.
- Nella pagina GeneDesign, scegliete Progettazione blocchi predefiniti (sovrapposizione lunghezza costante).
- Incollate le sequenze dei geni marcatori rari-codoni nella casella Sequenza.
- Definire la lunghezza di sovrapposizione tra gli oligos dell'assieme; tenere presente che il valore predefinito 40 bp funziona bene per la maggior parte delle sequenze.
NOTA: Consultare il manuale online per ulteriori istruzioni sulle impostazioni degli altri parametri. - Fare clic sul pulsante Progetta blocchi predefiniti e ordinare gli oligonucleotidi elencati nella pagina.
- Sintetizza i geni raramente modificati dal sistema di reazione a catena della polimerasi (PCR) sintesi accurata15.
- Ligate il raro-codon-rich kanR-RC al vettore pET-28a, gfp-RC a pSB1C3, e ppg-RC a CPB-37-44116.
NOTA: Le mappe pET-28a e pSB1C3 plasmid sono disponibili nel database di plasmide online SnapGene (tabella supplementare 1); la mappa plasmica CPB-37-441 è disponibile sul sito web delle proteine cromogeniche ATUM.- Sul ghiaccio, aggiungere il vettore e i frammenti del marcatore in un rapporto molare di 1:1 a 7,5 l di miscela di assemblaggio (vedere Tabella dei materiali) ad un volume totale di 10 l. Incubare il campione a 50 gradi centigradi per 1 h.
- Trasformare 5 -L del prodotto di assemblaggio in 50 -L di celle competenti (vedi Tabella deiMateriali) a 42 gradi centigradi per 30 s.
- Recuperare le cellule nel SOC (brodo super ottimale con repressione del catabolito) medio a 37 gradi centigradi per 1 h, piastrasu LB (brodo di lisogenia) mezzo di agar, e incubare a 37 gradi durante la notte.
- Inoculare la colonia nel mezzo LB e incubare a 37 gradi centigradi per 8 h.
- Isolare il plasmide utilizzando un kit commerciale preferito.
2. Ottimizzazione delle condizioni di selezione
- Rendere il ceppo genitore utilizzato per la mutagenesi in cellule competenti17.
- Trasformare 50 l delle celle competenti con 1 l of plasmid che trasporta il tipo selvaggio kanRe trasformare un altro insieme di celle competenti con il plasmide contenente kan R-RC29 con tutto il codone leucina sostituito dal raro codon CTA.
- Aggiungere 950 l di MEZZO SOC e incubare il campione in uno shaker a 250 giri/min a 37 gradi centigradi per 1 ora.
- Piastra 100 -L della coltura cellulare sul mezzo agar LB contenente 50 g-mL-1 kanamycin, e incubarlo a 37 gradi centigradi per circa 8 h fino a quando non appaiono colonie.
- Scegli le colonie che ospitano il kanR di tipo selvaggio e il kanR-RC29, ricco di codoni e usali per inoculare 5 mL di cinque volte mezzo LB diluito (0,2x LB) contenente 50 g-mL-1 kanamycin. Incubare i campioni in uno shaker a 250 giri/min a 37 gradi centigradi.
NOTA: il supporto è fondamentale per il sistema di selezione. Dovrebbe contenere abbastanza carbonio e azoto per consentire l'espressione della proteina di resistenza agli antibiotici dal gene wild-type piuttosto che dai derivati rari-codoni-modificati. In questo caso, la concentrazione di L-leucina nel mezzo 1x LB è troppo alta per inibire completamente l'espressione proteica dal kan Rraro-ricco di codone . Pertanto, un mezzo LB diluito con un fattore di diluizione come 5 viene utilizzato per generare una chiara differenza nelle espressioni proteiche dal tipo selvatico e dai geni rari-ricchi di codoni. - Trasferire 200 l di ciascuna coltura cellulare su una piastra di 96 pozzetto in triplice copia a punti temporali definiti (ad esempio, 8 h, 16 h e 24 h). Misurare l'OD600 (densità ottica a 600 nm) utilizzando un lettore di lamiere.
NOTA: Se non è possibile rilevare una diminuzione della cellula OD600 per i ceppi che ospitano i geni marcatori rari-codoni-ricchi rispetto all'OD600 del ceppo che ospita i geni marcatori di tipo selvatico, cercare di aumentare la quantità di codone raro nel o utilizzare un mezzo più diluito. - Eseguire il test dell'alimentazione degli amminoacidi per verificare se le espressioni dei geni marcatori rari-codoni-ricchi (ad esempio, kanR-RC29) possono essere ripristinate aumentando la concentrazione degli amminoacidi mirati.
NOTA: Per la selezione dei produttori di L-leucina, l-leucina viene utilizzata per l'alimentazione.- Inoculare i ceppi che ospitano il gene del marcatore kanR-RC29 in 5 mL di 0,2x LB (contenenti 50 g-mL-1 kanamycin) con o senza la fornitura di 1 g L-1 L-leucina. Inoculare altri 5 mL dello 0,2x LB con ceppi che ospitano il kanR di tipo selvaggio come controllo. Incubare i campioni in uno shaker a 250 giri/min a 37 gradi centigradi.
- Misurare l'OD600 per ogni coltura in punti temporali definiti (ad esempio, 15 h, 17 h, 19 h e 22 h).
3. Ottimizzazione delle condizioni di screening
- Trasformare 50 -L del ceppo padre utilizzato per la mutagenesi con 1 -L di plasmide (50ng-L -1) che trasporta il gfp di tipo selvatico o il tipo selvaggio ppg. Inoltre, trasformare la deformazione padre con i derivati rari-ricchi di codone dei geni marcatori.
- Aggiungere 950 l di MEZZO SOC e incubare il campione in uno shaker a 250 giri/min a 37 gradi centigradi per 1 ora.
- Piastra 100 L della coltura cellulare sul supporto agar LB contenente gli antibiotici appropriati (25 g-mL-1 clorambionico per il plasmide gfp che trasporta un marcatore R cm, o 50 g-mL -1 kanamycin per il ppg un indicatore kan R) e incubare durante la notte a 37 gradi centigradi.
- Scegli una colonia che ospita il tipo selvaggio gfp o ppg e una colonia che ospita il corrispondente derivato raro-ricco di codone, e trasferiscili a 5 mL del mezzo LB correttamente diluito individualmente. Incubare i campioni in uno shaker a 250 giri/min a 37 gradi centigradi.
NOTA: Per E. coli ceppi che ospitano la leucina rara-codon-ricca gfp-RC o ppg-RC,il mezzo LB non diluito (1x LB) può essere utilizzato per creare differenze significative nelle espressioni del tipo selvatico e il marcatore raro-codon-ricco Geni. Inoltre, i promotori inducibili vengono utilizzati per guidare le espressioni dei geni marcatori di screening. Iniziare l'induzione quando le cellule entrano nella fase esponenziale per ottenere migliori discriminazioni. - Per i marcatori di fluorescenza, trasferire 200 L di ciascuna coltura cellulare in una piastra nera a fondo chiaro 96 in triplice copia a punti temporali definiti (ad esempio, 2 h, 4 h e 6 h). Misurare l'OD600 e la fluorescenza e calcolare l'intensità della fluorescenza (il rapporto tra fluorescenza e OD600). Per i marcatori cromogenici, misurare lo sviluppo del colore delle colture cellulari.
NOTA: Se non è possibile rilevare intensità di fluorescenza inferiori per i ceppi che ospitano il gfp-RC rispetto a quello dei ceppi che ospitano il tipo selvaggio gfp, o se non è possibile rilevare un colore più chiaro per le cellule che esprimono la proteina viola dal ppg-RC che dal gene wild-type, cercare di aumentare il numero di codon rari sui geni marcatori o utilizzare un mezzo più diluito. - Eseguire il provvedimento di alimentazione (vedere i passaggi 2.7.1 e 2.7.2). Misurare l'intensità della fluorescenza o lo sviluppo del colore in punti temporali definiti (ad esempio, 12 h, 18 h e 24 h).
4. Identificazione dei sovraproduttori di amminoacidi
- Inoculare 100 - L della cultura dei mutanti in 5 mL di LB medio (2% v/v) e incubarlo in uno shaker a 250 rpm a 37 s fino a quando i valori di OD600 raggiungono 0,4.
- Rendere i mutanti in cellule competenti17.
- Trasformare 50 l delle cellule mutanti con 1 L del plasmide (50 ng-L-1) portando il marcatore di selezione kanR-RC29 o i marcatori di screening gfp-RC o ppg-RC. Aggiungere 950 l di mezzo SOC e ruotare il campione in uno shaker a 37 gradi centigradi per 1 h.
- Selezionare i prodotti amnominoacidi.
- Centrifugare la coltura cellulare a 4.000 x g per 5 min, eliminare il supernatante e aggiungere 5 mL di 0,2x lB medio contenente 50 g-mL-1 kanamycin. Incubare il campione in uno shaker a 37 gradi centigradi.
- Piastrare la coltura durante la notte (ad es., 100 l, dipende dalla densità cellulare della coltura) su un mezzo agar da 0,2 x LB contenente 50 -g-mL-1 kanamicina e incubare a 37 gradi centigradi per 12 h.
NOTA: Le colonie sviluppate sono i candidati degli amminoacidi mirati sovraproduttori e, in questo caso, gli sovraproduttori di L-leucina.
- Scherma i sovranni di amminoacidi.
- Piastrare il numero appropriato di cellule (ad es., 100 l) che ospitano il marcatore di screening (come descritto al punto 4.3) sul supporto lB agar contenente l'antibiotico appropriato (25 g-mL-1 cloramhenicol per lo screening con gfp-RC e 50 g-mL -1 kanamicicina per lo screening con ppg-RC) e incubare a 37 gradi centigradi per 8 h.
- Inoculare il supporto LB contenente l'antibiotico appropriato (vedere il passaggio 4.5.1) con ogni singola colonia dalla piastra. Incubare i campioni in uno shaker a 250 giri/min a 37 gradi centigradi.
- Misurare l'OD600 e la fluorescenza e calcolare l'intensità della fluorescenza se viene utilizzato gfp-RC. Misurare lo sviluppo del colore delle colture cellulari se si utilizza ppg-RC. Si noti che i ceppi che presentano una maggiore intensità di fluorescenza o un colore più profondo di quello del ceppo genitore sono i prodotti aminoacidi candidati e, in questo caso, gli overproduttori L-leucine.
NOTA: Lo smistamento cellulare attivato dalla fluorescenza è adatto anche per identificare i produttori alti monocellulari quando i rari geni delle proteine fluorescenti ricchi di codoni vengono utilizzati per lo screening.
- Verificare la prodottisia degli amminoacidi dei ceppi candidati.
- Inoculare 5 mL di lB medio con ciascuno dei ceppi candidati e lasciare che le cellule crescono durante la notte in uno shaker a 250 giri/mm a 37 .
- Raccogliere le cellule da 1 mL della coltura cellulare con centrifugazione a 4.000 x g per 2 min. Scartare il supernatante e riattendere il pellet con 1 mL di acqua sterile.
- Inoculare 20 mL di mezzo M9 contenente il 4% di glucosio con 200 l della sospensione cellulare e incubare in uno shaker da 250 mL a 250 giri/ma a 37 gradi centigradi per 24 ore.
- Centrifuga 1 mL del mezzo di coltura a 4.000 x g per 5 min. Trasferire 200 l del supernatante in un tubo pulito da 1,5 mL. Preparare soluzioni L-leucine (HPLC (cromatografia liquida ad alte prestazioni) di 0,01, 0,05, 0,1, 0,5 e 1 g L-1 come standard.
- Aggiungete 100 luna di 1 mM di triethylamine e 100-L di 1 M di isothiocyanate fenile al super-nato e agli standard, mescolarli delicatamente e incubarli a temperatura ambiente per 1 h18.
AVVISO: La trietilamina e l'isothiocyanato fenilpossono causare gravi ustioni cutanee e danni agli occhi ed è dannoso se inalato. Indossare guanti e una maschera e, se possibile, eseguire questo passaggio in un cappuccio fumi. - Aggiungete 400 l di n-exane allo stesso tubo e vortice per 10 s. Filtrare la fase inferiore contenente i derivati degli amminoacidi attraverso una membrana di politetrafluoroetilene di 0,2 m.
AVVISO: n-exane può causare irritazione della pelle. Indossare guanti e indumenti protettivi. Se viene a contatto con la pelle, sciacquare la pelle con molta acqua. - Preparare la fase a mobile mescolando acetato di sodio da 0,1 M (pH 6,5) e acetonitrile in un rapporto volumetrico 99.3:0.7. Preparare l'acetonitrile (80% v/v) come fase mobile B. Filtrare tutte le fasi mobili attraverso le membrane di politetrafluoroetilene da 0,2 m.
AVVISO: L'acetonitricolo è dannoso se inalato e può causare irritazioni cutanee e agli occhi. Indossare guanti e indumenti protettivi ed eseguire questo passaggio in una cappa di fumi. - Eseguire 1 l del campione su un ultra-HPLC dotato di una colonna C18 secondo il programma di elusione nella tabella 1 con una portata di 0,42 ml-min-1 e una temperatura della colonna di 40 gradi centigradi. Rileva gli amminoacidi mirati a 254 nm con un rilevatore di array di diodi e calcola le loro concentrazioni mappando le aree di picco alla curva standard.
| Tempo (min) | Fase Mobile A (%) | Fase mobile B (%) |
| 0 (in vie | 98 (di base) | 2 Il nome del sistema |
| 3.5 3: | 70 del sistema di | 30 milio |
| 7 (in questo stato | 43 (di cine) | 57 del sistema |
| 7.1 La | 0 (in vie | 100 del sistema |
| 11 Del sistema di | 98 (di base) | 2 Il nome del sistema |
Tabella 1: Programma di elusione per la quantificazione degli amminoacidi.
Risultati
Per il sistema di selezione, si osserva una forte diminuzione dell'OD600 per i ceppi che ospitano il gene di resistenza agli antibiotici, ricco di codoni rari, rispetto al ceppo che ospita il gene di resistenza agli antibiotici di tipo selvatico quando viene coltivato in un medio(Figura 1a). Nelle stesse condizioni, la diminuzione della cellula OD600 diventa più evidente con l'aumentare del numero di codoni rari nel gene di resistenza agli antibiotici (Figura 1a). Va notato che l'inibizione del codone raro sulle espressioni proteiche avviene principalmente in condizioni di fame. Pertanto, se il mezzo LB non viene diluito correttamente, non si osserverà una diminuzione significativa della cellula OD600 per il ceppo che ospita il gene marcatore raro-codon-ricco rispetto al ceppo che ospita il gene di tipo selvaggio (Figura 1b). Dopo l'alimentazione supplementare dell'amminoacido corrispondente, l'OD600 per il ceppo che ospita il gene di resistenza agli antibiotici raro-ricco di codoni aumenterà in modo significativo e si avvicinerà a quello del ceppo che ospita il gene di tipo selvaggio (Figura 1c).
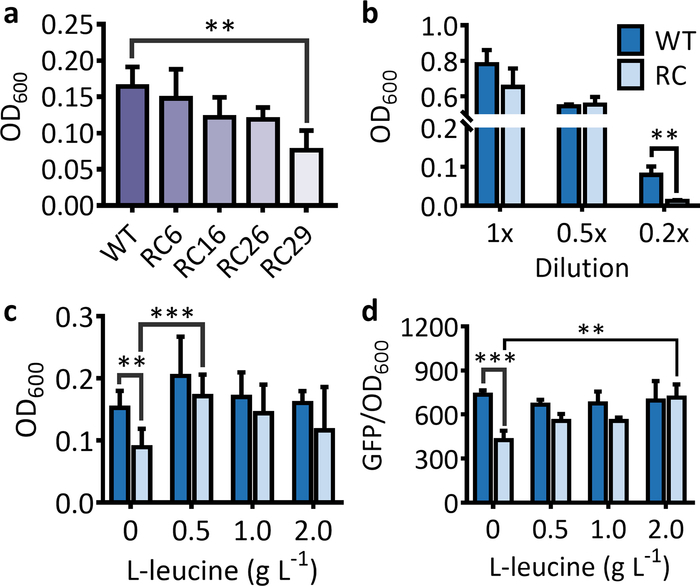
Figura 1: Effetti del codone raro sulle espressioni dei geni marcatori utilizzati per i sistemi di selezione e screening. (a) La cellula OD600 per un ceppo di E. coli che ospita il gene di resistenza agli antibiotici ( kanR) con 6, 16, 26 e 29 leucina rare-codon (RC6, RC16, RC26 e RC29) sostituisce dopo 5 h di incubazione. (b) La cella OD600 per un ceppo E. coli che ospita il tipo selvaggio (WT) e il kan R (RC) composto da codoni rari in 1x, 0,5x e 0,2x lB media dopo 5 h di incubazione. (c) Effetti dell'alimentazione della L-leucina sulla crescita cellulare per i ceppi di E. coli che ospitano il gene kan R composto da leuto raro-codoni dopo 5 h di incubazione. I valori e le barre di errore rappresentano la media e la SD (n - 6). L'alimentazione di L-leucine ha aumentato significativamente l'OD600 per le cellule che ospitano il kanR,ricco di codone raro. L'unica eccezione è stata per l'alimentazione di 2 g L-1 L-leucina a causa di un alto SD in OD600 per il trattamento di alimentazione. (d) Effetti dei geni rari-codon e L-leucine che si nutrono delle espressioni GFP trattate dai geni wild-type (WT) e leucine rare-codon-rich (RC) dopo 16 h di incubazione. L'alimentazione di 0,5–2 L-leucine ha aumentato significativamente l'intensità di fluorescenza per le cellule che ospitano il gfp raro-ricco di codoni. I valori e le barre di errore rappresentano la media e la SD (n - 3). P < 0,01, P < 0,001 come determinato dal test ta due code e sono stati visualizzati solo i risultati più significativi. Fare clic qui per visualizzare una versione più grande di questa figura.
Per il sistema di screening, l'intensità della fluorescenza e il numero di cellule fluorescenti saranno significativamente inferiori per il ceppo che esprime la proteina fluorescente dal gene raro-codoni-ricco rispetto al gene wild-type (Figura1d e figura 2). Quando si utilizza la proteina viola, il colore sviluppato dal ppg raro-codone-ricco dovrebbe essere più leggero di quello dal gene di tipo selvaggio quando espresso nelle stesse condizioni per lo stesso periodo di incubazione (Figura 3). L'alimentazione dell'amminoacido corrispondente ripristinerà le espressioni proteiche dai geni rari-ricchi di codone. Per i ceppi che ospitano il gfpricco di codoni rari, l'intensità di fluorescenza (Figura 1d) e il numero di cellule fluorescenti (Figura 2) dovrebbero aumentare in modo significativo e avvicinarsi a quello dei ceppi contenenti il tipo selvaggio gfp . Quando si utilizza LB non diluito, gli amminoacidi nel mezzo sarebbero sufficienti per consentire la lenta espressione del ppg raro-ricco di codoni anche senza alimentazione L-leucina supplementare, e la proteina viola espressa diventerebbe visibile una volta che le cellule sono pellettate ( Figura 3). Tuttavia, ciò non nasconde il fatto che l'espressione genica del ppg raro-codone è stata drammaticamente potenziata dall'alimentazione della L-leucina a 2 g L-1, specialmente se osservato nella coltura liquida (Figura 3). Pertanto, la coltura liquida è una scelta migliore per lo screening basato su proteine cromogene, e l'uso di un mezzo LB diluito porterebbe una differenza più significativa tra i fenotipi indotti dal tipo selvaggio e i geni rari-ricchi di codoni.

Figura 2: Il numero di cellule E. coli fluorescenti che ospitano il gfp di tipo selvatico o il raro gfp di codoni codoni (gfp-RC) dopo l'aggiunta di L-leucine. Le cellule sono state coltivate in 1x lB medio. Barra della scala: 20 m. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Sviluppo del colore per le cellule che ospitano il tipo selvaggio (WT) e i geni ppg di leucina rara e codoni (RC) che codificano una proteina viola nel mezzo 1x LB (pannello sinistro) e l'effetto dell'alimentazione L-leucina sullo sviluppo del colore della coltura cellulare ( pannello di destra). I geni ppg sono stati indotti quando le cellule sono entrate nella fase esponenziale e le immagini sono state catturate 3 h dopo l'induzione. La L-leucina è stata aggiunta al mezzo insieme all'induttore nel saggio di alimentazione. I cerchi colorati sono stati generati raccogliendo i colori delle colture cellulari e dei pellet cellulari. Fare clic qui per visualizzare una versione più grande di questa figura.
La strategia basata sul codone raro è in grado di identificare gli amminoacidi mirati dalla libreria di mutazioni, e questi mutanti dovrebbero produrre quantità più elevate di amminoacidi mirati rispetto ai ceppi dei genitori (Figura 4).

Figura 4: Amminoacidi prodotti dal tipo selvaggio e dai ceppi mutati identificati dalla strategia basata sul codone raro. (a) Produzioni L-leucine di ceppi di E. coli identificati dalle librerie di mutazioni dal kan R-RC29 (EL-1 a EL-5) e dal gfp-RC che ospita 29 e 19 codoni rari di leucina (rispettivamente da EL-6 a EL-10). (b) Le produzioni l-arginine di ceppi di Corynebacterium glutamicum selezionati dal kan R, ricco di codone rari, che contenevaotto otto codoni rari di arginina (AGG). Il gene marcatore è stato introdotto nelle librerie di mutazione C. glutamicum derivate dal ceppo di tipo selvatico ATCC13032. Il mezzo di selezione era 0,3x CGIII fornito con 25 g-mL-1 kanamycin. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il numero di codoni rari nei geni marcatori e nel mezzo di selezione o screening è fondamentale per inibire le espressioni proteiche dai geni marcatori raramente-modificato codoni. Se non è possibile rilevare alcuna differenza significativa tra le espressioni proteiche dei geni marcatori di tipo selvatico e i loro derivati, l'aumento del numero di codoni rari o l'utilizzo di un mezzo limitato dai nutrienti può amplificare le differenze. Tuttavia, se l'effetto di inibizione è troppo forte, le espressioni proteiche non possono essere recuperate anche con l'alimentazione supplementare degli amminoacidi corrispondenti. In questo caso, il numero di codoni rari nei geni marcatori dovrebbe essere ridotto per alleviare parte dello stress. Un altro modo per perfezionare la cordenza di selezione o screening consiste nel regolare i numeri di copia e i livelli di espressione dei geni marcatori rari-ricchi di codoni. La riduzione del numero di copie e dei livelli di espressione dei geni marcatori di solito porta a differenziazioni più forti tra gli amminoacidi sovraproduttori e i ceppi iniziali. Pertanto, devono essere utilizzati vettori contenenti le origini di replica del numero di copia bassa, ad esempio p15A o pSC101, nonché promotori deboli. Se il gene del marcatore è guidato da un promotore inducibile, si raccomanda una bassa induzione.
La strategia basata sul codone raro per la selezione o lo screening dei sovraproduttori di amminoacidi è un adattamento inverso della strategia comunemente utilizzata di "ottimizzazione del codone", che mira a facilitare le espressioni delle proteine esogene. Nell'ottimizzazione del codone, i codoni rari sui geni mirati vengono sostituiti da quelli comuni sinonimi rispetto all'ospite; pertanto, i geni di altri organismi potrebbero essere tradotti molto più rapidamente in proteine rispetto a quelli esogeni con alte proporzioni di codoni rari19. Pertanto, è ragionevole supporre che l'"ottimizzazione inversa", che passa i codoni comuni ai loro sinonimi rari, dovrebbe inibire le espressioni genetiche. Tuttavia, le espressioni genetiche dovrebbero essere ripristinate mediante una ricarica avanzata dei corrispondenti tRNA rari quando gli amminoacidi mirati si accumulano intracellulare. L'incorporazione di codoni rari aumenta la soglia della concentrazione di amminoacidi nelle espressioni proteiche, che offre una potenziale strategia per selezionare o vagliare i sovraproduttori di amminoacidi quando combinato con i geni marcatori appropriati.
Oltre ai geni di resistenza agli antibiotici, ai geni delle proteine fluorescenti e ai geni delle proteine cromogeniche utilizzati nel protocollo, potrebbero essere utilizzati vari geni marcatori per stabilire il sistema di selezione o screening basato su codone raro. Ad esempio, geni letali come tolC20 e sacB21 potrebbero essere usati per selezionare i sovraproduttori di amminoacidi. In questo caso, i codoni comuni sui geni che appartengono al sistema antidoto dovrebbero essere sostituiti dai codoni rari sinonimi degli amminoacidi mirati. I ceppi che sovraproducono gli amminoacidi mirati sono in grado di lanciare il sistema antidoto e, quindi, sopravvivono agli effetti tossici indotti dai geni letali.
Va notato che gli effetti collaterali possono verificarsi quando si utilizzano elevate quantità di aminoacidi nel saggio di alimentazione. Questo perché alcuni aminoacidi sono tossici per i microrganismi. Ad esempio, una concentrazione di circa 100 mg L-1 per L-serine è in grado di inibire la crescita di E. coli22. Tuttavia, anche se inferiore a quello del gene wild-type, abbiamo scoperto che l'alimentazione fino a 2 g L-1 L-serine potrebbe ancora ripristinare le espressioni dei geni della resistenza agli antibiotici ricchi di codon irare serine13. Pertanto, la tossicità degli amminoacidi, almeno per L-serine, non comprometterebbe l'affidabilità del saggio di alimentazione. Per superare i potenziali effetti negativi della tossicità degli amminoacidi sulla produttività dei ceppi mirati, potrebbero essere applicate strategie come la mutagenesi casuale e il potenziamento delle esportazioni di amminoacidi23. Infatti, il metodo a base di codone raro è adatto per identificare ceppi tolleranti in grado di resistere o sovraprodurre aminoacidi al di sopra dei livelli tossici. Le mutazioni chiave che conferiscono tolleranza agli amminoacidi potrebbero essere identificate e introdotte nei ceppi mirati, che sarebbero gli ospiti ideali per le costruzioni di sovraproduttori di amminoacidi.
Il sistema di selezione o screening basato su codone raro garantisce un'alta fedeltà. In altre parole, i ceppi identificati dal sistema dovrebbero essere gli eccessivamente produttori degli amminoacidi mirati. Tuttavia, in alcuni casi, i candidati che sopravvivono alla selezione di antibiotici non possono produrre quantità più elevate di aminoacidi mirati rispetto al ceppo genitore. Questo potrebbe essere attribuito alla resistenza agli antibiotici acquisita dai ceppi attraverso la mutagenesi e poi una perdita del plasmide di selezione24. Di conseguenza, i ceppi senza prodottidismidici aminoacidi accresciuti migliorati potrebbero sopravvivere allo stress antibiotico e sfuggire alla selezione. Questi ceppi falsi positivi potrebbero essere eliminati inserendo un altro marcatore di selezione nel plasmide di selezione, come un gene di tipo selvaggio che conferisce resistenza a un altro antibiotico. I ceppi che hanno perso il plasmide di selezione hanno meno probabilità di ottenere una doppia resistenza ai due antibiotici e saranno eliminati durante la selezione.
I mutanti identificati dal sistema basato su codone raro dovrebbero essere in grado di sovraprodurre gli amminoacidi mirati rispetto ai ceppi iniziali. Tuttavia, la produzione di amminoacidi per i ceppi selezionati può essere ancora inferiore ai requisiti industriali. Ciò non suggerisce un fallimento della strategia basata sul codone raro, in quanto le prestazioni di deformazione sono indipendenti dal processo di selezione o screening, ma dipendono da fattori quali le caratteristiche dello sforzo iniziale, l'approccio della mutagenesi, la dimensione del libreria di mutazioni e le condizioni di fermentazione. Al fine di ottenere ceppi ad alta produzione, occorre prestare attenzione alle strategie di ingegneria del ceppo, come ad esempio dalla mutagenesi casuale o attraverso la progettazione razionale dei percorsi biosintetici degli amminoacidi. La combinazione dell'evoluzione adattiva del laboratorio e della strategia basata sul codone raro faciliterebbe l'ottenimento di sovraproduttori di amminoacidi.
La methionina e il toptophan non hanno codoni alternativi tra i 20 aminoacidi proteici. Pertanto, questa strategia non può essere impiegata direttamente a questi aminoacidi. Una possibile soluzione è quella di utilizzare tRNA ingegnerizzati che sono in grado di riconoscere i codoni di arresto per trasportare questi aminoacidi. Così, i corrispondenti codoni di stop potrebbero essere adottati come i codoni rari artificiali di questi aminoacidi25,26.
Una delle più grandi carenze riguardanti la strategia convenzionale basata su analogici per la selezione dei sovraproduttori di amminoacidi è l'alto tasso di falsi positivi5,27. I ceppi che passano attraverso la mutagenesi potrebbero facilmente acquisire resistenza verso gli analoghi aminoacidi tossici, e la tolleranza può anche essere acquisita senza l'aiuto di mutageni27. Questi ceppi potrebbero facilmente sfuggire alle pressioni di selezione dagli analoghi aminoacidi e, di conseguenza, i ceppi selezionati di solito non sono i veri sovraproduttori di amminoacidi che sacrificano notevolmente l'efficienza del processo di selezione.
Al contrario, la strategia basata sul codone raro supera il tradizionale metodo analogico consentendo identificazioni accurate e rapide dei sovraproduttori di amminoacidi. Per quanto ne sappiamo, questa è la prima strategia che adotta la legge naturale del pregiudizio del codone. Si basa solo su un singolo gene marcatore raro-ricco di codone e, quindi, elimina l'uso di analoghi tossici. I geni marcatori sono generalmente non tossici per i ceppi ospiti, e le espressioni proteiche da geni rari ricchi di codone dipendono principalmente dalle concentrazioni intracellulari degli amminoacidi corrispondenti a causa della legge universale e rigorosa della distorsione del codone in tutte le specie. Ciò impedirebbe ai ceppi di sfuggire alle pressioni di selezione. Inoltre, a causa della grande diversità dei geni marcatori, la strategia basata sul codone raro potrebbe offrire varie scelte sia per la selezione che per lo screening dei sovraproduttori di amminoacidi.
A causa del fenomeno universale della distorsione della codone in tutti gli organismi viventi28, la strategia di selezione o screening basata su codone rara potrebbe teoricamente essere impiegata ad altri microrganismi oltre a E. coli, specialmente quelli con Potenziali. Quando si passa a un ospite diverso, la scelta di codoni rari utilizzati per la progettazione dei geni marcatori dovrebbe essere basata sulle frequenze di utilizzo del codone e sull'abbondanza dei tRNA corrispondenti per l'ospite specifico. Anche il mezzo utilizzato per la selezione o lo screening deve essere ottimizzato di conseguenza. Un esempio è il Comunemente usato C. glutamicum nelle fermentazioni di amminoacidi. Un gene kanR raramente modificato con otto codoni rari di arginina (AGG) è stato dimostrato efficace nella selezione di C. glutamicum L-argininina da uno studio precedente13 (Figura 4b). Le esplorazioni della strategia basata sul codone raro dovrebbero facilitare le costruzioni e le conoscenze dei sovraproduttori di amminoacidi. Oltre agli aminoacidi, la strategia a base di codone raro potrebbe anche essere impiegata con isobutanolo, 3-metile-1-butanolo, 2-metil-1-butanol, e altri prodotti che condividono gli stessi percorsi biosintetici con alcuni aminoacidi29. I ceppi identificati dai geni marcatori che ospitano i codoni rari di questi aminoacidi sono in grado di sovraprodurre i composti precursori, che potrebbero essere incanalati alla sintesi dei derivati degli amminoacidi. Pertanto, la strategia basata sul rara codoni potrebbe servire come metodo indiretto ma rapido per riflettere le potenzialità dei ceppi nell'accumulare queste sostanze chimiche sia intra- che extracellulare. Le mutazioni chiave che conferiscono una maggiore produzione di amminoacidi da vari produttori troppo produttori potrebbero essere identificate da sequenziamento profondo e possono essere introdotte singolarmente o simultaneamente in ceppi industriali per migliorare ulteriormente le produzioni di amminoacidi.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Il lavoro è stato sostenuto congiuntamente dalla National Natural Science Foundation of China (n. 21676026), dal National Key R&D Program of China (grant n. 2017YFD0201400) e dalla China Postdoctoral Science Foundation (n. 2017M620643). I lavori dell'UCLA Institute of Advancement (Suzhou) sono stati sostenuti dalle sovvenzioni interne della provincia di Jiangsu e del Parco Industriale di Suzhou.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Thermo | 51101 | |
| EasyPure HiPure Plasmid MiniPrep Kit | Transgen | EM111-01 | |
| EasyPure Quick Gel Extraction Kit | Transgen | EG101-01 | |
| Gibson assembly master mix | NEB | E2611S | |
| Isopropyl β-D-1-thiogalactopyranoside | Solarbio | I8070 | |
| L-leucine | Sigma | L8000 | |
| Microplate reader | Biotek | Synergy 2 | |
| n-hexane | Thermo | H3061 | |
| Phenyl isothiocyanate | Sigma | P1034 | |
| PrancerPurple CPB-37-441 | ATUM | CPB-37-441 | |
| TransStar FastPfu Fly DNA polymerase | Transgen | AP231-01 | |
| Triethylamine | Sigma | T0886 | |
| Ultra-high performance liquid chromatography | Agilent | 1290 Infinity II | |
| Wild type C. glutamicum | ATCC | 13032 | |
| XL10-Gold E. coli competent cell | Agilent | 200314 | |
| ZORBAX RRHD Eclipse Plus C18 column | Agilent | 959759-902K |
Riferimenti
- Tatsumi, N., Inui, M. . Corynebacterium glutamicum: biology and biotechnology. , (2012).
- Tonouchi, N., Ito, H., Yokota, A., Ikeda, M. Present global situation of amino acids in industry. Amino Acid Fermentation. , 3-14 (2017).
- Gusyatiner, M., Lunts, M., Kozlov, Y., Ivanovskaya, L., Voroshilova, E. . DNA coding for mutant isopropylmalate synthase, L-leucine-producing microorganism and method for producing L-leucine. , (2005).
- Park, J. H., Lee, S. Y. Towards systems metabolic engineering of microorganisms for amino acid production. Current Opinion in Biotechnology. 19 (5), 454-460 (2008).
- Norris, R., Lea, P. The use of amino acid analogues in biological studies. Science Progress. , 65-85 (1976).
- Park, J. H., Lee, S. Y. Fermentative production of branched chain amino acids: a focus on metabolic engineering. Applied Microbiology and Biotechnology. 85 (3), 491-506 (2010).
- Bach, T. M., Takagi, H. Properties, metabolisms, and applications of L-proline analogues. Applied Microbiology and Biotechnology. 97 (15), 6623-6634 (2013).
- Crick, F. H. C. On the genetic code. Science. 139 (3554), 461-464 (1963).
- Plotkin, J. B., Kudla, G. Synonymous but not the same: the causes and consequences of codon bias. Nature Reviews Genetics. 12 (1), 32-42 (2011).
- Dittmar, K. A., Sørensen, M. A., Elf, J., Ehrenberg, M., Pan, T. Selective charging of tRNA isoacceptors induced by amino‐acid starvation. EMBO Reports. 6 (2), 151-157 (2005).
- Elf, J., Nilsson, D., Tenson, T., Ehrenberg, M. Selective charging of tRNA isoacceptors explains patterns of codon usage. Science. 300 (5626), 1718-1722 (2003).
- Sørensen, M. A. Charging levels of four tRNA species in Escherichia coli Rel+ and Rel− strains during amino acid starvation: a simple model for the effect of ppGpp on translational accuracy. Journal of Molecular Biology. 307 (3), 785-798 (2001).
- Zheng, B., et al. Utilization of rare codon-rich markers for screening amino acid overproducers. Nature Communications. 9 (1), 3616 (2018).
- Richardson, S. M., Wheelan, S. J., Yarrington, R. M., Boeke, J. D. GeneDesign: rapid, automated design of multikilobase synthetic genes. Genome Research. 16 (4), 550-556 (2006).
- Xiong, A. -. S., et al. PCR-based accurate synthesis of long DNA sequences. Nature Protocols. 1 (2), 791-797 (2006).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Green, M. R., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Cohen, S. A., Bidlingmeyer, B. A., Tarvin, T. L. PITC derivatives in amino acid analysis. Nature. 320 (6064), 769-770 (1986).
- Zhou, J., Liu, W. J., Peng, S. W., Sun, X. Y., Frazer, I. Papillomavirus capsid protein expression level depends on the match between codon usage and tRNA availability. Journal of Virology. 73 (6), 4972-4982 (1999).
- Gregg, C. J., et al. Rational optimization of tolC as a powerful dual selectable marker for genome engineering. Nucleic Acids Research. 42 (7), 4779-4790 (2014).
- Pelicic, V., Reyrat, J. M., Gicquel, B. Expression of the Bacillus subtilis sacB gene confers sucrose sensitivity on mycobacteria. Journal of Bacteriology. 178 (4), 1197-1199 (1996).
- Avcilar-Kucukgoze, I., et al. Discharging tRNAs: a tug of war between translation and detoxification in Escherichia coli. Nucleic Acids Research. 44 (17), 8324-8334 (2016).
- Mundhada, H., Schneider, K., Christensen, H. B., Nielsen, A. T. Engineering of high yield production of L-serine in Escherichia coli. Biotechnology and Bioengineering. 113 (4), 807-816 (2016).
- Makosky, P. C., Dahlberg, A. E. Spectinomycin resistance at site 1192 in 16S ribosomal RNA of E. coli: an analysis of three mutants. Biochimie. 69 (8), 885-889 (1987).
- Feng, L., Tumbula-Hansen, D., Toogood, H., Söll, D. Expanding tRNA recognition of a tRNA synthetase by a single amino acid change. Proceedings of the National Academy of Sciences of the United States of America. 100 (10), 5676-5681 (2003).
- Naganuma, M., et al. The selective tRNA aminoacylation mechanism based on a single G• U pair. Nature. 510 (7506), 507 (2014).
- Hoesl, M. G., et al. Chemical evolution of a bacterial proteome. Angewandte Chemie International Edition. 54 (34), 10030-10034 (2015).
- Hershberg, R., Petrov, D. A. Selection on codon bias. Annual Review of Genetics. 42, 287-299 (2008).
- Huo, Y. -. X., et al. Conversion of proteins into biofuels by engineering nitrogen flux. Nature Biotechnology. 29, 346 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon