Method Article
使用稀有的科顿富标记识别氨基酸过度生产者
摘要
本研究提出了一种替代策略,即使用稀有富含柯顿标记物的氨基酸过度生产者识别氨基酸,同时实现精度、灵敏度和高通量。
摘要
为了满足不断增长的氨基酸市场,需要高性能生产菌株。氨基酸过度生产者通常是通过利用氨基酸与其模拟物之间的竞争来识别的。然而,这种基于模拟的方法精度较低,特定氨基酸的正确模拟是有限的。在这里,我们提出了一个替代策略,利用稀有富含柯顿标记的氨基酸过度生产者进行准确、敏感和高通量筛选。这一策略的灵感来自于蛋白质翻译中的柯顿使用偏差现象,根据在编码DNA中出现的频率,将科顿分为普通或稀有。稀有科顿子的翻译取决于其相应的罕见转移RNA(tRNA),在饥饿的情况下,这种RNA不能被共生氨基酸完全充电。从理论上讲,如果向同义的通用等接受器充电后,氨基酸过剩,则可以对稀有tRNA收费。因此,通过喂食或细胞内过度生产相应的氨基酸,可以恢复由稀有科顿引起的缓速翻译。根据这一假设,通过用抗生素耐药性基因或基因中的同义词稀有替代品取代靶向氨基酸的常见子细胞,建立了用于识别氨基酸过度生产者的选择或筛选系统编码荧光或致色蛋白。我们表明,蛋白质的表达会因稀有的科顿而受到很大阻碍,蛋白质水平与氨基酸浓度呈正相关。利用这个系统,多种氨基酸的过产可以很容易地从突变库中筛选出来。这种基于稀有的基于科顿的策略只需要一个修饰基因,并且宿主比其他方法更不可能逃脱选择。它提供了一种获得氨基酸过度生产者的替代方法。
引言
目前氨基酸的生产严重依赖发酵。然而,大多数氨基酸生产菌株的分度和产量低于全球氨基酸市场日益增长的需求,价值数十亿美元1,2。获得高性能氨基酸过度生产者是氨基酸产业升级的关键。
传统的识别氨基酸过度生产者的策略利用氨基酸和其模拟在蛋白质合成3,4之间的竞争。这些模拟物能够给识别相应氨基酸的tRNA充电,从而抑制肽链的增长,导致被抑制的生长或细胞死亡5。抵抗模拟应力的一种方法是增加细胞内氨基酸的浓度。富集氨基酸将超越有限tRNA的模拟,并确保功能蛋白的正确合成。因此,可以选择在模拟中存活的菌株,并且可能是相应氨基酸的过度生产者。
虽然证明在选择L-亮氨酸6等氨基酸的过度生产者方面是成功的,但基于模拟的策略存在严重缺陷。一个主要问题是来自诱变过程或自发突变的模拟阻力。具有电阻的应变可以通过阻塞、导出或降解模拟5来逃避选择。另一个担忧是模拟对其他细胞过程的毒性副作用7。因此,在模拟选择中幸存下来的菌株可能不是氨基酸过度生产者,而所需的过度生产者可能由于负面的副作用而被错误地消灭。
本文提出了一种基于科顿偏倚定律的新策略,以便准确、快速地识别氨基酸过度生产者。大多数氨基酸由多个核苷酸三联体编码,由宿主生物体8、9以不同的方式青睐。有些科顿很少在编码序列中使用,被称为稀有的科顿。它们转化为氨基酸依赖于携带相应氨基酸的干酪tRNA。然而,识别稀有科顿子的tRNA通常比普通科顿10、11的tRNA的丰度低得多。因此,这些稀有的tRNA不太可能在与其他异位接受器的竞赛中捕获游离氨基酸,当氨基酸的量有限时,稀有富含克顿序列的翻译开始减速甚至终止10.理论上,如果由于过度生产或额外喂食相应的氨基酸12而对同义的通用tRNA收费后,如果存在氨基酸过剩,翻译可以恢复。如果稀有的富含科顿的基因编码了选择或筛选标记,则表现出相应表型的菌株很容易被识别,并且很可能是靶向氨基酸的过度生产者。
应用上述策略建立氨基酸超标生产者的选型和筛选系统。选择系统使用抗生素抗性基因(例如kan R)作为标记,而筛选系统使用编码荧光(例如绿色荧光蛋白 [GFP]) 或色基因(例如 PrancerPurple)蛋白质的基因。通过用同义的稀有替代品替换目标氨基酸的常用通子的定义为号的标记基因,两个系统中的标记基因被修改。在突变库中,含有稀有的富含柯顿标记基因的菌株在适当的条件下被选择或筛选,并且靶向氨基酸的过度产生者很容易被识别。工作流程从构建稀有的富含柯顿标记基因系统开始,然后优化工作条件,然后对氨基酸过度生产者进行鉴定和验证。这种模拟独立策略基于蛋白质翻译中的教条,并已实际验证,能够准确、快速地识别氨基酸过度生产者。从理论上讲,它可以直接用于氨基酸与罕见的科顿和所有微生物。总之,当特定氨基酸无法使用适当的模拟时,或者当高误阳性率是主要问题时,基于稀有的节子策略将作为传统基于模拟的方法的有效替代方法。下面的协议使用亮氨酸稀有科顿来演示这一策略,以识别大肠杆菌L-亮氨酸过度生产者。
研究方案
1. 表达稀有富含柯顿标记基因的质粒构造
- 选择一个标记基因,其中包含目标氨基酸的相应数量的常用通子。
注:对于L-亮氨酸,卡那霉素抗基因kan R,其中含有29个亮氨酸科顿,其中27个是常见的科顿,用于构建选择系统13。gfp基因,包含17个普通科顿从19个亮子,或紫色蛋白质编码基因prancerpurple(ppg),其中含有14个亮氨酸常见的科顿,用于筛选系统(补充表1). - 用代名词稀有的科顿替换标记基因中的普通科顿。对于 L-亮氨酸,将其在 kan R、gfp或ppg中的科顿替换为稀有的柯顿 CTA,分别生成 kan R-RC、gfp-RC或ppg-RC(13) 补充表1。
注:标记基因中稀有科顿的频率将影响选择或筛选系统的严格性。一般来说,增加稀有的科顿数量将增加选择或筛选系统的严格性。为了达到适当的选择或筛选强度,设计一系列标记基因,其中含有不同数量的稀有科顿,并比较其效果。 - 使用GeneDesign14(http://54.235.254.95/gd/)等工具生成稀有富含科顿标记基因的构建基块,用于基因合成。或者,从商业基因合成服务订购标记基因。
- 在 GeneDesign 页上,选择构建基块设计(恒定长度重叠)。
- 将稀有富含密码子的标记基因的序列粘贴到序列框中。
- 定义装配寡核之间的重叠长度;请记住,默认的 40 bp适用于大多数序列。
注: 有关其他参数设置的更多说明,请参阅联机手册。 - 单击"设计构建基块"按钮,然后订购页面上列出的寡核苷酸。
- 通过聚合酶链反应(PCR)合成稀有的科顿修饰基因15。
- 利盖特稀有的康克顿丰富的kan R-RC向矢量pET-28a,gfp-RC到pSB1C3,ppg-RC到CPB-37-441 16。
注:pET-28a和pSB1C3质粒图可在SnapGene在线质粒数据库(补充表1)上查阅;CPB-37-441质粒图可在ATUM染色体蛋白网站上查阅。- 在冰上,将矢量和标记碎片以1:1至7.5 μL的装配组合(见材料表)的摩尔比添加到总体积为10 μL。在50°C孵育样品1小时。
- 将装配产物的 5 μL 转换为 50 μL 的合格电池(参见材料表),在 42°C 下 30 s。
- 在 SOC(具有催化素抑制的超级最佳肉汤)培养基中回收细胞,在 37°C 下1小时,在LB(乳原肉汤)琼脂培养基上进行板,并在37°C过夜孵育。
- 将菌落接种到LB介质中,在37°C孵育8小时。
- 使用首选商业试剂盒分离质粒。
2. 优化选择条件
- 使用于诱变的母性菌株转化为称职的细胞17。
- 用携带野生型kan R的1μL质粒转换50μL的合格细胞,用含有kan R-RC29的质粒改造另一组合格细胞,用稀有的亮氨酸科顿替换所有亮氨酸科顿科登CTA。
- 加入950μL的SOC介质,在37°C下以250rpm在摇床中孵育样品1小时。
- 将细胞培养物的板100μL板在含有50μgμmL-1卡那霉素的LB琼脂基培养基上,并在37°C孵育约8小时,直到菌落出现。
- 选择含有野生型kan R和富含金刚醇的康登-RC29的菌落,并分别使用接种5 mL的五倍稀释LB介质(0.2倍LB),含有50μg_mL-1卡那霉素。在 37°C 下以 250 rpm 的转速在摇床中孵育样品。
注: 介质对选择系统至关重要。它应该含有足够的碳和氮,允许从野生基因表达抗生素抗性蛋白,而不是从稀有的科顿修饰衍生物中表达。在这种情况下,1x LB介质中的L-亮氨酸浓度过高,无法完全抑制稀有富含康顿(kan R)的蛋白质表达。因此,使用稀释的LB介质,如5的稀释因子,在野生型和稀有的富含科顿基因的蛋白质表达上产生明显的差异。 - 在设定的时间点(例如,8 小时、16 小时和 24 小时)将每个细胞培养物的 200 μL 转移到三联的 96 孔板中。使用板式读取器测量 OD 600(600 nm 时的光密度)。
注意:如果与携带野生型标记基因的菌株OD600相比,无法检测出含有稀有科顿富标记基因的菌株的细胞OD600的减少,则尝试增加野生型标记基因中的稀有科顿数量。标记基因或使用更稀释的培养基。 - 执行氨基酸馈送测定,以测试稀有富含柯顿标记基因(例如kan R-RC29)的表达是否可以通过增加靶向氨基酸的浓度来恢复。
注:对于L-亮氨酸过度生产者的选择,L-亮氨酸用于喂养。- 将含有kan R-RC29标记基因的菌株接种到 0.2x LB 的 5 mL(包含 50 μgμmL-1 kanamycin),带或不提供 1 g*L-1 L-亮氨酸。用含有野生型kan R作为对照的菌株,为0.2倍LB的另外5 mL接种。在 37°C 下以 250 rpm 的转速在摇床中孵育样品。
- 在定义的时间点(例如,15 小时、17 小时、19 小时和 22 小时)测量每种区域性的 OD600。
3. 优化筛选条件
- 将用于诱变的母菌株的50 μL与携带野生型gfp或野生型ppg的1μL质粒(±50 ng_l-1)转化。此外,使用标记基因的稀有-富含柯顿衍生物来转换母菌株。
- 加入950μL的SOC介质,在37°C下以250rpm在摇床中孵育样品1小时。
- 将细胞培养物的板100μL板放在含有适当抗生素的LB琼脂介质上(25微克+mL-1氯霉素,用于携带厘米R标记的gfp质粒,或50μgμmL-1卡那霉素用于ppg携带kan R标记物的质粒,并在37°C孵育过夜。
- 选择一个窝藏野生型gfp或ppg的殖民地和一个窝窝,窝藏相应的稀有-富含科顿衍生物,并分别将它们转移到5mL的适当地稀释的LB介质。在 37°C 下以 250 rpm 的转速在摇床中孵育样品。
注:对于含有富含白细胞的大肠杆菌菌株,未稀释的LB介质(1x LB)可用于在野生型和稀有蜂腺富标记物的表达上产生显著差异基因。此外,诱导启动子用于驱动筛选标记基因的表达。当细胞进入指数阶段以实现更好的区分时,开始诱导。 - 对于荧光标记,在设定的时间点(例如,2 小时、4 小时和 6 小时)将每个细胞培养物的 200 μL 转移到一个 96 孔清底黑板中,以三联形式。测量 OD600和荧光并计算荧光强度(荧光与 OD600的比率)。对于染色体标记,测量细胞培养物的颜色发展。
注:如果与携带野生型gfp的菌株相比,无法检测出含有gfp-RC的菌株的荧光强度较低,或者对于表达紫色蛋白质的细胞,检测出较浅的颜色从ppg-RC比从野生型基因,试图增加在标记基因上的稀有科顿的数量或使用更稀释的介质。 - 执行进给测定(参见步骤 2.7.1 和 2.7.2)。在定义的时间点(例如,12 小时、18 小时和 24 小时)测量荧光强度或颜色发展。
4. 氨基酸过度生产者的鉴定
- 将突变体培养物的100 μL接种到5 mL的LB介质中(2%v/v),并在37°C下以250rpm的振动器孵育,直到OD600值达到0.4。
- 使突变体变成称职的细胞17。
- 使用 1 μL 的质粒 (+50 ng_l-1) 将 50 μL 的突变细胞转换为携带选择标记kan R-RC29或筛选标记gfp-RC或ppg-RC。加入 950 μL 的 SOC 介质,在 37°C 摇床中旋转样品 1 小时。
- 选择氨基酸过度生产者。
- 在4,000 x g下将细胞培养物离心5分钟,丢弃上清液,加入含有50μg_mL-1卡那霉素的5mL 0.2倍LB培养基。在37°C摇床中孵育样品过夜。
- 将隔夜培养物(例如,100 μL,取决于培养物的细胞密度)镀在含有50 μg_mL-1卡那霉素的0.2倍LB琼脂基培养基上,并在37°C孵育12小时。
注:开发的菌落是目标氨基酸过度生产者的候选,在这种情况下,L-亮氨酸过度生产者。
- 筛选氨基酸过度生产者。
- 将含有筛查标记的适量细胞(例如 100 μL)镀在含有适当抗生素(25 μg_mL-1 氯霉素)的LB琼脂介质上,用于用gfp-RC和50 μgμmL进行筛查-1卡那霉素,用于用ppg-RC进行筛选,并在37°C孵育8小时。
- 用板中的每一个菌群接种含有适当抗生素的LB介质(参见步骤4.5.1)。在 37°C 下以 250 rpm 的转速在摇床中孵育样品。
- 如果使用gfp-RC,则测量 OD600和荧光并计算荧光强度。如果使用ppg-RC,则测量细胞培养物的颜色发展。请注意,荧光强度高于母菌株的菌株是候选氨基酸过度培养,在这种情况下,L-亮氨酸过度生产者。
注:荧光活性细胞分拣也适合在使用稀有富含柯顿蛋白基因进行筛选时识别单细胞高生产者。
- 验证候选菌株的氨基酸产物。
- 用每个候选菌株接种5 mL的LB培养基,让细胞在37°C下以250rpm的振动器在摇床中生长过夜。
- 在4000 x g下离心2分钟,从细胞培养的1 mL中收获细胞,2分钟。丢弃上清液,用1 mL的无菌水重新悬浮颗粒。
- 用200μL的细胞悬浮液接种含有4%葡萄糖的M9介质,并在250mL摇床中孵育,在250rpm时在37°C下孵育24小时。
- 在 4,000 x g下将培养基的 1 mL 离心 5 分钟,将 200 μL 的上清液转移到干净的 1.5 mL 管中。制备 0.01、0.05、0.1、0.5 和 1 g* 的 L-亮氨酸溶液(HPLC(高性能液相色谱)等级L-1作为标准。
- 将100 μL的1mM三乙胺和100μL的1M苯异硫亚酸盐加入上清酸酯和标准中,轻轻混合,并在室温下孵育1h18。
注意:三乙胺和苯二恶英可引起严重的皮肤灼伤和眼睛损伤,吸入后有害。戴上手套和面罩,如果可能的话,在烟罩中执行此步骤。 - 将400μL的n-Hex烷加入同一管,涡旋10s。通过0.2μm聚四氟乙烯膜过滤含有氨基酸衍生物的下相。
注意:n-己毒可引起皮肤刺激。戴上手套和防护服。如果与皮肤接触,请用大量水冲洗皮肤。 - 以99.3:0.7体积比混合0.1M醋酸钠(pH 6.5)和醋酸酯,制备移动阶段A。制备醋酸酯(80%v/v)作为移动相B.通过0.2μm聚四氟乙烯膜过滤所有移动相。
注意:如果吸入,醋酸酯是有害的,并可能导致皮肤和眼睛刺激。戴上手套和防护服,在烟罩中执行此步骤。 - 根据表 1中的洗脱程序,在配备 C18 柱的超 HPLC 上运行 1 μL 样品,流速为 0.42 mLμmin-1,柱温度为 40°C。使用二极管阵列检测器检测 254 nm 处的目标氨基酸,并通过将峰值区域映射到标准曲线来计算其浓度。
| 时间(分钟) | 移动阶段 A (%) | 移动阶段 B (%) |
| 0 | 98 | 2 |
| 3.5 | 70 | 30 |
| 7 | 43 | 57 |
| 7.1 | 0 | 100 |
| 11 | 98 | 2 |
表1:氨基酸定量的洗脱方案。
结果
对于选择系统,与在合适的培养中培养野生型抗生素耐药性基因的菌株相比,应观察到携带稀有富柯顿抗生素耐药性基因的菌株的OD600的急剧减少。中等 (图 1a)。在相同的条件下,随着抗生素耐药性基因中稀有科顿数量的增加,细胞OD600的减少变得更加明显(图1a)。应该指出的是,对蛋白质表达的稀有科顿抑制大多发生在饥饿的条件下。因此,如果LB培养基未适当稀释,与携带野生型基因的菌株相比,携带稀有科顿富标记基因的菌株不会显著减少细胞OD600(图1b)。在额外喂养相应的氨基酸后,携带稀有富含柯顿抗生素抗药性基因的菌株的OD600将显著增加,并接近携带野生型基因的菌株的OD 600(图1c)。
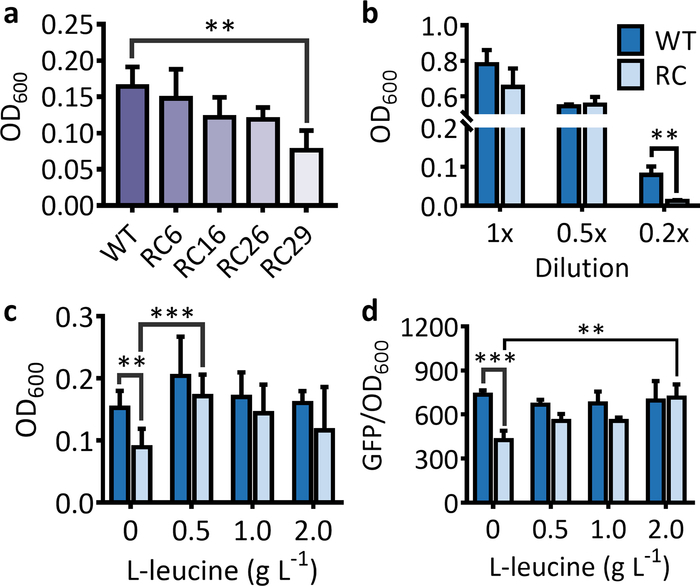
图1:稀有科顿对用于选择和筛选系统的标记基因表达的影响。(a) 含有抗生素抗性基因(kan R) 的大肠杆菌菌株(kanR)的细胞OD600在孵育5小时后更换6、16、26和29亮氨酸稀有红素(RC6、RC16、RC26和RC29)。(b) 大肠杆菌菌株的细胞OD 600在孵育5小时后,在1x、0.5x和0.2xLB培养基中含有野生型(WT)和稀有富含孔氏菌的kan R(RC)。(c) 喂养L-亮氨酸对含有潜伏在潜伏5小时后含有亮氨酸稀有-科顿-康登R基因的大肠杆菌菌株的细胞生长的影响。值和误差条表示平均值和 SD (n = 6)。L-亮氨酸的喂养显著增加了OD600的细胞窝藏稀有的康顿丰富的kan R。唯一的例外是喂食2克*L-1 L-亮氨酸,由于在OD600中的高SD用于喂食治疗。(d ) 在孵育16小时后,稀有科顿和L-亮氨酸对野生型(WT)和富含白细胞的亮氨酸稀有性(RC)基因的GFP表达的影响。0.5⁄2L-1 L-亮氨酸的喂养显著提高了含有稀有富含克顿gfp的细胞的荧光强度。值和误差条表示平均值和 SD (n = 3)。•P < 0.01, =P < 0.001 由双尾t检验确定,仅显示最重要的结果。请点击此处查看此图的较大版本。
对于筛选系统,荧光强度和荧光细胞的数量将明显低于从稀有-富通子基因表达荧光蛋白的菌株,而不是野生型基因(图1d和图 2.当使用紫色蛋白质时,从稀有的富含科顿蛋白的ppg中发展出的颜色应该比在相同潜伏期的相同条件下表达的野生型基因的颜色要轻(图3)。喂食相应的氨基酸会恢复稀有富含柯顿的基因的蛋白质表达。对于含有稀有克顿丰富的gfp的菌株,荧光强度(图1d)和荧光细胞的数量(图2)应显著增加,并接近含有野生型gfp的菌株。 .当使用未稀释的LB时,介质中的氨基酸将足以允许缓慢表达稀有富含柯顿ppg的ppg,即使没有额外的L-亮氨酸喂养,一旦细胞被造粒,表达的紫色蛋白质就会变得可见(图 3.然而,这并不掩盖一个事实,即从稀有的富含柯顿ppg的基因表达,通过喂养L-亮氨酸到2克,显着增强。L-1,特别是在液体培养中观察到时(图3)。因此,液体培养是基于色原蛋白筛选的更好选择,使用稀释的LB培养基会使野生型基因和稀有富通体基因引起的表型发生更显著的差异。

图 2:添加L-亮氨酸后含有野生型gfp或亮氨酸稀有红素-丰克顿(gfp-RC)的荧光大肠杆菌细胞的数量。 细胞在1xLB培养基培养。刻度条 = 20 μm。请点击此处查看此图的较大版本。

图3:含有野生型(WT)和亮氨酸稀有性红素(RC)的细胞的颜色发育,在1xLB培养基(左面板)中编码紫色蛋白质的ppg基因和L-亮氨酸喂养对细胞培养颜色发展的影响(右面板)。当细胞进入指数相时,诱导ppg基因,在诱导后3小时捕获图像。在喂食测定中,将L-亮氨酸与诱导剂一起添加到介质中。彩色圆圈是通过选择细胞培养物和细胞颗粒的颜色而生成的。请点击此处查看此图的较大版本。
基于稀有的基顿策略能够从突变库中识别目标氨基酸的过度生成,这些突变体应产生比母菌株数量更高的靶向氨基酸(图4)。

图4:野生型和变异菌株产生的氨基酸,由稀有基元策略确定。(a) 从突变库中分别由kan R-RC29(EL-1到 EL-5)和gfp-RC(分别含有 29 和 19 亮氨酸-6 到 EL-10)的大肠杆菌菌株的 L-亮氨酸生产。(b) 由稀有富含柯顿丰富的kan R选择的柯林杆菌谷氨基菌株的L-精氨酸生产,其中含有8种精氨酸稀有科顿(AGG)。标记基因被引入从野生型菌株ATCC13032衍生的C.谷氨酰氨基突变库中。选择介质为0.3xCGIII,提供25μgμmL-1卡那霉素。请点击此处查看此图的较大版本。
讨论
标记基因和选择或筛选培养基中的稀有科顿数量对于抑制稀有科顿修饰标记基因的蛋白质表达至关重要。如果野生型标记基因的蛋白质表达及其衍生物之间没有显著差异,增加稀有科顿的数量或使用营养有限的培养基可能会放大差异。然而,如果抑制作用太强,即使额外喂食相应的氨基酸,蛋白质表达也可能无法恢复。在这种情况下,标记基因中的稀有科子数量应减少,以减轻部分压力。微调选择或筛选严格性的另一种方法是调整稀有富含柯顿标记基因的拷贝数和表达水平。减少标记基因的拷贝数和表达水平通常会导致氨基酸过度生产者和初始菌株之间的更强分化。因此,应使用包含低拷贝数复制源(如 p15A 或 pSC101)以及弱启动子的矢量。如果标记基因由诱导启动子驱动,则建议低诱导。
氨基酸过度生产者的稀有-基于酮子的策略是反向调整常用的"科顿优化"策略,其目的是促进外源蛋白的表达。在科顿优化中,目标基因上的稀有科顿被与宿主同义的共性基因所取代;因此,来自其他生物体的基因可以比那些具有高比例的稀有科顿19的外源基因更快地转化为蛋白质。因此,可以合理地假设,将普通科顿切换到其同义词稀有的"反向优化"应抑制基因表达。然而,当靶向氨基酸在细胞内积累时,应通过增强对相应稀有tRNA的充电来恢复基因表达。稀有科顿加入会增加蛋白质表达中氨基酸浓度的阈值,这为与适当的标记基因结合时选择或筛选氨基酸过度生产者提供了一种潜在的策略。
除了协议中使用的抗生素抗性基因、荧光蛋白基因和染色体蛋白基因外,还可以利用各种标记基因建立稀有的基于科顿的选择或筛选系统。例如,致命基因,如tolC20和ssB21可用于选择氨基酸过度生产者。在这种情况下,属于解毒剂系统的基因上的普通科顿应该被目标氨基酸的同义词稀有的科顿所取代。过量生产靶向氨基酸的菌株能够启动解毒剂系统,从而在致命基因引起的毒性作用下存活下来。
应该注意的是,在喂食测定中使用大量氨基酸时可能会出现副作用。这是因为一些氨基酸对微生物有毒。例如,浓度约为100毫克*L-1为L-丝氨酸能够抑制大肠杆菌22的生长。然而,虽然低于野生型基因,我们发现喂养高达2克*L-1 L-丝氨酸仍然可以恢复富含丝氨酸稀有科顿13的抗生素耐药性基因的表达。因此,氨基酸毒性,至少对L-丝氨酸,不会危及喂养测定的可靠性。为了克服氨基酸毒性对靶向菌株的增产的潜在负面影响,可以采用随机诱变和增强氨基酸出口等策略。事实上,这种稀有的康酮法适用于识别能够承受或过度生产超过毒性水平的氨基酸的耐性菌株。可以识别赋予氨基酸耐受性的关键突变,并将其引入目标菌株中,而靶向菌株将是氨基酸过度生产者结构的理想宿主。
基于稀有的科顿选择或筛选系统可确保高保真度。换句话说,系统识别的菌株应该是靶向氨基酸的过度生产者。然而,在某些情况下,在抗生素选择中幸存下来的候选者不能产生比母菌株量更高的靶向氨基酸。这可以归因于菌株通过诱变获得的抗生素耐药性,然后失去选择质粒24。因此,没有增强氨基酸产物的菌株可以经受住抗生素的压力,并逃避选择。通过将另一个选择标记插入选择质粒,如对另一种抗生素产生抗药性的野生基因,可以消除这些假阳性菌株。失去选择质粒的菌株不太可能对两种抗生素产生双重耐药性,在选择过程中将消除。
与初始菌株相比,稀有基酮系统所识别的突变体应该能够过度生产靶向氨基酸。然而,所选菌株的氨基酸产物可能仍然低于工业要求。这并不意味着基于稀有的科顿策略失败,因为应变性能与选择或筛选过程无关,但取决于诸如初始应变的特征、诱变的方法、应变的大小等因素。突变库,和发酵条件。为了获得高产菌株,应注意应变工程的策略,如随机诱变或氨基酸生物合成途径的合理设计。结合适应性实验室进化和稀有的科顿为基础的策略,将有利于获得氨基酸过度生产者。
蛋氨酸和锥子在20种蛋白质氨基酸中没有替代的科顿。因此,这种策略可能不会直接用于这些氨基酸。一个可能的解决方案是使用能够识别停止科顿携带这些氨基酸的工程tRNA。因此,相应的停止科顿可以被采纳为这些氨基酸的人工稀有科顿25,26。
选择氨基酸过剩的传统模拟策略的最大缺点是高误率5,27。通过诱变的菌株很容易获得对有毒氨基酸类比的抗性,甚至无需诱变27获得耐受性。这些菌株可以很容易地从氨基酸模拟中逸出选择压力,因此,选择的菌株通常不是真正的氨基酸过度生产者,这极大地牺牲了选择过程的效率。
相比之下,基于稀有的康顿策略通过准确、快速地识别氨基酸过度生产者,胜过传统的基于模拟的方法。据我们所知,这是第一个采用科顿偏见的自然定律的策略。它仅依赖于一个稀有的富含柯顿标记基因,因此,消除了有毒类比的使用。标记基因通常对宿主菌株无毒,而稀有富含科顿基因的蛋白质表达主要取决于相应氨基酸的细胞内浓度,因为科顿偏倚的通用和严格规律跨越所有物种。这将防止应变逃避选择压力。此外,由于标记基因的多样性,稀有的科顿策略可以为氨基酸过度生产者的选择和筛选提供多种选择。
由于在所有生物体内普遍存在柯顿偏倚现象28,从理论上讲,这种稀有的基于科顿的选择或筛选策略可以应用于除大肠杆菌以外的其他微生物,特别是那些具有工业性电位。当更改为不同的宿主时,用于设计标记基因的稀有科顿的选择应基于柯登使用频率和特定宿主相应 tRNA 的丰度。用于选择或筛选的介质也应相应地进行优化。一个例子是氨基酸发酵中常用的谷氨酰氨基金。一种稀有的康顿修饰的kanR基因含有8个精氨酸稀有的科顿(AGG),通过先前的研究13(图4b)显示,在选择C.谷氨酰二醇L-精氨酸过度生产者方面是有效的。对稀有的、基于康顿的策略的探索应有助于氨基酸过度生产者的构建和理解。除氨基酸外,稀有的基于密码子的策略还可以与异丁醇、3-甲基-1-丁醇、2-甲基-1-丁醇以及其他与某些氨基酸共享相同生物合成通路的产品一起使用。由含有这些氨基酸的稀有科顿标记基因鉴定的菌株能够过度生产前体化合物,这些化合物可以引导到氨基酸衍生物的合成中。因此,基于稀有的康顿策略可以作为一种间接而快速的方法,以反映菌株在细胞内或细胞外积累这些化学品的潜力。通过深度测序,可以确定从各种过度生产者中增加氨基酸产量的关键突变,并单独或同时引入工业菌株,以进一步提高氨基酸产量。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了国家自然科学基金(授权号21676026)、国家重点研发项目(批号2017YFD0201400)和中国博士后科学基金会(批号2017M620643)共同支持。加州大学洛杉矶分校(苏州)的工程得到了江苏省和苏州工业园区的内部资助。
材料
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Thermo | 51101 | |
| EasyPure HiPure Plasmid MiniPrep Kit | Transgen | EM111-01 | |
| EasyPure Quick Gel Extraction Kit | Transgen | EG101-01 | |
| Gibson assembly master mix | NEB | E2611S | |
| Isopropyl β-D-1-thiogalactopyranoside | Solarbio | I8070 | |
| L-leucine | Sigma | L8000 | |
| Microplate reader | Biotek | Synergy 2 | |
| n-hexane | Thermo | H3061 | |
| Phenyl isothiocyanate | Sigma | P1034 | |
| PrancerPurple CPB-37-441 | ATUM | CPB-37-441 | |
| TransStar FastPfu Fly DNA polymerase | Transgen | AP231-01 | |
| Triethylamine | Sigma | T0886 | |
| Ultra-high performance liquid chromatography | Agilent | 1290 Infinity II | |
| Wild type C. glutamicum | ATCC | 13032 | |
| XL10-Gold E. coli competent cell | Agilent | 200314 | |
| ZORBAX RRHD Eclipse Plus C18 column | Agilent | 959759-902K |
参考文献
- Tatsumi, N., Inui, M. . Corynebacterium glutamicum: biology and biotechnology. , (2012).
- Tonouchi, N., Ito, H., Yokota, A., Ikeda, M. Present global situation of amino acids in industry. Amino Acid Fermentation. , 3-14 (2017).
- Gusyatiner, M., Lunts, M., Kozlov, Y., Ivanovskaya, L., Voroshilova, E. . DNA coding for mutant isopropylmalate synthase, L-leucine-producing microorganism and method for producing L-leucine. , (2005).
- Park, J. H., Lee, S. Y. Towards systems metabolic engineering of microorganisms for amino acid production. Current Opinion in Biotechnology. 19 (5), 454-460 (2008).
- Norris, R., Lea, P. The use of amino acid analogues in biological studies. Science Progress. , 65-85 (1976).
- Park, J. H., Lee, S. Y. Fermentative production of branched chain amino acids: a focus on metabolic engineering. Applied Microbiology and Biotechnology. 85 (3), 491-506 (2010).
- Bach, T. M., Takagi, H. Properties, metabolisms, and applications of L-proline analogues. Applied Microbiology and Biotechnology. 97 (15), 6623-6634 (2013).
- Crick, F. H. C. On the genetic code. Science. 139 (3554), 461-464 (1963).
- Plotkin, J. B., Kudla, G. Synonymous but not the same: the causes and consequences of codon bias. Nature Reviews Genetics. 12 (1), 32-42 (2011).
- Dittmar, K. A., Sørensen, M. A., Elf, J., Ehrenberg, M., Pan, T. Selective charging of tRNA isoacceptors induced by amino‐acid starvation. EMBO Reports. 6 (2), 151-157 (2005).
- Elf, J., Nilsson, D., Tenson, T., Ehrenberg, M. Selective charging of tRNA isoacceptors explains patterns of codon usage. Science. 300 (5626), 1718-1722 (2003).
- Sørensen, M. A. Charging levels of four tRNA species in Escherichia coli Rel+ and Rel− strains during amino acid starvation: a simple model for the effect of ppGpp on translational accuracy. Journal of Molecular Biology. 307 (3), 785-798 (2001).
- Zheng, B., et al. Utilization of rare codon-rich markers for screening amino acid overproducers. Nature Communications. 9 (1), 3616 (2018).
- Richardson, S. M., Wheelan, S. J., Yarrington, R. M., Boeke, J. D. GeneDesign: rapid, automated design of multikilobase synthetic genes. Genome Research. 16 (4), 550-556 (2006).
- Xiong, A. -. S., et al. PCR-based accurate synthesis of long DNA sequences. Nature Protocols. 1 (2), 791-797 (2006).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Green, M. R., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Cohen, S. A., Bidlingmeyer, B. A., Tarvin, T. L. PITC derivatives in amino acid analysis. Nature. 320 (6064), 769-770 (1986).
- Zhou, J., Liu, W. J., Peng, S. W., Sun, X. Y., Frazer, I. Papillomavirus capsid protein expression level depends on the match between codon usage and tRNA availability. Journal of Virology. 73 (6), 4972-4982 (1999).
- Gregg, C. J., et al. Rational optimization of tolC as a powerful dual selectable marker for genome engineering. Nucleic Acids Research. 42 (7), 4779-4790 (2014).
- Pelicic, V., Reyrat, J. M., Gicquel, B. Expression of the Bacillus subtilis sacB gene confers sucrose sensitivity on mycobacteria. Journal of Bacteriology. 178 (4), 1197-1199 (1996).
- Avcilar-Kucukgoze, I., et al. Discharging tRNAs: a tug of war between translation and detoxification in Escherichia coli. Nucleic Acids Research. 44 (17), 8324-8334 (2016).
- Mundhada, H., Schneider, K., Christensen, H. B., Nielsen, A. T. Engineering of high yield production of L-serine in Escherichia coli. Biotechnology and Bioengineering. 113 (4), 807-816 (2016).
- Makosky, P. C., Dahlberg, A. E. Spectinomycin resistance at site 1192 in 16S ribosomal RNA of E. coli: an analysis of three mutants. Biochimie. 69 (8), 885-889 (1987).
- Feng, L., Tumbula-Hansen, D., Toogood, H., Söll, D. Expanding tRNA recognition of a tRNA synthetase by a single amino acid change. Proceedings of the National Academy of Sciences of the United States of America. 100 (10), 5676-5681 (2003).
- Naganuma, M., et al. The selective tRNA aminoacylation mechanism based on a single G• U pair. Nature. 510 (7506), 507 (2014).
- Hoesl, M. G., et al. Chemical evolution of a bacterial proteome. Angewandte Chemie International Edition. 54 (34), 10030-10034 (2015).
- Hershberg, R., Petrov, D. A. Selection on codon bias. Annual Review of Genetics. 42, 287-299 (2008).
- Huo, Y. -. X., et al. Conversion of proteins into biofuels by engineering nitrogen flux. Nature Biotechnology. 29, 346 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。