Method Article
ES細胞におけるハイスループットスクリーニングを - マウスにおけるストレス抵抗性遺伝子を明らかにフォワード遺伝的アプローチ
要約
ストレス耐性は、長寿のための顕著な特徴の一つであり、遺伝的に支配されることが知られています。ここでは、寿命試験のためのマウスモデルを開発したとのES細胞におけるストレス耐性を付与する突然変異をスクリーニングするために公平な高スループットの方法を開発しました。
要約
Phenotype-driven genetic screens in mice is a powerful technique to uncover gene functions, but are often hampered by extremely high costs, which severely limits its potential. We describe here the use of mouse embryonic stem (ES) cells as surrogate cells to screen for a phenotype of interest and subsequently introduce these cells into a host embryo to develop into a living mouse carrying the phenotype. This method provides (1) a cost effective, high-throughput platform for genetic screen in mammalian cells; (2) a rapid way to identify the mutated genes and verify causality; and (3) a short-cut to develop mouse mutants directly from these selected ES cells for whole animal studies. We demonstrated the use of paraquat (PQ) to select resistant mutants and identify mutations that confer oxidative stress resistance. Other stressors or cytotoxic compounds may also be used to screen for resistant mutants to uncover novel genetic determinants of a variety of cellular stress resistance.
概要
長寿はストレス耐性との親密な関係を持っています。一般的には、長寿命の種は、多くの場合、過酸化水素、パラコート(PQ)、UV、熱、及び重金属1,2のような複数のストレスに向かって抵抗が増大し実証します。対照的に、ストレスに対する感受性の増大を短縮寿命および/または複数の疾患を起こしやすい表現型を予測する傾向があります。抗酸化掃気経路は長く、動物にストレス耐性を付与するに主要な役割を果たしていると推測されています。しかし、いくつかの例外を除いて、様々な抗酸化酵素 (例えば、SOD)での操作とトランスジェニック動物の多様からの研究では、酸化剤捕捉酵素のレベルを増加させると、寿命や健康スパン3を増加させないことを示しています。これらのデータは、一貫して長寿命の動物で観察されたストレス耐性形質がまだ発見される他の細胞経路によって媒介されることを示唆しています。
我々は公平な前進を取りました上に変異遺伝子を同定するための遺伝学的アプローチは、培養された胚性幹(ES)細胞におけるストレス耐性表現型を付与することができました。 ES細胞は、この研究において二つの大きな利点を提供する:(1)高度な遺伝子操作は、ES細胞のゲノムを改変するために利用可能です。 (2)画面から回収ストレス耐性ES細胞を直接全動物試験への急速な変換が寿命及び健康スパンを測定することができ、マウスの製造に使用することができます。
本稿では、BLMの対立遺伝子が、テトラサイクリン応答性エレメントの制御下にあったここでC9のES細胞株の使用を説明しました。ドキシサイクリン(DOX)の治療は、一時的に姉妹染色分体交換の増加事件につながるBLMの発現をオフになっています。この短期BLMノックアウトストレス耐性の劣性突然変異はスクリーニングプロセスで捕捉できるようにヘテロ集団内のホモ接合変異を生成するために許可されています。我々また、ランダムにゲノム中の遺伝子を変異させるポリトラップカセット(PB-UPA)を挿入するための変異原としてのpiggyBac(PB)、トランスポゾンの使用を記載しています。ポリトラップによる遺伝子の破壊を有する細胞はG418耐性となり、遺伝子トラップ変異体(遺伝子トラップライブラリー)の収集が行われ、その後、ストレス耐性た変異体クローンについてスクリーニングすることができるように回収することができました。
選択から回収したストレス耐性クローンは、挿入の数(定量PCR)の点で、分子技術によってかなり迅速に特徴付けることができ、挿入部位(splinkerette PCR)、破壊された遺伝子(BLAST)の正体、およびその発現レベル( RT-qPCRを)。 PBの挿入は、野生型DNA配列を復元するため、ストレス耐性の損失をテストするために、クローンでMPBトランスポザーゼの一過性発現によってremobilizedすることができます。これらは、前に行われるべきである変異の因果関係を確認する強力な方法であります高価なマウスの生産に。以前の研究では、ストレッサーにさらされた細胞は、その多能性4,5を失ったことを示しました。したがって、このプロトコルでは、ストレス因子で処理されない変異体細胞の複製セットの保存が成功したマウスの生産のために重要です。
私たちの研究室では、両方が要求に応じて他の研究者に利用可能であり、C9 ES細胞株およびPB-UPAベクトルを開発しました。プロトコルは、ここで報告されたストレス耐性クローン( 図1B)を分離するためにレプリカプレーティング、ストレスの選択に続いて、PB-UPA( 図1A)との遺伝子トラップされたES細胞のデノボライブラリーの生成によって開始されます。我々はパラコート、細胞内で強力なラジカル発生剤を用いた選択性を示しました。事実上、任意の細胞毒性化合物または毒素、例えば、ERのストレス要因(例えば、タプシガルギンおよびツニカマイシン)、神経細胞の酸化剤(例えば、MPP +、6-ヒドロキシドーパミン、およびロテノン)、熱、および重いです金属( 例えば、CD、SE)は、それぞれの耐性変異株を選択するための方法に適合させることができます。
プロトコル
1.ジーン・トラップされたES細胞ライブラリーの構築のpiggyBacトランスポゾンを使用します
- ES細胞培養用フィーダー等の一次マウス胚線維芽細胞(PMEF)を準備
- 37℃の水浴中でマイトマイシンC不活性化PMEF(5.0×10 6)の1バイアルを解凍します。 15%FBS、白血病阻害因子1,000単位/ ml、100μMの非必須アミノ酸、2mMグルタミン、55μM2-メルカプトエタノール、25単位/ mlのペニシリン/ストレプトマイシンを含有するES細胞培地(DMEM 5mlのに細胞を移します)、5分間100×gで遠心します。
- 上清を吸引除去し、2 T25フラスコ(それぞれ5ml)と2つの100 mmプレート(各10ml)の中に分散させることによって、続いてES細胞培地30mlに細胞を再懸濁。 24時間37℃、5%CO 2でインキュベートします。
- C9 ES細胞を培養し、拡大
- 37℃の水浴中でC9 ES細胞の1バイアル(0.5 mlの2.5×10 6細胞)を解凍し、転送ES細胞培地5mlに細胞。 5分間100×gで遠心分離します。
- T25フィーダフラスコにプレーティングすることによって、その後上清およびES細胞培地5ml中に再懸濁したES細胞を吸引します。 37℃、5%CO 2でインキュベートします。毎日のES細胞培地を交換してください。
- 培養2日後、ES細胞は、〜80%のコンフルエントになると、継代される準備ができている必要があります。 (2+及びMgのCaなし)5mlのPBSで細胞を洗浄した後、2.5ミリリットルを追加トリプシンEDTA、0.25%を予め温めておきました。 10分間37℃でインキュベートします。トリプシンを停止する2.5ミリリットルES細胞培地を追加します。細胞塊を破壊するまでピペット上下15回。 5分間100×gで遠心分離します。
- ES細胞培地4mlに上清および再懸濁細胞を吸引します。転送9ミリリットルのES細胞培地を含む100 mmのプレートあたりの細胞の1ミリリットル。 2つの100 mmプレートを準備します。 8通路:これは1です。 10 mLのES細胞培地で毎日細胞を養います。
- ライブラリの96ウェルフィーダープレートを準備
- 1.1.1で説明したように、マイトマイシンCの解凍3バイアルPMEFを不活性化し(それぞれ5×10 6個の細胞を含む)と33.3 mLのES細胞培地を各バイアルからの細胞を再懸濁します。 100mlの細胞懸濁液を得るために細胞を結合します。
- ウェルに10 96ウェルプレート当たり板100μlの細胞。これらの96ウェルフィーダープレートがPBトランスポゾンとC9 ES細胞をトランスフェクトする前に、少なくとも1日に準備する必要があります。
- PB-UPAベクターとC9 ES細胞のエレクトロポレーション
- トリプシン処理し、自動化細胞カウンターを使用して拡張C9 ES細胞をカウントします。 10mlのPBSでC9 ES細胞の各100 mmのプレートを洗浄します。 8 mlを加え、0.25%トリプシン-EDTAを予め加温し、10分間37℃でインキュベートします。
- 2ミリリットルのES細胞培地を追加することにより、トリプシンを停止します。細胞塊をバラバラ15 mlチューブとピペット上下15倍に細胞懸濁液を転送します。すぐに上清を吸引し、続いて5分間100×gで遠心します。
- 再懸濁細胞10 mLのES細胞培地中のSおよび血球計を使用してES細胞を数えます。 5×10 6個のES細胞を含む細胞懸濁液の6 15 mlチューブそれぞれを準備します。 5分間100×gで遠心分離します。上清を吸引除去し、0.8 mlのPBSで各細胞ペレットを再懸濁します。
- 6エレクトロポレーションキュベット(4ミリメートルギャップ)0.8ミリリットルの細胞を転送するための準備をします。各キュベットに、1μgのPB-UPAおよび20μgMPBトランスポゼース(mPBase)を追加します。ピペッティングにより穏やかに混合します。氷の上にキュベットを置きます。 250 Vで、指数の設定を使用してエレクトロポ、500μF、および無限大のΩでエレクトロポレーションを行ってください。
注:一般的には、時定数が5×10 6個の細胞あたり約1,500〜2,000 G418耐性形質転換体を得成功エレクトロポレーションのために約10〜12ミリ秒です。 - 98ミリリットルのES細胞培地を含むT75フラスコにトランスフェクトされた細胞を取得し、プール。穏やかなピペッティングにより細胞を混合した後、貯蔵容器(50ミリリットル)に細胞を移します。米国12チャネルピペットをる、(全部で10)先に調製した96ウェルフィーダープレートにウェル当たり細胞100μlを移します。 37℃、5%CO 2でインキュベートします。
- 24時間後、遺伝子トラップされたクローンを選択するために、新鮮なES細胞培地を含むG418(150μgの/ ml)で毎日細胞を養います。
注:一般的に、各ウェルについての10のG418耐性コロニーが含まれています。これらのコロニー(通常5-7日)十分な大きさに成長すると、96ウェルプレート(ライブラリ)は、それぞれストレージおよび選択のための2セットに複製されます。この場合、10個のサブライブラリーがあるので、ライブラリーの各プレートは、「サブライブラリー」と呼ばれます。
2.ライブラリレプリケーション
- マスタープレートを複製
- 24〜48時間50〜60%コンフルエントに達したG418耐性コロニーの前に、フィーダー(1.3節を参照)のような10の96ウェルプレート上にマイトマイシンC不活性化PMEFの別の3バイアルを解凍。ゼラチン少なくとも15 37℃で0.1%ゼラチン(100μl/ウェル)をインキュベートすることによって10の96ウェルプレートをIZE - 30分。 O / Nインキュベーションが許容されます。
- トリプシン処理により96ウェルプレート中に単一細胞懸濁液を調製します。吸引除去し、96ウェルプレートからの培地と( の Ca 2+、Mg 2+を含まない)等量のPBSで1回洗浄。予め温めておいた0.25%トリプシン-EDTAを50μl添加し、10分間、5%CO 2中、37℃でインキュベートします。 ES細胞培地50μlのトリプシンを停止します。マルチチャンネルピペットでダウン15回ピペッティングにより細胞塊をバラバラ。
- 転送すでに150μlのES細胞培地を含むフィーダープレート(マスター)と、すでに150μlのES細胞培地を含むゼラチン化プレート(レプリカ)の対応する行に残りの50μlに対応する行にトリプシン処理した細胞懸濁液の50μlの。マスタとレプリカのプレートへの細胞のそれぞれの行を処理します。今200μlのCを含む(プレートをインキュベート37℃、5%CO 2、O / Nでulture容量)。
注:20 96ウェルプレートが存在することになります。 - 翌日、150 / mlのG418を含有する100μlの新鮮なES細胞培地で細胞を養います。ウェルは80〜90%コンフルエントになるまで、毎日培地を交換してください。
- マスタープレートを凍結
- マスタープレートが約80%コンフルエントである場合(セクション2.1.2を参照してください)説明したように、細胞をトリプシン処理。
- プレートを取り外し、凍結培地の2倍の50μlのトリプシン反応を停止(60%ES細胞培地、20%FBS、20%DMSO)。ピペットアップおよびマルチチャンネルピペットで10回上下凍結培地の適切な混合を確実にします。
- 密閉発泡スチロールの箱に96ウェルプレートを入れて、最大4つのヶ月保存のために-80℃の冷凍庫に入れてください。四ヶ月は、ストレスの選択を完了すると、マスタープレートからのウェルが関心のクローンが含まれているかを識別するのに十分な時間を許可します。
注:-80&#での保存176; Cよりも長い4ヶ月は、細胞生存率の低下になります。一つは、貯蔵期間の延長のために液体窒素の気相中で保存することができる96ウェルフォーマットのクライオチューブを利用することができます。
- レプリカプレートからのDNAプレートおよびサブライブラリープールを準備します
- 24〜48時間前にDNAプレートおよびサブライブラリープールにさらに複製するには、サブライブラリープールのマイトマイシンC不活性化PMEFで10 100 mmプレートを準備し、10はプレートが複製DNAのための96ウェルプレートをゼラチン化。一般的な手順については、1.3節を参照してください。
- 96ウェルレプリカプレートで細胞をトリプシン処理(セクション2.1.2を参照してください)。 10分間のトリプシン処理工程と、DNAプレートから吸引ゼラチン中およびG418(150μgの/ ml)を含有する150μlのES細胞培地と交換してください。また、100 mmの給電板に「サブライブラリー」のプーリングのES細胞培地5mlを含む貯蔵を準備します。
- トリプシン処理後、試して停止12チャンネルピペットを使用して一度にES細胞培地1行の50μlのPSIN。ピペットは、最大10回上下細胞塊をバラバラにします。
- 転送50 DNAプレートの対応する行への細胞懸濁液のμL及び5 mLのES細胞培地を含む容器に残りの50μlを移します。つの完全96ウェルプレートを完了した後、プールした細胞が転送された100 mmの給電プレートの培地を吸引除去します。これらのプールされた突然変異誘発の細胞は「サブライブラリー」として指定されます。
- 残りの9の96ウェルプレートについて、この手順を繰り返します。 96ウェルDNAプレートを供給し、100-MMはG418を含むES細胞培地(150μgの/ mlで)を毎日「サブライブラリー」プレートをプールしました。
注:10の96ウェル「DNA」のプレート、および10の100ミリメートル「サブライブラリー」のプレートが存在することになります。
- DNAプレートをフリーズします。 DNAプレート内の細胞が80〜90%コンフルエントである場合、培地を吸引、100で細胞を2回洗浄後で、ゲノムDNA単離6 -20℃でPBSおよびストアをμlで。これらのDNAプレートは、「マスター」プレートにも対応する関心のクローンが含まれている識別するために、PCRによってスクリーニングされます。
3.ドキシサイクリン誘導性のホモ接合体変異体
- ドキシサイクリン処理のためのサブライブラリープール、継代細胞を凍結
- 70〜80%の密集度が得られるのに十分な大きコロニーを持つ100mmの「サブライブラリー」プレートの前に24時間は、ドキシサイクリン処理のために10糊化100 mmプレートのセットを準備します。
- 各サブライブラリーの単一細胞懸濁液を準備する(セクション1.4.1および詳細については、1.4.2を参照のこと)。ドキシサイクリン(1μg/ ml)とゼラチン化100 mmのプレートへの転送5×10 6細胞; 37℃、5%CO 2でインキュベートします。 1バイアル当たり5×10 6個の細胞で残りの非ドキシサイクリン処理したサブライブラリープールをフリーズします。
- 存在下で継代細胞BLMをノックアウトするためにドキシサイクリン
- パッセージ1のサブライブラリ:まで2週間ドキシサイクリン(1μg/ ml)の存在下で8ごとに2〜3日は、その間にノックアウトBLMのセルにヘテロ接合性の消失を促進するDOX誘導性の人口、ホモ接合変異体の生成を可能にします。
4.ストレスの選択と耐性クローンの単離
- ストレスの選択の対象とドキシサイクリン処理細胞
- 、準備前のストレスの選択にドキシサイクリン処理した細胞を継代する24時間の10は、37°C、5%CO 2加湿インキュベーター中、150 mmプレート、場所を糊化。
- トリプシン処理することにより、各サブライブラリーの単一細胞懸濁液を(詳細はセクション1.4.1と1.4.2を参照)を用意。細胞を均一に分配する、30ミリリットルの総培養容積の各150 mmのプレートに6.6×10 6個の細胞をシード。ストレッサー(10μMパラコートまたはβメルカプトエタノールの省略で細胞をインキュベートし、生存細胞は、ピッキングのためのコロニーに成長することを可能にするために、通常のES細胞培地と交換し、その後7日間FBSを不活性化し、7.5%の熱)を含むES細胞培地です。
- オプション:1バイアルあたり5.0×10 6個の細胞で、ドキシサイクリン処理細胞の上で左をフリーズします。
- ストレス耐性コロニーを選択してください
- 一週間の通常のメディアを復活した後、耐性コロニーが存在しているどのように多くのストレスを観察します。選ばれるべきコロニーの数に応じて、(詳細はセクション1.1を参照)事前に応じて96ウェルプレート1日にマイトマイシンC不活性化PMEFとの十分な井戸を準備します。
- 採取の日に、吸引及び100μlで96ウェルフィーダープレートに培地を交換し、新鮮なES細胞培地、37℃、5%CO 2の加湿インキュベーターに戻し置きます。この時点で、50μlの0.25%トリプシン-EDTAでU底96ウェルプレートを調製し、ウェルあたり。 150 mmのストレス選択プレートから培地を吸引し、電子メールを追加PBSのQUALボリューム。
- 2μlにマイクロピペットを設定し、ウェルあたり1つのコロニーを選んで、ウェルを含むU底トリプシンにコロニーを生き残った「選びます」。それはコロニーの所望の数をピッキングされるまでプレートを室温で放置することが許容可能です。その後、10分間、37℃、5%CO 2でU底プレートをインキュベートします。
注:一般的に、我々は96個のコロニーを選択する1時間を取ります。 - マルチチャンネルピペットを用いて50μlのES細胞培地で一度トリプシン反応を一行を停止します。ピペットは、上下に15回の単一細胞懸濁液を得て、すでにES細胞培地100μlを含むフィーダープレートの対応する行に100μlの細胞を転送します。プレート全体のための転送を完了します。 200μl中の培養細胞O / N。翌朝、培地を吸引し、100μlのES細胞培地と交換してください。
- 96ウェルプレートを複製
- 選んだコロニーのウェルは80〜90%のconflueある場合NT、2マッチングプレート、DNA単離のための1つの細胞のバックアップのため他に複製します。これらのプレートは、ゼラチン化され、マイトマイシンC不活性化PMEFが含まれていません。
- 培地を吸引し、100μlのPBSで洗浄します。 37℃で10分間インキュベートし、各ウェルに50μlの0.25%トリプシン-EDTAを加えます。 、150μlのES細胞培地でトリプシンを停止ピペッティングにより混合し、2つの96ウェルプレートの対応する行に100μlの細胞懸濁液を移します。毎日培地交換。
- プレートは80〜90%コンフルエントである場合には、「バックアップ」(詳細はセクション2.2を参照)のような一組のプレートを凍結します。プレートの他のセットは、以下にさらに記載されるように展開されます。
- ゲノムDNAの単離のために細胞を拡大
- 96ウェルプレートのDNA(詳細は2.1.2を参照のこと)で、単一細胞懸濁液を準備します。
- MICRを使用して24ウェルプレート(合計4)のウェルに、96ウェルプレートの各個々のウェルからの細胞懸濁液を転送しますopipette。 「選んだ」コロニーが起源の「クローン」であるため、任意のセルの交差汚染が存在しないことを確認してください。 0.5 mLのES細胞培地中の培養細胞、ウェルを80〜90%コンフルエントになるまで培地を毎日変えます。
注:24ウェルプレートは、PCR分析のためのゲノムDNAの十分な収量を可能にします。
PB挿入サイトとトラップされた遺伝子の同定5。
- 24ウェルプレートからのゲノムDNAの単離:24ウェルプレート中の細胞を溶解し、RNAフリーのゲノムDNAを単離します。
- 7,8に記載されているように 、PBトランスポゾン組込み部位に隣接する配列断片を生成するために、Splinkerette PCR(SpPCR)を実行します。
注:ハンズオンタイム毎日が最小ですがSpPCRは、5日間のプロセスです。
変異クローンの6遺伝子解析
- よくマスターを見つけ、興味のクローンを精製します。
- PBの組込み部位はされている場合にはマッピングされ、8 PBリバースプライマー(例えば、PB3'-1)で使用する組み込み部位の上流にフォワードプライマーを設計し、ストレス耐性クローンが選ばれたのと同じ「サブライブラリー」に対応するDNAプレートを選別。全体の96ウェルのDNAプレート全体に一つだけ陽性ウェルを見つけることを期待しています。フィーダー上の24ウェルプレートに「マスター」プレートから正確なだけでなく、これを解凍し、80〜90%の集密度になるまで細胞を増殖させます。
注:この時点では、この井戸は、遺伝子トラップされたクローンの混合物が含まれています。 - 24ウェルプレートを80-90%コンフルエントである場合、細胞をトリプシン処理し、(単一細胞懸濁液を確保する)100 mmの給電板1,000 ES細胞をプレート。航路拡張用フィーダーのT25フラスコに残った細胞。コロニーは80〜90%コンフルエントのときT25フラスコから細胞を凍結。バックアップとしてT25フラスコあたり4バイアルをフリーズします。
- コロニーは7日間増殖した後、96ウェルFEに96個のコロニーを選択することを許可しますエダープレート。手順を選ぶためのセクション4.2を参照してください。 「DNA」プレートにプレート準備を複製し、プレート「-master 第 2回」などの他の凍結(凍結については、セクション2.2を参照してください)。
- コロニーは80〜90%の密集度に達することを許可します。興味の精製されたクローンを含むウェルを見つけるために、DNAプレートを画面。 24ウェルに、その後T25フラスコ(すべてのフィーダ上)に96ウェルの第 2 -masterプレートから細胞を展開します。 T25フラスコあたり4バイアルをフリーズします。
注:この時点で、遺伝子トラップされた細胞株はクローンであり、ストレッサに曝露され、そしてマウスの生産に適していません。
- PBの組込み部位はされている場合にはマッピングされ、8 PBリバースプライマー(例えば、PB3'-1)で使用する組み込み部位の上流にフォワードプライマーを設計し、ストレス耐性クローンが選ばれたのと同じ「サブライブラリー」に対応するDNAプレートを選別。全体の96ウェルのDNAプレート全体に一つだけ陽性ウェルを見つけることを期待しています。フィーダー上の24ウェルプレートに「マスター」プレートから正確なだけでなく、これを解凍し、80〜90%の集密度になるまで細胞を増殖させます。
- ストレス耐性細胞株の1つのバイアルを解凍し、ゼラチン化T25フラスコに細胞を展開します。PB挿入の数を決定します。ゲノムDNAを分離し、PBトランスポゾンのコピー数を確認してください。これは、ネオマイシン遺伝子を標的定量PCRにより達成することができます。
- 遺伝子発現レベルを測定します。confi後関心の細胞株における単一のPB統合イベントがあるrming、各遺伝子に特異的なTaqManプローブを用いたRT-qPCRによりトラップされた遺伝子の発現レベルを測定します。
- 配列変異体のmRNA:遺伝子トラップカセットのスプライスアクセプター配列のエキソンおよび下流プライマーアニーリングに上流プライマーのアニーリングを用いてPCRによりcDNAアンプリコンを生成します。シーケンスcDNA断片は、予想されるスプライシングがトラップされた遺伝子で発生していることを確認します。
- トラップされた遺伝子とストレス耐性との因果関係をテストするためのpiggyBacトランスポゾンをRemobilize。
- 24〜48時間1 T25フラスコにPB remobilization、解凍プレートDR4フィーダーと2つの100 mmプレート用の細胞を解凍する前に。 DR4フィーダーは、T25フラスコ上にプレーティングされた後、少なくとも1日、PBの挿入を含む細胞系の1つのバイアルを解凍し、DR4フィーダーを含むT25フラスコに細胞をプレー。 ES細胞培地中で細胞を増殖させます48時間、培地を毎日変えます。
- ES細胞の単一細胞懸濁液を準備します。自動細胞カウンターで細胞をカウントし、15 mlチューブに5.0×10 6個のES細胞を移します。 100×gで遠心分離し、800μlのPBSで細胞を再懸濁します。 4ミリメートルのキュベットにサスペンションを転送します。
- ピペッティングにより混合し、キュベットに20μgのmPBase プーロを追加し、エレクトロポレーション細胞(詳細はセクション1.4を参照してください)。 2枚の100mm DR4フィーダープレート上にトランスフェクトされた細胞を配布します。 ES細胞培地中の培養細胞O / N。
- 翌朝、48時間に1μg/ mlのピューロマイシンと文化細胞とES培地で細胞を養います。 5日間細胞をピューロマイシン選択を撤回し、培養を継続します。コロニーはピッキングのために十分に大きい場合、96ウェルフィーダープレート(セクション4.2を参照)に選びます。
- 採取細胞が80-90%コンフルエントに達したとき、DNAプレートにプレートの1/6 番目に分割し、PLの1/6 番目(凍結のためのセクション2.2を参照してください)凍結する原版内のセルの2/3 RDSを残し 、G418プレートに食べました。
- ネオマイシン耐性の損失をテストするために150μg/ mlのG418の後のDNAの単離のためのES細胞培地、およびG418プレート文化のDNAプレート。 G418を含む培養液中で72〜96時間後には生存細胞を含むウェルは、遺伝子トラップPBトランスポゾンの成功remobilizationを示しています。
- 「DNA」プレートにおける対応するウェルは削除されましたPBトランスポゾンを確認するためにPCRによってスクリーニングされます。 DNAプレートを90%コンフルエントである場合には、野生型配列の回復と復帰を確認する手段として、ネオIRESの不在のためのPCRスクリーニングのため、96ウェルプレートフォーマット4でゲノムDNAを単離します。
- 24ウェルプレートにマスタープレートからマッチング井戸を解凍し、T25フラスコ(フィーダを持つすべての)に展開されます。バックアップとしてT25フラスコごとに4つのバイアルを凍結。
- マスタープレートから耐性ES細胞を回収し、キメラを生成するために、胚の注入のためにそれらを使用しています。約15-20 ES細胞は、個々のC57BL / 6胚盤胞に注入されます。偽妊娠の雌(株ICR)の子宮にマウスあたり15個の胚盤胞を転送します。生殖系列伝達をテストするために、および変異マウスのコロニーを構築するために、いくつかの若い処女C57BL / 6J雌とごみから回収された(ES細胞の特定のアグーチ毛色によって示されている)男性キメラメイト。
- 単離および活性酸素種(ROS)レベルの抵抗のためにF1突然変異マウスおよび野生型同腹子からテスト尾皮膚線維芽細胞パラコート9。
マウス変異体の7世代
結果
典型的な突然変異誘発実験では、トランスフェクションは、PB-UPAとMPBトランスポゼースで3×10 7 ES細胞の合計で構成されています。 2つの独立した実験で生成された遺伝子トラップされたES細胞の数を表1に要約されている。遺伝子取り込みの効率は約0.04%です。組み合わせ遺伝子トラップライブラリは、マウスゲノム(23000コードする遺伝子)を完全にカバー約22,400の独立した変異体を含みます。過剰なカバレッジを繰り返し変異誘発を介して複数の変異体を生成し、および/またはプロセスをスケールアップすることによって達成することができます。
遺伝子トラップライブラリーは、ランダム変異を含むので、ストレス耐性の表現型を有する変異体の数を予測することは不可能です。 PQ抵抗のための2つの独立した選択を行いました。 22440独立した遺伝子トラップ突然変異の組み合わせスクリーニングはPQに対する細胞の抵抗(0.08%)( 表1)を付与する17の変異を発見しました。
私たちは、さらなる分析とマウスの生産のためのマスタープレートから、3変異体のクローン、Pigl、Tiam1と、とRFFLを精製しています。ヘテロ接合RFFL変異を有する細胞は、正常にBLMをノックアウトすることにより、ホモ接合性子孫を産生するように誘導しました。しかし、我々はPiglとTiam1と突然変異体から、おそらくホモ接合連結細胞致死に任意のホモ接合変異体を生成するために失敗しました。 2-メルカプトエタノール(2-ME)、およびPQの省略( 図2)。これらの精製されたクローンのストレス耐性は、ストレスの2つの異なる種類を使用することによって確認しました。これらのクローンからのPBを除去する際に、関連するストレス耐性表現型は、遺伝子トラップ突然変異が原因因子であることが確認された、( 図2)を失いました。それが観察された表現型の原因としてストレス選択時に確率的病変を除外することになるとして、マスタープレートから回収されたクローンの特性は非常に重要です。3のうち、マウスの製造( 表2)のための胚盤胞に導入される突然変異体ES細胞、2行(PiglとTiam1との)生殖系列伝達を示します。 RFFL注入は、そもそもキメラの最適な数ではありませんでした唯一の女性キメラを生成します。したがって、RFFLの生殖系列伝達の失敗は、1無能なキメラの可能性があります。我々はPigl変異マウスから単離された皮膚線維芽細胞の細胞表現型をテストし、彼らはPQ( 図3)に減少し、内因性活性酸素種(ROS)のレベルだけでなく、ストレス耐性を維持することを示しました。
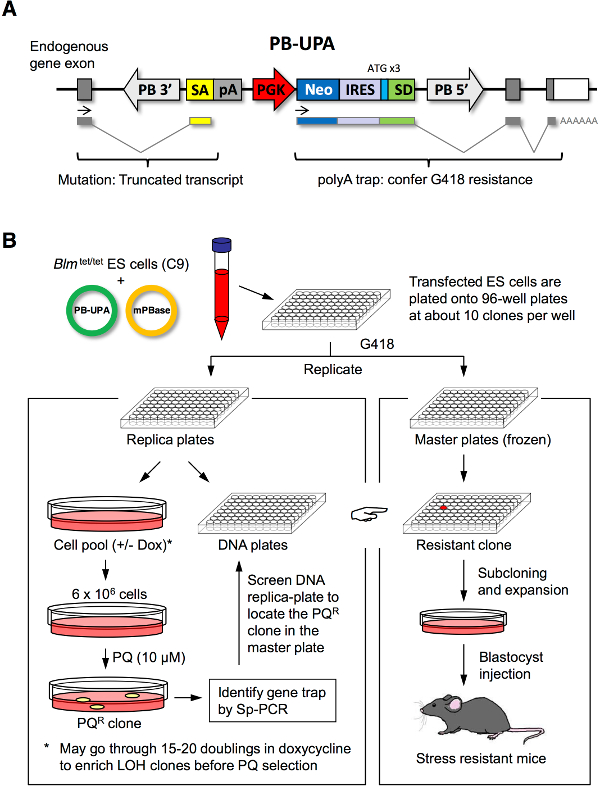
1 ES細胞の突然変異誘発およびストレス選択図。 (A)PB-UPAベクトル。スプライスアクセプター(SA)、ウシ成長ホルモンポリアデニル信号(PA)からなる公平なポリA(UPA)トラップベクター、phosphoglycerateキナーゼ(PGK)プロモーター、ネオマイシンホスホトランスフェラーゼ(ネオ)、内部リボソーム侵入部位(IRES)、及び(3つの異なるリーディングフレーム内)スプライスドナー人工ATGと(SD)が3 'で隣接し、 のpiggyBacトランスポゾンにクローニングしました。および5 'の長い末端反復(LTR)。ストレス耐性クローンについて(B)ランダムES細胞の突然変異誘発および選択。 ES細胞は、96ウェルプレート上にプレーティングすることにより、続いてPB-UPAとmPBaseとエレクトロポレーションによってコトランスフェクトしました。遺伝子トラップされたクローンは、G418耐性により選択しました。集密後、それらは2複製セットに分けました。一つのレプリカセットは、さらに、パラコート(PQ)によるDNAの分離とストレスの選択のための2つの半分に分割しました。他のレプリカセット(マスター)がダウンして凍結しました。ストレス処理から回収された生存しているES細胞コロニーは、SP-PCRによるPBを挿入するための分子解析しました。プライマーは、その後、そこからよくsibliを含むレプリカのDNAプレートをスクリーニングするために設計されましたNG PQ Rクローンを 、マスタープレート上に配置することができます。これらの細胞は、異なるストレス因子に対するストレス耐性アッセイのために、マウスの産生のためにです。チックら 7から変更された。 この図の拡大版をご覧になるにはこちらをクリックしてください。

突然変異体ES細胞内での図2ストレス耐性。 (A)2-メルカプトエタノール(2-ME)撤退への抵抗。 (B)パラコート(PQ)に対する抵抗性。以前の研究5から回収された:(赤4C11)、および3つの遺伝子トラップクローン(灰色)、ストレス耐性制御ES細胞クローン:親の野生型ES細胞(黄色C9)が示されています。細胞は、生存細胞の数をカウントした後、二日間のストレス処理を行いました。 < em>のPigl、Tiam1と、とRFFLヘテロ接合体 。(PB / +:ダークグレイ)これらのストレス要因の両方に展示抵抗がRFFLホモ接合体 (PB / PB:黒)はヘテロ接合体と比較して、より強いストレス耐性を示します。 (:ライトグレー+ / +)の両方のストレス要因に対する耐性は、PBの挿入が削除されたときに失われました。エラーバーは平均のSDを表す(N = 4)。 * P <0.001、#1、P = スチューデントのt検定によって評価遺伝子トラップクローンおよび野生型親C9、間に0.01。 PQ(10μM)治療中の(C)は 、ES細胞コロニー形成。ストレス耐性ESクローンは、野生型クローンからのコロニーの数およびサイズを大幅に低減しながらPQ処理下での培養中でコロニーを形成することができました。チックら 7から変更された。 この図の拡大版をご覧になるにはこちらをクリックしてください。
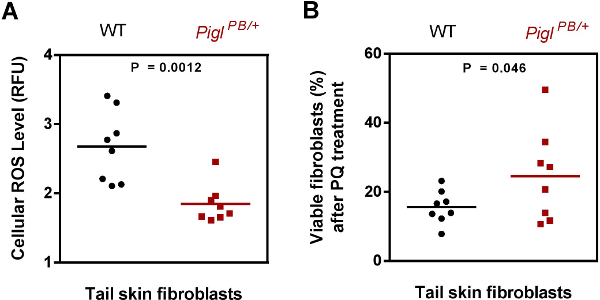
Pigl PB / +線維芽細胞の図3キャラ。(A)活性酸素種(ROS)レベル。野生型(WT)およびPigl PB / +マウスから単離した尾皮膚線維芽細胞を、CM-DCFCAで染色し、蛍光をヘキストに対して正規化しました。 ROS含有量は相対蛍光単位(RFU)として表しました。 P値は、両側検定、スチューデントのt検定により評価した(N = 8)。 (B)PQ抵抗。野生型(WT)およびPigl PB / +マウスからの尾皮膚線維芽細胞は、細胞の生存能力は、MTTアッセイによって測定した後の6時間、4 mMのPQに曝露しました。 PQ処理後の生細胞のパーセンテージを、PQ処置CEから得られた吸光度の比で計算しました。LLSおよび未処理細胞からの。 P値は、1つの両側スチューデントのt検定により評価した(N = 8)。チックら 7から変更された。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| ライブラリ | サブライブラリー | ES細胞株 | 遺伝子トラップされたクローンの番号 | PQ Rクローンの番号 |
| 1 | C9PA01 - C9PA10 | C9(BLM TET / TET) | 9,000 | 7 |
| 2 | C9PA11 - C9PA20 | C9(BLM TET / TET) | 13,440 | 10 |
| トータル | 22440 | 17 |
表1 のpiggyBac遺伝子トラップライブラリー構築およびPQ耐性クローンの回収。チックらから変更された。7。
| ES細胞クローンを注入 | キメラの号を回復 | 生殖系列伝達* |
| Pigl PB / Pigl + | 4 | はい |
| Tiam1とPB / Tiam1と+ | 2 | はい |
| RFFL PB / RFFL + | 1 | ノー |
マウスの表2世代。チックら 7から変更されました。
*生殖系列伝達は、PCR遺伝子型決定により検出される遺伝子トラップ対立遺伝子を遺伝することにより確認しました。
ディスカッション
Forward genetic analysis allows for an unbiased interrogation of the genome for genes responsible for a specific phenotype. This method is very powerful to uncover novel gene functions. It has been widely used in lower organisms but not in mammal, such as the mouse, mainly due to the extremely high cost associated with the infrastructure and logistics that would entail. Here, we moved the genetic screening process to the ES cell culture platform, greatly increasing the efficiency and throughput in generating mutants and identifying mutations.
To effectively select for resistant mutants, a lethal dose of stressor and period of treatment needs to be established such that all of the wild-type cells would be killed. The higher the stringency of the selection, the fewer occurrence of false positive will result. Fibroblast feeders have a higher tolerance to oxidative stress than ES cells; the presence of feeders would rescue the nearby ES cells which would otherwise be killed by the stressor. Thus, omission of feeders during stress selection was very critical for an effective killing. In skin fibroblasts, serum deprivation was necessary to reveal the stress resistance phenotype in long lived mice 10. However, this application was not feasible in ES cells because they cannot survive without serum. As an alternative, we discovered that the use of heat inactivated serum at reduced level (7.5%) achieved a similar result.
In this report, we employed poly-A trap rather than a promoter trap because promoter trap will only trap actively transcribed genes, which is estimated to be 60% of the genome in ES cells, under normal conditions 11. In contrast, a poly-A trap would trap genes regardless of their transcriptional activities, in theory giving us the potential to screen for mutations in all genes in the genome. A major drawback of the conventional poly-A trap is the strong bias towards trapping the last exon, appeared to be mediated by an endogenous nonsense-mediated decay (NMD) mechanism 12. By incorporating the unbiased poly-A trap (UPA) strategy in the PB-UPA transposon to suppresses NMD 13, we were able to eliminate biased trapping.
One distinguishing advantage of using ES cells is that these pluripotent cells make direct derivation of mice possible, speeding up the transition to in vivo studies. For this to happen, the replica plating strategy is critical, preserving a subset of clones sheltered from manipulations that can diminish pluripotency, such as knocking out of Blm and exposure to stressors. We have shown that mutant ES cells recovered from these screens were capable of participating in forming the entire mouse and transmitting the mutations to the germline. Most importantly, consistent with previous studies, the stress resistance phenotype was also transmitted and maintained in the adult mouse.
We and others showed that a variant phenotype observed in ES cells resulting from environmental induced lesion or deliberated engineered mutations was able to transmit to the differentiated progeny cells 4,5,9,14, and probably the whole mouse. This protocol describes the utilization of ES cells as surrogate cells to screen for stress resistance mutations in the mouse. In addition to screening for stress resistance genes, this method may have a broader implication to screen ES cells, identify targets, and to develop a variety of mouse models with a phenotype of interest.
開示事項
The authors have nothing to disclose.
謝辞
We would like to thank the Wellcome Trust Sanger Institute for the gifts of piggyBac transposon and piggyBac transposase. This work was supported by the Butcher grant of Colorado and the NIH R01 AG041801 (W.S.C).
資料
| Name | Company | Catalog Number | Comments |
| Vector | |||
| PB-UPA | |||
| mPBase | |||
| mPBasePuro | |||
| Tissue Culture | |||
| 500-ml Stericup filters | EMD Millipore | SCGPU05RE | |
| 250-ml Stericup filters | EMD Millipore | SCGPU02RE | |
| 50-ml Steriflip-GV filters | EMD Millipore | SE1M179M6 | |
| KO DMEM | Life Technologies | 10829-018 | |
| DMEM | Sigma-Aldrich | D6429 | |
| FBS | Tissue Culture Biologicals | 104 | |
| Heat Inactivated FBS | Sigma-Aldrich | F4135-500 | |
| LIF | EMD Millipore | ESG1107 | |
| Non-essential Amino Acids | Life Technologies | 11140-050 | |
| GlutaMAX | Life Technologies | 35050-061 | |
| Pen/Strep | Life Technologies | 15140148 | |
| β-Mercaptoethanol | Life Technologies | 21985-023 | |
| Methyl Viologen dichloride (Paraquat) | Sigma-Aldrich | 856177 | |
| Dimethyl Sulphoxide Hybri-MAX | Sigma-Aldrich | D2650 | |
| EmbryoMAX 0.1% gelatin | EMD Millipore | ES006B | |
| DPBS/Modified | HyClone | SH30028.02 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| T25 Flask | Corning | 353108 | |
| T75 flask | Corning | 353135 | |
| 100-mm plate | Corning | 353003 | |
| 150-mm plate | Corning | 430599 | |
| 96-well plate | Corning | 3585 | |
| 96-well U-bottom plate | Corning | 3799 | |
| 24-well plate | Corning | 3526 | |
| 50-ml reservoir | Corning | 4870 | |
| 15-ml tubes | VWR International, LLC | 82050-276 | |
| Primary Mouse Embryonic Fibroblasts | EMD Millipore | PMEF-NL | |
| DR4 Mouse Embryonic Fibroblasts | Applied StemCell | ASF-1001 | |
| Mitomycin C | Fisher BioReagents | BP25312 | |
| Geneticin (G418) | Life Technologies | 11811-023 | |
| Doxycycline | Fisher BioReagents | BP26531 | |
| Cryotubes | Thermo Scientific | 377267 | |
| Centrifuge | Eppendorf | Centrifuge 5702 | |
| TC10 cell counter | Bio-Rad | ||
| Counting Slides (for TC10) | Bio-Rad | 1450011 | |
| Electroporation | |||
| Gene Pulser Xcell | Bio-Rad | 1652611 | |
| Gene Pulser Cuvettes (4 mm gap) | Bio-Rad | 1652088 | |
| Molecular Biology | |||
| Thermal Cycler | Eppendorf | Mastercylcer ep Gradient S | |
| Puregene Core kit B | Qiagen | 158745 | |
| Topo-TA Cloning kit | Life Technologies | 450030 | |
| High Capacity cDNA synthesis kit | Applied Biosystems | 4368814 | |
| NaCl | Fisher BioReagents | BP358-212 | |
| 100% ethanol | Decon Laboratories, Inc. | 2716 | |
| Double Processed Tissue Culture Water | Sigma-Aldrich | W3500 | |
| Sau3A1 | New England BioLabs | R0169L | |
| T4 DNA Ligase | New England BioLabs | M0202T | |
| EcoRV | New England BioLabs | R3195S | |
| 96-well Lysis Buffer (Ramires-Solis et al. 1992) | |||
| Trizma Base | Sigma-Aldrich | T1503 | |
| Hydrochloric Acid | Fisher BioReagents | A144-212 | |
| EDTA | Sigma-Aldrich | E5134 | |
| N-Lauroylsarcosine sodium salt | Sigma-Aldrich | L5777 | |
| Proteinase-K | Fisher BioReagents | BP1700 | |
| Electrophoresis | |||
| Mini-Sub Cell GT | Bio-Rad | 170-4469EDU | |
| LE Agarose | GeneMate | E3120500 | |
| Ethidium Bromide | Fisher BioReagents | BP1302 | |
| 100 BP DNA Ladder | New England BioLabs | N3231S | |
| 1Kb DNA Ladder | New England BioLabs | N3232S | |
| 2-log DNA Ladder | New England BioLabs | N3200L |
参考文献
- Johnson, T. E., et al. Longevity genes in the nematode Caenorhabditis elegans also mediate increased resistance to stress and prevent disease. J Inherit Metab Dis. 25, 197-206 (2002).
- Murakami, S., Salmon, A., Miller, R. A. Multiplex stress resistance in cells from long-lived dwarf mice. Faseb J. 17, 1565-1566 (2003).
- Perez, V. I., et al. The overexpression of major antioxidant enzymes does not extend the lifespan of mice. Aging Cell. 8, 73-75 (2009).
- Martin, G. M. Genetic engineering of mice to test the oxidative damage theory of aging. Ann NY Acad Sci. 1055, 26-34 (2005).
- Chick, W. S., Drechsel, D. A., Hammond, W., Patel, M., Johnson, T. E. Transmission of mutant phenotypes from ES cells to adult mice. Mamm Genome. 20, 734-740 (2009).
- Ramirez-Solis, R., et al. Genomic DNA microextraction: a method to screen numerous samples. Anal Biochem. 201, 331-335 (1992).
- Uren, A. G., et al. A high-throughput splinkerette-PCR method for the isolation and sequencing of retroviral insertion sites. Nat Protoc. 4, 789-798 (2009).
- Wang, W., Bradley, A., Huang, Y. A piggyBac transposon-based genome-wide library of insertionally mutated Blm-deficient murine ES cells. Genome Res. 19, 667-673 (2009).
- Chick, W. S., et al. Screening for stress-resistance mutations in the mouse. Front Genet. 5, 310 (2014).
- Salmon, A. B., et al. Fibroblast cell lines from young adult mice of long-lived mutant strains are resistant to multiple forms of stress. Am J Physiol Endocrinol Metab. 289, 23-29 (2005).
- Skarnes, W. C., et al. A public gene trap resource for mouse functional genomics. Nat Genet. 36, 543-544 (2015).
- Baker, K. E., Parker, R. Nonsense-mediated mRNA decay: terminating erroneous gene expression. Curr Opin Cell Biol. 16, 293-299 (2004).
- Shigeoka, T., Kawaichi, M., Ishida, Y. Suppression of nonsense-mediated mRNA decay permits unbiased gene trapping in mouse embryonic stem cells. Nucleic Acids Res. 33, e20 (2004).
- Symula, D. J., Zhu, Y., Schimenti, J. C., Rubin, E. M. Functional annotation of mouse mutations in embryonic stem cells by use of expression profiling. Mamm Genome. 15, 1-13 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved