Method Article
Approche génétique de l'avant pour découvrir les gènes de résistance au stress chez la souris - un écran haute-débit dans les cellules ES
Dans cet article
Résumé
La résistance au stress est l'une des caractéristiques de longévité et est connu pour être génétiquement régie. Ici, nous avons développé une méthode à haut débit impartiale pour le dépistage de mutations qui confèrent la résistance au stress dans des cellules ES avec laquelle de développer des modèles de souris pour les études de longévité.
Résumé
Phenotype-driven genetic screens in mice is a powerful technique to uncover gene functions, but are often hampered by extremely high costs, which severely limits its potential. We describe here the use of mouse embryonic stem (ES) cells as surrogate cells to screen for a phenotype of interest and subsequently introduce these cells into a host embryo to develop into a living mouse carrying the phenotype. This method provides (1) a cost effective, high-throughput platform for genetic screen in mammalian cells; (2) a rapid way to identify the mutated genes and verify causality; and (3) a short-cut to develop mouse mutants directly from these selected ES cells for whole animal studies. We demonstrated the use of paraquat (PQ) to select resistant mutants and identify mutations that confer oxidative stress resistance. Other stressors or cytotoxic compounds may also be used to screen for resistant mutants to uncover novel genetic determinants of a variety of cellular stress resistance.
Introduction
Longévité a une relation intime avec la résistance au stress. En général, les espèces à long terme démontrent souvent une résistance accrue vers de multiples facteurs de stress, tels que le peroxyde d'hydrogène, le paraquat (PQ), UV, de la chaleur, et des métaux lourds 1,2. En revanche, la sensibilité accrue au stress a tendance à prédire la durée de vie réduite et / ou un phénotype de la maladie plus sujette. La voie de balayage anti-oxydant a longtemps été supposé jouer un rôle important pour conférer une résistance au stress à l'animal. Cependant, à quelques exceptions près, les études d'une variété d'animaux transgéniques à des manipulations dans divers enzymes anti-oxydantes (par exemple, SOD) indiquent que l'augmentation du niveau d'enzymes d'évacuation des oxydants ne pas augmenter la durée de vie ou durée de la santé 3. Ces données suggèrent que le caractère de résistance au stress constamment observés chez des animaux à long terme est médiée par d'autres voies cellulaires encore être découverts.
Nous avons pris une avance impartialeapproche génétique pour identifier des gènes, qui sur muté, peut conférer un phénotype de résistance au stress chez souches embryonnaires (ES) de culture des cellules. Les cellules ES offrent deux avantages majeurs dans cette étude: (1) les manipulations génétiques sophistiqués sont disponibles pour modifier le génome des cellules ES; et (2) les cellules ES résistantes au stress récupérés de l'écran peuvent être directement utilisés pour la production de la souris, ce qui permet la traduction rapide dans les études animales entières pour mesurer la durée de vie et la durée de la santé.
Dans ce rapport, nous avons décrit l'utilisation de la lignée cellulaire ES C9, dans laquelle les allèles BLM étaient sous le contrôle d'un élément sensible à la tétracycline. Traitement de la doxycycline (Dox) transitoirement éteint l'expression de Blm conduisant à une augmentation des incidents échange de chromatides sœurs. Ce court-terme Blm knock-out a permis la génération de mutations homozygotes sein de la population hétérozygote sorte que des mutations récessives pour la résistance au stress pourraient être capturés dans le processus de sélection. nousdécrit également l'utilisation de piggyBac (PB) en tant que le transposon mutagene au hasard pour insérer un poly-Cassette de piégeage (PB-UPA) pour muter des gènes dans le génome. Les cellules avec la perturbation d'un gène par la poly-Un piège est devenu résistant à G418 et pourraient être récupérés de sorte qu'une collection de mutants de piégeage de gènes (bibliothèque de piégeage de gènes) pourrait être faite, et ensuite pour produire des clones mutants qui étaient résistant au stress.
Les clones résistants au stress récupérés à partir de la sélection peuvent être caractérisés assez rapidement par des techniques moléculaires en ce qui concerne le nombre d'insertions (qPCR), le site d'insertion (splinkerette PCR), l'identité du gène perturbé (BLAST), et son niveau d'expression ( RT-qPCR). L'insertion de PB pourrait être remobilisé par expression transitoire de MPB transposase dans le clone de restaurer la séquence d'ADN de type sauvage et donc tester pour la perte de résistance au stress. Ce sont des moyens puissants pour confirmer la causalité de la mutation, ce qui devrait être fait avantpour la production de souris coûteux. Des études antérieures ont montré que les cellules exposées à des facteurs de stress ont perdu leur 4,5 pluripotence. Ainsi, dans ce protocole, la préservation d'un jeu de répliques de cellules mutantes, qui ne serait pas traitée avec les facteurs de stress est critique pour la production de la souris succès.
Notre laboratoire a conçu la lignée cellulaire ES C9 et le vecteur PB-UPA, les deux sont disponibles à d'autres enquêteurs sur demande. Le protocole présenté ici va commencer par la génération de novo bibliothèque de cellules ES gène piégé avec PB-UPA (figure 1A), suivis par des répliques et la sélection de stress pour isoler des clones résistants au stress (figure 1B). Nous avons démontré la sélection avec le paraquat, un puissant générateur de radicaux libres dans les cellules. Pratiquement, tout composé cytotoxique ou une toxine, par exemple, les facteurs de stress ER (par exemple, la thapsigargine et tunicamycine), oxydant neuronale (par exemple, MPP +, la dopamine 6-hydroxy, et la roténone), la chaleur, et lourdmétaux (par exemple, Cd, Se), pourraient être adaptés à la méthode de sélection de mutants résistants respectifs.
Protocole
1. Gene-piégé cellules ES Bibliothèque Construction Utilisation piggyBac Transposon
- Préparer les fibroblastes embryonnaires de souris primaire (PMEF) que les mangeoires pour la culture de cellules ES
- Décongelez un flacon de mitomycine C-inactivé PMEF (5,0 x 10 6) dans un bain d'eau à 37 ° C. Transférer les cellules dans 5 ml de milieu de cellules ES (DMEM contenant 15% de FBS, 1 000 unités / ml de facteurs inhibiteurs leucémiques, 100 uM d'acides aminés non essentiels, 2 mM de glutamine, 55 pM de 2-mercaptoéthanol, et 25 unités / ml de pénicilline / streptomycine ), et centrifuger à 100 g pendant 5 min.
- Aspirer le surnageant et remettre en suspension les cellules dans 30 ml de milieu de cellules ES, suivie par la distribution en deux flacons T25 (5 ml chacune) et deux plaques de 100 mm (10 ml chaque). Incuber à 37 ° C, 5% CO 2 pendant 24 heures.
- Culture et élargir cellules C9 ES
- Décongeler une fiole de cellules ES C9 (2,5 x 10 6 cellules dans 0,5 ml) dans un bain d'eau à 37 ° et transférer lecellules dans 5 ml de milieu de cellules ES. Centrifuger à 100 g pendant 5 min.
- Aspirer le surnageant et remettre en suspension les cellules ES dans 5 ml de milieu de cellules ES, puis en étalant dans le flacon T25 d'alimentation. Incuber à 37 ° C, 5% CO 2. Remplacer milieu cellulaire ES quotidienne.
- Après 2 jours de culture, les cellules ES devraient devenir ~ 80% de confluence, et sont prêtes à être repiquées. Laver les cellules avec 5 ml de PBS (sans Ca2 + et Mg2 +), puis ajouter 2,5 ml préchauffé 0,25% de trypsine-EDTA. Incuber à 37 ° C pendant 10 min. Ajouter 2,5 ml de milieu de cellules ES pour arrêter la trypsine; pipette de haut en bas 15 fois pour briser les amas de cellules. Centrifuger à 100 g pendant 5 min.
- Aspirer les cellules et remettre en suspension dans le surnageant de 4 ml de milieu de cellules ES. Transférer 1 ml de cellules par plaque de 100 mm contenant 9 ml ES milieu cellulaire; préparer deux plaques de 100 mm. Ceci est un 1: passage 8. Nourrir les cellules quotidien avec 10 ml ES milieu cellulaire.
- Préparer plaques à 96 puits d'alimentation pour la bibliothèque
- Comme décrit dans 1.1.1, décongeler trois flacons de la mitomycine-C (PMEF inactivés contenant chacun 5 x 10 6 cellules) et remettre les cellules à partir de chaque flacon avec 33,3 ml ES milieu cellulaire. Moissonneuse cellules pour obtenir une suspension de cellules de 100 ml.
- Planche 100 cellules par puits sur dix plaques à 96 puits. Ces plaques d'alimentation 96 puits devraient être préparés au moins un jour avant la transfection de cellules ES C9 avec PB transposon.
- Électroporation des cellules ES C9 avec le vecteur PB-UPA
- Trypsiniser et compter les cellules C9 ES élargis en utilisant un compteur de cellules automatisé. Laver chaque plaque 100 mm de cellules ES C9 avec 10 ml de PBS. Ajouter 8 ml préchauffé 0,25% de trypsine-EDTA et incuber à 37 ° C pendant 10 min.
- Arrêtez la trypsine en ajoutant 2 ml ES milieu cellulaire. Transférer la suspension de cellules dans un tube de 15 ml et pipette de haut en bas 15 fois pour briser amas de cellules. Centrifuger immédiatement à 100 g pendant 5 min, puis en aspirant le surnageant.
- Remettre en suspension cellulaires dans 10 ml de milieu de cellules ES et de compter les cellules ES à l'aide d'un hémocytomètre. Préparer six tubes de 15 ml de suspension cellulaire contenant chacun 5 x 10 6 cellules ES. Centrifuger à 100 g pendant 5 min. Aspirer le surnageant et remettre en suspension chaque culot cellulaire dans 0,8 ml de PBS.
- Préparer six cuvettes d'électroporation (gap de 4 mm) à laquelle les cellules sont transférées 0,8 ml. Pour chaque cuvette, ajouter 1 pg PB-UPA et 20 pg MPB transposase (mPBase); mélanger doucement par pipetage de haut en bas. Mettre les cuvettes sur la glace. Effectuer électroporation avec un électroporateur en utilisant le réglage exponentielle à 250 V, 500 pF, et l'infini ohms.
NOTE: En général, la constante de temps est d'environ 10 à 12 msec pour une électroporation réussie, qui donnent environ 1500 à 2000 transformants résistant à G418 par 5 x 10 6 cellules. - Récupérer et mettre en commun les cellules transfectées dans un ballon de 98 ml contenant T75 milieu de cellules ES. Mélanger les cellules par pipetage doux et ensuite transférer les cellules à un réservoir (50 ml). Noustion d'une pipette 12 canaux, transférer 100 pi de cellules par puits pour les plaques d'alimentation préalablement préparé 96 puits (10 au total). Incuber à 37 ° C, 5% CO 2.
- Après 24 h, tous les jours alimenter les cellules avec des produits frais ES milieu cellulaire contenant G418 (150 ug / ml) pour sélectionner des clones de gènes piégés.
REMARQUE: Typiquement chaque puits contiendra une dizaine de colonies résistantes à G418. Lorsque ces colonies se développent de taille suffisante (typiquement de 5 à 7 jours), les plaques à 96 puits (la bibliothèque) seront répliqués dans 2 ensembles de stockage et de sélection, respectivement. Chaque plaque dans la bibliothèque est désigné comme «sous-bibliothèque», dans ce cas, il ya 10 sous-bibliothèques.
2. Bibliothèque Replication
- Répliquer les plaques Master
- 24-48 h avant les colonies résistantes au G418 atteignant 50-60% de confluence, dégeler trois autres flacons de mitomycine C-inactivé PMEF sur dix plaques à 96 ainsi que les mangeoires (se référer à la section 1.3). Gélatineize dix plaques à 96 puits par incubation de 0,1% de gélatine (100 ul / puits) à 37 ° C pendant au moins 15 - 30 min; O / N incubation est acceptable.
- Préparer des suspensions de cellules simples dans des plaques à 96 puits par traitement à la trypsine. Aspirer moyen de plaques à 96 puits et laver une fois avec un volume égal de PBS (sans Ca 2+, Mg 2+). Ajouter 50 ul d'préchauffé 0,25% de trypsine-EDTA, incuber à 37 ° C dans 5% de CO2 pendant 10 min. Arrêtez la trypsine avec 50 pi de milieu de cellules ES. Briser les amas de cellules par pipetage de haut en bas 15 fois avec la pipette multicanaux.
- Transfert 50 pi de suspension de cellules trypsinisées à la ligne correspondante de la plaque d'alimentation (Master) contenant déjà 150 milieu cellulaire ul ES et les 50 pi restants à la ligne correspondante de la plaque gélatinisée (réplique) contenant déjà 150 pi de milieu de cellules ES. Traiter chaque rangée de cellules sur une plaque de maître et de réplication. Incuber les plaques (contenant maintenant 200 pi culture le volume) à 37 ° C, 5% CO 2 H / N.
NOTE: Il ya maintenant vingt plaques à 96 puits. - Le lendemain, nourrir les cellules avec 100 pi du milieu frais de cellules ES contenant 150 pg / ml de G418. Remplacez le support tous les jours jusqu'à les puits sont à 80-90% de confluence.
- Congeler les plaques Master
- Lorsque les plaques de base sont environ 80% de confluence, trypsiniser les cellules comme décrit (voir section 2.1.2).
- Retirer les plaques et arrêter la réaction avec de la trypsine 50 pi de 2 x milieu de congélation (60% de milieu de cellules ES, 20% de FBS, 20% de DMSO). Introduire à la pipette de haut en bas 10 fois avec une pipette multicanaux pour assurer un bon mélange du milieu de congélation.
- Mettre les plaques à 96 puits dans une boîte en polystyrène étanche et le placer dans le congélateur à -80 ° C pour un maximum de quatre mois de stockage. Quatre mois se laisser suffisamment de temps pour terminer la sélection de stress et d'identifier les puits de la plaque de Maître contiennent les clones d'intérêt.
REMARQUE: stockage à -80 & #176; C pendant plus de 4 mois se traduira par diminution de la viabilité cellulaire. On peut utiliser des tubes en cryo-format à 96 puits qui peuvent être stockées dans la phase vapeur de l'azote liquide pendant une longue période de stockage.
- Préparer plaques d'ADN et des piscines sous-bibliothèque à partir de plaques de réplique
- 24 à 48 heures avant de reproduire à la suite de plaques d'ADN et les piscines sous-bibliothèque, préparer dix 100 mm des plaques avec de la mitomycine C inactivé PMEF pour le pool de sous-bibliothèque, et dix gélatinisé plaques à 96 puits de la plaque d'ADN se réplique. Reportez-vous à la section 1.3 pour la procédure générale.
- Trypsiniser les cellules dans les 96 puits répliques de plaques (voir la section 2.1.2). Au cours de l'étape de traitement à la trypsine de 10 minutes, aspirer gélatine à partir des plaques d'ADN et le remplacer avec 150 pi de milieu de cellules ES contenant G418 (150 ug / ml). En outre, de préparer un réservoir contenant 5 ml de milieu de cellules ES pour la mise en commun des «sous-bibliothèques» dans la plaque d'alimentation de 100 mm.
- Après traitement à la trypsine, arrêtez d'essayerPSIN avec 50 ul de cellules ES moyen d'une ligne à la fois à l'aide d'une pipette 12 canaux. Pipette de haut en bas 10 fois pour se briser amas de cellules.
- Transfert 50 pi de la suspension de cellules à la ligne correspondante de la plaque d'ADN et transférer les 50 pi restants au réservoir contenant 5 ml ES milieu cellulaire. Après avoir terminé une plaque complète de 96 puits, aspirer le moyen d'une plaque d'alimentation de 100 mm à laquelle les cellules regroupées sont transférées. Ces cellules mutagénisées communs sont désignés comme un «sous-bibliothèque».
- Répétez l'opération pour les neuf plaques à 96 puits restants. Alimenter les plaques d'ADN de 96 puits et les plaques 100 mm réunies «sous-bibliothèque« tous les jours avec un milieu de cellules ES contenant G418 (150 ug / ml).
NOTE: Il ya maintenant dix plaques à 96 puits «ADN», et dix plaques de 100 mm «sous-bibliothèque».
- Congeler les plaques d'ADN. Lorsque les cellules de la plaque d'ADN sont de 80 à 90% de confluence, aspirer le milieu, laver les cellules deux fois avec 100ul de PBS et conserver à -20 ° C pour l'isolement de l'ADN génomique tard 6. Ces plaques d'ADN seront criblées par PCR pour identifier les puits correspondant de la plaque "Master" contient le clone d'intérêt.
3. mutants homozygotes induite par la doxycycline
- Geler les piscines sous-bibliothèque et les cellules de passage pour le traitement de la doxycycline
- 24 h avant les plaques de 100 mm «sous-bibliothèque» ayant colonies assez grandes pour obtenir 70-80% de confluence, préparer un ensemble de dix gélatinise plaques de 100 mm pour le traitement de la doxycycline.
- Préparer des suspensions de cellules uniques de chaque sous-bibliothèque (voir les sections 1.4.1 et 1.4.2 pour plus de détails). Transfert 5 x 10 6 cellules à la plaque 100 mm gélatinisé avec de la doxycycline (1 ug / ml); incuber à 37 ° C, 5% CO 2. Geler les non-doxycycline restants traités piscines sous-bibliothèque à 5 x 10 6 cellules par flacon.
- Passage des cellules en présencede la doxycycline pour assommer Blm
- Passage sous-bibliothèques à 1: 8 tous les 2-3 jours en présence de doxycycline (1 pg / ml) pendant deux semaines, période pendant laquelle dox-induites knock-out de BLM favoriser la perte d'hétérozygotie dans la cellule population, ce qui permet la production de mutants homozygotes.
4. Sélection stress et isolement de clones résistants
- Les cellules de la doxycycline sujet traité pour la sélection de stress
- 24 heures avant passage de cellules traitées à la doxycycline pour la sélection de stress, préparer dix gélatinise plaques 150 mm, place dans un 37 ° C, 5% de CO 2 incubateur humidifié.
- Préparer une suspension de cellule unique de chaque sous-bibliothèque par trypsinisation (se référer à la section 1.4.1 et 1.4.2 pour plus de détails). Ensemencer 6,6 x 10 6 cellules sur chaque plaque de 150 mm en 30 ml de volume total de culture, de distribuer les cellules uniformément. Incuber les cellules avec facteur de stress (10 uM paraquat ou omission de β-mercaptoéthanol,dans un milieu de cellules ES contenant 7,5% de FBS inactivé à la chaleur) pendant 7 jours, après quoi remplacer par un milieu de cellules ES normales pour permettre aux cellules dans survivantes de croître colonies pour le prélèvement.
- Facultatif: Geler la gauche par rapport aux cellules de doxycycline-traitée à 5,0 x 10 6 cellules par flacon.
- Prélever des colonies résistantes au stress
- Après le rétablissement des médias normale pendant une semaine, observer combien le stress colonies résistantes sont présents. Selon le nombre de colonies à cueillir, préparer suffisamment de puits avec la mitomycine C-inactivé PMEF sur des plaques à 96 puits un jour à l'avance en conséquence (voir la section 1.1 pour plus de détails).
- Le jour de la cueillette, aspirer et de le remplacer dans la plaque de 96 puits d'alimentation avec 100 pi de milieu frais de cellules ES moyen, placer de nouveau dans 37 ° C, 5% de CO 2 incubateur humidifié. A ce moment, préparer une plaque à 96 puits à fond en U à 50 ul de 0,25% de trypsine-EDTA par puits. Aspirer le milieu de la plaque de sélection de stress de 150 mm et d'ajouter ele volume de PBS qual.
- Réglez la micropipette à 2 pi, et 'ramasser' colonies survivantes dans la trypsine à fond en U contenant puits, ramasser une colonie par puits. Il est acceptable de laisser la plaque reposer à température ambiante jusqu'à ce que le nombre souhaité de colonies a été ramassé. Ensuite, incuber la plaque de fond en U à 37 ° C, 5% CO 2 pendant 10 minutes.
Remarque: En général, nous prenons 1 heure pour ramasser 96 colonies. - Arrêtez réaction trypsine une ligne à la fois avec 50 pi de milieu de cellules ES à l'aide d'une pipette multicanaux. Pipette de haut en bas 15 fois pour obtenir une suspension de cellules individuelles et transférer 100 pi de cellules à la ligne correspondante de la plaque d'alimentation contenant déjà 100 pi de milieu de cellules ES. Remplissez le transfert de la totalité de la plaque. Cellules de culture O / N dans 200 pi. Le lendemain matin, moyen d'aspiration et le remplacer par 100 pi de milieu de cellules ES.
- Répliquer les plaques à 96 puits
- Lorsque les puits des colonies cueillies sont 80 à 90% confluent, reproduire en deux lames d'adaptation, l'une pour l'isolement d'ADN et l'autre pour les cellules de secours. Ces plaques sont gélatinisé et ne contiennent pas de mitomycine C inactivé PMEF.
- Aspirer le milieu et laver avec 100 pi de PBS. Ajouter 50 pi de 0,25% de trypsine-EDTA à chaque puits, incuber à 37 ° C pendant 10 min. Arrêtez la trypsine avec 150 pi de milieu de cellules ES, mélanger par pipetage, et de transférer 100 pi de suspension cellulaire à la ligne correspondante de deux plaques à 96 puits. Changer le milieu de tous les jours.
- Lorsque les plaques sont 80-90% de confluence, geler un ensemble de plaques comme un «back-up» (se reporter à la section 2.2 pour plus de détails). L'autre série de plaques sont en outre étendu comme indiqué ci-dessous.
- Accroître les cellules d'isolement de l'ADN génomique
- Préparer une suspension cellulaire unique dans la plaque d'ADN de 96 puits (voir 2.1.2 pour plus de détails).
- Transfert suspension de cellules de chaque puits de la plaque de 96 puits pour le puits d'une plaque de 24 puits (4 au total) en utilisant un MICRopipette. Assurez-vous qu'il n'y a pas de contamination croisée de toutes les cellules, que les colonies «d'élite» sont «clonale 'origine. Cellules de culture en ml ES milieu cellulaire 0,5, l'évolution moyenne quotidienne jusqu'à ce puits sont 80-90% de confluence.
NOTE: La plaque de 24 puits permettra un rendement suffisant de l'ADN génomique pour l'analyse PCR.
5. Identification des sites Insertion de Pb et de gènes Trapped
- Isolement de l'ADN génomique à partir de plaque de 24 puits: Lyse des cellules dans la plaque de 24 puits et isoler l'ADN génomique exempt d'ARN.
- Effectuer Splinkerette PCR (SpPCR) pour générer des fragments de séquences flanquant le site d'insertion du transposon comme décrit PB 7,8.
REMARQUE: SpPCR est un processus de cinq jours, si les mains sur le temps chaque jour est minime.
6. Analyse génétique des clones mutants
- Localiser maître bien et purifier clone d'intérêt.
- Lorsque le site d'intégration est PBmappé, concevoir une amorce sens en amont du site d'intégration à utiliser avec une amorce inverse PB (par exemple, PB3'-1) 8 et l'écran de la plaque d'ADN correspondant à la même «sous-bibliothèque» à partir duquel le clone résistant au stress a été choisi. Attendez-vous à trouver un seul puits positif sur l'ensemble de plaque de l'ADN de 96 puits. Décongeler ce bien exact de la plaque "Master" à une plaque de 24 puits sur les départs et cultiver des cellules jusqu'à 80-90% de confluence.
NOTE: A ce stade, ce puits contient un mélange de clones de gènes piégés. - Lorsque la plaque de 24 puits est de 80-90% de confluence, Trypsiniser cellules et de la plaque de 1000 cellules ES dans une plaque d'alimentation de 100 mm (assurer la suspension cellulaire unique). Passage des cellules restantes à un flacon T25 sur les départs d'expansion. Geler les cellules du flacon T25 lorsque les colonies sont 80-90% de confluence. Congeler 4 flacons par flacon T25 comme back-up.
- Permettre aux colonies de se développer pendant 7 jours et ensuite choisir 96 colonies dans un 96 puits feplaque eder. Reportez-vous à la section 4.2 pour la cueillette procédure. Répliquer la plaque lorsqu'il est prêt dans une plaque «ADN» et figer les autres comme «2 ème -master 'plaque (voir la section 2.2 pour la congélation).
- Autoriser les colonies pour atteindre 80-90% de confluence. Cribler la plaque d'ADN pour trouver des puits contenant le clone purifié d'intérêt. Développer les cellules de la plaque de 96 puits 2 ème -master à 24 puits et par la suite à un flacon T25 (tout sur les départs). Congeler 4 flacons par flacon T25.
NOTE: A ce stade, la lignée cellulaire gène piégé est clonale, n'a pas été exposé à l'agent stressant, et est adapté pour la production de la souris.
- Lorsque le site d'intégration est PBmappé, concevoir une amorce sens en amont du site d'intégration à utiliser avec une amorce inverse PB (par exemple, PB3'-1) 8 et l'écran de la plaque d'ADN correspondant à la même «sous-bibliothèque» à partir duquel le clone résistant au stress a été choisi. Attendez-vous à trouver un seul puits positif sur l'ensemble de plaque de l'ADN de 96 puits. Décongeler ce bien exact de la plaque "Master" à une plaque de 24 puits sur les départs et cultiver des cellules jusqu'à 80-90% de confluence.
- Déterminer le nombre d'insertion PB: Décongelez un flacon de la lignée de cellules résistantes au stress et à étendre les cellules dans un flacon T25 gélatinisé. Isoler l'ADN génomique et de vérifier le nombre de copies du transposon PB. Ceci peut être réalisé par qPCR ciblant le gène de la néomycine.
- Mesurer le niveau d'expression des gènes: Après confirmant il y a un événement d'intégration de PB unique dans la lignée de cellules d'intérêt, mesurer le niveau d'expression du gène piégé par RT-PCR quantitative en utilisant des sondes Taqman spécifiques pour chaque gène.
- La séquence de l'ARNm mutant: générer un amplicon d'ADNc par PCR en utilisant une amorce en amont recuit à un exon et une hybridation des amorces en aval de la séquence accepteur d'épissage de la cassette de piégeage de gènes. La séquence de fragment d'ADNc pour confirmer que l'épissage se produit attendu dans le gène piégé.
- Remobiliser le transposon piggyBac pour tester relation de causalité entre le gène piégé et la résistance au stress.
- 24-48 h avant décongélation cellules pour PB remobilisation, dégel et la plaque DR4 mangeoires sur un flacon T25 et deux plaques de 100 mm. Au moins un jour après que les dispositifs d'alimentation DR4 sont étalées sur le flacon T25, décongeler un flacon de la lignée de cellules contenant l'insertion PB et plaque les cellules dans le flacon T25 contenant mangeoires DR4. Cultiver les cellules dans un milieu de cellules ESpendant 48 heures, en changeant moyenne quotidienne.
- Préparer une suspension cellulaire unique de cellules ES. Compter les cellules avec un compteur de cellules automatisé, et transférer 5,0 x 10 6 cellules ES dans un tube de 15 ml. Centrifuger à 100 xg, et remettre en suspension les cellules dans 800 pi de PBS. Transférer la suspension à un 4 mm cuvette.
- Ajouter 20 pg mPBase Puro à la cuvette, mélanger par pipetage, et les cellules électroporer (se reporter à la section 1.4 pour plus de détails). Distribuer les cellules transfectées sur deux plaques DR4 chargeur de 100 mm. Cellules de culture O / N dans un milieu de cellules ES.
- Le lendemain matin, à nourrir les cellules avec un milieu avec des cellules ES de puromycine et de la culture / ml 1 ug pendant 48 heures. Retirer sélection à la puromycine et continuer à cultiver les cellules pendant 5 jours. Lorsque les colonies sont assez grands pour la cueillette, choisir dans une plaque d'alimentation 96 puits (voir rubrique 4.2).
- Lorsque les cellules d'élite ont atteint 80-90% de confluence, divisé 1/6 ème de la plaque dans une plaque d'ADN et 1/6 ème de la plmangé dans une plaque de G418, laissant les 2/3 des cellules dans la plaque originale à congeler (voir la section 2.2 pour la congélation).
- Culture de la plaque de l'ADN dans un milieu de cellules ES pour l'isolement de l'ADN par la suite, et la plaque de G418 à 150 ug / ml de G418 à tester pour la perte de résistance à la néomycine. Les puits sans cellules viables après 72-96 heures dans la culture contenant G418 indiquent remobilisation réussie du PB transposon de piège à gènes.
- Les puits correspondant de la plaque «ADN» seront criblées par PCR pour confirmer le transposon de PB a été supprimée. Lorsque la plaque d'ADN est 90% de confluence, isoler l'ADN génomique dans un format de plaque à 96 puits pour le criblage par PCR 4 pour la restauration de la séquence de type sauvage et l'absence de néo-IRES en tant que moyen pour confirmer réversion.
- Décongeler les puits correspondants de la plaque de Maître à la plaque de 24 puits et d'étendre à un flacon T25 (tous avec les mangeoires). Congeler quatre flacons par flacon T25 comme back-up.
7. Génération de mutants de souris
- Récupérer les cellules ES résistantes à partir des plaques de base et de les utiliser pour l'injection d'embryons pour générer chimère. Environ 15-20 cellules ES sont injectés dans un individu C57BL / 6 blastocyste. Transfert de 15 blastocystes par la souris à l'utérus d'une femelle pseudo (souche ICR). Compagnon de la chimère mâle (indiqué par la couleur de cellules spécifiques agouti ES) récupéré de la litière avec plusieurs jeunes vierges C57BL / 6J femelles pour tester pour la transmission de la lignée germinale, et de construire une colonie de souris mutantes.
- Isoler et des fibroblastes de peau de la queue de test des souris mutantes de F1 et de type sauvage de la même portée pour les espèces réactives de l'oxygène (ROS) niveau et la résistance au paraquat 9.
Résultats
Dans une expérience typique de mutagenèse, la transfection se compose d'un total de 3 x 10 7 cellules ES avec PB-UPA et MPB transposase. Le nombre de cellules ES gènes piégés engendrés dans deux expériences indépendantes sont résumés dans le tableau 1. L'efficacité de piégeage de gènes est d'environ 0,04%. Les bibliothèques combinées de piégeage de gènes mutants contiennent 22.400 indépendants, soit environ une couverture complète de génome de la souris (23.000 gènes codant). La couverture excessive pourrait être atteint en générant plus de mutants par mutagénèse répétée et / ou mise à l'échelle le processus.
Parce que les bibliothèques de piégeage de gènes contiennent des mutations aléatoires, il est impossible de prédire le nombre de mutants possédant un phénotype de résistance au stress. Deux sélections indépendantes pour la résistance de PQ ont été effectuées; le dépistage combiné de 22,440 mutations de piégeage de gènes indépendants non couvert 17 mutations qui confèrent une résistance cellulaire à PQ (0,08%) (tableau 1).
Nous avons purifié 3 clones mutants, l'Pigl, Tiam1 et Rffl, à partir des plaques de base pour une analyse plus approfondie et la production de la souris. Les cellules avec hétérozygote mutation Rffl ont été induits avec succès pour produire une descendance homozygote en assommant Blm. Cependant, nous avons échoué à produire des mutants homozygotes des mutants Pigl et Tiam1, vraisemblablement due à une cellule de létalité homozygotie lié. La résistance au stress de ces clones purifiés a été confirmée en utilisant 2 types de facteurs de stress: omission de 2-mercaptoéthanol (2-ME) et PQ (Figure 2). Après élimination du PB à partir de ces clones, le phénotype de résistance au stress associé a également été perdu (figure 2), ce qui confirme que les mutations de piégeage de gènes sont le facteur causal. Caractérisation des clones récupérés à partir des plaques de base est cruciale car elle exclurait lésion stochastique lors de la sélection de stress comme la cause du phénotype observé.
Sur 3Les cellules ES mutantes étant introduites dans des blastocystes pour la production de souris (tableau 2), (2 lignes Pigl et Tiam1) montrent transmission de la lignée germinale. L'injection Rffl produit seulement une chimère femelle qui était pas un nombre optimal de chimère pour commencer. Ainsi, l'échec de la transmission germinale de Rffl est probablement due à un chimère incompétent. Nous avons testé le phénotype cellulaire de fibroblastes cutanés isolés à partir des souris mutantes Pigl et ont montré qu'ils ont maintenu le niveau réduit d'espèces oxygénées réactives endogènes (ROS), ainsi que la résistance au stress de PQ (figure 3).
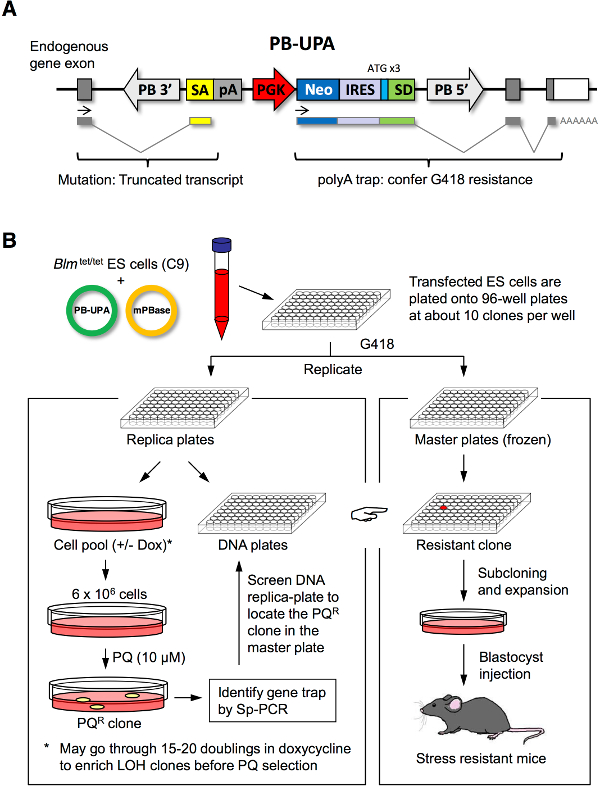
Figure 1. ES de mutagenèse cellulaire et la sélection de stress. Vecteur (A) PB-UPA. Un vecteur polyA (UPA) du piège impartiale comprenant un accepteur d'épissage (SA), signal de poly-cyclase de l'hormone de croissance bovine (PA), phosphoglycerate kinase (PGK), la néomycine phosphotransferase (néo), site d'entrée interne du ribosome (IRES), et un donneur d'épissage (SD) avec artificiel ATG (en 3 cadres de lecture différents) a été clone dans le transposon piggyBac, flanquée par la région 3 ' et 5 'la longue répétition terminale (LTR). (B) la mutagenèse aléatoire des cellules ES et la sélection de clones résistants au stress. Les cellules ES ont été cotransfectées par électroporation avec PB-UPA et mPBase suivie par étalement sur des plaques à 96 puits. Gènes clones pris au piège ont été sélectionnés par la résistance à G418; une fois confluentes, ils ont été divisés en 2 jeux de réplicas. Un jeu de réplicas a été divisée en deux demi pour l'isolement de l'ADN et la sélection de stress par le paraquat (PQ); l'autre jeu de réplicas (maître) a été gelé vers le bas. Les colonies de cellules ES survivants récupérés à partir du traitement du stress ont été analysés pour l'insertion moléculairement par PB Sp-PCR. Amorces ont ensuite été conçus pour dépister la plaque d'ADN réplique à partir de laquelle le puits contenant l'Sibling PQ R clone pourrait être situé sur la plaque de maître. Ces cellules sont pour le dosage de la résistance au stress sur les différents facteurs de stress et pour la production de la souris. Modifié à partir de Chick et al. 7. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 2. Résistance du stress dans les cellules ES mutantes. (A) Résistance à la 2-mercaptoéthanol (2-ME) retrait. (B) La résistance au paraquat (PQ). Sont représentés les cellules parentales de type sauvage ES (C9: jaune), un contrôle clone de cellules ES résistant au stress, (4C11: rouge), récupérées à partir d'une étude précédente, 5 et trois clones de piégeage de gènes (gris). Les cellules ont été soumises à un stress traitement pendant deux jours, après quoi le nombre de cellules viables a été compté. < em> hétérozygotes Pigl, Tiam1 et Rffl. (PB / +: gris foncé) présentent une résistance à la fois de ces facteurs de stress Rffl homozygotes (PB / PB: noir) présentent une résistance au stress plus forte par rapport aux hétérozygotes. Résistance à la fois les facteurs de stress a été perdu lorsque les insertions de PB ont été enlevés (+ / +: gris clair). Les barres d'erreur représentent SD de la moyenne (n = 4). * P <0,001, P = 0,01 # entre les clones de piégeage de gènes et la C9 parentale de type sauvage, évalués par le t-Test de Student. (C) de cellules ES formation de colonie sous PQ (10 M) traitement. ES clones résistant au stress ont pu former des colonies dans la culture sous traitement PQ alors que le nombre et la taille des colonies à partir du clone de type sauvage ont été considérablement réduits. Modifié à partir de Chick et al. 7. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
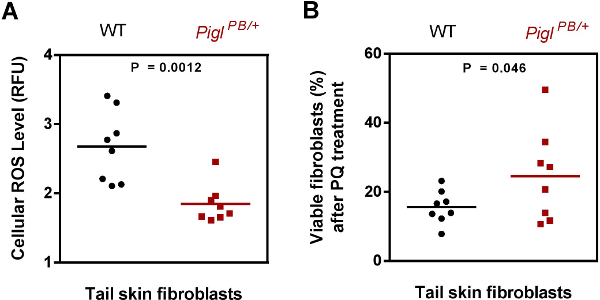
Figure 3. Caractérisation de Pigl PB / + fibroblastes. (A) Les espèces réactives de l'oxygène (ROS) niveau. Fibroblastes de peau de la queue isolées de type sauvage (WT) et Pigl PB / + souris ont été colorées avec CM-DCFCA et la fluorescence a été normalisée contre Hoechst. Le contenu ROS a été exprimée en unité de fluorescence relative (RFU). La valeur de P a été évaluée par le t -test de deux étudiants de Virginie (n = 8). (B) PQ résistance. Des fibroblastes de peau de queue de type sauvage (WT) et / Pigl PB + souris ont été exposées à 4 mM de PQ pendant 6 heures, après quoi la viabilité des cellules a été mesurée par des dosages MTT. Le pourcentage de cellules vivantes après traitement PQ été calculée par le rapport de l'absorbance obtenue à partir de la PQ traité CELLS et que des cellules non traités. La valeur de P a été évaluée par le t -test d'un élève de Virginie (n = 8). Modifié à partir de Chick et al. 7. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| Bibliothèque | Sous-bibliothèques | Souche de cellules ES | N ° de gène piégé clone | Nombre de clones PQ R |
| 1 | C9PA01 - C9PA10 | C9 (BLM tet / tet) | 9000 | 7 |
| 2 | C9PA11 - C9PA20 | C9 (BLM tet / tet) | 13440 | 10 |
| Le total | 22440 | 17 |
Tableau 1. PiggyBac construction de piégeage de gènes bibliothèque et la récupération des clones résistants PQ. Modifié à partir de Chick et al. 7.
| Clone de cellules ES injecté | Nombre de chimère récupéré | La transmission germinale * |
| Pigl PB / Pigl + | 4 | Oui |
| Tiam1 PB / Tiam1 + | 2 | Oui |
| Rffl PB / Rffl + | 1 | Non |
Tableau 2. Génération de souris. Modifié à partir de Chick et al. 7.
* Transmission germinale a été confirmée par l'héritage de l'allèle de piégeage de gènes détectés par génotypage par PCR.
Discussion
Forward genetic analysis allows for an unbiased interrogation of the genome for genes responsible for a specific phenotype. This method is very powerful to uncover novel gene functions. It has been widely used in lower organisms but not in mammal, such as the mouse, mainly due to the extremely high cost associated with the infrastructure and logistics that would entail. Here, we moved the genetic screening process to the ES cell culture platform, greatly increasing the efficiency and throughput in generating mutants and identifying mutations.
To effectively select for resistant mutants, a lethal dose of stressor and period of treatment needs to be established such that all of the wild-type cells would be killed. The higher the stringency of the selection, the fewer occurrence of false positive will result. Fibroblast feeders have a higher tolerance to oxidative stress than ES cells; the presence of feeders would rescue the nearby ES cells which would otherwise be killed by the stressor. Thus, omission of feeders during stress selection was very critical for an effective killing. In skin fibroblasts, serum deprivation was necessary to reveal the stress resistance phenotype in long lived mice 10. However, this application was not feasible in ES cells because they cannot survive without serum. As an alternative, we discovered that the use of heat inactivated serum at reduced level (7.5%) achieved a similar result.
In this report, we employed poly-A trap rather than a promoter trap because promoter trap will only trap actively transcribed genes, which is estimated to be 60% of the genome in ES cells, under normal conditions 11. In contrast, a poly-A trap would trap genes regardless of their transcriptional activities, in theory giving us the potential to screen for mutations in all genes in the genome. A major drawback of the conventional poly-A trap is the strong bias towards trapping the last exon, appeared to be mediated by an endogenous nonsense-mediated decay (NMD) mechanism 12. By incorporating the unbiased poly-A trap (UPA) strategy in the PB-UPA transposon to suppresses NMD 13, we were able to eliminate biased trapping.
One distinguishing advantage of using ES cells is that these pluripotent cells make direct derivation of mice possible, speeding up the transition to in vivo studies. For this to happen, the replica plating strategy is critical, preserving a subset of clones sheltered from manipulations that can diminish pluripotency, such as knocking out of Blm and exposure to stressors. We have shown that mutant ES cells recovered from these screens were capable of participating in forming the entire mouse and transmitting the mutations to the germline. Most importantly, consistent with previous studies, the stress resistance phenotype was also transmitted and maintained in the adult mouse.
We and others showed that a variant phenotype observed in ES cells resulting from environmental induced lesion or deliberated engineered mutations was able to transmit to the differentiated progeny cells 4,5,9,14, and probably the whole mouse. This protocol describes the utilization of ES cells as surrogate cells to screen for stress resistance mutations in the mouse. In addition to screening for stress resistance genes, this method may have a broader implication to screen ES cells, identify targets, and to develop a variety of mouse models with a phenotype of interest.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
We would like to thank the Wellcome Trust Sanger Institute for the gifts of piggyBac transposon and piggyBac transposase. This work was supported by the Butcher grant of Colorado and the NIH R01 AG041801 (W.S.C).
matériels
| Name | Company | Catalog Number | Comments |
| Vector | |||
| PB-UPA | |||
| mPBase | |||
| mPBasePuro | |||
| Tissue Culture | |||
| 500-ml Stericup filters | EMD Millipore | SCGPU05RE | |
| 250-ml Stericup filters | EMD Millipore | SCGPU02RE | |
| 50-ml Steriflip-GV filters | EMD Millipore | SE1M179M6 | |
| KO DMEM | Life Technologies | 10829-018 | |
| DMEM | Sigma-Aldrich | D6429 | |
| FBS | Tissue Culture Biologicals | 104 | |
| Heat Inactivated FBS | Sigma-Aldrich | F4135-500 | |
| LIF | EMD Millipore | ESG1107 | |
| Non-essential Amino Acids | Life Technologies | 11140-050 | |
| GlutaMAX | Life Technologies | 35050-061 | |
| Pen/Strep | Life Technologies | 15140148 | |
| β-Mercaptoethanol | Life Technologies | 21985-023 | |
| Methyl Viologen dichloride (Paraquat) | Sigma-Aldrich | 856177 | |
| Dimethyl Sulphoxide Hybri-MAX | Sigma-Aldrich | D2650 | |
| EmbryoMAX 0.1% gelatin | EMD Millipore | ES006B | |
| DPBS/Modified | HyClone | SH30028.02 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| T25 Flask | Corning | 353108 | |
| T75 flask | Corning | 353135 | |
| 100-mm plate | Corning | 353003 | |
| 150-mm plate | Corning | 430599 | |
| 96-well plate | Corning | 3585 | |
| 96-well U-bottom plate | Corning | 3799 | |
| 24-well plate | Corning | 3526 | |
| 50-ml reservoir | Corning | 4870 | |
| 15-ml tubes | VWR International, LLC | 82050-276 | |
| Primary Mouse Embryonic Fibroblasts | EMD Millipore | PMEF-NL | |
| DR4 Mouse Embryonic Fibroblasts | Applied StemCell | ASF-1001 | |
| Mitomycin C | Fisher BioReagents | BP25312 | |
| Geneticin (G418) | Life Technologies | 11811-023 | |
| Doxycycline | Fisher BioReagents | BP26531 | |
| Cryotubes | Thermo Scientific | 377267 | |
| Centrifuge | Eppendorf | Centrifuge 5702 | |
| TC10 cell counter | Bio-Rad | ||
| Counting Slides (for TC10) | Bio-Rad | 1450011 | |
| Electroporation | |||
| Gene Pulser Xcell | Bio-Rad | 1652611 | |
| Gene Pulser Cuvettes (4 mm gap) | Bio-Rad | 1652088 | |
| Molecular Biology | |||
| Thermal Cycler | Eppendorf | Mastercylcer ep Gradient S | |
| Puregene Core kit B | Qiagen | 158745 | |
| Topo-TA Cloning kit | Life Technologies | 450030 | |
| High Capacity cDNA synthesis kit | Applied Biosystems | 4368814 | |
| NaCl | Fisher BioReagents | BP358-212 | |
| 100% ethanol | Decon Laboratories, Inc. | 2716 | |
| Double Processed Tissue Culture Water | Sigma-Aldrich | W3500 | |
| Sau3A1 | New England BioLabs | R0169L | |
| T4 DNA Ligase | New England BioLabs | M0202T | |
| EcoRV | New England BioLabs | R3195S | |
| 96-well Lysis Buffer (Ramires-Solis et al. 1992) | |||
| Trizma Base | Sigma-Aldrich | T1503 | |
| Hydrochloric Acid | Fisher BioReagents | A144-212 | |
| EDTA | Sigma-Aldrich | E5134 | |
| N-Lauroylsarcosine sodium salt | Sigma-Aldrich | L5777 | |
| Proteinase-K | Fisher BioReagents | BP1700 | |
| Electrophoresis | |||
| Mini-Sub Cell GT | Bio-Rad | 170-4469EDU | |
| LE Agarose | GeneMate | E3120500 | |
| Ethidium Bromide | Fisher BioReagents | BP1302 | |
| 100 BP DNA Ladder | New England BioLabs | N3231S | |
| 1 Kb DNA Ladder | New England BioLabs | N3232S | |
| 2-log DNA Ladder | New England BioLabs | N3200L | |
Références
- Johnson, T. E., et al. Longevity genes in the nematode Caenorhabditis elegans also mediate increased resistance to stress and prevent disease. J Inherit Metab Dis. 25, 197-206 (2002).
- Murakami, S., Salmon, A., Miller, R. A. Multiplex stress resistance in cells from long-lived dwarf mice. Faseb J. 17, 1565-1566 (2003).
- Perez, V. I., et al. The overexpression of major antioxidant enzymes does not extend the lifespan of mice. Aging Cell. 8, 73-75 (2009).
- Martin, G. M. Genetic engineering of mice to test the oxidative damage theory of aging. Ann NY Acad Sci. 1055, 26-34 (2005).
- Chick, W. S., Drechsel, D. A., Hammond, W., Patel, M., Johnson, T. E. Transmission of mutant phenotypes from ES cells to adult mice. Mamm Genome. 20, 734-740 (2009).
- Ramirez-Solis, R., et al. Genomic DNA microextraction: a method to screen numerous samples. Anal Biochem. 201, 331-335 (1992).
- Uren, A. G., et al. A high-throughput splinkerette-PCR method for the isolation and sequencing of retroviral insertion sites. Nat Protoc. 4, 789-798 (2009).
- Wang, W., Bradley, A., Huang, Y. A piggyBac transposon-based genome-wide library of insertionally mutated Blm-deficient murine ES cells. Genome Res. 19, 667-673 (2009).
- Chick, W. S., et al. Screening for stress-resistance mutations in the mouse. Front Genet. 5, 310 (2014).
- Salmon, A. B., et al. Fibroblast cell lines from young adult mice of long-lived mutant strains are resistant to multiple forms of stress. Am J Physiol Endocrinol Metab. 289, 23-29 (2005).
- Skarnes, W. C., et al. A public gene trap resource for mouse functional genomics. Nat Genet. 36, 543-544 (2015).
- Baker, K. E., Parker, R. Nonsense-mediated mRNA decay: terminating erroneous gene expression. Curr Opin Cell Biol. 16, 293-299 (2004).
- Shigeoka, T., Kawaichi, M., Ishida, Y. Suppression of nonsense-mediated mRNA decay permits unbiased gene trapping in mouse embryonic stem cells. Nucleic Acids Res. 33, e20 (2004).
- Symula, D. J., Zhu, Y., Schimenti, J. C., Rubin, E. M. Functional annotation of mouse mutations in embryonic stem cells by use of expression profiling. Mamm Genome. 15, 1-13 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon