Method Article
Vorwärts genetische Ansatz zur Stressresistenz-Gene in Mäuse Entdecken - Hochdurchsatz-Bildschirm in ES-Zellen
In diesem Artikel
Zusammenfassung
Stressresistenz ist eines der Markenzeichen für Langlebigkeit und ist bekannt, genetisch geregelt. Hier entwickelten wir eine unvoreingenommene Hochdurchsatzverfahren für Mutationen, die Stress-Resistenz verleihen in ES-Zellen, mit dem Maus-Modelle für Langlebigkeit Studien entwickeln zu screenen.
Zusammenfassung
Phenotype-driven genetic screens in mice is a powerful technique to uncover gene functions, but are often hampered by extremely high costs, which severely limits its potential. We describe here the use of mouse embryonic stem (ES) cells as surrogate cells to screen for a phenotype of interest and subsequently introduce these cells into a host embryo to develop into a living mouse carrying the phenotype. This method provides (1) a cost effective, high-throughput platform for genetic screen in mammalian cells; (2) a rapid way to identify the mutated genes and verify causality; and (3) a short-cut to develop mouse mutants directly from these selected ES cells for whole animal studies. We demonstrated the use of paraquat (PQ) to select resistant mutants and identify mutations that confer oxidative stress resistance. Other stressors or cytotoxic compounds may also be used to screen for resistant mutants to uncover novel genetic determinants of a variety of cellular stress resistance.
Einleitung
Langlebigkeit hat eine intime Beziehung mit Stressresistenz. Im allgemeinen langlebige Spezies weisen häufig erhöhte Resistenz gegen multiple Stressoren, wie Wasserstoffperoxid, Paraquat (PQ), UV, Wärme und Schwermetalle 1,2. Im Gegensatz dazu erhöhte Empfindlichkeit gegenüber Stress verursacht verkürzten Lebensdauer und / oder eine Krankheit anfälligen Phänotyp zusagen. Die Antioxidationsmittel fang Weg war lange spekuliert worden, eine wichtige Rolle bei der Verleihung Stressresistenz für das Tier zu spielen. Mit wenigen Ausnahmen, Studien aus einer Vielzahl von transgenen Tieren mit Manipulationen in verschiedenen antioxidativen Enzymen (zB SOD) zeigen jedoch, dass die Erhöhung des Oxidationsfänger Enzyme nicht die Lebensdauer oder die Gesundheit Spannweite 3 zu erhöhen. Diese Daten legen nahe, dass die Stressresistenz Merkmal konsequent in langlebigen Tieren beobachtet wird von anderen zellulären Wege noch aufgedeckt werden vermittelt.
Wir nahmen eine unvoreingenommene vornegenetischer Ansatz für Gene, die auf mutierten, eine Stressresistenz-Phänotyp in kultivierten embryonalen Stammzellen (ES-Zellen) verleihen könnten. ES-Zellen bieten zwei wesentliche Vorteile in dieser Studie: (1) anspruchsvolle genetische Manipulationen sind vorhanden, um das Genom von ES-Zellen zu modifizieren; und (2) Stress resistente ES-Zellen aus dem Bildschirm wiedergewonnen können direkt für Maus Produktion verwendet werden, was eine schnelle Übersetzung in ganzen Tierstudien zur Lebenserwartung und Gesundheitsspanne zu messen.
In diesem Bericht beschrieben wir die Verwendung von C9 ES-Zelllinie, in welcher die BLM Allele unter Steuerung durch ein Tetracyclin ansprechende Element. Behandlung von Doxycyclin (DOX) vorübergehend von der Expression von Blm führt zu einer erhöhten Vorfall von Schwesterchromatidaustausch gedreht. Diese kurzfristige Blm Knock-out für die Erzeugung homozygoter Mutationen innerhalb der heterozygote Population erlaubt, so dass rezessive Mutationen für Stressresistenz könnte in das Screening-Verfahren erfasst werden. Wirbeschrieben die Verwendung von piggyBac (PB) Transposon als erbgutverändernd nach dem Zufallsprinzip setzen Sie einen Poly-A-Trap-Kassette (PB-UPA), um Gene in das Genom zu mutieren. Zellen, die mit Unterbrechung eines Gens durch die Poly-A-Falle wurden G418-resistente und konnte zurückgewonnen, so dass eine Sammlung von Gen-Trap-Mutanten (Gen-Trap-Bibliothek) können hergestellt werden, und in der Folge für die mutierten Klone, die Stress resistent waren zu sehen sein.
Stress-resistente Klone von der Selektion gewonnen könnte ziemlich rasch durch molekulare Techniken hinsichtlich der Anzahl von Einfügungen (qPCR) charakterisiert werden, der Ort der Insertion (splinkerette PCR), die Identität der unterbrochene Gen (BLAST) und sein Expressionsniveau ( RT-qPCR). Die PB Einsetzen könnte durch transiente Expression mPB Transposase in dem Klon wieder mobilisiert werden, um die Wildtyp-DNA-Sequenz wieder herzustellen und damit zu testen für den Verlust von Stressresistenz. Diese sind leistungsfähige Möglichkeiten, um die Kausalität der Mutation zu bestätigen, die vor getan werden sollteteure Maus Produktion. Frühere Studien haben gezeigt, dass Zellen, die Stressfaktoren ausgesetzt verloren ihre Pluripotenz 4,5. Somit wird in diesem Protokoll ist die Erhaltung einer Replikatgruppe von mutierten Zellen, die nicht mit Stressoren behandelt werden, würde die für den erfolgreichen Produktions Maus.
Unser Labor hat den C9 ES-Zelllinie und der PB-UPA-Vektor entwickelt, die beide zur Verfügung stehen, um anderen Forschern auf Anfrage. Das Protokoll die hier berichtet wird durch die Erzeugung von de novo Bibliothek von Gen-gefangen ES-Zellen mit PB-UPA (1A), gefolgt von Replika-Plattierung und Streßselektion auf Stress-resistente Klone (1B) zu isolieren, zu starten. Wir haben gezeigt, die Auswahl mit Paraquat, ein potenter Radikalerzeuger innerhalb der Zellen. Praktisch jede zytotoxische Verbindung oder ein Toxin, beispielsweise ER Stressoren (zB Thapsigargin und Tunicamycin), neuronale Oxidationsmittel (zB MPP +, 6-Hydroxy Dopamin und Rotenon), Wärme und SchwerMetalle (zB Cd, Se) könnten mit dem Verfahren geeignet ist, für die jeweiligen resistenten Mutanten auszuwählen.
Protokoll
1. Gene-gefangen ES Zellbibliothek Construction Mit piggyBac Transposon
- Bereiten primären embryonalen Maus-Fibroblasten (PMEF) als Zubringer für die ES-Zellkultur
- Tauwetter ein Fläschchen von Mitomycin C-inaktivierten PMEF (5,0 x 10 6) in einem 37 ° C Wasserbad. Übertragen der Zellen in 5 ml ES-Zellmedium (DMEM mit 15% FBS, 1000 Einheiten / ml Leukemia Inhibitory Faktoren, 100 & mgr; M nicht-essentiellen Aminosäuren, 2 mM Glutamin, 55 uM 2-Mercaptoethanol, und 25 Einheiten / ml Penicillin / Streptomycin ) und Zentrifugieren bei 100 xg für 5 min.
- Aspiration des Überstands und Resuspendieren der Zellen in 30 ml ES-Zellmedium, gefolgt von der Verteilung in zwei T25-Kolben (jeweils 5 ml) und zwei 100-mm-Platten (je 10 ml). Inkubieren bei 37 ° C, 5% CO 2 für 24 Std.
- Kultur und erweitern C9 ES-Zellen
- Tauwetter ein Fläschchen von C9 ES-Zellen (2,5 × 10 6 Zellen in 0,5 ml) in einem 37 ° C Wasserbad und übertragen Sie dieZellen in 5 ml ES-Zellmedium. Zentrifuge bei 100 × g für 5 min.
- Saugt den Überstand und resuspendiere ES-Zellen in 5 ml ES-Zellmedium, gefolgt von Plattieren in den T25-Kolben Zuführung. Inkubieren bei 37 ° C, 5% CO 2. Ersetzen Sie ES-Zellmedium täglich.
- Nach 2 Tagen in Kultur sollten die ES-Zellen ~ 80% konfluent werden und bereit sind, passagiert werden. Waschen Sie die Zellen mit 5 ml PBS (ohne Ca 2+ und Mg 2+) und fügen Sie 2,5 ml vorgewärmten 0,25% Trypsin-EDTA. Inkubieren bei 37 ° C für 10 min. In 2,5 ml ES-Zell-Medium, um Trypsin zu stoppen; Pipette nach oben und unten 15-mal zu brechen Zellklumpen. Zentrifuge bei 100 × g für 5 min.
- Absaugen Stand und resuspendiere Zellen in 4 ml ES-Zellmedium. 1 ml der Zellen pro 100 mm-Platte mit 9 ml ES-Zellmedium; bereiten zwei 100-mm-Platten. Dies ist ein 1: 8 Passage. Führen Sie die Zellen täglich mit 10 ml ES-Zellmedium.
- Bereiten Sie 96-Loch-Feeder-Platten für die Bibliothek
- Wie in Abschnitt 1.1.1 beschrieben, auftauen drei Fläschchen mit Mitomycin C inaktiviert PMEF (je 5 x 10 6 Zellen) und resuspendieren Zellen aus jeder Flasche mit 33,3 ml ES-Zellmedium. Kombinieren Zellen, um eine 100-ml-Zellsuspension zu erhalten.
- Platte 100 ul Zellen pro Vertiefung auf zehn Platten mit 96 Vertiefungen. Diese 96-Loch Nährplatten sollte mindestens einen Tag vor der Transfektion C9 ES-Zellen mit PB Transposons hergestellt werden.
- Elektroporation von C9 ES-Zellen mit dem PB-UPA-Vektor
- Trypsinize und zählen Sie die erweiterte C9 ES-Zellen unter Verwendung eines automatisierten Zellzählers. Waschen jede 100-mm-Platte von C9 ES-Zellen mit 10 ml PBS. 8 ml vorgewärmtes 0,25% Trypsin-EDTA und Inkubation bei 37 ° C für 10 min.
- Stoppen Trypsin durch Zugabe von 2 ml ES-Zellmedium. Übertragen Sie die Zellsuspension in ein 15-ml-Röhrchen und Pipette nach oben und unten 15-mal, um Zellklumpen auseinander brechen. Sofort bei 100 · g zentrifugieren für 5 min, gefolgt von Absaugen des Überstandes.
- Resuspendieren Zells in 10 ml ES-Zellmedium und zählen Sie die ES-Zellen mit einer Zählkammer. Bereiten Sie sechs 15-ml-Röhrchen Zellsuspension mit je 5 x 10 6 ES-Zellen. Zentrifuge bei 100 × g für 5 min. Saugen Sie den Überstand und resuspendieren jedes Zellpellet in 0,8 ml PBS.
- Bereiten Sie sechs Elektroporationsküvetten (4 mm Spalt), denen die 0,8 ml Zellen übertragen werden. Zu jeder Küvette, fügen Sie 1 & mgr; g PB-UPA und 20 ug mPB Transposase (mPBase); vorsichtig mischen durch Auf-und Abpipettieren. Setzen Sie die Küvetten auf Eis. Führen Elektroporation mit einem Elektroporator unter Verwendung des exponentiellen Einstellung bei 250 V, 500 uF und unendlich Ohm.
HINWEIS: In der Regel etwa 10 bis 12 ms für eine erfolgreiche Elektroporation, die etwa 1500 bis 2000 G418-resistenten Transformanten pro 5 x 10 6 Zellen liefern die Zeitkonstante. - Abrufen und bündeln die transfizierten Zellen mit einem T75-Kolben mit 98 ml ES-Zellmedium. Mischen der Zellen durch vorsichtiges Pipettieren und dann übertragen Zellen zu einem Reservoir (50 ml). Unsing eine 12-Kanal Pipette 100 ul Zellen pro Vertiefung zu der zuvor hergestellten 96-Well-Nährplatten (10 insgesamt). Inkubieren bei 37 ° C, 5% CO 2.
- Nach 24 Stunden zu füttern die Zellen täglich mit frischen ES-Zell-Medium, das G418 (150 ug / ml), um für die Gen-gefangen Klone auszuwählen.
HINWEIS: In der Regel jede Vertiefung wird etwa zehn G418 resistenten Kolonien enthalten. Wenn diese Kolonien wachsen, um eine ausreichende Größe (üblicherweise 5-7 Tage) werden die Platten mit 96 Vertiefungen (die Bibliothek) in 2 Sätzen zur Speicherung und Auswahl bzw. repliziert werden. Jede Platte in der Bibliothek als "Unter-Bibliothek" bezeichnet, so dass in diesem Fall gibt es 10 Teilbibliotheken.
2. Bibliotheks Replication
- Replizieren Sie die Master-Platten
- 24-48 Stunden vor der G418-resistenten Kolonien erreichte 50-60% Konfluenz, tauen noch drei Fläschchen mit Mitomycin C-inaktivierten PMEF auf zehn 96-Well-Platten als Zubringer (siehe Abschnitt 1.3). Gelatineize zehn Platten mit 96 Vertiefungen durch Inkubation 0,1% Gelatine (100 ul / Mulde) bei 37 ° C für mindestens 15 bis 30 min; O / N Inkubation ist akzeptabel.
- Vorbereiten eines einzelnen Zellsuspensionen in den 96-Well-Platten durch Trypsinierung. Aspirat Mediums von 96-Well-Platten und einmal mit einem gleichen Volumen PBS waschen (ohne Ca 2+, Mg 2+). Zugabe von 50 & mgr; l vorgewärmtes 0,25% Trypsin-EDTA, bei 37 ° C in 5% CO 2 für 10 min. Stoppen Trypsin mit 50 ul ES-Zellmedium. Auseinander brechen die Zellklumpen durch Auf- und Abpipettieren 15 Mal mit dem Mehrkanalpipette.
- Transfer 50 ul Trypsin Zellsuspension in die entsprechende Zeile der Zuführplatte (Master) 150 & mgr; ES-Zellmedium bereits enthalten, und die verbleibenden 50 ul in die entsprechende Zeile der gelatinierten Platte (Replik) 150 & mgr; ES-Zellmedium bereits enthalten. Verarbeiten jede Zeile von Zellen zu einem Master und Replikat-Platte. Die Inkubation (jetzt, die ein 200 & mgr; culture Volumen) bei 37 ° C, 5% CO & sub2; O / N.
HINWEIS: Es gibt jetzt zwanzig 96-Well-Platten. - Am nächsten Tag füttern die Zellen mit 100 ul frisch ES-Zell-Medium, das 150 ug / ml G418. Ersetzen Sie das Medium täglich, bis die Vertiefungen sind 80-90% konfluent.
- Frieren Sie die Master-Platten
- Wenn die Master-Platten sind etwa 80% konfluent, trypsinize die Zellen wie beschrieben (siehe Abschnitt 2.1.2).
- Entfernen Sie die Platten und Stop-Trypsin-Reaktion mit 50 ul 2x Gefriermedium (60% ES-Zellmedium, 20% FBS, 20% DMSO). Pipette nach oben und unten 10-mal mit einer Mehrkanalpipette um die richtige Mischung des Gefriermedium zu gewährleisten.
- Setzen Sie die Platten mit 96 Vertiefungen in einem dicht verschlossenen Styroporbox und legen Sie sie in -80 ° C Gefrierschrank für bis zu vier Monaten Lagerung. Vier Monate genug Zeit, um den Stress Auswahl abzuschließen und zu erkennen, welche Brunnen aus dem Master-Platte enthalten, die Klone von Interesse zu ermöglichen.
HINWEIS: Lagerung bei -80 & #176; C länger als 4 Monate in der Abnahme der Lebensfähigkeit der Zellen führen. Man kann Kryo-Röhrchen in 96-Well-Format, das in der Dampfphase von flüssigem Stickstoff für längere Lagerzeit gespeichert werden kann zu nutzen.
- Bereiten DNA Platten und Unterbibliothek Pools von Replikaplatten
- 24-48 h vor der weiteren replizierende DNA zu Platten und Unterbibliothek Pools herzustellen zehn 100 mm-Platten mit Mitomycin C inaktiviert PMEF für die Sub-Bankpool, und zehn gelatinierten Platten mit 96 Vertiefungen für die DNA Platte nachbildet. Siehe Abschnitt 1.3 für allgemeine Verfahren.
- Trypsinize die Zellen in der 96-Well-Platten replica (siehe Abschnitt 2.1.2). Während der 10 min Trypsinisierung Schritt Aspirat Gelatine von den DNA-Platten und ersetzen mit 150 ul ES-Zellmedium, das G418 (150 ug / ml). Außerdem bereiten ein Reservoir, das 5 ml ES-Zellmedium zur Bündelung der "Sub-Bibliotheken in die 100-mm-Feeder-Platte.
- Nach Trypsinierung, Stop versuchenPSIN mit 50 ul ES-Zellmedium einer Zeile zu einem Zeitpunkt unter Verwendung eines 12-Kanal-Pipette. Pipette nach oben und unten 10-mal auseinander zu brechen Zellklumpen.
- Dann werden 50 ul der Zellsuspension in der entsprechenden Zeile der DNA Platte mischen und die restlichen 50 & mgr; l in den Vorratsbehälter, die 5 ml ES-Zellmedium. Nach Abschluss einer vollen 96-Well-Platte, saugen das Medium aus einem 100-mm-Feeder-Platte, an der die vereinigten Zellen übertragen werden. Diese gepoolten mutagenisierten Zellen werden als "Unterbibliothek" bezeichnet.
- Wiederhole den restlichen neun Platten mit 96 Vertiefungen. Führen Sie die 96-Loch-DNA Platten und 100-mm gepoolt 'Unterbibliothek "Platten täglich mit ES-Zell-Medium, das G418 (150 ug / ml).
HINWEIS: Es gibt jetzt zehn 96-Loch 'DNA' Platten und zehn 100-mm "Unterbibliothek" Platten.
- Frieren Sie die DNA-Platten. Wenn die Zellen in der DNA Platte sind 80-90% konfluent, saugen das Medium, waschen Sie die Zellen zweimal mit 100mgr; l PBS und bei -20 ° C für die spätere Isolierung genomischer DNA 6. Diese DNA-Platten werden durch PCR gescreent werden, um festzustellen, welche entsprechend auch in der 'Master' Platte enthält den Klon von Interesse.
3. Doxycyclin-induzierten Homozygote Mutanten
- Frieren Sie die Unterbibliothek Pools und Durchgangszellen für Doxycyclin-Behandlung
- 24 Stunden vor der 100-mm "Unterbibliothek" Platten mit Kolonien groß genug, um 70-80% Konfluenz zu erhalten, bereiten Sie einen Satz von zehn verkleistert 100-mm-Platten für Doxycyclin Behandlung.
- Bereiten Einzelzellsuspensionen der einzelnen Teilbibliothek (die Abschnitte 1.4.1 und 1.4.2 für Details). Übertragungs 5 x 10 6 Zellen der gelierten 100 mm-Platte mit Doxycyclin (1 ug / ml); Inkubieren bei 37 ° C, 5% CO 2. Frieren Sie die verbleibenden nicht Doxycyclin behandelten Teilbibliothek Pools bei 5 × 10 6 Zellen pro Fläschchen.
- Passage-Zellen in GegenwartDoxycyclin, knock out Blm
- Durchgang der Teilbibliotheken, auf 1: 8 alle 2-3 Tage in Gegenwart von Doxycyclin (1 ug / ml) für bis zu zwei Wochen, während welcher Zeit dox-induzierte Knock-out von BLM Verlust der Heterozygotie in der Zelle zu fördern Population, so dass zur Erzeugung von homozygoten Mutanten.
4. Stress-Auswahl und Isolierung von resistenten Klonen
- Betreff Doxycyclin-behandelten Zellen zum Stress Auswahl
- 24 Stunden vor der Passage Doxycyclin behandelten Zellen zum Stress Auswahl, Vorbereitung zehn verkleistert 150-mm-Platten, in einem 37 ° C, 5% CO 2 befeuchteten Inkubator.
- Bereiten Sie eine Einzelzellsuspension der einzelnen Teilbibliothek durch Trypsinisierung (Abschnitt 1.4.1 und 1.4.2 für Details). Seed 6,6 x 10 6 Zellen auf jede 150-mm-Platte in 30 ml Kulturvolumen insgesamt, zu verbreiten Zellen gleichmäßig. Inkubieren Zellen mit Stressor (10 uM Paraquat oder Unterlassung des β-Mercaptoethanol,in ES-Zellmedium enthaltend 7,5% hitzeinaktiviertes FBS) für 7 Tage, nach dem Ersetzen mit normalen ES-Zellmedium um es überlebenden Zellen in Kolonien zum Aufnehmen wachsen.
- Optional: Frieren Sie den links auf Doxycyclin-behandelten Zellen mit 5,0 × 10 6 Zellen pro Fläschchen.
- Pick-Stress-resistente Kolonien
- Nach der Wiederherstellung normaler Medien für eine Woche, zu beobachten, wie viele Stress-resistente Kolonien vorhanden sind. Abhängig von der Anzahl der Kolonien, abgeholt werden, bereiten genug Brunnen mit Mitomycin C-inaktivierten PMEF auf 96-Well-Platten 1 Tag vorher entsprechend (siehe Abschnitt 1.1 für Details).
- Am Tag der Kommissionierung, absaugen und ersetzen Medium in der 96-Loch-Zuführplatte mit 100 ul frisch ES-Zellmedium, legen Sie wieder in 37 ° C, 5% CO 2 befeuchteten Inkubator. Zu dieser Zeit, bereiten Sie einen U-Boden-Platte mit 96 Vertiefungen mit 50 & mgr; l 0,25% Trypsin-EDTA pro Vertiefung. Saugen Sie Medium aus der 150-mm-Stress Selektionsplatte und fügen Sie eQual Volumen PBS.
- Stellen Sie die Mikropipette bis 2 & mgr; l und "Pick 'überlebenden Kolonien in die U-Boden Trypsin enthaltenden Vertiefungen, Kommissionierung eine Kolonie pro Vertiefung. Es ist akzeptabel, lassen Sie die Platte bei Raumtemperatur zu sitzen, bis die gewünschte Anzahl der Kolonien wurde abgeholt. Dann inkubieren Sie die U Bodenplatte bei 37 ° C, 5% CO 2 für 10 min.
HINWEIS: In der Regel übernehmen wir 1 Stunde auf 96 Kolonien holen. - Stoppen Trypsin Reaktions eine Zeile zu einem Zeitpunkt mit 50 ul ES-Zellmedium mit einer Mehrkanalpipette. Pipette nach oben und unten 15 Mal, um eine Einzelzellsuspension zu erhalten, und dann 100 ul der Zellen zu der entsprechenden Zeile der Zuführplatte bereits 100 ul ES-Zellmedium enthält. Die Übergabe für die gesamte Platte. Kulturzellen O / N in 200 & mgr; l. Am nächsten Morgen, absaugen Medium und ersetzen mit 100 ul ES-Zellmedium.
- Replizieren Sie die 96-Well-Platten
- Wenn die Vertiefungen der gepickten Kolonien sind 80-90% confluent, replizieren in zwei passenden Platten, eine für die DNA-Isolierung und die andere für Zellen, Back-up. Diese Platten sind gelatinierte und sich Mitomycin C inaktiviert PMEF enthalten.
- Absaugen Medium und waschen mit 100 ul PBS. In 50 ul 0,25% Trypsin-EDTA zu jeder Vertiefung, bei 37 ° C für 10 min. Stoppen Sie das Trypsin mit 150 ul ES-Zellmedium, mischen durch Pipettieren, und übertragen Sie 100 ul der Zellsuspension in die entsprechende Zeile aus zwei 96-Well-Platten. Ändern Medium täglich.
- Wenn die Platten 80-90% konfluent, Gefriert einem Satz von Platten als "Back-up" (siehe Abschnitt 2.2 für Details). Die andere Gruppe von Platten werden weiter ausgebaut, wie unten beschrieben.
- Ausbau Zellen für genomische DNA-Isolierung
- Bereiten Sie eine Einzelzellsuspension in der 96-Loch-DNA Platte (siehe 2.1.2 für Details).
- Bringen Zellsuspension aus jeder einzelnen Vertiefung der 96-Well-Platte in die Vertiefung einer 24-Well-Platte (4 gesamt) mit einem MICR-opipette. Stellen Sie sicher, es gibt keine Kreuzkontamination von beliebigen Zellen, da die 'abgeholt' Kolonien 'klonalen "Ursprungs. Kulturzellen in 0,5 ml ES-Zellmedium, Änderungsmedium täglich bis Brunnen sind 80-90% konfluent.
HINWEIS: Die 24-Well-Platte wird eine ausreichende Ausbeute an genomischer DNA für die PCR-Analyse zu ermöglichen.
5. Identifizierung der PB Insertion Seiten und Trapped Genes
- Genomische DNA isoliert von 24-Well-Platte: Lyse-Zellen in der 24-Well-Platte und zu isolieren RNA-freie genomische DNA.
- Führen Splinkerette PCR (SpPCR), um Sequenzfragmente flankierenden PB Transposon Integrationsstelle wie beschrieben 7,8 zu erzeugen.
HINWEIS: SpPCR ist ein Fünf-Tage-Prozess, wenn auch Hands-on-Zeit jeden Tag ist minimal.
6. Genetische Analyse der Mutant-Klone
- Suchen Stamm gut und zu reinigen Klon von Interesse.
- Wenn der PB Integrationsstelle wurdeabgebildet, die Gestaltung eines Forward-Primer stromaufwärts von der Integrationsstelle, um mit einem PB Rückwärtsprimer (zB PB3'-1) 8 und den Bildschirm Mithilfe der DNA-Platte entspricht, die gleiche "Unterbibliothek", aus dem der Stress-resistenten Klon abgeholt wurde. Erwarten Sie, um nur eine positive und über die gesamte 96-Loch-DNA Platte zu finden. Auftauen genau dieses auch von der 'Master' Platte auf eine Platte mit 24 Vertiefungen auf Feeder-Zellen und wachsen, bis 80-90% Konfluenz.
HINWEIS: An diesem Punkt enthält dieser auch eine Mischung aus Gen-Klone gefangen. - Wenn der 24-Well-Platte ist 80-90% konfluent, trypsinize Zellen und die Platte 1000 ES-Zellen in einem 100-mm-Zuführplatte (sicherzustellen, Einzelzellsuspension). Passage die restlichen Zellen in eine T25-Flasche auf Zubringer für Expansion. Einfrieren der Zellen von der T25-Flasche bei Kolonien 80-90% konfluent. Frieren Sie 4 Flaschen pro T25-Flasche als Back-ups.
- Lassen Sie die Kolonien für 7 Tage wachsen und dann holen 96 Kolonien in eine 96-Well-feeder Platte. Siehe Abschnitt 4.2 für die Kommissionierung Verfahren. Replizieren Sie die Platte, wenn Sie bereit zu einem 'DNA' Platte und frieren die anderen als "2. -master 'Platte (siehe Abschnitt 2.2 zum Einfrieren).
- Lassen Sie Kolonien auf 80-90% Konfluenz erreichen. Bildschirm des DNA Platte in die Vertiefungen, die das gereinigte Klon von Interesse zu finden. Erweitern Zellen aus dem 96-Loch 2. -master Platte mit 24 Vertiefungen und in der Folge zu einer T25-Flasche (alle auf Feeder). Frieren Sie 4 Flaschen pro T25-Flasche.
HINWEIS: An diesem Punkt ist das Gen-gefangen Zellinie klonalen, nicht zu dem Stressor ausgesetzt wurde, und ist für die Maus-Produktion.
- Wenn der PB Integrationsstelle wurdeabgebildet, die Gestaltung eines Forward-Primer stromaufwärts von der Integrationsstelle, um mit einem PB Rückwärtsprimer (zB PB3'-1) 8 und den Bildschirm Mithilfe der DNA-Platte entspricht, die gleiche "Unterbibliothek", aus dem der Stress-resistenten Klon abgeholt wurde. Erwarten Sie, um nur eine positive und über die gesamte 96-Loch-DNA Platte zu finden. Auftauen genau dieses auch von der 'Master' Platte auf eine Platte mit 24 Vertiefungen auf Feeder-Zellen und wachsen, bis 80-90% Konfluenz.
- Bestimmen Sie die Anzahl der PB Insertion: Tauwetter ein Fläschchen des spannungsresistenten Zelllinie und erweitern Sie die Zellen in einer T25-Flasche verkleistert. Isolieren Sie genomische DNA und überprüfen Sie die Kopienzahl des PB Transposons. Dies kann durch qPCR gezielt den Neomycin-Gen erreicht werden.
- Messen Sie Genexpression Ebene: Nach dem Konfirming es einen einzigen PB Integrationsereignis in der Zelllinie von Interesse ist, zu messen das Niveau der Expression des eingefangenen Gens durch RT-qPCR unter Verwendung von TaqMan-Sonden spezifisch für jedes Gen.
- Sequenz, die den mutierten mRNA: Erzeugen einer cDNA-Fragments durch PCR unter Verwendung eines Upstream-Primer-Annealing mit einem Exon und einem stromabwärtigen Primer-Annealing an die Spleiß-Akzeptor-Sequenz des Gen-Trap-Kassette. Sequenz die cDNA-Fragments zu bestätigen, dass das erwartete Spleißen des eingefangenen Gens kommt.
- Remobilisieren die piggyBac Transposon für kausale Beziehung zwischen dem eingefangenen Gens und Stressresistenz zu testen.
- 24-48 Stunden vor dem Auftauen Zellen für PB Remobilisierung, Tauwetter und Platte DR4 Zubringer auf einer T25-Flasche und zwei 100-mm-Platten. Mindestens einen Tag, nachdem die DR4 Anleger werden auf die T25-Kolben überzogen, Tauwetter ein Fläschchen der Zelllinie mit dem PB Einsetzen und Platte der Zellen in T25-Kolben mit DR4 Feeder. Wachsen die Zellen in ES-Zellmedium48 h, Änderungsmedium täglich.
- Vorbereitung einer Einzelzellsuspension von den ES-Zellen. Zählen von Zellen mit einem automatischen Zellzähler und über 5,0 x 10 6 ES-Zellen in ein 15-ml-Tube. Zentrifuge bei 100 xg und Resuspendieren der Zellen in 800 ul PBS. Übertragen Sie die Suspension auf eine 4 mm Küvette.
- In 20 ug mPBase Puro in die Küvette, mischen durch Pipettieren und elektroporieren Zellen (siehe Abschnitt 1.4 für Details). Verteilen Sie die transfizierten Zellen auf zwei 100-mm-DR4 Nährplatten. Kulturzellen O / N in ES-Zellmedium.
- Am nächsten Morgen, füttern Sie die Zellen mit ES-Medium mit 1 ug / ml Puromycin und Kultur-Zellen für 48 Stunden. Zurückzuziehen Puromycin-Selektion und weiter zu Kultur, die Zellen für 5 Tage. Wenn die Kolonien groß genug für Kommissionierung sind, wählen Sie in einer 96-Well-Zuführplatte (siehe Abschnitt 4.2).
- Wenn die aufgenommenen Zellen 80-90% Konfluenz erreichten, aufgeteilt 1/6 der Platte in eine DNA Platte und 1/6 die plaßen in ein G418 Platte, so dass 2/3 rds der Zellen in der Originalplatte eingefroren werden (siehe Abschnitt 2.2 zum Einfrieren).
- Kultur der DNA Platte in ES-Zellmedium für spätere DNA-Isolierung und die G418 Platte in 150 & mgr; g / ml G418, um den Verlust des Neomycin-Resistenz zu testen. Wells keine lebensfähigen Zellen nach 72-96 Stunden in Kultur G418 enthalten, ergaben erfolgreiche Remobilisierung von dem Gen-Trapping-PB Transposons.
- Die entsprechenden Vertiefungen in der "DNA" Platte wird durch PCR gescreent werden, um die PB Transposon wurde entfernt bestätigen. Wenn die DNA Platte beträgt 90% konfluent, Isolieren der genomischen DNA in 96-Well-Plattenformat 4 für PCR-Screening für die Wiederherstellung der Wildtyp-Sequenz und das Fehlen von Neo-IRES als Mittel zur Reversion zu bestätigen.
- Tauen die passenden Vertiefungen von der Master-Platte auf 24-Well-Platte und zu erweitern, um eine T25-Flasche (alle mit Feeder). Einfrieren vier Ampullen pro T25-Flasche als Back-up.
7. Generierung von Mausmutanten
- Recover resistenten ES-Zellen von den Masterplatten und nutzen sie für die Embryo-Injektion, um Chimären zu erzeugen. Etwa 15-20 ES-Zellen werden in ein Individuum C57BL / 6-Blastocysten injiziert. Transfer 15 Blastozysten pro Maus in die Gebärmutter einer scheinträchtigen weiblichen (Stamm ICR). Kamerad die männliche Chimäre (von der ES-Zellen spezifische Agouti-Fellfarbe angegeben) aus dem Wurf gewonnen mit mehreren Jungfrau C57BL / 6J Weibchen für Keimbahn-Übertragung zu testen und für den Aufbau einer Kolonie von mutierten Mäusen.
- Isolieren und Testschwanzhaut Fibroblasten von den F1 mutierten Mäusen und Wildtyp-Wurfgeschwister für reaktive Sauerstoffspezies (ROS) Ebene und Widerstand gegen Paraquat 9.
Ergebnisse
In einem typischen Experiment Mutagenese erfolgte die Transfektion besteht aus einer Gesamtzahl von 3 x 10 7 ES-Zellen mit PB-UPA und mPB Transposase. Die Zahl der Gene-gefangen ES-Zellen in zwei unabhängigen Experimenten erzeugt, sind in Tabelle 1 zusammengefasst. Die Effizienz der Gen-Einschluss etwa 0,04%. Die kombinierten Gen-Trap-Bibliotheken enthalten 22.400 unabhängigen Mutanten, über eine vollständige Abdeckung des Mausgenoms (23.000 kodierenden Gene). Übermäßige Abdeckung könnte durch Erzeugung von mehr Mutanten durch wiederholte Mutagenese und / oder Skalierung des Prozesses erreicht werden.
Weil die Gen-Trap-Bibliotheken enthalten zufälligen Mutationen, ist es nicht möglich, die Anzahl von Mutanten mit einer Stressresistenz-Phänotyp zusagen. Zwei unabhängige Selektionen für PQ Beständigkeit wurden durchgeführt; die kombinierte Screening von 22.440 unabhängige Gen-Trap-Mutationen entdeckt 17 Mutationen, die zelluläre Resistenz gegen PQ (0,08%) (Tabelle 1) zu übertragen.
Wir haben 3 mutierten Klone, die Pigl, Tiam1 und RFFL gereinigt, von den Master-Platten für die weitere Analyse und-Maus-Produktion. Zellen mit heterozygote Mutation RFFL erfolgreich induziert, um homozygote Nachkommen durch Ausschlagen Blm zu produzieren. Jedoch konnten wir keine homozygote Mutanten aus den Pigl und Tiam1 Mutanten, vermutlich aufgrund einer Homozygotie gebundenen Zell Tode führen. Weglassen von 2-Mercaptoethanol (2-ME) und PQ (Abbildung 2): die Beanspruchbarkeit dieser gereinigten Klonen wurde unter Verwendung von 2 verschiedenen Arten von Stressoren bestätigt. Beim Entfernen des PB aus diesen Klonen wurde die zugehörigen Stressresistenz-Phänotyp verloren (Figur 2), was bestätigt, dass das Gen-Trap-Mutationen sind die ursächlich. Charakterisierung von Klonen, die aus den Master-Platten gewonnen ist entscheidend, da sie während Streßselektion als Ursache des beobachteten Phänotyps ausschließen würde stochastischen Läsion.
Out of 3mutierten ES Zellen in Blastozysten für Maus Produktion (Tabelle 2), 2 Zeilen (Pigl und Tiam1) zeigen Keimbahnübertragung eingeführt. Die RFFL Injektion produziert nur eine weibliche Chimären, die keine optimale Anzahl der Chimäre von Anfang an war. Somit ist das Versagen der Keimbahntransmission des RFFL wahrscheinlich aufgrund einer inkompetenten Chimäre. Wir testeten die zelluläre Phänotyp Hautfibroblasten von den Pigl mutierten Mäusen isoliert und zeigten, dass sie erhalten die reduzierte Konzentration von endogener reaktiven Sauerstoffspezies (ROS) sowie Stressresistenz zu PQ (Abbildung 3).
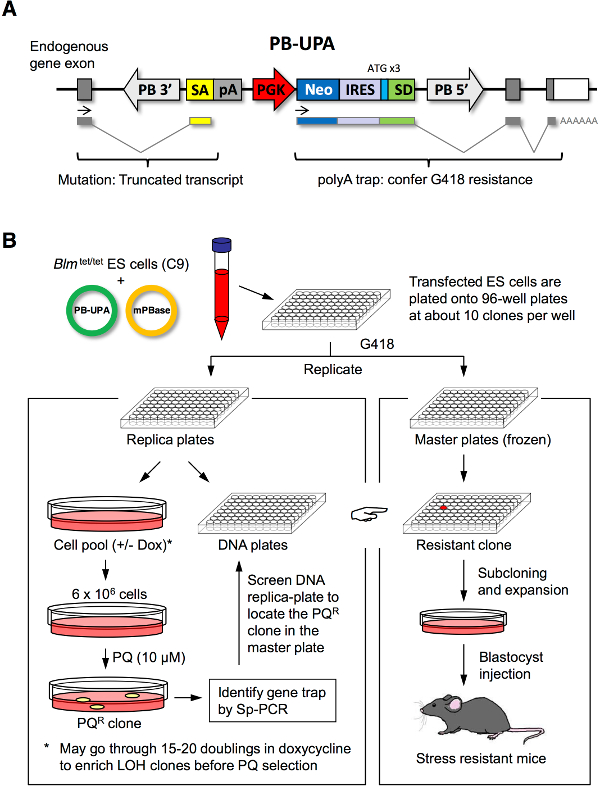
Abbildung 1. ES-Zell-Mutagenese und Stress Auswahl. (A) PB-UPA-Vektor. Eine unvoreingenommene polyA (UPA) Trap-Vektor, der eines Spleiß-Akzeptor (SA), Rinderwachstumshormon-Poly-Adenylat-Signal (pA), phosphoglycerate Kinase (PGK) Promotor, Neomycin-Phosphotransferase (neo), interne ribosomale Eintrittsstelle (IRES), und eine Splice-Donor (SD) mit Kunst ATG (in 3 verschiedenen Leserahmen) in die piggyBac Transposons kloniert, durch die 3 'flankiert und 5 'Long Terminal Repeat (LTR). (B) Zufallsmutagenese von ES-Zellen und Selektion auf Stress resistente Klone. ES-Zellen wurden kotransfiziert durch Elektroporation mit PB-UPA und mPBase gefolgt von Plattieren auf 96-Well-Platten. Gen eingeschlossen Klone wurden durch G418-Resistenz selektiert; einmal konfluent wurden sie in 2-Replikat-Gruppen unterteilt. Ein Replikat-Gruppe wurde weiter durch Paraquat (PQ) in zwei Halb zur DNA-Isolierung und Stress Auswahl unterteilt; das andere Replikat-Gruppe (Master) wurde verkleinert, eingefroren. Die überlebenden ES Zellkolonien aus der Stress-Behandlung gewonnen wurden analysiert molekular für die PB Einsetzen von Sp-PCR. Primer wurden dann entwickelt, um die Replik DNA Platte, von der die gut mit dem sibli Bildschirmng PQ R Klon könnte man auf der Masterplatte befinden. Diese Zellen sind für die Stress-Resistenz-Test auf verschiedenen Stressfaktoren und für Maus-Produktion. Geändert von Chick et al. 7. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 2. Beanspruchbarkeit in mutierten ES-Zellen. (A) Beständigkeit gegenüber 2-Mercaptoethanol (2-ME) Entzug. (B) Beständigkeit gegen Paraquat (PQ). Dargestellt sind die parentalen Wildtyp-ES-Zellen (C9: gelb), ein strapazierfähiges Steuerung ES-Zellklon, (4C11: rot), aus einer früheren Studie 5 gewonnen und drei Gen-Trap-Klone (grau). Zellen wurden einer Wärmebehandlung für zwei Tage, nach der die Zahl der lebensfähigen Zellen wurde gezählt betonen. < em> Pigl, Tiam1 und RFFL Heterozygoten. (PB / +: dunkelgrau) Resistenz gegenüber diesen beiden Stressoren RFFL Homozygoten (PB / PB: schwarz) weisen stärkere Stressresistenz im Vergleich zu den Heterozygoten. Resistenz gegen Stressfaktoren ging verloren, als die PB-Insertionen wurden entfernt (+ / +: hellgrau). Fehlerbalken stellen Standardabweichungen von Mittelwert (n = 4). * P <0,001, # P = 0,01 zwischen den Gen-Trap-Klone und der Wildtyp-Eltern C9, durch den Student-t-Test ausgewertet. (C) ES-Zellkoloniebildung unter PQ (10 uM) Behandlung. Stress-resistente ES-Klone waren in der Lage, Kolonien in Kultur unter PQ Behandlung bilden, während die Anzahl und Größe der Kolonien, die von der Wildtyp-Klon wurden deutlich verringert. Geändert von Chick et al. 7. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
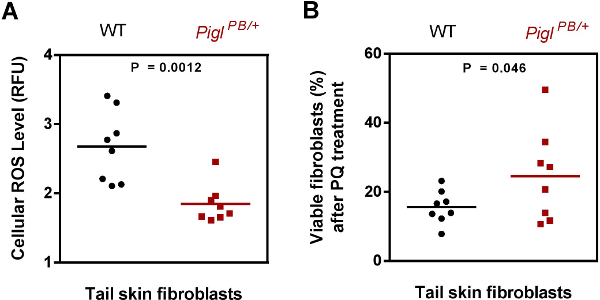
Abbildung 3. Charakterisierung Pigl PB / + Fibroblasten. (A) Reaktive Sauerstoffspezies (ROS) Ebene. Schwanzhaut Fibroblasten von Wildtyp (WT) und Pigl PB / + Mäusen isoliert wurden mit CM-DCFCA gefärbt und die Fluoreszenz wurde gegen Hoechst normalisiert. Die ROS-Gehalt wurde als relative Fluoreszenzeinheit (RFU) angegeben. Der P-Wert wurde von zwei tailed Student-t-Test ausgewertet (n = 8). (B) PQ Beständigkeit. Schwanzhaut Fibroblasten von Wildtyp (WT) und Pigl PB / + Mäuse wurden 4 mM PQ für 6 Stunden, nach der die Lebensfähigkeit der Zellen wurden durch MTT-Assays gemessen ausgesetzt. Der Prozentsatz der lebenden Zellen nach dem PQ-Behandlung wurde durch das Verhältnis der Extinktion von der PQ behandelt ce berechnetells, und dass von den unbehandelten Zellen. Der P-Wert wurde von einem tailed Student-t-Test ausgewertet (n = 8). Geändert von Chick et al. 7. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
| Bibilothek | Teilbibliotheken | ES-Zellstamm | Anzahl der Gen-Klon eingeschlossen | Anzahl der PQ R-Klone |
| 1 | C9PA01 - C9PA10 | C9 (BLM tet / tet) | 9.000 | 7 |
| 2 | C9PA11 - C9PA20 | C9 (BLM tet / tet) | 13.440 | 10 |
| Gesamt | 22.440 | 17 |
Tabelle 1 PiggyBac-Gen-Trap-Bibliothek Bau und die Rückgewinnung von PQ-resistenten Klone. Änderung von Chick et al. 7.
| ES-Zellklon eingespritzt | Anzahl der Chimäre erholt | Keimbahn-Übertragung * |
| Pigl PB / Pigl + | 4 | Ja |
| Tiam1 PB / Tiam1 + | 2 | Ja |
| RFFL PB / RFFL + | 1 | Nein |
Tabelle 2. Erzeugung von Mäusen. Geändert von Chick et al. 7.
* Keimbahntransmission wurde vom Erbe der Genotypisierung mittels PCR nachgewiesen Gen-Trap-Allel bestätigt.
Diskussion
Forward genetic analysis allows for an unbiased interrogation of the genome for genes responsible for a specific phenotype. This method is very powerful to uncover novel gene functions. It has been widely used in lower organisms but not in mammal, such as the mouse, mainly due to the extremely high cost associated with the infrastructure and logistics that would entail. Here, we moved the genetic screening process to the ES cell culture platform, greatly increasing the efficiency and throughput in generating mutants and identifying mutations.
To effectively select for resistant mutants, a lethal dose of stressor and period of treatment needs to be established such that all of the wild-type cells would be killed. The higher the stringency of the selection, the fewer occurrence of false positive will result. Fibroblast feeders have a higher tolerance to oxidative stress than ES cells; the presence of feeders would rescue the nearby ES cells which would otherwise be killed by the stressor. Thus, omission of feeders during stress selection was very critical for an effective killing. In skin fibroblasts, serum deprivation was necessary to reveal the stress resistance phenotype in long lived mice 10. However, this application was not feasible in ES cells because they cannot survive without serum. As an alternative, we discovered that the use of heat inactivated serum at reduced level (7.5%) achieved a similar result.
In this report, we employed poly-A trap rather than a promoter trap because promoter trap will only trap actively transcribed genes, which is estimated to be 60% of the genome in ES cells, under normal conditions 11. In contrast, a poly-A trap would trap genes regardless of their transcriptional activities, in theory giving us the potential to screen for mutations in all genes in the genome. A major drawback of the conventional poly-A trap is the strong bias towards trapping the last exon, appeared to be mediated by an endogenous nonsense-mediated decay (NMD) mechanism 12. By incorporating the unbiased poly-A trap (UPA) strategy in the PB-UPA transposon to suppresses NMD 13, we were able to eliminate biased trapping.
One distinguishing advantage of using ES cells is that these pluripotent cells make direct derivation of mice possible, speeding up the transition to in vivo studies. For this to happen, the replica plating strategy is critical, preserving a subset of clones sheltered from manipulations that can diminish pluripotency, such as knocking out of Blm and exposure to stressors. We have shown that mutant ES cells recovered from these screens were capable of participating in forming the entire mouse and transmitting the mutations to the germline. Most importantly, consistent with previous studies, the stress resistance phenotype was also transmitted and maintained in the adult mouse.
We and others showed that a variant phenotype observed in ES cells resulting from environmental induced lesion or deliberated engineered mutations was able to transmit to the differentiated progeny cells 4,5,9,14, and probably the whole mouse. This protocol describes the utilization of ES cells as surrogate cells to screen for stress resistance mutations in the mouse. In addition to screening for stress resistance genes, this method may have a broader implication to screen ES cells, identify targets, and to develop a variety of mouse models with a phenotype of interest.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We would like to thank the Wellcome Trust Sanger Institute for the gifts of piggyBac transposon and piggyBac transposase. This work was supported by the Butcher grant of Colorado and the NIH R01 AG041801 (W.S.C).
Materialien
| Name | Company | Catalog Number | Comments |
| Vector | |||
| PB-UPA | |||
| mPBase | |||
| mPBasePuro | |||
| Tissue Culture | |||
| 500-ml Stericup filters | EMD Millipore | SCGPU05RE | |
| 250-ml Stericup filters | EMD Millipore | SCGPU02RE | |
| 50-ml Steriflip-GV filters | EMD Millipore | SE1M179M6 | |
| KO DMEM | Life Technologies | 10829-018 | |
| DMEM | Sigma-Aldrich | D6429 | |
| FBS | Tissue Culture Biologicals | 104 | |
| Heat Inactivated FBS | Sigma-Aldrich | F4135-500 | |
| LIF | EMD Millipore | ESG1107 | |
| Non-essential Amino Acids | Life Technologies | 11140-050 | |
| GlutaMAX | Life Technologies | 35050-061 | |
| Pen/Strep | Life Technologies | 15140148 | |
| β-Mercaptoethanol | Life Technologies | 21985-023 | |
| Methyl Viologen dichloride (Paraquat) | Sigma-Aldrich | 856177 | |
| Dimethyl Sulphoxide Hybri-MAX | Sigma-Aldrich | D2650 | |
| EmbryoMAX 0.1% gelatin | EMD Millipore | ES006B | |
| DPBS/Modified | HyClone | SH30028.02 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| T25 Flask | Corning | 353108 | |
| T75 flask | Corning | 353135 | |
| 100-mm plate | Corning | 353003 | |
| 150-mm plate | Corning | 430599 | |
| 96-well plate | Corning | 3585 | |
| 96-well U-bottom plate | Corning | 3799 | |
| 24-well plate | Corning | 3526 | |
| 50-ml reservoir | Corning | 4870 | |
| 15-ml tubes | VWR International, LLC | 82050-276 | |
| Primary Mouse Embryonic Fibroblasts | EMD Millipore | PMEF-NL | |
| DR4 Mouse Embryonic Fibroblasts | Applied StemCell | ASF-1001 | |
| Mitomycin C | Fisher BioReagents | BP25312 | |
| Geneticin (G418) | Life Technologies | 11811-023 | |
| Doxycycline | Fisher BioReagents | BP26531 | |
| Cryotubes | Thermo Scientific | 377267 | |
| Centrifuge | Eppendorf | Centrifuge 5702 | |
| TC10 cell counter | Bio-Rad | ||
| Counting Slides (for TC10) | Bio-Rad | 1450011 | |
| Electroporation | |||
| Gene Pulser Xcell | Bio-Rad | 1652611 | |
| Gene Pulser Cuvettes (4 mm gap) | Bio-Rad | 1652088 | |
| Molecular Biology | |||
| Thermal Cycler | Eppendorf | Mastercylcer ep Gradient S | |
| Puregene Core kit B | Qiagen | 158745 | |
| Topo-TA Cloning kit | Life Technologies | 450030 | |
| High Capacity cDNA synthesis kit | Applied Biosystems | 4368814 | |
| NaCl | Fisher BioReagents | BP358-212 | |
| 100% ethanol | Decon Laboratories, Inc. | 2716 | |
| Double Processed Tissue Culture Water | Sigma-Aldrich | W3500 | |
| Sau3A1 | New England BioLabs | R0169L | |
| T4 DNA Ligase | New England BioLabs | M0202T | |
| EcoRV | New England BioLabs | R3195S | |
| 96-well Lysis Buffer (Ramires-Solis et al. 1992) | |||
| Trizma Base | Sigma-Aldrich | T1503 | |
| Hydrochloric Acid | Fisher BioReagents | A144-212 | |
| EDTA | Sigma-Aldrich | E5134 | |
| N-Lauroylsarcosine sodium salt | Sigma-Aldrich | L5777 | |
| Proteinase-K | Fisher BioReagents | BP1700 | |
| Electrophoresis | |||
| Mini-Sub Cell GT | Bio-Rad | 170-4469EDU | |
| LE Agarose | GeneMate | E3120500 | |
| Ethidium Bromide | Fisher BioReagents | BP1302 | |
| 100 BP DNA Ladder | New England BioLabs | N3231S | |
| 1Kb DNA Ladder | New England BioLabs | N3232S | |
| 2-log DNA Ladder | New England BioLabs | N3200L |
Referenzen
- Johnson, T. E., et al. Longevity genes in the nematode Caenorhabditis elegans also mediate increased resistance to stress and prevent disease. J Inherit Metab Dis. 25, 197-206 (2002).
- Murakami, S., Salmon, A., Miller, R. A. Multiplex stress resistance in cells from long-lived dwarf mice. Faseb J. 17, 1565-1566 (2003).
- Perez, V. I., et al. The overexpression of major antioxidant enzymes does not extend the lifespan of mice. Aging Cell. 8, 73-75 (2009).
- Martin, G. M. Genetic engineering of mice to test the oxidative damage theory of aging. Ann NY Acad Sci. 1055, 26-34 (2005).
- Chick, W. S., Drechsel, D. A., Hammond, W., Patel, M., Johnson, T. E. Transmission of mutant phenotypes from ES cells to adult mice. Mamm Genome. 20, 734-740 (2009).
- Ramirez-Solis, R., et al. Genomic DNA microextraction: a method to screen numerous samples. Anal Biochem. 201, 331-335 (1992).
- Uren, A. G., et al. A high-throughput splinkerette-PCR method for the isolation and sequencing of retroviral insertion sites. Nat Protoc. 4, 789-798 (2009).
- Wang, W., Bradley, A., Huang, Y. A piggyBac transposon-based genome-wide library of insertionally mutated Blm-deficient murine ES cells. Genome Res. 19, 667-673 (2009).
- Chick, W. S., et al. Screening for stress-resistance mutations in the mouse. Front Genet. 5, 310 (2014).
- Salmon, A. B., et al. Fibroblast cell lines from young adult mice of long-lived mutant strains are resistant to multiple forms of stress. Am J Physiol Endocrinol Metab. 289, 23-29 (2005).
- Skarnes, W. C., et al. A public gene trap resource for mouse functional genomics. Nat Genet. 36, 543-544 (2015).
- Baker, K. E., Parker, R. Nonsense-mediated mRNA decay: terminating erroneous gene expression. Curr Opin Cell Biol. 16, 293-299 (2004).
- Shigeoka, T., Kawaichi, M., Ishida, Y. Suppression of nonsense-mediated mRNA decay permits unbiased gene trapping in mouse embryonic stem cells. Nucleic Acids Res. 33, e20 (2004).
- Symula, D. J., Zhu, Y., Schimenti, J. C., Rubin, E. M. Functional annotation of mouse mutations in embryonic stem cells by use of expression profiling. Mamm Genome. 15, 1-13 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten