Method Article
蛍光灯と組み合わせることで染色体の複製タイミング
要約
染色体の複製タイミングの解析のための定量的な方法が記載されている。この方法は、蛍光との組み合わせでBrdUの取り込みを利用その場で哺乳類の染色体の複製タイミングを評価するためのハイブリダイゼーション(FISH)。この技術は、同じセル内に再配置および再配置非染色体の直接比較することができます。
要約
哺乳類のDNA複製は、時間的なレプリケーション·プログラムに続いて、S期に異なる時点で染色体に沿って、複数のサイトで開始されます。複製タイミングの仕様は、エピジェネティックな修飾に敏感な組織特異的、発達的な手がかりによって規制ダイナミックなプロセスであると考えられている。しかし、どこで、DNA複製が染色体に沿って開始したときに調節メカニズムはよくわかっていないままです。相同染色体は通常、同期複製は、しかし、この規則には注目すべき例外があります。例えば、女性の哺乳動物細胞内で2本のX染色体の一つがX不活性1として知られるプロセスを通じて後期複製になります。 2〜3時間であると推定複製タイミングでこの遅延に伴い、遺伝子の大部分は、転写一つのX染色体上に沈黙なる。また、X不活性化の中心地として知られ、離散シス作用性遺伝子座は、目を含めて、このX不活化工程を調節全体の不活性X染色体上の遅延複製タイミングの電子誘導。また、癌細胞では、細胞内に存在する特定の染色体再配列は、電離放射線にさらされる染色体全体に影響を及ぼす2,3> 3時間の複製タイミングで大幅な遅延が表示されます。我々の研究室から最近の研究では、離散と混乱シス作用性染色体全体の4に影響を与える非常に遅く複製表現型常染色体の遺伝子座の結果を示します。追加された'染色体工学"の研究では、すべての哺乳類の染色体は、個々の染色体の適切な複製タイミング5を制御離散シス作用性遺伝子座が含まれていることを示唆し、この異常な複製タイミングの表現型には多くの異なる染色結果に影響を与え、特定の染色体再配列を示す。
ここでは、蛍光in situハイブリダイゼーションと組み合わせた染色体複製タイミングの定量分析のための方法を提示する。このメソッドは、同じセル内の相同染色体間で複製タイミングの直接比較を可能にし、6から適応されました。さらに、このメソッドは、染色体全体に影響を与える複製タイミングの変化と相関染色体再編成の明確な識別を可能にします。この方法では、最近開発されたハイスループットマイクロアレイまたは相同対立遺伝子が再編成と非再配列染色体上に存在を区別することはできませんシーケンスプロトコルの上で利点があります。ここで説明する方法は、単一細胞を評価するためだけでなく、それは、集団中の細胞の一部のみに存在する染色体再配列で染色体複製タイミングの変化を検出することができます。
プロトコル
1。 BrdUの取り込み(末端標識)
- BrdUの添加する前に、150mmの組織培養ディッシュ24時間で約70%コンフルエンスに細胞をプレート。
- 収穫前に適切な時点で20μg/ mlののBrdU(Sigma)を含有する新鮮な完全培地でメディアを交換してください。時間セルの長さは、BrdUによる培地で培養する細胞の種類や種によって異なりますが、一般的にG2期は2〜5時間( 図1)が続きます。
2。単層細胞培養物の染色体の収穫
- プレートから培地を取り出して、15 mlコニカル遠心管に10ミリリットルを収集します。残りのメディアを捨ててください。
- 10ミリリットル正矢[またはHBSS(シグマ)]で一回細胞を洗浄します。
- 板に正矢(またはHBSS)で5ミリリットル、0.25%トリプシンを追加します。細胞がプレートから分離されるまで室温でインキュベートする。
- ステップ2.1からのメディアの10ミリリットルを含むチューブにトリプシン処理した細胞懸濁液を追加します
- 細胞を沈殿させ、10分間、400×gで遠心分離します。メディアプラス細胞ペレットを0.5mlのを残して、上清を吸引します。
- パスツールピペットでピペッティングでよく細胞を再懸濁する。
- 低張液を3滴追加(を0.075のKCl(Sigma社)を37℃に温め )、パスツールピペットを用いて細胞懸濁液を混ぜる。 0.5ミリリットル低張液を追加し、パスツールピペットを用いて再び混ぜる。 5ミリリットルに低張液の量を持参し、再度混ぜる。細胞の種類に応じて20から45分、37℃でインキュベートする。
- 10分間、400×gで遠心分離します。メディアプラス細胞ペレットを0.5mlのを残して、上清を吸引します。
- 優しくチューブをフリックすることによって、細胞ペレットを再懸濁します。浸透腫れ細胞はこの時点では壊れやすいとなりますので、注意が細胞膜を破壊しないように注意するべきです。
- カルノア固定液3滴(1:3氷酢酸:メタノール)を追加します;優しくパスツールpipettとペッティング電子。優しくパスツールピペットでピペッティングして0.5 mlの固定液と混ぜる。 5mlに固定液の量を持参し、穏やかにパスツールピペットでピペッティングにより再び混ぜる。固定された細胞は、数ヶ月のために-20℃で暗所に保存することができます。
- 遠心分離し、ステップ2.8のように吸引します。
- 新鮮な固定液で細胞ペレットを再懸濁し、ペレットとadd〜10xのカルノア固定液の容積を推定する。
- ぬれた、氷のように冷たい顕微鏡スライド上に細胞懸濁液を滴下して追加します。〜45°の角度でスライドを保持し、細胞懸濁液は、スライドの表面上を流れるようになります。ペーパータオルの上で平らなスライドや洪水固定でスライドを下にして置き、空気乾燥することができます。倒立顕微鏡を用いて細胞分裂スプレッドの存在のためのスライドを確認してください。
3。 RNase処理とエタノール脱水
- 37&ドでインキュベートし、10μgの/スライドごとに2×SSCでmlのRNase A(シグマ)の代わりに200μlのグラム、Cで1時間。
- 洗浄は、3分間ずつ室温で2×SSC、pH7.0で3回スライドします。
- 3分間ずつ室温でエタノール(シグマ)シリーズ(70%、90%、100%)を使用してスライドを脱水する。
- 空気は、室温でスライドを乾燥させてください。
4。 in situハイブリダイゼーションのBACプラス染色体に特異的なセントロメア列挙プローブ(CEP)、または全染色体塗料用のプローブのカクテルの調製
- BAC / CEP同時ハイブリダイゼーションのために、プレハイブリダイゼーションのために2つの別々のプローブのカクテルを用意してください。我々は、より少ないバックグラウンドと高いシグナル強度の異なるジェンの結果で個別BACおよびCEPのプローブをプレハイブリダイズすることを見出した。我々はまた、個々のフォスミドもBACクローンの代わりに使用することができることを見出した。後述のカクテル処方は、単一のスライド用のボリュームを表します。同じプローブを用いて、複数のスライドを処理する場合は、単にボリュームを上げるappropr iately。
* BAC DNAプローブのカクテル:
11.5μlのハイブリダイゼーション緩衝液(50%ホルムアミド(シグマ)/ 2×SSC /デキストラン硫酸(Sigma社製))を2μlのdH 2 O
のdH 2 Oに2.5μlのBAC/Cot1 DNA(プロトコルのラベリングについては下記を参照)15μlの合計体積
* CEPプローブカクテル:
7μlのCEPのハイブリダイゼーション緩衝液(65%Formamide/2x SSC /デキストラン硫酸)を2μlのdH 2 O
1μlのCEP/Cot1のDNA(スペクトラムOrange/Cy3/Texas赤で標識し購入)を10μlの合計体積
* 3:02比率を25μl/スライドとミックスプローブカクテルでCEPのプローブを用いてミックスのBACプローブを変性し、プレハイブリダイゼーション(ステップ5.1)の後。
OR
- チューブに全染色体ペイントプローブのアリコートを20μl/スライド(Cy3/Texas赤)。変性およびプレハイブリダイゼーションのステップに進みます。
- 75℃で変性プローブカクテル(S)℃で10分間。 COT1 DNAが反復配列にハイブリダイズするようにして染色体を分裂中期後続のハイブリダイゼーションを防止するために可能にするために30分間37℃で事前にハイブリダイズする。
- 72少なくとも70%Formamide/2x SSC、pH7.0中コプリンジャーに変性スライド℃で 3分間インキュベートする。
- すぐに3分間ずつ4でコプリンジャーにエタノールシリーズ(70%、90%、100%)℃を通してスライドを脱水する。
- スライドは、室温で空気乾燥させてください。
- 45〜プリ暖かいスライド°Cプローブプリhybridzation(ステップ5.1)の最後の10分間ウォーマースライド上。必要に応じて事前にハイブリダイズしたプローブのカクテルをミックスし、各スライドに25 ulを加える;優しくゴムセメントを持つすべてのエッジに沿って、各スライド及びシールにカバースリップを置く。蒸発を防ぐために加湿チャンバー中で37℃で一晩インキュベートする。
6。ポストハイブリダイゼーション洗浄の
- 3分ごとに38から40°Cで50パーセントformamide/2x SSC、pHは7.0でコプリンジャー3回でスライドを洗浄します。洗浄の温度はプローブ間で異なる場合があります。高いバックグラウンドハイブリダイゼーションがある場合は、洗浄の温度を上げることができます。信号が微弱でないと背景がない場合は逆に、あなたはこれらの洗浄の温度を下げることができます。
- 洗浄は、室温でのPN緩衝液中の1時間をスライド 3分間、およびBrdUの検出のステップに進みます。
7。 BrdUの検出
- 暗闇の中で、室温で10分間(200μL/スライド)のPNMバッファを持つブロックをスライドする。
- ペーパータオルの上にエッジをオンにすることでスライドを排出します。 37℃で30分間、PNMバッファに抗BrdU-FITC(50μg/ mlのロシュ社またはミリポア)100μLを°C。
- 3分間ずつ室温でPNバッファにスライドを3回洗浄します。
- ドレインペーパータオルの上にエッジをオンにすることにより、一度に1スライド上の過剰のPNバッファ。 20μlのDAPI /退色マウンティング溶液(Invitrogen)を追加します。 ペーパータオルでスライドをカバーし、気泡や過剰マウントソリューションを強制的にカバースリップを下に押します。画像解析に進みます。
8。画像をキャプチャし、BrdUの取り込みを定量化
- 画像はオリンパスBX61蛍光顕微鏡オリンパスCCDカメラ、100xの目的は、自動フィルターホイールとCytovisionソフトウェア(アプライドイメージング)で撮影されています。 BrdUはFITCフィルターを用いて捕獲され、染色体塗料(テキサスレッド、メタシステムまたはCytocell)、BACの(Cy3)とCEP(Vysisスペクトラムオレンジ)プローブは、テキサスレッドまたはCy3フィルタを使用してキャプチャされ、DAPIはDAPIフィルター(参照して撮影した図2、図3)。
- 興味のある個々の染色体は、BAC / CEPまたは染色体特異ペイントプローブ( 図2Bおよび3B)で識別されます。活用Cytovisionソフトウェアは、関心のある各染色体が全体として中期スプレッドから"カットアウト"ですが、ラインが長いアーム( 図2Cと3C)への短腕から染色体の長さ方向に沿って通って引き込まれる。 Cytovisionソフトウェアは、各染色体の長さ方向に沿ってピクセル強度プロファイルを定量化するために使用されます。 Cytovisionソフトウェアは、画素が占める面積を計算し、各単離染色体上でのBrdUまたはDAPI信号によって表されるピクセルの強度を定量化します。各染色体によって表される平均ピクセル強度は、その後の総画素数を( 表1、図4)を得るためにこれらの画素が占める面積で乗算されます。また、このプロトコルは、染色体の最新の複製領域の可視化を可能にします。したがって、BrdUの取り込みの縞状パターンが積極的に複製する染色体領域、およびchromos間の複製タイミングの違いを検出することができるOMEのペアがこのバンドパターン( 図2及び図3)の違いと見られている。最後に、結合パターンの違いは、相同染色体または染色体腕4間のレプリケーションのタイミング差の推定を可能にし、各染色体6,7、のために知られている複製タイミングのマップと比較することができます。例えば、二つの第6染色体の図3Aのは第6染色体4の正常な複製タイミングと比較した場合、> 2時間の横縞模様の違いを表示します。
また、ここで説明したものと同様の手順が正常に非アクティブとアクティブX染色体8,9間の非同期複製タイミングの研究のために使用されてきました。アクティブおよび非アクティブなX染色体はX染色体塗料を用いて同定した。画像は皮肉mFISHソフトウェア(Vysis)とX染色体によって占め画素数と蛍光ラによって占有さのピクセル数を用いて得たbeled BrdUはその後、NIHイメージ(使用して計算されたhttp://rsb.info.nih.gov/nih-image/index.htmlを )。
9。蛍光標識したBAC DNAのニック翻訳(Vysis)
- 冷やし微量遠心管に、以下のプローブミックスを追加します。
のdH 2 Oで70μlの(4μgのDNA)
10μlの0.2 mMのdUTPをオレンジまたはグリーン
20μlの0.1 mMのdTTPを
20μlの10xのニック変換バッファ
40μlのNTのdNTPの(マイナスのdTTP)
40μlのニックトランスレーション酵素
200μlの総量
インキュベート@ 16℃のCO / N 70℃で加熱することにより反応を停止℃で10分間。氷上で5分間冷やします。 - プローブミックスに以下を加えることで、エタノールでDNAを沈殿させる。
200μlのプローブ溶液
48μlの3 M NaOAc
160μlのCot1DNA(0.25μgの/ UL)
1200μlの100%EtOHを - O / N〜10分間-80℃で保存
- iをスピン20分間12,000 xgで冷n。 EtOHをオフデカント。 70%EtOHで1回洗浄します。前と同じようにスピン。 EtOHをオフデカント。空気だけでエタノールが蒸発するまで乾燥させます。 BAC DNAの100 ng /μlにの最終濃度を40μlのdH 2 Oに再懸濁する。
結果
ヒトの6番染色体の複製タイミング解析の例を図2に示します。 6q16.1に位置ASAR6遺伝子4の欠失を含む細胞は、5時間のBrdUにさらされる有糸分裂細胞のために収穫し、6番染色体ペイントプローブ(Vysis)で魚やBrdUの取り込みのために処理した。 2 6の、第6染色体の一つのために> 2時間の複製タイミングの遅延と一致している間にBrdUのバンドパターンに有意差があることに注意してください[前の6番染色体の横縞模様複製タイミングのために4を参照してください削除]。また、DAPI染色の同様の分析( 図2D)に比べてBrdUの取り込み(ピクセル)の合計金額に著しい差がある。
ヒトの6番染色体の複製タイミング解析のもう一つの例を図3に示します。 samを含むセル4は、 図2は 5時間のBrdUにさらされた場合、有糸分裂細胞のために採取し、示されており、第6染色体CEPプローブプラスASAR6遺伝子を含むBACとし、BrdUの取り込みのための魚のための処理ASAR6遺伝子のメールの削除。削除された染色体6(Δ6)が非欠失染色体6よりBrdUの取り込みとBrdUの取り込みをより拡張されたバンドパターンを表示することに注意してください。 7種類の細胞から分裂スプレッドは図3のように処理したとDAPIおよびBrdUの両方のための画素プロファイルは表1に示されている。

図1:BrdUの端子ラベルスキーム。 8から10時間の最後の、そしてG2の哺乳動物細胞における典型的なS期は、通常2〜5時間である。 BrdUを複製する染色体の最後の部分にラベルを付けるために時間の期間(緑の矢印)を増加させるために添加される。木曜日哺乳動物細胞における電子染色体は初期と後期の複製はそれぞれS期の開始時と終了時に発生する(黒線)と、時間的なプログラムに応じて複製します。不活性X染色体は、DNA合成の大部分は、S相(青線)の後半の間に起こると複製タイミングで表示されています。遅延複製タイミングを持つ染色体がアクティブなレプリケーションは、G2(赤線)まで継続して、DNA合成の開始と終了の両方で表示されています。非同期の培養は、有糸分裂細胞のために収穫し、BrdUの取り込みと魚のために処理されます。
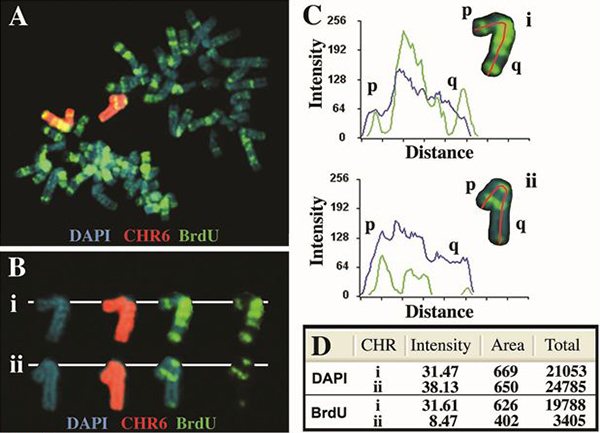
図2:ヒト染色体6の非同期レプリケーション。 ASAR6遺伝子4の人工欠失を含む細胞は、5時間のBrdUで処理された有糸分裂細胞のために収穫し、BrdUの取り込みとCHROMを用いたFISHのために処理したプローブとしてお染6ペイント。 DNAはDAPI(青)で染色した。 A)はBrdUの取り込み(緑)の典型的な "バンド"のパターンを含む分裂広がりが示されています。第6染色体塗料用プローブは2染色体6の(赤)このセル内にハイブリダイズした。 B)の2第6染色体の(IおよびII)を "切り取る"と異なる画像に区切られた三つの蛍光標識で表示されていました。 C)は第6染色体のCytovisionソフトウェアを使用して分析し、DAPI(青)およびBrdU(緑)の両方の信号強度プロファイルが表示されます。赤い線は、短腕(p)からのBrdUおよびDAPI両方の定量化のための長腕(q)に、使用されたパスを示します。距離は、短腕(p)からピクセル単位長腕(q)に、各染色体の長さを指します。 D)はDAPIおよびBrdUの蛍光の両方の全信号の定量。合計値は、それらのピクセルによって表される面積を乗じた平均ピクセル強度を表す。
igure 3は "src =" / files/ftp_upload/4400/4400fig3.jpg "/>
図3。ASAR6の削除を含むヒト第6染色体の複製を遅延させた。 ASAR6遺伝子4の人工欠失を含む細胞は、5時間のBrdUで処理された有糸分裂細胞のために収穫し、BrdUの取り込みおよび第6染色体CEPのプラスのプローブとしてASAR6遺伝子を含むBACを用いたFISHのために処理した。 DNAはDAPI(青)で染色した。 A)はBrdUの取り込み(緑)の典型的な "バンド"のパターンを含む分裂広がりが示されています。第6染色体CEPのプローブには、2つの染色体6の(赤い大きなセントロメア信号)にハイブリダイズし、BACはこのセル内の単一の染色体6(長腕上の小さな赤い信号)にハイブリダイズした。 2 6の間のBrdUのバンドパターンの違いに注意してください。 B)は削除された染色体6は6でΔ6と非削除6で表されます。 2 6の "カットアウト"と異なる画像に区切られた三つの蛍光標識で表示されていました。 C)がT彼は第6染色体のCytovisionソフトウェアを使用して分析し、DAPI(青)およびBrdU(緑)の両方の信号強度プロファイルが表示されます。赤い線は、短腕(p)からのBrdUおよびDAPI両方の定量化のための長腕(q)に、使用されたパスを示します。距離は、短腕(p)からピクセル単位長腕(q)に、各染色体の長さを指します。 D)はDAPIおよびBrdUの蛍光の両方の全信号の定量。合計値は、それらのピクセルによって表される面積を乗じた平均ピクセル強度を表す。
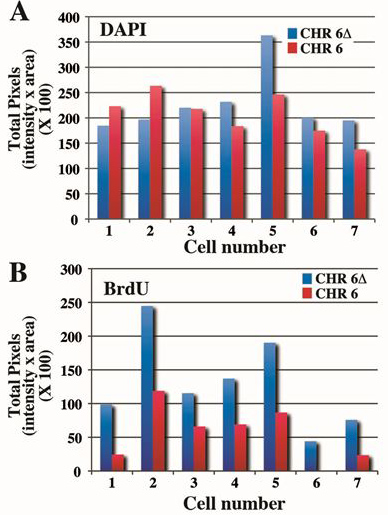
図4:第6染色体の間でのレプリケーションのタイミング差の定量化。 ASAR6遺伝子4の人工欠失を含む細胞は、染色体6 CEPプローブプラス使用して、5時間のBrdUで処理有糸分裂細胞のために収穫し、BrdUの取り込みおよびFISHのために処理したBACは、プローブとしてASAR6遺伝子を含む。分裂スプレッドは図3のように処理したと7種類のセルの値は、( 表1を参照)が示されている。 A)はDAPI染色は定量化され、総画素数は、各セルごとに表示されます。同じセル内の染色体の間だけ〜10-20%の差があることに注意してください。 B)はBrdUの取り込みは、上記のパネルBと同じ染色体から定量した。削除され、非削除第6染色体のための総画素値の> 2倍の差があることに注意してください。
ディスカッション
染色体スプレッドの調製は、ここで説明する正常な複製タイミングのアッセイのための重要なステップです。低張処理に先立っコルセミド前処理工程を含めることは、周波数と有糸分裂細胞の広がりを助けることができる。我々は通常、収穫前に1から3時間colcemdiに細胞を公開し、10μg/ mlの最終濃度でコルセミド使用します。しかし、コルセミド前処理工程を含めることは、G2の長さを変更することがあり、その結果、染色体3の凝縮の見かけの複製タイミングと状態を変更することがあります。
この手順は、細胞周期の継続時間に依存BrdUのインキュベーション、の長さを変えることによって、さまざまな種類の細胞や種に適用することができます。ほとんどのヒトとマウスの細胞株では、G2期は通常、3から6時間であるため、BrdUの治療は、この範囲にある。 BrdUへの代替手段は、5 - エチニル2'deoxyuridine(EDU)と、その使用方法とその後の検出である蛍光アジドと"クリックケミストリー"反応10。 eduの検出方式はBrdUの検出方式に比べていくつかの利点を持っています。例えば、EDUの検出は、試料の固定やDNA変性を必要としません。このように、ロバの複製タイミングにEDUの利用は、目的の染色体を識別するために、魚の代わりシンプルなG-バンディング技術と組み合わせることができます。
ここで説明する複製タイミングプロトコルは、具体的に、S検定後期プラスG2に伸びる任意のDNA合成に設計されています。さらに、DNA複製は6月10日時間の比較的長い期間続く追撃のBrdU(15-30分)短パルスを使用してこの手順を使用してS期を通してモニターすることができます。これは、S期初期と中期の両方においてBrdUの取り込みを可視化することができます。たとえば、特定の腫瘍由来の染色体再配列は、染色体3の全長に沿ってDNA合成の開始と終了の両方で遅延している(商標)。
この複製タイミング手順の一つの利点は、個々の細胞のそれアッセイは複製タイミングということです。そのため、同一セル内に含まれる相同染色体間で複製タイミングの差異を検出する能力を持っています。他の手順がありますが、 例えば 。 in situハイブリダイゼーション複製タイミング(ReTiSH; 11)の相同染色体上の特定の遺伝子座における対立遺伝子間の複製タイミングの違いを検出する能力を持っている、ここで説明する手順では、染色体の全長に沿って複製タイミングの違いを検出することができます。また、この手順では、人口3の細胞のごく一部に存在する染色体の複製タイミングでアッセイ相違することができます。例えば、多くの癌細胞株および原発腫瘍サンプルは、細胞の50%未満で存在している染色体再配列が含まれています。我々は現在、アッセイ染色体には、この手順を使用している原発腫瘍のサンプルであり、複数のサンプルの染色体の間で非同期レプリケーションを検出することができました。しかし、原発腫瘍サンプルは有糸分裂像の数が限られていることを考えると、初代培養の約3分の1は、有糸分裂スプレッドの十分な数を与えることができませんでした。
マイクロアレイやシーケンシングベースのアッセイを介してこの手順が持っているもう一つの利点は、個々の染色体ではなく、細胞のプールから免疫沈降したDNAよりも検定しているということです。対立遺伝子間の複製タイミングを区別するために、免疫沈降ベースのアッセイでは多型が同定されなければならず、特定の対立遺伝子にリンクされます。
さらに、多くのがん細胞は、多数の染色体再配列12、およびDNA複製ストレスが癌細胞13におけるゲノム不安定性に関連付けられているという観測が含まれていることを認識して、我々は、このプロトコルは便利でシンプルなツールfであると信じている癌細胞の染色体の複製タイミングのまたはルーチン分析。
開示事項
特別な利害関係は宣言されません。
謝辞
この作品は、米国国立がん研究所、CA131967からの助成金によってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名称 | 会社 | カタログ番号 | コメント(オプション) |
| 抗BrdU-FITC | ロシュミリポア | 11202593001 MAB326F | 50μg/μLの |
| ニックトランスレーションキット | アボット分子(Vysis) | 07J00-0001 | |
| スペクトラムオレンジdUTPを | アボット分子(Vysis) | 02N33-050 | |
| CEP | アボット分子(Vysis) | 変化する | |
| LSI / WCPハイブリダイゼーションバッファー | アボット分子(Vysis) | 06J67-011 | |
| CEPのハイブリダイゼーションバッファー | アボット分子(Vysis) | 07J36-001 | |
| 染色体パイNTS | メタシステムグループ | D-14NN-050-TR | |
| オリンパスBX61蛍光顕微鏡 | オリンポス | BX61TRF-1-5 | |
| 顕微鏡イメージングソフトウェアシステム | アプライドイメージング | Cytovision 3.93.1 | |
| デジタルカメラ | オリンポス | UCMAD3 | |
in situハイブリダイゼーションのレシピで 35ミリリットルホルムアミド*(シグマ) *これは、-20℃で保管されていることホルムアミドを使用することが重要です。長時間の室温貯蔵ではギ酸とpHワットを生成します病気低すぎるかも。 50パーセントFormamide/2x SSC 25mlのホルムアミド(シグマ) 20×SSC、4 L 702グラムのNaCl(シグマ) PNバッファ[0.1MのNaP0 4 0.1%NP_40(シグマ)] 0.1M溶液リン酸ナトリウムのそれぞれを(フィルター滅菌し、500mlアリコートで保存)してください。 、0.1MのNaH 2 PO 4、1L 13.8グラムのNaH 2 PO 4(シグマ): 、0.1MのNaH 2 PO 4、1L 14.2グラムのNaH 2 PO 4(シグマ) PN:0.1 MのNaH 2 PO 4と0.1Mの2 HP0 NA 4 pHは8.0のpHを調整する。殺菌フィルタリングおよびNP-40の1 mlを加える。 PNMは50ミリリットル 1.25グラム無脂肪ドライミルク(シグマ) 絶えず撹拌しながら15〜20分間混ぜる。 10分間、400×gで2回スピン。上清を使用し、析出した乳蛋白質を乱さないように注意してください。 |
参考文献
- Payer, B., Lee, J. T. X chromosome dosage compensation: how mammals keep the balance. Annu. Rev. Genet. 42, 733-772 (2008).
- Breger, K. S., Smith, L., Turker, M. S., Thayer, M. J. Ionizing radiation induces frequent translocations with delayed replication and condensation. Cancer Research. 64, 8231-8238 (2004).
- Smith, L., Plug, A., Thayer, M. Delayed Replication Timing Leads to Delayed Mitotic Chromosome Condensation and Chromosomal Instability of Chromosome Translocations. Proc. Natl. Acad. Sci. U.S.A. 98, 13300-13305 (2001).
- Stoffregen, E. P., Donley, N., Stauffer, D., Smith, L., Thayer, M. J. An autosomal locus that controls chromosome-wide replication timing and mono-allelicexpression. Hum. Mol. Genet. 20, 2366-2378 (2011).
- Breger, K. S., Smith, L., Thayer, M. J. Engineering translocations with delayed replication: evidence for cis control of chromosome replication timing. Hum. Mol. Genet. 14, 2813-2827 (2005).
- Camargo, M., Cervenka, J. Patterns of DNA replication of human chromosomes. II. Replication map and replication model. Am. J. Hum. Genet. 34, 757-780 (1982).
- Cohen, S. M., Cobb, E. R., Cordeiro-Stone, M., Kaufman, D. G. Identification of chromosomal bands replicating early in the S phase of normal human fibroblasts. Exp. Cell Res. 245 (98), 321-329 (1998).
- Diaz-Perez, S., et al. The element(s) at the nontranscribed Xist locus of the active X chromosome controls chromosomal replication timing in the mouse. Genetics. 171, 663-672 (2005).
- Diaz-Perez, S. V., et al. A deletion at the mouse Xist gene exposes trans-effects that alter the heterochromatin of the inactive X chromosome and the replication time and DNA stability of both X chromosomes. Genetics. 174, 1115-1133 (2006).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc. Natl. Acad. Sci. U.S.A. 105, 2415-2420 (2008).
- Schlesinger, S., Selig, S., Bergman, Y., Cedar, H. Allelic inactivation of rDNA loci. Genes Dev. 23, 2437-2447 (2009).
- Mitelman Database of Chromosome Aberrations in Cancer. , Available from: http://cgap.nci.nih.gov/Chromosomes/Mitelman (2006).
- Branzei, D., Foiani, M. The checkpoint response to replication stress. DNA Repair (Amst). 8, 1038-1046 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved