Method Article
Timing chromosome Réplication Combiné avec fluorescent
Dans cet article
Résumé
Procédé pour l'analyse quantitative de la réplication du chromosome de synchronisation est décrit. La méthode utilise l'incorporation de BrdU en combinaison avec fluorescent In situ Hybridation (FISH) afin d'évaluer le calendrier de réplication des chromosomes de mammifères. Cette technique permet la comparaison directe des chromosomes réarrangés et non réarrangé dans la même cellule.
Résumé
Réplication de l'ADN des mammifères initie à plusieurs endroits le long des chromosomes à différents moments au cours de la phase S, la suite d'un programme de réplication temporelle. La spécification de la synchronisation de réplication est considéré comme un processus dynamique régulée par des signaux spécifiques de tissus et de développement qui répondent aux modifications épigénétiques. Cependant, les mécanismes de régulation où et quand réplication de l'ADN initie le long des chromosomes reste mal comprise. Chromosomes homologues généralement se répliquer de manière synchrone, mais il ya des exceptions notables à cette règle. Par exemple, dans les cellules de mammifères femelles l'un des deux chromosomes X devient une réplication tardive par le biais d'un processus appelé inactivation du chromosome X 1. Avec ce retard dans le calendrier de réplication, estimé à 2-3 h, la majorité des gènes transcriptionnellement silencieux deviennent le seul chromosome X. En outre, un discret agissant en cis lieu, connu comme le centre inactivation du chromosome X, réglemente ce processus inactivation du chromosome X, y compris èmee induction de la réplication temps différé sur le chromosome X inactif entier. En outre, certains réarrangements chromosomiques trouvés dans les cellules cancéreuses et les cellules exposées à des rayonnements ionisants afficher un retard important dans le calendrier de la réplication> 3 heures qui affecte le chromosome entier 2,3. Des travaux récents de notre laboratoire indiquent que la perturbation de discret agissant en cis autosomique résultat loci dans un phénotype extrêmement réplication tardive qui affecte l'ensemble du chromosome 4. Études 'ingénierie chromosomique »supplémentaires indiquent que les réarrangements chromosomiques affectant certains nombre de résultats différents chromosomes anormal dans ce moment-phénotype de réplication, ce qui suggère que tous les chromosomes de mammifères contiennent discrets agissant en cis locus qui contrôlent le calendrier de réplication des chromosomes individuels 5.
Ici, nous présentons une méthode pour l'analyse quantitative de la synchronisation de réplication chromosomique associée à hybridation fluorescente in situ. Cetteméthode permet une comparaison directe de la synchronisation de la réplication entre chromosomes homologues au sein de la même cellule, et a été adapté à partir de 6. En outre, cette méthode permet l'identification sans ambiguïté des réarrangements chromosomiques qui sont en corrélation avec l'évolution du calendrier de réplication qui influent sur le chromosome entier. Cette méthode présente des avantages par rapport élaboré récemment débit micro-array haut ou le séquençage protocoles qui ne peuvent distinguer entre les allèles homologues présents sur réorganisées et non réorganisées-chromosomes. En outre, parce que la méthode décrite ici évalue cellules individuelles, il peut détecter des changements dans le calendrier sur la réplication des chromosomes réarrangements chromosomiques qui sont présents dans seulement une fraction des cellules dans une population.
Protocole
1. L'incorporation de BrdU (repérage des bornes)
- Cellules de la plaque à environ 70% de confluence dans une culture de tissu plat 150 mm 24 h avant l'addition de BrdU.
- Remplacez le support avec des produits frais milieu complet contenant 20 ug / ml de BrdU (Sigma) à des moments appropriés avant la récolte. La longueur de temps sont les cellules cultivées dans des milieux avec BrdU varient selon le type cellulaire et des espèces, généralement la phase G2 dure entre 2 et 5 heures (figure 1).
2. Récolte chromosomique des cultures monocouches de cellules
- Retirer des milieux de culture à partir de la plaque et reçois 10 ml dans un tube de centrifugeuse de 15 ml conique. Jeter les feuilles restantes.
- Rincer les cellules une fois avec 10 ml Versine [ou HBSS (Sigma)].
- Ajouter 5 ml de trypsine 0,25% dans Versine (ou HBSS) à la plaque. Incuber à la température ambiante jusqu'à ce que les cellules sont détachées de la plaque.
- Ajouter suspension de cellules trypsinisées dans le tube contenant 10 ml de médias de l'étape 2.1.
- Centrifuger à 400 xg pendant 10 min pour sédimenter les cellules. Aspirer le surnageant en laissant 0,5 ml de milieu ainsi que le culot cellulaire.
- Remettre en suspension les cellules à fond par pipetage avec une pipette Pasteur.
- Ajouter 3 gouttes de solution hypotonique (0,075 M de KCl (Sigma) chauffé à 37 ° C) et mélanger la suspension cellulaire avec une pipette Pasteur. Ajouter 0,5 ml de solution hypotonique et mélanger à nouveau avec une pipette Pasteur. Amener le volume de solution hypotonique à 5 ml et mélanger de nouveau. Incuber à 37 ° C pendant 20-45 min, en fonction du type cellulaire.
- Centrifuger à 400 xg pendant 10 min. Aspirer le surnageant en laissant 0,5 ml de milieu ainsi que le culot cellulaire.
- Reprendre le culot cellulaire en effleurant doucement le tube. Les cellules osmotiquement gonflées sera fragile à ce stade, donc il faut prendre soin de ne pas perturber les membranes cytoplasmiques.
- Ajouter 3 gouttes de fixateur de Carnoy (acide acétique glacial 01h03: Le méthanol); mélanger doucement par pipetage avec un pipett Pasteure. Ajouter 0,5 ml de fixateur et mélanger en pipetant doucement avec une pipette Pasteur. Amener le volume de fixateur à 5 ml et mélanger à nouveau en pipetant doucement avec une pipette Pasteur. Les cellules fixées peuvent être stockés dans l'obscurité à -20 ° C pendant plusieurs mois.
- Centrifuger et aspirer comme à l'étape 2.8.
- Remettre en suspension le culot cellulaire dans du fixateur frais; estimer le volume de la pastille et ajouter ~ 10x le volume de fixateur de Carnoy.
- Ajouter la suspension de cellules goutte à goutte sur des lames de microscope humides, glacées; maintenir la lame à un angle de 45 ° ~ et permettre la suspension de cellules à s'écouler sur la surface de la lame. Posez la lame à plat sur du papier absorbant et les inondations la diapositive avec un fixateur, laisser sécher. Vérifiez diapositives de la présence d'écarts mitotiques en utilisant un microscope inversé.
3. Traitement RNase et déshydratation de l'éthanol
- Lieu 200 ul de 10 pg / ml de RNase A (Sigma) dans 2x SSC à chaque diapositive; incuber à 37 ans & deg; C pendant 1 heure.
- Laver les lames 3 fois avec 2x SSC, pH 7,0 à température ambiante pendant 3 min chacun.
- Déshydrater les lames grâce à un mélange EtOH (Sigma) série (70%, 90% et 100%) à la température ambiante pendant 3 min chacun.
- Sécher les lames d'air à température ambiante.
4. Préparation de cocktails de sonde pour BAC + chromosomique spécifique énumération centromère Probe (CEP) ou pour les peintures chromosomiques entiers Hybridation in situ
- Pour l'hybridation BAC / CEP simultanée, préparer deux cocktails sondes séparées pour les pré-hybridation. Nous avons trouvé que la pré-hybridation des sondes d'alcoolémie et le SCEP séparément dans les résultats stringences différentes en moins d'arrière-plan et les intensités de signaux supérieures. Nous avons également constaté que fosmides individuels peuvent également être utilisés à la place de clones BAC. La formulation cocktail décrit ci-dessous représente les volumes pour une seule diapositive. Si le traitement de plusieurs diapositives en utilisant les mêmes sondes simplement augmenter le volume appropr iately.
* BAC sonde d'ADN cocktail:
11,5 ul de tampon d'hybridation (formamide 50% (Sigma) / 2x SSC / Dextran Sulfate (Sigma)) 2 ul dH 2 O
2,5 pi BAC/Cot1 ADN dans dH 2 O (voir ci-dessous pour l'étiquetage protocole) un volume de 15 ul au total
* CEP sonde cocktail:
7 pi de tampon d'hybridation CEP (65% Formamide/2x SSC / Dextran Sulfate) 2 ul dH 2 O
D'ADN de 1 ul CEP/Cot1 (acheté préalablement marqué avec Spectrum Orange/Cy3/Texas Rouge) 10 ul de volume total de
* Après dénaturation et de pré-hybridation (étape 5.1) mélange BAC sonde avec le CEP sonde à un 3:2 ratio de 25 cocktails sonde ul / slide et mélanger.
OU
- Aliquote de 20 ul / diapositive de la sonde chromosomique Peinture entier (Cy3/Texas rouge) dans un tube. Passez à la dénaturation et de pré-hybridation étape.
- Dénaturer sonde cocktail (s) à 75 ° C pendant 10 min. Pré-hybridation à 37 ° C pendant 30 min pour permettre l'ADN Cot1 de s'hybrider à des séquences répétitives et d'empêcher l'hybridation subséquente de chromosomes en métaphase.
- Diapositives dénaturer dans des jarres de Coplin à 70% Formamide/2x SSC, pH 7,0 à 72 ° C pendant 3 min.
- Immédiatement déshydrater diapositives à travers une série EtOH (70%, 90% et 100%) dans des jarres de Coplin à 4 ° C pendant 3 minutes chacun.
- Laisser les lames sécher à l'air à température ambiante.
- Préchauffer les diapositives à 45 ° C sur la lame chaude pour les 10 dernières minutes de la sonde pré-hybridzation (étape 5.1). Mélanger cocktails sonde pré-hybridés si nécessaire et ajouter 25 ul de chaque diapositive, placez doucement une lamelle sur chaque diapositive et joint le long de tous les bords avec la colle de caoutchouc. Incuber une nuit à 37 ° C dans une chambre humidifiée pour éviter l'évaporation.
6. Hybridation Message Lave
- Laver les lames dans des jarres de Coplin 3 fois dans 50% formamide/2x SSC, pH 7,0 à 38-40 ° C pendant 3 minutes chacun. La température des lavages peut varier entre sondes. S'il ya hybridation de fond élevé, vous pouvez augmenter la température des lavages. A l'inverse, si le signal est faible et il n'y a pas d'arrière-plan, vous pouvez diminuer la température de ces lavages.
- Laver les lames 1 fois dans du tampon de PN à la température ambiante pendant 3 min, puis passez à l'étape de détection du BrdU.
7. Détection BrdU
- Bloc glisse avec un tampon PNM pendant 10 min (200 pi / diapositive) à la température ambiante dans l'obscurité.
- Égoutter les lames en les tournant sur chant sur une serviette en papier. Ajouter 100 ul d'anti-BrdU-FITC (50 pg / ml Roche ou Millipore) dans un tampon PNM pendant 30 min à 37 ° C.
- Laver les lames 3 fois dans le tampon de PN à température ambiante pendant 3 min chacun.
- Drainerexcès de tampon de PN sur diapositives une à la fois en tournant sur chant sur une serviette en papier. Ajouter 20 ul DAPI / antifade montage solution (Invitrogen). Recouvrir la lame avec une serviette en papier et appuyez sur la lamelle pour forcer les bulles d'air et l'excès de solution de montage. Procéder à l'analyse d'image.
8. Capture d'images et de quantifier l'incorporation de BrdU
- Les images sont capturées avec un appareil Olympus BX61 microscope à fluorescence et Olympus Caméra CCD, objectif 100x, filtre automatique-roue et logiciels Cytovision (Applied Imaging). BrdU est capturé à l'aide d'un filtre FITC, les peintures chromosomiques (Texas Red, métasystèmes ou Cytocell), BAC (Cy3) et CEP (Vysis Spectrum Orange) sondes sont capturées à l'aide d'un Texas Red ou Cy3 filtre DAPI est capturée avec un filtre DAPI (voir Les figures 2 et 3).
- Chromosomes individuels d'intérêt sont identifiés avec le BAC / CEP ou sondes chromosomiques spécifiques de peinture (figures 2B et 3B). Utilisantle logiciel Cytovision, chaque chromosome d'intérêt est "découpé" en métaphase de la propagation dans l'ensemble, une ligne est tracée à travers le long de la longueur du chromosome de bras court à bras long (figures 2C et 3C). Le logiciel Cytovision est utilisé pour quantifier le profil d'intensité de pixel le long de la longueur de chaque chromosome. Le logiciel calcule Cytovision la zone occupée par les pixels et quantifie l'intensité des pixels représentés par les signaux de BrdU ou DAPI de chaque chromosome isolé. L'intensité moyenne des pixels représentés par chaque chromosome est alors multiplié par la surface occupée par les pixels pour obtenir le nombre total de pixels (tableau 1 et figure 4). En outre, ce protocole permet la visualisation des dernières régions réplication des chromosomes. Par conséquent, le motif à bandes d'incorporation de BrdU permet la détection des régions activement réplication des chromosomes, et les différences dans le calendrier de réplication entre les chromosertains paires sont considérées comme des différences dans ce modèle de bandes (figures 2 et 3). Enfin, ces différences dans les modes de liaison peuvent être comparées à des cartes connues de synchronisation de réplication pour chaque chromosome 6,7, ce qui permet une estimation de la différence de temps de réplication entre les chromosomes homologues ou des bras chromosomiques 4. Par exemple, le chromosome 6 les deux sur la figure 3A afficher une différence dans des bandes de motif> 2 heures par rapport à la normale de synchronisation de réplication du chromosome 6 4.
En outre, une procédure similaire à celle décrite ici a été utilisée avec succès pour l'étude de la chronologie de réplication asynchrone entre les chromosomes X actifs et inactifs 8,9. Les chromosomes X actifs et inactifs ont été identifiés en utilisant des peintures du chromosome X. Les images ont été acquises à l'aide du logiciel Quips mFISH (Vysis) et le nombre de pixels occupés par les chromosomes X et le nombre de pixels occupés par fluorescence labeled BrdU ont ensuite été calculées à l'aide NIH Image ( http://rsb.info.nih.gov/nih-image/index.html ).
9. Nick Traduction ADN de BAC pour le marquage fluorescent (Vysis)
- Ajouter le mélange refroidi sonde suivante pour un tube de centrifugeuse:
70 pi (4 pg d'ADN) dans dH 2 O
10 pl 0.2 mM dUTP-orange ou vert
20 pl 0.1 mM dTTP
20 ul de tampon 10x Traduction Nick
40 ul de dNTP NT (moins dTTP)
40 ul enzyme Traduction nick
Volume de 200 ul totale
Incuber à 16 ° CO / N. Arrêter la réaction par chauffage à 70 ° C pendant 10 min. Refroidir sur glace pendant 5 min. - Précipiter l'ADN avec de l'éthanol en ajoutant ce qui suit au mélange de sonde:
Solution 200 ul de sonde
48 ul de NaOAc 3 M
160 ul Cot1DNA (.25 g / ul)
1200 EtOH ul 100% - Conserver à -80 ° C pendant 10 min pour O / N.
- I Spinn froide à 12.000 xg pendant 20 min. Décanter EtOH. Laver 1 fois avec de l'EtOH 70%. Faites tourner comme avant. Décanter EtOH. Sécher à l'air jusqu'à ce que s'évapore EtOH. Remettre en suspension dans 40 ul dH 2 O pour une concentration finale de 100 ng / pl d'ADN de BAC.
Résultats
Un exemple de l'analyse de la synchronisation de réplication du chromosome 6 humain est montré dans la figure 2. Des cellules contenant une délétion du gène ASAR6 4, situé à 6q16.1, ont été exposés à BrdU pendant 5 heures, la récolte des cellules mitotiques et traitées de FISH avec une sonde de peinture chromosomique 6 (Vysis) et pour l'incorporation de BrdU. Notez qu'il ya une différence significative dans le modèle de BrdU bandes entre le deux 6, ce qui correspond à un retard dans le calendrier de réplication de> 2 heures pour l'un des chromosome 6 [voir 4 pour le moment la réplication de baguage modèle du chromosome 6 avant la suppression]. En outre, il existe une différence significative dans le montant total de l'incorporation de BrdU (pixels) par rapport à une analyse similaire de la coloration DAPI (figure 2D).
Un autre exemple de l'analyse de la synchronisation de réplication du chromosome 6 humain est montré dans la figure 3. Les cellules contenant le samsuppression électronique du gène ASAR6 4 représenté figure 2, si ont été exposés à la BrdU pendant 5 heures, la récolte des cellules mitotiques et traitées pour FISH avec une sonde chromosome 6 CEP plus un BAC contenant le gène ASAR6 et pour l'incorporation de BrdU. Notez que le chromosome supprimé 6 (Δ6) affiche plus d'incorporation de BrdU et un modèle plus étendu des bandes de l'incorporation de BrdU que le chromosome non supprimé 6. Spreads de 7 mitotiques des cellules différentes ont été traités comme dans la figure 3 et les profils de pixels à la fois pour DAPI et BrdU sont présentés dans le tableau 1.

Figure 1. Schéma de BrdU étiquette du terminal. Une phase S typique dans des cellules de mammifères derniers pour 8 à 10 h, et G2 est typiquement de 2 à 5 h. BrdU est ajouté pour augmenter les périodes de temps (flèches vertes) pour étiqueter les dernières portions de chromosomes à répliquer. The chromosomes dans les cellules de mammifères répliquer selon un programme temporel, avec réplication précoce et tardive se produisant au début et à la fin de la phase S, respectivement (courbe noire). Chromosomes X inactifs sont retardés dans le calendrier de réplication avec la majorité de la synthèse de l'ADN survenant au cours de la seconde moitié de la phase S (ligne bleue). Chromosomes avec une synchronisation de réplication différée sont retardés dans l'initiation et la réalisation de la synthèse de l'ADN, avec la réplication active continue par G2 (ligne rouge). Cultures asynchrones sont récoltées pour les cellules en mitose et traitées pour l'incorporation de BrdU et FISH.
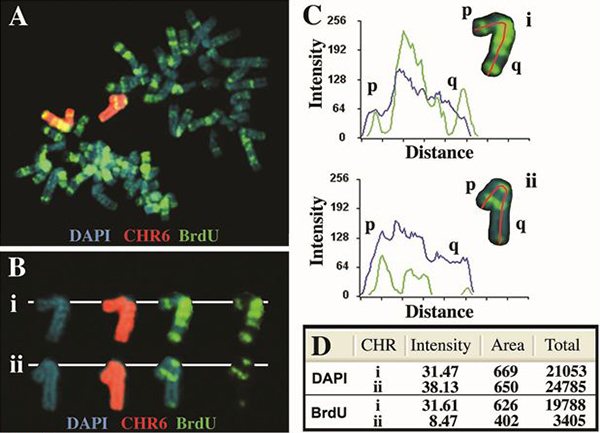
Figure 2. La réplication asynchrone du chromosome humain 6. Les cellules contenant une délétion du gène d'ingénierie ASAR6 4 ont été traités avec du BrdU pendant 5 heures, la récolte des cellules mitotiques et traitées pour l'incorporation de BrdU et FISH en utilisant un chrom OSOME 6 de la peinture en tant que sonde. L'ADN a été colorées avec du DAPI (bleu). A) Un écart mitotique contenant un typique "bandes" modèle de l'incorporation de BrdU (vert) est affiché. La sonde peinture chromosome 6 hybridé à deux chromosome 6 (rouge) dans cette cellule. B) Le chromosome deux 6 (i et ii) ont été "découpé" et affichée avec les trois marqueurs fluorescents séparés en images distinctes. C) Le chromosome 6 ont été analysées en utilisant le logiciel Cytovision et les profils d'intensité de signal pour les deux DAPI (bleu) et BrdU (vert) sont affichés. La ligne rouge indique le chemin d'accès utilisé, à partir du bras court (p) pour le bras long (q) pour la quantification de fois BrdU et DAPI. La distance correspond à la durée de chaque chromosome de bras court (p) pour le bras long (q) en pixels. D) Quantification du signal total pour les deux DAPI et BrdU fluorescence. Le total des valeurs d'intensité de pixel représentent la moyenne multipliée par la surface représentée par les pixels.
igure 3 "src =" / files/ftp_upload/4400/4400fig3.jpg "/>
La figure 3. Différé réplication du chromosome humain 6 contenant une délétion d'ASAR6. Les cellules contenant une délétion du gène d'ingénierie ASAR6 4 ont été traités avec du BrdU pendant 5 heures, la récolte des cellules mitotiques et traitées pour l'incorporation de BrdU et FISH en utilisant un chromosome 6 CEP plus un BAC contenant le gène ASAR6 comme sondes. L'ADN a été colorées avec du DAPI (bleu). A) Un écart mitotique contenant un typique "bandes" modèle de l'incorporation de BrdU (vert) est affiché. Le chromosome 6 CEP sonde hybridée à deux chromosome 6 de (centromérique grand signal rouge), et le BAC hybridé à un seul chromosome 6 (petit signal rouge sur le bras long) dans cette cellule. Notez la différence dans la structure de bandes BrdU entre les deux 6. B) Le chromosome supprimé 6 est représenté par la Δ6 et 6 non-supprimée par 6. Le 6 deux ont été "découpé" et affichée avec les trois marqueurs fluorescents séparés en images distinctes. C) Til chromosome 6 ont été analysées en utilisant le logiciel Cytovision et les profils d'intensité de signal pour les deux DAPI (bleu) et BrdU (vert) sont affichés. La ligne rouge indique le chemin d'accès utilisé, à partir du bras court (p) pour le bras long (q) pour la quantification de fois BrdU et DAPI. La distance correspond à la durée de chaque chromosome de bras court (p) pour le bras long (q) en pixels. D) Quantification du signal total pour les deux DAPI et BrdU fluorescence. Le total des valeurs d'intensité de pixel représentent la moyenne multipliée par la surface représentée par les pixels.
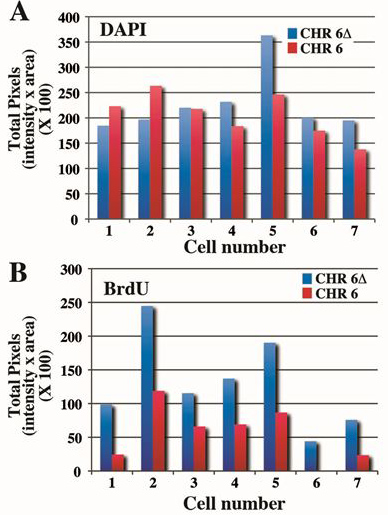
Figure 4. Quantification de la différence de temps entre la réplication du chromosome 6. Les cellules contenant une délétion du gène d'ingénierie ASAR6 4 ont été traités avec du BrdU pendant 5 heures, la récolte des cellules mitotiques et traitées pour l'incorporation de BrdU et FISH en utilisant un chromosome 6 CEP en plus d'une sondeBAC contenant le gène ASAR6 comme sonde. Spreads mitotiques ont été traités comme dans la figure 3 et les valeurs de 7 cellules différentes sont représentées (voir tableau 1). A) La coloration au DAPI est quantifiée, et le nombre total de pixels est affichée pour chaque cellule. Notez qu'il n'y a qu'une différence de ~ 10-20% entre les chromosomes au sein de la même cellule. B) L'incorporation de BrdU a été quantifié à partir des mêmes chromosomes que le panel B ci-dessus. Notez qu'il n'y a> 2 fois la différence entre les valeurs de pixels au total pour le chromosome supprimés et non supprimés 6 de.
Discussion
La préparation des étalements de chromosomes est une étape cruciale pour la réussite de la réplication de synchronisation essai décrit ici. L'inclusion d'une étape de prétraitement colcémide avant le traitement hypotonique peut aider à la fréquence et la propagation des cellules mitotiques. En général, nous exposer les cellules à colcemdi pendant 1-3 heures avant la récolte, et l'utilisation colcémide à une concentration finale de 10 ug / ml. Toutefois, l'inclusion d'une étape de prétraitement colcémide peut modifier la longueur de G2 et par conséquent peut modifier le calendrier de réplication apparente et l'état de condensation des 3 chromosomes.
Cette procédure peut être appliquée à de nombreux types cellulaires différents et des espèces en faisant varier la durée d'incubation BrdU, qui dépend de la durée du cycle cellulaire. Pour les lignées cellulaires les plus humaines et de souris, la phase G2 est généralement 3-6 heures, par conséquent, les traitements sont généralement BrdU dans cette gamme. Une alternative à la BrdU est le 5-éthynyl-2'deoxyuridine (EDU) et sa détection ultérieure à l'aideun azoture fluorescent et "click chemistry" réaction 10. Le système de détection EdU a plusieurs avantages sur la méthode de détection du BrdU. Par exemple, la détection de EdU ne nécessite pas de fixation échantillon ou dénaturation de l'ADN. Ainsi, l'utilisation de EdU du calendrier de réplication ânes peuvent être combinés avec de simples bandes G techniques au lieu de FISH pour identifier les chromosomes d'intérêt.
Le protocole de synchronisation de réplication décrit ici est spécifiquement conçu pour doser la fin de la phase S de synthèse d'ADN ainsi que tout s'étendant dans G2. En outre, réplication de l'ADN peuvent être suivis tout au long de la phase S en utilisant cette procédure à l'aide de courtes (15-30 min) des impulsions de BrdU suivies par des périodes relativement longues chase de 6-10 h. Cela permet la visualisation de l'incorporation de BrdU à la fois début et au milieu de la phase S. Par exemple, certaines tumeurs dérivées réarrangements chromosomiques sont décalées à la fois dans l'initiation et l'achèvement de la synthèse de l'ADN sur toute la longueur des chromosomes 3 .
Un avantage de cette procédure est que le calendrier de réplication de synchronisation de réplication qu'elle essais dans des cellules individuelles. Par conséquent, il a la capacité de détecter des différences dans le calendrier de réplication entre les chromosomes homologues contenues dans la même cellule. Bien qu'il existe d'autres procédures, par exemple. calendrier de réplication hybridation in situ (ReTiSH, 11), qui ont la capacité de détecter des différences dans le calendrier de réplication entre allèles à des loci spécifiques sur les chromosomes homologues, la procédure décrite ici peut détecter des différences dans le calendrier de réplication sur toute la longueur des chromosomes. En outre, cette procédure peut différences de dosage dans le calendrier de réplication des chromosomes qui sont présents dans seulement une fraction des cellules d'une population 3. Par exemple de nombreuses lignées cellulaires cancéreuses et tumorales primaires échantillons contiennent des réarrangements chromosomiques qui sont présents dans moins de 50% des cellules. Nous sommes en train d'utiliser cette procédure pour les chromosomes d'essaidans des échantillons de tumeurs primaires, et ont été en mesure de détecter la réplication asynchrone entre les chromosomes dans plusieurs échantillons. Toutefois, étant donné que les échantillons de tumeurs primaires ont un nombre limité de figures mitotiques, environ un tiers des cultures primaires omis de donner un nombre suffisant d'écarts mitotiques.
Un autre avantage de cette procédure a plus de microarray ou séquençage tests basés sur, c'est que les chromosomes individuels sont analysés plutôt que l'ADN immunoprécipité à partir de pools de cellules. Dans les dosages immuno-précipitation à base de polymorphismes doivent être identifiés et liés à des allèles spécifiques afin de distinguer le calendrier de réplication entre les allèles.
En outre, avec la reconnaissance du fait que de nombreuses cellules cancéreuses contiennent de nombreux réarrangements chromosomiques 12, et l'observation que le stress réplication de l'ADN est associé à une instabilité génomique dans 13 cellules cancéreuses, nous pensons que ce protocole est un f outil utile et simpleou l'analyse de routine de la synchronisation de réplication des chromosomes dans les cellules cancéreuses.
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été soutenu par une subvention de l'Institut national du cancer, CA131967.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires (optionnel) |
| Anti-BrdU-FITC | Roche Millipore | 11202593001 MAB326F | 50 ug / ul |
| Nick Translation Kit | Abbott Molecular (Vysis) | 07J00-0001 | |
| Spectrum Orange dUTP | Abbott Molecular (Vysis) | 02N33-050 | |
| CEP | Abbott Molecular (Vysis) | Varie | |
| Tampon d'hybridation INS / PCM | Abbott Molecular (Vysis) | 06J67-011 | |
| Tampon d'hybridation CEP | Abbott Molecular (Vysis) | 07J36-001 | |
| Chromosome paints | Métasystèmes Groupe | D-14NN-050-TR | |
| Olympus BX61 microscope à fluorescence | Olympe | BX61TRF-1-5 | |
| Système logiciel microscope d'imagerie | Applied Imaging | Cytovision 3.93.1 | |
| Appareil photo numérique | Olympe | UCMAD3 | |
EN RECETTES hybridation in situ 35 ml formamide * (Sigma) * Il est important d'utiliser le formamide qui a été stocké à -20 ° C. Stockage prolongé température ambiante va générer l'acide formique et le pH wmal-être trop bas. 50% Formamide/2x SSC 25 ml de formamide (Sigma) 20x SSC, 4 L 702 g de NaCl (Sigma) PN tampon [0,1 M NaP0 4 0,1% NP_40 (Sigma)] Préparer une solution à 0,1 M de phosphate de sodium chacun (Filtre stériliser et stocker en aliquots ml 500). 0,1 M de NaH 2 P0 4, 1 L 13,8 g de NaH 2 P0 4 (Sigma): 0,1 M de NaH 2 P0 4 1 L 14,2 g de NaH 2 P0 4 (Sigma) PN: Ajuster le pH de 0,1 M Na 2 HP0 4 à pH 8,0 avec 0,1 M NaH 2 P0 4. Stériliser par filtration et ajouter 1 ml de NP-40. PNM 50 ml 1,25 g Non lait écrémé en poudre (Sigma) Mélanger pendant 15-20 min en remuant constamment. Faites tourner 2 fois à 400 xg pendant 10 min. Utiliser le surnageant, et assurez-vous de ne pas déranger les protéines du lait précipités. |
Références
- Payer, B., Lee, J. T. X chromosome dosage compensation: how mammals keep the balance. Annu. Rev. Genet. 42, 733-772 (2008).
- Breger, K. S., Smith, L., Turker, M. S., Thayer, M. J. Ionizing radiation induces frequent translocations with delayed replication and condensation. Cancer Research. 64, 8231-8238 (2004).
- Smith, L., Plug, A., Thayer, M. Delayed Replication Timing Leads to Delayed Mitotic Chromosome Condensation and Chromosomal Instability of Chromosome Translocations. Proc. Natl. Acad. Sci. U.S.A. 98, 13300-13305 (2001).
- Stoffregen, E. P., Donley, N., Stauffer, D., Smith, L., Thayer, M. J. An autosomal locus that controls chromosome-wide replication timing and mono-allelicexpression. Hum. Mol. Genet. 20, 2366-2378 (2011).
- Breger, K. S., Smith, L., Thayer, M. J. Engineering translocations with delayed replication: evidence for cis control of chromosome replication timing. Hum. Mol. Genet. 14, 2813-2827 (2005).
- Camargo, M., Cervenka, J. Patterns of DNA replication of human chromosomes. II. Replication map and replication model. Am. J. Hum. Genet. 34, 757-780 (1982).
- Cohen, S. M., Cobb, E. R., Cordeiro-Stone, M., Kaufman, D. G. Identification of chromosomal bands replicating early in the S phase of normal human fibroblasts. Exp. Cell Res. 245 (98), 321-329 (1998).
- Diaz-Perez, S., et al. The element(s) at the nontranscribed Xist locus of the active X chromosome controls chromosomal replication timing in the mouse. Genetics. 171, 663-672 (2005).
- Diaz-Perez, S. V., et al. A deletion at the mouse Xist gene exposes trans-effects that alter the heterochromatin of the inactive X chromosome and the replication time and DNA stability of both X chromosomes. Genetics. 174, 1115-1133 (2006).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc. Natl. Acad. Sci. U.S.A. 105, 2415-2420 (2008).
- Schlesinger, S., Selig, S., Bergman, Y., Cedar, H. Allelic inactivation of rDNA loci. Genes Dev. 23, 2437-2447 (2009).
- Mitelman Database of Chromosome Aberrations in Cancer. , Available from: http://cgap.nci.nih.gov/Chromosomes/Mitelman (2006).
- Branzei, D., Foiani, M. The checkpoint response to replication stress. DNA Repair (Amst). 8, 1038-1046 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon