Method Article
Cromosoma Timing Replica combinazione con fluorescente
In questo articolo
Riepilogo
Un metodo per l'analisi quantitativa di temporizzazione cromosoma replica è descritto. Il metodo utilizza incorporazione di BrdU in combinazione con fluorescente In situ Ibridazione (FISH) per valutare la tempistica replicazione dei cromosomi di mammifero. Questa tecnica permette il confronto diretto dei cromosomi riarrangiati e un-riarrangiato all'interno della stessa cella.
Abstract
Replicazione del DNA di mammifero avvia in siti multipli lungo cromosomi in momenti diversi durante la fase S, seguendo un programma di replica temporale. La specifica dei tempi di replica è pensato per essere un processo dinamico regolato da stimoli tessuto-specifiche e di sviluppo che sono sensibili alle modificazioni epigenetiche. Tuttavia, i meccanismi che regolano dove e quando la replicazione del DNA inizia lungo cromosomi rimane poco chiaro. Cromosomi omologhi di solito replicare in modo sincrono, ma ci sono eccezioni a questa regola. Per esempio, in cellule di mammifero femmina uno dei due cromosomi X diventa tardiva replica attraverso un processo noto come inattivazione X 1. Insieme a questo ritardo nella temporizzazione replica, stimato a 2-3 ore, la maggior parte dei geni diventano trascrizionalmente tacere su un cromosoma X. In aggiunta, una discreta azione cis locus, noto come il centro inattivazione X, regola questo processo di inattivazione X, compresi thinduzione e dei tempi di replica ritardata sul cromosoma X inattivo intero. Inoltre, alcuni riarrangiamenti trovano nelle cellule tumorali e cellule esposte a radiazioni ionizzanti visualizzare un significativo ritardo nella temporizzazione replica di> 3 ore che interessa l'intero cromosoma 2,3. Un recente lavoro del nostro laboratorio indicano che l'alterazione di discreta azione cis autosomica risultato loci in un fenotipo estremamente tardiva replica che interessa l'intero cromosoma 4. Ulteriori studi 'di ingegneria cromosomica' indicano che alcuni riarrangiamenti cromosomici che interessano molti risultati diversi cromosomi in questa anomala replica-timing fenotipo, suggerendo che tutti i cromosomi di mammifero contengono discrete cis-che agiscono loci che controllano tempistica corretta replicazione dei cromosomi individuali 5.
Qui, presentiamo un metodo per l'analisi quantitativa di temporizzazione cromosoma replica combinato con ibridazione in situ fluorescente. Questometodo consente un confronto diretto di temporizzazione replica tra cromosomi omologhi all'interno della stessa cella, ed è stato adattato da 6. Inoltre, questo metodo consente l'identificazione univoca di riarrangiamenti cromosomici che correlano con modifiche nei tempi di replica che interessano l'intero cromosoma. Questo metodo ha dei vantaggi rispetto recentemente sviluppato un elevato throughput di micro-array o sequenza protocolli che non è possibile distinguere tra alleli omologhi presenti sul riarrangiati e poco risistemato cromosomi. Inoltre, poiché il metodo descritto qui valuta singole cellule, si può rilevare variazioni di temporizzazione sulla replicazione cromosomica riarrangiamenti cromosomici che sono presenti in una frazione di cellule in una popolazione.
Protocollo
1. Incorporazione di BrdU (Etichettatura Terminal)
- Cellule piastra per circa il 70% di confluenza di 150 mm in coltura tissutale piatto 24 ore prima dell'aggiunta di BrdU.
- Sostituire il supporto con nuovi mezzi completi contenenti 20 mg / ml di BrdU (Sigma) ad intervalli di tempo appropriati prima del raccolto. La lunghezza del tempo cellule vengono coltivate in mezzi con BrdU varierà con il tipo di cella e specie, tipicamente la fase G2 dura tra 2 e 5 ore (Figura 1).
2. Harvest Cromosoma di colture monostrato Celle
- Rimuovere terreni di coltura dalla piastra e raccogliere 10 ml in una provetta da centrifuga 15 ml. Gettare materiale rimanente.
- Risciacquare le cellule una volta con 10 ml Versine [o HBSS (Sigma)].
- Aggiungere 5 ml di tripsina 0,25% in Versine (o HBSS) alla piastra. Incubare a temperatura ambiente fino a quando le cellule vengono staccate dalla piastra.
- Aggiungi sospensione tripsinizzate cella a tubo contenente i 10 ml di mezzi di comunicazione a partire dal punto 2.1.
- Centrifugare a 400 xg per 10 minuti per ottenere il pellet. Aspirare il surnatante, lasciando 0,5 ml di supporti più il pellet di cellule.
- Risospendere le cellule a fondo pipettando con una pipetta Pasteur.
- Aggiungere 3 gocce di soluzione ipotonica (0,075 M KCl (Sigma) riscaldato a 37 ° C); miscelare la sospensione cellulare con una pipetta Pasteur. Aggiungere 0,5 ml di soluzione ipotonica e miscelare di nuovo con una pipetta Pasteur. Portare il volume di soluzione ipotonica a 5 ml e mescolare nuovamente. Incubare a 37 ° C per 20-45 minuti, a seconda del tipo di cellula.
- Centrifugare a 400 xg per 10 min. Aspirare il surnatante, lasciando 0,5 ml di supporti più il pellet di cellule.
- Risospendere il pellet di cellule delicatamente muovendo il tubo. Le cellule osmoticamente gonfie sarà fragile, a questo punto, per cui occorre prestare attenzione a non disturbare le membrane citoplasmatiche.
- Aggiungere 3 gocce di fissativo di Carnoy (1:3 acido acetico glaciale: Metanolo); mescolare delicatamente con una pipetta Pasteur pipette. Aggiungere 0,5 ml di fissativo e mescolare pipettando gentilmente con una pipetta Pasteur. Portare il volume di fissativo a 5 ml e mescolare ancora pipettando gentilmente con una pipetta Pasteur. Cellule fisse possono essere conservate al buio a -20 ° C per diversi mesi.
- Centrifugare e aspirare, come al punto 2.8.
- Risospendere il pellet cellulare in fissativo fresco; stimare il volume del pellet e aggiungere ~ 10 volte il volume del fissativo di Carnoy.
- Aggiungere la sospensione cellulare e goccia a goccia sul bagnato, gelide vetrini da microscopio; tenere il vetrino a un angolo di ~ 45 ° e consentire la sospensione cellulare a scorrere sulla superficie del vetrino. Appoggiare il vetrino piatto su carta assorbente e inondare il vetrino con fissativo e lasciare asciugare all'aria. Controllare vetrini per la presenza di spread mitotiche utilizzando un microscopio invertito.
3. RNasi trattamento e disidratazione etanolo
- Posto 200 pl di 10 pg / ml RNasi A (Sigma) in 2x SSC a ciascun vetrino; incubare a 37 & deg; C per 1 ora.
- Lavare i vetrini 3 volte con 2x SSC, 7,0 pH a temperatura ambiente per 3 min ciascuna.
- Disidratare diapositive attraverso un (Sigma) serie EtOH (70%, 90% e 100%) a temperatura ambiente per 3 min ciascuna.
- Far asciugare i vetrini a temperatura ambiente.
4. Preparazione di cocktail di misura per BAC Inoltre cromosomica specifica Centromero Enumerazione Probe (CEP) o per le vernici intero cromosoma ibridazione in situ
- Per la BAC / CEP ibridazione simultanea, preparare due cocktail sonda separata per la pre-ibridazione. Abbiamo trovato che la pre-ibridazione le sonde BAC e CEP separatamente in diversi risultati stringencies in meno background e intensità di segnale maggiore. Abbiamo anche trovato che Fosmids individuali possono anche essere utilizzati al posto di cloni BAC. La formulazione cocktail descritto di seguito rappresenta i volumi per una singola diapositiva. Se l'elaborazione più diapositive utilizzando le sonde stesse semplicemente aumentare la appropr volumi iately.
* BAC DNA della sonda cocktail:
11,5 microlitri tampone di ibridazione (50% formammide (Sigma) / 2x SSC / destrano solfato (Sigma)) 2 microlitri dH 2 O
2,5 microlitri BAC/Cot1 DNA in dH 2 O (vedi sotto per l'etichettatura di protocollo) volume di 15 microlitri totale
* CEP sonda cocktail:
7 CEP microlitri tampone di ibridazione (65% Formamide/2x SSC / destrano solfato) 2 microlitri dH 2 O
1 DNA microlitri CEP/Cot1 (acquistato prelabeled con Spectrum Orange/Cy3/Texas Rosso) 10 microlitri di volume totale
* Dopo la denaturazione e pre-ibridazione (punto 5.1) mix BAC sonda con CEP sonda ad un rapporto 3:2 25 cocktail sonda pl / diapositive e mescolare.
O
- Aliquotare 20 pl / slide di sonda tutto vernice Cromosoma (Cy3/Texas rosso) in una provetta. Procedere alla denaturazione e pre-ibridazione passo.
- Denaturare sonda cocktail (s) a 75 ° C per 10 min. Pre-ibridizzano a 37 ° C per 30 minuti per consentire la Cot1 DNA di ibridarsi a sequenze ripetitive e prevenire ibridazione successiva alla metafase cromosomi.
- Diapositive denaturate in vaschette Coplin nel 70% Formamide/2x SSC, pH 7,0 a 72 ° C per 3 min.
- Immediatamente disidratare diapositive attraverso una serie EtOH (70%, 90% e 100%) in vaschette Coplin a 4 ° C per 3 min ciascuna.
- Lasciare i vetrini per asciugare a temperatura ambiente.
- Pre-riscaldare vetrini a 45 ° C sul vetrino più caldo per gli ultimi 10 minuti della sonda pre-hybridzation (passo 5.1). Mescolare pre-ibridizzati cocktail sonda se necessario, e aggiungere 25 ul per ogni diapositiva, posizionare con cura un coprioggetto su ogni diapositiva e la guarnizione lungo tutti i bordi con mastice. Incubare per una notte a 37 ° C in una camera umidificata per evitare l'evaporazione.
6. Ibridazione Messaggio lava
- Lavare i vetrini in vaschette Coplin 3 volte nel 50% formamide/2x SSC, pH 7,0 a 38-40 ° C per 3 minuti ciascuno. La temperatura dei lavaggi può variare tra sonde. Se c'è ibridazione fondo elevato è possibile aumentare la temperatura dei lavaggi. Viceversa, se il segnale è debole e non c'è sfondo allora si può diminuire la temperatura di questi lavaggi.
- Lavare i vetrini 1 volta in tampone PN a temperatura ambiente per 3 min, e procedere alla fase di rilevamento BrdU.
7. BrdU rilevamento
- Vetrini con tampone di blocco PNM per 10 min (200 pl / vetrino) a temperatura ambiente al buio.
- Scaricare diapositive attivando bordo su un tovagliolo di carta. Aggiungere 100 ml di anti-BrdU-FITC (50 pg / ml Roche o Millipore) in tampone PNM per 30 min a 37 ° C.
- Lavare i vetrini tre volte in tampone PN a temperatura ambiente per 3 min ciascuna.
- Drenareeccesso di tampone PN sulle diapositive uno alla volta ruotando sul bordo su un tovagliolo di carta. Aggiungere 20 microlitri DAPI / antifade soluzione di montaggio (Invitrogen). Coprire il vetrino con un tovagliolo di carta e premere verso il basso sul vetrino per far fuoriuscire le bolle d'aria e l'eccesso di soluzione di montaggio. Procedere con l'analisi delle immagini.
8. Catturare Immagini e Quantificazione incorporazione BrdU
- Le immagini vengono acquisite con un microscopio a fluorescenza Olympus BX61 e Olympus CCD, obiettivo 100x, filtro automatico ruote e software Cytovision (Applied Imaging). BrdU viene catturato con un FITC filtro; vernici cromosomiche (Texas Red, Metasystems o Cytocell), BAC (Cy3) e CEP (Vysis Spectrum Orange) sonde sono state acquisite utilizzando un rosso Texas o Cy3 filtro; DAPI viene catturato con un filtro DAPI (vedi le figure 2 e 3).
- Cromosomi individuali di interesse sono identificati con BAC / CEP o cromosoma sonde specifiche vernici (Figure 2B e 3B). Utilizzandoil software Cytovision, ciascun cromosoma di interesse "cut-out" dalla diffusione metafase nel suo complesso, una linea disegnata attraverso il lungo la lunghezza del cromosoma dal braccio corto a braccio lungo (Figure 2C e 3C). Il software Cytovision viene utilizzato per quantificare il profilo di intensità di pixel lungo la lunghezza di ciascun cromosoma. Il software Cytovision calcola l'area occupata dai pixel e quantifica l'intensità dei pixel rappresentate dai segnali BrdU o DAPI di ciascun cromosoma isolato. L'intensità dei pixel media rappresentata da ciascun cromosoma viene quindi moltiplicata per l'area occupata da quei pixel per ottenere il numero totale di pixel (Tabella 1 e Figura 4). Inoltre, questo protocollo permette la visualizzazione delle ultime regioni replicazione dei cromosomi. Di conseguenza, il modello a bande di incorporazione di BrdU permette l'individuazione delle regioni replicazione attiva di cromosomi, e differenze di temporizzazione replica tra chromosome coppie sono visti come differenze in questo pattern di bande (figure 2 e 3). Infine, le differenze nei modelli di legame può essere paragonato a mappe replica noti temporizzazione per ciascun cromosoma 6,7, che consente una stima della differenza temporale tra replica cromosomi omologhi o bracci cromosoma 4. Ad esempio, i due cromosoma 6 è in Figura 3A presentano una differenza in bande schema di> 2 ore rispetto ai tempi di replica normale del cromosoma 6 4.
Inoltre, una procedura simile a quella descritta qui è stato usato con successo per lo studio della temporizzazione replica asincrona tra i cromosomi X inattiva ed attiva 8,9. I cromosomi X attivi e inattivi sono stati identificati utilizzando vernici cromosoma X. Le immagini sono state acquisite utilizzando Quips mFISH software (Vysis) e il numero di pixel occupati dai cromosomi X e il numero di pixel occupati da fluorescentemente laBeled BrdU sono stati poi calcolati utilizzando NIH Image ( http://rsb.info.nih.gov/nih-image/index.html ).
9. Nick Traduzione di BAC del DNA per l'etichettatura fluorescente (Vysis)
- Aggiungere la seguente miscela di sonde refrigerate una provetta per microcentrifuga:
70 microlitri (4 mg DNA) in dH 2 O
10 microlitri 0,2 mM dUTP-arancione o verde
20 pl 0,1 mM dTTP
20 pl 10x Nick Traduzione Buffer
40 microlitri NT dNTP (meno dTTP)
40 ml nick traduzione enzima
200 microlitri di volume totale
Incubare @ 16 ° CO / N. Fermare la reazione mediante riscaldamento a 70 ° C per 10 min. Raffreddare in ghiaccio per 5 min. - Precipitare il DNA con etanolo aggiungendo la seguente miscela di sonde:
200 microlitri soluzione della sonda
48 microlitri 3 M NaOAc
160 microlitri Cot1DNA (0,25 mg / ul)
1200 microlitri EtOH 100% - Conservare a -80 ° C per 10 min a O / N.
- Spin in fredda a 12.000 xg per 20 min. Decantare EtOH. Lavare 1 volta con il 70% EtOH. Spin come prima. Decantare EtOH. Asciugare solo fino evapora EtOH. Risospendere in 40 microlitri dH 2 O per la concentrazione finale di 100 ng / ml di BAC DNA.
Risultati
Un esempio di analisi replica temporizzazione per cromosoma umano 6 è mostrato in Figura 2. Cellule contenenti una delezione del gene ASAR6 4, situato in 6q16.1, sono stati esposti a BrdU per 5 ore, le cellule raccolte per mitotici e trattati per FISH con sonda cromosoma 6 vernice (Vysis) e per incorporazione di BrdU. Notare che vi è una differenza significativa nella configurazione BrdU bande tra i due 6, che è coerente con un ritardo nella temporizzazione replica di> 2 ore per uno dei il cromosoma 6 [vedere 4 per la temporizzazione replica banding modello di cromosoma 6 prima la cancellazione]. Inoltre, vi è una differenza significativa nella quantità totale di incorporazione di BrdU (pixel) rispetto ad una simile analisi della colorazione DAPI (Figura 2D).
Un altro esempio di analisi replica temporizzazione per cromosoma umano 6 è mostrato in Figura 3. Le celle contenenti il samsoppressione e del gene ASAR6 4 mostrato se la figura 2 sono stati esposti a BrdU per 5 ore, raccolto per le cellule mitotiche e trattati per FISH con un cromosoma 6 CEP più una sonda BAC contenente il ASAR6 genica e per incorporazione di BrdU. Si noti che il cromosoma cancellato 6 (Δ6) mostra più incorporazione di BrdU e un modello più esteso strisce di incorporazione di BrdU rispetto al cromosoma non-soppresso 6. Spread mitotiche da 7 diverse cellule sono state trattate come in figura 3 ed i profili di pixel sia per DAPI BrdU e sono mostrati nella Tabella 1.

Figura 1. BrdU terminale di un marchio di qualità. Una fase S tipica in cellule di mammifero ultimi per 8 a 10 ore, e G2 è tipicamente 2-5 ore. BrdU viene aggiunto per aumentare i periodi di tempo (frecce verdi) per etichettare le ultime porzioni dei cromosomi da replicare. Thcromosomi posta in cellule di mammifero replicare secondo un programma temporale, con la replica precoce e tardiva verificano all'inizio e alla fine della fase S, rispettivamente (linea nera). Cromosomi X inattivi sono in ritardo nei tempi di replica con la maggior parte della sintesi del DNA che si verificano durante la seconda metà di fase S (linea blu). Cromosomi con tempi di replica ritardo sono in ritardo sia in avvio e il completamento della sintesi del DNA, con replicazione attiva continuando con G2 (linea rossa). Culture asincroni vengono raccolti per le cellule mitotiche e trattati per incorporazione di BrdU e FISH.
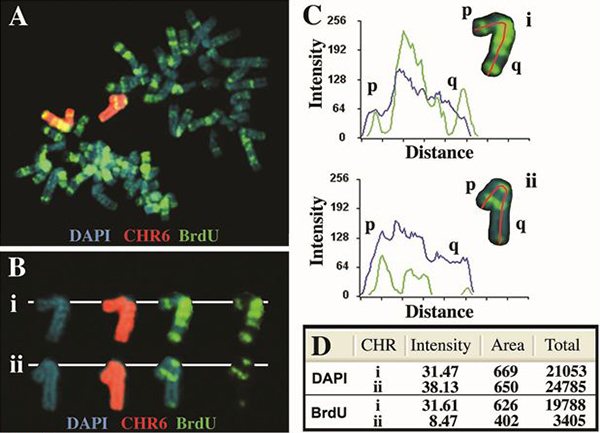
Figura 2. Replica asincrona del cromosoma umano 6. Le cellule contenenti una delezione del gene ingegnerizzato ASAR6 4 sono stati trattati con BrdU per 5 ore, raccolto per le cellule mitotiche e trattati per incorporazione di BrdU e FISH con un chrom osome 6 vernice come sonda. Il DNA è stato colorato con DAPI (blu). A) Uno spread mitotico contenente un tipico modello di "bande" di incorporazione di BrdU (verde) viene visualizzato. Il 6 sonda vernice cromosoma ibridato a due cromosoma 6 (rosso), in questa cella. B) I due cromosoma 6 (I e II) sono stati "tagliati" e visualizzati con le tre etichette fluorescenti separate in immagini distinte. C) Il cromosoma 6 sono stati analizzati utilizzando il software Cytovision ed i profili di intensità di segnale sia per DAPI (blu) e BrdU (verde) vengono visualizzati. La linea rossa indica il percorso utilizzato, dal braccio corto (p) a braccio lungo (q) per la quantificazione sia BrdU e DAPI. La distanza si riferisce alla lunghezza di ciascun cromosoma dal braccio corto (p) a braccio lungo (q) in pixel. D) Quantificazione del segnale totale sia per DAPI e BrdU fluorescenza. I valori totali rappresentano l'intensità media dei pixel moltiplicato per l'area rappresentata da quei pixel.
igura 3 "src =" / files/ftp_upload/4400/4400fig3.jpg "/>
Figura 3. Ritardati replicazione del cromosoma umano 6 contenente una delezione di ASAR6. Le cellule contenenti una delezione di ingegneria del gene ASAR6 4 sono stati trattati con BrdU per 5 ore, raccolto per le cellule mitotiche e trattati per incorporazione di BrdU e FISH con un cromosoma 6 CEP più un BAC contenente il gene ASAR6 come sonde. Il DNA è stato colorato con DAPI (blu). A) Uno spread mitotico contenente un tipico modello di "bande" di incorporazione di BrdU (verde) viene visualizzato. Il cromosoma 6 CEP sonda ibridata due cromosoma 6 di (grande segnale rosso centromerico), e il BAC ibridato ad un singolo cromosoma 6 (piccolo segnale rosso sul braccio lungo) in questa cella. Notare la differenza nel pattern BrdU tra le due bande di 6. B) Il cromosoma 6 è soppresso rappresentato dalla Δ6 e non cancellate 6 per 6. I due 6 sono stati "tagliati" e visualizzati con le tre etichette fluorescenti separate in immagini distinte. C) Tegli cromosoma 6 sono stati analizzati utilizzando il software Cytovision ed i profili di intensità di segnale sia per DAPI (blu) e BrdU (verde) vengono visualizzati. La linea rossa indica il percorso utilizzato, dal braccio corto (p) a braccio lungo (q) per la quantificazione sia BrdU e DAPI. La distanza si riferisce alla lunghezza di ciascun cromosoma dal braccio corto (p) a braccio lungo (q) in pixel. D) Quantificazione del segnale totale sia per DAPI e BrdU fluorescenza. I valori totali rappresentano l'intensità media dei pixel moltiplicato per l'area rappresentata da quei pixel.
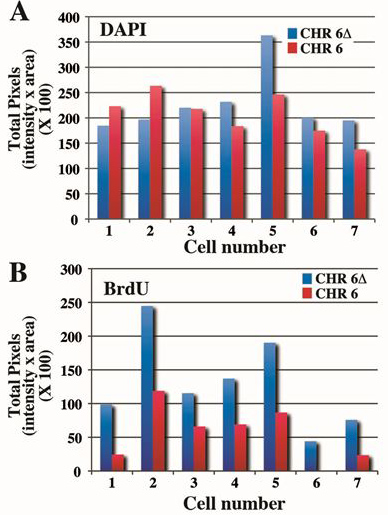
Figura 4. Quantificazione della differenza temporale tra replicazione del cromosoma 6. Le cellule contenenti una delezione di ingegneria del gene ASAR6 4 sono stati trattati con BrdU per 5 ore, raccolto per le cellule mitotiche e trattati per incorporazione di BrdU e FISH con un cromosoma 6 CEP sonda più unBAC contenente il gene ASAR6 come sonda. Spread mitotici sono stati trattati come in figura 3 e 7 i valori per celle diverse sono mostrati (vedi Tabella 1). A) La colorazione DAPI è stato quantificato e il numero totale di pixel viene visualizzato per ciascuna cella. Si noti che non vi è differenza solo ~ 10-20% tra cromosomi all'interno della stessa cella. B) L'incorporazione di BrdU è stata quantificata dai cromosomi stessi B pannello sopra. Si noti che non vi è> 2 volte differenza tra i valori dei pixel totali per il cromosoma cancellato e non cancellate i 6 anni.
Discussione
La preparazione degli spread cromosoma è un passo fondamentale per il successo di replica-timing saggio descritto qui. L'inclusione di una fase di pretrattamento Colcemid prima del trattamento ipotonico può facilitare la frequenza e la diffusione delle cellule mitotiche. Abbiamo tipicamente esporre le cellule ad colcemdi per 1-3 ore prima del raccolto, e utilizzare Colcemid ad una concentrazione finale di 10 ug / ml. Tuttavia, l'inclusione di una fase di pretrattamento Colcemid può modificare la lunghezza di G2 e di conseguenza possono alterare la replicazione e la temporizzazione apparente stato di condensazione dei cromosomi 3.
Questo procedimento può essere applicato a molti tipi cellulari e specie variando la durata dell'incubazione BrdU, che dipende dalla durata del ciclo cellulare. Per più linee cellulari umane e di topo, la fase G2 è tipicamente 3-6 hr, pertanto, trattamenti BrdU sono tipicamente in questo intervallo. Un'alternativa alla BrdU è 5-etinil-2'deoxyuridine (EDU) e la sua successiva rivelazione utilizzandoun azide fluorescente e "chimica click" reazione 10. Lo schema di rilevamento EdU ha diversi vantaggi rispetto al regime di rilevazione BrdU. Ad esempio, il rilevamento di EdU non richiede la fissazione del campione o denaturazione del DNA. Pertanto, l'uso di EdU di replica valutare temporizzazione può essere combinato con semplici G-banding tecniche invece di pesce per identificare i cromosomi di interesse.
Il protocollo di temporizzazione replica qui descritto è specificamente progettato per dosaggio fine S fase più qualsiasi sintesi del DNA estende in G2. Inoltre, la replicazione del DNA può essere monitorato durante tutta la fase S usando questa procedura utilizzando brevi (15-30 min) impulsi di BrdU seguiti da periodi relativamente lunghi inseguimenti di 6-10 ore. Questo permette la visualizzazione di incorporazione di BrdU sia prima e media S-fase. Ad esempio, alcuni riarrangiamenti cromosomici tumorali derivate sono ritardate sia inizio e di completamento della sintesi del DNA lungo l'intera lunghezza dei cromosomi 3 .
Un vantaggio di questa procedura è che replica temporizzazione temporizzazione replica essa saggi in cellule singole. Pertanto, ha la capacità di rilevare le differenze di temporizzazione replica tra cromosomi omologhi contenuti all'interno della stessa cella. Mentre ci sono altre procedure, ad esempio. temporizzazione replica ibridazione in situ (ReTiSH, 11), che hanno la capacità di rilevare le differenze di temporizzazione replica tra alleli a loci specifici su cromosomi omologhi, il procedimento qui descritto può rilevare differenze nei tempi di replica per tutta la lunghezza dei cromosomi. Inoltre, la procedura può essere saggio differenze nei tempi replicazione dei cromosomi che sono presenti in una frazione di cellule di una popolazione 3. Per esempio molte linee cellulari tumorali e primarie campioni tumorali contengono riarrangiamenti che sono presenti in meno del 50% delle cellule. Attualmente stiamo utilizzando questa procedura per cromosomi saggioprimarie in campioni tumorali, e sono stati in grado di rilevare la replica asincrona tra i cromosomi in più campioni. Tuttavia, dato che i campioni tumorali primarie hanno un numero limitato di figure mitotiche, circa un terzo delle colture primarie omesso di dare un numero sufficiente di spread mitotiche.
Un altro vantaggio che questa procedura ha più di microarray o sequenziamento test basati è che i cromosomi individuali sono analizzati invece di DNA immunoprecipitato da pool di cellule. Nei saggi basati immunoprecipitazione polimorfismi devono essere identificati e collegati a alleli specifici per distinguere i tempi di replica tra alleli.
Inoltre, con il riconoscimento che molte cellule cancerose contengono numerosi riarrangiamenti 12, nonché l'osservazione che la replicazione del DNA lo stress è associato ad instabilità genomica in cellule tumorali 13, riteniamo che questo protocollo è un f strumento utile e sempliceo l'analisi di routine di temporizzazione replica di cromosomi nelle cellule tumorali.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto da una sovvenzione da parte del National Cancer Institute, CA131967.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| Anti-BrdU-FITC | Roche Millipore | 11202593001 MAB326F | 50 pg / pl |
| Nick Traduzione Kit | Abbott Molecular (Vysis) | 07J00-0001 | |
| Spectrum Arancione dUTP | Abbott Molecular (Vysis) | 02N33-050 | |
| CEP | Abbott Molecular (Vysis) | Varia | |
| LSI / WCP ibridazione tampone | Abbott Molecular (Vysis) | 06J67-011 | |
| CEP ibridazione tampone | Abbott Molecular (Vysis) | 07J36-001 | |
| Cromosoma painti | MetaSystems Gruppo | D-14NN-050-TR | |
| Olympus BX61 microscopio a fluorescenza | Olimpo | BX61TRF-1-5 | |
| Microscopio di imaging del software di sistema | Applied Imaging | Cytovision 3.93.1 | |
| Macchina fotografica digitale | Olimpo | UCMAD3 | |
RICETTE IN ibridazione in situ 35 ml di formammide * (Sigma) * E 'importante utilizzare formammide che è stato conservato a -20 ° C. Prolungata conservazione a temperatura ambiente genera acido formico e il w pHill essere troppo bassa. 50% Formamide/2x SSC 25 ml formamide (Sigma) SSC 20x, 4 L 702 g NaCl (Sigma) PN Buffer [0,1 M NaP0 4 0,1% NP_40 (Sigma)] Effettuare una soluzione 0,1 M di fosfato di sodio ogni (Filtro sterilizzare e conservare in 500 ml aliquote). 0,1 M NaH 2 P0 4, 1 L 13,8 g di NaH 2 P0 4 (Sigma): 0,1 M NaH 2 P0 4 1 L 14,2 g di NaH 2 P0 4 (Sigma) PN: Regolare il pH di 0,1 M Na 2 HP0 4 a pH 8,0 con 0,1 M NaH 2 P0 4. Filtro sterilizzare e aggiungere 1 ml di NP-40. PNM 50 ml 1.25 g non grassi del latte secco (Sigma) Mescolare per 15-20 minuti mescolando costantemente. Spin 2 volte a 400 xg per 10 min. Utilizzare il surnatante, e fare attenzione a non disturbare proteine del latte precipitate. |
Riferimenti
- Payer, B., Lee, J. T. X chromosome dosage compensation: how mammals keep the balance. Annu. Rev. Genet. 42, 733-772 (2008).
- Breger, K. S., Smith, L., Turker, M. S., Thayer, M. J. Ionizing radiation induces frequent translocations with delayed replication and condensation. Cancer Research. 64, 8231-8238 (2004).
- Smith, L., Plug, A., Thayer, M. Delayed Replication Timing Leads to Delayed Mitotic Chromosome Condensation and Chromosomal Instability of Chromosome Translocations. Proc. Natl. Acad. Sci. U.S.A. 98, 13300-13305 (2001).
- Stoffregen, E. P., Donley, N., Stauffer, D., Smith, L., Thayer, M. J. An autosomal locus that controls chromosome-wide replication timing and mono-allelicexpression. Hum. Mol. Genet. 20, 2366-2378 (2011).
- Breger, K. S., Smith, L., Thayer, M. J. Engineering translocations with delayed replication: evidence for cis control of chromosome replication timing. Hum. Mol. Genet. 14, 2813-2827 (2005).
- Camargo, M., Cervenka, J. Patterns of DNA replication of human chromosomes. II. Replication map and replication model. Am. J. Hum. Genet. 34, 757-780 (1982).
- Cohen, S. M., Cobb, E. R., Cordeiro-Stone, M., Kaufman, D. G. Identification of chromosomal bands replicating early in the S phase of normal human fibroblasts. Exp. Cell Res. 245 (98), 321-329 (1998).
- Diaz-Perez, S., et al. The element(s) at the nontranscribed Xist locus of the active X chromosome controls chromosomal replication timing in the mouse. Genetics. 171, 663-672 (2005).
- Diaz-Perez, S. V., et al. A deletion at the mouse Xist gene exposes trans-effects that alter the heterochromatin of the inactive X chromosome and the replication time and DNA stability of both X chromosomes. Genetics. 174, 1115-1133 (2006).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc. Natl. Acad. Sci. U.S.A. 105, 2415-2420 (2008).
- Schlesinger, S., Selig, S., Bergman, Y., Cedar, H. Allelic inactivation of rDNA loci. Genes Dev. 23, 2437-2447 (2009).
- Mitelman Database of Chromosome Aberrations in Cancer. , Available from: http://cgap.nci.nih.gov/Chromosomes/Mitelman (2006).
- Branzei, D., Foiani, M. The checkpoint response to replication stress. DNA Repair (Amst). 8, 1038-1046 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon