Method Article
ヌクレオカプシドからウイルスDNAの調製
要約
我々は、感染細胞から高純度のヘルペスウイルスヌクレオカプシドDNAを単離するプロセスを説明します。ソリューションから取り込んだ最終的なDNAは、それが理想的にハイスループットシーケンシング、高忠実度PCR反応、および新たなウイルスの組換え体を生産するトランスフェクションに最適です、高濃度および純度である。
要約
ウイルスは絶対携帯寄生虫であり、そのため、DNAの研究では、離れて宿主細胞の汚染物質とDNAからのウイルス材料を分離する必要があります。いくつかの下流のアプリケーションでは、このプロトコルによって提供される純粋なウイルスDNA、大量のを必要とする。これらのアプリケーションは、ウイルスゲノムの宿主DNAの除去は、ウイルス配列のデータ出力を最適化するために重要であるシーケンシング、および新たなウイルスの組換え株の生産、プラスミドおよびリニアウイルスDNAの組換えを容易に精製されたの共トランスフェクション。1,2を、含まれています3
この手順では、感染細胞からリニアヘルペスウイルスヌクレオキャプシドのDNAを精製単離するために抽出し、密度ベースの遠心分離の組み合わせを採用しています。4,5初期精製ステップは、抽出と遠心操作中に含まれているとウイルスDNAを保護するウイルスキャプシドを、精製分離することを目指すことは、細胞タンパク質およびDNAを除去。ヌクレオカプシドの溶解は、ウイルスDNAを解放し、最後に2つのフェノール - クロロホルムの手順では、残りのタンパク質を除去。ソリューションから取り込んだ最後のDNAは1.90の平均OD 280分の260と、高濃度および純粋です。使用されて感染細胞の量に応じて、150から800μgの以上からウイルスDNAの範囲の収量。このDNAの純度は、4Cでの長期保管中にそれが安定したものになります。このDNAは、このように理想的にハイスループットシーケンシング、高忠実度PCR反応、およびトランスフェクションに適しています。
プロトコルを開始する前に、それは、ディッシュあたりの細胞(コンフルエント15cmの皿に8 × 10 6 PK - 15細胞の平均など)の平均数を知ることは重要です、そして、使用するウイルスストックの力価(例えば、ml当たり1 × 10 8プラーク形成単位を)。これらのプロトコルのために感染症の適切な多重度(MOI)を計算するために必要な。例えば6、上記のウイルスストックとPK - 15細胞の一つ15cmの皿に感染する、MOI 5の時には、400μlのを使用します。ウイルスストックと培地の3.6ミリリットル(一15cmのプレートに4 mlの総接種量)とそれを希釈する。
複数のウイルスDNAの調製物は、同時に調製することができる。同時の準備の数は、超遠心ローターで開催された管の数によって制限されます(ウイルスごとに、下記のステップ3.9を参照してください)。ここでは、つのウイルスのために行われているかのように手順を説明します。
プロトコル
1。一日目:ウイルス感染とバッファーの調製
- 感染の組織培養細胞の5-10皿(15cmの直径を)準備、仮性狂犬病ウイルス(PRV)またはヘルペスのためのVero細胞用例:PK - 15細胞(HSV)ウイルス単純。
- 細胞が95である場合 - 100%コンフルエント、(MOI)50から10の時にそれらを感染させる。これを行うには、プレート当たり4 mlの全体積にウイルスストックを用いて各プレートに接種し、37℃で1時間プレートをインキュベート℃に軽くロックプレート細胞単層はウイルス接種によって完全に被覆されたままであることを保証するために15分ごと。一方、次のステップのための培地を温める。
- 感染症の1時間後には、プレートからウイルス接種物を吸引除去暖かい培地15mlを追加し、37℃の加湿インキュベーター中、12〜20時間のために。
- LCMバッファ(表1参照)と° C完全に混合するための一晩4石を準備します。ウイルスDNAの再懸濁するためのTNEを準備する(表2参照)。
2。第二日目、フェーズ1:細胞の溶解および超遠心法
- 視覚的にその細胞の感染は、均一な細胞変性効果(CPE)を起こしていることを確認します。すべての細胞が切り上げているが、プレートから持ち上げていないまで、感染が進行することができます。
- 培地中に感染した細胞をこすり、および50 mlコニカルチューブに細胞や培地を組み合わせる。このステップに必要なコニカルチューブの数が上記のステップ1.2での感染のために使用される料理の数に依存する、 例えば、1つ50 mlコニカルチューブにペレットを15 mlの培地それぞれの3皿の最大を使用することができるオプション:。リンス10ミリリットルPBSでプレートは、細胞と培地との組み合わせ。
- 室温で2000年の相対centifugal力(RCF)で10分間、細胞と培地を遠心し、上清を吸引し、PBSの10mlにそれぞれのペレットを再懸濁します。つのウイルス感染からペレットをステップ2.2で複数のコニカルチューブに広がっていた場合、これらはこのステップに一つコニカルチューブに再結合することができます。遠心分離や吸引を繰り返して、 オプション:このペレットを-20℃で保存し、プロトコルは、後で継続することができる。
3。第二日目、フェーズ2:超遠心法
次の手順中に、可能な限り氷の上で細胞ペレットとLCMのバッファを保持します。
- 0%グリセロールにβ-メルカプトエタノールを追加 - LCMバッファ。に設定することによって超遠心機を(下のステップ3.9の)冷却開始4℃、真空をオンにする。
- 0%グリセロール5mlの細胞ペレットを - LCMのバッファを、それがこんもり茂ったれなくなるまで。必要に応じて、ボルテックスチューブはどんな溶解していない細胞ペレットを分割する。
- 1.5 mlのフロン(1,1,2 - トリクロロ-1,2,2 - トリフルオロエタン)を加えて激しくボルテックスで抽出する。すぐに4℃の温度で2000 RCFで10分間遠心操作ワイドボアピペットチップ(インタフェースを避ける)で最上位層を収集し、新しいチューブに移すオプション:。ペレットは、ゼラチン状の固体である場合は、すぐに新しいチューブに最上層を注ぐことができる。
- 再びフロンの抽出と遠心を繰り返します。
- 最上層(〜5 ml)を収集し、新しいポリアロマー超遠心チューブに移す。 LCMバッファ - 残りの2つのグリセロールにβ-メルカプトエタノールを追加。
- LCM(中間層)し、45%グリセロール2.5 mlで再び基礎となる - - LCM(底層)5%グリセロール3mlでフロンのエキス(最上層)の基礎となることによってポリアロマーの遠心管に勾配を準備します。試験管の内容のオーバーフローを避けるために、細いピペットを使用してください。
- 必要に応じて、LCMバッファの同じ比率を使用して同等のバランスチューブを準備する。
- 超遠心バケットにチューブを移す。最上位層(一度に〜50から100μL)にLCMバッファ - 優しく余分な0%のグリセロールを添加することにより、お互いに0.1gの内にバケツのバランスをとります。
- 水平またはスイングバケットローターで冷たい超遠心機を使用して、4℃の温度で77000 RCF(SW41Tiのローター用など25000 rpm)で1時間のバランスサンプルをスピン
- 超遠心分離中に、エタノール沈殿のステップ(下記4.6)に浮遊DNAを捕捉するためにガラスのフックを作る。ガラスパスツールピペットの両端を持ち、火炎上中央のセクションを中断。ガラスのセクションはほとんど溶融の場合は、しっかりと直角フック(<90 °)を作成し、そして終了を封印するために急激に引っ張って軽く曲げ、ガラスを伸ばすために慎重に引き抜いてください。ガラスのフックの先端が開いている場合、ガラスはそれを閉じるのに十分な溶けように炎を介して先端を保持する。
- 超遠心分離した後、中央のダークスポットと薄い不透明なペレットが表示されるはずです。慎重に両側に沿っ余滴を含めて、チューブから液体を吸引除去するが、チューブの底にペレットを避ける。
- 室温で0.5 mlのTNEを追加。
- ペレットは、ペレットの水和を可能にするために少なくとも10分間放置します。Optional:4で守ればペレットはまた、一晩TNEで再懸濁ができる℃の
4。第三日目:"ゴースト"DNAの沈殿
- 小口径のピペットチップ( 例えば、P200のピペットチップ)でピペッティングしてペレットを破る。ウイルスのDNAがまだこの時点でキャプシドに含まれているため、このステップでせん断を心配しないでください。キャプシドを破壊するために、室温で、ウイルスのペレットを含むチューブに以下の試薬を追加します。
- 4.25ミリリットルTNE
- 0.25ミリリットル10%SDS
- 2mgのプロティナーゼK
ここからはせん断用心、例えば大口径のピペットを使用してウイルスの材料にボルテックスしないでください
- フェノール - クロロホルム5mlを加えることによってウイルスタンパク質を抽出し、即座にエマルジョンを維持するために反転またはボルテックスを開始。室温で2000 rcfで5分間乳化物を遠心分離し、10秒間混ぜる。インタフェースを避け、ワイドボアピペットの先端と上部層を収集し、新しいチューブに移すオプション:使用する位相ロックゲル(PLG)チューブをこの段階でインターフェイスからの汚染を避けるために。
- 再びフェノール - クロロホルム抽出し、遠心を繰り返します。
- 第二フェノール - クロロホルム抽出後、30ミリリットルのガラスCorexチューブに最上層を回収し、10〜15分は-20℃でチューブを冷やし、それが凍結しないように気をつける。
- 、氷冷エタノール10mlを加え、チューブを(上からパラフィルムMの部分をストレッチするなど )カバー、そしてすぐに混在させる反転。細い縄状の沈殿物で構成されて表示されるようにDNA"ゴースト"、待ってください。さらにミックスし、粘着性DNAが一緒にクラスタに沈殿させるために軽くチューブを反転。
- DNAのゴースト(s)をキャプチャするためにガラスのフックを使用してください。慎重に余分な液体を除去するためにDNAをブロット、その後、1.5 mlチューブにフックを(下に傾ける)に置き、乾燥させます。 0.25を追加 - TEの0.5 mlをDNAのゴーストを溶解する。少なくとも1時間進行するDNAの再懸濁を許可する。 4℃店舗のDNA℃のオプション:1.5 mlチューブにガラスのフックを切り離し、DNAの収量を最大にするには、ガラスの破片からなるよう再懸濁は、時間をかけ続けることができます。
5。代表的な結果
MOI十分に高いと、細胞は野生型PRV株のために〜18時間の感染後(HPI)の中で完全なCPEを達成すべきである、と野生型HSV - 1株(図1)〜24 HPI。一度感染した細胞ペレットが収穫され、残りのプロトコルのステップは長い1日で完全になるか、2日間(図2)を介して実施することができます。
DNAのゴーストをキャプチャするためにガラスのフックを準備するとき、それは後には1.5 mlチューブ(図3)のベースに収まることができるように、90度未満となるフックの角度を作ることが重要です。このフックの先端部をシールするピペット内のDNAの損失を防ぎます。
DNAの幽霊は、沈殿工程中に形成し、白、粘着性の糸(図4)として表示されます。これらの集計とスティック相互に、かつ/またはDNAを捕捉するために使用されるガラスのフック。したがって、同一のガラスのフックは、降水量のソリューションの中から、DNAの複数のスレッドを収集するために使用することができます。
| 希望のボリュームに基づいて列を選択してください。 | 10ミリリットル | 15ミリリットル | 20ミリリットル | 40ミリリットル |
| 1MのKCl | 1.3ミリリットル | 1.9ミリリットル | 2.5ミリリットル | 5ミリリットル |
| 1Mトリス(pH 7.4) | 300μlの | 450μlの | 600μlの | 1.2ミリリットル |
| 1M MgCl 2の | 50μlの | 75μlの | 100μlの | 200μlの |
| 0.5 EDTA | 10μlの | 15μlの | 20μlの | 40μlの |
| NP40/IGEPAL | 50μlの | 75μlの | 100μlの | 200μlの |
| β-メルカプトエタノール(使用直前に追加) | 4.3μL | 6.5μL | 8.6μL | 17.2μL |
| 希望するパーセントのグリセロール(0,5、または45%)の割合を使用 | ||||
| 0%のグリセロール | なし | なし | なし | なし |
| 水を | 8.3ミリリットル | 12.5ミリリットル | 16.7ミリリットル | 33.4ミリリットル |
| 5%のグリセロール | 0.5ミリリットル | 0.8ミリリットル | 1.0ミリリットル | 2.0ミリリットル |
| 水を | 7.8ミリリットル | 11.7ミリリットル | 15.7ミリリットル | 31.4ミリリットル |
| 45%のグラムlycerol | 4.5ミリリットル | 6.8ミリリットル | 9.0ミリリットル | 18.0ミリリットル |
| 水を | 3.8ミリリットル | 5.7ミリリットル | 7.7ミリリットル | 15.4ミリリットル |
表1。 LCMバッファーの調製のためのレシピ
| 50mlの容積のための |
| 5ミリリットル1MのNaCl |
| 2.5ミリリットル1Mトリス、pH7.5の |
| 1ミリリットル0.5 EDTA |
| 41.5ミリリットルH 2 O |
表2。 TNEの準備 (0.1MのNaCl、50mMトリス、pH7.5の、10mMのEDTA)
| 1リットルの容積のための |
| 0.2グラムのKCl |
| 0.2グラムのKH 2 PO 4 |
| 8グラムのNaCl |
| 1.15グラムのNa 2 HPO 4 |
| 1LのH 2 O |
表3。 PBSの調製 (2.7 mMの塩化カリウム、1.5mMのKH 2 PO 4、137 mMのNaCl、8.1 mMののNa 2 HPO 4、pH7.0)で
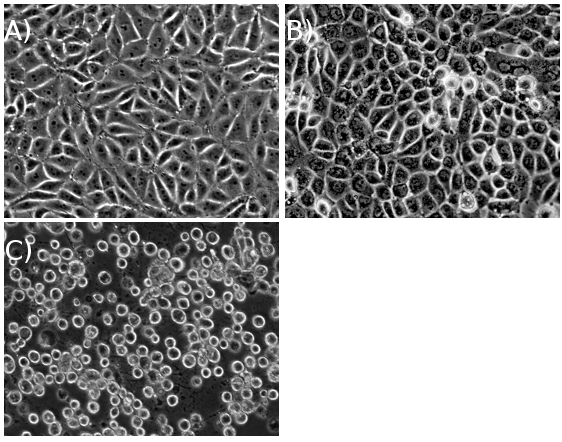
図感染を介して感染前に1。PK15細胞()、(B)の中間、および(C)細胞変性効果(CPE)で、感染の高い多重度の後。

図2プロトコルの概要。

図3:ガラスの3つの代表例はウイルスDNAの幽霊を収集するために使用されるフック。

図4。整形式と豊富なDNA"ゴースト"の代表例。
ディスカッション
このプロトコルの一部は、もともとBSL4条件におけるウイルスDNAの分離のために開発されたが、それは非BSL条件にも同様に適応された4我々は一般的にDNAゲノムを有するα-ヘルペスウイルスPRVおよびHSV - 1、からDNAを単離するためにこのプロトコルを使用キャプシドと脂質エンベロープに囲まれたタンパク質で囲みます。7,8しかし、それが含むβ-およびγ-ヘルペスウイルスやアデノウイルス、おそらく他の大型DNAウイルスに直接適応である。同様の抽出は、一般的にだけでなく、RNAウイルスのために使用されています。9
含まれている精製の複数のメソッドからこのDNA調製可能性が高い結果の頑健性。初期フロン抽出変性脂質膜、細胞成分を分離し、また、脂質エンベロープとヘルペスウイルスのキャプシドを囲む外側のテグメントタンパク質を放出する。これは、重いウイルスのヌクレオカプシドペレットは、2つの勾配のクッションを介して、効果的に他のほとんどの細胞成分からそれらを分離する遠心分離、が続いている。これらのキャプシドは再懸濁させ、そしてウイルスのヌクレオカプシドDNAは、プロテイナーゼKと洗剤でキャプシドを破裂によって解放されます。二つのフェノール - クロロホルム抽出は、徹底的にヌクレオカプシドコンポーネントと残りの細胞タンパク質を除去。最後に、溶液中での大規模なウイルスDNAゲノムの降水量は、塩や粒子状汚染物質のキャリーオーバーを低減します。
ウイルスのヌクレオカプシドDNAを単離するには、この手順は、ダウンストリームアプリケーションのための豊富な、高純度の材料を提供する。複数の抽出と遠心分離のステップの組み合わせは、DNAを10でより多くの細胞残渣や劣化したタンパク質産物を残す単純な単一の抽出のプロトコルからこのプロトコルを区別します。蛋白質の汚染物質は、ウイルスDNAの貯蔵安定性を低下させ、細胞にトランスフェクション効率を減らすことができます。細胞DNAの汚染物質は、マイナスのPCR反応およびハイスループットシーケンシングプロトコルに影響を与える。1は、このプロトコルのDNAの収量はそれがうまく制限断片長多型(RFLP)分析に適してサザンブロッティングこと、また高いです。5,11 Oneヌクレオカプシドの準備が十分に提供これらの分析のすべての材料。
感染のために使用される細胞の量は、ウイルスDNAの最終的な収量に影響を与えます。野生型の成長率とPRV株の場合は、PK - 15細胞(一つ15cmの直径の皿は、約8 × 10 6細胞を保持している)5〜10皿の入力は、通常、ウイルスDNAの250から500μgのが得られます。感染性ビリオンの減少利回りとウイルス変異体の場合は、入力の材料は、上向きに応じてスケーリングされます。料理の入力数を倍増して約収量を倍増する、とここで説明する試薬の同じ量を処理することができます。このポイントを超えて入力を向上させるために、我々はステップ3.13を介して2つの並列処理の流れ(例:2つの細胞ペレット二抽出チューブ)への入力材料を分離することをお勧めします。同じウイルス株の二つの超遠心ペレットは、この再懸濁のステップに一つのチューブに結合することができます。 DNAが正常手順の最後にゴーストを形成するために失敗した場合は、主要なトラブルシューティングのアプローチは、DNAの収量を増加させると、その降水量を向上させるセルの入力量を、増加させることです。
他の条件でもゴーストの形成の成功に影響を与える可能性があります。例えば、それはウイルスのヌクレオカプシドが豊富な点で感染症の収穫時には重要ですが、ほとんどは細胞内や細胞関連です。培地に放出されたビリオンは、このプロシージャでは捕捉されませんので、遅すぎる感染で(CPEは、細胞が培養皿から切り離し点に進行しているなど)細胞を採取すると、潜在的なDNAの収量やゴーストの形成を減少します。それは完全に、次の抽出工程をフルに活用するためにLCMバッファー(ステップ3.2)で収穫された細胞ペレットを溶解するためにも重要です。細胞ペレットは、前のステップで凍結されている場合は、この再懸濁は、より多くの時間と注意が必要です。マイナーな手続きの詳細は、同様にDNAの収量に影響を与えます。ステップ3と4は、チルドソリューションを使用し、氷上で全てのチューブを保持することなく実施することができる一方で、DNAのゴーストの成功率は、試薬は、コールドにまとめられていると非常に高くなります。同様に、ソリューションの時間の経過とともに低下し、したがって、使用がその削減容量を最適化する直前のLCMソリューションに追加する蛋白質の混乱を改善し、DNAの収量やゴーストの形成を増加させるのβ-メルカプトエタノールの安定性。これらの細部に細心の注意を払うことが、高品質なウイルスDNAの出力を向上します。
ウイルスDNAが粘性であるため、得られた溶液は不均質であることができる。ガラスのフックから再懸濁するためのより多くの時間を許可すると、有用なDNAの収量を増加させ、溶液の均一性を向上させます。標準的な分光光度法DNAの収量および純度を定量するために使用することができます。 DNAのfluorimetryは、DNAの収量のより正確な対策(例えば、Invitrogen社PicoGreen dsDNAを染色)を提供しています。
開示事項
利害の衝突は宣言されません。
謝辞
著者らは、このプロトコルを微調整するにグレッグスミス、リサPomeranz、マットライマン、マルリースエルドリッジ、ハリナStaniszewska Goraczniak、およびEnquistラボのメンバーの貢献を感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント |
| フロン(1,1,2 - トリクロロ-1,2,2 - トリフルオロエタン) | フィッシャー | T178 - 4 | フロン含有廃棄物の適正処理に関するガイドラインについては、貴機関に確認し、またはメンデスらを参照してください。潜在的なフロン代替用。9 |
| 位相ロックゲルチューブ、ヘビー15ミリリットル容量 | 5 PRIME | 2302850 | オプション |
| ポリアロマー超遠心管 | ベックマン* | 331372 * | *あなた自身の超遠心分離に適したチューブを選択し、これらは例としてのみ含まれています。 |
| NP - 40 / IGEPAL | シグマ | I - 3021 | |
| PK - 15細胞 | ATCC | CCL - 33 | |
| Vero細胞 | ATCC | CCL - 81 | |
| PBS | HyClone | SH30028.03 |
参考文献
- Szpara, M. L., Parsons, L., Enquist, L. W. Sequence variability in clinical and laboratory isolates of herpes simplex virus 1 reveals new mutations. J Virol. 84, 5303-5313 (2010).
- Banfield, B. W., Kaufman, J. D., Randall, J. A., Pickard, G. E. Development of pseudorabies virus strains expressing red fluorescent proteins: new tools for multisynaptic labeling applications. J Virol. 77, 10106-10112 (2003).
- Kobiler, O., Lipman, Y., Therkelsen, K., Daubechies, I., Enquist, L. W. Herpesviruses carrying a Brainbow cassette reveal replication and expression of limited numbers of incoming genomes. Nat Commun. 1, 146-146 (2010).
- Enquist, L. W., Madden, M. J., Schiop-Stanley, P., Vande Woude, G. F. Cloning of herpes simplex type 1 DNA fragments in a bacteriophage lambda vector. Science. 203, 541-544 (1979).
- Smith, G. A., Enquist, L. W. Construction and transposon mutagenesis in Escherichia coli of a full-length infectious clone of pseudorabies virus, an alphaherpesvirus. J Virol. 73, 6405-6414 (1999).
- Flint, S. J., Enquist, L. W., Racaniello, V. R., Skalka, A. M. Principles of virology. , 3rd edn, ASM Press. (2008).
- Pomeranz, L. E., Reynolds, A. E., Hengartner, C. J. Molecular biology of pseudorabies virus: impact on neurovirology and veterinary medicine. Microbiol Mol Biol Rev. 69, 462-500 (2005).
- Roizman, B., Pellett, P. E. Fields Virology. Knipe, D. M., Howley, P. M. , 2 ed, Lippincott Williams & Wilkins. 2381-2397 (2001).
- Mendez, I. I., Hermann, L. L., Hazelton, P. R., Coombs, K. M. A comparative analysis of freon substitutes in the purification of reovirus and calicivirus. J Virol Methods. 90, 59-67 (2000).
- Gharabaghi, F., Aymard, M., Trotemann, P., Gerdil, C. A rapid and simplified micromethod for subtyping varicella-zoster virus. J Med Virol. 31, 129-134 (1990).
- Granstedt, A. E., Szpara, M. L., Kuhn, B., Wang, S. S., Enquist, L. W. Fluorescence-based monitoring of in vivo neural activity using a circuit-tracing pseudorabies virus. PLoS One. 4, e6923-e6923 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved