Method Article
Préparation de l'ADN viral à partir des nucléocapsides
Dans cet article
Résumé
Nous décrivons le processus d'isolation de haute pureté nucléocapside herpèsvirus ADN à partir de cellules infectées. L'ADN capturé finale de la solution est de forte concentration et de pureté, qui le rend idéal pour séquençage à haut débit, haute fidélité réactions PCR, et transfections pour produire de nouveaux virus recombinants.
Résumé
Les virus sont des parasites cellulaires, et donc l'étude de leur ADN nécessite un matériau isolant virale loin de contaminants de la cellule hôte et de l'ADN. Plusieurs applications en aval nécessitent de grandes quantités d'ADN viral pur, qui est prévue par le présent protocole. Ces applications comprennent le séquençage du génome viral, où la suppression de l'ADN hôte est cruciale pour optimiser la production de données pour les séquences virales, et la production de nouvelles souches virales recombinantes, où la co-transfection de plasmide purifié et linéaire d'ADN viral facilite la recombinaison. 1,2, 3
Cette procédure utilise une combinaison d'extractions et de densité à base de centrifugation afin d'isoler l'ADN purifié herpèsvirus linéaire nucléocapside à partir de cellules infectées. 4,5 Les premières étapes de purification pour but d'isoler capsides virales purifiées, qui contient et protège l'ADN viral au cours de l'extraction et les étapes de centrifugation qui éliminent les protéines cellulaires et l'ADN. Lyse des nucleocapsides libère alors l'ADN viral, et deux finales de phénol-chloroforme étapes éliminer les protéines restantes. L'ADN capturé finale de la solution est très concentré et pur, avec une moyenne DO 260/280 de 1,90. En fonction de la quantité de cellules infectées utilisées, les rendements de l'ADN viral gamme de 150 à 800 mg ou plus. La pureté de cet ADN, il est stable pendant un stockage prolongé à 4 ° C. Cet ADN est donc parfaitement adapté pour séquençage à haut débit, haute fidélité réactions PCR, et transfections.
Avant de commencer le protocole, il est important de connaître le nombre moyen de cellules par boîte (par exemple, une moyenne de 8 x 10 6 cellules PK-15 confluentes dans un plat de 15 cm), et le titre du stock viral à utiliser ( par exemple 1 x 10 8 unités formant des plages par ml). Ceux-ci sont nécessaires pour calculer la multiplicité appropriée d'infection (MOI) pour le protocole. 6 Par exemple, pour infecter une de 15 cm plat de cellules PK-15 avec le stock au-dessus virale, àune MOI de 5, vous pouvez utiliser 400 ul de stock viral et le diluer avec 3,6 ml de milieu (volume inoculation total de 4 ml pour une plaque de 15 cm).
Plusieurs préparations d'ADN viraux peuvent être préparés en même temps. Le nombre de préparations simultanées n'est limité que par le nombre de tubes détenus par le rotor ultracentrifugeuse (un par le virus; voir l'étape 3.9 ci-dessous). Nous décrivons ici la procédure comme si elle était faite pour un virus.
Protocole
1. Premier jour: Infection virale et la préparation des tampons
- Préparez des plats 5-10 (15 cm de diamètre) de cellules de culture tissulaire de l'infection, par exemple cellules PK-15 pour le virus de la pseudo-rage (PRV) ou des cellules Vero pour l'herpès simplex virus (HSV).
- Lorsque les cellules sont de 95 - 100% de confluence, les infecter à un (MOI) de 5-10. Pour ce faire, inoculer chaque plaque en utilisant stock de virus dans un volume total de 4 ml par plaque, puis incuber les plaques pendant 1 heure à 37 ° C. Plaques rocheuses délicatement toutes les 15 minutes afin de s'assurer que la monocouche de cellules reste entièrement recouvert par le virus de l'inoculum. Pendant ce temps, faire chauffer le support pour la prochaine étape.
- Après une heure d'infection, aspirer inoculum viral à partir des plaques, ajouter 15 ml de milieu chaud, et incuber à 37 ° C pendant 12-20 heures dans un incubateur humidifié.
- Préparation des tampons LCM (voir tableau 1) et le rock toute la nuit à 4 ° C pour un mélange uniforme. Préparer TNE pour la remise en suspension de l'ADN viral (voir tableau 2).
2. Second Jour, Phase 1: la lyse cellulaire et ultracentrifugation
- Vérifier visuellement que l'infection des cellules a causé uniforme effet cytopathogène (ECP). Permettre à l'infection de progresser jusqu'à ce que toutes les cellules ont arrondi vers le haut, mais n'ont pas soulevé de la plaque.
- Racler les cellules infectées dans le milieu, et de combiner des cellules et du milieu dans des tubes coniques de 50 ml. Le nombre de tubes coniques nécessaires pour cette étape dépendra du nombre de plats utilisés pour l'infection à l'étape 1.2 ci-dessus, par exemple un tube conique de 50 ml peut être utilisée pour obtenir un culot d'un maximum de 3 plats de 15 ml de milieu de chaque option:. Rincer le plaques avec 10 ml de PBS et se combinent avec les cellules et le milieu.
- Centrifuger les cellules et le milieu pendant 10 minutes à 2000 centifugal vigueur relative (fcr) à température ambiante, puis aspirer le surnageant et remettre en suspension chaque culot dans 10 ml de PBS. Si le culot d'une infection virale a été étendue en de multiples tubes coniques à l'étape 2.2, ceux-ci peuvent être recombinés dans un tube conique à ce step. Répétez centrifugation et aspiration en option:. Cette pastille peut être conservé à -20 ° C et le protocole poursuivie à une date ultérieure.
3. Deuxième jour, Phase 2: Ultracentrifugation
Gardez le culot cellulaire et tampons LCM sur la glace chaque fois que possible pendant les étapes suivantes.
- Ajouter β-mercaptoéthanol à 0% de glycérol - mémoire tampon LCM. Commencez le refroidissement de la ultracentrifugeuse (pour l'étape 3.9 ci-dessous) en la fixant à 4 ° C et tournant sur le vide.
- Remettre en suspension le culot cellulaire dans 5 ml de glycérol 0% - mémoire tampon LCM jusqu'à ce qu'il ne soit plus touffue. Si nécessaire, le tube vortex de briser n'importe quelle cellule non dissous granulés.
- Extraire en ajoutant 1,5 ml fréon (1,1,2-trichloro-1 ,2,2-trifluoroéthane) et vortex vigoureusement. Immédiatement centrifuger pendant 10 min à 2000 rcf à une température de 4 ° C. Recueillir la couche supérieure avec une pointe de pipette de gros calibre (en évitant l'interface) et transférer dans un nouveau tube. Optionnel: Si la pastille est un solide gélatineux, vous pouvez verser rapidement la couche supérieure dans un nouveau tube.
- Répéter l'extraction fréon et centrifuger à nouveau.
- Recueillir la couche supérieure (~ 5 ml) et transférer dans un tube d'ultracentrifugation polyallomère nouvelle. Ajouter β-mercaptoéthanol au glycérol deux autres - tampons LCM.
- Préparer un dégradé dans le tube à centrifuger polyallomère par sous-jacente de l'extrait Fréon (couche supérieure) avec 3 ml de glycérol à 5% - LCM (couche intermédiaire), puis sous-jacente à nouveau avec 2,5 ml de glycérol 45% - LCM (couche inférieure). Utiliser une pipette fine pour éviter le débordement du contenu du tube.
- Si nécessaire, préparez un tube d'équilibrage équivalente en utilisant les mêmes proportions de tampons LCM.
- Transfert des tubes dans des seaux ultracentrifugation. Solde seaux à 0,1 g de l'autre, en ajoutant doucement supplémentaire glycérol 0% - mémoire tampon LCM à la couche supérieure (~ 50-100 ul à la fois).
- En utilisant une ultracentrifugeuse froid avec une roto godet horizontal ou oscillantr, tourner les échantillons équilibrés pendant 1 heure à 77.000 fcr (par exemple 25.000 rpm pour un rotor SW41Ti) à une température de 4 ° C.
- Au cours de l'ultracentrifugation, faire un crochet verre pour capturer l'ADN flottant à l'étape de précipitation à l'éthanol (4.6). Tenez les deux extrémités d'une pipette Pasteur en verre et de suspendre la partie médiane dessus d'une flamme. Quand une section de verre est presque fondu, tirez doucement pour étirer le verre, l'incliner légèrement pour créer un crochet à angle serré (<90 degrés), et tirez fermement pour sceller la fin. Si la pointe de l'hameçon en verre est ouvert, maintenez la pointe sur la flamme pour que le verre se fond assez pour la refermer.
- Après l'ultracentrifugation, vous devriez voir une mince opaque à granulés avec une tache sombre au milieu. Soigneusement aspirer le liquide du tube, y compris coulures sur les côtés, mais en évitant le culot au fond du tube.
- Ajouter 0,5 ml TNE à la température ambiante.
- Laissez le culot reposer pendant au moins 10 minutes pour permettre l'hydratation de la pastille. Optionnel: Le culot peut aussi remettre en suspension dans du TNE du jour au lendemain, si elles sont conservées à 4 ° C.
4. Troisième jour: Précipitations ADN "Ghost"
- Brisez le culot par pipetage avec une pointe de petit calibre pipette (par exemple une pointe de pipette p200). Ne vous inquiétez pas à propos de cisaillement au niveau de cette étape, parce que l'ADN viral est encore contenue dans des capsides à ce stade. Ajouter les réactifs suivants dans le tube contenant le culot viral, à la température ambiante, à détruire capsides:
- 4,25 ml TNE
- 0,25 ml SDS à 10%
- 2 mg protéinase K
Méfiez-vous de cisaillement à partir d'ici, par exemple, utiliser des pipettes de gros calibre et de ne pas vortexer le matériel viral
- Extraire des protéines virales en ajoutant 5 ml de phénol-chloroforme et commencer immédiatement inverser ou vortex de maintenir une émulsion. Mélanger pendant 10 secondes, puis centrifugerl'émulsion pendant 5 minutes à 2000 rcf à la température ambiante. Recueillir la couche supérieure avec une pointe de pipette de gros calibre, en évitant l'interface et le transfert dans un nouveau tube en option:. Utilisation verrouillage de phase gel (PLG) tubes de cette étape pour éviter la contamination de l'interface.
- Répéter l'extraction phénol-chloroforme et centrifuger à nouveau.
- Après la seconde extraction au phénol-chloroforme, recueillir la couche supérieure dans un tube de 30 ml en verre Corex et refroidir le tube à -20 ° C pendant 10-15 minutes, en prenant soin qu'il ne gèle pas.
- Ajouter 10 ml d'éthanol glacé, recouvrir le tube (par exemple en étirant un morceau de parafilm M sur le dessus), et mélanger par inversion immédiatement. Surveillez un ADN "fantôme" d'apparaître, qui se compose d'un précipité visqueux mince. Inverser le tube doucement pour mélanger encore et provoquer l'ADN précipite collant à se regrouper.
- Utiliser un crochet pour saisir le verre fantôme ADN (s). Soigneusement effacer l'ADN pour enlever l'excès de liquide, puis placez le crochet (pointe vers le bas)dans un tube de 1,5 ml et laisser sécher. Ajouter 0,25 - 0,5 ml de TE pour dissoudre le fantôme d'ADN. Permettre la remise en suspension de l'ADN se dérouler pendant au moins une heure. L'ADN se conserve à 4 ° C. Facultatif: Pour maximiser le rendement de l'ADN, brisez le crochet de verre dans le tube de 1,5 ml, de sorte que la remise en suspension au large des fragments de verre peuvent continuer au fil du temps.
5. Les résultats représentatifs
Avec une grande suffisamment MOI, les cellules doivent atteindre la pleine CPE dans les ~ 18 heures après l'infection (hpi) pour les souches de PRV de type sauvage, et ~ 24 hpi pour type sauvage des souches de HSV-1 (figure 1). Une fois la cellule infectée culot est récolté, les étapes restantes du protocole ne peut être complète en un seul jour long ou réalisé sur deux jours (Figure 2).
Lors de la préparation des crochets en verre pour capturer le fantôme d'ADN, il est important de faire l'angle d'attaque inférieur à 90 degrés, de sorte que plus tard, il peut s'adapter à la base d'un tube de 1,5 ml (figure 3). Scelle la pointe de ce crochet empêche la perte de l'ADN danscôté de la pipette.
Fantômes d'ADN se former pendant l'étape de précipitation et apparaîtra comme blancs, les discussions filant (figure 4). Ces granulats et le bâton à l'autre et / ou le crochet de verre utilisée pour capturer l'ADN. Ainsi, le crochet même verre peuvent être utilisés pour rassembler plusieurs threads d'ADN à partir de la solution de précipitation.
| Choisir la colonne en fonction du volume désiré: | 10 ml | 15 ml | 20 ml | 40 ml |
| 1M KCl | 1,3 ml | 1,9 ml | 2,5 ml | 5 ml |
| 1M Tris (pH 7,4) | 300 pl | 450 ul | 600 pi | 1,2 ml |
| 1M MgCl 2 | 50 pi | 75 pi | 100 pi | 200 pi |
| 0,5 EDTA | 10 pl | 15 pi | 20 pi | 40 pi |
| NP40/IGEPAL | 50 pi | 75 pi | 100 pi | 200 pi |
| β-mercaptoéthanol (ajouter immédiatement avant utilisation) | 4,3 pl | 6,5 pl | 8,6 pl | 17,2 ul |
| Utiliser des proportions pour le glycérol pour cent désirée (0, 5, ou 45%) | ||||
| 0% de glycérol | Aucun | Aucun | Aucun | Aucun |
| eau | 8,3 ml | 12,5 ml | 16,7 ml | 33,4 ml |
| Glycérol à 5% | 0,5 ml | 0,8 ml | 1,0 ml | 2,0 ml |
| eau | 7,8 ml | 11,7 ml | 15,7 ml | 31,4 ml |
| G 45%lycerol | 4,5 ml | 6,8 ml | 9,0 ml | 18,0 ml |
| eau | 3,8 ml | 5,7 ml | 7,7 ml | 15,4 ml |
Tableau 1. Recettes pour la préparation de tampons LCM
| Pour un volume de 50 ml |
| 5 ml de NaCl 1M |
| 2,5 ml Tris 1 M, pH 7,5 |
| 1 ml d'EDTA 0,5 |
| 41,5 ml de H 2 O |
Tableau 2. Préparation de TNE (0,1 M de NaCl, 50 mM de Tris, pH 7,5, EDTA 10 mM)
| Pour 1 litres |
| 0,2 g de KCl |
| 0,2 g KH 2 PO 4 |
| 8 g de NaCl |
| 1,15 g de Na 2 HPO 4 |
| H 2 O à 1L |
Tableau 3. Préparation de PBS (2,7 mM KCl, 1,5 mM KH 2 PO 4; 137 mM NaCl; 8,1 mM Na 2 HPO 4, pH 7,0)
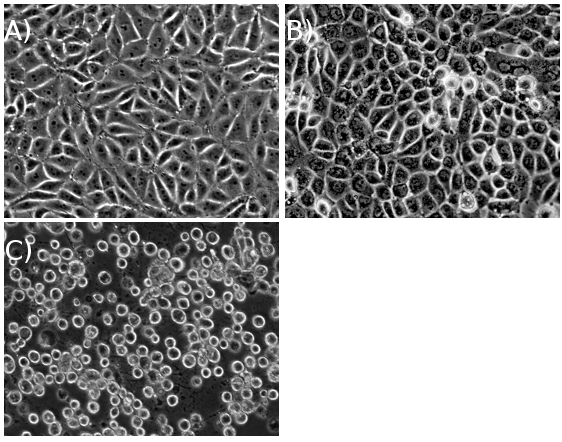
Figure 1. PK15 cellules (A) avant l'infection, (B) à mi-chemin à travers l'infection, et (C) à l'effet cytopathogène (CPE), après une multiplicité d'infection.

Figure 2. Vue d'ensemble du protocole.

Figure 3. Trois exemples représentatifs de verre crochets utilisés pour recueillir les fantômes d'ADN viral.

Figure 4. Exemple représentatif d'un ADN "fantôme" bien formé et abondante.
Discussion
Certaines parties de ce protocole ont été initialement développées pour l'isolement d'ADN viral dans des conditions de biosécurité L4, mais elle s'adapte aussi bien aux non-BSL conditions. 4 Nous communément utiliser ce protocole pour isoler l'ADN du PRV alpha-herpèsvirus et le HSV-1, qui possèdent des génomes d'ADN enfermé dans une capside protéique et entouré par une enveloppe lipidique. 7,8 Cependant, il est probable directement adaptable à d'autres virus à ADN de grande taille, dont le bêta-et gamma-herpèsvirus et les adénovirus. Extractions similaires sont couramment utilisés pour les virus à ARN et 9.
La robustesse de ces résultats préparation de l'ADN susceptibles de multiples méthodes de purification qui sont inclus. Les premières extractions membranes lipidiques Fréon dénaturer, séparation des composants cellulaires et publie également l'enveloppe lipidique et les protéines tégumentaires externes qui entourent capsides herpesvirus. Elle est suivie par ultracentrifugation, où les nucléocapsides virales lourds granulés pardeux coussins de gradient, ce qui les sépare de la plupart des autres composants cellulaires. Ces capsides sont remises en suspension, et l'ADN viral nucléocapside est libérée par rupture des capsides avec la protéinase K et du détergent. Deux extractions au phénol-chloroforme enlever complètement les composants nucléocapside et les protéines cellulaires restants. Enfin, la précipitation des grands génomes d'ADN viral dans une solution réduit report de sels ou de contaminants particulaires.
Cette procédure d'isoler l'ADN viral nucléocapside offre abondante, matériau de haute pureté pour des applications en aval. La combinaison de l'extraction multiple et les étapes de centrifugation distingue ce protocole d'extraction simples mono-protocoles qui laissent des débris cellulaires ou plusieurs produits protéiques dégradées avec l'ADN 10. Contaminants protéiques peuvent diminuer la stabilité au stockage de l'ADN viral, et réduire l'efficacité de la transfection dans des cellules. Contaminants d'ADN cellulaire impact négatif sur les réactions de PCR d'uneprotocoles d séquençage à haut débit. 1 Le rendement de l'ADN de ce protocole est également élevée, le rendant bien adapté au polymorphisme de longueur des fragments de restriction (RFLP) et Southern blot. 5,11 Une préparation nucléocapside fournit suffisamment de matière pour l'ensemble de ces analyses.
La quantité de cellules utilisées pour l'infection affecte le rendement final de l'ADN viral. Pour les souches de PRV avec un taux de croissance de type sauvage, de plats d'entrée de 5-10 cellules PK-15 (une de 15 cm de diamètre plat détient environ 8 x 10 6 cellules), on obtient généralement de 250 à 500 pg d'ADN viral. De mutants viraux avec un rendement diminué de virions infectieux, le matériel utilisé doit être mis à l'échelle à la hausse en conséquence. Doubler le nombre de plats d'entrée sera environ le double du rendement, et peuvent être traitées avec les mêmes quantités de réactifs décrits ici. Pour augmenter l'entrée au-delà de ce point, nous vous recommandons de séparer le matériau d'entrée en deux flux parallèles de traitement (e.g. deux pastilles cellulaires et deux tubes d'extraction) par étape 3.13. Deux pastilles d'ultracentrifugation de la même souche virale peuvent être combinés dans un tube à cette étape de remise en suspension. Si l'ADN ne parvient pas à proprement former un fantôme à la fin de la procédure, l'approche de dépannage premier est d'augmenter la quantité d'entrée des cellules, ce qui augmente le rendement en ADN et d'améliorer sa précipitation.
D'autres conditions peuvent également affecter le succès de la formation fantôme. Par exemple, il est important de temps de la récolte des infections à un moment où nucléocapsides virales sont abondantes, mais la plupart sont intracellulaire ou associé aux cellules. Virions libérés dans le milieu ne seront pas capturées par cette procédure, et donc la récolte des cellules trop tard dans l'infection (par exemple, lorsque CPE a progressé au point où les cellules se détachent de l'antenne) permettra de réduire le rendement en ADN potentiel et la formation fantôme. Il est également important pour dissoudre complètement la cellule récolté culot dans un tampon LCM (étape3.2) pour profiter pleinement de l'étape d'extraction suivante, ce qui nécessite la remise en suspension de plus de temps et de soins si le culot cellulaire a été congelée à l'étape précédente. Petits détails de procédure affecter le rendement de l'ADN ainsi. Alors que les étapes 3 et 4 peut être réalisée sans l'aide de solutions réfrigérées et de garder tous les tubes sur la glace, le taux de réussite des images fantômes ADN est beaucoup plus élevé lorsque les réactifs sont conservés au froid. De même, la stabilité de β-mercaptoéthanol dans des baisses solution au fil du temps, et donc il ajoutant aux solutions LCM immédiatement avant l'utilisation optimise son rendement en ADN réduction de la capacité, l'amélioration de la perturbation des protéines et l'augmentation et la formation fantôme. Une attention particulière à ces détails permettra d'améliorer la production de l'ADN viral de haute qualité.
Parce que l'ADN viral est visqueux, la solution obtenue peut être hétérogène. Accorder plus de temps pour la remise en suspension du crochet de verre augmente le rendement en ADN utile et améliore l'homogénéité de la solution. Spectrophotométrie standardpeut être utilisé pour quantifier l'ADN rendement et la pureté. Fluorimétrie ADN prévoit des mesures encore plus précises de rendement en ADN (ex.: Invitrogen PicoGreen ADNdb tache).
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Les auteurs ont apprécié les contributions de Greg Smith, Lisa Pomeranz, Matt Lyman, Marlies Eldridge, Halina Staniszewska Goraczniak, et des membres du laboratoire Enquist à peaufiner ce protocole.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Fréon (1,1,2-trichloro-1 ,2,2-trifluoroéthane) | Pêcheur | T178-4 | Vérifiez auprès de votre institution pour les directives concernant l'élimination appropriée des déchets contenant du fréon, ou de voir Mendez et al. des solutions de rechange potentielles Fréon 9. |
| Tubes à verrouillage de phase gel, lourd capacité 15ml | 5 PRIME | 2302850 | En option |
| Tubes ultracentrifugation polyallomère | Beckman * | 331372 * | * Sélectionnez tubes appropriés pour votre propre ultracentrifugeuse; ceux-ci sont inclus à titre d'exemple |
| NP-40 / IGEPAL | Sigma | Je-3021 | |
| Cellules PK-15 | ATCC | CCL-33 | |
| Les cellules Vero | ATCC | CCL-81 | |
| PBS | HyClone | SH30028.03 |
Références
- Szpara, M. L., Parsons, L., Enquist, L. W. Sequence variability in clinical and laboratory isolates of herpes simplex virus 1 reveals new mutations. J Virol. 84, 5303-5313 (2010).
- Banfield, B. W., Kaufman, J. D., Randall, J. A., Pickard, G. E. Development of pseudorabies virus strains expressing red fluorescent proteins: new tools for multisynaptic labeling applications. J Virol. 77, 10106-10112 (2003).
- Kobiler, O., Lipman, Y., Therkelsen, K., Daubechies, I., Enquist, L. W. Herpesviruses carrying a Brainbow cassette reveal replication and expression of limited numbers of incoming genomes. Nat Commun. 1, 146-146 (2010).
- Enquist, L. W., Madden, M. J., Schiop-Stanley, P., Vande Woude, G. F. Cloning of herpes simplex type 1 DNA fragments in a bacteriophage lambda vector. Science. 203, 541-544 (1979).
- Smith, G. A., Enquist, L. W. Construction and transposon mutagenesis in Escherichia coli of a full-length infectious clone of pseudorabies virus, an alphaherpesvirus. J Virol. 73, 6405-6414 (1999).
- Flint, S. J., Enquist, L. W., Racaniello, V. R., Skalka, A. M. Principles of virology. , 3rd edn, ASM Press. (2008).
- Pomeranz, L. E., Reynolds, A. E., Hengartner, C. J. Molecular biology of pseudorabies virus: impact on neurovirology and veterinary medicine. Microbiol Mol Biol Rev. 69, 462-500 (2005).
- Roizman, B., Pellett, P. E. Fields Virology. Knipe, D. M., Howley, P. M. , 2 ed, Lippincott Williams & Wilkins. 2381-2397 (2001).
- Mendez, I. I., Hermann, L. L., Hazelton, P. R., Coombs, K. M. A comparative analysis of freon substitutes in the purification of reovirus and calicivirus. J Virol Methods. 90, 59-67 (2000).
- Gharabaghi, F., Aymard, M., Trotemann, P., Gerdil, C. A rapid and simplified micromethod for subtyping varicella-zoster virus. J Med Virol. 31, 129-134 (1990).
- Granstedt, A. E., Szpara, M. L., Kuhn, B., Wang, S. S., Enquist, L. W. Fluorescence-based monitoring of in vivo neural activity using a circuit-tracing pseudorabies virus. PLoS One. 4, e6923-e6923 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon