Method Article
Screening CRISPR a livello di genoma per svelare geni radiosensibili e radioresistenti
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Viene fornito un approccio meticoloso e strutturato per selezionare geni resistenti e sensibili alle radiazioni attraverso l'applicazione di un metodo di screening CRISPR/Cas9 per l'intero genoma. Questo protocollo ha anche il potenziale per fungere da quadro versatile per altri sforzi di ricerca che studiano i meccanismi di resistenza ai farmaci chimici somministrati clinicamente.
Abstract
Il sistema CRISPR-Cas9 è stato sfruttato e riutilizzato in un potente strumento di editing del genoma. Sfruttando questa tecnologia, i ricercatori possono tagliare, incollare e persino riscrivere con precisione le sequenze di DNA all'interno delle cellule viventi. Tuttavia, l'applicazione della tecnologia dello schermo CRISPR va ben oltre la semplice sperimentazione. Funge da strumento fondamentale nella lotta contro le malattie genetiche, sezionando sistematicamente complessi paesaggi genetici, consentendo ai ricercatori di svelare i meccanismi molecolari alla base dei fenomeni biologici e consentendo agli scienziati di identificare e indirizzare le cause alla radice di malattie come il cancro, la fibrosi cistica e l'anemia falciforme. Tra tutti, il cancro rappresenta una sfida formidabile per la medicina, stimolando gli sforzi di eradicazione. La radioterapia, come trattamento tradizionale, dà risultati ma ha dei limiti. Sradica le cellule tumorali ma danneggia anche i tessuti sani, causando effetti avversi che riducono la qualità della vita. Inoltre, non tutte le cellule tumorali rispondono alla radioterapia e alcune possono sviluppare resistenza, peggiorando la condizione. Per affrontare questo problema, viene introdotta una tecnologia completa di screening CRISPR dell'intero genoma, in quanto consente l'identificazione efficiente di geni radiosensibili e radioresistenti, facendo così progredire il campo della ricerca e del trattamento del cancro. È stato condotto uno screening CRISPR a livello di genoma in cellule di adenocarcinoma polmonare esposte a irradiazione seguendo il protocollo descritto, attraverso il quale sono stati identificati sia i geni associati alla radioresistenza che quelli associati alla radiosensibilità.
Introduzione
L'indagine dei fenomeni biologici è intrinsecamente intrecciata con lo studio dei comportamenti cellulari e, a sua volta, l'esame dei comportamenti cellulari è fondamentalmente connesso all'esplorazione del suo genoma. Con la continua evoluzione della tecnologia moderna, i ricercatori medici stanno progressivamente reindirizzando la loro attenzione verso l'alterazione dei comportamenti cellulari attraverso l'editing genetico al fine di migliorare i risultati del trattamento di varie malattie. A questo proposito, la tecnologia CRISPR (clustered regularly interspaced short palindromic repeats) è emersa come uno strumento rivoluzionario per l'editing del genoma grazie alla sua applicazione relativamente semplice1. Il sistema CRISPR-Cas9 è costituito da nucleasi Cas9 e RNA a guida singola (sgRNA), che riconosce e si lega specificamente alla sequenza di DNA bersaglio, guidando la nucleasi Cas9 a tagliare in quella posizione, provocando una rottura a doppio filamento (DSB) nel DNA del genoma 2,3,4. Inoltre, l'introduzione di altre sostanze può portare a inserzioni, delezioni o mutazioni specifiche nel genoma, consentendo un editing genetico mirato.
Nella ricerca di genomica funzionale, lo screening dell'interferenza dell'RNA (RNAi) era un tempo un metodo ampiamente utilizzato per condurre esperimenti di perdita di funzione su larga scala per studiare i ruoli dei geni nel cancro. La tecnologia RNAi studia la funzione genica silenziando in modo specifico i geni bersaglio, aiutando i ricercatori a identificare i fattori oncogenici critici. Tuttavia, è limitato da effetti fuori bersaglio e da un'efficienza incompleta del knockdown genico. Gli effetti off-target possono portare al silenziamento di altri geni non bersaglio, compromettendo così l'accuratezza e l'affidabilità dei risultati sperimentali 1,2. Inoltre, l'RNAi mostra una bassa efficienza di knockdown per alcuni geni, non riuscendo potenzialmente a sopprimere completamente l'espressione genica bersaglio. A differenza dei tradizionali screening RNAi, lo screening CRISPR dimostra una maggiore specificità ed efficienza3. Questa tecnologia non solo consente l'editing preciso di geni specifici, ma consente anche lo screening su larga scala dell'intero genoma, fornendo un solido supporto per la ricerca sulla funzione genica. La tecnologia di screening CRISPR, un potente strumento di editing genetico basato sul sistema CRISPR-Cas9, viene utilizzata per lo screening efficiente e la rivelazione di funzioni sconosciute di geni specifici nelle cellule 5,6,7,8. I ricercatori progettano sgRNA in lotti per geni o regioni geniche specifici e preparano librerie di sgRNA corrispondenti con precisione e rigore, garantendone l'integrità e la funzionalità9. Queste librerie di sgRNA vengono quindi incapsulate in particelle lentivirali, che vengono utilizzate per infettare in modo efficiente le cellule ospiti. Dopo l'avvenuta infezione, le cellule infette vengono coltivate in condizioni di screening definite personalmente. Al momento dello screening, viene estratto il DNA genomico delle cellule sottoposte a screening, mantenendo elevati standard di purezza e quantità. Successivamente, le regioni bersaglio di interesse per l'sgRNA sono sottoposte ad amplificazione PCR, un processo che replica accuratamente i segmenti desiderati di acidi nucleici 3,9. Infine, viene eseguito il sequenziamento ad alto rendimento sui frammenti di DNA amplificati, consentendo un'analisi completa ed efficiente delle regioni bersaglio, fornendo così preziose informazioni sulla funzione e sul comportamento dei geni oggetto dello studio4.
Il cancro rappresenta una formidabile minaccia per la salute umana in quanto malattia complessa. In tutto il mondo, ricercatori e medici stanno compiendo sforzi concertati per svelare i meccanismi molecolari della cancerogenesi e sviluppare nuove strategie terapeutiche. Sono state stabilite collaborazioni internazionali per accelerare la traduzione dei risultati della ricerca di base in applicazioni cliniche, con l'obiettivo finale di migliorare i risultati dei pazienti. Sasmal et al. hanno proposto una strategia di assemblaggio bioortogonale basata su un sistema sintetico host-guest per il targeting preciso delle cellule tumorali metastatiche, che ha aiutato in modo significativo dozzine di scienziati a far progredire le tecnologie mediche. Il loro eccezionale lavoro di ricerca ha un'elevata innovazione e intuizioni uniche, che apportano contributi significativi alla comunità scientifica10. Il cancro è caratterizzato da uno stato tumultuoso di instabilità genomica, derivante dalla regolazione erratica delle risposte al danno del DNA 11-14. Il danno al DNA include difetti a singolo nucleotide, rotture a singolo filamento e DSB. La ricombinazione omologa (HR) e l'unione di estremità non omologa (NHEJ) partecipano alla riparazione di DSB in diversi stadi 15,16,17. Su questa base, la radioterapia è emersa come una valida opzione terapeutica, che utilizza raggi ad alta energia (come i raggi X e i raggi γ) per irradiare il tessuto tumorale, causando danni al DNA nelle cellule tumorali, interrompendo così la loro crescita e proliferazione18. Tuttavia, la radioterapia non sempre produce gli effetti desiderati in una percentuale significativa di pazienti oncologici, potenzialmente derivanti da danni ai tessuti paracancerosi e limitazioni imposte dalle caratteristiche intrinseche del tumore, come la bassa sensibilità alla radioterapia 19,20,21.
In teoria, qualsiasi tipo di cellula può essere utilizzato per uno schermo CRISPR. Tuttavia, mantenere una rappresentanza sufficiente nelle popolazioni mutate richiede un gran numero di cellule iniziali. I tipi di cellule con bassa abbondanza non sono particolarmente adatti per lo screening dell'intero genoma. Per quanto riguarda la scelta della libreria, la maggior parte delle librerie contiene 3-6 gRNA per gene bersaglio e mantenere la distribuzione di ciascun gRNA all'interno della popolazione è fondamentale18. La perdita di rappresentazione dovuta all'arricchimento o all'esaurimento di specifici gRNA può portare a una distribuzione non uniforme dei risultati. Per risolvere questo problema, potrebbe essere preferibile optare per librerie CRISPR disponibili in commercio che sono state testate sul mercato20. In vitro Lo screening CRISPR che utilizza linee cellulari tumorali omogenee potrebbe non catturare completamente l'eterogeneità genetica ed epigenetica dei tumori in vivo . Mentre lo screening in vitro ha rivelato geni chiave coinvolti nella riparazione dei danni al DNA e nella segnalazione autocrina indotta dalle radiazioni, non ha replicato completamente il microambiente tumorale, tra cui la radioresistenza indotta dall'ipossia (tramite ROS, adattamento metabolico e autofagia), gli effetti paracrini immuno-mediati e la modulazione delle citochine dipendente dalla MEC. Prima di utilizzare lo schermo CRISPR per esplorare i geni associati alla sensibilità o alla resistenza alle radiazioni, questi fattori devono essere attentamente considerati. Alla luce dell'attuale panorama terapeutico, è urgente identificare e studiare a fondo i fattori associati alla radioresistenza e alla radiosensibilità per migliorare efficacemente l'efficacia della radioterapia22. Dato il vantaggio chiave dello screening CRISPR nello studio delle funzioni di geni sconosciuti, viene fornita una tecnologia di screening CRISPR dell'intero genoma sistematicamente dettagliata per identificare in modo efficiente i geni radiosensibili e radioresistenti.
Protocollo
I reagenti e le attrezzature utilizzate in questo studio sono elencati nella Tabella dei materiali.
1. Selezione di una dose appropriata di radiazioni
- Preparazione e placcatura delle celle aderenti
- Regolare la densità cellulare a 5 x 105 cellule per mL con il terreno di coltura cellulare completo RPMI 1640 contenente il 10% di FBS per il passaggio e la crescita cellulare. Distribuire le cellule in piastre di coltura da 3,5 cm per la radiazione a dosi diverse. Aggiungere 2 mL (1 x 106 cellule) a ciascuna piastra e incubare per una notte a 37 °C con CO2 al 5%.
- Applicazione di diverse dosi di radiazioni
- Numerare i piatti di coltura da 3,5 cm da 1 a 5. Utilizza il Gruppo #1 come gruppo di controllo, con i restanti 4 gruppi designati come gruppi di trattamento.
- Sigillare i bordi delle piastre a 6 pozzetti con una membrana sigillante per gruppi di trattamento e somministrare dosi di radiazioni rispettivamente di 2, 4, 6 e 8 Gy. Dopo il trattamento, rimettere le piastre nell'incubatrice.
- Placcatura di linee cellulari dopo irradiazione
- Preparare piastre a 6 e 96 pozzetti. Lavare una volta le cellule nelle piastre di coltura da 3,5 cm con PBS, digerire le cellule in crescita logaritmica con lo 0,25% di tripsina e regolare la densità cellulare a 1 x 105 cellule per mL con il terreno completo RPMI 1640 contenente il 10% di FBS.
- Seminare 10 μL/pozzetto (1.000 cellule/100 μL) nelle piastre a 6 pozzetti a 3 repliche per ogni dose di radiazioni. Aggiungere 2 mL di terreno completo a ciascun pozzetto e contare dopo 14 giorni (quando ogni gruppo clone ha circa 50 cellule).
- Seminare 30 μL/pozzetto (3.000 cellule/100 μL) nelle piastre a 96 pozzetti a 5 repliche per ogni dose di radiazioni. Aggiungere 70 μl di terreno completo a ciascun pozzetto e misurare la vitalità cellulare dopo 72 ore.
- Misurazione della vitalità cellulare
NOTA: Tasso di inibizione clonogenica = 1 - (numero di cloni nel gruppo di trattamento/numero di cloni nel gruppo di controllo) x 100%. Il test CCK-8 utilizza un sale di tetrazolio solubile in acqua (WST-8) che può essere ridotto dalle deidrogenasi cellulari per generare un prodotto di formazan giallo altamente solubile in acqua11. La quantità di formazan prodotta è direttamente proporzionale al numero di cellule vitali e la sua densità ottica (misurata a 450 nm di lunghezza d'onda) riflette accuratamente l'attività metabolica cellulare e lo stato proliferativo11,13. Sulla base di questo principio consolidato, il test CCK-8 è stato ampiamente adottato per varie applicazioni, tra cui i saggi di proliferazione cellulare e i test di sensibilità ai farmaci tumorali. In questo protocollo, il test CCK-8 viene impiegato per valutare sistematicamente la vitalità cellulare sotto diverse dosi di radiazioni.- Miscelare il reagente CCK8 con il terreno RPMI 1640 senza FBS in un rapporto 1:9. Scartare il terreno nelle piastre a 96 pozzetti e aggiungere 100 μl di terreno contenente CCK8 a ciascun pozzetto.
- Incubare al buio per 1 ora e misurare il valore OD a 450 nm utilizzando un lettore di micropiastre.
NOTA: Tasso di sopravvivenza cellulare = [(Valore OD del gruppo di trattamento - Valore OD del pozzetto bianco) / (Valore OD del gruppo di controllo - Valore OD del pozzetto bianco)] x 100%
- Selezione della dose di radiazioni
- Integrare il tasso di inibizione clonogenica con il tasso di sopravvivenza cellulare al fine di selezionare una dose di radiazioni appropriata in base alle esigenze di ricerca. Selezionare una dose di radiazioni con un tasso di inibizione del 50% per lo screening di geni radioresistenti e radiosensibili.
2. Selezione del MOI e della concentrazione di puromicina appropriati
- Placcatura delle cellule aderenti
- Regolare la densità cellulare a 3 x 105 cellule per mL e inoculare 1 mL (3 x 105 cellule) per pozzetto in una piastra a 12 pozzetti. Incubare la piastra per una notte a 37 °C con CO2 al 5%.
- Infezione lentivirale
- Utilizzare una pipetta per aspirare il terreno dalla piastra a 12 pozzetti e sostituirlo con 1 mL di terreno completo RPMI 1640 contenente il 10% di FBS. Preparare una libreria lentivirale CRISPR dell'intero genoma di qualità controllata (si consigliano librerie disponibili in commercio) e impostare un gradiente di concentrazione logaritmico (ad esempio, 0-10-50-100-200-400-800)4,5,6,7,8.
- Aggiungere la quantità corrispondente di lentivirus a 2 μL/piatto di polibrene ed equilibrare a temperatura ambiente per 5 minuti. Versare lentamente la miscela di lentivirus e polibrene in ogni pozzetto, mescolare bene e incubare per una notte a 37 °C con il 5% di CO2.
- Determinazione della concentrazione minima di puromicina per l'uccisione cellulare
- Regolare la densità delle cellule parentali a 3 x 105 cellule/mL e inoculare 1 mL (3 x 105 cellule) per pozzetto in una piastra a 12 pozzetti. Incubare la piastra per una notte a 37 °C con CO2 al 5%.
- Aggiungere la puromicina a ciascun pozzetto della piastra a 12 pozzetti con un gradiente di concentrazione di 0-0,1-0,2-0,5-1-2-4-8 μM. Contare le cellule dopo 72 ore. La concentrazione minima che uccide tutte le cellule nel pozzetto è la concentrazione minima di puromicina per l'uccisione cellulare, che verrà utilizzata per il successivo screening delle cellule infettate viralmente.
- Selezione della puromicina dopo l'infezione
- Il secondo giorno dopo l'infezione, aspirare il terreno da ciascun pozzetto e sostituirlo con 1 mL di terreno completo RPMI 1640 contenente il 10% di FBS. Continuare la coltura per 48 h.
- Dopo 72 ore di infezione, sostituire il terreno esistente con un terreno completo contenente la concentrazione minima di puromicina per l'uccisione cellulare. Impostare 2 pozzetti nella piastra a 12 pozzetti senza infezione lentivirale come controlli negativi e positivi. Trattare il controllo negativo con la puromicina e lasciare il controllo positivo non trattato. Continuare la coltura per 72 ore.
- Dopo 72 ore di selezione della puromicina, tutte le cellule parentali nel controllo negativo saranno morte, mentre le cellule parentali nel controllo positivo mostreranno una morte minima. Calcola il MOI per ogni pozzetto.
NOTA: MOI = (Numero di cellule nel gruppo infetto da virus / Numero di cellule nel gruppo di controllo positivo) x 100%. Utilizzare la concentrazione del virus con un MOI di ~0,3 per lo screening successivo.
3. Infezione della libreria lentivirale CRISPR a livello di genoma
- Semina di linee cellulari aderenti
- Regolare la densità cellulare a 1 x 107 cellule per mL in terreno completo RPMI 1640 contenente il 10% di FBS per il passaggio e la crescita cellulare. Inoculare 1 mL di cellule (1 x 107 cellule) in ciascuna piastra di coltura da 15 cm a 37 °C con CO2 al 5% per 8 ore. Una volta che le cellule hanno aderito e raggiunto una confluenza del 70%-80%, erano pronte per l'infezione virale (facendo un numero di copie di circa 500).
- Infezione lentivirale
- Aspirare il terreno dalle piastre di coltura da 15 cm e sostituirlo con 15 mL di terreno completo RPMI 1640 contenente il 10% di FBS. Preparare una libreria lentivirale CRISPR dell'intero genoma di qualità controllata.
- Aggiungere la quantità corrispondente di lentivirus con il MOI = da 0,3 a 30 μL/piastra di polibrene ed equilibrare a temperatura ambiente per 5 minuti, far gocciolare lentamente la miscela di lentivirus e polibrene nei piatti di coltura da 15 cm, mescolare bene e incubare a 37 °C con il 5% di CO2 per una notte. Contemporaneamente, preparare una piastra di coltura di 15 cm con la cellula parentale come contrasto.
- Selezione della puromicina dopo l'infezione
- Il secondo giorno dopo l'infezione virale, aspirare il terreno dalle piastre di coltura da 15 cm e sostituirlo con 15 mL di terreno completo RPMI 1640 contenente il 10% di FBS. Continuare la coltura per 48 h.
- A 72 ore dall'infezione, sostituire il terreno con un terreno completo contenente la concentrazione letale minima di puromicina. Trattare le cellule parentali non infette come un controllo negativo allo stesso modo e continuare la coltura per 72 ore. Dopo 72 ore di selezione della puromicina, le cellule parentali nel gruppo di controllo negativo saranno tutte uccise e le cellule sopravvissute all'infezione lentivirale saranno considerate infettate con successo.
- Estrazione del genoma del giorno 0
- Digerire le cellule da una piastra di coltura da 15 cm utilizzando tripsina allo 0,25%, risospendere in un terreno completo RPMI 1640 contenente il 10% di FBS e contare il numero di cellule. Etichettare questo esempio come Giorno 0.
- Centrifugare a 300 x g per 5 minuti (a temperatura ambiente) ed eliminare il surnatante. Risospendere in 1 mL di PBS, centrifugare a 300 x g per 5 minuti ed eliminare il surnatante. Estrai il DNA genomico del giorno 0, misura la concentrazione e la purezza del DNA utilizzando uno spettrofotometro UV a nanogoccia.
- Per il rilevamento dell'integrità dell'sgRNA, utilizzare l'elettroforesi su gel di agarosio per analizzare la libreria di sgRNA amplificata, assicurandosi che le bande fossero chiare e non si degradassero in modo significativo, preservando così l'integrità della libreria23. Per la valutazione della copertura della libreria lentivirale, utilizzare la tecnologia di sequenziamento profondo per condurre analisi di sequenziamento sugli sgRNA all'interno della libreria 24,25.
- Nello screening CRISPR, l'amplificazione PCR funge da verifica preliminare dell'integrità del frammento di sgRNA e della qualità della libreria6. Per analizzare in modo più completo la copertura della libreria e i modelli di distribuzione degli sgRNA e per garantire un'adeguata rappresentazione degli sgRNA per ciascun gene bersaglio durante lo screening, eseguire l'NGS per approfondimenti sulla robustezza dello schermo9.
NOTA: Rilevando i cambiamenti nell'abbondanza di sgRNA pre e post-screening e identificando la potenziale integrazione off-target di sgRNA o la contaminazione della libreria, l'accuratezza e l'affidabilità dello screening CRISPR potrebbero essere ulteriormente migliorate. Questi campioni del Giorno 0 fungono da controlli negativi cruciali e vengono sottoposti a una valutazione completa della qualità attraverso l'amplificazione PCR e NGS per raggiungere 2 obiettivi chiave: (1) conferma della deplezione genica essenziale (che indica un'adeguata rappresentazione della libreria) e (2) dimostrazione di un'espressione stabile in geni non essenziali (stabilendo condizioni di base sperimentali)9. - Garantire che la copertura raggiunga il livello previsto per garantire la diversità e la rappresentatività della biblioteca ed evitare distorsioni nel processo di screening. Per la misurazione dell'efficienza dell'infezione, utilizzare l'imaging a fluorescenza per valutare l'efficienza dell'infezione, assicurandosi che soddisfi i requisiti dell'esperimento.
4. Applicazione delle radiazioni come condizione di schermatura
- Raggruppamento
- Regolare la densità delle cellule infettate da CRISPR a 1 x 107 cellule per ml e inoculare 1 ml in ciascuna piastra di Petri da 15 cm. Incubare per una notte a 37 °C con CO2 al 5%.
- Radioterapia
- Dividi casualmente le cellule in 2 gruppi: un gruppo di trattamento e un gruppo di controllo, con 6 piatti per gruppo. Somministrare una dose appropriata di radiazioni alle cellule del gruppo di trattamento, lasciando le cellule del gruppo di controllo non trattate per propagarsi normalmente. Dopo l'irradiazione, continuare a incubare a 37 °C con CO2 al 5% per 7 giorni.
- Nella seconda settimana, ripetere la somministrazione di una dose appropriata di radiazioni alle cellule del gruppo di trattamento, lasciando le cellule del gruppo di controllo non trattate e lasciandole propagare normalmente. Dopo la radioterapia, continuare a incubare a 37 °C con CO2 al 5% per altri 7 giorni.
5. Estrazione e sequenziamento del genoma
- Estrazione del giorno 14
- Dopo 14 giorni di trattamento, digerire le cellule dei gruppi di trattamento e di controllo con tripsina allo 0,25%, risospenderle utilizzando un terreno RPMI 1640 completo contenente il 10% di FBS ed etichettarle come Giorno 14-RT o Giorno 14-NC.
- Centrifugare le cellule a 300 x g per 5 minuti e scartare il surnatante. Risospendere in 1 mL di PBS, centrifugare a 300 x g per 5 minuti ed eliminare nuovamente il surnatante. Estrai il DNA genomico del giorno 14 e rileva la concentrazione e la purezza del DNA utilizzando la spettroscopia di assorbanza UV nanodrop.
- Amplificazione PCR
- Preparare i primer corrispondenti della sequenza (vedere la tabella dei materiali) e diluirli a 10 μM.Impostare un sistema di reazione da 20 μl aggiungendo i reagenti in una provetta da microcentrifuga sterile, centrifugare per 5 s a 300 x g e mescolare bene. Impostare i parametri dello strumento PCR in base alla condizione di amplificazione per ottenere prodotti PCR.
- Rilevamento dell'elettroforesi su gel di agarosio
- Preparare una piastra di colata in gel, sigillare i bordi dello stampo con agarosio, inserire il pettine e preparare un gel di agarosio di concentrazione appropriata in base alla lunghezza del DNA del campione.
- Pesare accuratamente una certa quantità di polvere di agarosio, aggiungere una quantità appropriata di tampone per elettroforesi, mescolare bene e scaldare in un forno a microonde per scioglierlo. Dopo un leggero raffreddamento, aggiungere una quantità adeguata di colorante per acidi nucleici, mescolare delicatamente e versare lentamente nello stampo per colata in gel. Lasciare solidificare il gel per 30 minuti.
- Rimuovere il pettine e aggiungere una quantità adeguata di tampone per elettroforesi al serbatoio per elettroforesi fino a coprire il gel. Aggiungere una quantità appropriata di tampone di caricamento al campione di DNA, mescolare bene e utilizzare una pipetta per aggiungere lentamente la miscela al pozzetto del campione.
- Impostare la tensione appropriata in base alle dimensioni del frammento di DNA e alla concentrazione del gel di agarosio. Dopo l'elettroforesi, rimuovere con cautela il gel e posizionarlo in un gel imager per osservare i risultati e verificare se l'amplificazione PCR ha avuto successo.
- Sequenziamento Illumina
- Raccogli il DNA genomico dai tre gruppi (gruppo del giorno 0, gruppo di controllo e gruppo di trattamento) e invialo a un'azienda per la costruzione della libreria e il sequenziamento Illumina. Eseguire analisi bioinformatiche e visualizzazione dei risultati del sequenziamento per ottenere geni sensibili alla radioterapia e resistenti alla radioterapia26.
- Impostare più esperimenti ripetuti per l'analisi dei dati e condurre analisi statistiche sui risultati sperimentali per garantire la coerenza e la riproducibilità dei dati27,28.
- Valutazione della qualità dei dati di sequenziamento
- Sottoporre i dati grezzi a rigorosi meccanismi di filtraggio e controllo della qualità per garantire la precisione delle informazioni di sequenziamento5. Calcola il punteggio Phred (Qphred) per ciascun nucleotide in base al tasso di errore di sequenziamento, utilizzando un algoritmo di conversione specificato e un modello che valuta la probabilità che si verifichi un errore durante la chiamata di base28.
- Mantenere il tasso di errore di sequenziamento per ciascuna posizione di base al di sotto dell'1% (equivalente a una soglia Q30) e assicurarsi che almeno l'80% dei dati di sequenziamento raggiunga questo standard Q30 per supportare le successive procedure analitiche.
- Elaborazione dei dati e controllo qualità a livello di campione
NOTA: Per i dettagli si rimanda ai rapporti precedentemente pubblicati29,30.- Allinea le letture filtrate dai dati di sequenziamento con le sequenze della libreria sgRNA. Riportare le statistiche, incluso il conteggio degli sgRNA nella libreria con allineamento perfetto, l'abbondanza media di sgRNA, il numero di sgRNA non rilevati e la percentuale di letture da singoli campioni mappati con successo nella libreria di sgRNA. Utilizzare un rapporto di mappatura più alto per indicare una maggiore copertura.
- Contare l'arricchimento delle reads per ciascun gene (bersaglio di diversi sgRNA) in ciascun campione. Normalizzare le letture di supporto per ogni sgRNA tra i campioni utilizzando il metodo di normalizzazione "mediana" di MAGeCK. Esegui il controllo di qualità a livello di campione valutando la distribuzione dei conteggi delle letture di sgRNA, generando box plot, conducendo analisi dei componenti principali (PCA) e costruendo mappe di calore di correlazione.
- Supponiamo che i conteggi delle letture degli sgRNA seguano una distribuzione di Poisson. Rappresentare i conteggi normalizzati degli sgRNA di diversi gruppi in box plot per visualizzare la distribuzione complessiva dei dati all'interno dei campioni e confrontare le distribuzioni tra i gruppi.
- Utilizzare la PCA per semplificare l'analisi dei dati riflettendo le differenze tra i componenti principali e il tasso di variazione all'interno di ciascun componente, facilitando l'osservazione delle variazioni intra e intergruppo. Utilizzare una mappa termica di correlazione per illustrare le relazioni tra i campioni.
- Analisi di base
- Eseguire l'analisi differenziale dell'sgRNA tra i gruppi e identificare i geni essenziali dopo il controllo di qualità e l'elaborazione preliminare dei dati. Condurre analisi di arricchimento funzionale dei geni rilevanti30. Classifica i geni essenziali in base al punteggio di aggregazione di rango robusto (RRA) calcolato da MAGeCK o altri metodi statistici come MLE, con un punteggio RRA più basso che indica una maggiore importanza.
- Utilizzare il linguaggio R o altri linguaggi di programmazione per eseguire analisi bioinformatiche sui risultati classificati e visualizzare i risultati utilizzando i diagrammi di analisi GO e KEGG30. Collegare geni o proteine ai corrispondenti termini OB (funzione molecolare, processo biologico e componente cellulare) attraverso la mappatura dell'ID o l'annotazione della sequenza.
- Utilizza KEGG come database per comprendere funzioni di alto livello e sistemi biologici, collegando il catalogo genico identificato alle funzioni del sistema a livello cellulare, di specie ed ecosistema. Seleziona i primi 10 o 20 percorsi dalle analisi GO e KEGG per una visualizzazione intuitiva delle direzioni dei percorsi.
Risultati
Il cancro del polmone, con il principale tasso di mortalità, rappresenta una malattia medica altamente aggressiva e prevalente. Utilizzando la linea cellulare di cancro del polmone A549 come esempio per condurre uno screening CRISPR a livello di genoma con radiazioni come condizione di screening, il flusso di lavoro schematico è mostrato nella Figura 1. In primo luogo, esplorare la sensibilità delle cellule A549 a diverse dosi di radiazioni attraverso la formazione di cloni e gli esperimenti CCK8 (Figura 2). Nel test clonogenico, la conta delle colonie era di 140 ± 5,35 a 2 Gy contro 303 ± 31,63 a 0 Gy, mentre nel test CCK-8, il valore di densità ottica (OD) era 0,65 ± 0,05 a 2 Gy contro 1,35 ± 0,08 a 0 Gy. Per studiare i geni candidati per la radiosensibilità e la radioresistenza, selezionare 2 Gy (IC50) come dose di radiazioni successiva. Le cellule A549 sono state trattate con diverse dosi di puromicina e contate dopo 72 ore. La concentrazione minima di uccisione della puromicina per le cellule A549 è stata rilevata come 1 μM (Figura 3). Le cellule A549 sono state infettate con lentivirus a diversi MOI di gradiente. Dopo 72 ore di infezione, l'efficienza dell'infezione è stata osservata al microscopio a fluorescenza e contata prima e dopo 72 ore di trattamento con puromicina. È stato ottenuto il MOI con un'efficienza di infezione di ~0,3 (Tabella 1). Il genoma umano contiene 19.050 geni, con 6 sgRNA corrispondenti a ciascun gene e un numero di copie di 500 (19.050 x 6 x 500 = 5,7 x 107 cellule). Utilizzando lentivirus con MOI = 0,3 e, di conseguenza, infettare 5,7 x 107 cellule A549. Dopo 72 ore, le cellule sono state trattate con 1 μM di puromicina per altre 72 ore. È stato raccolto il genoma del giorno 0 ed è stata eseguita l'amplificazione PCR. Le sequenze di sgRNA all'interno della libreria dell'intero genoma CRISPR mostrano una lunghezza di 231 coppie di basi, che è corroborata dai risultati dell'elettroforesi su gel di agarosio (Figura 4).
I prodotti PCR sono stati successivamente inviati a Novogene per il controllo della qualità dei dati e a livello di campione. Il rapporto di mappatura ha prodotto un tasso di copertura globale di circa il 60%, una metrica ritenuta adeguata per le procedure di screening dell'intero genoma, data l'assoluta complessità e l'inevitabile attrito durante l'elaborazione di un intero genoma (Figura 5). I conteggi di lettura dell'sgRNA hanno aderito a una distribuzione di Poisson, conforme alle aspettative teoriche. La successiva analisi attraverso il tracciamento PCA e la mappatura termica di correlazione ha ritratto vividamente la disparità distinguibile tra gruppi distinti, con variazioni tra gruppi che superano notevolmente le discrepanze all'interno del gruppo. Inoltre, il tasso di variazione all'interno dei campioni raggruppati rientrava nei limiti accettabili, comprovando il successo delle misure di controllo della qualità a livello di campione. Successivamente, le classifiche RRA ideate da MAGeCK saranno sfruttate per intraprendere una valutazione bioinformatica di base dei risultati della classifica utilizzando il linguaggio R. In particolare, i primi 15 percorsi in termini di OB presentavano in modo prominente meccanismi legati al danno al DNA, che si allineano perfettamente con i criteri di screening sottostanti dell'esperimento.

Figura 1: Schema dello screening CRISPR/Cas9 della radioterapia a livello di genoma in cellule di carcinoma polmonare A549. Una rappresentazione schematica del flusso di lavoro di screening nelle cellule A549 utilizzando CRISPR/Cas9 a livello di genoma e radioterapia. Clicca qui per visualizzare una versione più grande di questa figura.
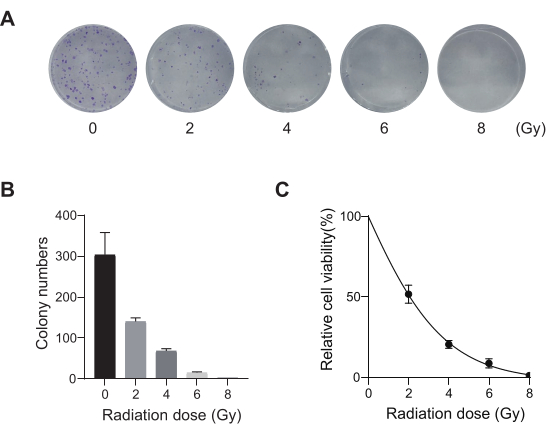
Figura 2: Ottimizzazione della dose di radiazioni per le cellule A549. (A) Le cellule A549 sono state esposte a dosi crescenti di radiazioni (0 Gy, 2 Gy, 4 Gy, 6 Gy e 8 Gy) e lasciate crescere per 14 giorni. Vengono mostrate le immagini del saggio di formazione delle colonie. I risultati sono rappresentativi di due esperimenti biologici indipendenti. (B) Risultati della quantificazione delle immagini del saggio di formazione clonata. Le barre di errore rappresentano la deviazione standard (n = 3). L'ANOVA unidirezionale ha prodotto P < 0,01. I risultati sono rappresentativi di due esperimenti biologici indipendenti. (C) Curve di sopravvivenza dose-risposta delle cellule A549 in seguito all'esposizione a diverse dosi di radiazioni. Le barre di errore rappresentano la deviazione standard (n = 3). L'ANOVA unidirezionale ha prodotto P < 0,01. I risultati sono rappresentativi di due esperimenti biologici indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
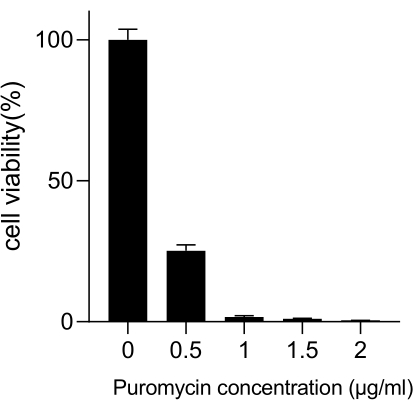
Figura 3: Determinazione della concentrazione minima di puromicina richiesta per la selezione delle cellule A549. Le cellule A549 sono state trattate con concentrazioni crescenti di puromicina e incubate per 72 ore. Le barre di errore rappresentano la deviazione standard (n = 3). L'ANOVA unidirezionale ha prodotto P < 0,01. I risultati sono rappresentativi di due esperimenti biologici indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
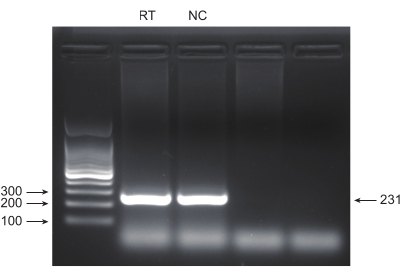
Figura 4: Validazione del prodotto PCR mediante elettroforesi su gel di agarosio. L'elettroforesi su gel di agarosio è stata utilizzata per esaminare la presenza e la qualità delle bande di prodotti PCR. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Controllo di qualità e analisi di base dei prodotti amplificati con PCR. (A) Il rapporto di mappatura indicava una copertura di circa il 60% del genoma di riferimento. (B) La distribuzione del conteggio delle letture degli sgRNA ha seguito una distribuzione di Poisson. (C) La maggior parte dei primi 10 termini di ontologia genica (GO) sono stati associati alla risposta al danno del DNA. (D) Grafico del vulcano che mostra i modelli di arricchimento genico, con geni arricchiti negativamente in rosso, geni arricchiti positivamente in blu e geni non significativi in grigio. Clicca qui per visualizzare una versione più grande di questa figura.
| Particella virale Numero (VP) | Numero di conteggio (prima del trattamento con puromicina) | Numero di conteggio (dopo il trattamento con puromicina) | Infezione efficienza |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
Tabella 1: Conta delle cellule A549 prima e dopo la selezione della puromicina sotto diverse particelle virali. Conta delle cellule A549 registrata prima e dopo il trattamento con puromicina sotto particelle virali variabili.
Discussione
In quanto tecnologia di editing genetico all'avanguardia, lo schermo CRISPR ha innescato profondi cambiamenti nel campo della ricerca scientifica5. Nata dal sistema CRISPR-Cas9, questa tecnologia è diventata uno strumento essenziale per lo studio delle funzioni geniche grazie alla sua elevata efficienza e precisione9. Il principio ingegneristico CRISPR/Cas9 prevede la progettazione e l'introduzione di specifici sgRNA con circa 20 nucleotidi per guidare la nucleasi Cas9 a localizzare e tagliare con precisione le sequenze di DNA bersaglio, consentendo l'editing genetico come il knockout, l'attivazione o la repressione6. Questo principio è alla base dello sviluppo di uno schermo CRISPR ad alta produttività e personalizzabile. Costruendo librerie contenenti un vasto numero di sgRNA, i ricercatori possono modificare e selezionare simultaneamente più geni, identificando rapidamente quelli associati a specifici fenotipi o stati patologici. Inoltre, lo schermo CRISPR consente la sperimentazione in vari tipi di cellule, tessuti e persino interi organismi, fornendo una comprensione più completa della funzione genica e dei meccanismi d'azione.
Con lo sviluppo accelerato della tecnologia, lo schermo CRISPR ha mostrato un enorme potenziale per l'applicazione in vari campi. Ad esempio, lo screening CRISPR può aiutare gli scienziati a scoprire nuovi bersagli farmacologici, accelerando così il processo di scoperta e sviluppo di farmaci. Nella ricerca sulle malattie, questa tecnologia rivela i meccanismi della patogenesi della malattia, offrendo nuove intuizioni e approcci per la diagnosi e il trattamento. Lo screening CRISPR differisce fondamentalmente dai metodi di screening tradizionali (come l'RNAi e gli inibitori di piccole molecole) in diversi aspetti chiave: meccanicisticamente, CRISPR raggiunge modifiche genetiche permanenti attraverso l'editing genetico, mentre l'RNAi sopprime solo temporaneamente l'espressione genica e gli inibitori di piccole molecole bloccano reversibilmente la funzione proteica28,29. In termini di specificità, CRISPR dimostra una precisione superiore attraverso un accurato targeting del DNA, mentre l'RNAi è soggetto a effetti fuori bersaglio 3,5. Per quanto riguarda la durata dell'effetto, CRISPR crea alterazioni genetiche permanenti, rispetto agli interventi transitori dei metodi convenzionali. Inoltre, CRISPR consente funzionalità di screening dell'intero genoma e può studiare contemporaneamente più geni/percorsi, mantenendo un'elevata efficienza in diversi tipi di cellule9. Gli approcci tradizionali sono in genere limitati all'analisi di percorsi specifici e spesso mostrano un'efficacia ridotta in alcune linee cellulari3. Queste caratteristiche distintive rendono lo screening CRISPR particolarmente prezioso per la ricerca di genomica funzionale e la scoperta di bersagli, in particolare negli studi che richiedono un'osservazione a lungo termine o una precisa regolazione genetica, dove dimostra vantaggi unici rispetto alle metodologie di screening convenzionali.
Tuttavia, la specificità di questo processo non è assoluta, ed è possibile che la proteina Cas9 riconosca e tagli erroneamente regioni del DNA che sono simili ma non del tutto identiche alla sequenza bersaglio prevista5. Questo evento è comunemente indicato come effetti fuori bersaglio. Questa scissione involontaria del DNA può portare a instabilità genomica e conseguenze genetiche imprevedibili, soprattutto se applicata in terapia umana. La tecnologia CRISPR fatica anche quando si tratta di complesse attività di editing genomico. Di fronte a richieste di editing che comprendono numerosi geni, cluster genici o intricate impostazioni genetiche, il sistema CRISPR può riscontrare limitazioni nell'anticipare e dirigere con precisione i risultati del processo di editing1.
Il principale ostacolo tecnico nella conduzione dello screening dell'intero genoma utilizzando la tecnologia CRISPR deriva dalla vasta complessità del genoma, che comprende decine di migliaia di geni, aumentando così significativamente il rischio di effetti fuori bersaglio9. Per garantire l'integrità e l'affidabilità dei successivi risultati del sequenziamento, è imperativo che ogni cellula sia trasfettata con un singolo sgRNA23. Ciò richiede il controllo meticoloso del MOI da parte dello sperimentatore, assicurandosi che rimanga al di sotto di 0,5 e contemporaneamente mantenendo un'adeguata soglia di numero di copie, che deve essere almeno 500 o superiore a24. Dato che ciascuna delle decine di migliaia di geni nell'intero genoma richiede l'utilizzo di 4-6 sgRNA, l'intero processo di screening richiede un'ampia quantità di cellule25. La durata prolungata dell'esperimento, il numero sostanziale di cellule coinvolte e l'elevato rischio richiedono che lo sperimentatore elabori strategie per alleviare queste preoccupazioni26. I geni potenziali devono essere ulteriormente convalidati con RNA-seq, nonché con saggi di guadagno e perdita di funzione. Negli esperimenti successivi, dopo aver ottenuto la sovraespressione e le cellule knockout dei geni identificati utilizzando l'editing CRISPR, sono stati eseguiti genomica a livello di genoma e trascrittomica per accedere a potenziali alterazioni off-target, in particolare a loci con parziale complementarità di sgRNA che possono subire una scissione non specifica di Cas9. Gli strumenti di previsione bioinformatica come COSMID potrebbero aiutare a identificare i siti fuori bersaglio ad alto rischio. I saggi di salvataggio potrebbero anche essere utilizzati per garantire la specificità del coinvolgimento dei geni identificati.
Nell'analisi successiva, i criteri per gli sgRNA espressi in modo differenziale si basavano sul punteggio di aggregazione di rango robusto (RRA) calcolato da MAGeCK, che comprendeva la significatività statistica, il cambiamento di piega e la coerenza della replicazione31,32. Per un set di dati, ogni gene è classificato in base a criteri completi che combinano i livelli di espressione genica e la significatività statistica. L'RRA utilizza il coefficiente di correlazione del rango di Spearman per il rango per valutare la situazione del rango completo di ciascun gene in tutti i set di dati e integra i risultati dell'ordinamento di più set di dati di espressione genica. Un gene con un rango relativamente alto nella maggior parte dei set di dati potrebbe essere considerato come un vero e proprio gene differenzialmente espresso33. Un valore p di RRA inferiore a 0,05 è stato considerato statisticamente significativo. Una variazione di almeno 1,5 volte o del 33% è stata considerata un'espressione differenziale. Inoltre, la consistenza di 3 repliche indipendenti doveva essere considerata come un'espressione statisticamente diversa. Dopo la classificazione RRA per i geni essenziali, un punteggio RRA più basso significa maggiore importanza.
Come potente strumento per l'editing del genoma, la tecnologia CRISPR-Cas9 dimostra un potenziale e un'accuratezza senza pari nello studio dell'area della radioresistenza26,27. Tuttavia, la sua applicazione presenta ancora dei limiti. Oltre al rischio di effetti fuori bersaglio, le complessità associate alla radiosensibilità introducono complessità nel processo di screening27. Questa sensibilità non è semplicemente governata da un gene solitario, ma piuttosto emerge come conseguenza dell'intricata interazione tra più geni e le loro reti regolatorie26. Quindi, nonostante il successo dell'identificazione di geni specifici relativi alla radiosensibilità, potrebbero rappresentare solo una frazione del quadro più ampio, ponendo sfide significative per chiarire in modo completo i meccanismi sottostanti 34,35,36. Di seguito sono riportate le soluzioni ai problemi comuni nello screening CRISPR, come la bassa efficienza dell'infezione, la degradazione dell'sgRNA e il fallimento del sequenziamento: poiché lo screening CRISPR richiede un valore MOI <0,5, l'aggiustamento dello stato cellulare è fondamentale per garantire che le cellule siano in una fase di crescita logaritmica o per valutare l'efficienza dell'infezione tramite saggi di fluorescenza; le potenziali cause della degradazione dell'sgRNA includono la contaminazione da nucleasi o difetti di progettazione del vettore, quindi dovrebbero essere evitati ripetuti cicli di congelamento-scongelamento delle librerie di sgRNA (conservare le aliquote a -80 °C) e il sequenziamento NGS dovrebbe essere utilizzato per verificare l'integrità della libreria di sgRNA; il fallimento del sequenziamento può derivare da una profondità di sequenziamento insufficiente o da problemi di progettazione del primer sgRNA, si consiglia il sequenziamento paired-end per migliorare l'accuratezza dell'identificazione della sequenza di sgRNA, con una profondità di sequenziamento suggerita di >500× copertura 37,38,39,40,41,42.
La radioterapia rimane una pietra miliare nel trattamento del cancro, ma mostra una notevole variabilità nell'efficacia terapeutica tra i diversi tipi di tumore e anche tra i pazienti con lo stesso cancro9. Questa eterogeneità interindividuale richiede spesso terapie combinate per ottenere un'eradicazione ottimale del tumore11,12. Nella pratica clinica, l'integrazione della radioterapia con l'immunoterapia - in particolare gli inibitori del checkpoint immunitario e i coniugati anticorpo-farmaco - è emersa come un importante paradigma terapeutico19. Questo protocollo presenta uno screening CRISPR in vitro utilizzando una linea cellulare tumorale relativamente omogenea, che potrebbe non riflettere totalmente l'eterogeneità genetica ed epigenetica dei tumori in vivo. Dopo aver determinato la dose appropriata di radiazioni per lo screening, i risultati forniscono potenziali geni radiosensibili o radioresistenti, che devono essere ulteriormente confermati sia in saggi cellulari in vitro che in modelli animali in vivo. Inoltre, lo screening CRISPR in vivo e la trascrittomica a singola cellula potrebbero essere utili per decifrare la complessità dell'eterogeneità tumorale. Lo screening CRISPR in vitro ha identificato geni importanti nella riparazione dei danni al DNA all'interno delle cellule, nonché nei meccanismi autocrini in risposta alle radiazioni, ma in realtà non ha replicato completamente l'eterogeneità del TME. Ad esempio, l'ipossia aumenta la produzione di ROS, innescando un ciclo di feedback che stimola l'adattamento metabolico, la generazione di antiossidanti e l'attivazione dell'autofagia, promuovendo in ultima analisi la radioresistenza18. Le cellule immunitarie potrebbero essere attivate nel meccanismo paracrino dopo la radiazione, inducendo radiosensibilità14. La MEC funge da serbatoio per i fattori di crescita e le citochine hanno effetti sia positivi che negativi sulla radiosensibilità21. Lo screening CRISPR in vivo deve essere eseguito per riflettere l'eterogeneità del TME. Inoltre, lo screening CRISPR in vitro ha potuto identificare solo geni importanti nella riparazione del danno al DNA all'interno delle cellule, così come nei meccanismi autocrini in risposta alle radiazioni, ma non ha catturato completamente l'influenza in vivo sulla radiosensibilità, compresa la vascolarizzazione tumorale e gli effetti sistemici20,21. Lo screening CRISPR in vivo deve essere eseguito per identificare i potenziali fattori coinvolti nella radiosensibilità in vivo.
Qui, viene ideato un protocollo completo volto a utilizzare lo screening dell'intero genoma CRISPR per individuare i geni implicati nella sensibilità o nella resistenza alla radioterapia. Svelando nuovi percorsi, questa metodologia cerca di aprire la strada ai progressi nella ricerca sulla radioterapia dei tumori. Questo protocollo potrebbe anche servire come riferimento per esplorare i meccanismi dei vincoli clinici nella chemioterapia e la progressione di agenti terapeutici alternativi. In sintesi, la tecnologia CRISPR incarna un potenziale e un valore significativi, ma i suoi vincoli intrinseci sottolineano l'importanza di un riconoscimento sobrio e di un approccio giudizioso. Man mano che la ricerca e le applicazioni future vengono approfondite, è imperativo sforzarsi incessantemente di esplorare e ottimizzare la tecnologia CRISPR per superare i suoi limiti e sfruttare il suo pieno potenziale.
Divulgazioni
Nessuno.
Riconoscimenti
Questo studio è stato supportato dal progetto regionale di innovazione scientifica e tecnologica della provincia di Hubei (2024EIA001) e dal progetto di supporto alla costruzione della piattaforma di innovazione scientifica e tecnologica medica dell'ospedale Zhongnan dell'Università di Wuhan (PTXM2025001). La Figura 1 è stata creata utilizzando Figdraw.
Materiali
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
Riferimenti
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon