Method Article
Écran CRISPR à l’échelle du génome pour dévoiler les gènes radiosensibles et radiorésistants
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Une approche méticuleuse et structurée est donnée pour sélectionner les gènes résistants et sensibles des rayonnements grâce à l’application d’une méthode de criblage CRISPR/Cas9 à l’échelle du génome. Ce protocole a également le potentiel de servir de cadre polyvalent pour d’autres efforts de recherche portant sur les mécanismes de résistance aux médicaments chimiques administrés cliniquement.
Résumé
Le système CRISPR-Cas9 a été exploité et transformé en un puissant outil d’édition du génome. En exploitant cette technologie, les chercheurs peuvent couper, coller et même réécrire avec précision des séquences d’ADN dans des cellules vivantes. Néanmoins, l’application de la technologie d’écran CRISPR va bien au-delà de la simple expérimentation. Il s’agit d’un outil essentiel dans la lutte contre les maladies génétiques, en disséquant systématiquement des paysages génétiques complexes, en permettant aux chercheurs de démêler les mécanismes moléculaires sous-jacents aux phénomènes biologiques et en permettant aux scientifiques d’identifier et de cibler les causes profondes de maladies telles que le cancer, la fibrose kystique et l’anémie falciforme. Parmi tous, le cancer représente un formidable défi pour la médecine, stimulant les efforts d’éradication. La radiothérapie, en tant que traitement traditionnel, donne des résultats mais a des limites. Il éradique les cellules cancéreuses mais endommage également les tissus sains, provoquant des effets néfastes qui réduisent la qualité de vie. De plus, toutes les cellules cancéreuses ne répondent pas à la radiothérapie, et certaines peuvent développer une résistance, aggravant la maladie. Pour y remédier, une technologie complète de criblage CRISPR du génome entier est introduite, car elle permet l’identification efficace des gènes radiosensibles et radiorésistants, faisant ainsi progresser le domaine de la recherche et du traitement du cancer. Un dépistage CRISPR à l’échelle du génome a été effectué dans des cellules d’adénocarcinome pulmonaire exposées à l’irradiation selon le protocole décrit, grâce auquel les gènes associés à la radiorésistance et à la radiosensibilité ont été identifiés.
Introduction
L’étude des phénomènes biologiques est intrinsèquement liée à l’étude des comportements cellulaires, et à son tour, l’examen des comportements cellulaires est fondamentalement lié à l’exploration de son génome. Au fur et à mesure que la technologie moderne continue d’évoluer, les chercheurs médicaux réorientent progressivement leur attention vers la modification des comportements cellulaires via l’édition de gènes afin d’améliorer les résultats du traitement de diverses maladies. À cet égard, la technologie CRISPR (Clustered regularly Interspaced Short Palindromic Repeats) est apparue comme un outil révolutionnaire pour l’édition du génome en raison de son application relativement simple1. Le système CRISPR-Cas9 se compose de la nucléase Cas9 et de l’ARN monoguide (ARNsg), qui reconnaît et se lie spécifiquement à la séquence d’ADN cible, guidant la nucléase Cas9 à couper à cet endroit, ce qui entraîne une cassure double brin (DSB) dans l’ADNdu génome 2,3,4. De plus, l’introduction d’autres substances peut entraîner des insertions, des délétions ou des mutations spécifiques dans le génome, permettant une édition génique ciblée.
Dans la recherche en génomique fonctionnelle, le dépistage de l’interférence ARN (ARNi) était autrefois une méthode largement utilisée pour mener des expériences de perte de fonction à grande échelle afin d’étudier les rôles des gènes dans le cancer. La technologie ARNi étudie la fonction des gènes en réduisant spécifiquement au silence les gènes cibles, aidant ainsi les chercheurs à identifier les facteurs oncogéniques critiques. Cependant, il est limité par des effets hors cible et une efficacité incomplète de l’inactivation des gènes. Les effets hors cible peuvent conduire à l’inactivation d’autres gènes non cibles, compromettant ainsi l’exactitude et la fiabilité des résultats expérimentaux 1,2. De plus, l’ARNi présente une faible efficacité d’inactivation pour certains gènes, ce qui pourrait ne pas réussir à supprimer complètement l’expression des gènes cibles. Contrairement aux cribles ARNi traditionnels, le crible CRISPR fait preuve d’une spécificité et d’une efficacité plusélevées3. Cette technologie permet non seulement l’édition précise de gènes spécifiques, mais permet également un dépistage à grande échelle à l’échelle du génome, fournissant un soutien solide à la recherche sur la fonction des gènes. La technologie d’écran CRISPR, un puissant outil d’édition de gènes basé sur le système CRISPR-Cas9, est utilisée pour cribler efficacement et dévoiler des fonctions inconnues de gènes spécifiques dans les cellules 5,6,7,8. Les chercheurs conçoivent des sgRNA par lots pour des gènes ou des régions de gènes spécifiques, et préparent les banques de sgRNA correspondantes avec précision et rigueur, en garantissant leur intégrité et leur fonctionnalité9. Ces banques d’ARNsg sont ensuite encapsulées dans des particules lentivirales, qui sont utilisées pour infecter efficacement les cellules hôtes. Après une infection réussie, les cellules infectées sont cultivées dans des conditions de dépistage définies personnellement. Lors du criblage, l’ADN génomique des cellules criblées est extrait, en maintenant des normes élevées de pureté et de quantité. Par la suite, les régions ciblées d’intérêt pour les ARNsg sont soumises à une amplification par PCR, un processus qui réplique avec précision les segments souhaités d’acides nucléiques 3,9. Enfin, un séquençage à haut débit est effectué sur les fragments d’ADN amplifiés, permettant une analyse complète et efficace des régions ciblées, fournissant ainsi des informations précieuses sur la fonction et le comportement des gènes étudiés4.
Le cancer représente une menace redoutable pour la santé humaine en tant que maladie complexe. Dans le monde entier, les chercheurs et les cliniciens déploient des efforts concertés pour démêler les mécanismes moléculaires de la cancérogenèse et développer de nouvelles stratégies thérapeutiques. Des collaborations internationales ont été établies pour accélérer l’application des résultats de la recherche fondamentale en applications cliniques, dans le but ultime d’améliorer les résultats pour les patients. Sasmal et al. ont proposé une stratégie d’assemblage bioorthogonale basée sur un système synthétique hôte-invité pour un ciblage précis des cellules cancéreuses métastatiques, ce qui a considérablement aidé des dizaines de scientifiques à faire progresser les technologies médicales. Leurs travaux de recherche exceptionnels sont caractérisés par une grande innovation et des idées uniques, apportant des contributions significatives à la communauté scientifique10. Le cancer est caractérisé par l’état tumultueux d’instabilité génomique, résultant de la régulation erratique des réponses aux dommages de l’ADN11-14. Les dommages à l’ADN comprennent les défauts mononucléotidiques, les cassures monocaténaires et les CDB. La recombinaison homologue (HR) et la jonction d’extrémité non homologue (NHEJ) participent à la réparation des DSB à différents stades 15,16,17. Sur cette base, la radiothérapie est apparue comme une option de traitement viable, qui utilise des rayons de haute énergie (tels que les rayons X et les rayons γ) pour irradier le tissu tumoral, causant des dommages à l’ADN dans les cellules tumorales, perturbant ainsi leur croissance et leur prolifération18. Cependant, la radiothérapie ne produit pas toujours les effets escomptés chez une proportion importante de patients atteints de cancer, résultant potentiellement de lésions des tissus paracancéreux et de limitations imposées par les caractéristiques inhérentes à la tumeur, telles qu’une faible sensibilité à la radiothérapie 19,20,21.
Théoriquement, n’importe quel type de cellule peut être utilisé pour un écran CRISPR. Cependant, le maintien d’une représentation suffisante dans les populations mutées nécessite un grand nombre de cellules de départ. Les types de cellules à faible abondance ne sont pas particulièrement adaptés au criblage à l’échelle du génome. En ce qui concerne le choix de la bibliothèque, la plupart des banques contiennent 3 à 6 ARNg par gène cible, et le maintien de la distribution de chaque ARNg au sein de la population est essentiel18. La perte de représentation due à l’enrichissement ou à l’épuisement d’ARNg spécifiques peut conduire à une distribution inégale des résultats. Pour résoudre ce problème, il peut être préférable d’opter pour des bibliothèques CRISPR disponibles dans le commerce et testées sur le marché20. In vitro Le criblage CRISPR utilisant des lignées cellulaires cancéreuses homogènes peut ne pas capturer pleinement l’hétérogénéité génétique et épigénétique des tumeurs in vivo . Bien que le dépistage in vitro ait révélé des gènes clés impliqués dans la réparation des dommages à l’ADN et la signalisation autocrine induite par les radiations, il n’a pas entièrement reproduit le microenvironnement tumoral, y compris la radiorésistance induite par l’hypoxie (via les ROS, l’adaptation métabolique et l’autophagie), les effets paracrines à médiation immunitaire et la modulation des cytokines dépendante de l’ECM. Avant d’utiliser l’écran CRISPR pour explorer les gènes associés à la sensibilité ou à la résistance aux rayonnements, ces facteurs doivent être soigneusement pris en compte. À la lumière du paysage thérapeutique actuel, il est urgent d’identifier et d’étudier en profondeur les facteurs associés à la radiorésistance et à la radiosensibilité afin d’améliorer efficacement l’efficacité de la radiothérapie22. Étant donné l’avantage clé de l’écran CRISPR dans l’étude des fonctions de gènes inconnus, une technologie d’écran CRISPR du génome entier systématiquement détaillée est fournie pour identifier efficacement les gènes radiosensibles et radiorésistants.
Protocole
Les réactifs et l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Choisir une dose appropriée de rayonnement
- Préparation et placage de cellules adhérentes
- Ajustez la densité cellulaire à 5 x 105 cellules par mL avec le milieu de culture cellulaire complet RPMI 1640 contenant 10 % de FBS pour le passage et la croissance cellulaire. Répartissez les cellules dans des boîtes de culture de 3,5 cm pour les radier à différentes doses. Ajouter 2 ml (1 x 106 cellules) dans chaque plat et incuber toute la nuit à 37 °C avec 5 % de CO2.
- Application de différentes doses de rayonnement
- Numérotez les boîtes de culture de 3,5 cm de 1 à 5. Utilisez le groupe #1 comme groupe témoin, les 4 autres groupes étant désignés comme groupes de traitement.
- Scellez les bords des plaques à 6 puits à l’aide d’une membrane d’étanchéité pour les groupes de traitement et administrez des doses de rayonnement de 2, 4, 6 et 8 Gy, respectivement. Après le traitement, remettez la vaisselle dans l’incubateur.
- Placage de lignées cellulaires après irradiation
- Préparez des plaques à 6 puits et à 96 puits. Lavez les cellules dans les boîtes de culture de 3,5 cm une fois avec du PBS, digérez les cellules en croissance logarithmique avec 0,25 % de trypsine et ajustez la densité cellulaire à 1 x 105 cellules par ml avec le milieu complet RPMI 1640 contenant 10 % de FBS.
- Ensemencez 10 μL/puits (1 000 cellules/100 μL) dans les plaques à 6 puits à 3 répétitions pour chaque dose de rayonnement. Ajouter 2 ml de milieu complet dans chaque puits et compter après 14 jours (lorsque chaque groupe de clones a environ 50 cellules).
- Implantez 30 μL/puits (3 000 cellules/100 μL) dans les plaques à 96 puits à raison de 5 répétitions pour chaque dose de rayonnement. Ajouter 70 μL de milieu complet dans chaque puits et mesurer la viabilité cellulaire après 72 h.
- Mesure de la viabilité cellulaire
REMARQUE : Taux d’inhibition clonogénique = 1 - (nombre de clones dans le groupe de traitement/nombre de clones dans le groupe de contrôle) x 100 %. Le test CCK-8 utilise un sel de tétrazolium soluble dans l’eau (WST-8) qui peut être réduit par les déshydrogénases cellulaires pour générer un produit de formazan jaune hautement soluble dans l’eau11. La quantité de formazan produite est directement proportionnelle au nombre de cellules viables, et sa densité optique (mesurée à une longueur d’onde de 450 nm) reflète avec précision l’activité métabolique cellulaire et l’état prolifératif11,13. Sur la base de ce principe bien établi, le test CCK-8 a été largement adopté pour diverses applications, notamment les tests de prolifération cellulaire et les tests de sensibilité aux médicaments tumoraux. Dans ce protocole, le test CCK-8 est utilisé pour évaluer systématiquement la viabilité cellulaire sous différentes doses de rayonnement.- Mélangez le réactif CCK8 avec le milieu RPMI 1640 sans FBS dans un rapport de 1:9. Jeter le milieu dans les plaques à 96 puits et ajouter 100 μL du milieu contenant la CCK8 dans chaque puits.
- Incuber dans l’obscurité pendant 1 h et mesurer la valeur de DO à 450 nm à l’aide d’un lecteur de microplaques.
REMARQUE : Taux de survie cellulaire = [(valeur DO du groupe traité - valeur DO du puits à blanc) / (valeur DO du groupe témoin - valeur DO du puits à blanc)] x 100 %
- Sélection de la dose de rayonnement
- Intégrer le taux d’inhibition clonogénique au taux de survie cellulaire afin de sélectionner une dose de rayonnement appropriée en fonction des besoins de la recherche. Choisir une dose de rayonnement avec un taux d’inhibition de 50 % pour le dépistage des gènes radiorésistants et radiosensibles.
2. Sélection de la concentration appropriée de MOI et de puromycine
- Placage de cellules adhérentes
- Ajustez la densité cellulaire à 3 x 105 cellules par mL et injectez 1 mL (3 x 105 cellules) par puits dans une plaque à 12 puits. Incuber la plaque pendant une nuit à 37 °C avec 5 % de CO2.
- Infection lentivirale
- À l’aide d’une pipette, aspirez le milieu de la plaque à 12 puits et remplacez-le par 1 mL de milieu complet RPMI 1640 contenant 10 % de FBS. Préparez une banque de lentiviraux CRISPR du génome entier dont la qualité a été vérifiée (les banques disponibles dans le commerce sont recommandées) et établissez un gradient de concentration logarithmique (p. ex., 0-10-50-100-200-400-800)4,5,6,7,8.
- Ajouter la quantité correspondante de lentivirus à 2 μL/boîte de polybrène et équilibrer à température ambiante pendant 5 min. Versez lentement le mélange de lentivirus et de polybrène dans chaque puits, mélangez bien et incubez toute la nuit à 37 °C avec 5 % de CO2.
- Détermination de la concentration minimale de puromycine pour la destruction cellulaire
- Ajuster la densité cellulaire des parents à 3 x 105 cellules/mL et inoculer 1 mL (3 x 105 cellules) par puits dans une plaque à 12 puits. Incuber la plaque pendant une nuit à 37 °C avec 5 % de CO2.
- Ajouter la puromycine dans chaque puits de la plaque à 12 puits dans un gradient de concentration de 0-0,1-0,2-0,5-1-2-4-8 μM. Compter les cellules après 72 h. La concentration minimale qui tue toutes les cellules dans le puits est la concentration minimale de puromycine pour la destruction des cellules, qui sera utilisée pour le dépistage ultérieur des cellules infectées par le virus.
- Sélection de la puromycine après l’infection
- Le deuxième jour après l’infection, aspirez le milieu de chaque puits et remplacez-le par 1 mL de milieu complet RPMI 1640 contenant 10 % de FBS. Poursuivre la culture pendant 48 h.
- Après 72 h d’infection, remplacer le milieu existant par un milieu complet contenant la concentration minimale de puromycine pour la destruction cellulaire. Mettre en place 2 puits dans la plaque de 12 puits sans infection lentivirale comme témoins négatifs et positifs. Traitez le témoin négatif avec de la puromycine et laissez le témoin positif non traité. Poursuivre la culture pendant 72 h.
- Après 72 h de sélection de la puromycine, toutes les cellules parentales du témoin négatif seront mortes, tandis que les cellules parentales du contrôle positif présentent une mort minimale. Calculez le moment d’inertie de chaque puits.
REMARQUE : MOI = (Nombre de cellules dans le groupe infecté par le virus / Nombre de cellules dans le groupe témoin positif) x 100 %. Utilisez la concentration du virus avec un MOI de ~0,3 pour le dépistage ultérieur.
3. Infection de la banque lentivirale CRISPR à l’échelle du génome
- Ensemencement de lignées cellulaires adhérentes
- Ajustez la densité cellulaire à 1 x 107 cellules par mL dans le milieu complet RPMI 1640 contenant 10 % de FBS pour le passage et la croissance cellulaire. Inocutionner 1 mL de cellules (1 x 107 cellules) dans chaque boîte de culture de 15 cm à 37 °C avec 5 % de CO2 pendant 8 h. Une fois que les cellules ont adhéré et ont atteint une confluence de 70 % à 80 %, elles étaient prêtes pour l’infection virale (ce qui fait un nombre de copies d’environ 500).
- Infection lentivirale
- Aspirez le milieu des boîtes de culture de 15 cm et remplacez-le par 15 mL de milieu complet RPMI 1640 contenant 10 % de FBS. Préparez une banque de lentiviraux CRISPR du génome entier dont la qualité a été vérifiée.
- Ajouter la quantité correspondante de lentivirus avec le MOI = 0,3 à 30 μL/boîte de polybrène et équilibrer à température ambiante pendant 5 min, verser lentement le mélange de lentivirus et de polybrène dans les boîtes de culture de 15 cm, bien mélanger et incuber à 37 °C avec 5 % de CO2 pendant la nuit. En même temps, préparez une boîte de culture de 15 cm avec la cellule parentale en contraste.
- Sélection de la puromycine après l’infection
- Le deuxième jour après l’infection virale, aspirer le milieu des boîtes de culture de 15 cm et le remplacer par 15 mL de milieu complet RPMI 1640 contenant 10 % de FBS. Poursuivre la culture pendant 48 h.
- 72 h après l’infection, remplacer le milieu par un milieu complet contenant la concentration létale minimale de puromycine. Traitez les cellules parentales non infectées comme un témoin négatif de la même manière et poursuivez la culture pendant 72 h. Après 72 h de sélection de la puromycine, les cellules parentales du groupe témoin négatif seront toutes tuées, et les cellules survivantes de l’infection lentivirale sont considérées comme infectées avec succès.
- Extraction du génome du jour 0
- Digérer les cellules d’une boîte de culture de 15 cm avec de la trypsine à 0,25 %, les remettre en suspension dans un milieu complet RPMI 1640 contenant 10 % de FBS et compter le nombre de cellules. Étiquetez cet échantillon comme Jour 0.
- Centrifuger à 300 x g pendant 5 min (à température ambiante) et jeter le surnageant. Remettre en suspension dans 1 mL de PBS, centrifuger à 300 x g pendant 5 min et jeter le surnageant. Extrayez l’ADN génomique du jour 0, mesurez la concentration et la pureté de l’ADN à l’aide d’un spectrophotomètre UV à nanogouttes.
- Pour la détection de l’intégrité de l’ARNsg, utilisez l’électrophorèse sur gel d’agarose pour analyser la banque d’ARNsg amplifiée, en s’assurant que les bandes étaient claires et qu’il n’y avait pas de dégradation significative, préservant ainsi l’intégrité de la banque23. Pour l’évaluation de la couverture de la banque de lentiviraux, utilisez la technologie de séquençage profond pour effectuer une analyse de séquençage sur les ARNsg de la banque24,25.
- Dans l’écran CRISPR, l’amplification PCR sert de vérification préliminaire de l’intégrité des fragments d’ARNsg et de la qualité de la banque6. Pour analyser de manière plus complète la couverture des banques et les modèles de distribution des ARNsg, et pour assurer une représentation adéquate des ARNsg pour chaque gène cible lors du dépistage, effectuez un NGS pour obtenir des informations approfondies sur la robustesse du criblage9.
REMARQUE : En détectant les changements dans l’abondance des ARNsg avant et après le criblage, et en identifiant l’intégration potentielle hors cible de l’ARNsg ou la contamination de la banque, la précision et la fiabilité du criblage CRISPR pourraient être encore améliorées. Ces échantillons du jour 0 servent de contrôles négatifs cruciaux et font l’objet d’une évaluation complète de la qualité par amplification PCR et NGS pour atteindre 2 objectifs clés : (1) la confirmation de l’épuisement des gènes essentiels (indiquant une représentation adéquate de la bibliothèque), et (2) la démonstration de l’expression stable dans les gènes non essentiels (établissant des conditions expérimentales de base)9. - S’assurer que la couverture atteint le niveau attendu pour assurer la diversité et la représentativité de la bibliothèque et éviter les biais dans le processus de sélection. Pour mesurer l’efficacité de l’infection, utilisez l’imagerie par fluorescence pour évaluer l’efficacité de l’infection, en vous assurant qu’elle répond aux exigences de l’expérience.
4. Application du rayonnement comme condition de dépistage
- Groupement
- Ajustez la densité des cellules infectées par CRISPR à 1 x 107 cellules par ml et injectez 1 ml dans chaque boîte de Pétri de 15 cm. Incuber toute la nuit à 37 °C avec 5 % de CO2.
- Radiothérapie
- Divisez au hasard les cellules en 2 groupes : un groupe de traitement et un groupe témoin, avec 6 plats par groupe. Administrer une dose appropriée de rayonnement aux cellules du groupe de traitement, tout en laissant les cellules du groupe témoin non traitées se propager normalement. Après la radiothérapie, poursuivre l’incubation à 37 °C avec 5 % de CO2 pendant 7 jours.
- Au cours de la deuxième semaine, répéter l’administration d’une dose appropriée de radiation aux cellules du groupe de traitement, en laissant les cellules du groupe témoin non traitées et en leur permettant de se propager normalement. Après la radiothérapie, continuer l’incubation à 37 °C avec 5 % de CO2 pendant 7 jours supplémentaires.
5. Extraction et séquençage du génome
- Extraction du Jour 14
- Après 14 jours de traitement, digérer les cellules des groupes de traitement et de contrôle avec de la trypsine à 0,25 %, les remettre en suspension à l’aide d’un milieu RPMI 1640 complet contenant 10 % de FBS et les étiqueter comme Day 14-RT ou Day 14-NC.
- Centrifugez les cellules à 300 x g pendant 5 min et jetez le surnageant. Remettre en suspension dans 1 mL de PBS, centrifuger à 300 x g pendant 5 min et jeter à nouveau le surnageant. Extrayez l’ADN génomique du jour 14 et détectez la concentration et la pureté de l’ADN à l’aide de la spectroscopie d’absorbance UV nanodrop.
- Amplification par PCR
- Préparez les amorces correspondantes de la séquence (voir tableau des matériaux) et diluez-les à 10 μM.Mettez en place un système de réaction de 20 μL en ajoutant les réactifs dans un tube de microcentrifugation stérile, centrifugez pendant 5 s à 300 x g et mélangez bien. Réglez les paramètres de l’instrument PCR en fonction de la condition d’amplification pour obtenir des produits PCR.
- Détection par électrophorèse sur gel d’agarose
- Préparez une plaque de coulée de gel, scellez les bords du moule avec de l’agarose, insérez le peigne et préparez un gel d’agarose de concentration appropriée en fonction de la longueur de l’ADN de l’échantillon.
- Pesez avec précision une certaine quantité de poudre d’agarose, ajoutez une quantité appropriée de tampon d’électrophorèse, mélangez bien et chauffez au four à micro-ondes pour faire fondre. Après avoir légèrement refroidi, ajoutez une quantité appropriée de colorant d’acide nucléique, mélangez doucement et versez lentement dans le moule de coulée de gel. Laisser le gel se solidifier pendant 30 min.
- Retirez le peigne et ajoutez une quantité appropriée de tampon d’électrophorèse dans le réservoir d’électrophorèse jusqu’à ce qu’il recouvre le gel. Ajoutez une quantité appropriée de tampon de chargement à l’échantillon d’ADN, mélangez bien et utilisez une pipette pour ajouter lentement le mélange à l’échantillon.
- Réglez la tension appropriée en fonction de la taille du fragment d’ADN et de la concentration du gel d’agarose. Après l’électrophorèse, retirez délicatement le gel et placez-le dans un imageur de gel pour observer les résultats et vérifier si l’amplification PCR a réussi.
- Séquençage Illumina
- Collectez l’ADN génomique des trois groupes (groupe du jour 0, groupe de contrôle et groupe de traitement) et envoyez-le à une entreprise pour la construction de la bibliothèque et le séquençage Illumina. Effectuer une analyse bioinformatique et une visualisation des résultats de séquençage pour obtenir des gènes sensibles et résistants à la radiothérapie26.
- Mettre en place plusieurs expériences répétées pour l’analyse des données et effectuer une analyse statistique des résultats expérimentaux pour assurer la cohérence et la reproductibilité des données27,28.
- Évaluation de la qualité des données de séquençage
- Soumettre les données brutes à des mécanismes rigoureux de filtrage et de contrôle de la qualité pour garantir la précision des informations de séquençage5. Calculez le score Phred (Qphred) pour chaque nucléotide en fonction du taux d’erreur de séquençage, à l’aide d’un algorithme de conversion spécifié et d’un modèle qui évalue la probabilité qu’une erreur se produise lors de l’appel de base28.
- Maintenir le taux d’erreur de séquençage pour chaque position de base en dessous de 1 % (ce qui équivaut à un seuil Q30) et s’assurer qu’au moins 80 % des données de séquençage atteignent cette norme Q30 pour appuyer les procédures analytiques ultérieures.
- Traitement des données et contrôle de la qualité au niveau de l’échantillon
REMARQUE : Pour plus de détails, veuillez vous référer aux rapportspubliés précédemment 29,30.- Alignez les lectures filtrées des données de séquençage avec les séquences de la bibliothèque sgRNA. Rapports statistiques : y compris le nombre d’ARNsg dans la banque avec un alignement parfait, l’abondance moyenne des ARNsg, le nombre d’ARNsg non détectés et la proportion de lectures d’échantillons individuels mappés avec succès à la banque d’ARNsg. Utilisez un rapport de mappage plus élevé pour indiquer une plus grande couverture.
- Comptez l’enrichissement des lectures pour chaque gène (ciblé par différents ARNsg) dans chaque échantillon. Normalisez les lectures de support pour chaque ARNsg dans les échantillons à l’aide de la méthode de normalisation « médiane » de MAGeCK. Effectuez un contrôle qualité au niveau de l’échantillon en évaluant la distribution du nombre de lectures d’ARNsg, en générant des boîtes à moustaches, en effectuant une analyse en composantes principales (ACP) et en construisant des cartes thermiques de corrélation.
- Supposons que le nombre de lectures d’ARNsg suive une distribution de Poisson. Représenter le nombre normalisé d’ARNsg de différents groupes dans des boîtes à moustaches pour visualiser la distribution globale des données dans les échantillons et comparer les distributions entre les groupes.
- Utilisez l’ACP pour simplifier l’analyse des données en reflétant les différences entre les composantes principales et le taux de variation au sein de chaque composante, facilitant ainsi l’observation des variations intra et intergroupes. Utilisez une carte thermique de corrélation pour illustrer les relations entre les échantillons.
- Analyse de base
- Effectuer une analyse différentielle des ARNsg entre les groupes et identifier les gènes essentiels après le contrôle de la qualité et le traitement préliminaire des données. Effectuer une analyse d’enrichissement fonctionnel des gènes pertinents30. Classez les gènes essentiels en fonction du score d’agrégation de rang robuste (RRA) calculé par MAGeCK ou d’autres méthodes statistiques telles que MLE, un score RRA plus faible indiquant une importance plus élevée.
- Utilisez le langage R ou d’autres langages de programmation pour effectuer une analyse bio-informatique sur les résultats classés, et visualisez les résultats à l’aide des diagrammes d’analyse GO et KEGG30. Lier des gènes ou des protéines à des termes GO correspondants (fonction moléculaire, processus biologique et composant cellulaire) par le biais de la cartographie d’identification ou de l’annotation de séquences.
- Utilisez KEGG comme base de données pour comprendre les fonctions de haut niveau et les systèmes biologiques, en reliant le catalogue de gènes identifié aux fonctions du système aux niveaux cellulaire, de l’espèce et de l’écosystème. Sélectionnez les 10 ou 20 premiers chemins à partir des analyses GO et KEGG pour un affichage intuitif des directions des chemins.
Résultats
Le cancer du poumon, avec le taux de mortalité le plus élevé, représente une maladie médicale très agressive et répandue. En utilisant la lignée cellulaire du cancer du poumon A549 comme exemple pour réaliser un dépistage CRISPR à l’échelle du génome avec le rayonnement comme condition de dépistage, le flux de travail schématique est illustré à la figure 1. Tout d’abord, explorez la sensibilité des cellules A549 à différentes doses de rayonnement par la formation de clones et les expériences CCK8 (Figure 2). Dans l’essai clonogénique, le nombre de colonies était de 140 ± 5,35 à 2 Gy contre 303 ± 31,63 à 0 Gy, tandis que dans l’essai CCK-8, la valeur de densité optique (DO) était de 0,65 ± 0,05 à 2 Gy contre 1,35 ± 0,08 à 0 Gy. Pour étudier les gènes candidats pour la radiosensibilité et la radiorésistance, sélectionnez 2 Gy (IC50) comme dose de rayonnement ultérieure. Les cellules A549 ont été traitées avec différentes doses de puromycine et comptées après 72 h. La concentration minimale de destruction de la puromycine pour les cellules A549 a été détectée à 1 μM (Figure 3). Les cellules A549 ont été infectées par des lentivirus à différents gradients d’intérêt. Après 72 h d’infection, l’efficacité de l’infection a été observée au microscope à fluorescence et comptée avant et après 72 h de traitement par puromycine. Le MOI avec une efficacité d’infection de ~0,3 a été obtenu (Tableau 1). Le génome humain contient 19 050 gènes, avec 6 ARNsg correspondant à chaque gène et un nombre de copies de 500 (19 050 x 6 x 500 = 5,7 x 107 cellules). Utilisation de lentivirus avec MOI = 0,3 et, par conséquent, infecter 5,7 x 107 cellules A549. Après 72 h, les cellules ont été traitées avec 1 μM de puromycine pendant 72 h supplémentaires. Le génome du jour 0 a été récolté et une amplification par PCR a été effectuée. Les séquences d’ARNsg de la bibliothèque du génome entier CRISPR présentent une longueur de 231 paires de bases, ce qui est corroboré par les résultats de l’électrophorèse sur gel d’agarose (Figure 4).
Les produits PCR ont ensuite été envoyés à Novogene pour le contrôle de la qualité des données et le contrôle de la qualité au niveau de l’échantillon. Le ratio de cartographie a donné un taux de couverture complet d’environ 60 %, une mesure jugée adéquate pour les procédures de dépistage du génome entier, compte tenu de la complexité et de l’attrition inévitable au cours du traitement d’un génome entier (Figure 5). Le nombre de lectures d’ARNsg a adhéré à une distribution de Poisson, conformément aux attentes théoriques. L’analyse ultérieure par le biais du tracé de l’ACP et de la cartographie thermique de corrélation a mis en évidence les disparités perceptibles entre les groupes distincts, les variations entre les groupes l’emportant nettement sur les écarts intragroupes. De plus, le taux de variation au sein des échantillons groupés se situait dans des limites acceptables, ce qui justifie le succès des mesures de contrôle de la qualité au niveau de l’échantillon. Par la suite, les classements RRA conçus par MAGeCK seront exploités pour entreprendre une évaluation bioinformatique fondamentale des résultats du classement en utilisant le langage R. Notamment, les 15 principales voies en termes de GO présentaient en bonne place des mécanismes liés aux dommages à l’ADN, ce qui s’aligne parfaitement sur les critères de dépistage sous-jacents de l’expérience.

Figure 1 : Schéma du crible CRISPR/Cas9 de radiothérapie à l’échelle du génome dans des cellules cancéreuses du poumon A549. Une représentation schématique du flux de travail de criblage dans les cellules A549 à l’aide de CRISPR/Cas9 à l’échelle du génome et de la radiothérapie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
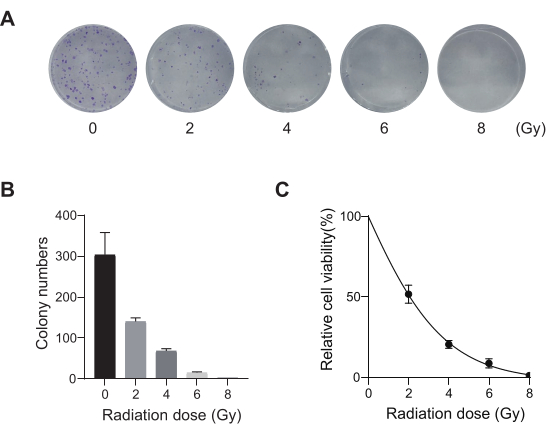
Figure 2 : Optimisation de la dose de rayonnement pour les cellules A549. (A) Les cellules A549 ont été exposées à des doses croissantes de rayonnement (0 Gy, 2 Gy, 4 Gy, 6 Gy et 8 Gy) et laissées croître pendant 14 jours. Des images d’essai de formation de colonie sont présentées. Les résultats sont représentatifs de deux expériences biologiques indépendantes. (B) Résultats de quantification des images d’essai de formation clonées. Les barres d’erreur représentent l’écart-type (n = 3). L’ANOVA à un facteur a donné un P < 0,01. Les résultats sont représentatifs de deux expériences biologiques indépendantes. (C) Courbes de survie dose-réponse des cellules A549 après exposition à différentes doses de rayonnement. Les barres d’erreur représentent l’écart-type (n = 3). L’ANOVA à un facteur a donné un P < 0,01. Les résultats sont représentatifs de deux expériences biologiques indépendantes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
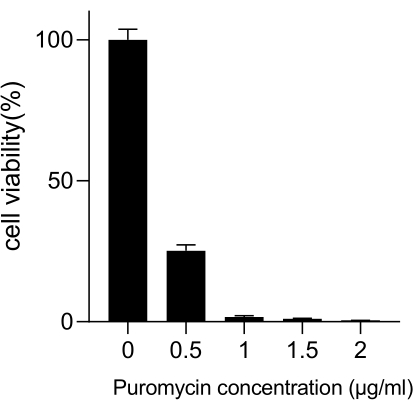
Figure 3 : Détermination de la concentration minimale de puromycine requise pour la sélection des cellules A549. Les cellules A549 ont été traitées avec des concentrations croissantes de puromycine et incubées pendant 72 h. Les barres d’erreur représentent l’écart-type (n = 3). L’ANOVA à un facteur a donné un P < 0,01. Les résultats sont représentatifs de deux expériences biologiques indépendantes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
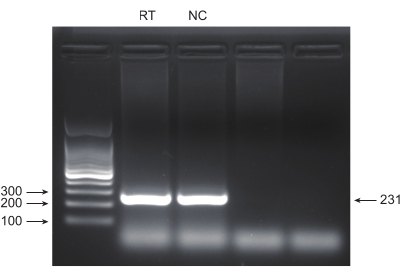
Figure 4 : Validation du produit par électrophorèse sur gel d’agarose. L’électrophorèse sur gel d’agarose a été utilisée pour examiner la présence et la qualité des bandes de produits PCR. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Contrôle de la qualité et analyse de base des produits amplifiés par PCR. (A) Le rapport de cartographie indiquait une couverture d’environ 60 % du génome de référence. (B) La distribution du nombre de lectures des ARNsg suivait une distribution de Poisson. (C) La plupart des 10 principaux termes de l’ontologie génétique (GO) étaient associés à la réponse aux dommages de l’ADN. (D) Graphique du volcan montrant les modèles d’enrichissement génétique, avec des gènes enrichis négativement en rouge, des gènes enrichis positivement en bleu et des gènes non significatifs en gris. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Particule virale Nombre (VP) | Nombre de comptes (avant le traitement par puromycine) | Nombre de comptes (après le traitement par puromycine) | Infection efficacité |
| 1,23 × 104 | 1,23 × 105 | 1,04 × 104 | 0.084552846 |
| 3,72 × 104 | 1,24 × 106 | 3,61 × 104 | 0.291129032 |
| 6,15 × 104 | 1,23 × 107 | 5,4 × 104 | 0.43902439 |
Tableau 1 : Nombre de cellules A549 avant et après la sélection de la puromycine sous différentes particules virales. Nombre de cellules A549 enregistrées avant et après le traitement par puromycine sous diverses particules virales.
Discussion
En tant que technologie d’édition génomique de pointe, l’écran CRISPR a déclenché de profonds changements dans le domaine de la recherchescientifique5. Issue du système CRISPR-Cas9, cette technologie est devenue un outil essentiel pour l’étude des fonctions des gènes en raison de sa grande efficacité et de sa précision9. Le principe d’ingénierie CRISPR/Cas9 implique la conception et l’introduction d’ARNsg spécifiques avec environ 20 nucléotides pour guider la nucléase Cas9 afin de localiser et de couper avec précision les séquences d’ADN cibles, permettant ainsi l’édition de gènes telle que l’inactivation, l’activation ou la répression6. Ce principe sous-tend le développement de l’écran CRISPR à haut débit et personnalisable. En construisant des banques contenant un grand nombre d’ARNsg, les chercheurs peuvent simultanément modifier et cribler plusieurs gènes, identifiant rapidement ceux associés à des phénotypes ou à des états pathologiques spécifiques. De plus, le crible CRISPR permet des expériences dans divers types de cellules, de tissus et même d’organismes entiers, offrant une compréhension plus complète de la fonction des gènes et des mécanismes d’action.
Avec l’accélération du développement de la technologie, l’écran CRISPR a montré un énorme potentiel d’application dans divers domaines. Par exemple, l’écran CRISPR peut aider les scientifiques à découvrir de nouvelles cibles médicamenteuses, accélérant ainsi le processus de découverte et de développement de médicaments. Dans la recherche sur les maladies, cette technologie révèle les mécanismes de la pathogenèse des maladies, offrant de nouvelles perspectives et approches pour le diagnostic et le traitement. Le criblage CRISPR diffère fondamentalement des méthodes de criblage traditionnelles (telles que l’ARNi et les inhibiteurs de petites molécules) sur plusieurs aspects clés : Mécaniquement, CRISPR réalise des modifications génétiques permanentes par l’édition de gènes, tandis que l’ARNi ne supprime que temporairement l’expression des gènes et que les inhibiteurs de petites molécules bloquent de manière réversible la fonction protéique28,29. En termes de spécificité, CRISPR démontre une précision supérieure grâce à un ciblage précis de l’ADN, tandis que l’ARNi est sujet à des effets hors cible 3,5. En ce qui concerne la durée de l’effet, CRISPR crée des altérations génétiques permanentes, par rapport aux interventions transitoires des méthodes conventionnelles. De plus, CRISPR permet des capacités de criblage à l’échelle du génome et peut étudier simultanément plusieurs gènes/voies, tout en maintenant une efficacité élevée sur divers types de cellules9. Les approches traditionnelles se limitent généralement à l’analyse de voies spécifiques et montrent souvent une efficacité réduite dans certaines lignées cellulaires3. Ces caractéristiques distinctives rendent le criblage CRISPR particulièrement précieux pour la recherche en génomique fonctionnelle et la découverte de cibles, en particulier dans les études nécessitant une observation à long terme ou une régulation génétique précise, où il présente des avantages uniques par rapport aux méthodologies de criblage conventionnelles.
Cependant, la spécificité de ce processus n’est pas absolue, et il est possible que la protéine Cas9 reconnaisse et clive par erreur des régions de l’ADN qui sont similaires mais pas entièrement identiques à la séquence cible5 prévue. Cet événement est communément appelé effets hors cible. Ce clivage involontaire de l’ADN peut entraîner une instabilité génomique et des conséquences génétiques imprévisibles, en particulier lorsqu’il est appliqué en thérapie humaine. La technologie CRISPR a également du mal à traiter des tâches complexes d’édition génomique. Lorsqu’il est confronté à des demandes d’édition qui englobent de nombreux gènes, des groupes de gènes ou des paramètres génétiques complexes, le système CRISPR peut rencontrer des limites dans l’anticipation et l’orientation précises des résultats du processus d’édition1.
Le principal obstacle technique à la réalisation d’un criblage à l’échelle du génome à l’aide de la technologie CRISPR provient de la grande complexité du génome, qui englobe des dizaines de milliers de gènes, augmentant ainsi considérablement le risque d’effets hors cible9. Pour garantir l’intégrité et la fiabilité des résultats de séquençage ultérieurs, il est impératif que chaque cellule soit transfectée avec un seul ARNsg23. Cela nécessite un contrôle méticuleux du MOI par l’expérimentateur, en veillant à ce qu’il reste inférieur à 0,5 tout en maintenant un seuil de nombre de copies adéquat, qui doit être d’au moins 500 ou supérieur à24. Étant donné que chacun des dizaines de milliers de gènes de l’ensemble du génome nécessite l’utilisation de 4 à 6 ARNsg, l’ensemble du processus de criblage nécessite une grande quantité de cellules25. La durée prolongée de l’expérience, le nombre important de cellules impliquées et le risque élevé nécessitent que l’expérimentateur élabore des stratégies pour atténuer ces préoccupations26. Les gènes potentiels doivent être validés davantage par le séquençage de l’ARN, ainsi que par des tests de gain et de perte de fonction. Dans les expériences suivantes, après avoir obtenu la surexpression et les cellules knock-out des gènes identifiés à l’aide de l’édition CRISPR, la génomique et la transcriptomique à l’échelle du génome ont été réalisées pour accéder à des altérations potentielles hors cible, en particulier au niveau des loci avec une complémentarité partielle de l’ARNsg qui peuvent subir un clivage non spécifique de Cas9. Les outils de prédiction bio-informatiques tels que COSMID pourraient aider à identifier les sites hors cible à haut risque. Des tests de sauvetage pourraient également être utilisés pour garantir la spécificité de l’implication des gènes identifiés.
Dans l’analyse ultérieure, les critères pour les ARNsg exprimés de manière différentielle reposaient sur le score d’agrégation de rang robuste (RRA) calculé par MAGeCK, qui impliquait la signification statistique, le changement de pli et la cohérence de réplication31,32. Pour un ensemble de données, chaque gène est classé selon des critères complets combinant les niveaux d’expression des gènes et la signification statistique. RRA utilise le coefficient de corrélation de rang de Spearman pour le rang pour évaluer la situation du rang complet de chaque gène dans tous les ensembles de données et intègre les résultats de tri de plusieurs ensembles de données d’expression génique. Un gène ayant un rang relativement élevé dans la plupart des ensembles de données pourrait être considéré comme un véritable gène33 exprimé de manière différentielle. Une valeur p de RRA inférieure à 0,05 a été considérée comme statistiquement significative. Une variation de pli d’au moins 1,5 fois ou une diminution de 33 % a été considérée comme une expression différentielle. De plus, la cohérence de 3 répétitions indépendantes devait être considérée comme une expression statistiquement différente. Après le classement RRA pour les gènes essentiels, un score RRA plus faible signifie une importance plus élevée.
En tant qu’outil puissant pour l’édition du génome, la technologie CRISPR-Cas9 démontre un potentiel et une précision inégalés dans l’étude du domaine de la radiorésistance26,27. Cependant, son application présente encore des limites. Outre le risque d’effets hors cible, les subtilités associées à la radiosensibilité introduisent des complexités dans le processus de dépistage27. Cette sensibilité n’est pas simplement régie par un gène solitaire, mais émerge plutôt comme la conséquence de l’interaction complexe entre plusieurs gènes et leurs réseaux régulateurs26. Par conséquent, malgré l’identification réussie de gènes spécifiques relatifs à la radiosensibilité, ils pourraient ne représenter qu’une fraction du tableau plus large, posant des défis importants pour élucider de manière exhaustive les mécanismes sous-jacents 34,35,36. Voici des solutions aux problèmes courants du criblage CRISPR, tels que la faible efficacité de l’infection, la dégradation de l’ARNsg et l’échec du séquençage : étant donné que le criblage CRISPR nécessite une valeur de MOI <0,5, l’ajustement de l’état cellulaire est essentiel pour s’assurer que les cellules sont dans une phase de croissance logarithmique ou évaluer l’efficacité de l’infection via des tests de fluorescence ; les causes potentielles de dégradation de l’ARNsg comprennent la contamination par des nucléases ou des défauts de conception des vecteurs, de sorte que les cycles répétés de gel-dégel des banques d’ARNsg doivent être évités (stocker les aliquotes à -80 °C), et le séquençage NGS doit être utilisé pour vérifier l’intégrité de la banque d’ARNsg ; L’échec du séquençage peut résulter d’une profondeur de séquençage insuffisante ou de problèmes de conception d’amorces d’ARNsg, le séquençage à extrémités appariées est recommandé pour améliorer la précision de l’identification de la séquence d’ARNsg, avec une profondeur de séquençage suggérée de >500× une couverture de 37,38,39,40,41,42.
La radiothérapie reste une pierre angulaire du traitement du cancer, mais présente une variabilité remarquable de l’efficacité thérapeutique entre différents types de tumeurs et même entre les patients atteints du même cancer9. Cette hétérogénéité interindividuelle nécessite fréquemment des thérapies combinées pour obtenir une éradication tumorale optimale11,12. Dans la pratique clinique, l’intégration de la radiothérapie à l’immunothérapie - en particulier les inhibiteurs de points de contrôle immunitaires et les conjugués anticorps-médicament - est apparue comme un paradigme thérapeutique de premier plan19. Ce protocole présente un criblage CRISPR in vitro utilisant une lignée cellulaire cancéreuse relativement homogène, qui ne pourrait pas refléter totalement l’hétérogénéité génétique et épigénétique des tumeurs in vivo. Après avoir déterminé la dose de rayonnement appropriée pour le dépistage, les résultats fournissent des gènes potentiellement radiosensibles ou radiorésistants, qui doivent être confirmés à la fois dans des essais cellulaires in vitro et dans des modèles animaux in vivo. De plus, le criblage CRISPR in vivo et la transcriptomique unicellulaire pourraient être utiles pour déchiffrer la complexité de l’hétérogénéité tumorale. Le crible CRISPR in vitro a identifié des gènes importants dans la réparation des dommages à l’ADN au sein des cellules, ainsi que dans les mécanismes autocrines en réponse aux radiations, mais n’a pas entièrement reproduit les TME hétérogènes. Par exemple, l’hypoxie augmente la production de ROS, déclenchant une boucle de rétroaction qui stimule l’adaptation métabolique, la génération d’antioxydants et l’activation de l’autophagie, favorisant finalement la radiorésistance18. Les cellules immunitaires pourraient être activées dans le mécanisme paracrine après une radiation, induisant une radiosensibilité14. L’ECM sert de réservoir pour les facteurs de croissance, et les cytokines ont des effets à la fois positifs et négatifs sur la radiosensibilité21. L’écran CRISPR in vivo doit être réalisé pour refléter l’hétérogénéité des TME. En outre, le crible CRISPR in vitro n’a pu identifier que des gènes importants dans la réparation des dommages à l’ADN dans les cellules, ainsi que dans les mécanismes autocrines en réponse aux radiations, mais n’a pas pleinement capturé l’influence in vivo sur la radiosensibilité, y compris la vascularisation tumorale et les effets systémiques20,21. L’écran CRISPR in vivo doit être effectué pour identifier les facteurs potentiels impliqués dans la radiosensibilité in vivo.
Ici, un protocole complet visant à utiliser le criblage CRISPR à l’échelle du génome est conçu pour identifier les gènes impliqués dans la sensibilité ou la résistance à la radiothérapie. En démêlant de nouvelles voies, cette méthodologie cherche à ouvrir la voie à des avancées dans la recherche sur la radiothérapie tumorale. Ce protocole pourrait également servir de référence pour explorer les mécanismes des contraintes cliniques en chimiothérapie et la progression des agents thérapeutiques alternatifs. En résumé, la technologie CRISPR incarne un potentiel et une valeur importants, mais ses contraintes inhérentes soulignent l’importance d’une reconnaissance sobre et d’une approche judicieuse. Au fur et à mesure que les recherches et les applications futures sont approfondies, il est impératif de s’efforcer sans relâche d’explorer et d’optimiser la technologie CRISPR pour surmonter ses contraintes et exploiter son plein potentiel.
Déclarations de divulgation
Aucun.
Remerciements
Cette étude a été soutenue par le Projet régional d’innovation scientifique et technologique de la province du Hubei (2024EIA001) et le Projet de soutien à la construction de la plateforme d’innovation en sciences et technologies médicales de l’hôpital Zhongnan de l’Université de Wuhan (PTXM2025001). La figure 1 a été créée à l’aide de Figdraw.
matériels
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
Références
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon