Method Article
מסך CRISPR רחב גנום לחשיפת גנים רגישים לקרינה ועמידים לרדיו
* These authors contributed equally
In This Article
Summary
ניתנת גישה קפדנית ומובנית לבחירת גנים עמידים ורגישים לקרינה באמצעות יישום שיטת סקר CRISPR/Cas9 כלל-גנומית. לפרוטוקול זה יש גם פוטנציאל לשמש מסגרת רב-תכליתית למאמצי מחקר אחרים החוקרים את מנגנוני העמידות לתרופות כימיות הניתנות קלינית.
Abstract
מערכת CRISPR-Cas9 נרתמה והפכה לכלי רב עוצמה לעריכת גנום. על ידי מינוף טכנולוגיה זו, חוקרים יכולים לחתוך, להדביק ואפילו לשכתב במדויק רצפי DNA בתוך תאים חיים. עם זאת, היישום של טכנולוגיית מסך CRISPR חורג הרבה מעבר לניסויים גרידא. הוא משמש ככלי מרכזי במאבק במחלות גנטיות, מנתח באופן שיטתי נופים גנטיים מורכבים, מאפשר לחוקרים לפענח את המנגנונים המולקולריים העומדים בבסיס תופעות ביולוגיות, ומאפשר למדענים לזהות ולמקד את הגורמים השורשיים למחלות כמו סרטן, סיסטיק פיברוזיס ואנמיה חרמשית. בין כולם, הסרטן מציב אתגר אדיר לרפואה, ומדרבן את מאמצי המיגור. רדיותרפיה, כטיפול מסורתי, מניבה תוצאות אך יש לה מגבלות. הוא מחסל תאים סרטניים אך גם פוגע ברקמות בריאות, וגורם לתופעות לוואי המפחיתות את איכות החיים. בנוסף, לא כל תאי הסרטן מגיבים לרדיותרפיה, וחלקם עלולים לפתח עמידות, מה שמחמיר את המצב. כדי להתמודד עם זה, מוצגת טכנולוגיית סקר CRISPR מקיפה של גנום שלם, מכיוון שהיא מאפשרת זיהוי יעיל של גנים רגישים לקרינה ועמידים לקרינה, ובכך מקדמת את תחום המחקר והטיפול בסרטן. בדיקת CRISPR כלל-גנומית נערכה בתאי אדנוקרצינומה של הריאות שנחשפו להקרנה בעקבות הפרוטוקול המתואר, שבאמצעותו זוהו גנים הקשורים לעמידות לקרינה ולרגישות לקרינה.
Introduction
חקירת תופעות ביולוגיות שזורה מטבעה בחקר התנהגויות תאיות, ובתורה, בחינת התנהגויות תאיות קשורה באופן מהותי לחקר הגנום שלה. ככל שהטכנולוגיה המודרנית ממשיכה להתפתח, חוקרים רפואיים מפנים בהדרגה את תשומת ליבם לשינוי התנהגויות תאיות באמצעות עריכת גנים על מנת לשפר את תוצאות הטיפול במחלות שונות. בהקשר זה, טכנולוגיית CRISPR התגלתה ככלי מהפכני לעריכת גנום בשל היישום הפשוט יחסית שלה1. מערכת CRISPR-Cas9 מורכבת מנוקלאז Cas9 ו-RNA מדריך יחיד (sgRNA), המזהה ונקשר באופן ספציפי לרצף ה-DNA המטרה, ומנחה את נוקלאז Cas9 לחתוך במיקום זה, וכתוצאה מכך לשבור גדיל כפול (DSB) ב-DNA הגנומי 2,3,4. בנוסף, הכנסת חומרים אחרים יכולה להוביל להוספות, מחיקות או מוטציות ספציפיות בגנום, מה שמאפשר עריכת גנים ממוקדת.
במחקר גנומי פונקציונלי, בדיקת הפרעות RNA (RNAi) הייתה בעבר שיטה בשימוש נרחב לביצוע ניסויים בקנה מידה גדול של אובדן תפקוד כדי לחקור תפקידי גנים בסרטן. טכנולוגיית ה-RNAi חוקרת את תפקוד הגנים על ידי השתקה ספציפית של גני מטרה, ומסייעת לחוקרים לזהות גורמים אונקוגניים קריטיים. עם זאת, הוא מוגבל על ידי השפעות מחוץ למטרה ויעילות לא שלמה של הפלת גנים. השפעות מחוץ למטרה עלולות להוביל להשתקה של גנים אחרים שאינם מטרה, ובכך לפגוע בדיוק ובאמינות של תוצאות הניסוי 1,2. בנוסף, RNAi מפגין יעילות נוקדאון נמוכה עבור גנים מסוימים, מה שעלול להיכשל בדיכוי מלא של ביטוי גן המטרה. בניגוד למסכי RNAi מסורתיים, מסך ה-CRISPR מפגין ספציפיות ויעילות גבוהות יותר3. טכנולוגיה זו לא רק מאפשרת עריכה מדויקת של גנים ספציפיים, אלא גם מאפשרת סינון בקנה מידה גדול ברחבי הגנום, ומספקת תמיכה חזקה למחקר תפקוד הגנים. טכנולוגיית מסך CRISPR, כלי רב עוצמה לעריכת גנים המבוסס על מערכת CRISPR-Cas9, משמשת לסינון יעיל וחשיפה של פונקציות לא ידועות של גנים ספציפיים בתאים 5,6,7,8. חוקרים מתכננים sgRNAs בקבוצות עבור גנים או אזורי גנים ספציפיים, ומכינים ספריות sgRNA תואמות בדיוק ובקפדנות, מה שמבטיח את שלמותן ופונקציונליותן9. ספריות sgRNA אלה עטופות לאחר מכן לחלקיקים לנטי-ויראליים, המשמשים להדבקה יעילה של תאי מארח. לאחר הדבקה מוצלחת, התאים הנגועים מעובדים בתנאי סקר מוגדרים אישית. לאחר ההקרנה, ה-DNA הגנומי של התאים המוקרנים מופק תוך שמירה על סטנדרטים גבוהים של טוהר וכמות. לאחר מכן, אזורים ממוקדים בעלי עניין sgRNA נתונים להגברת PCR, תהליך המשכפל במדויק את המקטעים הרצויים של חומצות גרעין 3,9. לבסוף, ריצוף תפוקה גבוהה מבוצע על מקטעי ה-DNA המוגברים, המאפשר ניתוח מקיף ויעיל של האזורים הממוקדים, ובכך מספק תובנות חשובות לגבי תפקודם והתנהגותם של הגנים הנחקרים4.
סרטן מהווה איום אדיר על בריאות האדם כמחלה מורכבת. ברחבי העולם, חוקרים וקלינאים עושים מאמצים מרוכזים לחשוף את המנגנונים המולקולריים של סרטן ולפתח אסטרטגיות טיפוליות חדשות. שיתופי פעולה בינלאומיים הוקמו כדי להאיץ את התרגום של ממצאי מחקר בסיסיים ליישומים קליניים, במטרה הסופית לשפר את תוצאות המטופלים. Sasmal ואחרים הציעו אסטרטגיית הרכבה ביו-אורתוגונלית המבוססת על מערכת סינתטית של מארח-אורח למיקוד מדויק של תאי סרטן גרורתיים, מה שסייע באופן משמעותי לעשרות מדענים בקידום טכנולוגיות רפואיות. לעבודת המחקר המצטיינת שלהם יש חדשנות גבוהה ותובנות ייחודיות, התורמות תרומה משמעותית לקהילה המדעית10. סרטן מאופיין במצב סוער של חוסר יציבות גנומית, הנובע מוויסות לא יציב של תגובות נזק ל-DNA 11-14. נזק ל-DNA כולל פגמים בנוקלאוטידים בודדים, שברים חד-גדיליים ו-DSBs. רקומבינציה הומולוגית (HR) והצטרפות קצה לא הומולוגית (NHEJ) משתתפים בתיקון DSBs בשלבים שונים 15,16,17. על בסיס זה, רדיותרפיה התגלתה כאפשרות טיפול בת קיימא, המשתמשת בקרניים בעלות אנרגיה גבוהה (כגון צילומי רנטגן וצילומי γ) כדי להקרין רקמת גידול, ולגרום לנזק ל-DNA בתאי הגידול, ובכך לשבש את צמיחתם והתפשטותם18. עם זאת, רדיותרפיה לא תמיד מניבה את ההשפעות הרצויות בחלק ניכר מחולי הסרטן, שעלולות לנבוע מנזקים לרקמות הפרא-סרטניות וממגבלות המוטלות על ידי המאפיינים האינהרנטיים של הגידול, כגון רגישות נמוכה לרדיותרפיה 19,20,21.
תיאורטית, ניתן להשתמש בכל סוג תא עבור מסך CRISPR. עם זאת, שמירה על ייצוג מספיק באוכלוסיות מוטנטיות דורשת מספר רב של תאי התחלה. סוגי תאים עם שפע נמוך אינם מתאימים במיוחד למסך רחב של הגנום. באשר לבחירת הספרייה, רוב הספריות מכילות 3-6 gRNAs לכל גן מטרה, ושמירה על ההתפלגות של כל gRNA בתוך האוכלוסייה היא קריטית18. אובדן ייצוג עקב העשרה או דלדול של gRNAs ספציפיים עלול להוביל להתפלגות תוצאות לא אחידה. כדי לטפל בבעיה זו, בחירה בספריות CRISPR זמינות מסחרית שנבדקו בשוק עשויה להיות בחירה עדיפה20. במבחנה מסך CRISPR המשתמש בקווי תאים סרטניים הומוגניים עשוי שלא ללכוד באופן מלא את ההטרוגניות הגנטית והאפיגנטית של גידולים in vivo . בעוד שסקר במבחנה חשף גנים מרכזיים המעורבים בתיקון נזק ל-DNA ואיתות אוטוקריני המושרה על ידי קרינה, הוא לא שכפל באופן מלא את המיקרו-סביבה של הגידול, כולל עמידות לקרינה הנגרמת על ידי היפוקסיה (באמצעות ROS, הסתגלות מטבולית ואוטופגיה), השפעות פרקריניות בתיווך חיסוני ואפנון ציטוקינים תלויי ECM. לפני השימוש במסך הקריספר כדי לחקור גנים הקשורים לרגישות או עמידות לקרינה, יש לשקול היטב גורמים אלה. לאור נוף הטיפול הנוכחי, דחוף לזהות ולחקור לעומק גורמים הקשורים לעמידות לקרינה ורגישות לקרינה כדי לשפר ביעילות את יעילות הרדיותרפיה22. בהתחשב ביתרון העיקרי של מסך ה-CRISPR בחקר התפקודים של גנים לא ידועים, מסופקת טכנולוגיית מסך CRISPR מפורטת באופן שיטתי לזיהוי יעיל של גנים רגישים לקרינה ועמידים לקרינה.
Protocol
הריאגנטים והציוד המשמשים במחקר זה מפורטים בטבלת החומרים.
1. בחירת מינון קרינה מתאים
- הכנה וציפוי של תאים דבקים
- התאם את צפיפות התאים ל-5 x 105 תאים למ"ל עם מדיום תרבית תאים שלם RPMI 1640 המכיל 10% FBS למעבר וצמיחת תאים. מחלקים את התאים לצלחות תרבית בגודל 3.5 ס"מ לקרינה במינונים שונים. מוסיפים 2 מ"ל (1 x 106 תאים) לכל מנה ודוגרים למשך הלילה בטמפרטורה של 37 מעלות צלזיוס עם 5% CO2.
- יישום מינוני קרינה שונים
- מספר את כלי התרבות בגודל 3.5 ס"מ מ -1 עד 5. השתמש בקבוצה #1 כקבוצת הביקורת, כאשר 4 הקבוצות הנותרות מוגדרות כקבוצות הטיפול.
- אטום את הקצוות של לוחות 6 הבארות עם קרום איטום לקבוצות טיפול ומתן מנות קרינה של 2, 4, 6 ו-8 Gy, בהתאמה. לאחר הטיפול מחזירים את הכלים לחממה.
- ציפוי קווי תאים לאחר קרינה
- מכינים צלחות 6 באר ו -96 בארות. שטפו את התאים בכלי התרבית בגודל 3.5 ס"מ פעם אחת עם PBS, עכלו את התאים הגדלים באופן לוגריתמי עם 0.25% טריפסין, והתאימו את צפיפות התאים ל-1 x 105 תאים למ"ל עם מדיום שלם RPMI 1640 המכיל 10% FBS.
- זרע 10 מיקרוליטר / באר (1,000 תאים / 100 מיקרוליטר) לתוך צלחות 6 הבארות ב -3 משכפל עבור כל מנת קרינה. הוסף 2 מ"ל של מדיום שלם לכל באר וספור לאחר 14 יום (כאשר בכל קבוצת שיבוטים יש כ -50 תאים).
- זרע 30 מיקרוליטר / באר (3,000 תאים / 100 מיקרוליטר) לתוך צלחות 96 הבארות ב -5 שכפולים עבור כל מנת קרינה. הוסף 70 מיקרוליטר של מדיום שלם לכל באר ומדוד את כדאיות התא לאחר 72 שעות.
- מדידת כדאיות התאים
הערה: שיעור עיכוב קלונוגני = 1 - (מספר השיבוטים בקבוצת הטיפול/מספר השיבוטים בקבוצת הביקורת) x 100%. בדיקת CCK-8 משתמשת במלח טטרזוליום מסיס במים (WST-8) שניתן להפחית על ידי דהידרוגנאזות תאיות כדי ליצור מוצר פורמזן צהוב מסיס מאודבמים 11. כמות הפורמזן המיוצרת עומדת ביחס ישר למספר התאים הקיימאים, והצפיפות האופטית שלו (הנמדדת באורך גל של 450 ננומטר) משקפת במדויק את הפעילות המטבולית התאית ואת מצב ההתפשטות11,13. בהתבסס על עיקרון מבוסס זה, בדיקת CCK-8 אומצה באופן נרחב ליישומים שונים, כולל בדיקות התפשטות תאים ובדיקות רגישות לתרופות גידול. בפרוטוקול זה, מבחן CCK-8 משמש להערכה שיטתית של כדאיות תאית במינוני קרינה שונים.- מערבבים מגיב CCK8 עם מדיום RPMI 1640 ללא FBS ביחס של 1:9. השליכו את המדיום בצלחות 96 הבארות והוסיפו 100 מיקרוליטר מהמדיום המכיל CCK8 לכל באר.
- דגירה בחושך למשך שעה אחת ומדידת ערך ה-OD ב-450 ננומטר באמצעות קורא מיקרו-פלטות.
הערה: שיעור הישרדות התאים = [(ערך OD של קבוצת הטיפול - ערך OD של הבאר הריקה) / (ערך OD של קבוצת הביקורת - ערך OD של הבאר הריקה)] x 100%
- בחירת מינון קרינה
- לשלב את קצב העיכוב הקלונוגני עם שיעור ההישרדות של התא על מנת לבחור מינון קרינה מתאים בהתאם לצרכי המחקר. בחר מנת קרינה עם שיעור עיכוב של 50% לבדיקת גנים עמידים לקרינה ורגישים לקרינה.
2. בחירת ה-MOI וריכוז הפורומיצין המתאימים
- ציפוי תאים נדבקים
- התאם את צפיפות התאים ל -3 x 105 תאים למ"ל וחסן 1 מ"ל (3 x 105 תאים) לבאר לצלחת של 12 בארות. דגרו את הצלחת למשך הלילה ב-37 מעלות צלזיוס עם 5%CO2.
- זיהום לנטי-ויראלי
- השתמש בפיפטה כדי לשאוב את המדיום מצלחת 12 הבארות והחלף אותו ב-1 מ"ל של מדיום שלם RPMI 1640 המכיל 10% FBS. הכן ספריית CRISPR lentiviral של גנום שלם שנבדקה באיכות (מומלץ להשתמש בספריות זמינות מסחרית) והגדר שיפוע ריכוז לוגריתמי (למשל, 0-10-50-100-200-400-800)4,5,6,7,8.
- הוסף את הכמות המתאימה של lentivirus ל-2 מיקרוליטר לצלחת פוליברן ואזן בטמפרטורת החדר למשך 5 דקות. מטפטפים לאט את תערובת הלנטי-וירוס והפוליברן לכל באר, מערבבים היטב ודוגרים למשך הלילה ב-37 מעלות צלזיוס עם 5% CO2.
- קביעת ריכוז הפורומיצין המינימלי להרג תאים
- התאם את צפיפות התאים ההורית ל-3 x 105 תאים/מ"ל וחסן 1 מ"ל (3 x 105 תאים) לבאר לצלחת של 12 בארות. דגרו את הצלחת למשך הלילה ב-37 מעלות צלזיוס עם 5%CO2.
- הוסף פורומיצין לכל באר בצלחת 12 הבארות בשיפוע ריכוז של 0-0.1-0.2-0.5-1-2-4-8 מיקרומטר. ספרו את התאים לאחר 72 שעות. הריכוז המינימלי שהורג את כל התאים בבאר הוא ריכוז הפורומיצין המינימלי להרג תאים, שישמש להקרנה לאחר מכן של תאים נגועים ויראליים.
- בחירת פורומיצין לאחר הדבקה
- ביום השני לאחר ההדבקה יש לשאוב את המדיום מכל באר ולהחליף אותו ב-1 מ"ל של מדיום שלם RPMI 1640 המכיל 10% FBS. המשך בתרבות במשך 48 שעות.
- לאחר 72 שעות של הדבקה, החלף את המדיום הקיים במדיום שלם המכיל את ריכוז הפורומיצין המינימלי להרג תאים. הגדר 2 בארות בצלחת 12 הבארות ללא זיהום לנטי-ויראלי כבקרות שליליות וחיוביות. טפל בשליטה השלילית עם פורומיצין, והשאיר את השליטה החיובית ללא טיפול. המשך בתרבות במשך 72 שעות.
- אחרי 72 שעות של בחירת פורומיצין, כל תאי ההורים בביקורת השלילית ימותו, בעוד שתאי ההורים בביקורת החיובית מראים מוות מינימלי. חשב את ה-MOI עבור כל באר.
הערה: MOI = (מספר התאים בקבוצה הנגועה בנגיף / מספר התאים בקבוצת הביקורת החיובית) x 100%. השתמש בריכוז הנגיף עם MOI של ~0.3 לבדיקה הבאה.
3. זיהום בספריית CRISPR lentiviral ברחבי הגנום
- זריעה של קווי תאים דבקים
- התאם את צפיפות התאים ל-1 x 107 תאים למ"ל במדיום שלם RPMI 1640 המכיל 10% FBS למעבר וצמיחת תאים. חסנו 1 מ"ל תאים (1 x 107 תאים) לכל צלחת תרבית בגודל 15 ס"מ ב-37 מעלות צלזיוס עם 5% CO2 למשך 8 שעות. ברגע שהתאים נדבקו והגיעו למפגש של 70%-80%, הם היו מוכנים לזיהום ויראלי (מספר עותקים של כ-500).
- זיהום לנטי-ויראלי
- שאפו את המדיום מכלי התרבות בגודל 15 ס"מ והחליפו אותו ב-15 מ"ל של מדיום שלם RPMI 1640 המכיל 10% FBS. הכן ספריית CRISPR lentiviral של גנום שלם שנבדקה באיכות.
- מוסיפים את הכמות המתאימה של לנטי-וירוס עם MOI = 0.3 עד 30 מיקרוליטר לצלחת פוליברן ומאזן בטמפרטורת החדר למשך 5 דקות, מטפטפים לאט את תערובת הלנטי-וירוס והפוליברן לתוך כלי התרבית בגודל 15 ס"מ, מערבבים היטב ודוגרים ב-37 מעלות צלזיוס עם 5% CO2 למשך הלילה. במקביל, הכינו צלחת תרבית בגודל 15 ס"מ עם תא ההורה כניגוד.
- בחירת פורומיצין לאחר הדבקה
- ביום השני לאחר ההדבקה הנגיפית, שאפו את המדיום מכלי התרבות בגודל 15 ס"מ והחליפו אותו ב-15 מ"ל של מדיום שלם RPMI 1640 המכיל 10% FBS. המשך בתרבות במשך 48 שעות.
- לאחר 72 שעות לאחר ההדבקה, החלף את המדיום במדיום שלם המכיל את הריכוז הקטלני המינימלי של פורומיצין. התייחסו לתאי ההורים הלא נגועים כאל ביקורת שלילית באותו אופן והמשיכו בתרבית במשך 72 שעות. לאחר 72 שעות של בחירת פורומיצין, תאי ההורים בקבוצת הביקורת השלילית ייהרגו כולם, והתאים ששרדו מהזיהום הלנטי-ויראלי נחשבים נגועים בהצלחה.
- מיצוי הגנום של יום 0
- עכל את התאים מצלחת תרבית אחת בגודל 15 ס"מ באמצעות 0.25% טריפסין, השהה מחדש במדיום שלם RPMI 1640 המכיל 10% FBS, וספור את מספר התא. סמן את הדגימה הזו כיום 0.
- צנטריפוגה בחום של 300 x גרם למשך 5 דקות (בטמפרטורת החדר) והשליכו את הסופרנטנט. השעו מחדש ב-1 מ"ל PBS, צנטריפוגה ב-300 x גרם למשך 5 דקות, והשליכו את הסופרנטנט. לחלץ DNA גנומי של יום 0, למדוד את ריכוז ה-DNA והטוהר באמצעות ספקטרופוטומטר UV ננו-טיפה.
- לזיהוי שלמות sgRNA, השתמש באלקטרופורזה של ג'ל אגרוז כדי לנתח את ספריית ה-sgRNA המוגברת, ולהבטיח שהרצועות היו ברורות וללא השפלה משמעותית, ובכך לשמור על שלמות הספרייה23. להערכת כיסוי הספרייה הלנטיויראלית, השתמש בטכנולוגיית ריצוף עמוק כדי לבצע ניתוח ריצוף על ה-sgRNAs בתוך הספרייה24,25.
- במסך CRISPR, הגברת PCR משמשת כאימות ראשוני של תקינות מקטעי sgRNA ואיכות הספרייה6. כדי לנתח בצורה מקיפה יותר את כיסוי הספרייה ואת דפוסי התפלגות ה-sgRNA, וכדי להבטיח ייצוג הולם של sgRNAs עבור כל גן יעד במהלך ההקרנה, בצע NGS לתובנות מעמיקות לגבי החוסן של מסך9.
הערה: על ידי זיהוי שינויים בשפע sgRNA לפני ואחרי ההקרנה, וזיהוי אינטגרציה פוטנציאלית של sgRNA מחוץ למטרה או זיהום ספרייה, ניתן לשפר עוד יותר את הדיוק והאמינות של מסך ה-CRISPR. דגימות יום 0 אלה משמשות כבקרות שליליות מכריעות ועוברות הערכת איכות מקיפה באמצעות הגברת PCR ו-NGS כדי להשיג 2 יעדים מרכזיים: (1) אישור של דלדול גנים חיוני (המצביע על ייצוג ספרייה הולם), ו-(2) הדגמה של ביטוי יציב בגנים לא חיוניים (קביעת תנאי בסיס ניסיוניים)9. - ודא שהכיסוי מגיע לרמה המצופה כדי להבטיח את הגיוון והייצוגיות של הספרייה ולמנוע הטיות בתהליך הסינון. למדידת יעילות הזיהום, השתמש בהדמיה פלואורסצנטית כדי להעריך את יעילות הזיהום, ולהבטיח שהיא עומדת בדרישות הניסוי.
4. יישום קרינה כתנאי סקר
- קיבוץ
- התאם את צפיפות התאים הנגועים בקריספר ל-1 x 107 תאים למ"ל וחסן 1 מ"ל לכל צלחת פטרי בגודל 15 ס"מ. דגירה למשך הלילה ב-37 מעלות צלזיוס עם 5%CO2.
- טיפול בקרינה
- חלקו את התאים באופן אקראי ל-2 קבוצות: קבוצת טיפול וקבוצת ביקורת, עם 6 מנות בקבוצה. מתן מינון מתאים של קרינה לתאים בקבוצת הטיפול, תוך השארת תאי קבוצת הביקורת ללא טיפול להתפשטות רגילה. לאחר ההקרנה, המשיכו לדגור בטמפרטורה של 37 מעלות צלזיוס עם 5%CO2 למשך 7 ימים.
- בשבוע השני, חזור על מתן מינון מתאים של קרינה לתאי קבוצת הטיפול, והשאיר את תאי קבוצת הביקורת ללא טיפול ואפשר להם להתפשט כרגיל. לאחר ההקרנות, יש להמשיך לדגור בטמפרטורה של 37 מעלות צלזיוס עם 5% CO2 למשך 7 ימים נוספים.
5. מיצוי וריצוף גנום
- מיצוי היום ה-14
- לאחר 14 יום של טיפול, עכל את התאים בקבוצות הטיפול והביקורת עם 0.25% טריפסין, השהה אותם מחדש באמצעות מדיום RPMI 1640 מלא המכיל 10% FBS, ותייג אותם כ-Day 14-RT או Day 14-NC.
- צנטריפוגה של התאים ב-300 x גרם למשך 5 דקות והשליכו את הסופרנטנט. השעו מחדש ב-1 מ"ל של PBS, צנטריפוגה ב-300 x גרם למשך 5 דקות, והשליכו שוב את הסופרנטנט. חלץ את ה-DNA הגנומי של יום 14, וזהה את הריכוז והטוהר של ה-DNA באמצעות ספקטרוסקופיה של ספיגת UV nanodrop.
- הגברת PCR
- הכן פריימרים מתאימים של הרצף (ראה טבלת חומרים) ודלל אותם ל-10 מיקרומטר.הגדר מערכת תגובה של 20 מיקרוליטר על ידי הוספת הריאגנטים לצינור מיקרו-צנטריפוגה סטרילי, צנטריפוגה למשך 5 שניות ב-300 x גרם, וערבב היטב. הגדר את פרמטרי מכשיר ה-PCR בהתאם למצב ההגברה כדי להשיג מוצרי PCR.
- זיהוי אלקטרופורזה של ג'ל אגרוז
- מכינים צלחת יציקת ג'ל, אוטמים את שולי התבנית באגרוז, מכניסים את המסרק ומכינים ג'ל אגרוז בריכוז מתאים לפי אורך ה-DNA של הדגימה.
- שקלו במדויק כמות מסוימת של אבקת אגרוז, הוסיפו כמות מתאימה של מאגר אלקטרופורזה, ערבבו היטב וחממו במיקרוגל להמסה. לאחר קירור קל, מוסיפים כמות מתאימה של צבע חומצת גרעין, מערבבים בעדינות ויוצקים לאט לתבנית יציקת הג'ל. הניחו לג'ל להתמצק למשך 30 דקות.
- הסר את המסרק והוסף כמות מתאימה של מאגר אלקטרופורזה למיכל האלקטרופורזה עד שהוא מכסה את הג'ל. הוסף כמות מתאימה של מאגר טעינה לדגימת ה-DNA, ערבב היטב והשתמש בפיפטה כדי להוסיף לאט את התערובת לבאר הדגימה.
- הגדר את המתח המתאים על סמך גודל שבר ה-DNA וריכוז ג'ל האגרוז. לאחר האלקטרופורזה, הסר בזהירות את הג'ל והנח אותו בהדמיית ג'ל כדי לצפות בתוצאות ולבדוק אם הגברת ה-PCR הצליחה.
- ריצוף אילומינה
- אסוף את ה-DNA הגנומי משלוש הקבוצות (קבוצת יום 0, קבוצת ביקורת וקבוצת טיפול), ושלח אותו לחברה לבניית ספריות וריצוף אילומינה. בצע ניתוח ביואינפורמטי והדמיה של תוצאות הריצוף כדי להשיג גנים רגישים לרדיותרפיה ועמידים לרדיותרפיה26.
- הגדר מספר ניסויים חוזרים לניתוח נתונים, ובצע ניתוח סטטיסטי על תוצאות הניסוי כדי להבטיח את העקביות והשחזור של הנתונים27,28.
- הערכת איכות של נתוני ריצוף
- הכפיף את הנתונים הגולמיים למנגנוני סינון ובקרת איכות קפדניים כדי להבטיח את הדיוק של מידע הרצף5. חשב את ציון ה-Phred (Qphred) עבור כל נוקלאוטיד על סמך שיעור שגיאות הרצף, באמצעות אלגוריתם המרה שצוין ומודל המעריך את הסבירות להתרחשות שגיאה במהלך קריאת בסיס28.
- שמור על שיעור שגיאות הריצוף עבור כל מיקום בסיס מתחת ל-1% (שווה ערך לסף Q30), וודא שלפחות 80% מנתוני הריצוף משיגים תקן Q30 זה כדי לתמוך בהליכים אנליטיים עוקבים.
- עיבוד נתונים ובקרת איכות ברמת המדגם
הערה: לפרטים, עיין בדוחות שפורסמו בעבר29,30.- יישר את הקריאות המסוננות מנתוני רצף עם רצפי ספריית sgRNA. דווח על סטטיסטיקה, כולל ספירת ה-sgRNAs בספרייה עם יישור מושלם, השפע הממוצע של sgRNAs, מספר ה-sgRNAs שלא זוהו, ושיעור הקריאות מדגימות בודדות שמופו בהצלחה לספריית sgRNA. השתמש ביחס מיפוי גבוה יותר כדי לציין כיסוי גדול יותר.
- ספרו את העשרת הקריאות עבור כל גן (ממוקד על ידי sgRNAs שונים) בכל דגימה. נרמל את הקריאות התומכות עבור כל sgRNA על פני דגימות באמצעות שיטת הנורמליזציה "החציונית" של MAGeCK. בצע בקרת איכות ברמת המדגם על ידי הערכת התפלגות ספירות הקריאה של sgRNA, יצירת עלילות תיבה, ביצוע ניתוח רכיבים עיקריים (PCA) ובניית מפות חום מתאם.
- נניח שספירת הקריאה של sgRNA עוקבת אחר התפלגות פואסון. תאר ספירות sgRNA מנורמלות מקבוצות שונות בעלילות קופסאות כדי לדמיין את התפלגות הנתונים הכוללת בתוך דגימות ולהשוות התפלגויות בין קבוצות.
- השתמש ב-PCA כדי לפשט את ניתוח הנתונים על-ידי שיקוף ההבדלים בין המרכיבים העיקריים וקצב השונות בתוך כל רכיב, מה שמקל על התבוננות בווריאציות תוך-קבוצתיות ובין-קבוצתיות. השתמש במפת חום של מתאם כדי להמחיש את הקשרים בין דגימות.
- ניתוח בסיסי
- לבצע ניתוח sgRNA דיפרנציאלי בין קבוצות ולזהות גנים חיוניים לאחר בקרת איכות ועיבוד נתונים ראשוני. לבצע ניתוח העשרה תפקודית של הגנים הרלוונטיים30. דרג גנים חיוניים על סמך ציון צבירת הדירוג החזק (RRA) המחושב על ידי MAGeCK או שיטות סטטיסטיות אחרות כגון MLE, עם ציון RRA נמוך יותר המצביע על חשיבות גבוהה יותר.
- השתמש בשפת R או בשפות תכנות אחרות כדי לבצע ניתוח ביואינפורמטי על התוצאות המדורגות, והמחיש את הממצאים באמצעות דיאגרמות ניתוח GO ו-KEGG30. קשר גנים או חלבונים למונחי GO מתאימים (תפקוד מולקולרי, תהליך ביולוגי ומרכיב תאי) באמצעות מיפוי מזהה או הערת רצף.
- השתמש ב-KEGG כמסד נתונים להבנת פונקציות ברמה גבוהה ומערכות ביולוגיות, תוך חיבור קטלוג הגנים המזוהה לתפקודי מערכת ברמת התא, המינים והמערכת האקולוגית. בחר את 10 או 20 המסלולים המובילים מניתוחי GO ו-KEGG לתצוגה אינטואיטיבית של כיווני המסלול.
תוצאות
סרטן הריאות, עם שיעור התמותה המוביל, מייצג מחלה רפואית אגרסיבית ונפוצה ביותר. תוך שימוש בקו תאי סרטן הריאה A549 כדוגמה לביצוע מסך CRISPR בכל הגנום עם קרינה כתנאי ההקרנה, זרימת העבודה הסכמטית מוצגת באיור 1. ראשית, חקרו את הרגישות של תאי A549 למינונים שונים של קרינה באמצעות היווצרות שיבוטים וניסויי CCK8 (איור 2). בבדיקה הקלונוגנית, ספירת המושבה הייתה 140 ± 5.35 ב-2 Gy לעומת 303 ±-31.63 ב-0 Gy, בעוד שבבדיקת CCK-8, ערך הצפיפות האופטית (OD) היה 0.65 ±-0.05 ב-2 Gy לעומת 1.35 ±-0.08 ב-0 Gy. כדי לחקור גנים מועמדים לרגישות לקרינה ועמידות לקרינה, בחר 2 Gy (IC50) כמינון הקרינה הבא. תאי A549 טופלו במינונים שונים של פורומיצין ונספרו לאחר 72 שעות. ריכוז ההרג המינימלי של פורומיצין עבור תאי A549 זוהה כ-1 מיקרומטר (איור 3). תאי A549 נדבקו ב-lentiviruses ב-MOIs שיפועים שונים. לאחר 72 שעות של הדבקה, יעילות ההדבקה נצפתה על ידי מיקרוסקופ פלואורסצנטי ונספרה לפני ואחרי 72 שעות של טיפול בפורומיצין. ה-MOI עם יעילות זיהום של ~0.3 התקבל (טבלה 1). הגנום האנושי מכיל 19,050 גנים, עם 6 sgRNA המתאימים לכל גן ומספר עותק של 500 (19,050 x 6 x 500 = 5.7 x 107 תאים). שימוש ב-lentiviruses עם MOI = 0.3 ובהתאם לכך מדביק 5.7 x 107 תאי A549. לאחר 72 שעות, התאים טופלו ב-1 מיקרומטר פורומיצין למשך 72 שעות נוספות. הגנום של יום 0 נקצר, ובוצעה הגברת PCR. רצפי ה-sgRNA בספריית הגנום השלם של CRISPR מציגים אורך של 231 זוגות בסיסים, אשר מאושש על ידי תוצאות האלקטרופורזה של ג'ל אגרוז (איור 4).
מוצרי ה-PCR נשלחו לאחר מכן לנובוגן לבקרת איכות נתונים ובקרת איכות ברמת המדגם. יחס המיפוי הניב שיעור כיסוי מקיף של כ-60%, מדד שנחשב מתאים להליכי סינון גנום שלם, בהתחשב במורכבות העצומה והשחיקה הבלתי נמנעת במהלך עיבוד גנום שלם (איור 5). ספירת קריאת ה-sgRNA דבקה בהתפלגות פואסון, בהתאם לציפיות התיאורטיות. הניתוח שלאחר מכן באמצעות תרשים PCA ומיפוי חום מתאם תיאר בצורה חיה את הפער הניתן להבחנה בין קבוצות נפרדות, כאשר שונות בין-קבוצתית עולה באופן ניכר על פערים בתוך קבוצות. יתר על כן, שיעור השונות בתוך המדגם המקובץ נפל בגבולות מקובלים, מה שמוכיח את הצלחת אמצעי בקרת האיכות ברמת המדגם. לאחר מכן, דירוגי ה-RRA שפותחו על ידי MAGeCK יירתמו כדי להתחיל בהערכה ביואינפורמטית בסיסית של תוצאות הדירוג תוך שימוש בשפת R. יש לציין כי 15 המסלולים המובילים במונחי GO הציגו באופן בולט מנגנונים הקשורים לנזק ל-DNA, מה שמתיישב בצורה חלקה עם קריטריוני הסינון הבסיסיים של הניסוי.

איור 1: סכמטי של מסך CRISPR/Cas9 של רדיותרפיה כלל-גנומית בתאי סרטן ריאות A549. ייצוג סכמטי של זרימת העבודה של הסינון בתאי A549 באמצעות CRISPR/Cas9 ברחבי הגנום והקרנות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
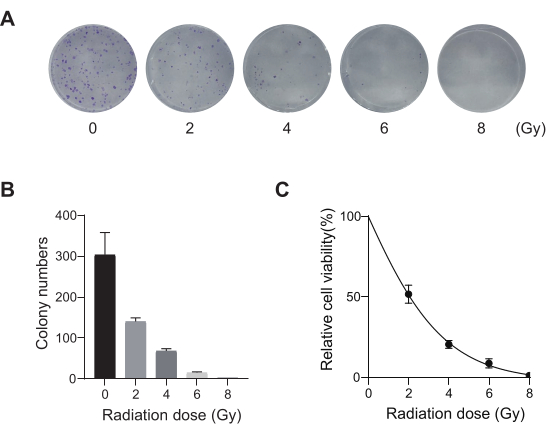
איור 2: אופטימיזציה של מינון קרינה עבור תאי A549. (A) תאי A549 נחשפו למינונים הולכים וגדלים של קרינה (0 Gy, 2 Gy, 4 Gy, 6 Gy ו-8 Gy) ואפשרו להם לגדול במשך 14 ימים. מוצגות תמונות בדיקת היווצרות מושבה. התוצאות מייצגות שני ניסויים ביולוגיים עצמאיים. (B) תוצאות כימות של תמונות בדיקת היווצרות משובטות. קווי שגיאה מייצגים סטיית תקן (n = 3). ANOVA חד כיוונית הניבה P < 0.01. התוצאות מייצגות שני ניסויים ביולוגיים עצמאיים. (C) עקומות הישרדות מינון-תגובה של תאי A549 בעקבות חשיפה למינוני קרינה שונים. קווי שגיאה מייצגים סטיית תקן (n = 3). ANOVA חד כיוונית הניבה P < 0.01. התוצאות מייצגות שני ניסויים ביולוגיים עצמאיים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
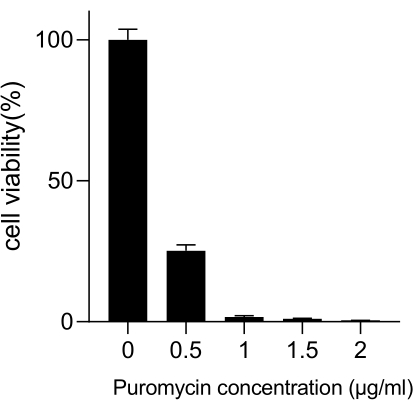
איור 3: קביעת ריכוז הפורומיצין המינימלי הנדרש לבחירת תאי A549. תאי A549 טופלו בריכוזים הולכים וגדלים של פורומיצין והודגרו במשך 72 שעות. קווי שגיאה מייצגים סטיית תקן (n = 3). ANOVA חד כיוונית הניבה P < 0.01. התוצאות מייצגות שני ניסויים ביולוגיים עצמאיים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
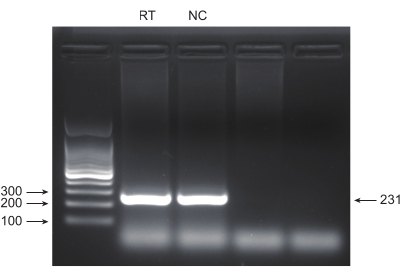
איור 4: אימות מוצר PCR על ידי אלקטרופורזה של ג'ל אגרוז. נעשה שימוש באלקטרופורזה של ג'ל אגרוז כדי לבחון את הנוכחות והאיכות של רצועות מוצר PCR. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 5: בקרת איכות וניתוח בסיסי של מוצרים מוגברים PCR. (A) יחס המיפוי הצביע על כיסוי של כ-60% של גנום הייחוס. (B) התפלגות ספירת קריאה של sgRNAs עקבה אחר התפלגות פואסון. (C) רוב 10 המונחים המובילים באונטולוגיה גנטית (GO) היו קשורים לתגובה לנזק ל-DNA. (D) תרשים הר געש שמראה דפוסי העשרה של גנים, עם גנים מועשרים שלילית באדום, גנים מועשרים חיובית בכחול וגנים לא משמעותיים באפור. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| חלקיק וירוס מספר (סמנכ"ל) | מספר ספירה (לפני טיפול בפורומיצין) | מספר ספירה (לאחר טיפול בפורומיצין) | זיהום יעילות |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
טבלה 1: ספירת תאים A549 לפני ואחרי בחירת פורומיצין תחת חלקיקי וירוס שונים. ספירות של תאי A549 שתועדו לפני ואחרי הטיפול בפורומיצין תחת חלקיקי נגיף משתנים.
Discussion
כטכנולוגיה מתקדמת לעריכת גנים, מסך הקריספר עורר שינויים עמוקיםבתחום המחקר המדעי. טכנולוגיה זו, הנובעת ממערכת CRISPR-Cas9, הפכה לכלי חיוני לחקר תפקודי גנים בשל יעילותהודיוקה הגבוהים. העיקרון ההנדסי של CRISPR/Cas9 כולל תכנון והכנסת sgRNAs ספציפיים עם כ-20 נוקלאוטידים כדי להנחות את נוקלאז Cas9 לאתר ולחתוך במדויק רצפי DNA מטרה, מה שמאפשר עריכת גנים כגון נוקאאוט, הפעלה או דיכוי6. עיקרון זה עומד בבסיס הפיתוח של מסך CRISPR בעל תפוקה גבוהה וניתן להתאמה אישית. על ידי בניית ספריות המכילות מספר עצום של sgRNAs, חוקרים יכולים לערוך ולסנן בו זמנית גנים מרובים, ולזהות במהירות את אלה הקשורים לפנוטיפים ספציפיים או למצבי מחלה. יתר על כן, מסך הקריספר מאפשר ניסויים בסוגי תאים שונים, רקמות ואפילו אורגניזמים שלמים, ומספק הבנה מקיפה יותר של תפקוד הגנים ומנגנוני הפעולה.
עם התפתחות הטכנולוגיה המואצת, מסך הקריספר הראה פוטנציאל אדיר ליישום בתחומים שונים. לדוגמה, מסך CRISPR יכול לסייע למדענים בגילוי מטרות תרופות חדשות, ובכך להאיץ את תהליך הגילוי והפיתוח של תרופות. בחקר מחלות, טכנולוגיה זו חושפת את המנגנונים של פתוגנזה של מחלות, ומציעה תובנות וגישות חדשות לאבחון וטיפול. מסך CRISPR שונה באופן מהותי משיטות סינון מסורתיות (כגון RNAi ומעכבי מולקולות קטנות) בכמה היבטים מרכזיים: מבחינה מכנית, CRISPR משיג שינויים גנטיים קבועים באמצעות עריכת גנים, בעוד ש-RNAi מדכא רק באופן זמני את ביטוי הגנים ומעכבי מולקולות קטנות חוסמים באופן הפיך את תפקוד החלבון28,29. מבחינת ספציפיות, CRISPR מפגין דיוק מעולה באמצעות מיקוד DNA מדויק, בעוד ש-RNAi נוטה להשפעות מחוץ למטרה 3,5. לגבי משך ההשפעה, CRISPR יוצר שינויים גנטיים קבועים, בהשוואה להתערבויות החולפות של שיטות קונבנציונליות. יתר על כן, CRISPR מאפשר יכולות סינון בכל הגנום ויכול לחקור בו זמנית גנים/מסלולים מרובים, תוך שמירה על יעילות גבוהה על פני סוגי תאים מגוונים9. גישות מסורתיות מוגבלות בדרך כלל לניתוח מסלולים ספציפיים ולעתים קרובות מראות יעילות מופחתת בשורות תאים מסוימות3. מאפיינים ייחודיים אלה הופכים את מסך הקריספר לבעל ערך מיוחד למחקר גנומי פונקציונלי וגילוי מטרות, במיוחד במחקרים הדורשים תצפית ארוכת טווח או בקרה גנטית מדויקת, שם הוא מדגים יתרונות ייחודיים על פני מתודולוגיות סקר קונבנציונליות.
עם זאת, הספציפיות של תהליך זה אינה מוחלטת, וייתכן שחלבון Cas9 יזהה בטעות ויבקע אזורי DNA הדומים אך לא זהים לחלוטין לרצף המטרההמיועד 5. התרחשות זו מכונה בדרך כלל אפקטים מחוץ למטרה. מחשוף DNA לא מכוון זה יכול להוביל לחוסר יציבות גנומית ולהשלכות גנטיות בלתי צפויות, במיוחד כאשר הוא מיושם בטיפול בבני אדם. טכנולוגיית CRISPR מתקשה גם בהתמודדות עם משימות עריכה גנומיות מורכבות. כאשר מתמודדים עם דרישות עריכה המקיפות גנים רבים, אשכולות גנים או הגדרות גנטיות מורכבות, מערכת הקריספר עשויה להיתקל במגבלות בחיזוי מדויק ובהכוונה של תוצאות תהליך העריכה1.
המכשול הטכני העיקרי בביצוע סריקה כלל-גנומית באמצעות טכנולוגיית CRISPR נובע מהמורכבות העצומה של הגנום, המקיף עשרות אלפי גנים, ובכך מגדיל משמעותית את הסיכוןלהשפעות מחוץ למטרה. כדי להבטיח את השלמות והאמינות של תוצאות הריצוף הבאות, הכרחי שכל תא יועבר עם sgRNA23 יחיד. זה מחייב בקרה קפדנית של ה-MOI על ידי הנסיין, להבטיח שהוא יישאר מתחת ל-0.5 ובמקביל לשמור על סף מספר עותק הולם, שחייב להיות לפחות 500 או יותר24. בהתחשב בעובדה שכל אחד מעשרות אלפי הגנים בגנום כולו מחייב ניצול של 4-6 sgRNAs, כל תהליך הסינון מצריך כמות גדולה של תאים25. משך הניסוי הממושך, המספר המשמעותי של התאים המעורבים והסיכון המוגבר מחייבים את הנסיין לתכנן אסטרטגיות כדי להקל על חששות אלה26. הגנים הפוטנציאליים נדרשים לעבור אימות נוסף באמצעות RNA-seq, כמו גם מבחני רווח ואובדן תפקוד. בניסויים הבאים, לאחר השגת ביטוי יתר ותאי נוקאאוט של גנים שזוהו באמצעות עריכת CRISPR, בוצעו גנומיקה וטרנסקריפטומיקה ברחבי הגנום כדי לגשת לשינויים פוטנציאליים מחוץ למטרה, במיוחד בלוקוסים עם השלמת sgRNA חלקית שעשויים לעבור מחשוף Cas9 לא ספציפי. כלי חיזוי ביואינפורמטיקה כגון COSMID עשויים לסייע בזיהוי אתרים בסיכון גבוה מחוץ למטרה. ניתן להשתמש במבחני הצלה גם כדי להבטיח את הספציפיות של מעורבות הגנים המזוהים.
בניתוח שלאחר מכן, הקריטריונים ל-sgRNAs המתבטאים באופן דיפרנציאלי הסתמכו על ציון צבירת הדירוג החזק (RRA) שחושב על ידי MAGeCK, שכלל מובהקות סטטיסטית, שינוי קיפול ועקביות שכפול31,32. עבור מערך נתונים, כל גן מדורג על פי קריטריונים מקיפים המשלבים רמות ביטוי גנים ומובהקות סטטיסטית. RRA משתמש במקדם מתאם הדירוג של ספירמן כדי להעריך את מצב הדירוג המקיף של כל גן בכל מערכי הנתונים ומשלב את תוצאות המיון של מערכי נתונים מרובים של ביטוי גנים. גן בעל דירוג גבוה יחסית ברוב מערכי הנתונים עשוי להיחשב כגן אמיתי המתבטא באופן דיפרנציאלי33. ערך p של RRA נמוך מ-0.05 נחשב מובהק סטטיסטית. שינוי של עלייה של לפחות פי 1.5 או ירידה של 33% נחשב כביטוי דיפרנציאלי. בנוסף, העקביות של 3 שכפולים בלתי תלויים נדרשה להיחשב כביטוי שונה מבחינה סטטיסטית. לאחר דירוג RRA עבור גנים חיוניים, ציון RRA נמוך יותר מסמל חשיבות גבוהה יותר.
ככלי רב עוצמה לעריכת גנום, טכנולוגיית CRISPR-Cas9 מדגימה פוטנציאל ודיוק שאין שני להם בחקירת תחום העמידות לקרינה26,27. עם זאת, ליישום שלו עדיין יש מגבלות. מלבד הסיכון להשפעות מחוץ למטרה, המורכבויות הקשורות לרגישות לקרינה מכניסות מורכבויות לתהליך הסינון27. רגישות זו אינה נשלטת רק על ידי גן בודד, אלא מופיעה כתוצאה מיחסי הגומלין המורכבים בין גנים מרובים ורשתות הבקרה שלהם26. לפיכך, למרות הזיהוי המוצלח של גנים ספציפיים הקשורים לרגישות לקרינה, הם עשויים לייצג רק חלק קטן מהתמונה הרחבה יותר, מה שמציב אתגרים משמעותיים להבהרה מקיפה של המנגנונים הבסיסיים 34,35,36. להלן פתרונות לבעיות נפוצות במסך CRISPR, כגון יעילות זיהום נמוכה, פירוק sgRNA וכשל בריצוף: מכיוון שמסך CRISPR דורש ערך MOI <0.5, התאמת מצב התא היא קריטית כדי להבטיח שהתאים נמצאים בשלב צמיחה לוגריתמי או להעריך את יעילות הזיהום באמצעות מבחני פלואורסצנטיות; הגורמים הפוטנציאליים לפירוק sgRNA כוללים זיהום נוקלאז או פגמים בתכנון וקטור, ולכן יש להימנע ממחזורי הקפאה-הפשרה חוזרים ונשנים של ספריות sgRNA (לאחסן ציטוטים ב-80 מעלות צלזיוס), ויש להשתמש בריצוף NGS כדי לאמת את תקינות ספריית sgRNA; כשל בריצוף עלול לנבוע מעומק ריצוף לא מספיק או מבעיות בתכנון פריימר sgRNA, רצף קצה מזווג מומלץ כדי לשפר את דיוק זיהוי רצף sgRNA, עם עומק ריצוף מוצע של כיסוי >500× 37,38,39,40,41,42.
רדיותרפיה נותרה אבן פינה בטיפול בסרטן, אך מציגה שונות יוצאת דופן ביעילות הטיפולית בין סוגי גידולים שונים ואפילו בקרב חולים עם אותו סרטן9. הטרוגניות בין-אישית זו מחייבת לעתים קרובות טיפולים משולבים כדי להשיג מיגור אופטימלי של הגידול11,12. בפרקטיקה הקלינית, השילוב של רדיותרפיה עם אימונותרפיה - במיוחד מעכבי נקודות ביקורת חיסוניות וצימודי נוגדנים-תרופות - התגלה כפרדיגמה טיפולית בולטת19. פרוטוקול זה מציג מסך CRISPR במבחנה באמצעות קו תאי סרטן הומוגני יחסית, שלא יכול היה לשקף לחלוטין את ההטרוגניות הגנטית והאפיגנטית של גידולים in vivo. לאחר קביעת מינון הקרינה המתאים להקרנה, התוצאות מספקות גנים פוטנציאליים רגישים לקרינה או עמידים לקרינה, אותם יש לאשר עוד יותר הן במבחני תאים במבחנה והן במודלים של בעלי חיים in vivo. יתר על כן, מסך CRISPR in vivo בתוספת טרנסקריפטומיקה של תא בודד עשויים להועיל בפענוח המורכבות של הטרוגניות הגידול. מסך ה-CRISPR במבחנה זיהה גנים חשובים בתיקון נזקי DNA בתוך תאים, כמו גם במנגנונים אוטוקריניים בתגובה לקרינה, אך אכן לא שחזר באופן מלא TME הטרוגני. לדוגמה, היפוקסיה מגבירה את ייצור ה-ROS, ומפעילה לולאת משוב המגרה הסתגלות מטבולית, יצירת נוגדי חמצון והפעלת אוטופגיה, ובסופו של דבר מקדמת עמידות לקרינה18. תאי חיסון עשויים להיות מופעלים במנגנון הפרקרין לאחר הקרינה, ולגרום לרגישות לקרינה14. ה-ECM משמש כמאגר לגורמי גדילה, ולציטוקינים יש השפעות חיוביות ושליליות על רגישות לקרינה21. יש לבצע את מסך ה-CRISPR in vivo כדי לשקף את ההטרוגניות של TME. חוץ מזה, מסך ה-CRISPR במבחנה יכול היה לזהות רק גנים חשובים בתיקון נזק ל-DNA בתוך תאים, כמו גם במנגנונים אוטוקריניים בתגובה לקרינה, אך לא לכד באופן מלא את ההשפעה in vivo על רגישות לקרינה, כולל כלי דם של הגידול והשפעות מערכתיות20,21. יש לבצע את מסך ה-CRISPR in vivo כדי לזהות גורמים פוטנציאליים המעורבים ברגישות לקרינה in vivo.
כאן, פרוטוקול מקיף שמטרתו להשתמש בסריקה כלל-גנומית של CRISPR תוכנן כדי לאתר גנים המעורבים ברגישות או בעמידות לרדיותרפיה. על ידי חשיפת מסלולים חדשים, מתודולוגיה זו מבקשת לסלול את הדרך להתקדמות במחקר הרדיותרפיה של גידולים. פרוטוקול זה יכול לשמש גם כהתייחסות לבחינת מנגנונים של אילוצים קליניים בכימותרפיה והתקדמות של חומרים טיפוליים אלטרנטיביים. לסיכום, טכנולוגיית הקריספר מגלמת פוטנציאל וערך משמעותיים, אך האילוצים המובנים בה מדגישים את החשיבות של הכרה מפוכחת וגישה נבונה. ככל שמתעמקים במחקר ויישומים עתידיים, הכרחי לנסות ללא לאות לחקור ולייעל את טכנולוגיית הקריספר כדי להתגבר על אילוציה ולרתום את מלוא הפוטנציאל שלה.
Disclosures
ללא.
Acknowledgements
מחקר זה נתמך על ידי פרויקט החדשנות המדעית והטכנולוגית האזורי של מחוז חוביי (2024EIA001), ופרויקט התמיכה בבניית פלטפורמת חדשנות במדע וטכנולוגיה רפואית של בית החולים ג'ונגנאן של אוניברסיטת ווהאן (PTXM2025001). איור 1 נוצר באמצעות Figdraw.
Materials
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
References
- Barrangou, R., Marraffini, L. A. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell. 54 (2), 234-244 (2014).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nat Methods. 11 (8), 783-784 (2014).
- Arroyo, J. D., et al. A Genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metab. 24 (6), 875-885 (2016).
- Sidik, S. M., et al. A genome-wide CRISPR screen in toxoplasma identifies essential apicomplexan genes. Cell. 166 (6), 1423-1435 (2016).
- Jing, Y., et al. Genome-wide CRISPR activation screening in senescent cells reveals SOX5 as a driver and therapeutic target of rejuvenation. Cell Stem Cell. 30 (11), 1452-1471 (2023).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human t cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971 (2018).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Katti, A., Diaz, B. J., Caragine, C. M., Sanjana, N. E., Dow, L. E. CRISPR in cancer biology and therapy. Nat Rev Cancer. 22 (5), 259-279 (2022).
- Sasmal, R., et al. Synthetic host-guest assembly in cells and tissues: Fast, stable, and selective bioorthogonal imaging via molecular recognition. Anal Chem. 90 (19), 11305-11314 (2018).
- Roos, W. P., Thomas, A. D., Kaina, B. DNA damage and the balance between survival and death in cancer biology. Nat Rev Cancer. 16 (1), 20-33 (2016).
- Huang, R., Zhou, P. K. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 6 (1), 254(2021).
- O'Connor, M. J. Targeting the DNA damage response in cancer. Mol Cell. 60 (4), 547-560 (2015).
- Lord, C. J., Ashworth, A. The DNA damage response and cancer therapy. Nature. 481 (7381), 287-294 (2012).
- Carusillo, A., Mussolino, C. DNA Damage: From threat to treatment. Cells. 9 (7), 1665(2020).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Res. 18 (1), 99-113 (2008).
- Chang, H., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol. 18 (8), 495-506 (2017).
- Andratschke, N., et al. European society for radiotherapy and oncology and European organization for research and treatment of cancer consensus on re-irradiation: Definition, reporting, and clinical decision making. Lancet Oncol. 23 (10), e469-e478 (2022).
- Olivares-Urbano, M. A., Grinan-Lison, C., Marchal, J. A., Nunez, M. I. CSC radioresistance: A therapeutic challenge to improve radiotherapy effectiveness in cancer. Cells. 9 (7), 1651(2020).
- Schaue, D., McBride, W. H. Opportunities and challenges of radiotherapy for treating cancer. Nat Rev Clin Oncol. 12 (9), 527-540 (2015).
- Allen, C., Her, S., Jaffray, D. A. Radiotherapy for cancer: Present and future. Adv Drug Deliv Rev. 109, 1-2 (2017).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-Mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Tzelepis, K., et al. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia. Cell Rep. 17 (4), 1193-1205 (2016).
- Rottner, A. K., et al. A genome-wide CRISPR screen identifies CALCOCO2 as a regulator of beta cell function influencing type 2 diabetes risk. Nat Genet. 55 (1), 54-65 (2023).
- Chen, M., et al. CRISPR-Cas9 for cancer therapy: Opportunities and challenges. Cancer Lett. 447, 48-55 (2019).
- Gupta, D., et al. CRISPR-Cas9 system: A new-fangled dawn in gene editing. Life Sci. 232, 116636(2019).
- Li, B., et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat Commun. 11 (1), 164(2020).
- Hung, K. L., et al. Targeted profiling of human extrachromosomal DNA by CRISPR-CATCH. Nat Genet. 54 (11), 1746-1754 (2022).
- Bhattacharya, D., Van Meir, E. G. A simple genotyping method to detect small CRISPR-Cas9 induced indels by agarose gel electrophoresis. Sci Rep. 9 (1), 4437(2019).
- Razavi, Z., Soltani, M., Souri, M., Van Wijnen, A. J. CRISPR innovations in tissue engineering and gene editing. Life Sci. 358, 123120(2024).
- Wang, W., et al. A genome-wide CRISPR-based screen identifies KAT7 as a driver of cellular senescence. Sci Transl Med. 13 (575), eabd2655(2021).
- Xu, X., et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell. 81 (20), 4333-4345 (2021).
- Huang, M., et al. FACS-based genome-wide CRISPR screens define key regulators of DNA damage signaling pathways. Mol Cell. 83 (15), 2810-2828 (2023).
- Feng, J., et al. Genome-wide CRISPR screen identifies synthetic lethality between DOCK1 inhibition and metformin in liver cancer. Protein Cell. 13 (11), 825-841 (2022).
- Saurat, N., et al. Genome-wide CRISPR screen identifies neddylation as a regulator of neuronal aging and AD neurodegeneration. Cell Stem Cell. 31 (8), 1162-1174 (2024).
- Du, Y., Liu, Y., Hu, J., Peng, X., Liu, Z. CRISPR/Cas9 systems: Delivery technologies and biomedical applications. Asian J Pharm Sci. 18 (6), 100854(2023).
- Shi, J., et al. A genome-wide CRISPR screen identifies WDFY3 as a regulator of macrophage efferocytosis. Nat Commun. 13 (1), 7929(2022).
- Liu, N., Olson, E. N. CRISPR modeling and correction of cardiovascular disease. Circ Res. 130, (2022).
- Feng, X., et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 8 (19), 6638(2022).
- Ghaemi, A., et al. CRISPR-Cas9 genome editing delivery systems for targeted cancer therapy. Life Sci. 267, 118969(2021).
- Derry, W. B. CRISPR: Development of a technology and its applications. FEBS J. 288 (2), 358-359 (2021).
- Sharma, G., Sharma, A. R., Bhattacharya, M., Lee, S. S., Chakraborty, C. CRISPR-Cas9: A preclinical and clinical perspective for the treatment of human diseases. Mol Ther. 29 (2), 571-586 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved