Method Article
Cribado CRISPR de todo el genoma para revelar genes radiosensibles y radiorresistentes
* Estos autores han contribuido por igual
En este artículo
Resumen
Se ofrece un enfoque meticuloso y estructurado para seleccionar genes resistentes y sensibles a la radiación mediante la aplicación de un método de cribado CRISPR/Cas9 de todo el genoma. Este protocolo también tiene el potencial de servir como un marco versátil para otros esfuerzos de investigación que investigan los mecanismos de resistencia a los medicamentos químicos administrados clínicamente.
Resumen
El sistema CRISPR-Cas9 ha sido aprovechado y reutilizado en una poderosa herramienta de edición del genoma. Al aprovechar esta tecnología, los investigadores pueden cortar, pegar e incluso reescribir con precisión secuencias de ADN dentro de células vivas. Sin embargo, la aplicación de la tecnología de pantalla CRISPR va mucho más allá de la mera experimentación. Sirve como una herramienta fundamental en la lucha contra las enfermedades genéticas, diseccionando sistemáticamente paisajes genéticos complejos, permitiendo a los investigadores desentrañar los mecanismos moleculares que subyacen a los fenómenos biológicos y permitiendo a los científicos identificar y abordar las causas fundamentales de enfermedades como el cáncer, la fibrosis quística y la anemia de células falciformes. Entre todos, el cáncer representa un desafío formidable para la medicina, ya que estimula los esfuerzos de erradicación. La radioterapia, como tratamiento tradicional, da resultados pero tiene limitaciones. Erradica las células cancerosas pero también daña los tejidos sanos, causando efectos adversos que reducen la calidad de vida. Además, no todas las células cancerosas responden a la radioterapia, y algunas pueden desarrollar resistencia, empeorando la afección. Para abordar esto, se introduce una tecnología integral de detección CRISPR de genoma completo, ya que permite la identificación eficiente de genes radiosensibles y radiorresistentes, avanzando así en el campo de la investigación y el tratamiento del cáncer. Se llevó a cabo un cribado CRISPR de todo el genoma en células de adenocarcinoma de pulmón expuestas a irradiación siguiendo el protocolo descrito, a través del cual se identificaron los genes asociados a la radiorresistencia y a la radiosensibilidad.
Introducción
La investigación de los fenómenos biológicos está inherentemente entrelazada con el estudio de los comportamientos celulares y, a su vez, el examen de los comportamientos celulares está fundamentalmente relacionado con la exploración de su genoma. A medida que la tecnología moderna continúa evolucionando, los investigadores médicos están redirigiendo progresivamente su atención hacia la alteración de los comportamientos celulares a través de la edición de genes para mejorar los resultados del tratamiento de diversas enfermedades. En este sentido, la tecnología de repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) se ha convertido en una herramienta revolucionaria para la edición del genoma debido a su aplicación relativamente sencilla1. El sistema CRISPR-Cas9 consta de nucleasa Cas9 y ARN de guía única (sgRNA), que reconoce y se une específicamente a la secuencia de ADN objetivo, guiando a la nucleasa Cas9 para que corte en ese lugar, lo que resulta en una rotura de doble cadena (DSB) en el ADN del genoma 2,3,4. Además, la introducción de otras sustancias puede dar lugar a inserciones, deleciones o mutaciones específicas en el genoma, lo que permite la edición genética dirigida.
En la investigación de la genómica funcional, la detección de ARN de interferencia (ARNi) fue una vez un método ampliamente utilizado para realizar experimentos de pérdida de función a gran escala para investigar las funciones de los genes en el cáncer. La tecnología de ARNi estudia la función de los genes silenciando específicamente los genes objetivo, lo que ayuda a los investigadores a identificar factores oncogénicos críticos. Sin embargo, está limitado por los efectos fuera del objetivo y la eficiencia incompleta de la eliminación de genes. Los efectos fuera del objetivo pueden conducir al silenciamiento de otros genes no objetivo, comprometiendo así la precisión y fiabilidad de los resultados experimentales 1,2. Además, el ARNi exhibe una baja eficiencia de eliminación para ciertos genes, lo que potencialmente no logra suprimir completamente la expresión génica objetivo. A diferencia de los cribados de ARNi tradicionales, el cribado CRISPR demuestra una mayor especificidad y eficiencia3. Esta tecnología no solo permite la edición precisa de genes específicos, sino que también permite el cribado a gran escala de todo el genoma, lo que proporciona un sólido apoyo a la investigación de la función génica. La tecnología de cribado CRISPR, una potente herramienta de edición genética basada en el sistema CRISPR-Cas9, se utiliza para cribar y revelar de forma eficiente funciones desconocidas de genes específicos en las células5,6,7,8. Los investigadores diseñan sgRNAs en lotes para genes específicos o regiones génicas, y preparan las correspondientes bibliotecas de sgRNAs con precisión y rigor, asegurando su integridad y funcionalidad9. Estas bibliotecas de sgRNA se encapsulan en partículas lentivirales, que se utilizan para infectar eficazmente las células huésped. Después de una infección exitosa, las células infectadas se cultivan bajo condiciones de detección definidas personalmente. Tras el cribado, se extrae el ADN genómico de las células cribadas, manteniendo altos estándares de pureza y cantidad. Posteriormente, las regiones objetivo de interés para el sgRNA se someten a la amplificación por PCR, un proceso que replica con precisión los segmentos deseados de los ácidos nucleicos 3,9. Por último, se realiza una secuenciación de alto rendimiento en los fragmentos de ADN amplificados, lo que permite un análisis exhaustivo y eficiente de las regiones objetivo, proporcionando así información valiosa sobre la función y el comportamiento de los genes estudiados4.
El cáncer representa una amenaza formidable para la salud humana como enfermedad compleja. En todo el mundo, los investigadores y los médicos están realizando esfuerzos concertados para desentrañar los mecanismos moleculares de la carcinogénesis y desarrollar nuevas estrategias terapéuticas. Se han establecido colaboraciones internacionales para acelerar la traducción de los hallazgos de la investigación básica en aplicaciones clínicas, con el objetivo final de mejorar los resultados de los pacientes. Sasmal et al. propusieron una estrategia de ensamblaje bioortogonal basada en un sistema sintético de huésped-huésped para dirigirse con precisión a las células cancerosas metastásicas, que ha ayudado significativamente a docenas de científicos en el avance de las tecnologías médicas. Su destacado trabajo de investigación tiene una alta innovación y conocimientos únicos, haciendo contribuciones significativas a la comunidad científica10. El cáncer se caracteriza por el tumultuoso estado de inestabilidad genómica, derivado de la regulación errática de las respuestas al daño del ADN11-14. El daño en el ADN incluye defectos de un solo nucleótido, roturas de una sola cadena y DSBs. La recombinación homóloga (HR) y la unión de extremos no homólogos (NHEJ) participan en la reparación de DSBs en diferentes etapas 15,16,17. Sobre esta base, la radioterapia ha surgido como una opción de tratamiento viable, que utiliza rayos de alta energía (como rayos X y rayos γ) para irradiar el tejido tumoral, causando daño en el ADN de las células tumorales, interrumpiendo así su crecimiento y proliferación18. Sin embargo, la radioterapia no siempre produce los efectos deseados en una proporción significativa de pacientes con cáncer, lo que puede resultar de daños en los tejidos paracancerosos y limitaciones impuestas por las características inherentes al tumor, como la baja sensibilidad a la radioterapia 19,20,21.
Teóricamente, se puede utilizar cualquier tipo de célula para una pantalla CRISPR. Sin embargo, mantener una representación suficiente en las poblaciones mutadas requiere un gran número de células iniciales. Los tipos de células con baja abundancia no son particularmente adecuados para el cribado de todo el genoma. En cuanto a la elección de la biblioteca, la mayoría de las bibliotecas contienen de 3 a 6 ARNg por gen diana, y es fundamental mantener la distribución de cada ARNg dentro de la población18. La pérdida de representación debido al enriquecimiento o agotamiento de ARNg específicos puede dar lugar a una distribución desigual de los resultados. Para abordar este problema, optar por bibliotecas CRISPR disponibles comercialmente que hayan sido probadas en el mercado puede ser una opción preferible20. In vitro Es posible que el cribado CRISPR que utiliza líneas celulares cancerosas homogéneas no capture completamente la heterogeneidad genética y epigenética de los tumores in vivo . Si bien el cribado in vitro reveló genes clave implicados en la reparación del daño del ADN y la señalización autocrina inducida por la radiación, no replicó completamente el microambiente tumoral, incluida la radiorresistencia inducida por la hipoxia (a través de ROS, adaptación metabólica y autofagia), los efectos paracrinos mediados por el sistema inmunitario y la modulación de citocinas dependiente de la MEC. Antes de emplear el cribado CRISPR para explorar los genes asociados con la sensibilidad o la resistencia a la radiación, estos factores deben considerarse cuidadosamente. A la luz del panorama actual del tratamiento, es urgente identificar y estudiar en profundidad los factores asociados con la radiorresistencia y la radiosensibilidad para mejorar eficazmente la eficacia de la radioterapia22. Dada la ventaja clave de la pantalla CRISPR en el estudio de las funciones de genes desconocidos, se proporciona una tecnología de detección CRISPR de genoma completo sistemáticamente detallada para identificar de manera eficiente los genes radiosensibles y radiorresistentes.
Protocolo
Los reactivos y el equipo utilizado en este estudio se enumeran en la Tabla de Materiales.
1. Selección de una dosis adecuada de radiación
- Preparación y siembra de células adherentes
- Ajuste la densidad celular a 5 x 105 células por ml con el medio de cultivo celular completo RPMI 1640 que contiene 10% de FBS para el paso y crecimiento celular. Distribuya las células en placas de cultivo de 3,5 cm para la radiación a diferentes dosis. Añadir 2 mL (1 x 106 células) a cada plato e incubar durante la noche a 37 °C con 5% de CO2.
- Aplicación de diferentes dosis de radiación
- Numere los platos de cultivo de 3,5 cm del 1 al 5. Utilice el Grupo # 1 como grupo de control, con los 4 grupos restantes designados como grupos de tratamiento.
- Selle los bordes de las placas de 6 pocillos con una membrana de sellado para los grupos de tratamiento y administre dosis de radiación de 2, 4, 6 y 8 Gy, respectivamente. Después del tratamiento, regrese los platos a la incubadora.
- Siembra de líneas celulares después de la radiación
- Prepare platos de 6 y 96 pocillos. Lave las células en las placas de cultivo de 3,5 cm una vez con PBS, digiera las células en crecimiento logarítmico con tripsina al 0,25% y ajuste la densidad celular a 1 x 105 células por ml con el medio completo RPMI 1640 que contiene 10% de FBS.
- Siembre 10 μL/pocillo (1.000 células/100 μL) en las placas de 6 pocillos a 3 repeticiones por cada dosis de radiación. Agregue 2 mL de medio completo a cada pocillo y cuente después de 14 días (cuando cada grupo de clones tenga aproximadamente 50 celdas).
- Siembre 30 μL/pocillo (3.000 células/100 μL) en las placas de 96 pocillos a 5 repeticiones por cada dosis de radiación. Agregue 70 μL de medio completo a cada pocillo y mida la viabilidad celular después de 72 h.
- Medición de la viabilidad celular
NOTA: Tasa de inhibición clonogénica = 1 - (número de clones en el grupo de tratamiento/número de clones en el grupo de control) x 100%. El ensayo CCK-8 utiliza una sal de tetrazolio soluble en agua (WST-8) que puede ser reducida por las deshidrogenasas celulares para generar un producto de formazano amarillo altamente soluble en agua11. La cantidad de formazano producido es directamente proporcional al número de células viables, y su densidad óptica (medida a una longitud de onda de 450 nm) refleja con precisión la actividad metabólica celular y el estado proliferativo11,13. Sobre la base de este principio bien establecido, el ensayo CCK-8 ha sido ampliamente adoptado para diversas aplicaciones, incluidos los ensayos de proliferación celular y las pruebas de sensibilidad a los medicamentos tumorales. En este protocolo, el ensayo CCK-8 se emplea para evaluar sistemáticamente la viabilidad celular bajo diferentes dosis de radiación.- Mezcle el reactivo CCK8 con el medio RPMI 1640 sin FBS en una proporción de 1:9. Deseche el medio en las placas de 96 pocillos y agregue 100 μL del medio que contiene CCK8 a cada pocillo.
- Incubar en la oscuridad durante 1 h y medir el valor de OD a 450 nm con un lector de microplacas.
NOTA: Tasa de supervivencia celular = [(Valor OD del grupo de tratamiento - Valor OD del pocillo blanco) / (Valor OD del grupo control - Valor OD del pocillo blanco)] x 100%
- Selección de la dosis de radiación
- Integrar la tasa de inhibición clonogénica con la tasa de supervivencia celular para seleccionar una dosis de radiación adecuada en función de las necesidades de investigación. Seleccione una dosis de radiación con una tasa de inhibición del 50% para el cribado de genes radiorresistentes y radiosensibles.
2. Selección del MOI y la concentración de puromicina adecuados
- Siembra de células adherentes
- Ajuste la densidad de células a 3 x 105 células por ml e inocule 1 ml (3 x 105 células) por pocillo en una placa de 12 pocillos. Incubar la placa durante la noche a 37 °C con 5% de CO2.
- Infección lentiviral
- Utilice una pipeta para aspirar el medio de la placa de 12 pocillos y sustitúyalo por 1 ml de medio completo RPMI 1640 que contenga un 10 % de FBS. Prepare una biblioteca lentiviral CRISPR de genoma completo de calidad verificada (se recomiendan bibliotecas disponibles comercialmente) y configure un gradiente de concentración logarítmico (por ejemplo, 0-10-50-100-200-400-800)4,5,6,7,8.
- Añadir la cantidad correspondiente de lentivirus a 2 μL/plato de policeno y equilibrar a temperatura ambiente durante 5 min. Gotee lentamente la mezcla de lentivirus y polibreno en cada pocillo, mezcle bien e incube durante la noche a 37 °C con 5% de CO2.
- Determinación de la concentración mínima de puromicina para la destrucción celular
- Ajuste la densidad de células parentales a 3 x 105 células/ml e inocule 1 ml (3 x 105 células) por pocillo en una placa de 12 pocillos. Incubar la placa durante la noche a 37 °C con 5% de CO2.
- Agregue puromicina a cada pocillo de la placa de 12 pocillos en un gradiente de concentración de 0-0.1-0.2-0.5-1-2-4-8 μM. Cuente las células después de 72 h. La concentración mínima que mata a todas las células en el pocillo es la concentración mínima de puromicina para la destrucción celular, que se utilizará para el cribado posterior de células infectadas por el virus.
- Selección de puromicina después de la infección
- Al segundo día después de la infección, aspire el medio de cada pocillo y reemplácelo con 1 mL de medio completo RPMI 1640 que contenga 10% de FBS. Continuar el cultivo durante 48 h.
- Después de 72 h de infección, reemplace el medio existente con un medio completo que contenga la concentración mínima de puromicina para la eliminación celular. Configure 2 pocillos en la placa de 12 pocillos sin infección lentiviral como controles negativos y positivos. Trate el control negativo con puromicina y deje el control positivo sin tratar. Continuar cultivando durante 72 h.
- Después de 72 h de selección de puromicina, todas las células parentales del control negativo estarán muertas, mientras que las células parentales del control positivo muestran una muerte mínima. Calcule el MOI de cada pozo.
NOTA: MOI = (Número de células en el grupo infectado por el virus / Número de células en el grupo de control positivo) x 100%. Utilice la concentración del virus con un MOI de ~0,3 para el cribado posterior.
3. Infección de la biblioteca lentiviral CRISPR en todo el genoma
- Siembra de líneas celulares adherentes
- Ajuste la densidad celular a 1 x 107 celdas por mL en medio completo RPMI 1640 que contiene 10% de FBS para el paso y el crecimiento celular. Inocular 1 mL de células (1 x 107 células ) en cada placa de cultivo de 15 cm a 37 °C con 5% de CO2 durante 8 h. Una vez que las células se adhirieron y alcanzaron una confluencia del 70%-80%, estaban listas para la infección viral (haciendo un número de copias de aproximadamente 500).
- Infección lentiviral
- Aspire el medio de las placas de cultivo de 15 cm y reemplácelo con 15 mL de medio completo RPMI 1640 que contenga un 10% de FBS. Preparar una biblioteca lentiviral CRISPR de genoma completo de calidad comprobada.
- Añadir la cantidad correspondiente de lentivirus con el MOI = 0,3 a 30 μL/placa de polibreno y equilibrar a temperatura ambiente durante 5 min, gotear lentamente la mezcla de lentivirus y polibreno en las placas de cultivo de 15 cm, mezclar bien e incubar a 37 °C con 5% de CO2 durante la noche. Simultáneamente, prepare una placa de cultivo de 15 cm con la célula parental como contraste.
- Selección de puromicina después de la infección
- Al segundo día después de la infección viral, aspire el medio de las placas de cultivo de 15 cm y reemplácelo con 15 mL de medio completo RPMI 1640 que contenga 10% de FBS. Continuar el cultivo durante 48 h.
- A las 72 h después de la infección, sustituya el medio por un medio completo que contenga la concentración letal mínima de puromicina. Trate las células parentales no infectadas como un control negativo de la misma manera y continúe el cultivo durante 72 h. Después de 72 h de selección de puromicina, todas las células parentales del grupo de control negativo serán eliminadas, y las células supervivientes de la infección lentiviral se considerarán infectadas con éxito.
- Extracción del genoma del día 0
- Digiera las células de una placa de cultivo de 15 cm con tripsina al 0,25%, vuelva a suspender en un medio completo RPMI 1640 que contenga un 10% de FBS y cuente el número de células. Etiquete este ejemplo como Día 0.
- Centrifugar a 300 x g durante 5 min (a temperatura ambiente) y desechar el sobrenadante. Vuelva a suspender en 1 mL de PBS, centrifugue a 300 x g durante 5 min y deseche el sobrenadante. Extraiga el ADN genómico del día 0, mida la concentración y la pureza del ADN utilizando un espectrofotómetro UV de nanogotas.
- Para la detección de la integridad del sgRNA, utilice la electroforesis en gel de agarosa para analizar la biblioteca de sgRNA amplificada, asegurándose de que las bandas estén claras y no se degraden significativamente, preservando así la integridad de la biblioteca23. Para la evaluación de la cobertura de la biblioteca lentiviral, emplee la tecnología de secuenciación profunda para realizar análisis de secuenciación en los sgRNAs dentro de la biblioteca24,25.
- En el cribado CRISPR, la amplificación por PCR sirve como verificación preliminar de la integridad del fragmento de sgRNA y de la calidad de la biblioteca6. Para analizar de manera más exhaustiva la cobertura de la biblioteca y los patrones de distribución de sgRNA, y para garantizar una representación adecuada de los sgRNA para cada gen objetivo durante el cribado, realice NGS para obtener información detallada sobre la solidez del cribado9.
NOTA: Al detectar cambios en la abundancia de sgRNA antes y después de la selección, e identificar la posible integración de sgRNA fuera del objetivo o la contaminación de la biblioteca, la precisión y la confiabilidad de la pantalla CRISPR podrían mejorarse aún más. Estas muestras del día 0 sirven como controles negativos cruciales y se someten a una evaluación integral de la calidad mediante amplificación por PCR y NGS para lograr 2 objetivos clave: (1) confirmación de la depleción de genes esenciales (lo que indica una representación adecuada de la biblioteca) y (2) demostración de expresión estable en genes no esenciales (establecimiento de condiciones experimentales de referencia)9. - Asegurar que la cobertura alcance el nivel esperado para asegurar la diversidad y representatividad de la biblioteca y evitar sesgos en el proceso de selección. Para la medición de la eficiencia de la infección, utilice imágenes de fluorescencia para evaluar la eficiencia de la infección, asegurándose de que cumple con los requisitos del experimento.
4. Aplicación de radiación como condición de detección
- Agrupación
- Ajuste la densidad de las células infectadas con CRISPR a 1 x 107 células por ml e inocule 1 ml en cada placa de Petri de 15 cm. Incubar durante la noche a 37 °C con 5% de CO2.
- Tratamiento con radiación
- Divida aleatoriamente las células en 2 grupos: un grupo de tratamiento y un grupo de control, con 6 platos por grupo. Administre una dosis adecuada de radiación a las células del grupo de tratamiento, mientras que deje las células del grupo de control sin tratar para que se propaguen normalmente. Después de la radiación, continúe incubando a 37 °C con 5% de CO2 durante 7 días.
- En la segunda semana, repita la administración de una dosis adecuada de radiación a las células del grupo de tratamiento, dejando las células del grupo de control sin tratar y permitiendo que se propaguen normalmente. Después de la radioterapia, continuar incubando a 37 °C con 5% de CO2 durante otros 7 días.
5. Extracción y secuenciación del genoma
- Extracción del día 14
- Después de 14 días de tratamiento, digiera las células de los grupos de tratamiento y control con tripsina al 0,25%, vuelva a suspenderlas utilizando un medio RPMI 1640 completo que contenga 10% de FBS y etiquételas como Día 14-RT o Día 14-NC.
- Centrifugar las células a 300 x g durante 5 min y desechar el sobrenadante. Vuelva a suspender en 1 mL de PBS, centrifugue a 300 x g durante 5 min y deseche nuevamente el sobrenadante. Extraiga el ADN genómico del día 14 y detecte la concentración y la pureza del ADN mediante espectroscopía de absorbancia UV de nanogotas.
- Amplificación por PCR
- Prepare los cebadores correspondientes de la secuencia (consulte la Tabla de materiales) y dilúyalos hasta 10 μM. Configure un sistema de reacción de 20 μL agregando los reactivos en un tubo de microcentrífuga estéril, centrifugue durante 5 s a 300 x g y mezcle bien. Configure los parámetros del instrumento de PCR de acuerdo con la condición de amplificación para obtener productos de PCR.
- Detección de electroforesis en gel de agarosa
- Prepare una placa de fundición de gel, selle los bordes del molde con agarosa, inserte el peine y prepare un gel de agarosa de concentración adecuada de acuerdo con la longitud de la muestra de ADN.
- Pese con precisión una cierta cantidad de polvo de agarosa, agregue una cantidad adecuada de tampón de electroforesis, mezcle bien y caliente en un horno de microondas para que se derrita. Después de enfriar un poco, agregue una cantidad adecuada de tinte de ácido nucleico, mezcle suavemente y vierta lentamente en el molde de fundición en gel. Deja que el gel se solidifique durante 30 min.
- Retire el peine y agregue una cantidad adecuada de tampón de electroforesis al tanque de electroforesis hasta que cubra el gel. Agregue una cantidad adecuada de tampón de carga a la muestra de ADN, mezcle bien y use una pipeta para agregar lentamente la mezcla al pocillo de la muestra.
- Establezca el voltaje adecuado en función del tamaño del fragmento de ADN y la concentración del gel de agarosa. Después de la electroforesis, retire con cuidado el gel y colóquelo en un generador de imágenes en gel para observar los resultados y verificar si la amplificación por PCR fue exitosa.
- Secuenciación de Illumina
- Recoja el ADN genómico de los tres grupos (grupo de día 0, grupo de control y grupo de tratamiento) y envíelo a una empresa para la construcción de la biblioteca y la secuenciación de Illumina. Realizar análisis bioinformáticos y visualización de los resultados de la secuenciación para obtener genes sensibles y resistentes a la radioterapia26.
- Establecer múltiples experimentos repetidos para el análisis de datos y realizar análisis estadísticos sobre los resultados experimentales para garantizar la consistencia y reproducibilidad de los datos27,28.
- Evaluación de la calidad de los datos de secuenciación
- Someter los datos brutos a rigurosos mecanismos de filtrado y control de calidad para garantizar la precisión de la secuenciación de la información5. Calcule la puntuación Phred (Qphred) para cada nucleótido en función de la tasa de errores de secuenciación, utilizando un algoritmo de conversión especificado y un modelo que evalúe la probabilidad de que se produzca un error durante la llamada base28.
- Mantener la tasa de error de secuenciación para cada posición base por debajo del 1% (equivalente a un umbral Q30) y asegurarse de que al menos el 80% de los datos de secuenciación cumplan con este estándar Q30 para respaldar los procedimientos analíticos posteriores.
- Procesamiento de datos y control de calidad a nivel de muestra
NOTA: Para más detalles, consulte los informes publicados anteriormente 29,30.- Alinee las lecturas filtradas de los datos de secuenciación con las secuencias de la biblioteca de sgRNA. Estadísticas de informes, incluido el recuento de sgRNAs en la biblioteca con una alineación perfecta, la abundancia promedio de sgRNAs, el número de sgRNAs no detectados y la proporción de lecturas de muestras individuales mapeadas con éxito a la biblioteca de sgRNAs. Utilice una relación de asignación más alta para indicar una mayor cobertura.
- Cuente el enriquecimiento de las lecturas de cada gen (dirigido por diferentes sgRNAs) en cada muestra. Normalice las lecturas de soporte para cada sgRNA en todas las muestras utilizando el método de normalización de la "mediana" de MAGeCK. Realice un control de calidad a nivel de muestra mediante la evaluación de la distribución de los recuentos de lecturas de sgRNA, la generación de diagramas de caja, la realización de análisis de componentes principales (PCA) y la construcción de mapas de calor de correlación.
- Supongamos que los recuentos de lectura de sgRNA siguen una distribución de Poisson. Represente recuentos normalizados de sgRNA de diferentes grupos en diagramas de caja para visualizar la distribución general de datos dentro de las muestras y comparar las distribuciones entre grupos.
- Utilice el ACP para simplificar el análisis de datos al reflejar las diferencias entre los componentes principales y la tasa de variación dentro de cada componente, lo que facilita la observación de las variaciones intra e intergrupales. Utilice un mapa de calor de correlación para ilustrar las relaciones entre las muestras.
- Análisis básico
- Realice análisis diferenciales de sgRNA entre grupos e identifique genes esenciales después del control de calidad y el procesamiento preliminar de datos. Realizar análisis de enriquecimiento funcional de los genes relevantes30. Clasifique los genes esenciales en función de la puntuación de agregación de rangos (RRA) robusta calculada por MAGeCK u otros métodos estadísticos como MLE, con una puntuación de RRA más baja que indica una mayor importancia.
- Utilice el lenguaje R u otros lenguajes de programación para realizar análisis bioinformáticos en los resultados clasificados y visualice los hallazgos utilizando diagramas de análisis GO y KEGG30. Vincular genes o proteínas a los términos GO correspondientes (función molecular, proceso biológico y componente celular) a través del mapeo de ID o la anotación de secuencias.
- Utilice KEGG como base de datos para comprender las funciones de alto nivel y los sistemas biológicos, conectando el catálogo de genes identificados con las funciones del sistema a nivel celular, de especies y de ecosistema. Seleccione las 10 o 20 rutas principales de los análisis GO y KEGG para una visualización intuitiva de las direcciones de las rutas.
Resultados
El cáncer de pulmón, con la tasa de mortalidad más alta, representa una enfermedad médica altamente agresiva y prevalente. En la Figura 1 se muestra el flujo de trabajo esquemático utilizando la línea celular de cáncer de pulmón A549 para llevar a cabo un cribado CRISPR de todo el genoma con radiación como condición de cribado. En primer lugar, explorar la sensibilidad de las células A549 a diferentes dosis de radiación a través de la formación de clones y experimentos con CCK8 (Figura 2). En el ensayo clonogénico, el recuento de colonias fue de 140 ± 5,35 a 2 Gy frente a 303 ± 31,63 a 0 Gy, mientras que en el ensayo CCK-8, el valor de densidad óptica (OD) fue de 0,65 ± 0,05 a 2 Gy frente a 1,35 ± 0,08 a 0 Gy. Para investigar genes candidatos para radiosensibilidad y radiorresistencia, seleccione 2 Gy (IC50) como dosis de radiación subsiguiente. Las células A549 se trataron con diferentes dosis de puromicina y se contaron después de 72 h. La concentración mínima de puromicina para las células A549 se detectó en 1 μM (Figura 3). Las células A549 se infectaron con lentivirus a diferentes MOI de gradiente. Después de 72 h de la infección, la eficacia de la infección se observó mediante microscopio de fluorescencia y se contó antes y después de 72 h de tratamiento con puromicina. Se obtuvo el MOI con una eficiencia de infección de ~0,3 (Tabla 1). El genoma humano contiene 19.050 genes, con 6 sgRNAs correspondientes a cada gen y un número de copias de 500 (19.050 x 6 x 500 = 5,7 x 107 células). Utilizar lentivirus con MOI = 0,3 y, en consecuencia, infectar 5,7 x 107 células A549. Después de 72 h, las células se trataron con 1 μM de puromicina durante 72 h adicionales. Se recolectó el genoma del día 0 y se realizó la amplificación por PCR. Las secuencias de sgRNA dentro de la biblioteca del genoma completo de CRISPR exhiben una longitud de 231 pares de bases, lo que se corrobora con los resultados de la electroforesis en gel de agarosa (Figura 4).
Posteriormente, los productos de PCR se enviaron a Novogene para el control de calidad de los datos y el control de calidad a nivel de muestra. La proporción de mapeo arrojó una tasa de cobertura completa de aproximadamente el 60%, una métrica considerada adecuada para los procedimientos de detección del genoma completo, dada la gran complejidad y el inevitable desgaste durante el procesamiento de un genoma completo (Figura 5). Los recuentos de lectura de sgRNA se adhirieron a una distribución de Poisson, ajustándose a las expectativas teóricas. El análisis posterior a través de gráficos de PCA y mapas de calor de correlación retrató vívidamente la disparidad discernible entre los distintos grupos, con la variación entre grupos superando notablemente las discrepancias intragrupales. Además, la tasa de variación dentro de las muestras agrupadas se situó dentro de límites aceptables, lo que corrobora el éxito de las medidas de control de calidad a nivel de muestra. Posteriormente, se aprovecharán las clasificaciones RRA ideadas por MAGeCK para embarcarse en una evaluación bioinformática fundamental de los resultados de la clasificación utilizando el lenguaje R. En particular, las 15 vías principales en términos de GO presentaron de manera destacada mecanismos relacionados con el daño en el ADN, lo que se alinea a la perfección con los criterios de detección subyacentes del experimento.

Figura 1: Esquema del cribado CRISPR/Cas9 de radioterapia de todo el genoma en células de cáncer de pulmón A549. Una representación esquemática del flujo de trabajo de cribado en células A549 utilizando CRISPR/Cas9 de todo el genoma y radioterapia. Haga clic aquí para ver una versión más grande de esta figura.
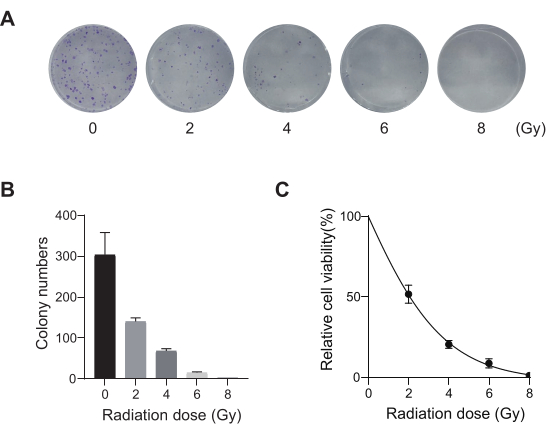
Figura 2: Optimización de la dosis de radiación para las células A549. (A) Las células A549 se expusieron a dosis crecientes de radiación (0 Gy, 2 Gy, 4 Gy, 6 Gy y 8 Gy) y se les permitió crecer durante 14 días. Se muestran las imágenes del ensayo de formación de colonias. Los resultados son representativos de dos experimentos biológicos independientes. (B) Resultados de cuantificación de imágenes de ensayos de formación clonadas. Las barras de error representan la desviación estándar (n = 3). El ANOVA de un factor arrojó P < 0.01. Los resultados son representativos de dos experimentos biológicos independientes. (C) Curvas de supervivencia dosis-respuesta de las células A549 después de la exposición a diferentes dosis de radiación. Las barras de error representan la desviación estándar (n = 3). El ANOVA de un factor arrojó P < 0.01. Los resultados son representativos de dos experimentos biológicos independientes. Haga clic aquí para ver una versión más grande de esta figura.
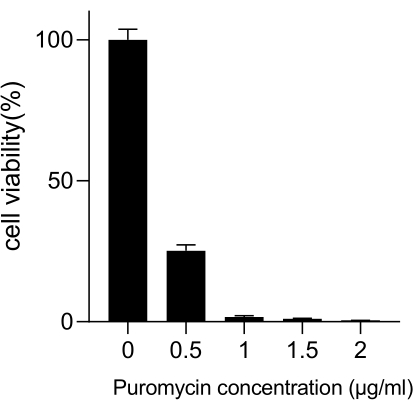
Figura 3: Determinación de la concentración mínima de puromicina necesaria para la selección de células A549. Las células A549 se trataron con concentraciones crecientes de puromicina y se incubaron durante 72 h. Las barras de error representan la desviación estándar (n = 3). El ANOVA de un factor arrojó P < 0.01. Los resultados son representativos de dos experimentos biológicos independientes. Haga clic aquí para ver una versión más grande de esta figura.
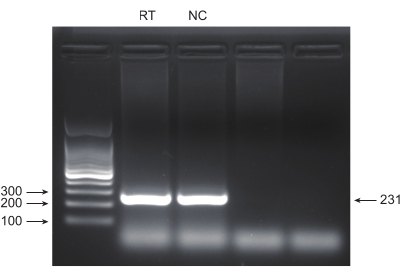
Figura 4: Validación del producto de PCR por electroforesis en gel de agarosa. Se utilizó electroforesis en gel de agarosa para examinar la presencia y la calidad de las bandas de productos de PCR. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Control de calidad y análisis básico de productos amplificados por PCR. (A) La proporción de mapeo indicó una cobertura de aproximadamente el 60% del genoma de referencia. (B) La distribución del conteo de lecturas de sgRNAs siguió una distribución de Poisson. (C) La mayoría de los 10 términos principales de la Ontología Genética (GO) se asociaron con la respuesta al daño del ADN. (D) Gráfico volcánico que muestra patrones de enriquecimiento de genes, con genes enriquecidos negativamente en rojo, genes enriquecidos positivamente en azul y genes no significativos en gris. Haga clic aquí para ver una versión más grande de esta figura.
| Partícula de virus Número (VP) | Número de conteo (antes del tratamiento con puromicina) | Número de conteo (después del tratamiento con puromicina) | Infección eficacia |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
Tabla 1: Recuentos de células A549 antes y después de la selección de puromicina bajo diferentes partículas de virus. Recuentos de células A549 registrados antes y después del tratamiento con puromicina bajo diferentes partículas virales.
Discusión
Como tecnología de edición genética de vanguardia, la pantalla CRISPR ha provocado cambios profundos en el campo de la investigación científica5. Derivada del sistema CRISPR-Cas9, esta tecnología se ha convertido en una herramienta esencial para el estudio de las funciones de los genes debido a su alta eficiencia y precisión9. El principio de ingeniería de CRISPR/Cas9 implica el diseño y la introducción de sgRNAs específicos con aproximadamente 20 nucleótidos para guiar a la nucleasa Cas9 para localizar y cortar con precisión las secuencias de ADN objetivo, lo que permite la edición de genes como la eliminación, la activación o la represión. Este principio subyace en el desarrollo de pantallas CRISPR personalizables y de alto rendimiento. Mediante la construcción de bibliotecas que contienen un gran número de sgRNAs, los investigadores pueden editar y examinar simultáneamente múltiples genes, identificando rápidamente aquellos asociados con fenotipos específicos o estados de enfermedad. Además, la pantalla CRISPR permite la experimentación en varios tipos de células, tejidos e incluso organismos completos, proporcionando una comprensión más completa de la función de los genes y los mecanismos de acción.
Con el desarrollo acelerado de la tecnología, la pantalla CRISPR ha demostrado un tremendo potencial para su aplicación en varios campos. Por ejemplo, el cribado CRISPR puede ayudar a los científicos a descubrir nuevas dianas farmacológicas, acelerando así el proceso de descubrimiento y desarrollo de fármacos. En la investigación de enfermedades, esta tecnología revela los mecanismos de la patogénesis de la enfermedad, ofreciendo nuevos conocimientos y enfoques para el diagnóstico y el tratamiento. El cribado CRISPR difiere fundamentalmente de los métodos tradicionales de cribado (como el ARNi y los inhibidores de moléculas pequeñas) en varios aspectos clave: Mecánicamente, CRISPR logra modificaciones genéticas permanentes a través de la edición de genes, mientras que el ARNi solo suprime temporalmente la expresión génica y los inhibidores de moléculas pequeñas bloquean de forma reversible la función de las proteínas28,29. En términos de especificidad, CRISPR demuestra una precisión superior a través de una orientación precisa del ADN, mientras que el ARNi es propenso a efectos fuera del objetivo 3,5. En cuanto a la duración del efecto, CRISPR crea alteraciones genéticas permanentes, en comparación con las intervenciones transitorias de los métodos convencionales. Además, CRISPR permite capacidades de cribado en todo el genoma y puede investigar simultáneamente múltiples genes/vías, al tiempo que mantiene una alta eficiencia endiversos tipos de células. Los enfoques tradicionales suelen limitarse al análisis de vías específicas y, a menudo, muestran una eficacia reducida en ciertas líneas celulares3. Estas características distintivas hacen que el cribado CRISPR sea especialmente valioso para la investigación de la genómica funcional y el descubrimiento de objetivos, especialmente en estudios que requieren observación a largo plazo o una regulación genética precisa, donde demuestra ventajas únicas sobre las metodologías de cribado convencionales.
Sin embargo, la especificidad de este proceso no es absoluta, y es posible que la proteína Cas9 reconozca y escinda erróneamente regiones de ADN que son similares pero no completamente idénticas a la secuencia diana prevista. Esta ocurrencia se conoce comúnmente como efectos fuera del objetivo. Esta escisión involuntaria del ADN puede provocar inestabilidad genómica y consecuencias genéticas impredecibles, especialmente cuando se aplica en terapia humana. La tecnología CRISPR también tiene dificultades cuando se trata de tareas complejas de edición genómica. Cuando se enfrenta a demandas de edición que abarcan numerosos genes, grupos de genes o configuraciones genéticas intrincadas, el sistema CRISPR puede experimentar limitaciones para anticipar y dirigir con precisión los resultados del proceso de edición1.
El principal obstáculo técnico para llevar a cabo el cribado de todo el genoma mediante la tecnología CRISPR se deriva de la gran complejidad del genoma, que abarca decenas de miles de genes, lo que aumenta significativamente el riesgo de efectos fuera del objetivo9. Para garantizar la integridad y fiabilidad de los resultados de la secuenciación posterior, es imperativo que cada célula se transfecte con un sgRNA23 singular. Esto requiere un control meticuloso del MOI por parte del experimentador, asegurándose de que permanezca por debajo de 0,5 y, al mismo tiempo, manteniendo un umbral de número de copias adecuado, que debe ser de al menos 500 o más24. Dado el hecho de que cada una de las decenas de miles de genes en el genoma completo requiere la utilización de 4-6 sgRNAs, todo el proceso de cribado requiere una gran cantidad de células25. La duración prolongada del experimento, el número sustancial de células involucradas y el riesgo elevado requieren que el experimentador diseñe estrategias para aliviar estas preocupaciones26. Es necesario validar aún más los genes potenciales con RNA-seq, así como con ensayos de ganancia y pérdida de función. En los siguientes experimentos, después de obtener la sobreexpresión y las células knockout de los genes identificados mediante edición CRISPR, se realizaron genómicas de todo el genoma y transcriptómica para acceder a posibles alteraciones fuera del objetivo, particularmente en loci con complementariedad parcial de sgRNA que pueden sufrir una escisión inespecífica de Cas9. Las herramientas de predicción bioinformática, como COSMID, podrían ayudar a identificar sitios fuera del objetivo de alto riesgo. Los ensayos de rescate también podrían utilizarse para garantizar la especificidad de la implicación de los genes identificados.
En el análisis posterior, los criterios para los sgRNAs expresados diferencialmente se basaron en la puntuación de agregación de rangos robusta (RRA) calculada por MAGeCK, que involucró significación estadística, cambio de pliegue y consistencia replicada31,32. Para un conjunto de datos, cada gen se clasifica de acuerdo con criterios integrales que combinan los niveles de expresión génica y la significación estadística. RRA utiliza el coeficiente de correlación de rango de Spearman para evaluar la situación del rango completo de cada gen en todos los conjuntos de datos e integra los resultados de clasificación de múltiples conjuntos de datos de expresión génica. Un gen con un rango relativamente alto en la mayoría de los conjuntos de datos podría considerarse como un gen genuino expresado diferencialmente33. Un valor p de RRA menor de 0,05 se consideró estadísticamente significativo. Un cambio de pliegue de al menos 1,5 veces o una disminución del 33% se consideró una expresión diferencial. Además, se requirió la consistencia de 3 réplicas independientes para ser considerada como una expresión estadísticamente diferente. Después de la clasificación de RRA para los genes esenciales, una puntuación de RRA más baja significa una mayor importancia.
Como potente herramienta para la edición del genoma, la tecnología CRISPR-Cas9 demuestra un potencial y una precisión sin precedentes en la investigación del área de la radiorresistencia26,27. Sin embargo, su aplicación aún tiene limitaciones. Aparte del riesgo de efectos fuera del objetivo, las complejidades asociadas con la radiosensibilidad introducen complejidades en el proceso de detección27. Esta sensibilidad no está meramente gobernada por un gen solitario, sino que surge como consecuencia de la intrincada interacción entre múltiples genes y sus redes reguladoras26. Por lo tanto, a pesar de la identificación exitosa de genes específicos relacionados con la radiosensibilidad, es posible que solo representen una fracción del panorama más amplio, lo que plantea desafíos significativos para dilucidar de manera integral los mecanismos subyacentes 34,35,36. Estas son las soluciones a los problemas comunes en el cribado CRISPR, como la baja eficiencia de la infección, la degradación del SGRNA y el fallo de secuenciación: dado que el cribado CRISPR requiere un valor de MOI <0,5, el ajuste del estado celular es fundamental para garantizar que las células se encuentran en una fase de crecimiento logarítmico o evaluar la eficacia de la infección mediante ensayos de fluorescencia; las posibles causas de la degradación del sgRNA incluyen la contaminación por nucleasas o defectos de diseño del vector, por lo que deben evitarse los ciclos repetidos de congelación y descongelación de las bibliotecas de sgRNA (almacenar alícuotas a -80 °C), y se debe utilizar la secuenciación NGS para verificar la integridad de la biblioteca de sgRNA; El fracaso de la secuenciación puede deberse a una profundidad de secuenciación insuficiente o a problemas de diseño del cebador de sgRNA, se recomienda la secuenciación de extremo emparejado para mejorar la precisión de la identificación de la secuencia de sgRNA, con una profundidad de secuenciación sugerida de >500× de cobertura 37,38,39,40,41,42.
La radioterapia sigue siendo una piedra angular en el tratamiento del cáncer, pero presenta una notable variabilidad en la eficacia terapéutica entre los diferentes tipos de tumores e incluso entre los pacientes con el mismo cáncer9. Esta heterogeneidad interindividual requiere frecuentemente de terapias combinadas para lograr una erradicación óptima del tumor11,12. En la práctica clínica, la integración de la radioterapia con la inmunoterapia, en particular con los inhibidores de puntos de control inmunitario y los conjugados anticuerpo-fármaco, se ha convertido en un paradigma terapéutico destacado19. Este protocolo presenta un cribado CRISPR in vitro utilizando una línea celular cancerosa relativamente homogénea, que no pudo reflejar totalmente la heterogeneidad genética y epigenética de los tumores in vivo. Después de determinar la dosis apropiada de radiación para el cribado, los resultados proporcionan posibles genes radiosensibles o radiorresistentes, que deben confirmarse aún más tanto en ensayos celulares in vitro como en modelos animales in vivo. Además, el cribado CRISPR in vivo más la transcriptómica unicelular podrían ser útiles para descifrar la complejidad de la heterogeneidad tumoral. El cribado CRISPR in vitro identificó genes importantes en la reparación del daño del ADN dentro de las células, así como en los mecanismos autocrinos en respuesta a la radiación, pero de hecho no replicó completamente el TME heterogéneo. Por ejemplo, la hipoxia aumenta la producción de ROS, desencadenando un ciclo de retroalimentación que estimula la adaptación metabólica, la generación de antioxidantes y la activación de la autofagia, promoviendo en última instancia la radiorresistencia18. Las células inmunitarias podrían activarse en el mecanismo paracrino después de la radiación, induciendo radiosensibilidad14. El MEC sirve como reservorio de factores de crecimiento, y las citocinas tienen efectos positivos y negativos sobre la radiosensibilidad21. El cribado CRISPR in vivo debe realizarse para reflejar la heterogeneidad de la TME. Además, el cribado CRISPR in vitro solo pudo identificar genes importantes en la reparación del daño del ADN dentro de las células, así como en los mecanismos autocrinos en respuesta a la radiación, pero no capturó completamente la influencia in vivo sobre la radiosensibilidad, incluida la vasculatura tumoral y los efectos sistémicos20,21. El cribado CRISPR in vivo debe realizarse para identificar los posibles factores implicados en la radiosensibilidad in vivo.
En este caso, se diseña un protocolo exhaustivo destinado a utilizar el cribado de todo el genoma CRISPR para identificar los genes que están implicados en la sensibilidad o la resistencia a la radioterapia. Al desentrañar nuevas vías, esta metodología busca allanar el camino para los avances en la investigación de la radioterapia tumoral. Este protocolo también podría servir como referencia para explorar los mecanismos de las restricciones clínicas en la quimioterapia y la progresión de agentes terapéuticos alternativos. En resumen, la tecnología CRISPR incorpora un potencial y un valor significativos, pero sus limitaciones inherentes subrayan la importancia de un reconocimiento sobrio y un enfoque juicioso. A medida que se profundiza en la investigación y las aplicaciones futuras, es imperativo esforzarse incansablemente por explorar y optimizar la tecnología CRISPR para superar sus limitaciones y aprovechar todo su potencial.
Divulgaciones
Ninguno.
Agradecimientos
Este estudio contó con el apoyo del Proyecto Regional de Innovación Científica y Tecnológica de la provincia de Hubei (2024EIA001) y el Proyecto de Apoyo a la Construcción de la Plataforma de Innovación Científica y Tecnológica Médica del Hospital Zhongnan de la Universidad de Wuhan (PTXM2025001). La Figura 1 se creó con Figdraw.
Materiales
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
Referencias
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados