Method Article
Sfruttare il potere dei carichi di microRNA in piccole vescicole extracellulari rilasciate da sezioni di cervello umano appena congelate
In questo articolo
Riepilogo
Qui, stabiliamo un protocollo che utilizza quantità minime di sezioni di tessuto cerebrale fresco congelate e un metodo di centrifugazione ad alta velocità accessibile accoppiato con la cromatografia ad esclusione dimensionale per ottenere piccole vescicole extracellulari come fonti di biomarcatori di microRNA (miRNA) per disturbi neurologici.
Abstract
Le piccole vescicole extracellulari (sEV) sono mediatori cruciali della comunicazione cellula-cellula, trasportando carichi diversi come proteine, lipidi e acidi nucleici (microRNA, mRNA, DNA). Il carico di microRNA sEV ha una potenziale utilità come potente biomarcatore di malattia non invasivo grazie alla capacità di sEV di attraversare le barriere biologiche (ad esempio, la barriera emato-encefalica) e diventare accessibile attraverso vari fluidi corporei. Nonostante i numerosi studi sui biomarcatori delle sEV nei fluidi corporei, l'identificazione di sottopopolazioni di sEV specifiche per i tessuti o le cellule rimane impegnativa, in particolare dal cervello. Il nostro studio affronta questa sfida adattando i metodi esistenti per isolare le sEV da quantità minime di sezioni di cervello umano congelate utilizzando la cromatografia ad esclusione dimensionale (SEC).
Dopo l'approvazione etica, circa 250 μg di tessuto cerebrale umano fresco congelato (ottenuto dalla Manchester Brain Bank [UK]) sono stati tagliati dai 3 tessuti donatori e incubati in soluzione di collagenasi di tipo 3/Hibernate-E, con agitazione intermedia, seguita da fasi seriali di centrifugazione e filtrazione. Quindi, le sEV sono state isolate utilizzando il metodo SEC e caratterizzate seguendo le linee guida MISEV. Prima di isolare l'RNA dall'interno di queste sEV, la soluzione è stata trattata con proteinasi-K e RNasi-A per rimuovere qualsiasi RNA extracellulare non sEV. La quantità e la qualità dell'RNA sono state controllate ed elaborate ulteriormente per esperimenti di qPCR e sequenziamento di piccoli RNA.
La presenza di sEV è stata confermata attraverso l'analisi di tracciamento delle nanoparticelle di fluorescenza (fNTA) e il western blot per i marcatori di superficie (CD9, CD63, CD81). La distribuzione dimensionale (50-200 nm) è stata confermata dalla NTA e dalla microscopia elettronica. La concentrazione totale di RNA all'interno delle sEV lisate variava da 3 a 9 ng/μL ed è stata utilizzata per una quantificazione di successo mediante qPCR per microRNA candidati selezionati. Il sequenziamento di piccoli RNA su MiSeq ha fornito dati di alta qualità (Q >32) con 1,4-5 milioni di letture per campione.
Questo metodo consente l'isolamento e la caratterizzazione efficiente delle sEV da volumi minimi di tessuto cerebrale, facilitando la ricerca non invasiva sui biomarcatori ed è promettente per studi equi sui biomarcatori di malattia, offrendo approfondimenti sulle malattie neurodegenerative e potenzialmente su altri disturbi.
Introduzione
Le vescicole extracellulari (EV) sono uno dei principali attori della comunicazione intercellulare in tutti gli organismi multicellulari1. Le EV sono particelle di membrana a doppio strato lipidico derivate da cellule che possono facilitare il trasferimento di una varietà di carichi di carico, come proteine, lipidi e acidi nucleici, alle cellule riceventi. Le vescicole extracellulari possono avere un ampio intervallo di dimensioni che vanno da 30 nm fino a 1 μM. Le vescicole extracellulari piccole (sEV), definite come vescicole legate ai lipidi con un diametro medio di <200 nm, hanno la capacità di attraversare la barriera emato-encefalica; pertanto, sono stati implicati nella diffusione e nell'esacerbazione di malattie neurodegenerative simili ai prioni e di altre condizioni come il morbo di Alzheimer (AD), la demenza frontotemporale (FTD), il morbo di Parkinson (PD) e i tumori 2,3. Inoltre, poiché le sEV possono essere trovate in una serie di biofluidi come il sangue, il liquido cerebrospinale (CSF), la saliva e persino l'urina, il loro uso benefico può abbracciare il campo dei biomarcatori e della diagnostica non invasiva. Ad esempio, la ricerca sull'AD può indicare l'uso di rapporti proteici patogeni biofluidi, come tau/Aβ, o anche Aβ42/Aβ404.
Un carico noto di sEV è il microRNA (miRNA), un gruppo di piccole molecole di RNA non codificanti di circa 22 nucleotidi di lunghezza che si legano alle regioni 3'-UTR dell'mRNA e di solito regolano negativamente l'espressione proteica. Coinvolti in molti ruoli cellulari, i miRNA sono stati anche implicati nella patogenesi di varie malattie, tra cui tumori e malattie neurodegenerative. Cheng et al. hanno condotto un'analisi di sequenziamento ad alto rendimento delle firme di espressione dei miRNA sEV derivate dal siero da pazienti con AD5. Una volta accoppiati con i registri di neuroimaging e i fattori di rischio noti di età, sesso e presentazione dell'allele APOE ε4, i risultati sono risultati predittivi di AD con una sensibilità dell'87% e una specificità del 77%. Inoltre, la ricerca ha identificato due miRNA upregolati nel liquido cerebrospinale (miR-151a-3p, let-7f-5p) e 3 miRNA sottoregolati (miR-27a-3p, miR-125a-5p e miR-423-5p) che possono potenzialmente diagnosticare la PD6 in stadio iniziale. Con le malattie patologiche, lo stato patologico può precedere alcuni sintomi caratteristici delle malattie, mentre nella neurodegenerazione, l'accumulo di segni patologici si verifica molto prima del declino cognitivo.
I microRNA sono potenzialmente un biomarcatore più efficace delle proteine, a causa delle loro diverse funzioni e della regolazione epigenetica di ordine superiore. Utilizzando il tessuto cerebrale, i ricercatori possono potenzialmente identificare specifiche firme di miRNA sEV derivate dal cervello (BDsEV) per le malattie e i loro sottotipi. Ad esempio, le sEV con marcatori neuronali e gliali possono presentare un diverso carico di miRNA e l'analisi può portare a metodi più precisi di rilevamento della malattia. Inoltre, si ritiene che le BDsEV svolgano un ruolo importante nella diffusione transsinaptica delle proteine neuropatogene7. Rapporti precedenti hanno suggerito l'immunoprecipitazione e l'ultracentrifugazione con gradiente di densità (gradiente di saccarosio) per ottenere sEV da tessuto cerebrale appena congelato 8,9. Tuttavia, questi approcci richiedono un'infrastruttura specifica con ultracentrifuga e metodi di purificazione a valle per ottenere campioni di sEV di alta qualità10. Rapporti più recenti hanno suggerito diverse modifiche e miglioramenti all'approccio 11,12,13; tuttavia, nonostante ciò, l'isolamento e lo studio di microRNA derivati da sEV da tessuti umani non sono ancora ampiamente applicati. L'approccio descritto in questo protocollo mira a fornire un protocollo raffinato e graduale per lo studio delle sEV derivate dal cervello per migliorare l'accessibilità a questa tecnica. Abbiamo stabilito un protocollo utilizzando quantità minime di tessuto cerebrale, da cui abbiamo isolato le sEV pure utilizzando la cromatografia ad esclusione dimensionale e mostriamo dati di sequenziamento di nuova generazione di alta qualità dal carico di microRNA di queste sEV.
Protocollo
Il lavoro è stato approvato eticamente dalla Manchester Brain Bank (riferimento REC 09/H0906/52) e dal comitato etico dell'Università di Salford (ID domanda: 3408).
1. Rottura della matrice intracellulare mediante collagenasi su sezioni di tessuto cerebrale congelato
- Affettare sottilmente 250 mg di tessuto cerebrale congelato (frammenti spessi circa 0,4 mm) utilizzando un bisturi sterilizzato su una piastra fredda e aggiungere 2 mL di soluzione di collagenasi di tipo III/ibernato 75 U/mL.
- Incubare ogni campione in un bagno d'acqua a 37 °C per 20 minuti. Al punto di 5 minuti, capovolgere ogni campione due volte per mescolare; al punto di 10 minuti, pipettare delicatamente ogni campione tre volte utilizzando una stripette monouso in plastica da 10 mL.
- Mettere ogni campione sul ghiaccio e aggiungere 1x cocktail di inibitori della proteasi e 1x inibitore della fosfatasi. Questo interrompe la reazione enzimatica ed è il punto finale della digestione utilizzando la collagenasi di tipo III.
- Centrifugare ogni campione di tessuto cerebrale a 300x g per 10 minuti a 4 °C. Raccogliere ogni surnatante e centrifugare ulteriormente a 2000 x g per 15 minuti a 4 °C.
- Raccogliere nuovamente ogni surnatante e filtrarlo attraverso un filtro da 0,22 μm. Centrifugare ciascun filtrato a 10 000 x g per 48 minuti a 4 °C.
- Raccogliere il surnatante e aggiungere il tampone di precipitazione in un rapporto di 2:1 per ciascun campione. Incubare per una notte a 4 °C.
- Centrifugare ciascun campione a 10 000 x g per 96 minuti a 4 °C e risospendere il pellet in 100 μl di PBS libero da vescicole extracellulari (EV).
2. Preparazione delle colonne cromatografiche ad esclusione dimensionale
- Prima dell'uso, equilibrare la colonna SEC pre-preparata a temperatura ambiente (RT) per 15 minuti.
NOTA: Il tappo inferiore deve essere rimosso prima del tappo a vite. - Dopo l'equilibratura, rimuovere il tampone conservante dalla parte superiore della colonna e lavare la colonna due volte utilizzando 250 μl di PBS senza EV. Ogni lavaggio PBS viene lasciato entrare nella colonna per gravità.
3. Isolamento di piccole vescicole extracellulari mediante cromatografia ad esclusione dimensionale
- Aggiungere il pellet risospeso alla sommità della colonna SEC e centrifugare la colonna a 50 x g per 30 s. Scartare il flusso.
- Aggiungere 180 μl di PBS privo di EV alla parte superiore della colonna SEC e centrifugare la colonna a 50 x g per 1 minuto, consentendo alle sEV derivate dal cervello di eluire dalla colonna.
NOTA: La parte del protocollo descritta sopra è riepilogata nella Figura 1.
4. Conferma di piccoli marcatori di vescicole extracellulari mediante western blotting
- Per lisare le sEV prima dell'analisi western blot (per garantire l'analisi di tutti i carichi di membrana e citosolici), diluire il campione di sEV isolato 1:1 in tampone di lisi ed estrazione (1x inibitore della proteasi e 1x inibitore della fosfatasi).
- Agitare il campione a 4 °C per 30 minuti.
- Centrifugare il campione a 14 000 x g per 15 min.
- Raccogli il surnatante proteico e misura le concentrazioni proteiche utilizzando un kit per il dosaggio delle proteine.
- Mescolare i campioni con 4 tamponi Laemmli e caricare 20 μg di proteine in ciascun pozzetto contro una scala proteica precolorata.
- Una volta risolte, trasferire le proteine su una membrana di nitrocellulosa da 0,45 μm. Dopo il trasferimento, bloccare la membrana per 2 ore al 5% BSA.
- Incubare le membrane con anticorpi primari (Tabella 1) durante la notte.
- Lavare la membrana tre volte con un tampone di lavaggio (1% Tween20 in PBS) e colorare con un anticorpo secondario anti-topo. Successivamente, lavare tre volte con tampone di lavaggio (1% Tween20 in PBS).
- Segui i passaggi precedenti nel manoscritto prima di immaginare le macchie.
- Immagine utilizzando il substrato della perossidasi di rafano (HRP).
5. Conferma di piccole vescicole extracellulari mediante analisi di tracciamento delle nanoparticelle (NTA)
- Eseguire l'NTA per analizzare la concentrazione e le dimensioni di tutte le particelle nel campione. A tale scopo, diluire ciascun campione in PBS con un fattore di diluizione di 1:1000.
- Iniettare il campione nello strumento NTA e leggere il campione a una lunghezza d'onda di 550 nm. In questa fase, utilizzare la quantità di particelle per misurare la quantità di proteine per particella (solitamente espressa in femtogramma) come raccomandato nelle linee guida MISEV.
- Successivamente, per l'NTA fluorescente, diluire la maschera cellulare arancione (CMO) (colora tutte le particelle biologiche all'interno di un campione) in PBS con un fattore di diluizione di 1:1000. Da questo, diluire il colorante CMO utilizzando il campione sEV con un fattore di diluizione di 1:10 - Incubare questa fase di diluizione al buio per 30 minuti a 4 °C.
- Diluire la seconda diluizione CMO utilizzando PBS con un fattore di diluizione di 1:1000.
NOTA: La diluizione finale del colorante OCM deve essere 1:10 000 000. - Leggere il CMO di ciascun campione di sEV a una lunghezza d'onda di 550 nm utilizzando lo strumento NTA.
- Per gli anticorpi fluorescenti con tetraspanina (vedi Tabella dei materiali), in primo luogo, diluire ciascun rispettivo anticorpo in PBS con un fattore di diluizione di 1:10. Da questo, diluire ciascuno dei coloranti utilizzando il campione sEV con un fattore di diluizione di 1:10 - Incubare la fase di diluizione al buio per 2 ore a 4 °C.
- Diluire la seconda diluizione anticorpale utilizzando PBS con un fattore di diluizione di 1:1000.
NOTA: La diluizione finale di ciascun anticorpo tetraspanina deve essere 1:100 000. - Leggere le letture degli anticorpi fluorescenti del campione sEV a una lunghezza d'onda di 550 nm utilizzando lo strumento NTA.
6. Conferma di piccole vescicole extracellulari mediante microscopia elettronica a trasmissione (TEM)
NOTA: Questo protocollo è stato eseguito dalla Biomedical Microscopy Facility dell'Università di Liverpool.
- In primo luogo, fissare i campioni di sEV utilizzando glutaraldeide e colorare con acetato di uranile.
- Attraverso una serie di alcoli graduati, disidratare il campione pronto per la TEM.
- Immagini di campioni utilizzando un microscopio elettronico a trasmissione adatto.
7. Trattamento con proteinasi K e RNasi A
- A 50 μL di EV isolate derivate dal cervello, aggiungere 1 μL di 20 μg/μL di proteinasi K e incubare a 37 °C per 30 minuti.
- Arresta ogni reazione aggiungendo 1x cocktail di inibitori della proteinasi e incuba con ghiaccio per 10 minuti.
- Dopo l'incubazione, aggiungere 1 μL di 10 μg/μL di RNasi A a ciascun campione e incubare ulteriormente a 37 °C per 30 minuti.
NOTA: Passare immediatamente alla sezione 8.
8. Isolamento totale dell'RNA da piccole vescicole extracellulari
- Per isolare l'RNA totale, aggiungere 700 μL di reagente di lisi di recupero dell'RNA di alta qualità a 50 μL di campione di EV derivato dal cervello (BDsEV). Agitare ogni campione per 1 ora.
- Aggiungere 140 μl di cloroformio, agitare energicamente ogni campione per 20 s e lasciare incubare a RT per 3 minuti.
- Centrifugare il campione a 12 000 x g per 15 minuti a 4 °C e raccogliere l'interfase dal campione. Aggiungere 1,5 volte il volume di etanolo al 100%.
- Aggiungere 700 μL di colonne di isolamento dell'RNA della miscela interfase/etanolo e centrifugare a ≥8000 x g per 15 s a RT.
NOTA: Ripetere questo passaggio utilizzando tutti i campioni di interfase/etanolo. - Alla colonna, aggiungere 700 μL di tampone di lavaggio e centrifugare a ≥8000 x g per 15 s. Scartare il flusso.
- Aggiungere 500 μl di tampone RPE e centrifugare nuovamente a ≥8000 x g per 15 s. Scartare il flusso.
- Aggiungere 500 μl di etanolo all'80% e centrifugare a ≥8000 x g per 2 minuti. Scartare il flusso.
- A questo punto, asciugare la membrana della colonna centrifugando le colonne alla massima velocità (>8000 x g) per 5 min con il coperchio aperto.
- Trasferire la colonna in una nuova provetta di raccolta, aggiungere 14 μL di acqua priva di RNasi direttamente alla membrana della colonna e centrifugare a ≥8000 x g per 1 minuto per eluire l'RNA totale dalle BDsEV.
- Quantificare il microRNA totale con i campioni utilizzando un kit di dosaggio per la quantificazione dei miRNA, in cui i campioni sono stati misurati su un fluorimetro per quantificazione, selezionando il tipo di test per i miRNA.
9. Sequenziamento di piccoli RNA
- Dopo la quantificazione del miRNA all'interno del campione, sintetizzare una libreria di cDNA utilizzando il kit di preparazione della libreria Small RNA-Seq.
NOTA: Utilizzando questo protocollo, la generazione della libreria includeva quattro passaggi: legatura dell'adattatore 3', purificazione, legatura dell'adattatore 5' e trascrizione inversa - dove, alla fine, viene formata una libreria di RNA/cDNA. Dopo la generazione della libreria, l'amplificazione PCR dell'endpoint avviene con l'aggiunta di primer indice (utilizzati per scopi di multiplexing), dopodiché i campioni vengono purificati. - Per testare la qualità dell'RNA, utilizzare un test applicativo di controllo qualità che descriva in dettaglio le dimensioni di ciascuna preparazione e l'intensità di picco.
- Per verificare la concentrazione di DNA, misurare la quantificazione del DNA su un fluorimetro di quantificazione, selezionando il tipo di saggio dsDNA.
- Preparare i campioni per Small RNA Seq utilizzando il kit di reagenti, dopodiché i campioni vengono caricati nella cella a flusso.
Risultati
Per confermare la presenza di BDsEVsEV, sono state utilizzate tre tecniche: western blotting, NTA e TEM (Figura 3). I risultati del Western blot (Figura 3A e File supplementare 1) mostrano la presenza di tutti e cinque i marcatori positivi (CD9, CD63, CD81, Flot-1 e TSG101) e l'assenza di Calnexina nelle sEV (utilizzate come controllo negativo), confermando l'assenza di contaminazione con il contenuto cellulare. Come previsto, gli omogeneizzati cerebrali (BH) mostrano più proteine di quelle osservate nelle sEV per i marcatori testati. Le immagini TEM hanno confermato la morfologia delle BDsEV e la loro dimensione relativa all'interno del campione (Figura 3B), che è stata ulteriormente confermata dall'analisi del tracciamento delle nanoparticelle (Figura 3C).
La Figura 3C (pannello superiore) mostra le dimensioni delle particelle isolate comprese tra 50 e 200 nm. Dai dati NTA, prendendo il risultato della dispersione come il 100% delle particelle rappresentate, CMO rappresenta circa il 52,35% delle particelle (Figura 3C, pannello inferiore) che hanno un doppio strato lipidico. Di queste particelle colorate con CMO, il 34,66% conteneva CD9, il 15,49% conteneva CD63 e il 12,01% conteneva CD81. Fare riferimento al file supplementare 2 per i dati di controllo NTA.
I dati di quantificazione dell'RNA isolato (Tabella 2) mostrano livelli compresi tra 3,24 e 8,76 (ng/μL), valori che sono stati normalizzati durante la generazione delle librerie di cDNA. I risultati del test TapeStation (Figura 4) mostrano picchi importanti di circa 120-160 bp, con picchi più piccoli inferiori a circa 50 bp e altri picchi più piccoli superiori a circa 484 bp.
I controlli di qualità per i dati di sequenziamento di nuova generazione mostrano letture di altissima qualità dopo il taglio dell'adattatore (Figura 5A). Tutte le posizioni di sequenza mostrano un punteggio Phred di >30, che indica tassi di errore inferiori a 1 su 1000 nucleotidi. La Figura 5B mostra che la maggior parte delle sequenze si aggira intorno a 21-22 bp, corrispondendo all'intervallo di dimensioni previsto per i microRNA. Attraverso un'ulteriore mappatura a valle (utilizzando BowTie2), utilizzando il biotool di analisi flagstat di Samtools, su 1.304.100 sequenze allineate, 1.116.264 (85%) sono state mappate nelle regioni dei microRNA nel genoma umano (build hg38; Tabella 3).
Del totale dei miRNA, 808 miRNA sono stati espressi attraverso tutti e 3 i campioni, con 344 miRNA condivisi tra tutti e tre i campioni (~43%), 5 condivisi tra il controllo 1 e il controllo 2, 183 condivisi tra il controllo 1 e il controllo 3 e 16 condivisi tra il controllo 2 e il controllo 3 (Figura 6). Da questi dati, i miRNA espressi più alti in tutti e tre i BDsEV sono has-let-7b-5p, has-miR-143-3p, has-miR-30a-5p, has-miR-221-3p e has-let7i-5p. Allo stesso modo, i primi 5 miRNA espressi in modo meno differenziale nei campioni di BDsEV (letture >10) sono has-miR-128-2-5p, has-miR-182-5p, has-miR-193b-5p, has-miR-448 e has-miR-505-3p. Tutti i conteggi delle letture grezze e normalizzati sono riportati nel file supplementare 3.

Figura 1: Flusso di lavoro per l'elaborazione dei tessuti. La figura mostra il processo graduale per l'isolamento di sEV derivate dal cervello (BDsEV) da sezioni cerebrali appena congelate utilizzando la cromatografia ad esclusione dimensionale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Flusso di lavoro per l'isolamento dell'RNA da BDsEVsEVs. La figura mostra il processo graduale di isolamento dell'RNA totale dalle BDsEVsEV, inclusa la fase di pre-trattamento di rimozione di qualsiasi RNA extracellulare utilizzando la proteinasi K e la RNasi A. Fare clic qui per visualizzare una versione più grande di questa figura.
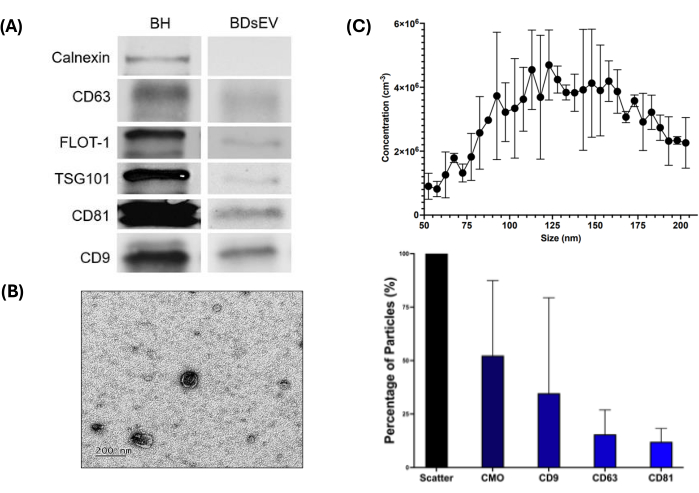
Figura 3: Flusso di lavoro di caratterizzazione BDsEV. La figura mostra vari metodi per caratterizzare i BDsEV. (A) Western blot per rilevare marcatori proteici specifici per sEV. BH Cervello omogeneizzato. La calnexina è un controllo negativo per le sEV: l'assenza in sEV e la presenza in BH confermano la purezza del campione di sEV. Per altri marcatori, come previsto, la quantità di proteine è maggiore in BH rispetto ai campioni sEV (20 μg di proteina caricata per pozzetto). (B) Immagine rappresentativa al microscopio elettronico a trasmissione (TEM) per BDsEV su scala 200 μM, che conferma la morfologia attesa con una membrana a doppio strato lipidico. (C) Grafico della distribuzione dimensionale per l'analisi di tracciamento delle nanoparticelle (NTA) per tutte le particelle (pannello superiore). Ogni punto dati rappresenta la variazione tra tre repliche. Proporzioni normalizzate per particelle biologiche (sEV) e proporzioni relative per specifici marcatori di superficie (pannello inferiore) (dati mostrati come media ± SEM dei pozzetti triplicati (n = 3)). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Controllo di qualità per la preparazione della libreria di cDNA. Analisi con tapestation per singoli campioni dopo la preparazione della libreria di cDNA. I picchi visualizzati in ciascun grafico mostrano la dimensione di ciascuna preparazione (asse orizzontale, in bp) e la rispettiva intensità di ciascun picco (asse verticale, FU). La libreria di piccoli RNA prevista raggiunge picchi intorno a 120-160 bp, come si vede nella figura. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Controllo di qualità per il sequenziamento miRnome. (A) Grafico FASTQC per la qualità di lettura dei singoli campioni dopo il taglio dell'adattatore. L'asse orizzontale è per la posizione nucleotidica e l'asse verticale è per i punteggi Phred. I box plot per ogni posizione mostrano la distribuzione del punteggio Phred in tutte le letture per ogni posizione. Tutte le distribuzioni all'interno della zona verde confermano dati di altissima qualità (>Q30) che implicano una preparazione del campione di alta qualità. (B) La distribuzione della lunghezza della sequenza per ciascun campione mostra che per tutti e tre i campioni, la maggior parte delle letture era di circa 21-22 nucleotidi, l'intervallo di dimensioni previsto per i microRNA. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Sovrapposizione dell'espressione dei MicroRNA in tutti i campioni. Il numero di miRNA rilevati da ciascun campione (conteggio delle letture ≥ 10) e i microRNA specifici condivisi in più di un campione sono mostrati nel diagramma di Venn. Tutte le letture non elaborate e normalizzate sono mostrate nel file supplementare 3. Clicca qui per visualizzare una versione più grande di questa figura.
| Epitopo anticorpale | Fattore di diluizione dell'anticorpo |
| Mouse Anti-CD9 | 1:2000 |
| Mouse Anti-CD63 | 1:2000 |
| Mouse Anti-CD81 | 1:2000 |
| Topo Anti-Flottiglia 1 | 1:2000 |
| Mouse Anti-TSG101 | 1:1000 |
| Mouse Anti-Calnexin | 1:10 000 |
| Anticorpo secondario anti-topo | 1:3000 |
Tabella 1: Epitopi anticorpali utilizzati per la caratterizzazione delle BDsEV e dei rispettivi fattori di diluizione.
| Campione | Concentrazione di miRNA (ng/μL) |
| 1 | 8.76 |
| 2 | 3.24 |
| 3 | 5.33 |
Tabella 2: Dati di quantificazione dell'RNA isolato.
| Campione | Sequenze allineate | MiRNA mappato | Percentuale (%) |
| Campione di controllo 1 | 1 444 665 | 1 133 437 | 78.46 |
| Campione di controllo 2 | 442 808 | 381 631 | 86.18 |
| Campione di controllo 3 | 2 024 828 | 1 983 726 | 97.97 |
| Nella media | 1 304 100.33 | 1 166 264.67 | 89.43 |
Tabella 3: Quantità di sequenze di miRNA allineate e mappate con successo.
File supplementare 1: Dati supplementari dell'analisi del western blot. Clicca qui per scaricare questo file.
File supplementare 2: Dati di controllo NTA supplementari. Clicca qui per scaricare questo file.
File supplementare 3: Conteggi delle letture grezze e normalizzate dell'analisi dell'espressione dei miRNA. Clicca qui per scaricare questo file.
File supplementare 4: Dati supplementari sulla concentrazione proteica. Clicca qui per scaricare questo file.
Discussione
Questo protocollo modificato e migliorato per l'isolamento di piccole vescicole extracellulari derivate dal cervello e del loro carico di microRNA dimostra la fattibilità dell'utilizzo di una quantità minima di tessuto senza compromettere la qualità e la quantità dei prodotti a valle. Nel campo della scoperta di biomarcatori, l'identificazione di identificatori molecolari specifici per i tipi di cellule e tessuti può portare a mezzi più accessibili per test diagnostici non invasivi che utilizzano i fluidi corporei. Inoltre, l'approccio qui descritto fornisce il quadro per l'identificazione di specifiche sottopopolazioni di sEV all'interno di diversi tipi di cellule (neuronali vs. gliali), tipi di tessuto (tessuto ippocampale vs tessuto della corteccia prefrontale) e biofluidi, che offriranno una maggiore comprensione delle firme di carico del carico, della fisiopatologia e dell'estensione della malattia. Come precedentemente delineato in Vella et al., è stato suggerito che da 450 mg a 1 g dovrebbero essere utilizzati per l'isolamento di BDsEV, accoppiato con l'ultracentrifugazione a gradiente di densità (utilizzando velocità fino a 180.000 x g)8. In questa metodologia, le sEV sono state isolate con successo utilizzando quantità inferiori (riduzione del >50%) di campioni di tessuto cerebrale fresco congelato (250-450 mg). La riduzione della quantità di tessuto cerebrale utilizzato per isolare le sEV porta a una riduzione dei costi e dei materiali di consumo. Allo stesso modo, il protocollo è stato reso più accessibile attraverso l'applicazione del SEC abbinato alla centrifugazione ad alta velocità (10.000 x g) invece dell'ultracentrifugazione (180.000 x g).
Utilizzando fNTA e TEM, abbiamo stabilito che il profilo dell'intervallo di dimensioni delle sEV rientra nell'intervallo di dimensioni caratteristico di 30-200 nm, come raccomandato dalle linee guida MISEV 13,14, con TEM che fornisce conferma morfologica delle sEV ottenute. Come mostrato nella Figura 3C e nel File Supplementare 4, il >87% delle particelle ha dimensioni <250 nm e i campioni, in media, mostrano 10,56 fg protein/BDsEV. Specie specifiche di piccole vescicole extracellulari variano in dimensioni e forma, dove si osserva che sottopopolazioni tipicamente più piccole, come gli esosomi, hanno una morfologia uniforme rispetto alle microvescicole che si dice abbiano una struttura irregolare a causa della loro natura di biogenesi15. Per l'fNTA, è stata prodotta una lettura di dispersione (520 nm) per analizzare le particelle totali nei campioni, fornendo dettagli sul profilo dimensionale e sulle concentrazioni. Le letture mostrano che oltre il 50% delle particelle erano biologiche (attraverso la colorazione di CMO), con popolazioni di particelle biologiche che rientrano nell'intervallo di dimensioni delle sEV che mostrano marcatori di tetraspanina, con CD9 che è il più predominante nei nostri campioni di controllo (34,66%). Mentre per il western blotting, la presenza di carichi proteici caratteristici di membrana e citosolici ha fornito evidenza della presenza di sEV, con l'assenza di calnexina che conferma che le BDsEV sono prive di qualsiasi contaminazione cellulare. Per garantire ulteriormente che il miRNA disponibile per l'elaborazione a valle facesse parte del carico di sEV (e non di altro RNA extracellulare), abbiamo trattato le sEV con gli enzimi proteinasi K e RNasi A prima di interrompere le membrane delle sEV per degradare qualsiasi proteina e RNA presenti esternamente nello spazio extracellulare al di fuori delle BDsEV16.
L'RNA totale è stato estratto dalle sEV e preparato per applicazioni a valle come la qRT-PCR e il sequenziamento di nuova generazione (per piccoli RNA). I controlli standard che utilizzano saggi di controllo qualità hanno mostrato campioni di alta qualità e librerie di cDNA pronti per il sequenziamento. Le librerie di cDNA raggiungono un picco di circa 120-160 bp, attribuendo picchi di cDNA derivati da miRNA con sequenze adattatrici di lunghezze variabili, con eventuali picchi più piccoli che potenzialmente rappresentano dimeri adattatori.
L'analisi dei dati di sequenziamento di nuova generazione utilizzando FASTQC e CutAdapt mostra risultati di alta qualità ottenuti dai miRNA BDsEV. Come mostrato nella Figura 5, ogni posizione letta ha un punteggio Q superiore a 30 (Q30), mostrando una probabilità di errore di 0,001-0,0001 (1 su 1000-10.000). Inoltre, l'analisi della distribuzione della lunghezza delle sequenze (Figura 6) mostra picchi predominanti che rientrano nell'intervallo caratteristico dei miRNA (19-25 bp), suggerendo un'elevata qualità e specificità dei dati ottenuti.
Numerosi studi futuri trarranno vantaggio da questo metodo grazie alla sua migliore accessibilità, oltre a risparmiare costi e risorse grazie alla riduzione non solo della quantità di campioni di tessuto, ma anche del costo dei reagenti. La riduzione (>50%) della quantità di tessuto necessaria rende questo metodo anche più etico. In linea con gli sforzi globali per la salute di precisione, il flusso di lavoro migliorato qui descritto fornisce strumenti relativamente più convenienti per intraprendere biomarcatori accurati specifici per tipo di cellula attraverso percorsi non invasivi (o minimamente invasivi) utilizzando BDsEV e il loro carico. Questo può essere utilizzato per un'ampia gamma di condizioni neurologiche. Una possibile limitazione di questo metodo è che il protocollo dovrà essere aggiustato se i ricercatori vogliono utilizzare un tessuto diverso dal cervello umano per l'isolamento delle sEV. A lungo termine, questo metodo migliorato renderà la scoperta e la convalida di biomarcatori tessuto-specifici non invasivi più equi e accessibili in tutto il mondo.
Divulgazioni
Non ci sono conflitti di interesse per nessuno degli autori.
Riconoscimenti
Questo lavoro è stato finanziato dalla borsa di dottorato per Joseph Morgan dell'Alzheimer's Society UK (Grant number 549/SERA-52) e dai fondi per la strategia di innovazione dell'Università di Salford (grant SEFA-39). Il tessuto cerebrale è stato ottenuto dalla banca del cervello di Manchester (riferimento REC 09/H0906/52) del Brains for Dementia Network.
Materiali
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin | Merck | A9418-100G | |
| Cell Mask Orange Plasma Membrane Stain | ThermoFisher Scientific | C10045 | |
| Collagenase Type-III | StemCell Technologies | 07422 | |
| ExoSpin Columns and Buffer | Cell Guidance Systems Ltd. | EX01-50 | This kit contains SEC columns used in this experiment, precipitation buffer and EV free PBS. |
| Halt Protease Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78429 | |
| Hibernate-E Medium | ThermoFisher Scientific | A1247601 | |
| Laemmli Sample Buffer (4x) | BioRad | 1610747 | |
| Lexogen Small RNA-Seq Library Prep Kit | Lexogen | 052.24 | This kit contains Small RNA preparation reagent box with i7 Index primer plate. |
| miRNeasy Micro Kit (50) | Qiagen | 217084 | This kit contains high-quality RNA recovery lysis reagent (Qiazol), RNA isolation columns, isolation buffers (RWT, RPE) and RNase free water. |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3001 | This is the Illumina Preparation Kit |
| Nitrocellulose Membrane, 0.45 μm | ThermoFisher Scientific | 88018 | |
| PE/Dazzle 594 anti-human CD63 Antibody | BioLegend | 143914 | Used for fNTA Analysis |
| PE/Dazzle 594 anti-human CD81 Antibody | BioLegend | 349520 | Used for fNTA Analysis |
| PE/Dazzle 594 anti-human CD9 Antibody | BioLegend | 312118 | Used for fNTA Analysis |
| PhosSTOP | Merck | 4906845001 | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23225 | |
| Proteinase K | ThermoFisher Scientific | 25530049 | |
| Qubit microRNA Assay Kit | ThermoFisher Scientific | Q32880 | |
| Qubit 1X dsDNA HS assay kit | ThermoFisher Scientific | Q33230 | |
| Qubit 3.0 Fluorometer | ThermoFisher Scientific | Q33216 | |
| RIPA Lysis and Extraction Buffer | ThermoFisher Scientific | 89901 | |
| RNase A | ThermoFisher Scientific | EN0531 | |
| SuperSignal West Femto Maximum Sensitivity Substrate | ThermoFisher Scientific | 34094 |
Riferimenti
- Jia, Y., et al. Small extracellular vesicles isolation and separation: Current techniques, pending questions and clinical applications. Theranostics. 12 (15), 6548-6575 (2022).
- Fornari, S., Schäfer, A., Jucker, M., Goriely, A., Kuhl, E. Prion-like spreading of Alzheimer's disease within the brain's connectome. J R Soc Interface. 16 (159), 20190356(2019).
- Zhang, P., et al. Tumor-derived small extracellular vesicles in cancer invasion and metastasis: molecular mechanisms, and clinical significance. Mol Cancer. 23 (1), 18(2024).
- Constantinides, V., et al. CSF Aβ42 and Aβ42/ Aβ40 ratio in Alzheimer's disease and frontotemporal dementia. Diagnostics. 13 (4), 783(2023).
- Cheng, L., et al. Prognostic serum miRNA biomarkers associated with Alzheimer's disease shows concordance with neuropsychological and neuroimaging assessment. Mol Psychiatry. 20 (10), 1188-1196 (2015).
- Roser, A. E., Caldi Gomes, L., Schünemann, J., Maass, F., Lingor, P. Circulating miRNAs as diagnostic biomarkers for Parkinson's disease. Front Neurosci. 12, 625(2018).
- Jackson, N., Guerrero-Muñoz, M. J., Castillo-Carranza, D. L. The prion-like transmission of tau oligomers via exosomes. Front Aging Neurosci. 14, 974414(2022).
- Vella, L., et al. A rigorous method to enrich for exosomes from brain tissue. J Extracell Vesicles. 6 (1), 1348885(2017).
- Yousif, G., Qadri, S., Parray, A., Akhthar, N., Shuaib, A., Haik, Y. Exosomes derived neuronal markers: Immunoaffinity isolation and characterization. Neuromolecular Med. 24 (3), 339-351 (2022).
- Kumar, K., et al. Recent advances in microfluidic approaches for the isolation and detection of exosomes. TRAC-Trend Anal Chem. 159, 116912(2023).
- Ransom, L., et al. Human brain small extracellular vesicles contain selectively packaged, full-length mRNA. Cell Rep. 43 (4), 114061(2024).
- Gomes, P., et al. A novel isolation method for spontaneously released extracellular vesicles from brain tissue and its implication for stress-driven brain pathology. Cell Commun Signal. 21 (1), 35(2023).
- Welsh, J., et al. Minimal information for studies of extracellular vesicles (MISEV 2023): From basic to advanced approaches. J Extracell Vesicles. 13 (2), e12404(2023).
- Théry, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750(2018).
- Pirisinu, M. The long journey of extracellular vesicles towards global scientific acclamation. Adv Pharm Bull. 13 (3), 489-501 (2023).
- Bender, A., Sullivan, B. P., Lillis, L., Posner, J. D. Enzymatic and chemical-based methods to inactivate endogenous blood ribonucleases for nucleic acid diagnostics. J Mol Diagn. 22 (8), 1030-1040 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon