Method Article
Nutzung der Kraft von microRNA-Ladungen in kleinen extrazellulären Vesikel, die aus frisch gefrorenen menschlichen Gehirnabschnitten freigesetzt werden
In diesem Artikel
Zusammenfassung
Hier etablieren wir ein Protokoll mit minimalen Mengen an frisch gefrorenen Hirngewebeschnitten und einer zugänglichen Hochgeschwindigkeits-Zentrifugationsmethode in Verbindung mit Größenausschlusschromatographie, um kleine extrazelluläre Vesikel als Quellen für microRNA (miRNA)-Biomarker für neurologische Erkrankungen zu gewinnen.
Zusammenfassung
Kleine extrazelluläre Vesikel (sEVs) sind wichtige Mediatoren der Zell-Zell-Kommunikation und transportieren verschiedene Frachten wie Proteine, Lipide und Nukleinsäuren (microRNA, mRNA, DNA). Die microRNA-sEV-Fracht hat einen potenziellen Nutzen als leistungsstarker nicht-invasiver Biomarker für Krankheiten, da sEV biologische Barrieren (z. B. die Blut-Hirn-Schranke) überwinden und durch verschiedene Körperflüssigkeiten zugänglich werden kann. Trotz zahlreicher Studien zu sEV-Biomarkern in Körperflüssigkeiten bleibt die Identifizierung von gewebe- oder zellspezifischen sEV-Subpopulationen, insbesondere aus dem Gehirn, eine Herausforderung. Unsere Studie geht diese Herausforderung an, indem sie bestehende Methoden zur Isolierung von sEVs aus minimalen Mengen gefrorener menschlicher Gehirnschnitte mittels Größenausschlusschromatographie (SEC) anpasst.
Nach der ethischen Genehmigung wurden etwa 250 μg frisch gefrorenes menschliches Hirngewebe (von der Manchester Brain Bank [UK]) aus den 3 Spendergeweben geschnitten und in Kollagenase Typ 3/Hibernate-E-Lösung inkubiert, mit Zwischenrührung, gefolgt von seriellen Zentrifugations- und Filtrationsschritten. Anschließend wurden die sEVs mit der SEC-Methode isoliert und unter Befolgung der MISEV-Richtlinien charakterisiert. Vor der Isolierung von RNA aus diesen sEVs wurde die Lösung mit Proteinase-K und RNase-A behandelt, um extrazelluläre RNA ohne sEV zu entfernen. Die RNA-Menge und -Qualität wurden für qPCR- und Small-RNA-Sequenzierungsexperimente überprüft und weiterverarbeitet.
Das Vorhandensein von sEVs wurde durch Fluoreszenz-Nanopartikel-Tracking-Analyse (fNTA) und Western Blot für Oberflächenmarker (CD9, CD63, CD81) bestätigt. Die Größenverteilung (50-200 nm) wurde durch NTA und Elektronenmikroskopie bestätigt. Die Gesamt-RNA-Konzentration in lysierten sEVs lag im Bereich von 3-9 ng/μL und wurde für die erfolgreiche Quantifizierung ausgewählter microRNA-Kandidaten mittels qPCR verwendet. Die Small-RNA-Sequenzierung auf MiSeq lieferte qualitativ hochwertige Daten (Q >32) mit 1,4-5 Millionen Reads pro Probe.
Diese Methode ermöglicht die effiziente Isolierung und Charakterisierung von sEVs aus minimalen Hirngewebevolumina, erleichtert die nicht-invasive Biomarkerforschung und ist vielversprechend für gerechte Biomarker-Studien, die Einblicke in neurodegenerative Erkrankungen und potenziell andere Erkrankungen bieten.
Einleitung
Extrazelluläre Vesikel (EVs) sind einer der Hauptakteure der interzellulären Kommunikation in allen mehrzelligen Organismen1. EVs sind aus Zellen gewonnene Lipid-Doppelschicht-Membranpartikel, die die Übertragung einer Vielzahl von Frachtladungen wie Proteinen, Lipiden und Nukleinsäuren auf Empfängerzellen erleichtern können. EVs können einen breiten Größenbereich von 30 nm bis 1 μM haben. Kleine EVs (sEVs), definiert als lipidgebundene Vesikel mit einer durchschnittlichen Durchmessergröße von <200 nm, haben die Fähigkeit, die Blut-Hirn-Schranke zu überwinden; Daher wurden sie mit der prionenartigen Ausbreitung und Verschlimmerung von neurodegenerativen Erkrankungen und anderen Erkrankungen wie Alzheimer (AD), frontotemporaler Demenz (FTD), Parkinson (PD) und Krebs in Verbindung gebracht 2,3. Da sEVs in einer Reihe von Bioflüssigkeiten wie Blut, Liquor cerebrospinalis (CSF), Speichel und sogar Urin zu finden sind, kann sich ihr vorteilhafter Nutzen über den Bereich der Biomarker und der nicht-invasiven Diagnostik erstrecken. Zum Beispiel könnte die AD-Forschung auf die Verwendung von biofluiden pathogenen Proteinverhältnissen wie tau/Aβ oder sogar Aβ42/Aβ404 hinweisen.
Eine bekannte Ladung von sEVs ist microRNA (miRNA), eine Gruppe kleiner, nicht-kodierender RNA-Moleküle mit einer Länge von etwa 22 Nukleotiden, die an die 3'-UTR-Regionen der mRNA binden und die Proteinexpression normalerweise negativ regulieren. miRNAs sind an vielen zellulären Rollen beteiligt und wurden auch mit der Pathogenese verschiedener Krankheiten, einschließlich Krebs und neurodegenerativer Erkrankungen, in Verbindung gebracht. Cheng et al. führten eine Hochdurchsatz-Sequenzierungsanalyse von serumabgeleiteten sEV miRNA-Expressionssignaturen von Alzheimer-Patientendurch 5. In Verbindung mit Neuroimaging-Aufzeichnungen und bekannten Risikofaktoren wie Alter, Geschlecht und APOE ε4-Allelpräsentation zeigten die Ergebnisse eine Vorhersage der Alzheimer-Krankheit mit einer Sensitivität von 87 % und einer Spezifität von 77 %. Darüber hinaus hat die Forschung zwei hochregulierte Liquor-miRNAs (miR-151a-3p, let-7f-5p) und 3 herunterregulierte miRNAs (miR-27a-3p, miR-125a-5p und miR-423-5p) identifiziert, die möglicherweise PD6 im Frühstadium diagnostizieren können. Bei pathologischen Erkrankungen kann der pathologische Status bestimmten charakteristischen Krankheitssymptomen vorausgehen, während bei der Neurodegeneration die Anhäufung pathologischer Merkmale viel früher erfolgt als der kognitive Verfall.
MicroRNAs sind aufgrund ihrer vielfältigen Funktionen und einer epigenetischen Regulation höherer Ordnung potenziell ein wirksamerer Biomarker als Proteine. Anhand von Hirngewebe können Forscher möglicherweise spezifische vom Gehirn abgeleitete sEV (BDsEV) miRNA-Signaturen für Krankheiten und ihre Subtypen identifizieren. Zum Beispiel können sEVs mit neuronalen und glialen Markern unterschiedliche miRNA-Fracht aufweisen, und die Analyse kann zu präziseren Methoden zur Krankheitserkennung führen. Darüber hinaus wird vermutet, dass BDsEVs eine große Rolle bei der transsynaptischen Ausbreitung neuropathogener Proteine spielen7. Frühere Berichte haben Immunpräzipitation und Dichtegradienten-Ultrazentrifugation (Saccharosegradient) vorgeschlagen, um sEVs aus frisch gefrorenem Hirngewebe zu erhalten 8,9. Diese Ansätze erfordern jedoch eine spezifische Infrastruktur mit Ultrazentrifuge und nachgeschalteten Aufreinigungsmethoden, um qualitativ hochwertige sEV-Proben zu gewinnen10. In neueren Berichten wurden mehrere Änderungen und Verbesserungen des Ansatzes vorgeschlagen 11,12,13; Trotzdem sind die Isolierung und Untersuchung von sEV-abgeleiteter microRNA aus menschlichen Geweben immer noch nicht weit verbreitet. Der in diesem Protokoll beschriebene Ansatz zielt darauf ab, ein verfeinertes Schritt-für-Schritt-Protokoll für sEV-Studien aus dem Gehirn bereitzustellen, um den Zugang zu dieser Technik zu verbessern. Wir haben ein Protokoll mit minimalen Mengen an Hirngewebe etabliert, aus dem wir reine sEVs mittels Größenausschlusschromatographie isoliert haben und qualitativ hochwertige Next-Generation-Sequencing-Daten aus der microRNA-Fracht dieser sEVs gezeigt haben.
Protokoll
Die Arbeit wurde von der Manchester Brain Bank (REC-Referenz 09/H0906/52) und von der Ethikkommission der University of Salford (Bewerbungs-ID: 3408) ethisch genehmigt.
1. Abbau der intrazellulären Matrix mittels Kollagenase an gefrorenen Hirngewebeschnitten
- 250 mg gefrorenes Hirngewebe (ca. 0,4 mm dicke Fragmente) mit einem sterilisierten Skalpell auf einer Kühlplatte in dünne Scheiben schneiden und 2 ml 75 U/ml-Kollagenase Typ-III/Hibernate-E-Lösung hinzufügen.
- Jede Probe wird 20 Minuten lang in einem Wasserbad bei 37 °C inkubiert. Bei 5 Minuten invertieren Sie jede Probe zweimal, um sie zu mischen; Pipettieren Sie jede Probe nach 10 Minuten dreimal vorsichtig mit einer 10-ml-Einweg-Stripette aus Kunststoff.
- Legen Sie jede Probe auf Eis und fügen Sie 1x Proteasehemmer-Cocktail und 1x Phosphatase-Hemmer hinzu. Dadurch wird die Enzymreaktion gestoppt und es ist der Endpunkt der Verdauung mittels Kollagenase Typ-III.
- Jede Hirngewebeprobe wird bei 300x g für 10 min bei 4 °C zentrifugiert. Jeder Überstand wird aufgefangen und bei 2000 x g 15 min bei 4 °C weiter zentrifugiert.
- Jeder Überstand wird erneut aufgefangen und durch einen 0,22-μm-Filter filtriert. Jedes Filtrat wird bei 10 000 x g 48 Minuten lang bei 4 °C zentrifugiert.
- Den Überstand auffangen und Fällungspuffer im Verhältnis 2:1 zu jeder Probe hinzufügen. Über Nacht bei 4 °C inkubieren.
- Jede Probe wird 96 Minuten lang bei 4 °C bei 10 000 x g zentrifugiert und das Pellet in 100 μl extrazellulärem Vesikel (EV)-freiem PBS resuspendiert.
2. Vorbereitung von Größenausschluss-Chromatographiesäulen
- Vor der Verwendung die vorbereitete SEC-Säule 15 min lang bei Raumtemperatur (RT) äquilibrieren.
HINWEIS: Die untere Kappe muss vor dem Schraubverschluss entfernt werden. - Entfernen Sie nach der Äquilibrierung den Konservierungsmittelpuffer von der Oberseite der Säule und waschen Sie die Säule zweimal mit 250 μl EV-freiem PBS. Bei jedem PBS-Waschgang gelangt er durch die Schwerkraft in die Säule.
3. Isolierung kleiner extrazellulärer Vesikel mittels Größenausschlusschromatographie
- Geben Sie das resuspendierte Pellet auf die Oberseite der SEC-Säule und zentrifugieren Sie die Säule bei 50 x g für 30 s. Verwerfen Sie den Durchfluss.
- Geben Sie 180 μl EV-freies PBS auf die Oberseite der SEC-Säule und zentrifugieren Sie die Säule 1 Minute lang bei 50 x g , so dass die aus dem Gehirn stammenden sEVs aus der Säule eluieren können.
HINWEIS: Der oben beschriebene Teil des Protokolls ist in Abbildung 1 zusammengefasst.
4. Bestätigung kleiner extrazellulärer Vesikelmarker durch Western Blot
- Um sEVs vor der Western-Blot-Analyse zu lysieren (um sicherzustellen, dass alle Membran- und zytosolischen Ladungen analysiert werden), verdünnen Sie die isolierte sEV-Probe 1:1 in Lyse- und Extraktionspuffer (1x Proteaseinhibitor und 1x Phosphatase-Inhibitor).
- Die Probe wird 30 Minuten lang bei 4 °C gerührt.
- Zentrifugieren Sie die Probe bei 14 000 x g für 15 min.
- Sammeln Sie den Proteinüberstand und messen Sie die Proteinkonzentrationen mit einem Protein-Assay-Kit.
- Mischen Sie die Proben mit 4x Laemmli-Puffer und laden Sie 20 μg Protein in jede Vertiefung gegen eine vorgefärbte Proteinleiter.
- Sobald sie aufgelöst sind, übertragen Sie die Proteine auf eine 0,45 μm große Nitrozellulosemembran. Blockieren Sie die Membran nach dem Transfer für 2 h in 5 % BSA.
- Inkubieren Sie die Membranen über Nacht mit Primärantikörpern (Tabelle 1).
- Waschen Sie die Membran dreimal mit Waschpuffer (1 % Tween20 in PBS) und färben Sie sie mit einem Anti-Maus-Sekundärantikörper. Danach dreimal mit Waschpuffer (1 % Tween20 in PBS) waschen.
- Befolgen Sie die obigen Schritte im Manuskript, bevor Sie die Blots abbilden.
- Bild mit Meerrettichperoxidase (HRP)-Substrat.
5. Nachweis kleiner extrazellulärer Vesikel durch Nanopartikel-Tracking-Analyse (NTA)
- Führen Sie eine NTA durch, um die Konzentration und Größe aller Partikel in der Probe zu analysieren. Dazu wird jede Probe in PBS mit einem Verdünnungsfaktor von 1:1000 verdünnt.
- Injizieren Sie die Probe in das NTA-Gerät und lesen Sie die Probe bei einer Wellenlänge von 550 nm ab. Verwenden Sie zu diesem Zeitpunkt die Partikelmenge, um die Proteinmenge pro Partikel (normalerweise in Femtogramm ausgedrückt) zu messen, wie in den MISEV-Richtlinien empfohlen.
- Als nächstes verdünnen Sie für fluoreszierendes NTA die orangefarbene Zellmaske (CMO) (färbt alle biologischen Partikel in einer Probe) in PBS mit einem Verdünnungsfaktor von 1:1000. Daraus wird der CMO-Farbstoff mit einer sEV-Probe mit einem Verdünnungsfaktor von 1:10 verdünnt - Inkubieren Sie diesen Verdünnungsschritt 30 min lang im Dunkeln bei 4 °C.
- Die zweite GMO-Verdünnung mit PBS um einen Verdünnungsfaktor von 1:1000 verdünnen.
HINWEIS: Die endgültige Verdünnung des CMO-Farbstoffs muss 1:10 000 000 betragen. - Lesen Sie den CMO jeder sEV-Probe bei einer Wellenlänge von 550 nm mit dem NTA-Gerät ab.
- Für die Tetraspanin-Fluoreszenzantikörper (siehe Materialtabelle) wird zunächst der jeweilige Antikörper in PBS mit einem Verdünnungsfaktor von 1:10 verdünnt. Daraus verdünnen Sie jeden der Farbstoffe mit einer sEV-Probe um einen Verdünnungsfaktor von 1:10 - Inkubieren Sie den Verdünnungsschritt im Dunkeln für 2 h bei 4 °C.
- Verdünnen Sie die zweite Antikörperverdünnung mit PBS um einen Verdünnungsfaktor von 1:1000.
HINWEIS: Die endgültige Verdünnung jedes Tetraspanin-Antikörpers muss 1:100 000 betragen. - Lesen Sie die Fluoreszenz-Antikörper-Messwerte der sEV-Probe bei einer Wellenlänge von 550 nm mit dem NTA-Gerät.
6. Nachweis kleiner extrazellulärer Vesikel durch Transmissionselektronenmikroskopie (TEM)
HINWEIS: Dieses Protokoll wurde von der Biomedical Microscopy Facility an der University of Liverpool durchgeführt.
- Fixieren Sie zunächst sEV-Proben mit Glutaraldehyd und färben Sie sie mit Uranylacetat.
- Dehydrieren Sie die Probe durch eine abgestufte Reihe von Alkoholen, die für die TEM bereit sind.
- Nehmen Sie Proben mit einem geeigneten Transmissionselektronenmikroskop ab.
7. Behandlung mit Proteinase K und RNase A
- Zu 50 μl isolierten aus dem Gehirn gewonnenen EVs wird 1 μl 20 μg/μl Proteinase K hinzugefügt und 30 Minuten lang bei 37 °C inkubiert.
- Stoppen Sie jede Reaktion, indem Sie 1x Proteinase-Inhibitor-Cocktail hinzufügen und 10 Minuten auf Eis inkubieren.
- Nach der Inkubation 1 μl 10 μg/μl RNase A zu jeder Probe geben und 30 Minuten lang bei 37 °C weiter inkubieren.
HINWEIS: Fahren Sie sofort mit Abschnitt 8 fort.
8. Vollständige RNA-Isolierung aus kleinen extrazellulären Vesikeln
- Um die Gesamt-RNA zu isolieren, fügen Sie 700 μl hochwertiges RNA-Rückgewinnungs-Lysereagenz zu 50 μl einer Probe aus dem Gehirn (BDsEV) hinzu. Jede Probe 1 h lang rühren.
- 140 μl Chloroform zugeben, jede Probe 20 s lang kräftig schütteln und 3 Minuten bei RT inkubieren lassen.
- Die Probe wird bei 15 min bei 4 °C bei 12 000 x g zentrifugiert und die Interphase aus der Probe entnommen. Fügen Sie 1,5x Volumen 100% Ethanol hinzu.
- 700 μl Interphase/Ethanol-Gemisch, RNA-Isolationssäulen zugeben und bei ≥8000 x g für 15 s bei RT zentrifugieren.
HINWEIS: Wiederholen Sie diesen Schritt mit allen Interphase-/Ethanolproben. - Geben Sie 700 μl Waschpuffer in die Säule und zentrifugieren Sie 15 s lang bei ≥8000 x g . Verwerfen Sie den Durchfluss.
- Fügen Sie 500 μl Puffer RPE hinzu und zentrifugieren Sie erneut bei ≥8000 x g für 15 s. Verwerfen Sie den Durchfluss.
- 500 μl 80%iges Ethanol zugeben und bei ≥8000 x g für 2 min zentrifugieren. Verwerfen Sie den Durchfluss.
- Trocknen Sie zu diesem Zeitpunkt die Säulenmembran, indem Sie die Säulen bei voller Geschwindigkeit (>8000 x g) 5 Minuten lang bei geöffnetem Deckel zentrifugieren.
- Übertragen Sie die Säule in ein neues Sammelröhrchen, geben Sie 14 μl RNase-freies Wasser direkt auf die Säulenmembran und zentrifugieren Sie 1 Minute lang bei ≥8000 x g , um die Gesamt-RNA aus BDsEVs zu eluieren.
- Quantifizieren Sie die gesamte microRNA mit Proben unter Verwendung eines miRNA-Quantifizierungs-Assay-Kits, bei dem die Proben auf einem Quantifizierungsfluorometer gemessen wurden, wobei der miRNA-Assay-Typ ausgewählt wurde.
9. Sequenzierung kleiner RNAs
- Nach der Quantifizierung der miRNA in der Probe synthetisieren Sie eine cDNA-Bibliothek mit dem Small RNA-Seq Library Prep Kit.
HINWEIS: Bei Verwendung dieses Protokolls umfasste die Bibliotheksgenerierung vier Schritte: 3'-Adapterligation, Aufreinigung, 5'-Adapterligation und reverse Transkription - wobei am Ende eine RNA/cDNA-Bibliothek gebildet wird. Nach der Generierung der Bibliothek erfolgt die Endpunkt-PCR-Amplifikation, bei der Indexprimer hinzugefügt werden (die für Multiplexzwecke verwendet werden), wonach die Proben aufgereinigt werden. - Um die RNA-Qualität zu testen, verwenden Sie einen Qualitätskontroll-Anwendungsassay, in dem die Größe jedes Präparats und die Spitzenintensität aufgeführt sind.
- Um die DNA-Konzentration zu testen, messen Sie die DNA-Quantifizierung auf einem Quantifizierungsfluorometer und wählen Sie den dsDNA-Assay-Typ aus.
- Bereiten Sie die Proben mit dem Reagenzienkit für die Small RNA Seq vor, wonach die Proben in die Durchflusszelle geladen werden.
Ergebnisse
Um das Vorhandensein von BDsEVs zu bestätigen, wurden drei Techniken verwendet: Western Blotting, NTA und TEM (Abbildung 3). Die Western-Blot-Ergebnisse (Abbildung 3A und ergänzende Datei 1) zeigen das Vorhandensein aller fünf positiven Marker (CD9, CD63, CD81, Flot-1 und TSG101) und das Fehlen von Calnexin in sEVs (als Negativkontrolle verwendet), was bestätigt, dass keine Kontamination mit zellulärem Inhalt vorliegt. Erwartungsgemäß weisen die Gehirnhomogenate (BH) mehr Protein auf, als in den sEVs für die getesteten Marker beobachtet wurde. TEM-Bilder bestätigten die Morphologie der BDsEVs und ihre relative Größe innerhalb der Probe (Abbildung 3B), was durch eine Nanopartikel-Tracking-Analyse weiter bestätigt wurde (Abbildung 3C).
Abbildung 3C (oberes Bild) zeigt die isolierten Partikelgrößen, die zwischen 50 und 200 nm liegen. Aus den NTA-Daten, die das Streuergebnis als 100 % der dargestellten Partikel annehmen, repräsentiert CMO etwa 52,35 % der Partikel (Abbildung 3C, unteres Bild), die eine Lipiddoppelschicht aufweisen. Von diesen CMO-gefärbten Partikeln enthielten 34,66 % CD9, 15,49 % CD63 und 12,01 % CD81. Informationen zu den NTA-Steuerdaten finden Sie in der Ergänzungsdatei 2 .
Die Quantifizierungsdaten der isolierten RNA (Tabelle 2) zeigen Werte zwischen 3,24 und 8,76 (ng/μL), Werte, die bei der Generierung der cDNA-Bibliotheken normalisiert wurden. Die Analyseergebnisse von TapeStation (Abbildung 4) zeigen große Spitzen von etwa 120-160 bp, mit kleineren Spitzen darunter von etwa 50 bp und anderen kleineren Spitzen, die höher bei etwa 484 bp liegen.
Qualitätsprüfungen für die Next-Generation-Sequencing-Daten zeigen nach dem Trimmen des Adapters eine sehr hohe Qualität der Lesevorgänge (Abbildung 5A). Alle Sequenzpositionen weisen einen Phred-Score von >30 auf, was auf Fehlerraten von weniger als 1 zu 1000 Nukleotiden hinweist. Abbildung 5B zeigt, dass die meisten Sequenzen etwa 21-22 bp groß sind, was dem erwarteten Größenbereich für microRNAs entspricht. Durch weitere nachgelagerte Kartierung (unter Verwendung von BowTie2) unter Verwendung des Flagstat-Analyse-Biotools Samtools wurden von 1.304.100 ausgerichteten Sequenzen 1.116.264 (85%) auf die microRNA-Regionen im menschlichen Genom kartiert (build hg38; Tabelle 3).
Von den gesamten miRNAs wurden 808 miRNAs in allen 3 Proben exprimiert, wobei 344 miRNAs auf alle drei Proben (~43 %), 5 auf Kontrolle 1 und Kontrolle 2, 183 auf Kontrolle 1 und Kontrolle 3 und 16 auf Kontrolle 2 und Kontrolle 3 aufgeteilt wurden (Abbildung 6). Aus diesen Daten geht hervor, dass die am höchsten exprimierten miRNAs über alle drei BDsEVs has-let-7b-5p, has-miR-143-3p, has-miR-30a-5p, has-miR-221-3p und has-let7i-5p sind. Ebenso sind die Top 5 der am wenigsten differentiell exprimierten miRNA in BDsEVs-Proben (Reads >10) has-miR-128-2-5p, has-miR-182-5p, has-miR-193b-5p, has-miR-448 und has-miR-505-3p. Alle rohen und normalisierten Lesezahlen sind in der Zusatzdatei 3 angegeben.

Abbildung 1: Arbeitsablauf bei der Gewebeverarbeitung. Die Abbildung zeigt den schrittweisen Prozess zur Isolierung von aus dem Gehirn stammenden sEVs (BDsEVs) aus frisch gefrorenen Hirnschnitten mittels Größenausschlusschromatographie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Arbeitsablauf bei der RNA-Isolierung aus BDsEVs. Die Abbildung zeigt den schrittweisen Prozess der Isolierung der Gesamt-RNA aus den BDsEVs, einschließlich des Vorbehandlungsschritts der Entfernung extrazellulärer RNA mit Proteinase K und RNase A. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
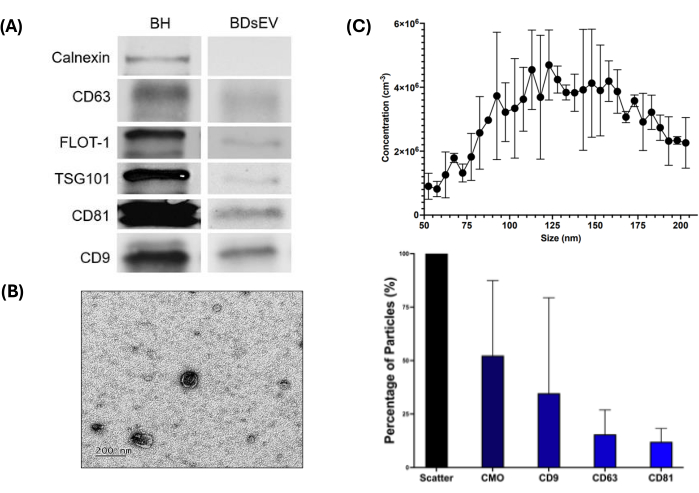
Abbildung 3: Arbeitsablauf für die Charakterisierung von BDsEV. Die Abbildung zeigt verschiedene Methoden zur Charakterisierung der BDsEVs. (A) Western Blot zum Nachweis von sEV-spezifischen Proteinmarkern. BH Gehirn homogenat. Calnexin ist eine Negativkontrolle für sEVs - das Fehlen in sEV und das Vorhandensein in BH bestätigen die Reinheit der sEV-Probe. Bei anderen Markern ist die Proteinmenge in BH erwartungsgemäß höher als in den sEV-Proben (20 μg Protein pro Well). (B) Repräsentatives Transmissionselektronenmikroskopie-Bild (TEM) für BDsEVs im 200 μM-Maßstab, das die erwartete Morphologie mit einer Lipid-Doppelschichtmembran bestätigt. (C) Größenverteilungsdiagramm für die Nanopartikel-Tracking-Analyse (NTA) für alle Partikel (oberes Bild). Jeder Datenpunkt stellt die Variation über drei Replikate dar. Normalisierte Anteile für biologische Partikel (sEVs) und relative Anteile für spezifische Oberflächenmarker (unteres Bild) (Daten als Mittelwert ± SEM von dreifachen Vertiefungen (n = 3)). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Qualitätsprüfung für die Vorbereitung von cDNA-Bibliotheken. Tapestation-Analyse für Einzelproben nach der Erstellung der cDNA-Bibliothek. Die in jedem Diagramm angezeigten Peaks zeigen die Größe jeder Präparation (horizontale Achse, in bp) und die jeweilige Intensität jedes Peaks (vertikale Achse, FU) an. Die erwartete kleine RNA-Bibliothek erreicht ihren Höhepunkt bei etwa 120-160 bp, wie in der Abbildung zu sehen ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Qualitätsprüfung für die miRnome-Sequenzierung. (A) FASTQC-Plot für die Lesequalität der einzelnen Proben nach dem Adaptertrimmen. Die horizontale Achse ist für die Nukleotidposition und die vertikale Achse für die Phred-Scores. Boxplots für jede Position zeigen die Verteilung des Phred-Scores über alle Lesevorgänge für jede Position. Alle Verteilungen innerhalb des grünen Bereichs bestätigen sehr hochwertige Daten (>Q30), was auf eine qualitativ hochwertige Probenvorbereitung hindeutet. (B) Die Sequenzlängenverteilung für jede Probe zeigt, dass bei allen drei Proben die Mehrheit der Reads bei etwa 21-22 Nukleotiden lag - dem erwarteten Größenbereich für microRNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Überlappung der MicroRNA-Expression über alle Proben hinweg. Die Anzahl der miRNAs, die aus jeder Probe nachgewiesen wurden (Lesezahl ≥ 10) und der spezifischen microRNAs, die in mehr als einer Probe geteilt werden, sind im Venn-Diagramm dargestellt. Alle rohen und normalisierten Lesevorgänge werden in der Zusatzdatei 3 angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Antikörper-Epitop | Verdünnungsfaktor des Antikörpers |
| Maus Anti-CD9 | 1:2000 |
| Maus Anti-CD63 | 1:2000 |
| Maus Anti-CD81 | 1:2000 |
| Maus Anti-Flotillin 1 | 1:2000 |
| Maus Anti-TSG101 | 1:1000 |
| Maus Anti-Calnexin | 1:10 000 |
| Anti-Maus-Sekundärantikörper | 1:3000 |
Tabelle 1: Antikörper-Epitope, die zur Charakterisierung von BDsEVs verwendet werden, und ihre jeweiligen Verdünnungsfaktoren.
| Probe | miRNA-Konzentration (ng/μL) |
| 1 | 8.76 |
| 2 | 3.24 |
| 3 | 5.33 |
Tabelle 2: Quantifizierungsdaten von isolierter RNA.
| Probe | Ausgerichtete Sequenzen | Kartierte miRNA | Prozentsatz (%) |
| Kontrollprobe 1 | 1 444 665 | 1 133 437 | 78.46 |
| Kontrollprobe 2 | 442 808 | 381 631 | 86.18 |
| Kontrollprobe 3 | 2 024 828 | 1 983 726 | 97.97 |
| Durchschnitt | 1 304 100.33 | 1 166 264.67 | 89.43 |
Tabelle 3: Anzahl der ausgerichteten und erfolgreich kartierten miRNA-Sequenzen.
Ergänzende Datei 1: Ergänzende Daten zur Western-Blot-Analyse. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: Ergänzende NTA-Kontrolldaten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: Rohe und normalisierte Lesezahlen der miRNA-Expressionsanalyse. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: Daten zur ergänzenden Proteinkonzentration. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Dieses modifizierte und verbesserte Protokoll zur Isolierung von kleinen extrazellulären Vesikeln aus dem Gehirn und ihrer microRNA-Fracht demonstriert die Machbarkeit, minimales Gewebe zu verwenden, ohne die Qualität und Quantität der nachgelagerten Produkte zu beeinträchtigen. Im Bereich der Biomarker-Entdeckung kann die Identifizierung molekularer Identifikatoren, die spezifisch für Zell- und Gewebetypen sind, zu leichter zugänglichen Mitteln für nicht-invasive diagnostische Tests mit Körperflüssigkeiten führen. Darüber hinaus bietet der hier beschriebene Ansatz den Rahmen für die Identifizierung spezifischer sEV-Subpopulationen innerhalb verschiedener Zelltypen (neuronal vs. glial), Gewebetypen (Hippocampusgewebe vs. präfrontales Kortexgewebe) und Bioflüssigkeiten - was ein besseres Verständnis der Frachtlastsignaturen, der Pathophysiologie und des Krankheitsausmaßes ermöglicht. Zuvor wurde in Vella et al. vorgeschlagen, dass 450 mg bis 1 g für die BDsEV-Isolierung in Verbindung mit einer Dichtegradienten-Ultrazentrifugation (mit Geschwindigkeiten von bis zu 180.000 x g) verwendet werden sollten8. Bei dieser Methode wurden sEVs erfolgreich mit geringeren Mengen (>50 % Reduktion) von frisch gefrorenen Hirngewebeproben (250-450 mg) isoliert. Die Reduzierung der Menge an Hirngewebe, die zur Isolierung von sEVs verwendet wird, führt zu reduzierten Kosten und Verbrauchsmaterialien. Ebenso wurde das Protokoll durch die Anwendung von SEC in Verbindung mit Hochgeschwindigkeitszentrifugation (10.000 x g) anstelle von Ultrazentrifugation (180.000 x g) zugänglicher gemacht.
Unter Verwendung von fNTA und TEM stellten wir fest, dass das Größenbereichsprofil der sEVs in den charakteristischen Größenbereich von 30-200 nm fällt, wie in den MISEV-Richtlinien13,14 empfohlen, wobei die TEM eine morphologische Bestätigung der erhaltenen sEVs liefert. Wie in Abbildung 3C und in der Zusatzdatei 4 gezeigt, haben >87 % der Partikel einen Größenbereich von <250 nm, und die Proben zeigen im Durchschnitt 10,56 fg Protein/BDsEV. Bestimmte Spezies kleiner extrazellulärer Vesikel variieren in Größe und Form, wobei charakteristischerweise kleinere Subpopulationen, wie z. B. Exosomen, eine einheitliche Morphologie im Vergleich zu Mikrovesikeln aufweisen, von denen gesagt wird, dass sie aufgrund ihrer Art der Biogenese eine unregelmäßige Struktur aufweisen15. Für die fNTA wurde ein Streuwert (520 nm) erstellt, um die Gesamtpartikel in den Proben zu analysieren und Details zum Größenprofil und zu den Konzentrationen zu liefern. Die Messwerte zeigen, dass über 50 % der Partikel biologisch waren (durch die Färbung von CMO), wobei Populationen biologischer Partikel, die in den Größenbereich von sEVs fallen, Tetraspanin-Marker aufwiesen, wobei CD9 in unseren Kontrollproben am stärksten vorherrschend war (34,66 %). Während beim Western Blot das Vorhandensein charakteristischer Membran- und zytosolischer Proteinladungen einen Hinweis auf das Vorhandensein von sEV lieferte, wobei das Fehlen von Calnexin bestätigte, dass BDsEVs frei von jeglicher zellulärer Kontamination sind. Um weiter sicherzustellen, dass die für die nachgelagerte Verarbeitung verfügbare miRNA Teil der sEV-Fracht (und keine andere extrazelluläre RNA) ist, behandelten wir die sEVs mit Proteinase K- und RNase A-Enzymen, bevor wir die sEV-Membranen aufbrachen, um alle Proteine und RNA abzubauen, die extern im extrazellulären Raum außerhalb der BDsEVs vorhanden waren16.
Die Gesamt-RNA wurde aus den sEVs extrahiert und für nachgelagerte Anwendungen wie qRT-PCR und Next-Generation-Sequencing (für kleine RNA) vorbereitet. Standardkontrollen mit Qualitätskontroll-Assays zeigten qualitativ hochwertige Proben und cDNA-Bibliotheken, die für die Sequenzierung bereit waren. Die cDNA-Bibliotheken erreichen ihren Höhepunkt bei etwa 120-160 bp, was auf miRNA-abgeleitete cDNA-Peaks mit Adaptersequenzen unterschiedlicher Länge zurückzuführen ist, wobei kleinere Peaks möglicherweise Adapterdimere darstellen.
Die Analyse der Next-Generation-Sequencing-Daten mit FASTQC und CutAdapt zeigt qualitativ hochwertige Ergebnisse, die mit BDsEV-miRNAs erzielt wurden. Wie in Abbildung 5 gezeigt, hat jede gelesene Position einen Q-Wert von mehr als 30 (Q30), was eine Fehlerwahrscheinlichkeit von 0,001-0,0001 (1 zu 1000-10.000) zeigt. Darüber hinaus zeigt die Analyse der Sequenzlängenverteilung (Abbildung 6) vorherrschende Peaks, die innerhalb des charakteristischen Bereichs der miRNA (19-25 bp) liegen, was auf eine hohe Qualität und Spezifität der erhaltenen Daten hindeutet.
Zahlreiche zukünftige Studien werden von dieser Methode profitieren, da sie besser zugänglich ist und Kosten und Ressourcen spart, indem nicht nur die Menge der Gewebeproben, sondern auch die Kosten für Reagenzien eingespart werden. Die Reduzierung (>50%) der benötigten Gewebemenge macht diese Methode auch ethischer. Im Einklang mit den globalen Bemühungen im Bereich der Präzisionsmedizin bietet der hier beschriebene verbesserte Arbeitsablauf vergleichsweise kostengünstigere Werkzeuge, um genaue zelltypspezifische Biomarker über nicht-invasive (oder minimal-invasive) Wege mit BDsEVs und ihrer Fracht zu entwickeln. Dies kann bei einer Vielzahl von neurologischen Erkrankungen eingesetzt werden. Eine mögliche Einschränkung dieser Methode besteht darin, dass das Protokoll angepasst werden muss, wenn Forscher ein anderes Gewebe als das menschliche Gehirn für die sEV-Isolierung verwenden möchten. Langfristig wird diese verbesserte Methode die nicht-invasive, gewebespezifische Entdeckung und Validierung von Biomarkern gerechter und weltweit zugänglicher machen.
Offenlegungen
Es gibt keine Interessenkonflikte für einen der Autoren.
Danksagungen
Diese Arbeit wurde durch das Doktorandenstipendium für Joseph Morgan der Alzheimer's Society UK (Fördernummer 549/SERA-52) und durch die Innovationsstrategiefonds der Universität Salford (Fördermittel SEFA-39) finanziert. Das Hirngewebe wurde aus der Gehirnbank Manchester (REC Reference 09/H0906/52) des Brains for Dementia Network gewonnen.
Materialien
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin | Merck | A9418-100G | |
| Cell Mask Orange Plasma Membrane Stain | ThermoFisher Scientific | C10045 | |
| Collagenase Type-III | StemCell Technologies | 07422 | |
| ExoSpin Columns and Buffer | Cell Guidance Systems Ltd. | EX01-50 | This kit contains SEC columns used in this experiment, precipitation buffer and EV free PBS. |
| Halt Protease Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78429 | |
| Hibernate-E Medium | ThermoFisher Scientific | A1247601 | |
| Laemmli Sample Buffer (4x) | BioRad | 1610747 | |
| Lexogen Small RNA-Seq Library Prep Kit | Lexogen | 052.24 | This kit contains Small RNA preparation reagent box with i7 Index primer plate. |
| miRNeasy Micro Kit (50) | Qiagen | 217084 | This kit contains high-quality RNA recovery lysis reagent (Qiazol), RNA isolation columns, isolation buffers (RWT, RPE) and RNase free water. |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3001 | This is the Illumina Preparation Kit |
| Nitrocellulose Membrane, 0.45 μm | ThermoFisher Scientific | 88018 | |
| PE/Dazzle 594 anti-human CD63 Antibody | BioLegend | 143914 | Used for fNTA Analysis |
| PE/Dazzle 594 anti-human CD81 Antibody | BioLegend | 349520 | Used for fNTA Analysis |
| PE/Dazzle 594 anti-human CD9 Antibody | BioLegend | 312118 | Used for fNTA Analysis |
| PhosSTOP | Merck | 4906845001 | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23225 | |
| Proteinase K | ThermoFisher Scientific | 25530049 | |
| Qubit microRNA Assay Kit | ThermoFisher Scientific | Q32880 | |
| Qubit 1X dsDNA HS assay kit | ThermoFisher Scientific | Q33230 | |
| Qubit 3.0 Fluorometer | ThermoFisher Scientific | Q33216 | |
| RIPA Lysis and Extraction Buffer | ThermoFisher Scientific | 89901 | |
| RNase A | ThermoFisher Scientific | EN0531 | |
| SuperSignal West Femto Maximum Sensitivity Substrate | ThermoFisher Scientific | 34094 |
Referenzen
- Jia, Y., et al. Small extracellular vesicles isolation and separation: Current techniques, pending questions and clinical applications. Theranostics. 12 (15), 6548-6575 (2022).
- Fornari, S., Schäfer, A., Jucker, M., Goriely, A., Kuhl, E. Prion-like spreading of Alzheimer's disease within the brain's connectome. J R Soc Interface. 16 (159), 20190356(2019).
- Zhang, P., et al. Tumor-derived small extracellular vesicles in cancer invasion and metastasis: molecular mechanisms, and clinical significance. Mol Cancer. 23 (1), 18(2024).
- Constantinides, V., et al. CSF Aβ42 and Aβ42/ Aβ40 ratio in Alzheimer's disease and frontotemporal dementia. Diagnostics. 13 (4), 783(2023).
- Cheng, L., et al. Prognostic serum miRNA biomarkers associated with Alzheimer's disease shows concordance with neuropsychological and neuroimaging assessment. Mol Psychiatry. 20 (10), 1188-1196 (2015).
- Roser, A. E., Caldi Gomes, L., Schünemann, J., Maass, F., Lingor, P. Circulating miRNAs as diagnostic biomarkers for Parkinson's disease. Front Neurosci. 12, 625(2018).
- Jackson, N., Guerrero-Muñoz, M. J., Castillo-Carranza, D. L. The prion-like transmission of tau oligomers via exosomes. Front Aging Neurosci. 14, 974414(2022).
- Vella, L., et al. A rigorous method to enrich for exosomes from brain tissue. J Extracell Vesicles. 6 (1), 1348885(2017).
- Yousif, G., Qadri, S., Parray, A., Akhthar, N., Shuaib, A., Haik, Y. Exosomes derived neuronal markers: Immunoaffinity isolation and characterization. Neuromolecular Med. 24 (3), 339-351 (2022).
- Kumar, K., et al. Recent advances in microfluidic approaches for the isolation and detection of exosomes. TRAC-Trend Anal Chem. 159, 116912(2023).
- Ransom, L., et al. Human brain small extracellular vesicles contain selectively packaged, full-length mRNA. Cell Rep. 43 (4), 114061(2024).
- Gomes, P., et al. A novel isolation method for spontaneously released extracellular vesicles from brain tissue and its implication for stress-driven brain pathology. Cell Commun Signal. 21 (1), 35(2023).
- Welsh, J., et al. Minimal information for studies of extracellular vesicles (MISEV 2023): From basic to advanced approaches. J Extracell Vesicles. 13 (2), e12404(2023).
- Théry, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750(2018).
- Pirisinu, M. The long journey of extracellular vesicles towards global scientific acclamation. Adv Pharm Bull. 13 (3), 489-501 (2023).
- Bender, A., Sullivan, B. P., Lillis, L., Posner, J. D. Enzymatic and chemical-based methods to inactivate endogenous blood ribonucleases for nucleic acid diagnostics. J Mol Diagn. 22 (8), 1030-1040 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten