Method Article
Aprovechamiento del poder de las cargas de microARN en pequeñas vesículas extracelulares liberadas de secciones de cerebro humano recién congeladas
En este artículo
Resumen
Aquí, establecemos un protocolo utilizando cantidades mínimas de secciones de tejido cerebral recién congelado y un método de centrifugación de alta velocidad accesible junto con cromatografía de exclusión por tamaño para obtener pequeñas vesículas extracelulares como fuentes de biomarcadores de microARN (miARN) para trastornos neurológicos.
Resumen
Las pequeñas vesículas extracelulares (sEV) son mediadores cruciales de la comunicación célula-célula, transportando diversas cargas como proteínas, lípidos y ácidos nucleicos (microARN, ARNm, ADN). La carga de microARN sEV tiene una utilidad potencial como un poderoso biomarcador de enfermedad no invasiva debido a la capacidad de sEV para atravesar barreras biológicas (por ejemplo, barrera hematoencefálica) y ser accesible a través de varios fluidos corporales. A pesar de los numerosos estudios sobre los biomarcadores de sEV en los fluidos corporales, la identificación de subpoblaciones de sEV específicas de tejidos o células sigue siendo un desafío, particularmente desde el cerebro. Nuestro estudio aborda este desafío mediante la adaptación de los métodos existentes para aislar sEV de cantidades mínimas de secciones de cerebro humano congeladas utilizando cromatografía de exclusión por tamaño (SEC).
Después de la aprobación ética, se cortaron aproximadamente 250 μg de tejido cerebral humano fresco congelado (obtenido del Banco de Cerebros de Manchester [Reino Unido]) de los 3 tejidos donantes y se incubaron en una solución de colagenasa tipo 3/Hibernate-E, con agitación intermedia, seguida de pasos de centrifugación y filtración en serie. A continuación, se aislaron los sEV mediante el método SEC y se caracterizaron siguiendo las directrices de MISEV. Antes de aislar el ARN del interior de estos sEV, la solución se trató con proteinasa-K y RNasa-A para eliminar cualquier ARN extracelular no sEV. La cantidad y la calidad del ARN se comprobaron y se procesaron posteriormente para experimentos de qPCR y secuenciación de ARN pequeño.
La presencia de sEVs se confirmó mediante análisis de seguimiento de nanopartículas de fluorescencia (fNTA) y Western blot para marcadores de superficie (CD9, CD63, CD81). La distribución del tamaño (50-200 nm) se confirmó mediante NTA y microscopía electrónica. La concentración total de ARN dentro de los sEV lisados osciló entre 3 y 9 ng/μL y se utilizó para la cuantificación exitosa mediante qPCR para los microARN candidatos seleccionados. La secuenciación de ARN pequeño en MiSeq proporcionó datos de alta calidad (Q >32) con 1,4-5 millones de lecturas por muestra.
Este método permite el aislamiento y la caracterización eficientes de los sEV a partir de volúmenes mínimos de tejido cerebral, lo que facilita la investigación no invasiva de biomarcadores y es prometedor para los estudios equitativos de biomarcadores de enfermedades, ya que ofrece información sobre enfermedades neurodegenerativas y potencialmente otros trastornos.
Introducción
Las vesículas extracelulares (VE) son uno de los actores clave de la comunicación intercelular en todos los organismos multicelulares1. Los EV son partículas de membrana bicapa lipídica derivadas de células que pueden facilitar la transferencia de una variedad de cargas de carga, como proteínas, lípidos y ácidos nucleicos, a las células receptoras. Los VE pueden tener una amplia gama de tamaños que van desde 30 nm hasta 1 μM. Los VE pequeños (sEV), definidos como vesículas unidas a lípidos con un diámetro promedio de <200 nm, tienen la capacidad de cruzar la barrera hematoencefálica; por lo tanto, se han implicado en la propagación y exacerbación de enfermedades neurodegenerativas y otras afecciones como la enfermedad de Alzheimer (EA), la demencia frontotemporal (DFT), la enfermedad de Parkinson (EP) y los cánceres 2,3. Además, dado que los sEV se pueden encontrar en una variedad de biofluidos como la sangre, el líquido cefalorraquídeo (LCR), la saliva e incluso la orina, su uso beneficioso puede abarcar el campo de los biomarcadores y el diagnóstico no invasivo. Por ejemplo, la investigación de la EA puede apuntar al uso de proporciones de proteínas patógenas biofluidas, como tau/Aβ, o incluso Aβ42/Aβ404.
Un cargamento conocido de sEV es el microARN (miARN), un grupo de pequeñas moléculas de ARN no codificantes de alrededor de 22 nucleótidos de longitud que se unen a las regiones 3'-UTR del ARNm y generalmente regulan negativamente la expresión de proteínas. Implicados en muchas funciones celulares, los miARN también se han implicado en la patogénesis de diversas enfermedades, incluidos los cánceres y las enfermedades neurodegenerativas. Cheng et al. llevaron a cabo un análisis de secuenciación de alto rendimiento de las firmas de expresión de miARN sEV derivadas del suero de pacientes con EA5. Una vez combinados con los registros de neuroimagen y los factores de riesgo conocidos de la edad, el sexo y la presentación del alelo APOE ε4, se encontró que los resultados predicen la EA con una sensibilidad del 87% y una especificidad del 77%. Además, la investigación ha identificado dos miRNAs de LCR regulados al alza (miR-151a-3p, let-7f-5p) y 3 miRNAs regulados a la baja (miR-27a-3p, miR-125a-5p y miR-423-5p) que potencialmente pueden diagnosticar la EP6 en etapa temprana. En las enfermedades patológicas, el estado patológico puede preceder a ciertos síntomas característicos de las enfermedades, mientras que en la neurodegeneración, la acumulación de características patológicas se produce mucho antes que el deterioro cognitivo.
Los microARN son potencialmente un biomarcador más eficaz que las proteínas, debido a sus diversas funciones y a su regulación epigenética de orden superior. Utilizando tejido cerebral, los investigadores pueden identificar potencialmente firmas específicas de miARN de sEV (BDsEV) derivadas del cerebro para enfermedades y sus subtipos. Por ejemplo, los sEV con marcadores neuronales y gliales pueden presentar diferentes cargas de miARN, y el análisis puede dar lugar a métodos más precisos de detección de enfermedades. Además, se sugiere que las BDsEV desempeñan un papel importante en la propagación transsináptica de proteínas neuropatógenas7. Informes previos han sugerido inmunoprecipitación y ultracentrifugación en gradiente de densidad (gradiente de sacarosa) para obtener sEVs a partir de tejido cerebral fresco congelado 8,9. Sin embargo, estos enfoques requieren una infraestructura específica con ultracentrífuga y métodos de purificación posteriores para obtener muestras de sEV de alta calidad10. Informes más recientes han sugerido varias modificaciones y mejoras al enfoque 11,12,13; sin embargo, a pesar de esto, el aislamiento y el estudio de los microARN derivados de sEV a partir de tejidos humanos aún no se aplican ampliamente. El enfoque descrito en este protocolo tiene como objetivo proporcionar un protocolo refinado y paso a paso para el estudio de sEV derivado del cerebro para mejorar la accesibilidad a esta técnica. Establecimos un protocolo utilizando cantidades mínimas de tejido cerebral, a partir del cual aislamos sEV puros mediante cromatografía de exclusión por tamaño y mostramos datos de secuenciación de próxima generación de alta calidad a partir de la carga de microARN de estos sEV.
Protocolo
El trabajo ha sido aprobado éticamente por el Manchester Brain Bank (referencia REC 09/H0906/52) y por el comité de ética de la Universidad de Salford (ID de solicitud: 3408).
1. Ruptura de la matriz intracelular mediante colagenasa en secciones de tejido cerebral congelado
- Cortar en rodajas finas 250 mg de tejido cerebral congelado (fragmentos de alrededor de 0,4 mm de grosor) con un bisturí esterilizado en una placa fría y añadir 2 mL de solución de colagenasa tipo III/Hibernación E de 75 U/mL.
- Incubar cada muestra en un baño de agua a 37 °C durante 20 min. En el punto de 5 minutos, invierta cada muestra dos veces para mezclar; en el punto de 10 minutos, pipetee cada muestra tres veces suavemente con una tira desechable de plástico de 10 ml.
- Coloque cada muestra en hielo y agregue 1x cóctel de inhibidores de proteasa y 1x inhibidor de fosfatasa. Esto detiene la reacción enzimática y es el punto final de la digestión utilizando colagenasa tipo III.
- Centrifugar cada muestra de tejido cerebral a 300x g durante 10 min a 4 °C. Recoja cada sobrenadante y centrifuga a 2000 x g durante 15 min a 4 °C.
- Vuelva a recoger cada sobrenadante y filtre a través de un filtro de 0,22 μm. Centrifugar cada filtrado a 10 000 x g durante 48 min a 4 °C.
- Recoja el sobrenadante y agregue tampón de precipitación en una proporción de 2:1 a cada muestra. Incubar durante la noche a 4 °C.
- Centrifugar cada muestra a 10 000 x g durante 96 min a 4 °C y resuspender el pellet en 100 μL de PBS libre de vesículas extracelulares (EV).
2. Preparación de columnas de cromatografía de exclusión por tamaño
- Antes de usar, equilibre la columna SEC preparada previamente a temperatura ambiente (RT) durante 15 min.
NOTA: La tapa inferior debe quitarse antes que la tapa de rosca. - Después del equilibrio, retire el tampón conservante de la parte superior de la columna y lave la columna dos veces con 250 μL de PBS sin EV. Cada lavado de PBS se deja entrar en la columna por gravedad.
3. Aislamiento de pequeñas vesículas extracelulares mediante cromatografía de exclusión por tamaño
- Añada el pellet resuspendido a la parte superior de la columna SEC y centrifugue la columna a 50 x g durante 30 s. Deseche el flujo continuo.
- Agregue 180 μL de PBS libre de EV a la parte superior de la columna SEC y centrifugue la columna a 50 x g durante 1 minuto, permitiendo que los sEV derivados del cerebro eluyan de la columna.
NOTA: La parte del protocolo detallada anteriormente se resume en la Figura 1.
4. Confirmación de marcadores de vesículas extracelulares pequeñas mediante Western blot
- Para lisar las sEV antes del análisis de Western blot (para garantizar que se analicen todas las cargas de membrana y citosólicas), diluya la muestra de sEV aislada 1:1 en tampón de lisis y extracción (1x inhibidor de la proteasa y 1x inhibidor de la fosfatasa).
- Agitar la muestra a 4 °C durante 30 min.
- Centrifugar la muestra a 14 000 x g durante 15 min.
- Recoja el sobrenadante de proteínas y mida las concentraciones de proteínas utilizando un kit de ensayo de proteínas.
- Mezcle las muestras con 4 tampones Laemmli y cargue 20 μg de proteína en cada pocillo contra una escalera de proteínas previamente teñida.
- Una vez resueltas, transfiera las proteínas a una membrana de nitrocelulosa de 0,45 μm. Después de la transferencia, bloquee la membrana durante 2 h en BSA al 5%.
- Incubar las membranas con anticuerpos primarios (Tabla 1) durante la noche.
- Lavar la membrana tres veces con tampón de lavado (1% Tween20 en PBS) y teñir con un anticuerpo secundario anti-ratón. Después, lavar tres veces con tampón de lavado (1% Tween20 en PBS).
- Siga los pasos anteriores en el manuscrito antes de obtener imágenes de las manchas.
- Imagen utilizando sustrato de peroxidasa de rábano picante (HRP).
5. Confirmación de pequeñas vesículas extracelulares mediante análisis de seguimiento de nanopartículas (NTA)
- Realice NTA para analizar la concentración y el tamaño de todas las partículas de la muestra. Para ello, diluya cada muestra en PBS con un factor de dilución de 1:1000.
- Inyecte la muestra en el instrumento NTA y lea la muestra a una longitud de onda de 550 nm. En esta etapa, use la cantidad de partícula para medir la cantidad de proteína por partícula (generalmente expresada en femtogramo) como se recomienda en las pautas de MISEV.
- A continuación, para NTA fluorescente, diluya la mascarilla celular naranja (CMO) (tiñe todas las partículas biológicas dentro de una muestra) en PBS por un factor de dilución de 1:1000. A partir de esto, diluya el colorante CMO usando una muestra de sEV por un factor de dilución de 1:10 - Incube este paso de dilución en la oscuridad durante 30 min a 4 °C.
- Diluir la segunda dilución de CMO usando PBS en un factor de dilución de 1:1000.
NOTA: La dilución final del tinte CMO debe ser 1:10 000 000. - Lea el CMO de cada muestra de sEV a una longitud de onda de 550 nm utilizando el instrumento NTA.
- Para los anticuerpos fluorescentes de tetraspanina (ver Tabla de Materiales), en primer lugar, diluya cada anticuerpo respectivo en PBS por un factor de dilución de 1:10. A partir de esto, diluya cada uno de los tintes usando una muestra de sEV por un factor de dilución de 1:10 - Incubar el paso de dilución en la oscuridad durante 2 h a 4 °C.
- Diluir la segunda dilución de anticuerpos usando PBS en un factor de dilución de 1:1000.
NOTA: La dilución final de cada anticuerpo de tetraspanina debe ser de 1:100 000. - Lea las lecturas de anticuerpos fluorescentes de la muestra de sEV a una longitud de onda de 550 nm utilizando el instrumento NTA.
6. Confirmación de pequeñas vesículas extracelulares por microscopía electrónica de transmisión (TEM)
NOTA: Este protocolo fue realizado por el Centro de Microscopía Biomédica de la Universidad de Liverpool.
- En primer lugar, fije las muestras de sEV con glutaraldehído y tiña con acetato de uranilo.
- A través de una serie graduada de alcoholes, deshidrate la muestra y prepárela para TEM.
- Imágenes de muestras utilizando un microscopio electrónico de transmisión adecuado.
7. Tratamiento con proteinasa K y RNasa A
- A 50 μL de EV derivadas del cerebro aisladas, añadir 1 μL de 20 μg/μL de proteinasa K e incubar a 37 °C durante 30 min.
- Detenga cada reacción agregando 1x cóctel de inhibidores de proteinasa e incube en hielo durante 10 minutos.
- Después de la incubación, añadir 1 μL de 10 μg/μL de RNasa A a cada muestra y seguir incubando a 37 °C durante 30 min.
NOTA: Pase inmediatamente a la sección 8.
8. Aislamiento de ARN total a partir de pequeñas vesículas extracelulares
- Para aislar el ARN total, agregue 700 μL de reactivo de lisis de recuperación de ARN de alta calidad a 50 μL de muestra de EV derivado del cerebro (BDsEV). Agite cada muestra durante 1 h.
- Añadir 140 μL de cloroformo, agitar enérgicamente cada muestra durante 20 s y dejar incubar a RT durante 3 min.
- Centrifugar la muestra a 12 000 x g durante 15 min a 4 °C y recoger la interfase de la muestra. Agregue 1,5 veces el volumen de etanol al 100%.
- Añadir 700 μL de mezcla de interfase/etanol, columnas de aislamiento de ARN y centrífuga a ≥8000 x g durante 15 s a RT.
NOTA: Repita este paso utilizando todas las muestras de interfase/etanol. - A la columna, añadir 700 μL de tampón de lavado y centrifugar a ≥8000 x g durante 15 s. Deseche el flujo continuo.
- Añadir 500 μL de TPE RPE y volver a centrifugar a ≥8000 x g durante 15 s. Deseche el flujo continuo.
- Agregue 500 μL de etanol al 80% y centrifugue a ≥8000 x g durante 2 min. Deseche el flujo continuo.
- En este punto, seque la membrana de la columna centrifugando las columnas a toda velocidad (>8000 x g) durante 5 min con la tapa abierta.
- Transfiera la columna a un nuevo tubo de recolección, agregue 14 μL de agua libre de ARNasa directamente a la membrana de la columna y centrifugue a ≥8000 x g durante 1 minuto para eluir el ARN total de los BDsEV.
- Cuantificar el microARN total con muestras utilizando un kit de ensayo de cuantificación de miARN, donde las muestras se midieron en un fluorómetro de cuantificación, seleccionando el tipo de ensayo de miARN.
9. Secuenciación de ARN pequeño
- Después de la cuantificación del miARN dentro de la muestra, sintetice una biblioteca de ADNc utilizando el kit de preparación de biblioteca Small RNA-Seq.
NOTA: Con este protocolo, la generación de bibliotecas incluía cuatro pasos: ligadura del adaptador 3', purificación, ligadura del adaptador 5' y transcripción inversa, donde, al final, se forma una biblioteca de ARN/ADNc. Después de la generación de la biblioteca, se produce la amplificación de PCR de punto final con la adición de cebadores de índice (utilizados con fines de multiplexación), después de lo cual se purifican las muestras. - Para probar la calidad del ARN, utilice un ensayo de aplicación de control de calidad que detalle el tamaño de cada preparación y la intensidad máxima.
- Para probar la concentración de ADN, mida la cuantificación de ADN en un fluorómetro de cuantificación, seleccionando el tipo de ensayo dsDNA.
- Prepare las muestras para Small RNA Seq utilizando el kit de reactivos, después de lo cual las muestras se cargan en la celda de flujo.
Resultados
Para confirmar la presencia de BDsEVs, se utilizaron tres técnicas: western blot, NTA y TEM (Figura 3). Los resultados del Western blot (Figura 3A y Archivo Suplementario 1) muestran la presencia de los cinco marcadores positivos (CD9, CD63, CD81, Flot-1 y TSG101) y la ausencia de Calnexina en los sEV (utilizados como control negativo), lo que confirma que no hay contaminación con el contenido celular. Como era de esperar, los homogeneizados cerebrales (BH) muestran más proteínas de las observadas en los sEV para los marcadores analizados. Las imágenes TEM confirmaron la morfología de los BDsEV y su tamaño relativo dentro de la muestra (Figura 3B), lo que se confirmó aún más mediante el análisis de seguimiento de nanopartículas (Figura 3C).
La figura 3C (panel superior) muestra los tamaños de partícula aislados que caen entre 50 y 200 nm. A partir de los datos de NTA, tomando el resultado de la dispersión como el 100% de las partículas representadas, el CMO representa alrededor del 52,35% de las partículas (Figura 3C, panel inferior) que tienen una bicapa lipídica. De estas partículas teñidas con CMO, el 34,66% contenía CD9, el 15,49% CD63 y el 12,01% CD81. Consulte el Archivo Suplementario 2 para ver los datos de control NTA.
Los datos de cuantificación del ARN aislado (Tabla 2) muestran niveles que oscilan entre 3,24 y 8,76 (ng/μL), valores que se normalizaron al generar las bibliotecas de ADNc. Los resultados del ensayo TapeStation (Figura 4) muestran picos principales de alrededor de 120-160 pb, con picos más pequeños por debajo de esto de alrededor de 50 pb y otros picos más pequeños que son más altos de alrededor de 484 pb.
Las comprobaciones de calidad de los datos de secuenciación de próxima generación muestran lecturas de muy alta calidad después del recorte del adaptador (Figura 5A). Todas las posiciones de la secuencia muestran una puntuación Phred de >30, lo que indica tasas de error de menos de 1 en 1000 nucleótidos. La Figura 5B muestra que la mayoría de las secuencias están en torno a los 21-22 pb, coincidiendo con el rango de tamaño esperado para los microARN. A través de un mapeo posterior (usando BowTie2), utilizando la bioherramienta de análisis de flagstat de Samtools, de 1.304.100 secuencias alineadas, 1.116 264 (85%) se mapearon a las regiones de microARN en el genoma humano (build hg38; Tabla 3).
Del total de miRNAs, 808 miRNAs se expresaron a través de las 3 muestras, con 344 miRNAs compartidos entre las tres muestras (~43%), 5 compartidos entre el control 1 y el control 2, 183 compartidos entre el control 1 y el control 3, y 16 compartidos entre el control 2 y el control 3 (Figura 6). A partir de estos datos, los miRNA más expresados en los tres BDsEV son has-let-7b-5p, has-miR-143-3p, has-miR-30a-5p, has-miR-221-3p y has-let7i-5p. Del mismo modo, los 5 miARN menos expresados diferencialmente en las muestras de BDsEV (lecturas >10) son has-miR-128-2-5p, has-miR-182-5p, has-miR-193b-5p, has-miR-448 y has-miR-505-3p. Todos los recuentos de lecturas brutos y normalizados se dan en el Archivo Suplementario 3.

Figura 1: Flujo de trabajo de procesamiento de tejidos. La figura muestra el proceso escalonado para el aislamiento de sEV derivados del cerebro (BDsEV) de secciones de cerebro recién congeladas utilizando cromatografía de exclusión por tamaño. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Flujo de trabajo de aislamiento de ARN de BDsEVs. La figura muestra el proceso escalonado de aislar el ARN total de los BDsEV, incluido el paso previo al tratamiento para eliminar cualquier ARN extracelular utilizando proteinasa K y ARNasa A. Haga clic aquí para ver una versión más grande de esta figura.
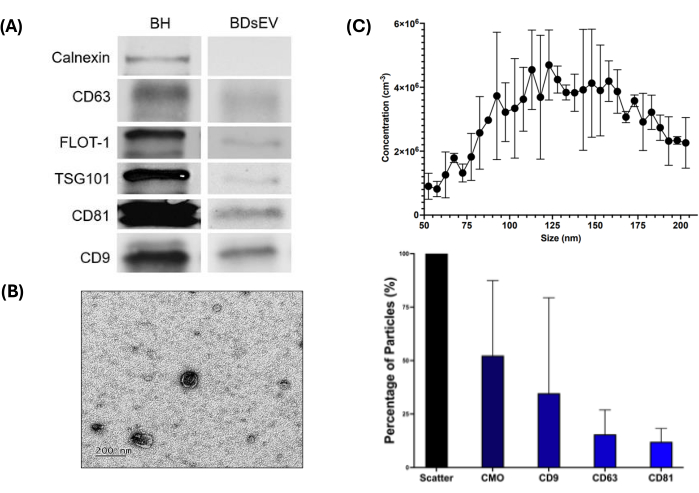
Figura 3: Flujo de trabajo de caracterización de BDsEV. La figura muestra varios métodos para caracterizar los BDsEV. (A) Western blot para detectar marcadores de proteínas específicos de sEV. BH Homogeneizado cerebral. La calnexina es un control negativo para los sEV: la ausencia en el sEV y la presencia en BH confirman la pureza de la muestra del sEV. Para otros marcadores, como se esperaba, la cantidad de proteína es mayor en BH que en las muestras de sEV (20 μg de proteína cargada por pocillo). (B) Imagen representativa de microscopía electrónica de transmisión (TEM) para BDsEVs a escala de 200 μM, que confirma la morfología esperada con una membrana de bicapa lipídica. (C) Gráfico de distribución de tamaño para el análisis de seguimiento de nanopartículas (NTA) para todas las partículas (panel superior). Cada punto de datos representa la variación en tres réplicas. Proporciones normalizadas para partículas biológicas (sEVs) y proporciones relativas para marcadores de superficie específicos (panel inferior) (datos mostrados como media ± SEM de pocillos triplicados (n = 3)). Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Control de calidad para la preparación de la biblioteca de ADNc. Análisis de cintas para muestras individuales después de la preparación de la biblioteca de ADNc. Los picos que se muestran en cada gráfico muestran el tamaño de cada preparación (eje horizontal, en pb) y la intensidad respectiva de cada pico (eje vertical, FU). Se espera que la pequeña biblioteca de ARN alcance un máximo de alrededor de 120-160 pb, como se ve en la figura. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Control de calidad para la secuenciación de miRnome. (A) Gráfico FASTQC para la calidad de lectura de las muestras individuales después del recorte del adaptador. El eje horizontal es para la posición de los nucleótidos y el eje vertical es para las puntuaciones de Phred. Los diagramas de caja para cada posición muestran la distribución de la puntuación Phred en todas las lecturas para cada posición. Todas las distribuciones dentro de la zona verde confirman datos de muy alta calidad (>Q30) que implican una preparación de muestras de alta calidad. (B) La distribución de la longitud de la secuencia para cada muestra muestra que para las tres muestras, la mayoría de las lecturas fueron de alrededor de 21-22 nucleótidos, el rango de tamaño esperado para el microARN. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Superposición de la expresión de microARN en todas las muestras. En el diagrama de Venn se muestra el número de miARN detectados en cada muestra (recuento de lecturas ≥ 10) y los microARN específicos compartidos en más de una muestra. Todas las lecturas sin procesar y normalizadas se muestran en el Archivo Suplementario 3. Haga clic aquí para ver una versión más grande de esta figura.
| Epítopo de anticuerpos | Factor de dilución del anticuerpo |
| Ratón Anti-CD9 | 1:2000 |
| Ratón Anti-CD63 | 1:2000 |
| Ratón Anti-CD81 | 1:2000 |
| Antiflotillina para ratones 1 | 1:2000 |
| Ratón anti-TSG101 | 1:1000 |
| Anti-Calnexina para ratones | 1:10 000 |
| Anticuerpo secundario anti-ratón | 1:3000 |
Tabla 1: Epítopos de anticuerpos utilizados para la caracterización de BDsEVs y sus respectivos factores de dilución.
| Muestra | Concentración de miARN (ng/μL) |
| 1 | 8.76 |
| 2 | 3.24 |
| 3 | 5.33 |
Tabla 2: Datos de cuantificación de ARN aislado.
| Muestra | Secuencias alineadas | MiARN mapeado | Porcentaje (%) |
| Ejemplo de control 1 | 1 444 665 | 1 133 437 | 78.46 |
| Ejemplo de control 2 | 442 808 | 381 631 | 86.18 |
| Ejemplo de control 3 | 2 024 828 | 1 983 726 | 97.97 |
| Promedio | 1 304 100.33 | 1 166 264.67 | 89.43 |
Tabla 3: Cantidad de secuencias de miRNA alineadas y mapeadas con éxito.
Archivo complementario 1: Datos complementarios del análisis de Western blot. Haga clic aquí para descargar este archivo.
Fichero complementario 2: Datos complementarios de control de NTA. Haga clic aquí para descargar este archivo.
Archivo complementario 3: Recuentos de lectura brutos y normalizados de análisis de expresión de miARN. Haga clic aquí para descargar este archivo.
Fichero complementario 4: Datos suplementarios de concentración de proteínas. Haga clic aquí para descargar este archivo.
Discusión
Este protocolo modificado y mejorado para aislar pequeñas vesículas extracelulares derivadas del cerebro y su carga de microARN demuestra la viabilidad de utilizar un mínimo de tejido sin comprometer la calidad y cantidad de los productos posteriores. En el campo del descubrimiento de biomarcadores, la identificación de identificadores moleculares específicos de los tipos de células y tejidos puede conducir a medios más accesibles de pruebas diagnósticas no invasivas que utilizan fluidos corporales. Además, el enfoque descrito aquí proporciona el marco para la identificación de subpoblaciones específicas de sEV dentro de diferentes tipos de células (neuronales frente a gliales), tipos de tejidos (tejido del hipocampo frente a tejido de la corteza prefrontal) y biofluidos, lo que ofrecerá una mayor comprensión de las firmas de carga de carga, la fisiopatología y el alcance de la enfermedad. Como se ha señalado previamente en Vella et al., se sugirió que se deberían utilizar 450 mg a 1 g para el aislamiento de BDsEV, junto con la ultracentrifugación en gradiente de densidad (utilizando velocidades de hasta 180.000 x g)8. En esta metodología, los sEV se han aislado con éxito utilizando cantidades más bajas (>50% de reducción) de muestras de tejido cerebral recién congeladas (250-450 mg). La reducción de la cantidad de tejido cerebral utilizado para aislar los sEV conduce a una reducción de costos y consumibles. Del mismo modo, el protocolo se ha hecho más accesible mediante la aplicación de SEC junto con la centrifugación de alta velocidad (10.000 x g) en lugar de la ultracentrifugación (180.000 x g).
Utilizando fNTA y TEM, establecimos que el perfil de rango de tamaño de los sEV se encuentra dentro del rango de tamaño característico de 30-200 nm, según lo recomendado por las guías MISEV13,14, con TEM proporcionando una confirmación morfológica de los sEV obtenidos. Como se muestra en la Figura 3C y en el Archivo Suplementario 4, el >87% de las partículas tienen un rango de tamaño de <250 nm, y las muestras, en promedio, muestran 10.56 fg proteína/BDsEV. Las especies específicas de pequeñas vesículas extracelulares varían en tamaño y forma, donde se observa que las subpoblaciones característicamente más pequeñas, como los exosomas, tienen una morfología uniforme en comparación con las microvesículas que se dice que tienen una estructura irregular debido a su naturaleza de biogénesis15. Para el fNTA, se produjo una lectura de dispersión (520 nm) para analizar el total de partículas en las muestras, proporcionando detalles del perfil de tamaño y las concentraciones. Las lecturas muestran que más del 50% de las partículas eran biológicas (a través de la tinción de CMO), con poblaciones de partículas biológicas que se encuentran dentro del rango de tamaño de las sEV que muestran marcadores de tetraspanina, siendo CD9 la más predominante en nuestras muestras de control (34,66%). Mientras que para el western blot, la presencia de cargas características de proteínas citosólicas y de membrana proporcionó evidencia de la presencia de sEV, y la ausencia de calnexina confirmó que los BDsEV estaban libres de cualquier contaminación celular. Para asegurarnos aún más de que el miARN disponible para el procesamiento posterior era parte de la carga de sEV (y no de otro ARN extracelular), tratamos los sEV con enzimas proteinasa K y RNasa A antes de interrumpir las membranas de sEV para degradar cualquier proteína y ARN que estuvieran presentes externamente en el espacio extracelular fuera de los BDsEV16.
El ARN total se extrajo de los sEV y se preparó para aplicaciones posteriores como qRT-PCR y secuenciación de próxima generación (para ARN pequeño). Los controles estándar mediante ensayos de control de calidad mostraron muestras de alta calidad y bibliotecas de ADNc listas para la secuenciación. Las bibliotecas de ADNc alcanzan su punto máximo en torno a los 120-160 pb, lo que se atribuye a picos de ADNc derivados de miARN con secuencias adaptadoras de longitudes variables, y los picos más pequeños pueden representar dímeros adaptadores.
El análisis de los datos de secuenciación de próxima generación utilizando FASTQC y CutAdapt muestra resultados de alta calidad obtenidos a partir de miARN BDsEV. Como se muestra en la Figura 5, cada posición leída tiene una puntuación Q superior a 30 (Q30), lo que muestra una probabilidad de error de 0,001-0,0001 (1 en 1000-10.000). Además, el análisis de la distribución de la longitud de la secuencia (Figura 6) muestra picos predominantes que se encuentran dentro del rango característico del miRNA (19-25 pb), lo que sugiere una alta calidad y especificidad para los datos obtenidos.
Numerosos estudios futuros se beneficiarán de este método debido a su mejor accesibilidad, además de ahorrar costos y recursos al reducir no solo la cantidad de muestras de tejido, sino también el costo de los reactivos. La reducción (>50%) de la cantidad necesaria de tejido hace que este método sea también más ético. En línea con los esfuerzos globales en salud de precisión, el flujo de trabajo mejorado que se detalla aquí proporciona herramientas comparativamente más asequibles para embarcarse en biomarcadores precisos específicos del tipo de célula a través de rutas no invasivas (o mínimamente invasivas) que utilizan BDsEV y su carga. Esto se puede utilizar para una amplia gama de afecciones neurológicas. Una posible limitación de este método es que el protocolo necesitará ajustes si los investigadores quieren utilizar un tejido distinto del cerebro humano para el aislamiento de sEV. A largo plazo, este método mejorado hará que el descubrimiento y la validación de biomarcadores no invasivos y específicos de tejidos sean más equitativos y accesibles en todo el mundo.
Divulgaciones
No hay conflictos de intereses para ninguno de los autores.
Agradecimientos
Este trabajo ha sido financiado por la beca de doctorado Joseph Morgan de la Alzheimer's Society UK (subvención número 549/SERA-52) y por los fondos de Estrategia de Innovación de la Universidad de Salford (subvención SEFA-39). El tejido encefálico se obtuvo del banco de cerebros de Manchester (referencia REC 09/H0906/52) de la Red de Cerebros para la Demencia.
Materiales
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin | Merck | A9418-100G | |
| Cell Mask Orange Plasma Membrane Stain | ThermoFisher Scientific | C10045 | |
| Collagenase Type-III | StemCell Technologies | 07422 | |
| ExoSpin Columns and Buffer | Cell Guidance Systems Ltd. | EX01-50 | This kit contains SEC columns used in this experiment, precipitation buffer and EV free PBS. |
| Halt Protease Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78429 | |
| Hibernate-E Medium | ThermoFisher Scientific | A1247601 | |
| Laemmli Sample Buffer (4x) | BioRad | 1610747 | |
| Lexogen Small RNA-Seq Library Prep Kit | Lexogen | 052.24 | This kit contains Small RNA preparation reagent box with i7 Index primer plate. |
| miRNeasy Micro Kit (50) | Qiagen | 217084 | This kit contains high-quality RNA recovery lysis reagent (Qiazol), RNA isolation columns, isolation buffers (RWT, RPE) and RNase free water. |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3001 | This is the Illumina Preparation Kit |
| Nitrocellulose Membrane, 0.45 μm | ThermoFisher Scientific | 88018 | |
| PE/Dazzle 594 anti-human CD63 Antibody | BioLegend | 143914 | Used for fNTA Analysis |
| PE/Dazzle 594 anti-human CD81 Antibody | BioLegend | 349520 | Used for fNTA Analysis |
| PE/Dazzle 594 anti-human CD9 Antibody | BioLegend | 312118 | Used for fNTA Analysis |
| PhosSTOP | Merck | 4906845001 | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23225 | |
| Proteinase K | ThermoFisher Scientific | 25530049 | |
| Qubit microRNA Assay Kit | ThermoFisher Scientific | Q32880 | |
| Qubit 1X dsDNA HS assay kit | ThermoFisher Scientific | Q33230 | |
| Qubit 3.0 Fluorometer | ThermoFisher Scientific | Q33216 | |
| RIPA Lysis and Extraction Buffer | ThermoFisher Scientific | 89901 | |
| RNase A | ThermoFisher Scientific | EN0531 | |
| SuperSignal West Femto Maximum Sensitivity Substrate | ThermoFisher Scientific | 34094 |
Referencias
- Jia, Y., et al. Small extracellular vesicles isolation and separation: Current techniques, pending questions and clinical applications. Theranostics. 12 (15), 6548-6575 (2022).
- Fornari, S., Schäfer, A., Jucker, M., Goriely, A., Kuhl, E. Prion-like spreading of Alzheimer's disease within the brain's connectome. J R Soc Interface. 16 (159), 20190356(2019).
- Zhang, P., et al. Tumor-derived small extracellular vesicles in cancer invasion and metastasis: molecular mechanisms, and clinical significance. Mol Cancer. 23 (1), 18(2024).
- Constantinides, V., et al. CSF Aβ42 and Aβ42/ Aβ40 ratio in Alzheimer's disease and frontotemporal dementia. Diagnostics. 13 (4), 783(2023).
- Cheng, L., et al. Prognostic serum miRNA biomarkers associated with Alzheimer's disease shows concordance with neuropsychological and neuroimaging assessment. Mol Psychiatry. 20 (10), 1188-1196 (2015).
- Roser, A. E., Caldi Gomes, L., Schünemann, J., Maass, F., Lingor, P. Circulating miRNAs as diagnostic biomarkers for Parkinson's disease. Front Neurosci. 12, 625(2018).
- Jackson, N., Guerrero-Muñoz, M. J., Castillo-Carranza, D. L. The prion-like transmission of tau oligomers via exosomes. Front Aging Neurosci. 14, 974414(2022).
- Vella, L., et al. A rigorous method to enrich for exosomes from brain tissue. J Extracell Vesicles. 6 (1), 1348885(2017).
- Yousif, G., Qadri, S., Parray, A., Akhthar, N., Shuaib, A., Haik, Y. Exosomes derived neuronal markers: Immunoaffinity isolation and characterization. Neuromolecular Med. 24 (3), 339-351 (2022).
- Kumar, K., et al. Recent advances in microfluidic approaches for the isolation and detection of exosomes. TRAC-Trend Anal Chem. 159, 116912(2023).
- Ransom, L., et al. Human brain small extracellular vesicles contain selectively packaged, full-length mRNA. Cell Rep. 43 (4), 114061(2024).
- Gomes, P., et al. A novel isolation method for spontaneously released extracellular vesicles from brain tissue and its implication for stress-driven brain pathology. Cell Commun Signal. 21 (1), 35(2023).
- Welsh, J., et al. Minimal information for studies of extracellular vesicles (MISEV 2023): From basic to advanced approaches. J Extracell Vesicles. 13 (2), e12404(2023).
- Théry, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750(2018).
- Pirisinu, M. The long journey of extracellular vesicles towards global scientific acclamation. Adv Pharm Bull. 13 (3), 489-501 (2023).
- Bender, A., Sullivan, B. P., Lillis, L., Posner, J. D. Enzymatic and chemical-based methods to inactivate endogenous blood ribonucleases for nucleic acid diagnostics. J Mol Diagn. 22 (8), 1030-1040 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados