Method Article
Isolamento di cellule con informazioni morfologiche e spaziali da campioni di fibrosi sottomucosa orale mediante microdissezione a cattura laser
In questo articolo
Riepilogo
La microdissezione a cattura laser dei tessuti di fibrosi sottomucosa orale consente l'estrazione precisa di cellule dalle regioni istologiche di interesse per l'analisi di dati multi-omici con informazioni morfologiche e spaziali.
Abstract
La fibrosi sottomucosa orale (OSF) è un tipo comune di disturbo potenzialmente maligno nella cavità orale. L'atrofia dell'epitelio e la fibrosi della lamina propria e della sottomucosa si riscontrano spesso sui vetrini istopatologici. È stato proposto che la displasia epiteliale, l'atrofia epiteliale e i fibroblasti senescenti siano associati alla trasformazione maligna della OSF. Tuttavia, a causa dell'eterogeneità delle malattie orali potenzialmente maligne e del carcinoma orale a cellule squamose, è difficile identificare i meccanismi molecolari specifici della trasformazione maligna nella OSF. Qui, presentiamo un metodo per ottenere un piccolo numero di cellule epiteliali o mesenchimali che trasportano dati morfologici e informazioni spaziali mediante microdissezione a cattura laser su vetrini di tessuto fissati in formalina e inclusi in paraffina. Utilizzando un microscopio, possiamo catturare con precisione il tessuto epiteliale displastico o atrofico su microscala (~500 cellule) e il tessuto subepiteliale fibrotico. Le cellule estratte possono essere valutate mediante sequenziamento del genoma o del trascrittoma per acquisire dati genomici e trascrittomici con informazioni morfologiche e spaziali. Questo approccio rimuove l'eterogeneità del sequenziamento di tessuti OSF e l'interferenza causata dalle cellule in aree non lesionate, consentendo un'analisi spazio-omica precisa del tessuto OSF.
Introduzione
La fibrosi sottomucosa orale (OSF) è una malattia cronica e insidiosa che si sviluppa principalmente nella mucosa vestibolare e provoca una limitazione dell'apertura della bocca1. Mentre l'OSF è una malattia multifattoriale, la masticazione della noce di areca o della noce di betel è la causa principale dell'OSF 2,3. A causa di questa abitudine geograficamente specifica, l'OSF è prevalentemente concentrata nelle popolazioni del sud-est e del sud dell'Asia3. Le caratteristiche istologiche comuni dell'OSF includono la deposizione anomala di collagene nel tessuto connettivo sotto l'epitelio della mucosa orale, la stenosi vascolare e l'occlusione1. Il tessuto epiteliale OSF può presentarsi con manifestazioni di atrofia o iperplasia e persino displasia se concomitante con leucoplachia orale 4,5.
L'OSF è definita dall'Organizzazione Mondiale della Sanità come una comune malattia orale potenzialmente maligna (OPMD) che presenta il potenziale per progredire verso il carcinoma orale a cellule squamose con un tasso di trasformazione maligna del 4%-6%6,7,8,9. Il meccanismo alla base della trasformazione maligna di OSF è complesso10. La crescita anormale dell'epitelio, inclusa la displasia e l'atrofia, aumenta il potenziale di cancerogenesi e i fibroblasti senescenti nello stroma possono essere coinvolti nella progressione maligna dell'OSF inducendo la transizione epiteliale-mesenchimale (EMT) attraverso le specie reattive dell'ossigeno (ROS) e altre molecole10.
Le tecnologie per le analisi spazio-omiche hanno generato dati multi-omici con informazioni morfologiche e spaziali che hanno fornito approfondimenti sui meccanismi del cancro11,12,13. Qui, presentiamo un protocollo per catturare popolazioni cellulari correlate alla morfologia da tessuti OSF inclusi in paraffina fissati in formalina mediante microdissezione laser. Le analisi multi-omiche di questi campioni possono superare le sfide dell'eterogeneità intratissutale e aumentare la comprensione della patologia molecolare e dei meccanismi di trasformazione maligna in OSF14.
Protocollo
Questo studio è stato approvato dal comitato di revisione istituzionale della Scuola e dell'Ospedale dell'Università di Pechino. Il consenso informato è stato ottenuto dai pazienti. I campioni di tessuto utilizzati in questo studio sono stati resi anonimi. Lo schema di studio è illustrato nella Figura 1.
1. Preparazione del campione
- Tagliare i tessuti di fibrosi sottomucosa orale fissati in formalina e inclusi in paraffina in sezioni continue di 3 μm e 10 μm di spessore su un microtomo.
- Aprire le sezioni in acqua e poi pescare sugli scivoli. Fissare le sezioni da 3 μM e 10 μm rispettivamente sui vetrini del microscopio ad adesione (vetrino A) e sui vetrini a membrana PEN (vetrino B).
- Mettere i vetrini su una piastra calda a 60 °C per 2 h.
2. Colorazione ematossilina-eosina
- Posizionare i vetrini A e B nello xilene (ATTENZIONE) per 10 minuti e ripetere questo passaggio tre volte.
- Idratare i vetrini A e B in soluzioni di etanolo graduate nell'ordine del 100%, 100%, 90%, 80% e 70% per 1 minuto in ciascuna concentrazione.
- Immergere i vetrini A e B in acqua distillata ultrapura per 30 s
- Posizionare i vetrini A e B nella soluzione di colorante ematossilina Harris per 90 s.
- Immergere i vetrini A e B in acqua distillata ultrapura per 30 secondi e ripetere questo passaggio tre volte.
- Mettere i vetrini A e B in una soluzione di div-ematossilina (ATTENZIONE) per 2 s.
- Immergere i vetrini A e B in acqua distillata ultrapura per 30 secondi e ripetere questo passaggio tre volte.
- Posizionare i vetrini in A e B in una soluzione riblu all'1% per 2 s.
- Immergere i vetrini A e B in acqua distillata ultrapura per 30 secondi e ripetere questo passaggio tre volte.
- Mettere i vetrini in soluzione di eosina per 2 s.
- Immergere i vetrini A e B in acqua distillata ultrapura per 30 secondi e ripetere questo passaggio tre volte.
- Disidratare i vetrini in soluzioni di etanolo graduate nell'ordine del 70%, 80%, 90%, 100% e 100% per 2 secondi in ciascuna concentrazione.
- Metti il vetrino A nello xilene per 3 minuti e ripeti questo passaggio due volte.
- Aggiungere una goccia di mezzo di montaggio (ATTENZIONE) per far scorrere A e coprirlo con un vetro di copertura.
3. Osservazione della morfologia istologica e microdissezione a cattura laser
- Avviare il sistema e il software di microdissezione laser (Figura 2).
- Posizionare il vetrino A sul tavolino e osservare la morfologia istologica per identificare l'area di interesse per la microdissezione laser.
- Rimuovere la diapositiva A e posizionare la diapositiva B in senso inversamente sul tavolino di scorrimento.
- Fare clic sul pulsante Scarica (Figura 2) per estrarre il dispositivo di raccolta. Inserire una provetta di reazione a catena della polimerasi (PCR) nel dispositivo di raccolta e assicurarsi che la provetta sia tenuta fissa. Fare clic sul pulsante OK (Figura 3).
- Selezionare un tappo facendo clic sul cerchio rosso corrispondente che contrassegna il dispositivo di raccolta: Tappi per tubi. Il cerchio selezionato diventerà verde.
- Fare clic sul pulsante Disegna e sul pulsante Disegna + Taglia e utilizzare il mouse per disegnare l'area di interesse.
- Fare clic sul pulsante Avvia taglio per acquisire l'area di interesse; il campione prelevato sarà raccolto dal tappo (Figura 4).
- Fare clic sul pulsante Inferiore per assicurarsi che il campione acquisito venga raccolto (Figura 5).
- Fare clic sul pulsante Campione per acquisire il campione successivo.
- Fare clic sul pulsante Scarica per scaricare la provetta PCR con il campione acquisito.
Risultati
Eseguendo la microdissezione laser dei tessuti OSF, abbiamo catturato campioni di epitelio displastico, stroma sotto l'epitelio displastico, epitelio atrofico e stroma sotto il tessuto epiteliale atrofico (Figura 1). Attraverso l'estrazione del DNA e il sequenziamento dell'intero genoma a bassa profondità, siamo stati in grado di analizzare le alterazioni del numero di copie (CNA) correlate alla morfologia15. La CNA è una forma comune di instabilità genomica associata ad un aumentato rischio di trasformazione maligna nell'OPMD15,16. Sono stati rilevati diversi modelli di CNA tra quattro tipi di campioni. Come mostrato nella Figura 6, la CNA era presente nei campioni epiteliali ma non nei campioni di stroma. Sebbene i campioni provenissero dallo stesso paziente, il modello CNA nell'epitelio displastico non era lo stesso di quello nell'epitelio atrofico. Il CNA nei cromosomi 3 e 8 è stato rilevato nell'epitelio displastico, mentre il CNA è stato rilevato con una frequenza inferiore nel cromosoma 8 nell'epitelio atrofico.
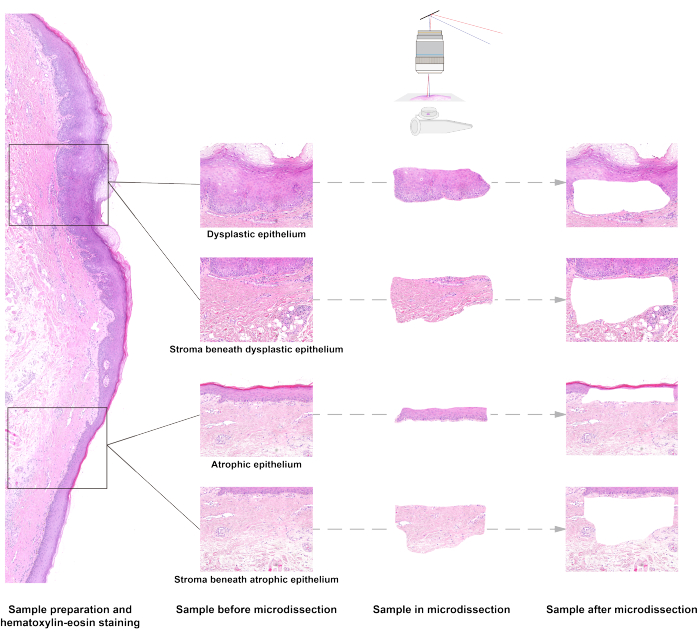
Figura 1: Lo schema della microdissezione a cattura laser di campioni di fibrosi sottomucosa orale. I tessuti dell'epitelio e dello stroma con modelli diversi vengono catturati dal laser al microscopio. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Software di microdissezione laser. Uno screenshot del software che mostra il pannello live. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Finestra del dispositivo di raccolta. Uno screenshot del software che mostra la finestra del dispositivo di raccolta per la selezione del dispositivo di raccolta. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Acquisizione del campione nell'area di interesse. Facendo clic sul pulsante Avvia taglio sarà possibile catturare l'area di interesse. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Campione acquisito. Il campione catturato può essere visto nel tappo della provetta PCR. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Modifiche del numero di copie. I risultati rappresentativi di diversi modelli di alterazioni del numero di copie tra i vari campioni. Esistono diverse alterazioni del numero di copie tra l'epitelio displastico e l'epitelio atrofico. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Questo protocollo ha riportato una pipeline per catturare campioni di tessuto OSF con informazioni morfologiche e spaziali per ulteriori analisi spazio-omiche attraverso la microdissezione laser. Dai risultati rappresentativi, abbiamo identificato diversi modelli CNA tra vari campioni correlati alla morfologia.
L'OSF, un tipo di OPMD, è una condizione precancerosa comune del carcinoma orale a cellule squamose6. È stato riportato che l'instabilità genomica è associata allo sviluppo e alla trasformazione maligna di OPMD17,18. Diversi studi hanno riportato alterazioni cromosomiche, espressione genica differenziale e variazioni epigenetiche associate alla progressione e alla trasformazione maligna di OSF dai dati di genomica, trascrittomica e proteomica 19,20,21,22,23,24. I componenti cancerogeni della noce di areca, come l'arecolina, non solo hanno portato allo sviluppo di OSF, ma la stimolazione a lungo termine ha anche indotto la senescenza dei fibroblasti e dell'EMT e la produzione continua di specie reattive dell'ossigeno (ROS), con conseguente trasformazione maligna regolando il microambiente immunitario e le vie di segnalazione come i segnali TGF-β e NF-κB5, 10,21. Tuttavia, nei tessuti OSF sono presenti sia alterazioni istologiche epiteliali che stromali e quali alterazioni tissutali o cellulari guidino la trasformazione maligna in OSF rimane poco chiaro. Gli studi precedenti hanno utilizzato principalmente analisi di sequenziamento di massa, in cui il DNA o l'RNA sono stati estratti da un blocco di tessuto per analizzare ulteriormente l'espressione molecolare e le vie coinvolte. Tuttavia, questa analisi mancava di una correlazione morfologica con l'istologia e ignorava l'eterogeneità intratissutale; Inoltre, questo approccio non è in grado di identificare se ci sono alterazioni molecolari differenziali in diverse aree istologiche.
Studi recenti hanno riportato l'analisi spazio-omica in molte malattie11,13,25. Attraverso una precisa analisi spazio-omica, si sta scoprendo un numero crescente di molecole e bersagli e si stanno chiarendo le interazioni cellulari e il clustering11,12,13. Tuttavia, c'è ancora una mancanza di analisi omiche spazialmente risolte di OSF. I campioni di OSF sono stati solitamente prelevati da biopsie della mucosa orale e sono stati preparati come piccoli volumi di campioni fissati in formalina e inclusi in paraffina, rendendo difficile l'esecuzione del sequenziamento omico spaziale a singola cellula, che richiede campioni di grandi dimensioni. Pertanto, lo sviluppo di metodi con risoluzione spaziale che utilizzano campioni fissati in formalina e inclusi in paraffina potrebbe essere un vantaggio significativo. Ci aspettiamo che l'attuale protocollo vada a beneficio dei ricercatori che lavorano su OSF e altri tipi di OPMD e contribuisca a comprendere i meccanismi dello sviluppo e della trasformazione maligna dell'OPMD.
Questo metodo presenta alcune limitazioni. In primo luogo, il protocollo è stato eseguito utilizzando campioni fissati in formalina e inclusi in paraffina, rendendo difficile l'estrazione dell'RNA per l'analisi di sequenziamento; tuttavia, l'analisi del sequenziamento dell'RNA correlata alla morfologia può essere eseguita quando si utilizza la colorazione viola cristallina e si mantengono pratiche asettiche15,26. I campioni attualmente catturati non sono in grado di soddisfare il livello di risoluzione di una singola cellula perché quando il numero di cellule è troppo piccolo, le cellule potrebbero essere scomposte dal raggio laser, influenzando l'estrazione del DNA.
Divulgazioni
Gli autori dichiarano di non avere alcun conflitto di interessi.
Riconoscimenti
Questo lavoro è stato supportato da sovvenzioni di ricerca della National Nature Science Foundation of China (81671006, 81300894), CAMS Innovation Fund for Medical Sciences (2019-I2M-5-038), National clinical key discipline construction project (PKUSSNKP-202102), Innovation Fund for Outstanding Doctoral Candidate of Peking University Health Science Center (BMU2022BSS001).
Materiali
| Name | Company | Catalog Number | Comments |
| Adhesion microscope slides | CITOTEST | REF.188105 | |
| Div-haematoxylin | YiLi | 20230326 | |
| Eosin solution | BASO | BA4098 | |
| Ethanol | PEKING REAGENT | No.32061 | |
| Harris hematoxylin dye solution | YiLi | 20230326 | |
| Hot plate | LEICA | HI1220 | |
| Laser capture microdissection system | LEICA | LMD7 | Machine |
| Laser microdissection microsystem | LEICA | 8.2.3.7603 | Software |
| Micromount mounting medium | LEICA | REF.3801731 | |
| Microscope cover glass | CITOTEST | REF.10212450C | |
| Microtome | LEICA | RM2235 | |
| PCR tubes | AXYGEN | 16421959 | |
| PEN-membrane slides | LEICA | No.11505158 | |
| Re-blue solution | YiLi | 20230326 | |
| Ultrapure distilled water | Invitrogen | REF.10977-015 | |
| Xylene | PEKING REAGENT | No.33535 |
Riferimenti
- Cai, X., et al. Oral submucous fibrosis: A clinicopathological study of 674 cases in China. Journal of Oral Pathology & Medicine. 48 (4), 321-325 (2019).
- Cai, X., Huang, J. Clinicopathological factors associated with progression of oral submucous fibrosis: A population-based retrospective study. Oral Oncology. 130, 105949(2022).
- Ray, J. G., Chatterjee, R., Chaudhuri, K. Oral submucous fibrosis: A global challenge. Rising incidence, risk factors, management, and research priorities. Periodontology 2000. 80 (1), 200-212 (2019).
- Shih, Y. H., Wang, T. H., Shieh, T. M., Tseng, Y. H. Oral submucous fibrosis: A Review on etiopathogenesis, diagnosis, and therapy. International Journal of Molecular Sciences. 20 (12), 2940(2019).
- Cai, X., et al. The preliminary exploration of immune microenvironment in oral leukoplakia concomitant with oral submucosal fibrosis: A comparative immunohistochemical study. Journal of Oral Pathology & Medicine. , (2023).
- Warnakulasuriya, S., et al. Oral potentially malignant disorders: A consensus report from an international seminar on nomenclature and classification, convened by the WHO Collaborating Centre for Oral Cancer. Oral Diseases. 27 (8), 1862-1880 (2021).
- Cai, X., et al. Development and validation of a nomogram prediction model for malignant transformation of oral potentially malignant disorders. Oral Oncology. 123, 105619(2021).
- Murthy, V., et al. Malignant transformation rate of oral submucous fibrosis: A systematic review and meta-analysis. Journal of Clinical Medicine. 11 (7), 1793(2022).
- Kujan, O., Mello, F. W., Warnakulasuriya, S. Malignant transformation of oral submucous fibrosis: A systematic review and meta-analysis. Oral Diseases. 27 (8), 1936-1946 (2020).
- Qin, X., Ning, Y., Zhou, L., Zhu, Y. Oral submucous fibrosis: Etiological mechanism, malignant transformation, therapeutic approaches and targets. International Journal of Molecular Sciences. 24 (5), 4992(2023).
- Sun, L., et al. Single-cell and spatial dissection of precancerous lesions underlying the initiation process of oral squamous cell carcinoma. Cell Discovery. 9 (1), 28(2023).
- Zhu, J., et al. Delineating the dynamic evolution from preneoplasia to invasive lung adenocarcinoma by integrating single-cell RNA sequencing and spatial transcriptomics. Experimental & Molecular Medicine. 54 (11), 2060-2076 (2022).
- Ji, A. L., et al. Multimodal analysis of composition and spatial architecture in human squamous cell carcinoma. Cell. 182 (2), 497-514 (2020).
- Van den Bossche, V., et al. Microenvironment-driven intratumoral heterogeneity in head and neck cancers: clinical challenges and opportunities for precision medicine. Drug Resistance Updates. 60, 100806(2022).
- Li, X., et al. Improvement in the risk assessment of oral leukoplakia through morphology-related copy number analysis. Science China. Life sciences. 64 (9), 1379-1391 (2021).
- Watkins, T. B. K., et al. Pervasive chromosomal instability and karyotype order in tumour evolution. Nature. 587 (7832), 126-132 (2020).
- Odell, E. W. Aneuploidy and loss of heterozygosity as risk markers for malignant transformation in oral mucosa. Oral Diseases. 27 (8), 1993-2007 (2021).
- Wood, H. M., et al. The genomic road to invasion-examining the similarities and differences in the genomes of associated oral pre-cancer and cancer samples. Genome Medicine. 9 (1), 53(2017).
- Venugopal, D. C., et al. Integrated proteomics based on 2D gel electrophoresis and mass spectrometry with validations: Identification of a biomarker compendium for oral submucous fibrosis-An indian study. Journal of Personalized Medicine. 12 (2), 208(2022).
- Kundu, P., Pant, I., Jain, R., Rao, S. G., Kondaiah, P. Genome-wide DNA methylation changes in oral submucous fibrosis. Oral Diseases. 28 (4), 1094-1103 (2022).
- Cai, X., Zhang, H., Li, T. Multi-target pharmacological mechanisms of Salvia miltiorrhiza against oral submucous fibrosis: A network pharmacology approach. Archives of Oral Biology. 126, 105131(2021).
- Xiao, X., Hu, Y., Li, C., Shi, L., Liu, W. DNA content abnormality in oral submucous fibrosis concomitant leukoplakia: A preliminary evaluation of the diagnostic and clinical implications. Diagnostic Cytopathology. 48 (11), 1111-1114 (2020).
- Zhou, S., et al. Long non-coding RNA expression profile associated with malignant progression of oral submucous fibrosis. Journal of Oncology. 2019, 6835176(2019).
- Lunde, M. L., et al. Profiling of chromosomal changes in potentially malignant and malignant oral mucosal lesions from South and Southeast Asia using array-comparative genomic hybridization. Cancer Genomics & Proteomics. 11 (3), 127-140 (2014).
- Sun, C., et al. Spatially resolved metabolomics to discover tumor-associated metabolic alterations. Proceedings of the National Academy of Sciences of the United States of America. 116 (1), 52-57 (2019).
- Ma, M., et al. Copy number alteration profiling facilitates differential diagnosis between ossifying fibroma and fibrous dysplasia of the jaws. International Journal of Oral Science. 13 (1), 21(2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon