Method Article
Imaging tissutale altamente multiplexato con coloranti Raman
In questo articolo
Riepilogo
L'imaging a diffusione Raman (epr-SRS) stimolato dalla pre-risonanza elettronica di coloranti Raman simili a arcobaleno è una nuova piattaforma per l'imaging proteico a base di epitopi altamente multiplexati. Qui, presentiamo una guida pratica che include la preparazione degli anticorpi, la colorazione del campione di tessuto, l'assemblaggio del microscopio SRS e l'imaging tissutale epr-SRS.
Abstract
La visualizzazione di una vasta gamma di biomarcatori specifici nei tessuti svolge un ruolo vitale nell'esplorazione delle intricate organizzazioni di sistemi biologici complessi. Pertanto, le tecnologie di imaging altamente multiplexed sono state sempre più apprezzate. Qui, descriviamo una piattaforma emergente di imaging vibrazionale altamente multiplexato di proteine specifiche con sensibilità comparabile all'immunofluorescenza standard tramite imaging a diffusione Raman (epr-SRS) stimolato dalla pre-risonanza elettronica di coloranti Raman simili all'arcobaleno. Questo metodo aggira il limite dei canali risolvibili spettralmente nell'immunofluorescenza convenzionale e fornisce un approccio ottico one-shot per interrogare più marcatori nei tessuti con risoluzione subcellulare. È generalmente compatibile con i preparati tissutali standard, compresi i tessuti fissati alla paraformaldeide, i tessuti congelati e i tessuti umani incorporati nella paraffina fissata alla formalina (FFPE). Prevediamo che questa piattaforma fornirà un quadro più completo delle interazioni proteiche dei campioni biologici, in particolare per i tessuti intatti spessi. Questo protocollo fornisce il flusso di lavoro dalla preparazione degli anticorpi alla colorazione del campione di tessuto, all'assemblaggio del microscopio SRS, all'imaging tissutale epr-SRS.
Introduzione
I sistemi tissutali complessi sono composti da distinte sottopopolazioni cellulari le cui posizioni spaziali e reti di interazione sono profondamente intrecciate con le loro funzioni e disfunzioni 1,2. Per rivelare l'architettura del tessuto e interrogarne la complessità, è essenziale la conoscenza delle posizioni spaziali delle proteine a risoluzione monocellulare. Quindi, le tecnologie di imaging proteico altamente multiplexed sono state sempre più apprezzate e potrebbero diventare una pietra miliare per lo studio della biologia dei tessuti 3,4,5. Gli attuali metodi comuni di imaging delle proteine multiplexate possono essere classificati in due categorie principali. Uno è l'imaging a immunofluorescenza seriale che si basa su più cicli di colorazione e imaging dei tessuti, e l'altro è la citometria di massa accoppiata con anticorpi marcati con metalli pesanti 6,7,8,9,10,11,12.
Qui viene introdotta una strategia alternativa per l'imaging proteico a base di anticorpi multiplexati. A differenza della modalità di imaging a fluorescenza prevalente, che può visualizzare solo 4-5 canali contemporaneamente a causa degli ampi spettri di eccitazione ed emissione (larghezza intera a metà massimo (FWHM) ~ 500 cm-1), la microscopia Raman presenta una larghezza di linea spettrale molto più stretta (FWHM ~ 10 cm-1) e quindi fornisce multiplextà scalabile. Recentemente, sfruttando lo spettro ristretto, è stato sviluppato un nuovo schema di microscopia Raman chiamato microscopia elettronica a dispersione Raman stimolata dalla pre-risonanza (epr-SRS), fornendo una potente strategia per l'imaging multiplexato13. Sondando le modalità vibrazionali accoppiate elettronicamente dei coloranti Raman, epr-SRS ottiene un drastico effetto di miglioramento di10 13 volte sulle sezioni trasversali Raman e supera il collo di bottiglia di sensibilità della microscopia Raman convenzionale (Figura 1A)13,14,15. Di conseguenza, il limite di rilevazione di epr-SRS è stato spinto al di sotto di μM, il che consente il rilevamento Raman di interessanti marcatori molecolari come proteine e organelli specifici all'interno delle cellule13,16. In particolare, utilizzando anticorpi coniugati con colorante Raman, l'imaging epr-SRS di proteine specifiche in cellule e tessuti (chiamato immuno-eprSRS) è stato dimostrato con sensibilità comparabile all'immunofluorescenza standard (Figura 1B)13,17. Regolando la lunghezza d'onda della pompa di soli 2 nm, il segnale epr-SRS sarà completamente spento (Figura 1B), che mostra un elevato contrasto vibrazionale.
Sul lato della sonda, è stata sviluppata una serie di sonde Raman arcobaleno chiamate coloranti Manhattan Raman scattering (MARS) per la coniugazione anticorpale 13,18,19,20. Questa tavolozza Raman unica è composta da nuovi coloranti con tripli legami coniugati π (materiale supplementare), ognuno dei quali mostra un picco epr-SRS singolo e stretto nell'intervallo spettrale Raman bioortogonale (Figura 1C). Modificando la struttura del cromoforo del nucleo e modificando isotopicamente entrambi gli atomi del triplo legame (Materiale Supplementare), sono state sviluppate sonde Raman separate spettralmente. Sfruttando la multiplexità scalabile, la microscopia epr-SRS abbinata alla tavolozza dei coloranti MARS offre una strategia ottica per l'imaging di proteine multiplex one-shot in cellule e tessuti.
Immuno-eprSRS fornisce una strategia alternativa agli attuali metodi di imaging delle proteine multiplex con punti di forza unici. Rispetto agli approcci di fluorescenza con colorazione ciclica, imaging e rimozione del segnale, questa piattaforma basata su Raman garantisce colorazione e imaging a singolo tondo. Pertanto, aggira la complessità pratica nelle procedure cicliche e semplifica in gran parte il protocollo, aprendo così nuovi territori di imaging di proteine multiplexate. Ad esempio, sfruttando un protocollo di compensazione dei tessuti su misura per il colorante Raman, l'immuno-eprSRS è stato esteso a tre dimensioni per la mappatura di proteine altamente multiplexate in tessuti intatti spessi17. Oltre 10 bersagli proteici sono stati visualizzati lungo tessuti cerebrali di topo spessi millimetri17. Più recentemente, l'accoppiamento di immuno-eprSRS con un protocollo di microscopia di espansione a ritenzione di biomolecole ottimizzato (ExM)protocollo 21, è stato anche dimostrato l'imaging su scala nanometrica one-shot di più bersagli22. Rispetto alla spettroscopia di massa 4,9, epr-SRS non è distruttivo e ha intrinsecamente capacità di sezionamento ottico. Inoltre, epr-SRS è più efficiente in termini di tempo nella scansione dei tessuti. In genere, una regione di tessuto di 0,25 mm2 con una dimensione dei pixel di 0,5 μm richiede solo pochi minuti per l'immagine per un singolo canale epr-SRS. Ad esempio, il tempo totale di imaging di quattro canali SRS più quattro canali di fluorescenza nella Figura 4 è di circa 10 minuti.
Protocollo
Il protocollo è stato condotto in conformità con il protocollo sperimentale animale (AC-AABD1552) approvato dall'Institutional Animal Care and Use Committee della Columbia University.
1. Preparazione di anticorpi coniugati con colorante Raman
- Preparare il tampone di coniugazione come ~0,1 M NaHCO3 in tampone PBS, pH = 8,3, conservare a 4 °C.
- Preparare la soluzione di sonda MARS (materiale supplementare) con funzione estere N-idrossisuccinimidy (NHS) come 3 mM in DMSO anidro. La sintesi delle sonde MARS può essere riferita ai rapporti precedenti 13,17,18.
NOTA: Ai fini della conservazione, la soluzione di estere colorante NHS deve essere protetta dalla luce e mantenuta a -20 °C. - Sciogliere i solidi anticorpali nel tampone di coniugazione ad una concentrazione di 2 mg/mL. Per gli anticorpi disciolti in altri tamponi, scambiarli nel tampone di coniugazione ad una concentrazione di 1-2 mg/mL con filtri centrifughi.
NOTA: gli anticorpi secondari altamente adsorbiti sono preferiti per la colorazione multiplex per ridurre al minimo la reattività tra specie. Gli anticorpi secondari utilizzati sono elencati nella Tabella 1 e nella Tabella dei materiali. - Eseguire la reazione di coniugazione.
- Per gli anticorpi secondari, aggiungere 15 volte l'eccesso molare di soluzione di colorante alla soluzione anticorpale in un flaconcino di vetro lentamente mescolando. Ad esempio, in 0,5 mL 2 mg/mL di soluzione anticorpale, aggiungere 35 μL 3 mM di soluzione colorante.
- Incubare la miscela di reazione a temperatura ambiente (RT) con agitazione per 1 ora. Proteggere la reazione dalla luce.
- Purificazione.
- Preparare il liquame di resina di filtrazione del gel (Tabella dei materiali) nel tampone PBS, seguendo i passaggi 1.5.2-1.5.4.
- Aggiungere 10 mL di polvere di resina filtrante gel in 40 mL di tampone PBS all'interno di un tubo da 50 mL.
- Conservare la soluzione a bagnomaria a 90 °C per 1 ora.
- Decantare il surnatante e riaggiungere PBS a 40 ml. Conservare il liquame a 4 °C.
- Imballare la colonna di esclusione delle dimensioni (diametro di 1 cm, colonne a flusso gravitazionale) con la soluzione di liquame all'altezza di 10-15 cm.
- Risciacquare e lavare la colonna con ~ 10 ml di tampone PBS per imballare ulteriormente la resina.
- Pipettare la miscela di reazione di coniugazione (~0,5 mL) alla colonna. Aggiungere immediatamente 1 mL di tampone PBS come tampone di eluizione quando viene caricata tutta la miscela di reazione. Riempire costantemente il buffer di eluizione (PBS) sulla colonna.
- Raccogliere l'eluato della soluzione coniugata osservando il colore sulla colonna (i coloranti MARS hanno colori da verde chiaro a blu) o misurando l'assorbanza a 280 nm (A280).
- Concentrare la soluzione raccolta a 1-2 mg/mL con un filtro centrifugo.
- Determinare la concentrazione e il grado medio di etichettatura (DOL, rapporto colorante-proteina) misurando lo spettro ultravioletto-visibile (UV-Vis) della soluzione coniugata con un lettore di piastre Nano.
NOTA: il materiale supplementare fornisce le proprietà degli esteri NHS del colorante MARS per il calcolo. Il DOL normale per l'anticorpo secondario è di circa 3.
2. Preparazione del campione di tessuto
- Paraformaldeide fissa tessuti cerebrali di topo.
- Anestetizzare i topi (C57BL/6J, Femmina, 25 d postnatale) con isoflurano. Valutare la corretta anestesia con un test del pizzico della punta.
- Uccidi i topi per spostamento cervicale. Perfondere immediatamente i topi con il 4% di paraformaldeide (PFA) in PBS transcardialmente.
- Raccogliere il cervello del topo, seguendo i passaggi 2.1.4-2.1.5
- Tagliare verso l'alto dal tronco cerebrale lungo la sutura sagittale. Sbuccia le due metà del cranio di lato e tira fuori il cervello con una pinzetta.
- Fissare il cervello raccolto in PFA al 4% in PBS a 4 °C per 24 ore. Quindi, lavare il cervello nel tampone PBS a 4 °C per 24 ore per rimuovere l'eccesso di PFA.
- Introdurre l'agarosio solido in acqua fino ad una concentrazione finale del 7% (p/v) in un becher, con un coperchio sciolto. Mescolare la soluzione con un'asta di agitazione del vetro. Riscaldare il liquame nel microonde fino a quando la soluzione è limpida.
- Lasciare raffreddare l'agarosio a 45-55 °C.
- Versare l'agarosio liquido in una piccola camera, quindi trasferire il cervello da PBS all'agarosio liquido e orientarlo con una spatola per incorporare il cervello. Attendere che il blocco tessuto-agarosio si indurisca.
- Sezionare il tessuto-agrose in fette coronali spesse 40 μm usando un vibratoma.
- Trasferire il tessuto in una piastra a 4 pozzetti per la successiva colorazione. Rimuovere l'agarosio con una pinzetta. Lavare il tessuto con 1 ml di PBS, tre volte.
- Tessuti pancreatici di topo congelati fissi.
- Fissare la pancreata del mouse in PFA al 4% in PBS a 4 °C con oscillazione per 16-20 ore.
- Lavare il campione in 1 mL di PBS (4 °C) tre volte per rimuovere il PFA.
- Incorporare il campione (~ 0,3-0,5 cm di dimensione) in blocchi composti a temperatura di taglio ottimale (OCT). Metti 2 gocce di OCT in un criostampo di plastica. Posizionare il tessuto nel corretto orientamento e versare OCT sopra i tessuti fino a quando nessuno dei tessuti rimane esposto.
- Sezionare la pancreata a fette spesse 8 μm e immobilizzarle su un vetrino di vetro legante il tessuto, conservarle a -80 °C.
- Prima di colorare, equilibrare il campione a RT. Lavare il tessuto con PBS per rimuovere i blocchi OCT.
- Campioni FFPE.
- Cuocere il vetrino ffpe a 60 °C per 10 min.
- Deparaffinazione e reidratazione: Posizionare i campioni in sequenza nelle seguenti soluzioni in un tubo da 50 ml a RT per 3 minuti ogni volta con lieve agitazione:
xilene due volte,
etanolo due volte,
95% (vol/vol) di etanolo in acqua deionizzata due volte,
70% (vol/vol) di etanolo in acqua deionizzata due volte,
50% (vol/vol) di etanolo in acqua deionizzata una volta,
acqua deionizzata una volta. - Trasferire il campione in 20 mM di citrato di sodio (pH 8,0) a 100 °C in un barattolo di vetro. Assicurarsi che i tessuti siano immersi nella soluzione.
- Mettere il barattolo a bagnomaria a 60 °C per 45 min.
- Lavare il campione con acqua deionizzata a RT per 5 minuti.
3. Colorazione immuno-eprSRS tissutale
- Utilizzare una penna idrofoba per disegnare un limite attorno alle sezioni di tessuto sul vetrino.
NOTA: un barattolo di colorazione del vetrino viene utilizzato per seguire le fasi di incubazione del tessuto sul vetrino. I tessuti galleggianti (sezioni cerebrali di topo spesse 40 μm) sono macchiati in piastre di pozzo. - Incubare i tessuti con 0,3-0,5% PBST (Triton X-100 in PBS) per 10 min.
- Incubare i tessuti con tampone bloccante (5% siero d'asino, 0,5% Triton X-100 in PBS) per 30 min.
- Preparare la soluzione colorante primaria: aggiungere tutti gli anticorpi primari a 200-500 μL di tampone colorante (2% siero d'asino, 0,5% Triton X-100 in PBS) alle concentrazioni desiderate. Centrifugare la soluzione colorante primaria a 13.000 x g per 5 min. Utilizzare il surnatante solo se si formano precipitati.
- Incubare il tessuto nella soluzione anticorpale primaria a 4 °C per 1-2 giorni.
NOTA: per colorare le sezioni di tessuto sul vetrino, mettere il campione in una scatola di colorazione con una salvietta umidificata per mantenere l'umidità. - Lavare le diapositive tre volte con 0,3-0,5% PBST a RT per 5 minuti ciascuna. Utilizzare 1 mL PBST per tessuti galleggianti. Per i tessuti sul vetrino, lavare i vetrini in un barattolo di colorazione del vetrino e assicurarsi che i tessuti siano tutti immersi nella soluzione.
- Incubare il tessuto in 200-500 μL di tampone bloccante per 30 min.
- Preparare la soluzione colorante secondaria: aggiungere tutti gli anticorpi secondari (e le lectine se necessario) a 200-500 μL di tampone colorante con le concentrazioni desiderate (normalmente 10 μg/mL). Centrifugare la soluzione colorante secondaria a 13.000 x g per 5 min. Utilizzare il surnatante solo se si formano precipitati.
- Incubare i tessuti in 200-500 μL di soluzione anticorpale secondaria a 4 °C per 1-2 giorni.
- Lavare le diapositive due volte con 0,3-0,5% PBST a RT per 5 minuti ciascuna.
- Incubare con 200-500 μL di soluzione DAPI per 30 min.
- Lavare le diapositive tre volte con PBS a RT per 5 minuti ciascuna.
- Per le sezioni di tessuto galleggianti, trasferirle su vetrini con una pipetta a goccia di vetro. Stendere il tessuto con una spazzola per tessuti e pulire l'ambiente circostante con salviette se necessario.
- Montare il tessuto in una goccia di reagenti antideflagranti con un coperchio di vetro e fissarlo con lo smalto per unghie.
4. Assemblaggio del microscopio SRS
NOTA: un sistema di fluorescenza confocale commerciale viene utilizzato nell'imaging a fluorescenza SRS in tandem. Ulteriori descrizioni sono disponibili in un rapporto precedente17. Questo protocollo si concentrerà sul lato di imaging SRS utilizzando l'eccitazione a banda stretta.
- Preparare un tavolo ottico isolato dalle vibrazioni in una stanza con controllo della temperatura.
- Posizionare un sistema a doppio laser sincronizzato (pompa e Stokes) sul tavolo ottico (Figura 2A) con un refrigeratore collegato.
NOTA: Il laser fondamentale nel sistema dual-laser fornisce un treno di impulsi di uscita a 1064 nm con larghezza di impulso di 6 ps e frequenza di ripetizione di 80 MHz. Il raggio di Stokes proviene dal laser fondamentale. L'intensità del fascio di Stokes è stata modulata sinusoidalemente da un modulatore di ampiezza elettro-ottica (EOM) incorporato a 8 MHz con una profondità di modulazione superiore al 90%. L'altra porzione del laser fondamentale è raddoppiata in frequenza a 532 nm, che viene ulteriormente utilizzata per seminare in modo sincrono un oscillatore ottico parametrico (OPO) a picosecondi per produrre un treno di impulsi bloccato in modalità con larghezza di impulso di 5-6 ps (il fascio inattivo dell'OPO è bloccato con un filtro interferometrico). La lunghezza d'onda di uscita dell'OPO è sintonizzabile da 720-950 nm, che funge da fascio della pompa. - Montare gli specchi (gamma di lunghezze d'onda: 750-1100 nm), gli splitter a fascio dicroico (DBS, filtro passa-lungo da 980 nm, rettangolare) e la lente (acromatico, rivestimento AR per 650-1050 nm) sui rispettivi supporti. Utilizzare supporti a specchio cinematici molto stabili per gli specchi e gli splitter a fascio dicroico.
- Misurare l'altezza dell'uscita laser e le dimensioni del fascio della pompa e dei raggi Stokes. Regola l'altezza degli specchi e delle lenti per assicurarti che la luce colpisca il centro di tutti gli elementi ottici.
- Posizionare lo specchio M1 sul tavolo ottico e renderlo ~ 45 ° all'uscita laser (Figura 2B). Utilizzare le manopole sul supporto cinematico per effettuare regolazioni di punta e inclinazione. Assicurarsi che la luce viaggi alla stessa altezza lungo la lunghezza del tavolo e una linea retta rispetto al tavolo.
- Posizionare e allineare gli splitter a fascio dicroico (filtri long-pass da 980 nm) e gli specchi per dividere la pompa e le travi di Stokes (Figura 2A-B).
- Posizionare e allineare le coppie di lenti (L1, L2 e L3, L4) su ciascuno dei percorsi del fascio per collimare i raggi ed espandere i diametri del fascio in modo che corrispondano alla pupilla posteriore dell'obiettivo (Figura 2A-B).
- Utilizzare specchi M7 e M8 per allineare i raggi laser combinati al microscopio (Figura 2C). Allineare prima un raggio nel microscopio e utilizzare le coppie di specchi sull'altro raggio per garantire la sovrapposizione spaziale dei due fasci.
- Impostare la parte di rilevamento.
- Indossare un condensatore di olio rivestito a infrarossi (1.4-NA) per raccogliere la pompa in avanti e le travi di Stokes dopo aver attraversato i campioni (Figura 2C).
- Montare un fotodiodo Si di grandi dimensioni su una scatola schermata con connettori BNC (Figura 2E). Aggiungere un alimentatore a 64 V CC al fotodiodo montato per aumentarne la soglia di saturazione e la larghezza di banda di risposta.
- Rifletti la luce in avanti con uno specchio da 2 pollici. Rifocalizzare la luce sul fotodiodo dopo un filtro ottico per bloccare il fascio di Stokes modulante (Figura 2D).
- Invia la corrente di uscita del fotodiodo a un amplificatore lock-in veloce terminato con 50 per la demodulazione del segnale. Invia un trigger a 8 MHz all'amplificatore lock-in come segnale di riferimento.
- Inviare il componente X in fase dell'amplificatore lock-in nella scatola di interfaccia analogica del microscopio.
- Ottimizzare la sovrapposizione temporale con lo stadio di ritardo motorizzato integrato misurando il segnale SRS del liquido D2O puro al microscopio.
5. Acquisizione e analisi delle immagini
- Esegui l'imaging epr-SRS multicanale con la regolazione sequenziale della lunghezza d'onda della pompa.
- Impostare la potenza del laser su pompa P = 10-40 mW e PStokes = 40-80 mW sul pannello di controllo laser.
- Impostare il tempo di permanenza dei pixel a 2-4 μs e utilizzare più fotogrammi con una media di 10-20 fotogrammi sul software di microscopia.
NOTA: Evitare un uso combinatorio di alta potenza laser (pompa P> 40 mW, PStokes> 80 mW) e dimensioni ridotte dei pixel (<0,2 μm), che probabilmente causano "effetto sbiancamento" dei coloranti Raman a causa dell'eccitazione multifotonica. - Impostare le costanti temporali dell'amplificatore lock-in su metà del tempo di permanenza dei pixel.
- Unmixing spettrale lineare.
NOTA: epr-SRS segue una rigorosa dipendenza lineare segnale-concentrazione sull'intero intervallo di concentrazione; pertanto, l'unmixing spettrale lineare è efficace per rimuovere qualsiasi potenziale cross-talk tra canali. Per la misura epr-SRS a canale N con sonde N MARS, i segnali misurati (S) possono essere espressi come S = MC, dove C è la concentrazione della sonda MARS e M è una matrice N x N determinata dalle sezioni trasversali Raman delle sonde MARS.- Misurare la matrice M su campioni immuno-eprSRS monocolore etichettati con diverse sonde MARS.
- Usare l'equazione C = M−1· S per determinare la matrice di concentrazione della sonda MARS con la misurazione del segnale del campione multiplex S.
Risultati
La Figura 3 mostra immagini di esempio di epr-SRS in diversi campioni, tra cui cellule fisse (Figura 3A), tessuti di topo fissati per paraformaldeide (PFA) (Figura 3B) e campioni umani incorporati in paraffina fissa in formalina (FFPE) (Figura 3C). La risoluzione spaziale della microscopia SRS è limitata alla diffrazione, la risoluzione laterale tipica è di ~ 300 nm e la risoluzione assiale è di 1-2 μm utilizzando la luce del vicino infrarosso per l'eccitazione. Di conseguenza, le strutture subcellulari fini come i microtubuli nelle cellule HeLa sono state rivelate fedelmente con l'imaging immuno-eprSRS di α-tubulina (Figura 3A). Inoltre, epr-SRS è generalmente compatibile con i tessuti FFPE (Figura 3C), che è una forma comune di campioni di biopsia per la diagnosi clinica e la ricerca patologica. Simile alla microscopia a fluorescenza a due fotoni, come processo non lineare, epr-SRS ha capacità di sezionamento ottico per visualizzare modelli tridimensionali con risoluzione subcellulare (Figura 3D-E).
Per prima cosa abbiamo mostrato l'utilità di imaging della proteina multiplex di epr-SRS su campioni di tessuto congelato fisso di isole di topo di Langerhans nel pancreas. Sono stati selezionati diversi bersagli interessati, tra cui l'espressione ormonale (ad esempio, insulina, agonista del glucosio (glucagone), polipeptide pancreatico (PP) e somatostatina) per la classificazione del tipo cellulare (cellule β e cellule non β (cellule α, δ)) e fattori di trascrizione che sono noti per essere correlati con l'eterogeneità delle cellule β23. Da notare, poiché il rilevamento della fluorescenza è ortogonale al rilevamento SRS, epr-SRS è pienamente compatibile con la fluorescenza confocale e la fluorescenza a due fotoni. Come proof-of-concept, l'imaging tandem SRS-fluorescenza a 7 colori su una singola isolotto è stato prontamente raggiunto (Figura 4) con un buon contrasto e modelli corretti. I bersagli a bassa espressione come il fattore di trascrizione Pdx1 sono stati ripresi con un contrasto sufficiente.
Abbiamo anche dimostrato un imaging tandem SRS-fluorescenza a otto colori in tessuti cervellettosi di topo fissati con PFA (Figura 5). Attraverso biomarcatori stabiliti, è possibile identificare diversi tipi di cellule, come i neuroni del granulo cerebellare (NeuN), i neuroni di Purkinje (Calbindin), gli astrociti (GFAP), gli oligodendrociti (MBP) e i neuroni GABAergici (recettore GABA B2 ).

Figura 1: Microscopia Epr-SRS per l'imaging di proteine altamente multiplexate. (A) Diagramma energetico per Raman spontaneo, SRS non risonante e SRS elettronico pre-risonante (epr-SRS). Il tasso di transizione vibrazionale dei cromofori sarà migliorato in epr-SRS fino a 1013 volte. (B) L'immuno-imaging a base di epitopi di α-tubulina è stato dimostrato in cellule COS-7 colorate da ATTO740 con elevato contrasto vibrazionale da epr-SRS. Il segnale epr-SRS scompare completamente quando la lunghezza d'onda del laser della pompa è fuori risonanza di soli 2 nm (a destra). Barre di scala, 20 μm. (C) Spettri Epr-SRS delle sonde MARS coniugate con estere NHS come elencato in Materiale supplementare. Fare clic qui per visualizzare una versione più grande di questa figura.
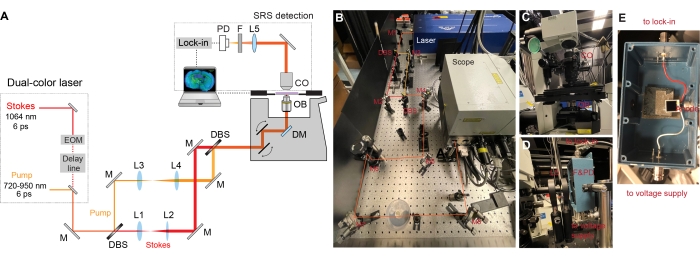
Figura 2: La progettazione del set-up del microscopio SRS. (A) Diagramma schematico della configurazione SRS. EOM = modulatore elettro-ottico, M = specchio, L = lente, DBS = splitter a fascio dicroico, DM = specchio dicroico, OB = obiettivo obiettivo, CO = condensatore, F = filtro, PD = fotodiodo. (B) Questo pannello mostra la parte di eccitazione laser. Il raggio a doppio colore dall'uscita laser viene prima separato con ogni raggio collimato ed espanso e successivamente combinato e diretto nel corpo del microscopio. (C) Questo pannello mostra la raccolta trasmessa con un condensatore. (D) Questo pannello mostra la parte di rilevamento SRS. Il fotodiodo e il filtro sono montati su una scatola schermata con due connettori BNC femmina. Il connettore BNC inferiore è per la tensione di polarizzazione inversa e il connettore BNC più alto è per l'uscita del segnale di corrente all'amplificatore lock-in terminato con 50 Ω. (E) Questo pannello mostra come il fotodiodo Si è montato all'interno della scatola schermata. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Imaging con colorante Raman di marcatori proteici distinti tramite immunolabeling. (A) Imaging immuno-eprSRS di α-tubulina in cellule HeLa. (B) Imaging immuno-eprSRS di NeuN nella corteccia cerebrale di topo fissata da PFA. (C) Imaging immuno-eprSRS di Vimentina nel tessuto FFPE renale umano. (D) Immagine volumetrica di GFAP colorato con MARS2145 in tessuto cerebrale di topo spesso 100 μm. La dimensione del passo in z era di 2 μm. (E) L'immagine renderizzata in volume di NEUN colorato di MARS2228 in tessuto cerebrale di topo spesso 40 μm. La dimensione del passo in z era di 1 μm. Barre di scala, 20 μm in (A), 50 μm in (B-C), 30 μm in (D-E). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Risultati rappresentativi dell'imaging tandem a 7 colori di ormoni e fattori di trascrizione sul tessuto insulare di topo congelato. Epr-SRS: Insulina (rilevata da Cy5, marcatore a cellule β, verde), Pdx1 (rilevata da MARS2228, fattore di trascrizione, rosso), Glucagone (rilevato da MARS2216, marcatore a cellule α, giallo), PP (rilevato da MARS2147, marcatore a cellule PP, blu). Fluorescenza: Somatostatina (Alexa488, marcatore a cellule δ, arancione), Nkx2.2 (Cy3, fattore di trascrizione, magenta), DAPI (nucleo, blu scuro). Barra della scala, 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
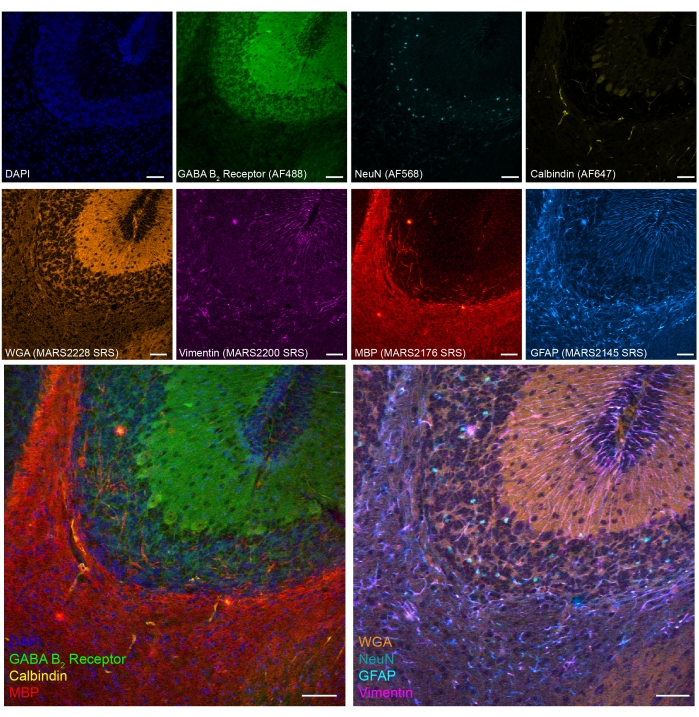
Figura 5: Risultati rappresentativi dell'imaging tandem a 8 colori di marcatori di tipo cellulare sulla sezione cerebrale di topo fissata da PFA. Fluorescenza: DNA (DAPI), GABA (acido γ-aminobutirrico) recettore B 2 (neuroni GABAergici, Alexa Fluor 488), nuclei neuronali (NeuN; neuroni, Alexa Fluor 568) e Calbindin (neuroni Purkinje, Alexa Fluor 647); epr-SRS: agglutinina del germe di grano (WGA; MARS2228), Vimentina (MARS2200), proteina basica mielinica (MBP; oligodendrociti, MARS2176) e GFAP (astrociti e cellule staminali neurali, MARS2145). Barra della scala, 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Anticorpi convalidati per immuno-eprSRS. Fare riferimento alla Tabella dei materiali per maggiori dettagli. Fare clic qui per scaricare questa tabella.
Materiale supplementare: proprietà di 8 sonde MARS funzionalizzate NHS-ester utilizzate. λabs e coefficienti di eccitazione dei coloranti MARS sono stati misurati in soluzione DMSO su spettrometro UV-Vis utilizzando una cuvetta di vetro da 1 cm come contenitore. Le sezioni trasversali Raman assolute dei coloranti MARS sono state determinate in DMSO confrontando il segnale epr-SRS dei coloranti MARS con la modalità di allungamento C−O standard (1030 cm-1) di metanolo. La sezione trasversale Raman assoluta per la modalità di allungamento C−O standard (1030 cm-1) del metanolo è stata riportata come 2,1 x 10-30 cm2 a 785 nm. Una sezione trasversale di 0,9 x 10-30 cm2 è stata stimata sotto la lunghezza d'onda della pompa di 860 nm per estrapolazione. Fare clic qui per scaricare questo file.
Discussione
Qui, presentiamo il protocollo immuno-eprSRS che è ampiamente applicabile ai tipi di tessuto comuni, compresi i tessuti di topo appena conservati, i tessuti umani FFPE e i tessuti di topo congelati. Immuno-eprSRS è stato convalidato per un pannello di epitopi in cellule e tessuti, come elencato nella Tabella 1. Questa piattaforma one-shot è particolarmente adatta per applicazioni in cui le strategie cicliche non funzionano bene. Ad esempio, la fluorescenza ciclica è impegnativa per i tessuti spessi poiché più cicli di immunolabeling 3D sono poco praticie lunghi 17. È anche molto probabile che introduca errori di registrazione a causa di cambiamenti istologici 3D non lineari11,17. Immuno-eprSRS supera le barriere pratiche della fluorescenza ciclica in un tale scenario e offre l'opportunità di rivelare reti di interazione proteica attraverso un grande volume17.
L'attuale multiplexity è principalmente limitata dalla disponibilità di anticorpi secondari. Mentre in questo protocollo, ci siamo concentrati sull'immunolabeling indiretto, in cui le sonde MARS sono coniugate agli anticorpi secondari, l'immunolabeling diretto e la colorazione delle lectine sono fattibili17. Dopo un'ulteriore validazione degli anticorpi primari con coloranti Raman, sono attesi 20 canali con coloranti Raman attualmente sviluppati 13,18,24. Inoltre, l'imaging di bersagli a bassissima abbondanza potrebbe essere difficile per epr-SRS a causa della sua sensibilità leggermente compromessa rispetto al sistema di fluorescenza confocale. A questo proposito, raccomandiamo di assegnare bersagli relativamente poco abbondanti a coloranti MARS più luminosi e bersagli a bassa espressione ai canali di fluorescenza.
Un aspetto critico del protocollo è l'accessibilità di strumenti e sonde. Per quanto riguarda la strumentazione, un microscopio SRS è generalmente composto da una sorgente laser a doppio colore con un modulatore ottico, un microscopio, un rilevatore di fotodiodi e un amplificatore lock-in per la demodulazione25. Ogni componente è disponibile in commercio con un costo totale leggermente superiore rispetto a un microscopio a fluorescenza a scansione laser a due fotoni. Un microscopio di ricerca SRS / fluorescenza multimodale completamente integrato è stato commercializzato26 utilizzando un laser a picosecondi simile a quello qui per l'eccitazione SRS e i set laser a onda continua (CW) per la fluorescenza. Questo sistema è facilmente applicabile per l'imaging vibrazionale multiplex nella ricerca biologica quotidiana. Per quanto riguarda le sonde MARS, le sonde MARS non sono ancora state commercializzate e richiedono alcune capacità di sintesi. In alternativa, molti fluorofori commerciali far-red (fare riferimento alla Extended Data Table 1 in L. Wei et al. Nature 201713) possono essere utilizzati per epr-SRS. Tuttavia, la multiplexity potrebbe essere compromessa. Inoltre, poiché le sonde MARS per natura sono piccole molecole organiche, l'immuno-eprSRS è simile all'immunofluorescenza in termini di colorazione dei tessuti. Pertanto, l'archivio di reagenti di affinità convalidati come gli anticorpi in immunofluorescenza può essere facilmente trasferito in applicazioni immuno-eprSRS.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Ruth A. Singer e Richard K.P. Benninger per aver fornito tessuti pancreatici di topo. W.M. riconosce il supporto di NIH R01 (GM128214), R01 (GM132860), R01 (EB029523) e US Army (W911NF-19-1-0214).
Materiali
| Name | Company | Catalog Number | Comments |
| 16% Paraformaldehyde, EM Grade | Electron Microscopy Sciences | 15710 | |
| α-tubulin | Abcam | ab18251 | Primary antibodies |
| α-tubulin | BioLegend | 625902 | Primary antibodies |
| β-III-tubulin | BioLegend | 657402 | Primary antibodies |
| β-III-tubulin | Abcam | ab41489 | Primary antibodies |
| β-tubulin | Abcam | ab131205 | Primary antibodies |
| Agarose, low gellling temperature | Sigma Aldrich | A9414 | For brain embedding |
| Anti-a-tubulin antibody produced in rabbit (α-tubulin) | Abcam | ab52866 | Primary antibodies |
| Anti-Calbindin antibody produced in mouse (Calbindin) | Abcam | ab82812 | Primary antibodies |
| Anti-GABA B receptor R2 antibody produced in guinea pig (GABA B receptor R2) | Millipore Sigma | AB2255 | Primary antibodies |
| Anti-GFAP antibody produced in goat (GFAP) | Thermo Scientific | PA5-18598 | Primary antibodies |
| Anti-Glucagon antibody produced in mouse (Glucagon) | Santa Cruz Biotechnology | sc-514592 | Primary antibodies |
| Anti-insulin antibody produced in guinea pig (insulin) | DAKO | IR00261-2 | Primary antibodies |
| Anti-MBP antibody produced in rat (MBP) | Abcam | ab7349 | Primary antibodies |
| Anti-NeuN antibody produced in rabbit (NeuN) | Thermo Scientific | PA5-78639 | Primary antibodies |
| Anti-Pancreatic polypeptide (PP) antibody produced in goat- Pancreatic polypeptide (PP) | Sigma Aldrich | SAB2500747 | Primary antibodies |
| Anti-Pdx1 antibody produced in rabbit (Pdx1) | Milipore | 06-1379 | Primary antibodies |
| Anti-Somatostatin antibody produced in rat (Somatostatin) | Abcam | ab30788 | Primary antibodies |
| Anti-Vimentin antibody produced in chicken (Vimentin) | Abcam | ab24525 | Primary antibodies |
| Band-pass filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| BNC cable | Thorlabs | 2249-C | Coaxial Cable, BNC Male / Male |
| Broadband dielectric mirror | Thorlabs | BB1-E03 | 750 - 1100 nm |
| C57BL/6J mice | Jackson Laboratory | 000664 | |
| Centrifuge | |||
| Condenser | Olympus | oil immersion, 1.4 N.A. | |
| Cytokeratin 18 | Abcam | ab7797 | Primary antibodies |
| Cytokeratin 18 | Abcam | ab24561 | Primary antibodies |
| DC power supply | TopWard | 6302D | Bias voltage is 64 V |
| Dichroic mount | Thorlabs | KM100CL | Kinematic Mount for up to 1.3" (33 mm) Tall Rectangular Optics, Left Handed |
| Donkey anti-Chicken IgY (H+L) | Jackson ImmunoResearch | 703-005-155 | Secondary antibodies for MARS conjugation |
| Donkey anti-Goat IgG (H+L) | Jackson ImmunoResearch | 705-005-147 | Secondary antibodies for MARS conjugation |
| Donkey anti-Guinea Pig IgG (H+L) | Jackson ImmunoResearch | 706-005-148 | Secondary antibodies for MARS conjugation |
| Donkey anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-005-151 | Secondary antibodies for MARS conjugation |
| Donkey anti-Rabbit IgG (H+L) | Jackson ImmunoResearch | 711-005-152 | Secondary antibodies for MARS conjugation |
| Donkey anti-Rat IgG (H+L) | Jackson ImmunoResearch | 712-005-153 | Secondary antibodies for MARS conjugation |
| Donkey anti-Sheep IgG (H+L) | Jackson ImmunoResearch | 713-005-147 | Secondary antibodies for MARS conjugation |
| DPBS | Fisher Scientific | 14-190-250 | |
| EpCAM | Abcam | ab71916 | Primary antibodies |
| Ethanol | Sigma Aldrich | 443611 | |
| Fast-speed look-in amplifier | Zurich Instruments | HF2LI | DC - 50 MHz |
| FFPE Kidney Sample | USBiomax | HuFPT072 | |
| Fibrillarin | Abcam | ab5821 | Primary antibodies |
| Giantin | Abcam | ab24586 | Primary antibodies |
| Glucagon | Santa Cruz Biotechnology | sc-514592 | Primary antibodies |
| H2B | Abcam | ab1790 | Primary antibodies |
| HeLa | ATCC | ATCC CCL-2 | |
| High O.D. bandpass filter | Chroma Technology | ET890/220m | Filter the Stokes beam and transmit the pump beam |
| Hydrophobic pen | Fisher Scientific | NC1384846 | |
| Insulin | ThermoFisher | 701265 | Primary antibodies |
| Integrated SRS laser system | Applied Physics & Electronics, Inc. | picoEMERALD | picoEMERALD provides an output pulse train at 1,064 nm with 6-ps pulse width and 80-MHz repetition rate, which serves as the Stokes beam. The frequency doubled beam at 532 nm is used to synchronously seed a picosecond optical parametric oscillator (OPO) to produce a mode-locked pulse train with five~6 ps pulse width (the idler beam of the OPO is blocked with an interferometric filter). The output wavelength of the OPO is tunable from 720–950 nm, which serves as the pump beam. The intensity of the 1,064-nm Stokes beam is modulated sinusoidally by a built-in EOM at 8 MHz with a modulation depth of more than 90%. The pump beam is spatially overlapped with the Stokes beam by using a dichroic mirror inside picoEMERALD. The temporal overlap between pump and Stokes pulse trains is achieved with a built-in delay stage and optimized by the SRS signal of pure D2O at the microscope. |
| Inverted laser-scanning microscope | Olympus | FV1200MPE | |
| Kinematic mirror mount | Thorlabs | POLARIS-K1-2AH | 2 Low-Profile Hex Adjusters |
| Lectin from Triticum vulgaris (wheat) | Sigma Aldrich | L0636-5 mg | |
| Long-pass dichroic beam splitter | Semrock | Di02-R980-25x36 | 980 nm laser BrightLine single-edge laser-flat dichroic beamsplitter |
| MAP2 | BioLegend | 801810 | Primary antibodies |
| Microscopy imaging software | Olympus | FluoView | |
| NanoQuant Plate | Tecan | For absorbance-based, small volume analyses in a plate reader. | |
| Normal donkey serum | Jackson ImmunoResearch | 017-000-121 | |
| NucBlue Fixed Cell ReadyProbes Reagent (DAPI) | Thermo Scientific | R37606 | |
| Nunc 4-Well Dishes | Fisher Scientific | 12-566-300 | |
| Objective lens | Olympus | XLPlan N | x25, 1.05-NA, MP, working distance = 2 mm |
| Paint brush | |||
| Periscope assembly | Thorlabs | RS99 | includes the top and bottom units, Ø1" post, and clamping fork. |
| pH meter | |||
| Plate reader | Tecan | Infinite 200 PRO | An easy-to-use multimode plate reader. Absorbance measurement capabilities over a spectral range of 230–1000 nm. |
| ProLong Gold antifade reagent | Thermo Scientific | P36930 | |
| PSD95 | Invitrogen | 51-6900 | Primary antibodies |
| Sephadex G-25 Medium | GE Life Sciences | 17-0033-01 | gel filtration resin for desalting and buffer exchange |
| Shielded box with BNC connectors | Pomona Electronics | 2902 | Aluminum Box With Cover, BNC Female/Female |
| Si photodiode | Thorlabs | FDS1010 | 350–1100 nm, 10 mm x 10 mm Active Area |
| Synapsin 2 | ThermoFisher | OSS00073G | Primary antibodies |
| Tissue Path Superfrost Plus Gold Slides | Fisher Scientific | 22-035813 | Adhesive slide to attract and chemically bond fresh or formalin-fixed tissue sections firmly to the slide surface (tiisue bindling glass slides) |
| Triton X-100 | Fisher Scientific | BP151-500 | |
| Vibratome | Leica | VT1000 | |
| Vimentin | Abcam | ab8069 | Primary antibodies |
| Xylenes | Sigma Aldrich | 214736 |
Riferimenti
- Goltsev, Y., et al. Deep profiling of mouse splenic architecture with CODEX multiplexed imaging. Cell. 174 (4), 968-981 (2018).
- Taube, J. M., et al. The Society for Immunotherapy of Cancer statement on best practices for multiplex immunohistochemistry (IHC) and immunofluorescence (IF) staining and validation. Journal for Immunotherapy of Cancer. 8 (1), 000155 (2020).
- Lewis, S. M., et al. Spatial omics and multiplexed imaging to explore cancer biology. Nature Methods. 18 (9), 997-1012 (2021).
- Bodenmiller, B. Multiplexed epitope-based tissue imaging for discovery and healthcare applications. Cell Systems. 2 (4), 225-238 (2016).
- Hickey, J. W., et al. Spatial mapping of protein composition and tissue organization: a primer for multiplexed antibody-based imaging. Nature Methods. , (2021).
- Lin, J. -. R., et al. Highly multiplexed immunofluorescence imaging of human tissues and tumors using t-CyCIF and conventional optical microscopes. eLife. 7, 31657 (2018).
- Black, S., et al. CODEX multiplexed tissue imaging with DNA-conjugated antibodies. Nature Protocols. 16 (8), 3802-3835 (2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. MIBI-TOF: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Science Advances. 5 (10), 5851 (2019).
- Gerdes, M. J., et al. Highly multiplexed single-cell analysis of formalin-fixed, paraffin-embedded cancer tissue. Proceedings of the National Academy of Sciences of the United States of America. 110 (29), 11982 (2013).
- Angelo, M., et al. Multiplexed ion beam imaging of human breast tumors. Nature Medicine. 20 (4), 436-442 (2014).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 117 (52), 33455 (2020).
- Wei, L., et al. Super-multiplex vibrational imaging. Nature. 544, 465 (2017).
- Wei, L., Min, W. Electronic preresonance stimulated Raman scattering microscopy. The Journal of Physical Chemistry Letters. 9 (15), 4294-4301 (2018).
- Shi, L., et al. Electronic resonant stimulated Raman scattering micro-spectroscopy. The Journal of Physical Chemistry B. 122 (39), 9218-9224 (2018).
- Fujioka, H., et al. Multicolor activatable Raman probes for simultaneous detection of plural enzyme activities. Journal of the American Chemical Society. 142 (49), 20701-20707 (2020).
- Shi, L., et al. Highly-multiplexed volumetric mapping with Raman dye imaging and tissue clearing. Nature Biotechnology. , (2021).
- Miao, Y., Qian, N., Shi, L., Hu, F., Min, W. 9-Cyanopyronin probe palette for super-multiplexed vibrational imaging. Nature Communications. 12 (1), 4518 (2021).
- Miao, Y., Shi, L., Hu, F., Min, W. Probe design for super-multiplexed vibrational imaging. Physical Biology. 16 (4), 041003 (2019).
- Qian, N., Min, W. Super-multiplexed vibrational probes: Being colorful makes a difference. Current Opinion in Chemical Biology. 67, 102115 (2022).
- Klimas, A., et al. Nanoscale imaging of biomolecules using molecule anchorable gel-enabled nanoscale in-situ fluorescence microscopy. Nature Portfolio. , (2021).
- Shi, L., et al. Super-resolution vibrational imaging using expansion stimulated Raman scattering microscopy. bioRxiv. , (2021).
- Benninger, R. K. P., Hodson, D. J. New understanding of β-cell heterogeneity and in situ islet function. Diabetes. 67 (4), 537 (2018).
- Hu, F., et al. Supermultiplexed optical imaging and barcoding with engineered polyynes. Nature Methods. 15 (3), 194-200 (2018).
- Hu, F., Shi, L., Min, W. Biological imaging of chemical bonds by stimulated Raman scattering microscopy. Nature Methods. 16 (9), 830-842 (2019).
- . Coherent Raman Scattering Microscope Available from: https://www.leica-microsystems.com/products/confocal-microscopes/p/leica-tcs-sp8-cars/ (2022)
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Tutti i diritti riservati
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.
