Method Article
In Utero Electroporation of Multiaddressable Genome-Integrating Color (MAGIC) Markers to Individualize Cortical Mouse Astrocytes
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Gli astrociti piastrellano uniformemente la corteccia cerebrale, rendendo difficile l'analisi della loro complessa morfologia a livello cellulare. Il protocollo qui fornito utilizza l'etichettatura multicolore basata sull'elettroporazione in utero per singolo astrociti corticali e analizzarne il volume e la morfologia con una pipeline di analisi delle immagini intuitiva.
Abstract
Gli astrociti protoplasmatici (PrA) situati nella corteccia cerebrale del topo sono strettamente giustapposti, formando una matrice tridimensionale apparentemente continua negli stadi adulti. Finora, nessuna strategia di immunosottenzione può distinguerli e segmentarne la morfologia negli animali maturi e nel corso della corticogenesi. I PrA corticali provengono da progenitori situati nel pallio dorsale e possono essere facilmente mirati usando in utero l'elettroporazione di vettori integrativi. Qui viene presentato un protocollo per etichettare queste cellule con la strategia MAGIC (Genome-Integrating Color) Markers multiindirittabile, che si basa sulla trasposizione piggyBac/Tol2 e sulla ricombinazione Cre/lox per esprimere stocasticamente proteine fluorescenti distinte (blu, ciano, giallo e rosso) indirizzate a specifici compartimenti subcellulari. Questa strategia di mappatura del destino multicolore consente di contrassegnare in situ i progenitori corticali vicini con combinazioni di marcatori di colore prima dell'inizio della gliogenesi e di tracciare i loro discendenti, compresi gli astrociti, dagli stadi embrionali a quelli adulti a livello di singola cellula. L'etichettatura semi-sparsa ottenuta regolando la concentrazione di vettori elettroporati e i contrasti di colore forniti dai marcatori di colore multiindirizzabili per l'integrazione del genoma (MAGIC Markers o MM) consentono di individualizzare gli astrociti e di distinguerne il territorio e la morfologia complessa nonostante la loro densa disposizione anatomica. Qui è presentato un flusso di lavoro sperimentale completo che include i dettagli della procedura di elettroporazione, l'acquisizione di stack di immagini multicanale tramite microscopia confocale e la segmentazione tridimensionale assistita da computer che consentirà allo sperimentatore di valutare il volume e la morfologia dei singoli PrA. In sintesi, l'elettroporazione dei marcatori MAGIC fornisce un metodo conveniente per etichettare individualmente numerosi astrociti e ottenere l'accesso alle loro caratteristiche anatomiche in diversi stadi di sviluppo. Questa tecnica sarà utile per analizzare le proprietà morfologiche degli astrociti corticali in vari modelli di topi senza ricorrere a croci complesse con linee di reporter transgeniche.
Introduzione
Gli astrociti svolgono numerose funzioni vitali nello sviluppo cerebrale e nella fisiologia1. Oltre al loro ruolo alla barriera ematico-encefalica dove regolano l'assorbimento dei nutrienti e il flusso sanguigno, contribuiscono attivamente alla formazione e alla funzione della sinapsi producendo neuromodulatori in grado di alterare l'attività e il comportamento neuronali2. Inoltre, la disfunzione degli astrociti contribuisce a una varietà di disturbineurologici 3. Gli astrociti situati nella corteccia cerebrale mostrano una morfologia elaborata che consente un ampio contatto con i processi neuronali. Questi contatti, essenziali per la funzione del circuito, controllano anche la morfogenesi degli astrociti e la sinaptogenesi attraverso le proteine di adesione cellulare4. I neuroscienziati hanno bisogno di strumenti convenienti e robusti per indagare lo sviluppo degli astrociti e la morfogenesi nei loro modelli neurologici di interesse. Tuttavia, a causa della stretta apposizione di astrociti ai loro vicini e della loro piastrellatura tridimensionale uniforme, è difficile trovare astrociti corticali e valutare in modo completo la loro morfologia usando immunomarcatori.
Attualmente, due principali strategie di ingegneria genetica consentono l'etichettatura e l'individualizzazione degli astrociti corticali in situ: scarsa attivazione del reporter nelle linee transgeniche del topo o transgenesi somatica usando l'elettroporazione dei plasmidi reporter. La prima strategia si basa sull'allevamento di una linea di topi reporter floxed con topi che esprimono una forma induttiva di Cre ricombinasi attivata specificamente negli astrociti al momento della consegna del tamoxifene (ad esempio, Aldh1l1-CreERT25). Diversi svantaggi sono associati a questa strategia. In primo luogo, l'allevamento di topi transgenici richiede un gran numero di animali e sono tipicamente necessari test multipli per determinare la dose corretta di tamoxifene per fornire un'etichettatura adeguatamente scarsa degli astrociti corticali. L'analisi dei fenotipi di astrociti corticali in un modello genetico di interesse del topo richiederà ancora più allevamento e consumo di topi. Inoltre, in utero l'iniezione di tamoxifene è nota per interferire con il parto, rendendo questa strategia difficile da applicare allo studio delle prime fasi dello sviluppo degli astrociti. L'elettroporazione del DNA in vivo è una strategia alternativa priva di tamoxifene che si basa su un numero minimo di animali6. Eseguito sia allo stadio embrionale che postnatale, questo approccio consiste nell'iniettare plasmidi reporter nei ventricoli laterale dei roditori seguiti da impulsi elettrici che creano pori nella membrana cellulare, permettendo così al DNA di entrare nelle cellule progenitrici che rivestono il ventricolo. Successivamente, i transgeni reporter trasportati dai plasmidi elettroporati vengono lavorati dalla macchina cellulare mirata ed espressi7. Due metodi di elettroporazione sono stati precedentemente descritti per etichettare gli astrociti corticali del topo: 1) Etichettatura postnatale degli astrociti mediante elettroporazione (PALE), che si basa sull'elettroporazione di 1-2 plasmidi reporter episomiali monocolore nelle prime fasi postnatali4; 2) La strategia StarTrack basata sull'elettroporazione in utero (IUE) di più plasmidi reporter integrativi monocolore8,9,10. Sebbene queste due tecniche etichettino efficacemente pra nella corteccia cerebrale, presentano anche alcune limitazioni. Nella loro versione iniziale, entrambi i metodi si basano su un promotore di proteine acide fibrillari gliali (GFAP) per guidare l'espressione negli astrociti, il che può polarizzare l'etichettatura verso glia radiali così come astrociti piali e reattivi che esprimono GFAP più fortemente del normale riposo PrA11,12. Per quanto riguarda PALE, altri svantaggi sono la fase avanzata dell'elettroporazione, che impedisce l'etichettatura del PrA primogenito (o di quelli originati da progenitori di delaminazione precoce) e l'analisi delle prime fasi dello sviluppo di astroglia, e l'uso di vettori episomici che si diluiscono attraverso successive divisioni durante la massiccia proliferazione che PrA subisce durante la prima settimana postnatale13,14. A differenza di PALE, StarTrack si basa sull'elettroporazione embrionale dei plasmidi reporter integrativi che consentono di tracciare il contributo dei progenitori sia embrionali che postnatali a PrA. Uno schema StarTrack aggiornato basato sul promotore ubiquitina C (UbC-StarTrack) raggiunge una più ampia espressione di reporter fluorescenti sia nella discesa neuronale che gliale (astrociti inclusi) dei progenitori neurali15,16,17. Tuttavia, nella sua versione attuale, l'implementazione di questo approccio è complessa, in quanto si basa su una miscela equimolare di 12 plasmidi distinti che esprimono sei proteine fluorescenti (FP) con eccitazione parziale e sovrapposizione di spettri di emissione.
Presentato qui è un semplice metodo di etichettatura multicolore basato sull'elettroporazione in utero utilizzando costrutti di reporter integrativi guidati da un promotore forte e ampiamente attivo per intasare gli astrociticorticali 14. Inoltre, viene fornita una semplice pipeline di analisi delle immagini utilizzando sia il software di analisi delle immagini concesso in licenza (ad esempio, Imaris) che l'accesso aperto (Vaa3D18 , 19,20) per segmentare rispettivamente il volume territoriale degli astrociti e l'arborizzazione. Rispetto ai metodi descritti in precedenza, questa strategia si basa esclusivamente su 1-2 transgeni integrativi multicolore Multiaddressable Genome-Integrating Color Markers (MAGIC Markers o MM21) diretti al compartimento cellulare citoplasmatico e (opzionalmente) nucleare la cui espressione è guidata da un promotore CAG sintetico composto da un potenziatore di citomegalovirus, promotore di pollo β-actina e coniglio β-globin splice acceptor site22. Ciò consente l'etichettatura e il tracciamento degli astrociti corticali, dagli stadi postnatali embrionali a quello tardo, indipendentemente dall'espressioneGFAP 14,23. Ognuno di questi transgeni porta i seguenti quattro FP distinti: eBFP, mTurquoise2/mCerulean, EYFP e tdTomato/mCherry, che mostrano una sovrapposizione spettrale minima che può essere facilmente aggirata con 1) Acquisizione sequenziale del canale; 2) Potenza di eccitazione ottimizzata e guadagno di raccolta; e 3) Filtri dicroici specifici per raccogliere finestre di emissione FP strette. La strategia MM utilizza laricombinazione Cre/ lox con una ricombinasi cre auto-incisibile (seCre) per guidare l'espressione stocastica di FP in una popolazione cellulare. Una singola copia del transgene MM esprime FP in modo reciprocamente esclusivo, mentre più transgeni danno origine a combinazioni FP, creando dozzine di tonalità distinte. L'integrazione genomica dei transgeni è guidata dal sistema di trasposizione piggyBac (PB) o Tol224,25,26. Pertanto, attraverso l'elettroporazione utero, il toolkit MM e il 'mosaico' multicolore che genera consentono la marcatura simultanea di più progenitori corticali adiacenti e il tracciamento della loro discesa gliale, compresi gli astrociti corticali, per lunghi periodi. I contrasti di colore risultanti dall'espressione di FP distinti consentono la delimitazione del contorno di PrA e successivamente estraggono informazioni chiave sul loro volume territoriale (usando IMARIS) e sulla morfologia complessa (usando Vaa3D). La strategia multicolore presentata in dettaglio qui è un metodo comodo e robusto che dà un accesso rapido e facile alla superficie e alla morfologia degli astrociti corticali nei topi di tipo selvaggio in varie fasi dello sviluppo, ed è facilmente adattabile per indagare le caratteristiche anatomiche degli astrociti nei modelli di topi di malattie neurologiche senza utilizzare linee di reporter transgeniche.
Protocollo
Tutte le procedure relative agli animali qui descritte sono state eseguite in conformità con gli orientamenti istituzionali. I protocolli sugli animali sono stati approvati dal consiglio etico per la sperimentazione animale di Charles Darwin (CEEACD/N°5).
1. Preparazione di plasmidi senza endotossina per marcatori MAGIC in elettroporazione utero
- Trasformazione batterica
- Sul ghiaccio, scongelare le cellule competenti DH5 alpha immagazzinate a -70 °C.
- Riscaldare le piastre di agar contenenti l'antibiotico appropriato (100 μg/mL di ampicillina o 50 μg/mL di kanamicina) a 37 °C.
- Aggiungere 1 μL di 5-50 ng di DNA plasmide magic markers in 10 μL di cellule alfa competenti DH5 scongelate e incubare sul ghiaccio per 10 minuti senza miscelazione.
- Per la trasformazione dello shock termico, posizionare l'aliquota in un bagno d'acqua di 42 °C per 45 s, quindi posizionare immediatamente sul ghiaccio e attendere 3-5 minuti.
- In condizioni sterili, aggiungere 230 μL di mezzo SOC e incubare per 1 h a 37 °C.
- Stendere il contenuto dell'aliquota sulla piastra di agar e incubare durante la notte a 37 °C.
- Coltura plasmide
- La mattina seguente, in condizioni sterili, prendere una colonia dalla piastra di agar e metterla in un tubo da 14 ml contenente 2 ml di mezzo LB con antibiotico appropriato. Lasciare incubare per la giornata a 37 °C in un'incubatrice di scuotimento a 300 giri/min.
- Alla fine della giornata, semina 300 mL di LB con antibiotico usando la coltura iniziale di 2 mL dal passo 1.2.1 e incubare durante la notte a 37 °C in un'incubatrice di scuotimento a 300 giri/min.
- Preparazione del DNA plasmide
- La mattina seguente procedere con la purificazione del DNA plasmide magic markers (cioè PB-CAG-Cytbow e Tol2-CAG-Nucbow, nonché dei plasmidi guidati da CAG che esprimono trasposasi PB e Tol2 e Cre ricombinasi) utilizzando un kit maxiprep privo di endotossina seguendo il protocollo del produttore.
- Elute il DNA in 200 μL di acqua sterile e stimarne la concentrazione utilizzando uno spettrofotometro prima dello stoccaggio a -20 °C.
2. Preparazione per marcatori MAGIC in elettroporazione utero (MM IUE)
- Preparazione della soluzione
- Riscaldare 30 mL di soluzione salina allo 0,9% a 37 °C in un bagno d'acqua e tenerlo caldo per tutta la durata dell'intervento chirurgico.
- Preparare un mix plasmide contenente PB-CAG-Cytbow e Tol2-CAG-Nucbow (concentrazione finale, 0,8 μg/μL ciascuno), trasposasi PB e Tol2 (concentrazione finale, 0,4 μg/μL ciascuna), CAG-seCre (concentrazione finale, 0,16 μg/μL) e colorante verde veloce allo 0,01% in PBS senza Ca2+ e Mg2+.
NOTA: Ai fini della ricostruzione anatomica degli astrociti, il costrutto Tol2-CAG-Nucbow può essere omesso. Tuttavia, questo plasmide è utile per distinguere doppietti di astrociti strettamente giustapposti e quando si utilizzano marcatori MAGIC per sondare le relazioni clonali tra gli astrociti14. - Preparare una soluzione anestetica contenente 100 μL di ketamina (100 mg/mL) e 100 μL di xiazina (20 mg/mL) diluita in 2 mL di soluzione salina.
- Preparare la soluzione analgesica diluire la soluzione di 0,3 mg/mL di soluzione di buprenorfina 1:10 in soluzione salina.
- Preparazione del materiale chirurgico
- Sterilizzare gli utensili chirurgici (vedi Tabella dei materiali)ad alta temperatura in uno sterilizzatore di perline di vetro o equivalente.
- Posizionare una goccia di gel elettrodo in un piatto da 35 mm.
- Inserire la micropipetta nel supporto del microiniettore, rompere la punta della micropipetta e aspirare alla soluzione di DNA.
- Posizionare un calo di 1 μL della soluzione Fast Green (0,01%) nel coperchio di un piatto da 3 cm e, utilizzandolo come riferimento, regolare il diametro della punta della micropipetta rompendo la punta con forcep fini. Regolare il parametro di pressione in modo che le gocce di dimensioni equivalenti siano prodotte dal microiniettore, consentendo così l'erogazione di circa 1 μL di soluzione di DNA per iniezione.
- Preparazione del topo femmina incinta
- Pesare il topo incinta RjOrl:SWISS.
- Eseguire un'iniezione intraperitoneale con 12,5 μL per grammo di peso corporeo (BW) di soluzione anestetica. Attendere 5 minuti e verificare che il mouse sia addormentato pizzicando il dito del mouse.
- Una volta che l'animale non risponde per pizzicare, iniettarlo per via sottocutanea con 1,6 μL / g di BW di soluzione analgesica.
- Aggiungere una goccia di gel oculare su ogni occhio per evitare l'asciugatura durante l'intervento chirurgico e posizionare la pancia animale sul tampone riscaldante.
- Radere delicatamente l'addome, pulirlo con un tampone imbevuto di iodio e sanificare l'area rasata con un tampone alcolico.
- Disporre un campo operatorio posizionando impacchi sterili intorno all'area rasata, pulita e igienizzata.
3. In utero elettroporazione (IUE)
- Iniezione intraventricolare
- Tagliare un'incisione verticale di 2 cm lungo la linea mediana a partire dalla parte inferiore dell'addome, attraverso la pelle e quindi attraverso il muscolo sottostante. Esporre le corna uterine manipolando delicatamente i sacchetti embrionali e valutare la posizione della cervice e il numero di sacchetti embrionali su ciascun lato della cervice.
- Orientare il cervello dell'embrione E15.5 per essere elettroporato per vedere il bregma, facilmente riconoscibile come la sua posizione corrisponde alla giunzione dei tre principali vasi sanguigni che corrono lungo le fessure cerebrali.
- Immaginate una linea virtuale tra il bregma (visibile attraverso il cranio) e l'occhio; introdurre la micropipetta tra la linea virtuale e la fessura longitudinale, quindi premere il pedale dell'iniettore per fornire 1 μL di soluzione di DNA nel ventricolo laterale dell'emisfero mirato.
NOTA: Quando viene iniettato nella posizione corretta, il ventricolo laterale riempito appare blu, indicando che è stato riempito con la soluzione di DNA.
- Elettroporazione
- Applicare gli elettrodi (vedi Tabella dei materiali),precedentemente immerso nel gel dell'elettrodo, su entrambi i lati dell'embrione iniettato con l'anodo che copre l'emisfero iniettato. Premere il pedale dell'elettroporatore per fornire una serie di quattro impulsi da 50 ms da 35 V, ciascuno separato da un intervallo di 950 ms.
- Smorzare l'embrione con soluzione salina riscaldata.
NOTA: Gli embrioni devono essere mantenuti umidi utilizzando una soluzione salina riscaldata durante l'intera operazione chirurgica e l'utero non deve asciugarsi. - Ripetere i passaggi 3.1.2–3.2.2 per ogni embrione.
- Una volta che tutti gli embrioni mirati sono stati elettroporati, sostituire le corna uterine nell'addome spingendole delicatamente con le flessione nella loro posizione originale. Riempire la cavità addominale con soluzione salina riscaldata per evitare che l'utero si asciughi mentre vengono fatte le suture.
- Prima chiudi il muscolo addominale con una sutura assorbibile continua, quindi la pelle con più punti individuali (~ 10) usando la sutura 4-0.
- Metti l'animale in una gabbia pulita, sdraiato su un tovagliolo di carta pulito e gira l'animale dall'altra parte ogni 5-10 minuti fino a quando non si sveglia e inizia a muoversi da solo.
- Valuta lo stato dell'animale il giorno seguente, specialmente se è stato fatto un nido.
NOTA: Non è richiesto alcun trattamento post-chirurgico. In assenza di un nido, qualsiasi segno di dolore (ad esempio, prostrazione, pelliccia arruffata) e / o sanguinamento pesante, l'animale deve essere eutanasiato senza indugio.
4. Raccolta e sessatura dei tessuti
- Raccolta dei tessuti
- Iniettare fenobarbital (100 mg/kg di PW) intraperitonealmente per l'anestesia terminale al momento della raccolta desiderato. In questo caso, i tempi di raccolta erano ai giorni postnatali (P) P4, P7 e P21.
- Eseguire la perfusione intracardiaca utilizzando una soluzione a base di paraformaldeide prefabbrota a freddo.
- Sezionare il cervello e posizionarlo durante la notte a 4 °C nella soluzione a base di paraformaldeide per la postfissazione.
- Istologia
- La mattina seguente sciacquare il cervello 3 volte per 10 minuti con 1x PBS.
- Incorporare il cervello nel 3% di agarosio sciolto in 1x PBS.
- Tagliare sezioni da 80 μm utilizzando un microtomo a lama vibrante.
- Raccogliere sezioni in una piastra da 24 pozza di pozzo precompilato con 1x PBS.
- Montare sezioni nel supporto di montaggio (vedere Tabella dei materiali) tra la diapositiva e il coverslip. Mantenere le diapositive montate a -20 °C per una conservazione ottimale delle proteine fluorescenti e una conservazione a lungo termine.
5. Imaging confocale multicanale
- Impostazioni del microscopio
- Impostare la configurazione del microscopio confocale per eccitare separatamente le linee laser mCerulean/mTurquoise2, EYFP, tdTomato/mCherry utilizzando rispettivamente linee laser da 440, 515 e 559 nm.
- Utilizzare un obiettivo NA 20x 0,8 (o NA superiore) e regolare il campionamento XY e la dimensione del passo Z in base ai criteri di Nyquist.
NOTA: Le immagini acquisite con una risoluzione più elevata (ad esempio, l'obiettivo petrolifero 60x 1.4 NA) e gli algoritmi di deconvoluzione consentiranno la ricostruzione dei dettagli più fini degli arbitri astrociti. Tuttavia, lo sperimentatore dovrebbe tenere presente che i migliori processi di astrociti potrebbero non essere risolti dall'imaging ottico convenzionale.
- Acquisizione di immagini
- Trova gli astrociti più luminosi nell'area elettroporata.
- Poiché le celle etichettate situate vicino alla superficie potrebbero apparire più luminose di quelle più profonde nella sezione, regolare le impostazioni di acquisizione sulle celle di superficie per evitare di saturare le immagini.
- Regola le impostazioni di acquisizione separatamente per ciascuno dei tre canali, assicurandoti al contempo di evitare la saturazione dei pixel usando HiLo LUT, che visualizza valori zero come valori di pixel blu e massimi come rossi.
NOTA: un'immagine adeguatamente bilanciata dovrebbe visualizzare solo pochi pixel blu e quasi nessun pixel rosso. - Acquisire pile Z piastrellate da 1.024 x 1.024 pixel utilizzando lo stadio motorizzato del microscopio, con una sovrapposizione del 10% tra pile adiacenti per consentire successivamente ricostruzioni a mosaico dell'area elettroporata o della zona di interesse.
6. Segmentazione del volume territoriale degli astrociti
NOTA: Questo viene eseguito utilizzando un programma software commerciale (ad esempio, IMARIS).
-
Preparazione del set di dati
- Ritagliare gli astrociti etichettati all'interno della ricostruzione 3D (250 x 250 pixel) selezionando le celle interamente racchiuse nella sezione del tessuto immaginato.
NOTA: È anche possibile lavorare direttamente su volumi più grandi se il computer utilizzato è in grado di gestirli. - Fate clic sul pulsante Superficie blu (Blue Surface) nella barra degli strumenti Oggetto (Object) della vista Supera (Surpass) per creare una nuova superficie per ogni astrocita.
- Per ottenere un migliore contrasto di visualizzazione, regolare innanzitutto i valori minimi e massimi di contrasto del colore facendo clic su Modifica | Visualizzate l'opzione Regolazione visualizzazione (Display Adjustment) e trascinate entrambe le maniglie. Se necessario, modificare il colore del canale facendo clic direttamente sul nome del canale nella finestra Regolazione visualizzazione per selezionare un nuovo colore.
NOTA: Vengono sempre lavorati con lo stesso display a colori per evitare distorsioni visive. - Clicca su Modifica | Proprietà immagine per indicare manualmente la dimensione voxel in micrometri. Se la dimensione del voxel non è accurata, i calcoli del volume non saranno corretti.
- Salvare le nuove impostazioni.
- Ritagliare gli astrociti etichettati all'interno della ricostruzione 3D (250 x 250 pixel) selezionando le celle interamente racchiuse nella sezione del tessuto immaginato.
-
Segmentazione superficiale
- Utilizzate l'icona Blu per introdurre una superficie. Nell'elenco Scena verrà visualizzata un'icona e una casella Superficie. Una casella Volume consente di visualizzare/nascondere il set di dati.
- Selezionate la linea di superficie e fate clic su Salta creazione automatica (Skip Automatic Creation) | Modificare manualmente.
- Clicca su Contour | Visibilità e fare clic su Nessuno. Quindi passare alla visualizzazione Seleziona e fare clic sul pulsante Disegno.
- Fate clic su Modalità (Mode) per selezionare la modalità di disegno.
- Utilizzate lo strumento di disegno semiautomatico Isoline rapido ed efficiente. Definire il contorno della cella spostando il puntatore del mouse su di esso. Se l'anteprima dell'isolinea non corrisponde al contorno dell'astrocita, regolare la posizione del puntatore del mouse. Per convalidare l'anteprima, fare clic con il pulsante sinistro del mouse sulla selezione. Per correggere potenziali errori, utilizzare la finestra Scheda per eliminare il contorno del piano Z corrente oppure premere CTRL+Z.
- Uso della sezione | Posizionate per navigare tra i piani Z, spostate il piano successivo modificando il numero del piano Z e iniziate un nuovo contorno. Iniziare preferenzialmente dal centro della cella e quindi spostarsi alle estremità.
- Dopo aver disegnato i contorni in tutti i piani Z contenenti l'astrocita, fate clic su Crea superficie (Create Surface). Prima di creare una superficie, verificare che tutti i componenti siano collegati. In caso meno, selezionare quelli più grandi o connetterli prima di esportare i dati del volume.
- Controllare i dati quantitativi visualizzati nel Pannello dati ed esportare le statistiche in formato foglio di calcolo, risparmiando valore e unità di volume.
- Salvate la superficie con un nuovo nome per accedervi in un secondo momento.
7. Tracciare l'arborizzazione degli astrociti
NOTA: Questo viene fatto utilizzando il programma software ad accesso aperto Vaa3D.
- Preparazione del set di dati
- Carica lo stack di immagini e cerca astrociti isolati o astrociti vicini che mostrano colori distinti.
NOTA: La risoluzione della ricostruzione può essere migliorata aumentando l'obiettivo NA e il campionamento. Alla fine, tuttavia, a causa del limite di risoluzione della microscopia confocale, i migliori processi di astrociti non possono essere tracciati con precisione. Si dovrebbe fare attenzione a evitare un sovrasegmentazione oltre la risoluzione delle immagini acquisite. - Usando fiji, ritaglia pile di immagini da 250 x 250 pixel intorno a ogni astrocita.
- Poiché la traccia Vaa3D viene eseguita su un solo colore, selezionare un canale e disattivare gli altri canali.
- Converti l'immagine in RGB e salvala in .tiff formato.
- Carica lo stack di immagini e cerca astrociti isolati o astrociti vicini che mostrano colori distinti.
- Tracciamento arbor
- Apri l'immagine RGB in Vaa3D. Passare a Immagine | Dati | La geometria quindi il ricampionamentodelle immagini e la regolazione dei valori delle dimensioni voxel X/Y e Z.
NOTA: Durante la ricostruzione degli arbitri di astrociti, si deve fare attenzione a non tracciare i dettagli più fini della risoluzione fornita dalle immagini. - Aprire una finestra 3D facendo clic su Visualizza | visualizzatore 3D per l'intera immagine. Fare clic con il pulsante destro del mouse su un'immagine 3D e scegliere 1 clic con il pulsante destro del mouse per definire il marcatore. Posizionare il puntatore del mouse con il cursore al centro dell'astrocita e quindi fare clic con il pulsante destro del mouse per impostare un marcatore.
NOTA: clicca su Escape per avere di nuovo accesso alla vista 3D. - Vai a Advanced | 3D tracing | Vaa3D-Neuron2-auto-tracing.
- Nella finestra appena aperta selezionare il canale su cui applicare il plug-in.
- Scegli una soglia di sfondo (spesso tra 30 e 90). Adatta questo valore per ogni astrocita, se necessario.
- Deselezionare Raggio da 2D e mantenere selezionato l'esempio di ridimensionamento automatico e ricampionare automaticamente.
- Impostare cnn_type a 3, length_thresh a 1 e SR_ratio a 0,1.
- Dopo aver fatto clicsu OK , assicurarsi che il plug-in generi automaticamente due file in base al nome originale. Verrà aggiunto il suffisso –ini.swc e -coordinateX-coordinateY-coordinateZ-app2.swc. Aggiungere manualmente un'etichetta distintiva a questi file di nomi per non sovrascriverli mentre si continua a eseguire il plug-in.
- Aprire Object Manager, passare a Neuron | Struttura linea, selezionare l'astrocita tracciato, fare clic su Modalità divisualizzazione e selezionare Modalità linea sempre.
- Torna alla finestra 3D, dove appare uno scheletro dell'astrocita.
NOTA: se la segmentazione e il segnale non corrispondono, ripetere i passaggi da 7.2.3 a 7.2.10 e regolare la soglia fino a quando non lo fanno. Assicurarsi di reinsediare gli altri parametri man mano che vengono reimpostati al riavvio del plug-in. - Fare clic con il pulsante destro del mouse sullo scheletro e selezionare il neurone di prima | linea #1... APP2_Tracing per accedere alle misurazioni quantitative che appariranno su una nuova finestra Superficie| Annotazione oggetto.
NOTA: A causa della limitata risoluzione della microscopia ottica, le misurazioni visualizzate sotto gli elementi elencati Numero di rami e numero di biforcazioni forniscono solo una stima approssimativa della complessità dell'arbor degli astrociti.
- Apri l'immagine RGB in Vaa3D. Passare a Immagine | Dati | La geometria quindi il ricampionamentodelle immagini e la regolazione dei valori delle dimensioni voxel X/Y e Z.
Risultati
L'elettroporazione dei marcatori MAGIC nei progenitori corticali embrionali consente l'etichettatura degli astrociti dalle prime alle ultime fasi dello sviluppo della corteccia cerebrale (Figura 1). Questi astrociti sono stati trovati in tutti gli strati corticali in vari stadi postnatali (P4, P7, P21) mentre si disperdevano ampiamente nell'intera corteccia cerebrale. Sono stati valutati con immagini confocali piastrellate acquisite con un obiettivo 20x 0.8 NA (o NA superiore) e assemblate come ricostruzioni Z-stack(Figura 2). L'etichettatura combinatoria dei marcatori MAGIC ha permesso l'individualizzazione degli astrociti corticali e l'estrazione di informazioni riguardanti il loro volume e morfologia. Utilizzando il software di analisi delle immagini commerciali, il contorno dei singoli astrociti è stato delineato su ogni singola sezione ottica di pile di immagini confocali per segmentare e ricostruire il dominio territoriale occupato da ciascun astrocita (Figura 3). Dagli stessi stack di immagini Z, la morfologia ramificata degli astrociti corticali singled-out è stata segmentata utilizzando il software ad accesso aperto che consente l'estrazione dello scheletro dei principali processi astrociti (Figura 4). Questi strumenti di segmentazione e tracciamento hanno fornito una valutazione semiquantitativa dell'aumento del volume territoriale(figura 3)e della complessità morfologica(figura 4)che si è verificata per i singoli astrociti corticali dalle fasi postnatali precoci a quella tardiva14. Questi approcci hanno anche rivelato l'eterogeneità del volume e della morfologia mostrata da astrociti corticali distinti nello stesso stadio di sviluppo, come illustrato nella figura 2, figura 3e figura 4.

Figura 1: Rappresentazione schematica dei marcatori MAGIC (MM) in elettroporazione utero (IUE) per etichettare i progenitori corticali e la loro discesa durante lo sviluppo cerebrale. (A) Il toolkit MM comprende diversi plasmidi che codificano le trasposasi PB-Cytbow, Tol2-Nucbow, PB e Tol2 e la cre ricombinasi autoeccitabile (seCre). Nei costrutti MM, tre coppie di siti lox incompatibili (loxN, lox2272e loxP) fiancheggiano quattro distinte sequenze di codifica FP (EBFP2, mTurquoise2/mCerulean, EYFP, tdTomato/mCherry) e creano possibilità di escissione reciprocamente esclusive al momento della ricombinazione cre. Prima dell'azione di Cre, viene espresso solo il primo gene (EBFP2). Dopo l'escissione mediata da Cre indotta da seCre, viene espresso tdTomato/mCherry (FP rosso), EYFP (FP verde) o mTurquoise2/mCerulean (FP ciano). La co-espressione di FP da più copie di MAGIC Markers produce combinazioni di colori nel citoplasma (PB-CAG-Cytbow) o nel nucleo (Tol2-CAG-Nucbow) delle cellule etichettate. Lafet finale di trasposizione PB e Tol2 che inquadra le cassette MM consente la loro integrazione nel genoma dei progenitori corticali quando i costrutti MM vengono coelettroporati insieme ai plasmidi codificanti PB e Tol2. (B–G) Illustrazione grafica delle fasi successive dell'IUE, tra cui la laparotomia dei topi gravidi anestetizzati (B), l'iniezione della miscela plasmide MM(C) nei ventricoli laterale degli embrioni (D), la somministrazione di impulsi elettrici attraverso tweezertrodes accuratamente posizionati (E) per colpire i progenitori corticali in uno dei due emisferi cerebrali (F) e la sutura dell'addome del topo incinta (G). Clicca qui per visualizzare una versione più grande di questa figura.
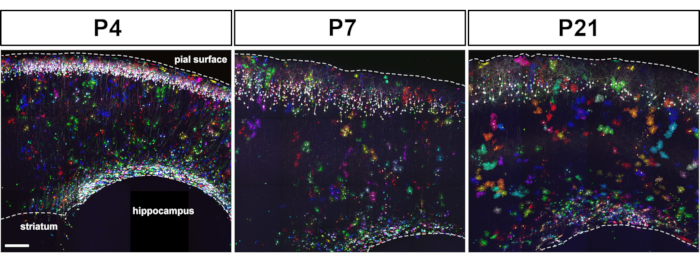
Figura 2: A seguito dell'elettroporazione in utero del toolkit MAGIC Markers, sono stati trovati astrociti multicolori sparsi nell'intera corteccia cerebrale nelle fasi postnatali. L'IUE dei plasmidi che guidano l'espressione delle trasposasi MM, seCre, PB e Tol2 nei progenitori corticali del topo E15.5 ha portato all'etichettatura di neuroni e astrociti di livello 2-3 a P4, P7 e P21. Il livello di espressione e la tavolozza dei colori dipendevano dal numero di transgeni MM integrati nel genoma dei progenitori corticali. Montaggio di proiezioni di massima intensità da pile di immagini confocali piastrellate acquisite su sezioni cerebrali sagittali da 80 μm. Barra di scala: 200 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Segmentazione del dominio territoriale degli astrociti corticali in fasi di sviluppo distinte. Proiezioni di intensità massima del confocale Z-stack ritagliato che inquadra i singoli astrociti (A-F) e il loro dominio territoriale associato segmentato con il software commerciale (A'-F') in tre fasi di sviluppo distinte: P4 (A-B, A'-B'), P7 (C-D, C'-D') e P21 (E-F, E'-F'),rispettivamente. Barra di scala: 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
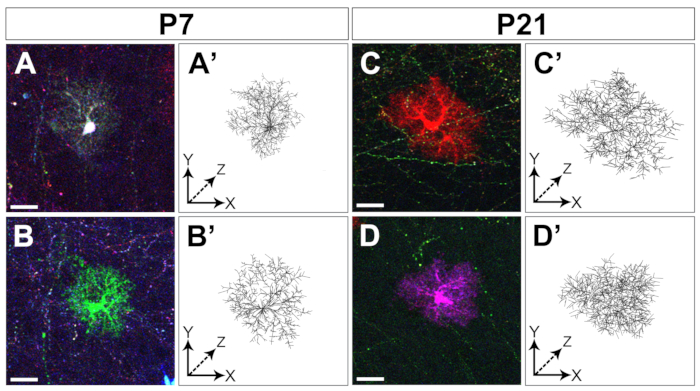
Figura 4: Tracciamento dell'arborizzazione degli astrociti corticali. Esempi di due astrociti distinti ritagliati da z-stack confocali (A-D) e ricostruzione della loro arborizzazione grossolanamente segmentata con Vaa3D (A'-D') raccolti in due fasi di sviluppo distinte: P7 (A-B, A'-B') e P21(C-D, C'-D'),rispettivamente. Barra di scala: 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
In utero l'elettroporazione (IUE) dei marcatori MAGIC nei progenitori corticali(Figura 1)ha permesso l'etichettatura degli astrociti in tutta la corteccia cerebrale postnatale in diversi stadi postnatali (P4-P7-P21, Figura 2). È interessante notare che lo stadio dell'IUE non è critico, in quanto l'elettroporazione eseguita da E13.5 a E15.5 produce modelli di etichettatura simili per quanto riguarda gli astrociticorticali 14. Tuttavia, la posizione dei neuroni piramidali etichettati nel parenchima corticale varia con lo stadio di elettroporazione. Infatti, iUE ha eseguito a E15 segni strato 2-3 neuroni mentre IUE ha eseguito a etichette E13 neuroni piramidali in tutti gli strati corticali, dallo strato 5 allo strato 2-36,27. Questa etichettatura congiunta dei neuroni piramidali corticali a seguito di MM IUE è la principale limitazione di questo metodo, in quanto impedisce la segmentazione semiautomatica della morfologia degli astrociti negli strati in cui l'etichettatura neuronale densa interferisce con il riconoscimento dei processi astrociti. Se questo fosse un problema, MM potrebbe essere elettroporata nelle fasi postnatali come in PALE. In P4, l'etichettatura delle fibre di glia radiali ancora presenti in quella fase può anche interferire con la segmentazione del pergolato Vaa3D. Sebbene ingombrante, una soluzione se si desidera procedere con la ricostruzione dell'arbor dell'astrocita in questa fase è quella di rimuovere manualmente le fibre gliali radiali sostituendo progressivamente il segnale in fibra radiale con pixel neri intorno all'astrocita di interesse utilizzando Adobe Photoshop.
Nonostante questa limitazione, MM IUE è una tecnica potente se adeguatamente eseguita. Alcuni passaggi critici devono essere maneggiati con cura: 1) gli embrioni devono essere mantenuti umidi durante l'intera procedura chirurgica e accuratamente manipolati per aumentarne la sopravvivenza; 2) l'utilizzo di capillari in vetro di grande diametro o la spremitura troppo stretta di sacchi embrionali può portare alla rottura del sacchetto e quindi alla morte degli embrioni; 3) durante l'iniezione di DNA, i vasi sanguigni devono essere evitati per prevenire il sanguinamento; 4) l'intera procedura non deve durare più di 40 minuti dall'anestesia alla sutura al fine di massimizzare la sopravvivenza dell'embrione; 5) lo stress svolge un ruolo fondamentale nel successo dell'IUE e quindi ulteriori fonti di stress come il cambio di gabbia, il trasporto, i rumori e le vibrazioni devono essere evitate da 5 giorni prima dell'intervento chirurgico a 7 giorni dopo la nascita al fine di prevenire l'aborto e il cannibalismo.
Da notare che gli sperimentatori che desiderano indirizzare specificamente le cellule nate in una data fase possono utilizzare il toolkit MM senza aggiungere le trasposasi piggyBac e Tol2 in modo tale che solo le cellule nate al momento dell'elettroporazione esprimano le etichette combinatoriali. Un altro vantaggio del metodo è la flessibilità che conferisce in termini di densità delle cellule etichettate e della loro posizione in varie regioni cerebrali. Infatti, un'etichettatura più densa degli astrociti corticali può essere ottenuta aumentando la concentrazione totale di transgeni MM mantenendo costante il rapporto plasmidi (rapporto 1:10 per i costrutti Cre ricombinasi/MM e rapporto 1:2 per i costrutti transposasi/MM). Contrariamente agli approcci monocromatici, come l'elettroporazione dei trasposoni monocolore o l'elettroporazione Cre nei topi Ai9, dove la capacità di insaccare i singoli astrociti richiede un'etichettatura sparsa, i contrasti di colore offerti dalla strategia MAGIC Markers consentono l'individualizzazione degli astrociti su una vasta gamma di densità di etichettatura. Inoltre, il posizionamento delle sonde degli elettrodi in orientamenti distinti consente di colpire distinte regioni cerebrali come lo striato prospettico (anodo in posizione ventrale, opposto alla posizione dorsale richiesta per ottenere l'elettroporazione nella corteccia cerebrale), o l'ippocampo (anodo in posizione mediale)28. Infine, l'IUE può essere eseguita in diversi ceppi di topi come topi di razza (OF1, svizzera) e inbred (C57BL/6J o N), il che apre la strada all'uso del toolkit MM nei modelli di malattie animali transgeniche. Tuttavia, per ottenere con successo l'IUE nei topi inbred, si dovrebbe adattare il numero di impulsi (tre impulsi per i topi C57BL/6 rispetto a cinque impulsi in svizzero), tensione (30 V invece di 35 V) e dose analgesica (soluzione di stock di buprenorfina mg/mL e un volume iniettato di 0,8 μL/g BW).
Rispetto all'allevamento di animali transgenici o agli approcci PALE e StarTrack, questo metodo offre diversi vantaggi. Per cominciare, a differenza della strategia di allevamento, utilizza pochi animali. Permette anche l'etichettatura degli astrociti corticali fin dalle prime fasi del loro sviluppo, compresi gli stadi embrionali, a differenza di PALE4, che si basa sull'elettroporazione postnatale. Inoltre, rispetto ai dodici costrutti reporter utilizzati nell'approccio StarTrack8, questa strategia si basa solo su due transgeni multicolore, rendendo così più semplice la preparazione del mix di DNA e l'imaging. Inoltre, l'equilibrio tra i diversi colori stocasticamente espressi da MM è intrinsecamente determinato dalla struttura dei transgeni e non dipende dalla miscelazione di diversi componenti da parte dello sperimentatore. Inoltre, questa strategia può estendersi oltre la semplice considerazione anatomica e può essere applicata con successo per l'analisi multiclonale dello sviluppo degli astrociti, come dimostrato nel lavoroprecedentemente pubblicato 14. Questo lavoro, utilizzando rare combinazioni di colori di marcatori citoplasmatici e nucleari per definire cloni di astrociti corticali, ha dimostrato che mostrano un'ampia variabilità in termini di distribuzione spaziale, organizzazione strutturale, numero e sottotipo di cellule generate.
Gli astrociti corticali nati da progenitori corticali etichettati MM mostravano un contrasto cromatico significativo e si dispersero ampiamente nell'intera corteccia cerebrale (Figura 2). Semplici acquisizioni di Z-stack multicanale mediante microscopia confocale sono state utilizzate per accedere a caratteristiche chiave degli astrociti come il loro volume territoriale (Figura 3) e la loro complessità morfologica (Figura 4) in diversi stadi postnatali. Oltre agli astrociti, questa metodologia può essere adattata per studiare la morfologia di altre cellule gliali come gli oligodendrociti. Tuttavia, va tenuto presente che la risoluzione limitata offerta dalla microscopia confocale può fornire solo una resa parziale della complessità morfologica degli astrociti. Mentre le immagini ottenute con una risoluzione più elevata (ad esempio, obiettivo olio 63x 1.4 NA) e algoritmi di deconvoluzione possono essere utilizzate per ricostruire dettagli più fini degli arbitri di astrociti14, i processi più raffinati non possono essere risolti con l'imaging ottico convenzionale. Tuttavia, la strategia qui presentata sarà interessante per migliorare in modo efficiente un potenziale fenotipo che influisce sul volume o sulla morfologia degli astrociti corticali nei modelli di topo delle malattie neurologiche.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo S. Fouquet e le strutture per l'imaging e il nucleo animale dell'Institut de la Vision e dell'Institut des Neurosciences de Montpellier (MRI e RAM) per l'assistenza tecnica. Questo lavoro è stato sostenuto da borse di studio della Région Ile-de-France e della Fondation ARC pour la Recherche sur le Cancer a S.C e dall'Université Paris-Saclay (Initiatives Doctorales Interdisciplinaires) a Los Angeles, finanziando il Consiglio europeo della ricerca (ERC-SG 336331, PI J. Valette) a E.H., da Agence Nationale de la Recherche con contratti ANR-10-LABX-65 (LabEx LifeSenses), ANR-11-EQPX-0029 (Equipex Morphoscope2), ANR-10-INBS-04 (France BioImaging) , di Fondation pour la Recherche Médicale (Rif. DBI20141231328), dal Consiglio Europeo delle Ricerche (ERC-CoG 649117, PI J. Livet) e dal programma ATIP-Avenir (PI K. Loulier).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.1 Bacteria transformation | |||
| Ampicillin | Euromedex | EU0400-C | |
| DH5 alpha competent cells | Fisher Scientitic | 11563117 | |
| Ice box | Dutscher | 139959 | |
| Kanamycin | Sigma | 60615 | |
| LB Agar | Sigma | L2897 | |
| SOC medium | Fisher Scientitic | 11563117 | |
| Sterile petri dish- 10 cm | Thermo Fisher | 150350 | |
| Water bath | VWR | 462-0556H | |
| 1.2 Plasmid culture | |||
| 14 ml culture tube | Dutscher | 187262 | |
| Glass erlenmeyer- 2L | Fisher Scientitic | 11383454 | |
| LB medium | Sigma | L3522 | |
| 1.3 Plasmid DNA preparation | |||
| NucleoBond Xtra Maxi Plus EF | Macherey-Nagel | 740426.10 | |
| 2.1 Preparation of the solutions | |||
| 26 G x 1/2 needle | Terumo | 8AN2613R1 | |
| 30 G x 1/2 needle | Terumo | 8AN3013R1 | |
| Fast Green | Sigma Aldrich | F7272 | |
| NaCl | VWR | 27810.295 | |
| Single-use polypropylene syringe, 1 mL | Dutscher | 50002 | |
| 2.2 Preparation of the surgery material | |||
| Adson Forceps - DeBakey Pattern- 12.5 cm | FST | 11617-12 | |
| Arched tip Forceps- 10 cm | FST | 11071-10 | |
| Glass bead sterilizer Steri 250 | Sigma | Z378569 | |
| Glass micropipette 1 mm diameter | FHC | 10-10-L | |
| Graefe Forceps - Titanium 1 mm Tips Slight Curve- 10 cm | FST | 11651-10 | |
| Graefe Forceps - Titanium 1 mm Tips Straight- 10 cm | FST | 11650-10 | |
| Iris Scissors - Delicate Straight- 9 cm | FST | 14060-09 | |
| Laboratory tape | Fisher Scientitic | 11730454 | |
| Microinjector | INJECT+MATIC | No catalog number | |
| Olsen-Hegar Needle Holder - 12 cm | FST | 12002-12 | |
| Optical fiber | VWR | 631-1806 | |
| Plastic-coated white paper | Distrimed | 700103 | |
| Signagel electrode gel | Free-Med | 15-60 | |
| Sterile Petri dish- 35 mm | Dutscher | 056714 | |
| Sterilizer, glass dry bead, Steri 250 | Sigma | Z378569 | |
| 2.3 Preparation of the pregnant female mouse | |||
| Alcohol pad | Alcomed | 1731000 | |
| Buprecare | Axience | 0.3 mg/ml | |
| Compress | tRAFFIN | 70189 | |
| Ketamine | Merial | Imalgene 1000 | |
| Ocular gel | tvm lab | Ocry-gel | |
| RjOrl:SWISS mice | Janvier Labs | ||
| Vetadine, 10% solution | Vetoquinol | 4337400113B | |
| Warming pad | Harvard Apparatus | 72-0493 | |
| Xylazine | Bayer | Rompun 2% | |
| 3.2 Electroporation | |||
| Absorbable suture Size 4-0 45 cm Suture 1-Needle 19 mm Length 3/8 Circle Reverse | Novosyn | C0068220 | |
| Electroporateur Sonidel | Sonidel | NEPA 21 | |
| Sterile transfer pipets (individually wrapped) | Dutscher | 043202S | |
| Tweezers with 3 mm platinium disk electrodes | Sonidel | CUY650P3 | |
| 4.1 Tissue harvesting and sectioning | |||
| 24-well plate | Falcon | 353047 | |
| Agarose | Lonza | 50004 | |
| Antigenfix | Microm Microtech | U/P0014 | |
| Coverslip | Dutscher | 100266 | |
| Dolethal | Vetoquinol | DOL202 | |
| DPBS (10X), no calcium, no magnesium | Fisher Scientific | 11540486 | |
| Nail polish | EMS | 72180 | |
| Slide | Dutscher | 100001 | |
| Vectashield | Vectorlabs | H-1000 | |
| Vibratome | Leica | VT1000S | |
| 5. Multichannel confocal imaging | |||
| 20X oil NA 0.85 | Olympus | ||
| Confocal Laser Scanning Microscope | Carl Zeiss | LSM880 | |
| Confocal Laser Scanning Microscope | Olympus | FV1000 | |
| Plan Apochromat 20x/0.8 M27 | Carl Zeiss | ||
| 6. Astrocyte territorial volume segmentation | |||
| IMARIS 8.3 and later versions | Bitplane | ||
| 7. Astrocyte arborization tracing | |||
| 3D Visualization-Assisted Analysis software suite (Vaa3D) | HHMI - Janelia Research Campus /Allen Institute for Brain Science |
Riferimenti
- Clarke, L. E., Barres, B. A. Emerging roles of astrocytes in neural circuit development. Nature Reviews Neuroscience. 14 (5), 311-321 (2013).
- Ma, Z., Stork, T., Bergles, D. E., Freeman, M. R. Neuromodulators signal through astrocytes to alter neural circuit activity and behaviour. Nature. 539 (7629), 428-432 (2016).
- Blanco-Suárez, E., Caldwell, A. L. M., Allen, N. J. Role of astrocyte-synapse interactions in CNS disorders: Astrocyte-synapse disease. The Journal of Physiology. 595 (6), 1903-1916 (2017).
- Stogsdill, J. A., et al. Astrocytic neuroligins control astrocyte morphogenesis and synaptogenesis. Nature. 551 (7679), 192-197 (2017).
- Srinivasan, R., et al. New Transgenic Mouse Lines for Selectively Targeting Astrocytes and Studying Calcium Signals in Astrocyte Processes in Situ and In Vivo. Neuron. 92 (6), 1181-1195 (2016).
- Tabata, H., Nakajima, K. Efficient in utero gene transfer system to the developing mouse brain using electroporation: visualization of neuronal migration in the developing cortex. Neuroscience. 103 (4), 865-872 (2001).
- Shimogori, T., Ogawa, M. Gene application with in utero electroporation in mouse embryonic brain. Development, Growth & Differentiation. 50 (6), 499-506 (2008).
- García-Marqués, J., López-Mascaraque, L. Clonal Identity Determines Astrocyte Cortical Heterogeneity. Cerebral Cortex. 23 (6), 1463-1472 (2013).
- Martín-López, E., García-Marques, J., Núñez-Llaves, R., López-Mascaraque, L. Clonal Astrocytic Response to Cortical Injury. PLoS ONE. 8 (9), 74039(2013).
- Gutiérrez, Y., et al. Sibling astrocytes share preferential coupling via gap junctions. Glia. 67 (10), 1852-1858 (2019).
- Lee, Y., Messing, A., Su, M., Brenner, M. GFAP promoter elements required for region-specific and astrocyte-specific expression. Glia. 56 (5), 481-493 (2008).
- Yoon, H., Walters, G., Paulsen, A. R., Scarisbrick, I. A. Astrocyte heterogeneity across the brain and spinal cord occurs developmentally, in adulthood and in response to demyelination. PloS One. 12 (7), 0180697(2017).
- Ge, W. P., Miyawaki, A., Gage, F. H., Jan, Y. N., Jan, L. Y. Local generation of glia is a major astrocyte source in postnatal cortex. Nature. 484 (7394), 376-380 (2012).
- Clavreul, S., et al. Cortical astrocytes develop in a plastic manner at both clonal and cellular levels. Nature Communications. 10 (1), 4884(2019).
- Figueres-Oñate, M., García-Marqués, J., López-Mascaraque, L. UbC-StarTrack, a clonal method to target the entire progeny of individual progenitors. Scientific Reports. 6 (1), 33896(2016).
- Figueres-Oñate, M., García-Marqués, J., Pedraza, M., De Carlos, J. A., López-Mascaraque, L. Spatiotemporal analyses of neural lineages after embryonic and postnatal progenitor targeting combining different reporters. Frontiers in Neuroscience. 9, 87(2015).
- Figueres-Oñate, M., Sánchez-Villalón, M., Sánchez-González, R., López-Mascaraque, L. Lineage Tracing and Cell Potential of Postnatal Single Progenitor Cells In Vivo. Stem Cell Reports. 13 (4), 700-712 (2019).
- Peng, H., Ruan, Z., Long, F., Simpson, J. H., Myers, E. W. V3D enables real-time 3D visualization and quantitative analysis of large-scale biological image data sets. Nature Biotechnology. 28 (4), 348-353 (2010).
- Peng, H., Bria, A., Zhou, Z., Iannello, G., Long, F. Extensible visualization and analysis for multidimensional images using Vaa3D. Nature Protocols. 9 (1), 193-208 (2014).
- Peng, H., et al. Virtual finger boosts three-dimensional imaging and microsurgery as well as terabyte volume image visualization and analysis. Nature Communications. 5 (1), 4342(2014).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Niwa, H., Yamamura, K., Miyazaki, J. Efficient selection for high-expression transfectants with a novel eukaryotic vector. Gene. 108 (2), 193-199 (1991).
- Abdeladim, L., et al. Multicolor multiscale brain imaging with chromatic multiphoton serial microscopy. Nature Communications. 10 (1), 1662(2019).
- Chen, F., LoTurco, J. A method for stable transgenesis of radial glia lineage in rat neocortex by piggyBac mediated transposition. Journal of Neuroscience Methods. 207 (2), 172-180 (2012).
- Siddiqi, F., et al. Fate Mapping by PiggyBac Transposase Reveals That Neocortical GLAST+ Progenitors Generate More Astrocytes Than Nestin+ Progenitors in Rat Neocortex. Cerebral Cortex. 24 (2), 508-520 (2014).
- Yoshida, A., et al. Simultaneous expression of different transgenes in neurons and glia by combining in utero electroporation with the Tol2 transposon-mediated gene transfer system. Genes to Cells. 15 (5), 501-502 (2010).
- LoTurco, J., Manent, J. B., Sidiqi, F. New and Improved Tools for In Utero Electroporation Studies of Developing Cerebral Cortex. Cerebral Cortex. 19, suppl 1 120-125 (2009).
- Pacary, E., et al. Visualization and Genetic Manipulation of Dendrites and Spines in the Mouse Cerebral Cortex and Hippocampus using in utero Electroporation. Journal of Visualized Experiments. (65), e4163(2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon