Method Article
Un modello di espressione della chemiochina ectopica per testare il reclutamento macrofrofo in vivo
In questo articolo
Riepilogo
Per testare l'effetto di una chemiochina sul reclutamento di macrofagi in vivo, l'intero montaggio in situ ibridazione è stato utilizzato per rilevare l'espressione ectopica della chemiochina, e l'immunostainizzazione è stata utilizzata per etichettare i macrofagi. L'imaging dal vivo è stato utilizzato per l'osservazione in tempo reale della migrazione dei macrofafi.
Abstract
I pesci zebra sono ampiamente utilizzati nella ricerca di base e biomedica. Molte linee transgeniche di pesci zebra sono attualmente disponibili per etichettare vari tipi di cellule. A causa del corpo embrionale trasparente del pesce zebra, è conveniente per noi studiare l'effetto di una chemiochina sul comportamento di un certo tipo di cellule in vivo. Qui abbiamo fornito un flusso di lavoro per studiare la funzione di una chemiochina sulla migrazione dei macrofaghi in vivo. Abbiamo costruito un plasmide di sovraespressione per itessuti specifici dei tessuti per sovraesprimere l'IL-34 e abbiamo iniettato il plasmide in embrioni di pesci transgenici con stadio unicellulare i cui macrofagi sono stati specificamente etichettati da una proteina fluorescente. Abbiamo quindi usato l'intero montaggio fluorescente in situ ibridazione e immunostaining per rilevare il modello dell'espressione chemiochina e il numero o la posizione dei macrofagi. Gli embrioni di WT iniettati sono stati sollevati per generare una linea transgenica stabile. Infine, abbiamo usato l'imaging dal vivo confocale per osservare direttamente il comportamento dei macrofagi nel pesce transgenico stabile per studiare la funzione dell'IL-34 sui macrofagi in vivo.
Introduzione
Il pesce zebra è un piccolo pesce dolce tropicale che ha avuto origine in India. Per quanto riguarda la conservazione genica, i pesci zebra hanno una somiglianza dell'87% con l'umano1. Può fornirci informazioni su argomenti correlati dell'uomo studiando la regolazione genica, la funzione delle proteine e il comportamento cellulare come la migrazione, la proliferazione et.al nel pesce zebra. L'embrione di pesce zebra può essere utilizzato per osservare lo sviluppo di embrioni precoci in diverse fasi dopo aver inibito il pigmento. Nel frattempo, ci vogliono solo tre mesi perché il pesce zebra si sviluppi in maturità sessuale, quindi il pesce zebra può produrre centinaia di uova ogni 4 giorni. Mini-dimensioni, allevamento semplice, forte capacità riproduttiva, questi vantaggi rendono la cultura del pesce zebra molto risparmio di spazio, favorevole alla cultura su larga scala. Il topo modello tradizionale di mammiferi ha costi di manutenzione più elevati rispetto al pesce zebra, limitando così la scala di sollevamento del mouse. Nell'aspetto dello sviluppo precoce dell'embrione, l'embrione di topo è difficile da osservare in condizioni vive a causa delle caratteristiche dello sviluppo dell'embrione di topo nel grembo materno. Al contrario, gli embrioni di pesce zebra si sviluppano esternamente e sono trasparenti, quindi sono facili da osservare al microscopio. Inoltre, il pesce zebra è molto facile da costruire una varietà di linee transgeniche per la ricerca sulle funzioni geniche correlate. Attualmente, sono disponibili varie linee transgeniche di pesci zebra per etichettare diversi tipi di cellule. Ora è molto conveniente costruire linee transgeniche per sovraesprimere le chemiochine in luoghi specifici e studiare la funzione delle chemiochine sul comportamento cellulare nel pesce zebra.
Qui, abbiamo fornito un flusso di lavoro per utilizzare la linea transgenica di pesce zebra per studiare la funzione di IL-34 sul comportamento del macrofago in vivo2,3,4,5,6,7. In primo luogo, abbiamo costruito una plasmida di sovraespressione specifica del fegato del gene il34 e iniettato il plasmide in uno stadio unicellulare Tg (mpeg1: GFP) embrioni di pesce che specificamente etichettavano i macrofagi dalla proteina fluorescente GFP. Quindi, abbiamo usato l'intero monte fluorescente in situ ibridazione e immunostaining per rilevare il modello dell'espressione il34 e il numero o la posizione dei macrofagi. Gli embrioni di WT iniettati sono stati sollevati per generare una linea transgenica stabile. In questi passaggi, abbiamo stabilito e convalidato la linea di produzione della citochina e valutato visivamente gli effetti che si possono vedere sulla distribuzione dei macrofafi. Infine, per studiare il comportamento dei macrofagi in risposta alla citochina, abbiamo usato l'imaging dal vivo confocale per osservare direttamente la migrazione dei macrofagi per confermare la funzione di il34 sulla migrazione del macrofago in vivo.
Protocollo
NOT: Tutti i campioni sono stati trattati con l'acqua uovo fenilthiourea (PTU) per inibire il pigmento.
1. Generazione di Tg (fabp10a:il34) Costrutti transgenici e iniezione di pesce
- Clonare il promotore di 2,8 kb fabp10a 8 e le aree di codifica IL-34 (ENSDART00000126460.3) del pesce zebra nel vettore pTol2 per generare il costrutto fabp10a-il34. Iniettare i costrutti nello stadio a una cellula Tg (mpeg1: GFP) e negli embrioni di pesce WT insieme all'mRNA traposase. Sollevare gli embrioni WT iniettati fabp10a-il34 agli adulti9 e identificare il fondatore transgenico mediante l'ibridazione in situ.
NOT: L'iniezione del costrutto Tol2 direttamente in un altro transgenico potrebbe essere problematica se l'altra linea transgenica fosse fatta con lo stesso sistema di trasposoni. Una pratica generale sarebbe quella di fare una linea transgenica indipendente e successivamente attraversare la nuova linea con un'altra linea reporter. Questo assicura che non ci saranno effetti della nuova transgenesi su un transgene precedentemente inserito.
2. Combinazione dell'intero monte fluorescente in situ (WISH) con immunostaining
- Fissazione del campione
- Raccogliere gli embrioni di iniezione transitoria o stabile linea transgenica IL-34 che ha attraversato con Tg (mpeg1: GFP) nelle fasi desiderate.
NOT: In questo caso, gli embrioni sono stati raccolti a 4 d dopo la fecondazione (dpf). (Se necessario) rimuovere il chorion con la siringa. - Fissare gli embrioni con il 4% di paraformaldeide (PFA) durante la notte a 4 o 2 h a temperatura ambiente (RT) (circa 25 gradi centigradi).
- Lavare gli embrioni con fosfato tamponato salina più Tween 20 (PBST) 3x 5 min.
- Disidratare gli embrioni separatamente con il 50% di metanolo in PBST (50% metanolo/PBST) e 100% metanolo, 1x 5 min ciascuno. Quindi, passare a fresco 100% metanolo e conservare a -20 gradi centigradi (almeno 2 h).
NOT: Il protocollo può essere messo in pausa qui.
- Raccogliere gli embrioni di iniezione transitoria o stabile linea transgenica IL-34 che ha attraversato con Tg (mpeg1: GFP) nelle fasi desiderate.
- Ibridazione della sonda (giorno I)
- Reidratare gli embrioni nei passaggi precedenti con il 50% di metanolo in PBST (50% metanolo/PBST), quindi lavare con PBST 3x 5 min.
- Digerire gli embrioni con proteinasi K in PBST a RT (concentrazione finale: 10 g/mL; 1:2000 in PBST).
NOT: Il tempo di digestione dipende dallo stadio embrioni: meno di 36 h dopo la fecondazione (hpf), non c'è bisogno; 36 hpf-2 dpf embrione, 3-5 min; 2-3 dpf embrione, 10 min; 3-4 dpf embrione, 15 min; 4-5 dpf embrionale, 15-20 min; 5-6 dpf embrione, 20-27 min; >6 dpf embrione, 25-30 min a RT (circa 25 gradi centigradi). - Eliminare la soluzione di digestione ed eseguire nuovamente la fissazione con il 4% di PFA, per 20 min a RT.
- Lavare gli embrioni con PBST 2x 10 min.
- Scartare il PBST, eseguire la pre-ibridazione con buffer di ibridazione riscaldata (HB buffer) a 65 gradi centigradi per 5 min, riciclare il buffer HB nel tubo originale.
- Eseguire la pre-ibridazione con il nuovo buffer HB riscaldato a 65 gradi centigradi almeno 1 h.
- Preriscaldare la sonda9 (per questo caso era una sonda il34, 1 ng/mL) a 65 gradi centigradi almeno 10 min. Quindi riciclare il buffer HB nel tubo originale. Eseguire l'ibridazione con la sonda preriscaldata a 65 gradi durante la notte.
- Trattamento anticorpale (giorno II)
- Preriscaldare il citrato di sodio salina al 50% formamide/2x più Tween 20 (SSCT), 2x SSCT, 0,2x SSCT a 65 gradi centigradi.
- Riciclare la sonda nel tubo originale e conservare la sonda a -20 gradi centigradi.
- Lavare gli embrioni separatamente con il 50% di formamide/2x SSCT; 2x SSCT; 0,2x SSCT, 3x 20 min o 2x 30 min ciascuno a 65 gradi centigradi.
- Lavare gli embrioni con PBST 3x 5 min.
- Bloccare i campioni con 600 l di buffer di blocco (5% di siero bovino fetale filtrato (FBS) in PBST) per 1 h a RT.
- Aggiungere 400 -L di anticorpi Anti-digossina-HRP (1:1.000-1:2.000 nel buffer di blocco) e incubare gli embrioni a 4 gradi durante la notte. Se i segnali sono deboli, utilizzare 1:500 diluizione dell'anticorpo.
- Colorazione e incubazione primaria di anticorpi (Giorno III)
- Rimuovere l'anticorpo; lavare gli embrioni con PBST, 6x 20 min a RT.
- Sciacquare il campione con 30 : L di 1x Plus Amplification Diluent per 5 min a RT.
- Scartare il diluente con il pipeting out; diluire Fluorophore Tyramide Stock Solution (Cyanine 3 Plus Amplification Reagent (Cy3) o Cyanine 5 Plus Amplification Reagent (Cy5), per questo caso è stato utilizzato Cy3) 1:50 in 1x Plus Amplification Diluent per realizzare la Soluzione di lavorazione Fluorophorami Tyde. Preparare 50-100 l di soluzione di lavoro per ogni campione.
- Incubare il campione nella soluzione di lavoro Fluorophore Tyramide per 5-15 min al buio a RT. Se i segnali sono deboli, estendere il tempo di incubazione a 30 min.
- Interrompere la reazione modificando la soluzione di lavoro con PBST ed esaminare i segnali.
- Lavare gli embrioni con PBST 3x 10 min a RT.
- Incubare il campione con anticorpo primario a 4 gradi durante la notte. Per questo caso, utilizzare l'anticorpo Goat-Anti-GFP come anticorpo primario.
- Colorazione anticorpale secondaria (giorno IV)
- Lavare gli embrioni con PBST per 4x 30 min.
- Incubare gli embrioni con anticorpi secondari a 4 gradi centigradi durante la notte. In questo caso, utilizzare l'anticorpo Alexa 488-Anti-Goat come anticorpo secondario.
- Scattare foto (Giorno V)
- Lavare gli embrioni con PBST 3x 10 min a RT.
- Conservare gli embrioni nel 70% glicerolo al buio a 4 gradi centigradi o a -20 gradi centigradi più a lungo.
3. Immagine dal vivo
-
Selezione del campione
NOTA: Utilizzare l'immagine dal vivo per osservare direttamente se i macrofagi di Tg (fabp10a: il34; fabp10a: DsRed; mpeg1: GFP) i pesci migrarebbero nel fegato sotto induzione di IL-34 durante 3-3,5 dpf. Qui la linea transgenica Tg (fabp10a-DsRed) viene utilizzata per etichettare la regione del fegato e renderla visibile, per facilitare la localizzazione del fegato e per determinare se i macrofagi migrano nel fegato. Prima dell'imaging, utilizzare un microscopio a fluescenza per selezionare gli embrioni doppi positivi d'DsRed e GFP. -
Montaggio dei pesci
- Utilizzare un bagno di metallo per riscaldare 1 mL di 1% agarose di fusione bassa a oltre 90 gradi per scioglierlo completamente.
- Dopo che l'agarose a bassa fusione viene raffreddata a temperatura corporea, aggiungere 50 - L di 0,2% di tricaina, e mescolare uniformemente la tricaina con l'agarose.
- Spostare gli embrioni anestesittati in un piccolo piatto montato con uno scivolo di copertura sul fondo, rimuovere l'acqua circostante, rilasciare lentamente la bassa agarose di fusione sugli embrioni, impostare con attenzione la posizione del pesce prima che l'agarose sia solidificato, mantenere l'area del fegato vicino la diapositiva di copertura sul fondo del piatto.
- Dopo che l'agarose a bassa fusione è solidificata, coprirla con cura con un altro strato di agarose per rinforzarla.
- Posizionare il piatto sul tavolo portamicroscopio confocale, coprire il pesce con la soluzione E210 con la tricaina e iniziare l'imaging.
-
Funzionamento software del microscopio confocale
- Aprire il software 2.3 nero EN, installare il banco di lavoro cellulare vivente sul tavolo portante del microscopio.
- Fare clic su Individua . Incubazione Temperatura per impostare la temperatura a 29 gradi centigradi.
- Posizionare il piatto al centro della panca di lavoro della cellula vivente, coprire il pesce con la soluzione E210 con tricaina.
- Fare clic sul menu Acquisizione, selezionare la modalità di scansione e i laser richiesti nel menu Smart Setup, quindi selezionare Stack e Posizione.
- Fai clic sul menu Progettazione esperimenti, seleziona Abilita esperimento a blocchi multi (Enable Multi Block Experiment)nel primo blocco per trovare il campione sotto il basso ingrandimento, quindi passa all'ingrandimento elevato, lascia che l'area osservata al centro del campo visivo.
- Impostare la posizione e le informazioni di stack z, selezionare l'intensità del laser appropriata, i livelli di scansione e la velocità di imaging.
- Creare un nuovo blocco e ripetere i passaggi precedenti. Dopo aver impostato tutti i blocchi, impostare il numero appropriato di cicli e avviare la registrazione (Figura 1).
Risultati
I passaggi coinvolti nel protocollo del pesce zebra sono illustrati nella Figura 2. In primo luogo, è stato generato il costrutto pBLK-fabp10a-il34-sv40 in cui il34 è stato guidato dal promotore fabp10a (Figura 2). Il costrutto è stato microiniettato in embrioni di pesce zebra Tg (mpeg1: GFP) che possono etichettare i macrofagi con embrioni GFP e WT che sono stati innalzati agli adulti per generare una linea stabile transgenica (Figura 2). L'espressione di il34 è stata analizzata da tutta la fluorescenza di montaggio nell'ibridazione situ(Figura 2 e Figura 3). I macrofagi etichettati dalla GFP sono stati analizzati mediante immunostaining (Figura 2 e Figura 3). Abbiamo usato l'imaging dal vivo per osservare direttamente se i macrofagi migrerebbero nel fegato sotto l'induzione di il34 durante 3-3.5 dpf (Figura 2, Figura 4, Supplementary Movie 1 e Supplementary Movie 2) .
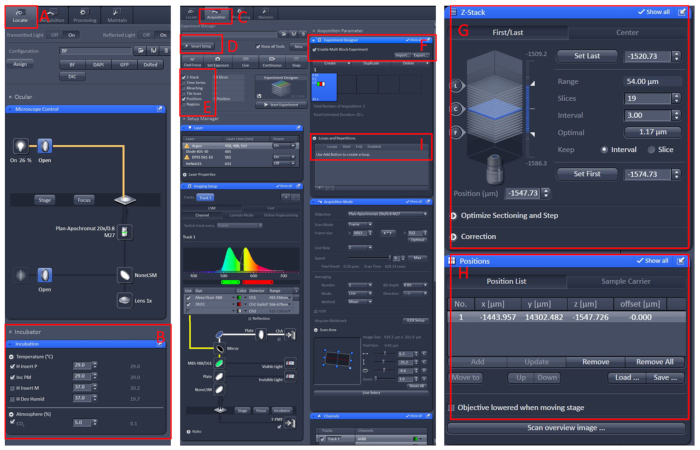
Figura 1: Funzionamento software dell'imaging live al microscopio confocale. Aprire il software di 2.3 nero, installare il banco di lavoro della cella vivente nella tabella portascopio, quindi fare clic su Individua (A) Incubazione Temperatura (B) per impostare la temperatura a 29 . Posizionare il piatto al centro della panca di lavoro della cellula vivente, coprire il pesce con la soluzione E210 con tricaina. Dopo tutte queste operazioni, fare clic sul menu Acquisizione (C), selezionare la modalità di scansione e i laser richiesti nel menu Smart Setup (D),quindi selezionare zo-Stack e Position (E). Infine, fare clic sul menu Progettazione esperimenti (F), selezionare l'opzione Abilita esperimentomulti blocco , nel primo blocco, per trovare il campione sotto il basso ingrandimento, quindi passare all'ingrandimento elevato, lasciare l'area osservata nella al centro del campo visivo, impostare la posizione e le informazioni di stack(G e H),selezionare l'intensità del laser appropriata, i livelli di scansione e la velocità di imaging. Creare un nuovo blocco e ripetere i passaggi precedenti. Dopo aver impostato tutti i blocchi, impostare il numero appropriato di loop (I) e avviare la registrazione. Fare clic qui per visualizzare una versione più grande di questa figura.
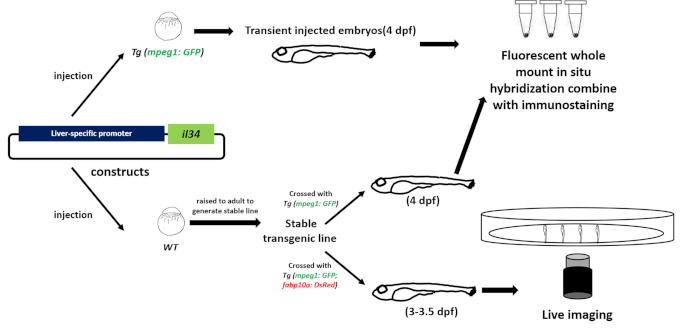
Figura 2: Flusso di lavoro per analizzare la funzione di una chemiochina sulla migrazione dei macrofaghi in vivo. Abbiamo costruito un plasmide di sovraespressione per sovraespressione del tessuto (fegato) per sovraesprimere l'IL-34 e iniettato la plasmide in embrioni di pesce transgenici a uno stadio a celle i cui macrofagi sono stati specificamente etichettati da una proteina fluorescente (Tg: (mpeg1: GFP )). Gli embrioni di WT iniettati sono stati sollevati per generare una linea transgenica stabile. Abbiamo quindi utilizzato l'interi insiorescenti in situ e l'immunostaining per rilevare il modello dell'espressione genica e il numero o la posizione dei macrofagi degli embrioni iniettati transitori o degli embrioni di linea stabile (4 dpf). Infine, abbiamo usato l'imaging dal vivo confocale per osservare direttamente il comportamento dei macrofagi nel pesce transgenico stabile (3-3,5 dpf) per studiare la funzione dell'IL-34 sui macrofagi in vivo. Fare clic qui per visualizzare una versione più grande di questa figura.
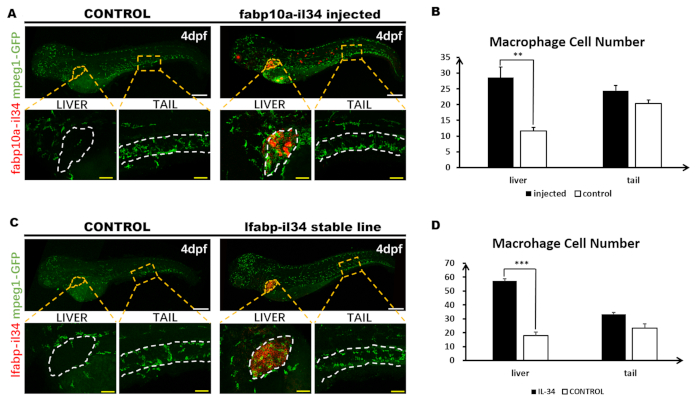
Figura 3: WISH fluorescente si combinano con l'immunostaining. Questa cifra è stata modificata da Jiang et al.11. Un totale di 1,8 nL (30 ng/l) del costrutto pBLK-fabp10a-il34-sv40 è stato microiniettato negli embrioni di pesce zebra Tg (mpeg1: GFP) a uno stadio cellulare. (A) WISH dell'espressione il34 (rosso) e della colorazione anticorpale integrale dell'espressione GFP (verde) nell'embrione 4 dpf (6x). L'intera immagine del corpo del pesce è costituita da due immagini separate scattate da confocal e cucite insieme in Photoshop. Gli inset sono ad alto ingrandimento (20x) delle regioni boxed corrispondenti (aree punteggiate arancioni). (B) Analisi quantitativa del numero di cellule dei macrofaci nel fegato non iniettato e costruibile degli embrioni iniettati (mostrato nella zona punteggiata bianca) e della regione della coda (circa tra la 13a e la 17a somitetica, dimostrata tra due linee tratteggiate bianche). I dati sono stati analizzati dal test di Mann Whitney U, p < 0,01 rispetto al controllo. n 5, 5 per i 4 dpf iniettati e pesci di controllo. Barre: 200 m (linea bianca); 50 m (linea gialla). (C) WISH dell'espressione il34 e colorazione anticorpale integrale dell'espressione GFP in embrione di linea stabile 4 dpf (6x). L'intera immagine del corpo del pesce è costituita da due immagini separate scattate da confocal e cucite insieme in Photoshop. Gli inset sono ad alto ingrandimento (20x) delle regioni boxed corrispondenti (aree punteggiate arancioni). (D) Analisi quantitativa del numero di cellule macrofagi in Tg (mpeg1: GFP) e Tg (fabp10a: il34; mpeg1: GFP) fegato degli embrioni (mostrato nella zona punteggiata bianca) e regione della coda (circa tra il 13 e la diciassettesima somite, mostrata tra due linee tratteggiate bianche). Fare clic qui per visualizzare una versione più grande di questa figura.
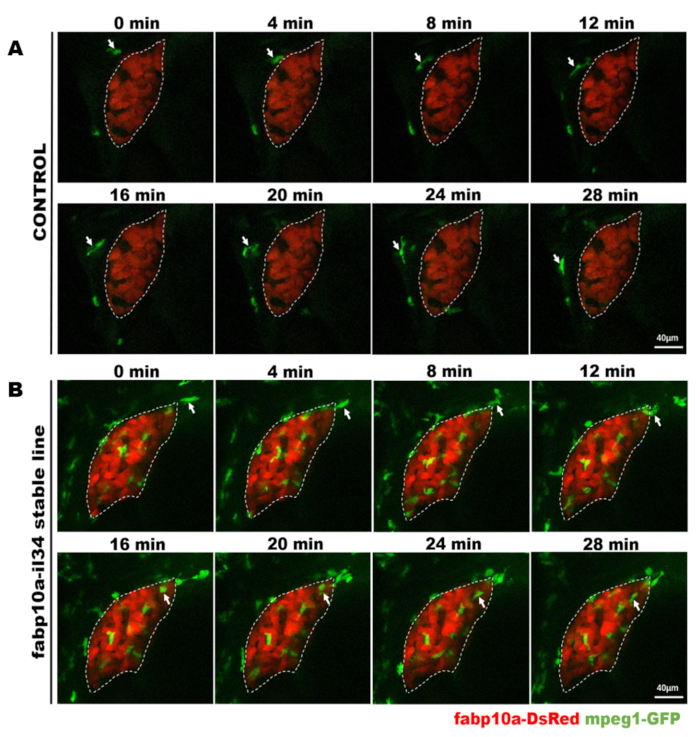
Figura 4: Imaging dal vivo confocale per osservare direttamente il comportamento dei macrofafi nei pesci transgenici stabili. Questa cifra è stata modificata da Jiang et al.11. Le micrografie di imaging dal vivo mostrano il processo di un macrofago (verde, etichettato da frecce bianche) che passa dal fegato (rosso) entro 28 min nel pesce di controllo (A) e il processo di un macrofago (verde, etichettato da frecce bianche) che migra nel fegato (rosso) entro 28 in IL-34 sovraesprimenti pesci (B). Barre della scala: 40 m (linea bianca). Fare clic qui per visualizzare una versione più grande di questa figura.
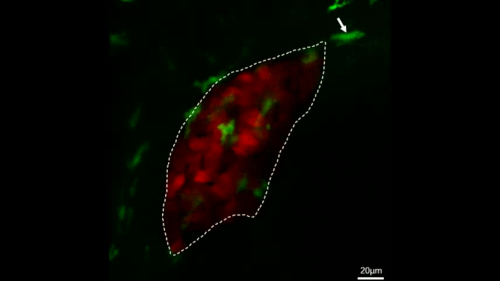
Filmo supplementare 1: Immagini dal vivo che mostrano il processo dei macrofagi (verdi, etichettati da frecce bianche) che migrano nel fegato (rosso) entro 2 h nel pesce IL-34. Barre della scala: 20 m (linea bianca). Questo film è stato ripubblicato da Jiang et al.11. Clicca qui per vedere questo video. (Fare clic con il pulsante destro del mouse per scaricare.)
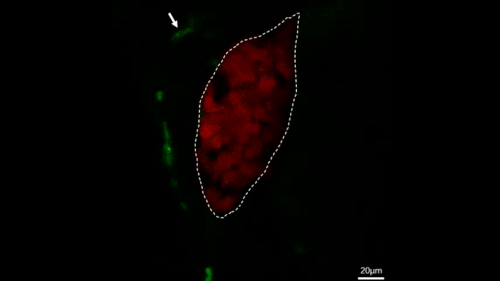
Filmato supplementare 2: Immagini dal vivo che mostrano il processo dei macrofagi (verde, etichettati da frecce bianche) che vagano intorno al fegato (rosso) entro 2 h nel pesce di controllo. Barre della scala: 20 m (linea bianca). Questo film è stato ripubblicato da Jiang et al.11. Clicca qui per vedere questo video. (Fare clic con il pulsante destro del mouse per scaricare.)
Discussione
Il protocollo qui descritto ci permette di indagare la funzione di una chemiochina sul comportamento di macrophagein vivo e la procedura richiede una certa competenza tecnica. In sintesi, ci sono diversi passaggi critici per evitare complicazioni nel protocollo: 1) selezionare una linea transgenica adatta che mostra un segnale transgenico specifico e forte per etichettare la cellula di interesse; 2) selezionare un tessuto appropriato accessibile per l'imaging e la sovraespressione genica transgenica; 3) fare una sonda di RNA sensibile e specifica; 4) selezionare un intervallo di tempo di osservazione appropriato per catturare con precisione il comportamento della cella.
Nella procedura di ibridazione in situ intera di supporto combinata con l'immunostaining, la sonda RNA utilizzata per rilevare l'espressione genica dovrebbe essere sensibile e il segnale deve essere abbastanza forte. Per catturare la funzione genica sul comportamento cellulare, è necessario testare una serie di punti temporali. Ad esempio, osservando l'effetto del il34 sulla migrazione dei macrofagi, anche se il promotore di fabp10a ha cominciato ad esprimere a 2-3 dpf, l'accumulo di macrofagi nel fegato non era evidente in quel momento. È solo da 4 dpf che l'arricchimento del macrofago nel fegato diventa evidente. Inoltre, dopo l'ibridazione in situ, l'intensità del segnale della successiva immunostainizzazione sarà influenzata. Ad esempio, confrontandolo con GFP, DsRed è difficile da colorare nella colorazione immunofluorescenza dopo l'ibridazione in situ, probabilmente a causa delle diverse strutture proteiche. In generale, l'intensità del segnale dell'immunostaining dopo l'intera fluorescenza in situ nell'ibridazione situ sarebbe inferiore a quella dell'immunostaining singolo.
Nella fase di imaging dal vivo con il microscopio confocale, è necessario tenere il campione vicino al fondo del piatto. Quando gli embrioni galleggiano in agarose, la distanza di lavoro dell'obiettivo può essere insufficiente, inoltre, l'agarose tra l'obiettivo e il campione influenzerebbe la qualità dell'imaging. Inoltre, il numero di campioni per l'imaging in ogni momento deve essere impostato correttamente. Si deve fare in modo che l'intervallo di tempo tra due scansioni di ogni pesce non sarebbe troppo lungo per perdere i dettagli del comportamento cellulare. Quindi questo metodo non è adatto per il monitoraggio delle cellule che si muovono velocemente nei tessuti spessi.
In conclusione, questo protocollo può essere utilizzato per osservare la funzione delle chemiochine sul comportamento di una varietà di cellule come macrofagi, neutrofili e cellule T. Qui, abbiamo usato IL-34, un ligando recentemente identificato della funzione CSF-1R in chemotassico6,7, come una chemiochina espressa ectopica per indurre la migrazione dei macrofagi. La maggior parte dei modelli sperimentali esistenti di chemiotassi cellulari si basano su esperimenti a cellule in vitro, ma gli esperimenti in vitro a volte sono troppo semplici per modellare l'ambiente complesso in vivo. Inoltre, è difficile immaginare la capacità chemio-attrazionein vivo quando si guarda semplicemente la situazione in vitro. Questo metodo ha utilizzato i vantaggi specifici del pesce zebra per l'osservazione diretta del comportamento cellulare che è difficile per i topi. Il metodo attuale ci ha permesso di testare rapidamente le funzioni della chemiochina sui comportamenti cellulari entro diversi giorni e di rendere il pesce zebra un modello potente per studiare la biologia molecolare e cellulare.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo il Dr. Jingrong Peng per aver condiviso la linea transgenica Tg (fabp10a: DsRed); Dr. .ilong Wen per la condivisione delle linee transgeniche Tg (mpeg1: GFP); Dr. Koichi Kawakami per aver fornito il vettore pTol2. Questo lavoro è stato sostenuto dalla National Natural Science Foundation of China (31771594), dai progetti del Guangdong Science and Technology Plan (2019A030317001) e dai Fondi fondamentali di ricerca per le università centrali (D2191450).
Materiali
| Name | Company | Catalog Number | Comments |
| Antibody | |||
| Alexa 488-Anti-Goat antibody | Invitrogen | A11055 | |
| Anti-Digoxigenin-HRP | perkinelmer | NEF832001EA | |
| Goat-Anti-GFP antibody | Abcam | ab6658 | |
| Reagent | |||
| CaCl2· 2H2O | Sigma | 21097 | |
| Cyanine 3 Plus Amplification Reagent | perkinelmer | NEL745001KT | |
| E2 solution | 15 mM NaCl +0.5 mM KCl +1.0 mM MgSO4+150 µM KH2PO4 + 50 µM Na2HPO4 +1.0 mM CaCl2 + 0.7 mM NaHCO3 | ||
| Fetal Bovine Serum (FBS) | Life | 10099-133 | |
| Formamide | Diamond | A100314 | |
| Glycerol | Sigma | V900860 | |
| Heparin sodium | Sigma | H3149 | |
| Hybridization buffer(HB) | 50% formamide+ 5×SSC+9 mM sodium citrate+50 μg/ml heparin sodium+ 500 μg/mL tRNA+ 0.1% Tween20 | ||
| KCl | Sigma | P5405 | |
| KH2PO4 | Sigma | P5655 | |
| Low melting agarose | Sigma | A9414 | |
| Methanol | GHTECH | 1.17112.023 | |
| Methylene blue | Sigma | M9140 | |
| MgSO4 | Sigma | M2643 | |
| Na2HPO4 | Sigma | S5136 | |
| NaCl | Sigma | S5886 | |
| NaHCO3 | Sigma | S5761 | |
| Paraformaldehyde (PFA) | Sigma | 158127 | Suspend 16 g of PFA in 400 ml of 1x PBS, heat at 60 °C to dissolve about 30 min. This solution can be prepared in advance and stored at -4 °C. Caution. Manipulate with mask. |
| 10×PBS | 14.2 g Na2HPO4+80 g NaCl+2 g KCl+ 2.4 g KH2PO4 in 1L ddH2O | ||
| Phenylthiourea (PTU) | Sigma | P7629 | |
| 1×Plus Amplification Diluent | perkinelmer | NEL745001KT | |
| Proteinase K | Fermentas | E00492 | |
| 20×Saline sodium citrate(SSC) | 175.3 g NaCl+ 88.2 g sodium citrate in 1 L ddH2O, PH 7.0 | ||
| Sodium citrate | Sigma | A5040 | |
| Tricaine | Sigma | E10521 | |
| tRNA | Sigma | R6625 | |
| Tween20 | Sigma | P2287 | |
| Plasmid | |||
| pBLK-fabp10a-il34-sv40 | For Tg (fab10a:il34) transgenic line generation | ||
| pBSK-il34 | For il34 probe preparation | ||
| Fish | |||
| Tg (mpeg1: GFP) | Label macrophages with GFP | ||
| Tg (fabp10a: DsRed) | Label liver cells with DsRed | ||
| Tg (fab10a:il34) | Over-expression IL-34 in liver cells |
Riferimenti
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Wang, Y., et al. IL-34 is a tissue-restricted ligand of CSF1R required for the development of Langerhans cells and microglia. Nature Immunology. 13 (8), 753-760 (2012).
- Lin, H., et al. Discovery of a cytokine and its receptor by functional screening of the extracellular proteome. Science. 320 (5877), 807-811 (2008).
- Wei, S., et al. Functional overlap but differential expression of CSF-1 and IL-34 in their CSF-1 receptor-mediated regulation of myeloid cells. Journal of leukocyte biology. 88 (3), 495-505 (2010).
- Etienne, D., Foucher, S. B. L. P., Norbert Ifrah, P. G. Y. D. IL-34 Induces the Differentiation of Human Monocytes into Immunosuppressive Macrophages. Antagonistic Effects of GM-CSF and IFNc. PLoS One. 8 (2), e56045 (2013).
- Segaliny, A. I., et al. Syndecan-1 regulates the biological activities of interleukin-34. Biochimica et Biophysica Acta. 1853 (5), 1010-1021 (2015).
- Zhou, S. L., et al. miR-28-5p-IL-34-macrophage feedback loop modulates hepatocellular carcinoma metastasis. Hepatology. 63 (5), 1560-1575 (2016).
- Gordon, J. I., et al. Tissue specific expression and developmental regulation of two genes coding for rat fatty acid binding proteins. Journal of Biological Chemistry. 260 (4), 1995-1998 (1985).
- Westerfield, M. . The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio rerio). , (2000).
- Nüsslein-Volhard, C., Dahm, R. . Zebrafish: a practical approach. , (2002).
- Jiang, Y., Chen, J., Yen, K., Xu, J. Ectopically Expressed IL-34 Can Efficiently Induce Macrophage Migration to the Liver in Zebrafish. Zebrafish. 16 (2), 165-170 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon