Method Article
Un modelo de expresión de chemocina ectópico para probar el reclutamiento de macrófagos in Vivo
En este artículo
Resumen
Para probar el efecto de una quimiocina en el reclutamiento in vivo de macrófagos, se utilizó toda la hibridación in situ del monte para detectar la expresión ectópica de la quimiocina, y se utilizó inmunomanchado para etiquetar los macrófagos. Las imágenes en vivo se utilizaron para la observación en tiempo real de la migración de macrófagos.
Resumen
Los peces cebra son ampliamente utilizados en la investigación básica y biomédica. Muchas líneas transgénicas de pez cebra están actualmente disponibles para etiquetar varios tipos de células. Debido al cuerpo embrionario transparente del pez cebra, es conveniente para nosotros estudiar el efecto de una quimiocina en el comportamiento de un cierto tipo de células in vivo. Aquí proporcionamos un flujo de trabajo para investigar la función de una quimiocina en la migración de macrófagos in vivo. Construimos un plásmido de sobreexpresión específico del tejido para sobreexpresar IL-34 e inyectamos el plásmido en embriones de peces transgénicos de una célula cuyos macrófagos fueron etiquetados específicamente por una proteína fluorescente. A continuación, utilizamos hibridación fluorescente in situ de montaje completo e inmunomancha para detectar el patrón de la expresión de quimiocina y el número o ubicación de los macrófagos. Los embriones WT inyectados fueron levantados para generar una línea transgénica estable. Finalmente, utilizamos imágenes en vivo confocales para observar directamente el comportamiento de los macrófagos en los peces transgénicos estables para estudiar la función de IL-34 en los macrófagos in vivo.
Introducción
Zebrafish es un pequeño pez tropical de agua dulce de huesos duros originario de la India. En cuanto a la conservación del gen, el pez cebra tiene una similitud del 87% con el humano1. Puede proporcionarnos información sobre temas relacionados con el ser humano mediante el estudio de la regulación genética, la función proteica y el comportamiento celular, como la migración, la proliferación et.al en el pez cebra. El embrión zebrafish se puede utilizar para observar el desarrollo de embriones tempranos en diferentes etapas después de inhibir el pigmento. Mientras tanto, el pez cebra tarda sólo tres meses en convertirse en madurez sexual, entonces el pez cebra puede producir cientos de huevos cada 4 días. Mini-tamaño, simple cría, fuerte capacidad reproductiva, estas ventajas hacen que el cultivo de peces cebra muy ahorro de espacio, propicio para el cultivo a gran escala. El ratón modelo de mamífero tradicional tiene costos de mantenimiento más altos que el pez cebra, lo que limita la escala de la elevación del ratón. En el aspecto del desarrollo temprano del embrión, el embrión de ratón es difícil de observar en estado vivo debido a las características del desarrollo del embrión de ratón en el útero materno. Por el contrario, los embriones de pez cebra se desarrollan externamente y son transparentes, por lo que son fáciles de observar bajo un microscopio. Además, el pez cebra es muy fácil de construir una variedad de líneas transgénicas para la investigación de la función génica relacionada. Actualmente, varias líneas transgénicas de pez cebra están disponibles para etiquetar diferentes tipos de células. Es muy conveniente ahora construir líneas transgénicas para sobreexpresar quimiolinas en lugares específicos y estudiar la función de las quimiolinas en el comportamiento celular en el pez cebra.
Aquí, proporcionamos un flujo de trabajo para utilizar la línea transgénica de pez cebra para investigar la función de IL-34 sobre el comportamiento de los macrófagos in vivo2,3,4,5,6,7. En primer lugar, construimos un plásmido de sobreexpresión específico del hígado del gen il34 e inyectamos el plásmido en embriones de peces Tg (mpeg1: GFP) de una célula que etiquetaron específicamente los macrófagos por proteína fluorescente GFP. Luego, utilizamos hibridación fluorescente in situ de montaje completo e inmunomancha para detectar el patrón de la expresión il34 y el número o ubicación de los macrófagos. Los embriones WT inyectados fueron levantados para generar una línea transgénica estable. En estos pasos, establecimos y validamos la línea productora de citoquinas y evaluamos visualmente los efectos que se pueden ver en la distribución de los macrófagos. Por último, para investigar el comportamiento de los macrófagos en respuesta a la citoquina, utilizamos imágenes en vivo confocales para observar directamente la migración de macrófagos para confirmar la función de il34 en la migración de macrófagos in vivo.
Protocolo
NOTA: Todas las muestras fueron tratadas con agua de huevo de feniltiourea (PTU) para inhibir el pigmento.
1. Generación de Tg (fabp10a:il34) Construcciones transgénicas e inyección de pescado
- Clonar el promotor fabp10a 8 de 2,8 kb y las regiones de codificación IL-34 (ENSDART00000126460.3) del pez cebra en el vector pTol2 para generar la construcción fabp10a-il34. Inyectar las construcciones en la etapa de una célula Tg (mpeg1: GFP) y WT embriones de peces junto con el ARNm transposasa. Elevar los embriones WT inyectados en fabp10a-il34 aadultos 9 e identificar al fundador transgénico mediante la hibridación in situ.
NOTA: La inyección de la construcción Tol2 directamente en otro transgénico podría ser problemática si la otra línea transgénica se hace con el mismo sistema de transposon. Una práctica general sería hacer una línea transgénica independiente y posteriormente cruzar la nueva línea con otra línea de reportero. Esto garantiza que no habrá efectos de la nueva transgénesis en un transgén insertado previamente.
2. Montaje entero fluorescente in Situ Hybridization (WISH) Combinar con inmunostaining
- Fijación de muestras
- Recoger embriones de inyección transitoria o línea transgénica IL-34 estable que se cruzó con Tg (mpeg1: GFP) en las etapas deseadas.
NOTA: Para este caso, los embriones fueron recogidos a 4 d después de la fertilización (dpf). (Si es necesario) retire el coro con jeringa. - Fijar los embriones con 4% de paraformaldehído (PFA) durante la noche a 4 oC o 2 h a temperatura ambiente (RT) (alrededor de 25 oC).
- Lavar los embriones con solución salina tamponada de fosfato más Tween 20 (PBST) 3x 5 min.
- Deshidratar los embriones por separado con 50% de metanol en PBST (50% metanol/PBST) y 100% metanol, 1x 5 min cada uno. A continuación, cambiar a metanol fresco 100% y almacenar a -20 oC (al menos 2 h).
NOTA: El protocolo se puede pausar aquí.
- Recoger embriones de inyección transitoria o línea transgénica IL-34 estable que se cruzó con Tg (mpeg1: GFP) en las etapas deseadas.
- Hibridación de sondas (Día I)
- Rehidratar los embriones en los pasos anteriores con 50% de metanol en PBST (50% Metanol/PBST), luego lavar con PBST 3x 5 min.
- Digerir los embriones con proteinasa K en PBST a RT (concentración final: 10 g/ml; 1:2000 en PBST).
NOTA: El tiempo de digestión depende de la etapa de los embriones: Menos de 36 h después de la fertilización (hpf), sin necesidad; 36 hpf-2 dpf embrión, 3-5 min; 2-3 dpf embrión, 10 min; 3-4 dpf embrión, 15 min; 4-5 dpf embrión, 15-20 min; 5-6 dpf embrión, 20-27 min; >6 dpf embrión, 25-30 min a RT (alrededor de 25oC). - Deseche la solución de digestión y vuelva a realizar la fijación con 4% de PFA, durante 20 min a RT.
- Lavar los embriones con PBST 2x 10 min.
- Deseche el PBST, realice la prehibridación con búfer de hibridación calentado (buffer HB) a 65 oC durante 5 min, recicle el búfer HB en el tubo original.
- Realizar la pre-hibridación con el nuevo búfer HB calentado a 65 oC al menos 1 h.
- Precalentar la sonda9 (para este caso era una sonda il34, 1 ng/mL) a 65 oC al menos 10 min. A continuación, recicle el búfer HB en el tubo original. Realice la hibridación con la sonda precalentada a 65 oC durante la noche.
- Tratamiento de anticuerpos (Día II)
- Precalienta el 50% de formamida/2x citrato de sodio salino más Tween 20 (SSCT), 2x SSCT, 0.2x SSCT a 65oC.
- Recicle la sonda en el tubo original y almacene la sonda a -20 oC.
- Lavar los embriones por separado con 50% de formamida/2x SSCT; 2x SSCT; 0,2x SSCT, 3x 20 min o 2x 30 min cada uno a 65oC.
- Lavar los embriones con PBST 3x 5 min.
- Bloquear las muestras con 600 l de tampón de bloqueo (5% de suero bovino fetal filtrado (FBS) en PBST) durante 1 h a RT.
- Añadir 400 l de solución de anticuerpos antidigoxigenina-HRP (1:1.000-1:2.000 en tampón de bloqueo) e incubar los embriones a 4 oC durante la noche. Si las señales son débiles, utilice 1:500 dilución de anticuerpos.
- Incubación de anticuerpos primarios y colorantes (Día III)
- Retire el anticuerpo; lavar los embriones con PBST, 6x 20 min a RT.
- Enjuague la muestra con 30 s l de diluyente de amplificación 1x Plus durante 5 min a RT.
- Deseche el diluyente pipeteando; Diluir fluorofore Tyramid stock Solution (Cyanine 3 Plus Amplification Reagent (Cy3) o Cyanine 5 Plus Amplification Reagent (Cy5), para este caso se utilizó Cy3) 1:50 en 1x Plus Amplification Diluent para hacer la solución de trabajo de tiramida de fluoróforo. Prepare 50-100 l de solución de trabajo para cada muestra.
- Incubar la muestra en la solución de trabajo de tiramida de fluoróforo durante 5-15 minutos en la oscuridad a RT. Si las señales son débiles, amplíe el tiempo de incubación a 30 min.
- Detenga la reacción cambiando la solución de trabajo con PBST y examine las señales.
- Lavar los embriones con PBST 3x 10 min a RT.
- Incubar la muestra con anticuerpoprimario a 4oC durante la noche. Para este caso, utilice el anticuerpo Goat-Anti-GFP como anticuerpo primario.
- Tinción secundaria de anticuerpos (Día IV)
- Lavar los embriones con PBST durante 4x 30 min.
- Incubar los embriones con anticuerposecundario a 4oC durante la noche. Para este caso, utilice el anticuerpo Alexa 488-Anti-Goat como anticuerpo secundario.
- Tomar fotos (Día V)
- Lavar los embriones con PBST 3x 10 min a RT.
- Almacenar los embriones en 70% glicerol en la oscuridad a 4 oC durante la noche o -20 oC durante más tiempo.
3. Imágenes en vivo
-
Selección de muestras
NOTA: Utilice la imagen en vivo para observar directamente si los macrófagos de Tg (fabp10a: il34; fabp10a: DsRed; mpeg1: GFP) peces migrarían al hígado bajo IL-34 inducción durante 3-3.5 dpf. Aquí la línea transgénica Tg (fabp10a-DsRed) se utiliza para etiquetar la región hepática y hacerla visible, para facilitar la localización del hígado y para determinar si los macrófagos migran al hígado. Antes de tomar imágenes, utilice un microscopio de fluorescencia para seleccionar los embriones dobles positivos DsRed y GFP. -
Montaje de peces
- Utilice un baño de metal para calentar 1 ml de agarosa de fusión baja al 1% a más de 90 oC para derretirla por completo.
- Después de que la baja agarosa de fusión se enfríe a la temperatura corporal, agregue 50 ml de tricaína al 0,2% y mezcle uniformemente la tricaína con la agarosa.
- Mover los embriones anestesiados a un pequeño plato montado con un tobogán de cubierta en la parte inferior, quitar el agua circundante, soltar lentamente la baja agarosa de fusión en los embriones, establecer cuidadosamente la posición del pez antes de que la agarosa se solidifica, mantener el área del hígado cerca de la cubierta se desliza en la parte inferior del plato.
- Después de que la baja agarosa de fusión se solidifica, cúbrala cuidadosamente con otra capa de agarosa para reforzarla.
- Coloque el plato en la mesa portadora del microscopio confocal, cubra el pescado con la solución E210 con tricaína y comience la toma de imágenes.
-
Funcionamiento del software del microscopio confocal
- Abra el software ZEN black 2.3, instale el banco de trabajo de células vivas en la mesa portadora del microscopio.
- Haga clic en Localizar (Localizar) Incubación ? Temperatura para ajustar la temperatura a 29oC.
- Coloque el plato en el centro de la mesa de trabajo de la célula viva, cubra el pescado con la solución E210 con tricaína.
- Haga clic en el menú Adquisición, seleccione el modo de escaneado y los láseres necesarios en el menú Configuración inteligente y, a continuación, seleccione Z-Stack y Posición.
- Haga clic en el menú Diseñador de experimentos, seleccione Habilitar experimentode bloque múltiple , en el primer bloque, para encontrar la muestra bajo la ampliación baja y, a continuación, cambie a la ampliación alta y, a continuación, deje que el área observada en el centro del campo visual.
- Establezca la posición y la información de Z-Stack, seleccione la intensidad láser adecuada, las capas de escaneo y la velocidad de imagen.
- Cree un nuevo bloque y repita los pasos anteriores. Después de configurar todos los bloques, establezca el número adecuado de bucles y comience a grabar(Figura 1).
Resultados
Los pasos involucrados en el protocolo de pez cebra se ilustran en la Figura 2. En primer lugar, generamos la construcción pBLK-fabp10a-il34-sv40 en la que il34 fue impulsado por el promotor fabp10a (Figura 2). La construcción fue microinyectada en la etapa unicelular Tg (mpeg1: GFP) embriones de pez cebra que pueden etiquetar macrófagos con embriones GFP y WT que fueron elevados a adultos para generar línea estable transgénica(Figura 2). La expresión de il34 fue analizada por hibridación in situ de fluorescencia de montaje completo(Figura 2 y Figura 3). Los macrófagos etiquetados por GFP se analizaron mediante inmunomancha (Figura 2 y Figura 3). Usamos imágenes en vivo para observar directamente si los macrófagos migrarían al hígado bajo la inducción il34 durante 3-3.5 dpf(Figura 2, Figura 4, Película Suplementaria 1 y Película Suplementaria 2) .
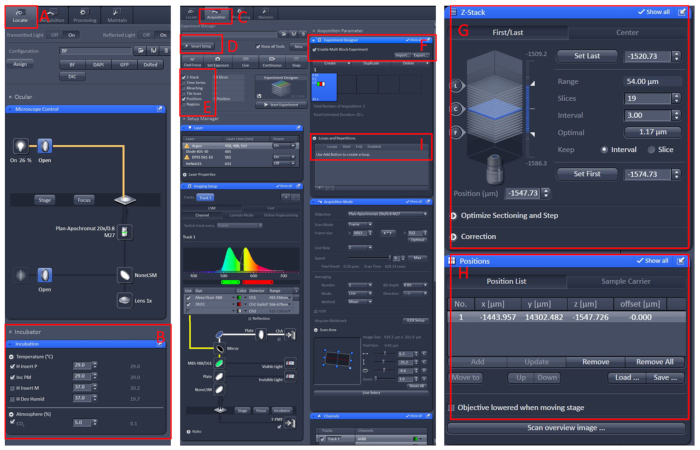
Figura 1: Funcionamiento del software de la imagen en vivo del microscopio confocal. Abra el software ZEN black 2.3, instale el banco de trabajo de la célula viva en la mesa portadora del microscopio y, a continuación, haga clic en Localizar (A) Incubación ? Temperatura (B) para ajustar la temperatura a 29 oC. Coloque el plato en el centro de la mesa de trabajo de la célula viva, cubra el pescado con la solución E210 con tricaína. Después de todo esto, haga clic en el menú Adquisición (C), seleccione el modo de escaneo y los láseres necesarios en el menú Configuración inteligente (D) y, a continuación, seleccione Z-Stack y Posición (E). Por último, haga clic en el menú Diseñador de experimentos (F), seleccione Habilitar experimento de bloque múltiple, en el primer bloque, para encontrar la muestra bajo el aumento bajo, luego cambie a la ampliación alta, deje que el área observada en el centro del campo visual, establezca la posición y la información de Z-Stack (G y H), seleccione la intensidad del láser adecuada, las capas de escaneo y la velocidad de imagen. Cree un nuevo bloque y repita los pasos anteriores. Después de configurar todos los bloques, establezca el número adecuado de bucles (I) y comience a grabar. Haga clic aquí para ver una versión más grande de esta figura.
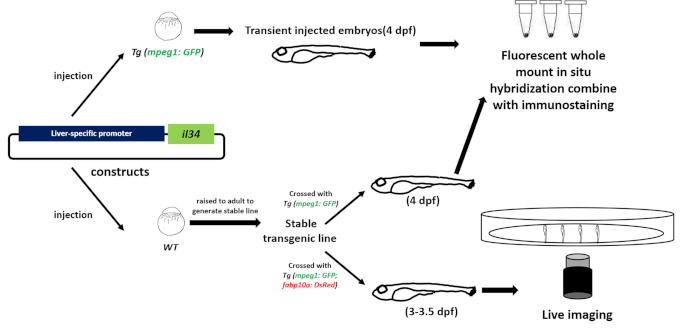
Figura 2: Un flujo de trabajo para investigar la función de una quimiocina en la migración de macrófagos in vivo. Construimos un plásmido de sobreexpresión específico del tejido (hígado) para sobreexpresar IL-34 e inyectamos el plásmido en embriones de peces transgénicos de una fase celular cuyos macrófagos fueron etiquetados específicamente por una proteína fluorescente (Tg: ( )). Los embriones WT inyectados fueron levantados para generar una línea transgénica estable. A continuación, utilizamos hibridación e inmunoutilización fluorescente in situ fluorescentes de montaje entero para detectar el patrón de la expresión génica y el número o ubicación de los macrófagos de los embriones inyectados transitorios o embriones de línea estable (4 dpf). Finalmente, utilizamos imágenes en vivo confocales para observar directamente el comportamiento de los macrófagos en el pez transgénico estable (3-3.5 dpf) para estudiar la función de IL-34 en los macrófagos in vivo. Haga clic aquí para ver una versión más grande de esta figura.
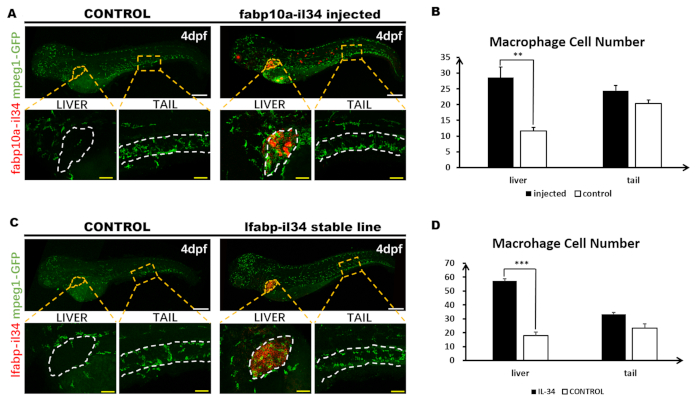
Figura 3: Fluorescent WISH combinar con inmunostaining. Esta cifra ha sido modificada de Jiang et al.11. Un total de 1,8 nL (30 ng/L) de la construcción pBLK-fabp10a-il34-sv40 fue microinyectado en embriones de pez cebra Tg (mpeg1: GFP) en etapa de una célula. (A) WISH de la expresión il34 (rojo) y la tinción de anticuerpos de montaje completo de la expresión GFP (verde) en un embrión de 4 dpf (6x). La imagen de todo el cuerpo del pez se compone de dos imágenes separadas tomadas por confocales y cosidas juntas en Photoshop. Los recuadros son de gran aumento (20x) de las regiones en caja correspondientes (áreas de puntos naranjas). (B) Análisis cuantitativo de los números de células de macrófagos en el hígado de embriones inyectados y construir el hígado de los embriones inyectados (mostrado en el área de puntos blancos) y la región de la cola (aproximadamente entre la somita 13 y la 17a, que se muestra entre dos líneas de puntos blancas). Los datos fueron analizados por la prueba Mann Whitney U, ** p < 0.01 en comparación con el control. n 5, 5 para los peces inyectados de 4 dpf y de control. Barras: 200 m (línea blanca); 50 m (línea amarilla). (C) WISH de expresión il34 y tinción de anticuerpos de montaje completo de la expresión GFP en embrión de línea estable de 4 dpf (6x). La imagen de todo el cuerpo del pez se compone de dos imágenes separadas tomadas por confocales y cosidas juntas en Photoshop. Los recuadros son de gran aumento (20x) de las regiones en caja correspondientes (áreas de puntos naranjas). (D) Análisis cuantitativo de los números de células de macrófagos en Tg (mpeg1: GFP) y Tg (fabp10a: il34; mpeg1: GFP) hígado de embriones (mostrado en el área de puntos blancos) y la región de la cola (aproximadamente entre el 13o y la 17a essomita, que se muestra entre dos líneas de puntos blancas). Haga clic aquí para ver una versión más grande de esta figura.
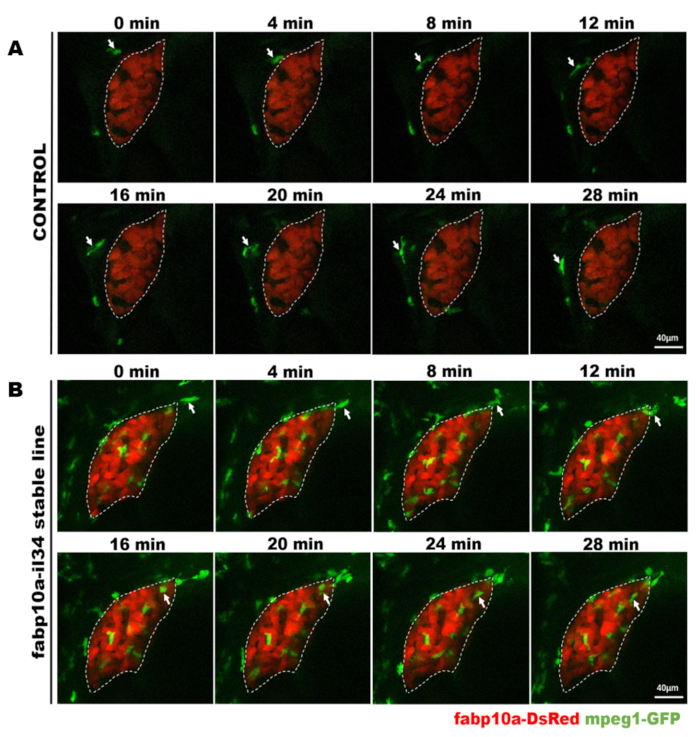
Figura 4: Imágenes en vivo confocales para observar directamente el comportamiento de los macrófagos en los peces transgénicos estables. Esta cifra ha sido modificada de Jiang et al.11. Las micrografías de imágenes en vivo muestran el proceso de un macrófago (verde, etiquetado por flechas blancas) pasando por el hígado (rojo) dentro de 28 min en peces de control (A) y el proceso de un macrófago (verde, etiquetado por flechas blancas) migrando al hígado (rojo) dentro de 28 min en IL-34 pez sobre-expresión (B). Barras de escala a 40 m (línea blanca). Haga clic aquí para ver una versión más grande de esta figura.
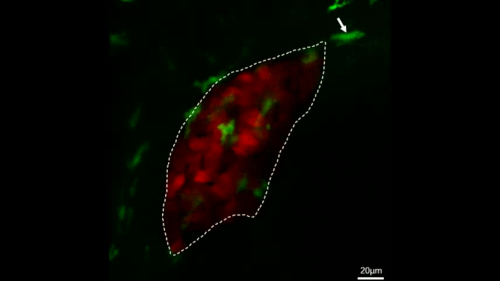
Película suplementaria 1: Imágenes en vivo que muestran el proceso de macrófagos (verde, etiquetados por flechas blancas) migrando al hígado (rojo) dentro de las 2 h en IL-34 sobreexpresando peces. Barras de escala a 20 m (línea blanca). Esta película ha sido reeditada de Jiang et al.11. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)
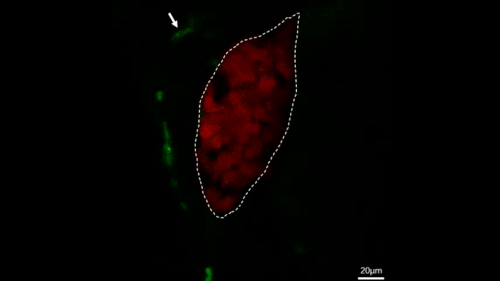
Película suplementaria 2: Imágenes en vivo que muestran el proceso de macrófagos (verde, etiquetados por flechas blancas) vagando por el hígado (rojo) dentro de 2 h en peces de control. Barras de escala a 20 m (línea blanca). Esta película ha sido reeditada de Jiang et al.11. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)
Discusión
El protocolo descrito aquí nos permite investigar la función de una quimiocina sobre el comportamiento de los macrofagoin vivo y el procedimiento requiere cierta experiencia técnica. En resumen, hay varios pasos críticos para evitar complicaciones en el protocolo: 1) seleccionar una línea transgénica adecuada que muestre una señal transgénica específica y fuerte para etiquetar la célula de interés; 2) seleccionar un tejido apropiado al que se pueda acceder a imágenes y sobreexpresión de genes transgénicos; 3) hacer una sonda de ARN sensible y específica; 4) seleccione una ventana de tiempo de observación apropiada para capturar con precisión el comportamiento celular.
En el procedimiento de hibridación in situ de fluorescencia de montaje completo combinada con inmunomanchas, la sonda de ARN utilizada para detectar la expresión génica debe ser sensible y la señal debe ser lo suficientemente fuerte. Con el fin de capturar la función génica en el comportamiento celular, se deben probar una serie de puntos de tiempo. Por ejemplo, al observar el efecto de il34 en la migración de macrófagos, aunque el promotor fabp10a comenzó a expresarse a 2-3 dpf, la acumulación de macrófagos en el hígado no era obvia en ese momento. Es sólo por 4 dpf que el enriquecimiento de macrófagos en el hígado se hace evidente. Además, después de la hibridación in situ, la intensidad de la señal de la inmunomancha posterior se verá afectada. Por ejemplo, en comparación con GFP, DsRed es difícil de colorear en la tinción de inmunofluorescencia después de la hibridación in situ, probablemente debido a las diferentes estructuras proteicas. En términos generales, la intensidad de la señal de inmunomancha después de la hibridación in situ de fluorescencia de montaje completo sería menor que la de la inmunomancha única.
En el paso de imagen en vivo con el microscopio confocal, es necesario mantener la muestra cerca de la parte inferior del plato. Cuando los embriones flotan en agarosa, la distancia de trabajo del objetivo puede ser insuficiente, también, la agarosa entre el objetivo y la muestra afectaría a la calidad de la imagen. Además, el número de muestras para la toma de imágenes en cada momento debe establecerse correctamente. Uno debe asegurarse de que el lapso de tiempo entre dos escaneos de cada pez no sería demasiado largo para perder los detalles del comportamiento celular. Así que este método no es adecuado para el seguimiento de las células que se mueven rápidamente en los tejidos gruesos.
En conclusión, este protocolo se puede utilizar para observar la función de las quimiolinas en el comportamiento de una variedad de células como macrófagos, neutrófilos y células T. Aquí, utilizamos IL-34, un ligando recientemente identificado de la función CSF-1R en quimiotaxis6,7, como un ectópico expresado quimioquina para inducir la migración de macrófagos. La mayoría de los modelos experimentales existentes de quimiotaxis celulares se basan en experimentos con células in vitro, pero los experimentos in vitro a veces son demasiado simples para modelar el complejo entorno in vivo. Además, es difícil imaginar la capacidad de quimio-atracciónin vivo cuando simplemente mira la situación in vitro. Este método utilizó las ventajas específicas del pez cebra para la observación directa del comportamiento celular, que es difícil para los ratones. El método actual nos permitió probar rápidamente las funciones de la quimiocina en los comportamientos celulares dentro de varios días y hacer del pez cebra un modelo poderoso para estudiar la biología molecular y celular.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al Dr. Jingrong Peng por compartir la línea transgénica Tg (fabp10a: DsRed); Dr. Zilong Wen por compartir las líneas transgénicas Tg (mpeg1: GFP); Dr. Koichi Kawakami por proporcionar el vector pTol2. Este trabajo fue apoyado por la Fundación Nacional de Ciencias Naturales de China (31771594), los proyectos del Plan de Ciencia y Tecnología de Guangdong (2019A030317001) y los Fondos Fundamentales de Investigación para las Universidades Centrales (D2191450).
Materiales
| Name | Company | Catalog Number | Comments |
| Antibody | |||
| Alexa 488-Anti-Goat antibody | Invitrogen | A11055 | |
| Anti-Digoxigenin-HRP | perkinelmer | NEF832001EA | |
| Goat-Anti-GFP antibody | Abcam | ab6658 | |
| Reagent | |||
| CaCl2· 2H2O | Sigma | 21097 | |
| Cyanine 3 Plus Amplification Reagent | perkinelmer | NEL745001KT | |
| E2 solution | 15 mM NaCl +0.5 mM KCl +1.0 mM MgSO4+150 µM KH2PO4 + 50 µM Na2HPO4 +1.0 mM CaCl2 + 0.7 mM NaHCO3 | ||
| Fetal Bovine Serum (FBS) | Life | 10099-133 | |
| Formamide | Diamond | A100314 | |
| Glycerol | Sigma | V900860 | |
| Heparin sodium | Sigma | H3149 | |
| Hybridization buffer(HB) | 50% formamide+ 5×SSC+9 mM sodium citrate+50 μg/ml heparin sodium+ 500 μg/mL tRNA+ 0.1% Tween20 | ||
| KCl | Sigma | P5405 | |
| KH2PO4 | Sigma | P5655 | |
| Low melting agarose | Sigma | A9414 | |
| Methanol | GHTECH | 1.17112.023 | |
| Methylene blue | Sigma | M9140 | |
| MgSO4 | Sigma | M2643 | |
| Na2HPO4 | Sigma | S5136 | |
| NaCl | Sigma | S5886 | |
| NaHCO3 | Sigma | S5761 | |
| Paraformaldehyde (PFA) | Sigma | 158127 | Suspend 16 g of PFA in 400 ml of 1x PBS, heat at 60 °C to dissolve about 30 min. This solution can be prepared in advance and stored at -4 °C. Caution. Manipulate with mask. |
| 10×PBS | 14.2 g Na2HPO4+80 g NaCl+2 g KCl+ 2.4 g KH2PO4 in 1L ddH2O | ||
| Phenylthiourea (PTU) | Sigma | P7629 | |
| 1×Plus Amplification Diluent | perkinelmer | NEL745001KT | |
| Proteinase K | Fermentas | E00492 | |
| 20×Saline sodium citrate(SSC) | 175.3 g NaCl+ 88.2 g sodium citrate in 1 L ddH2O, PH 7.0 | ||
| Sodium citrate | Sigma | A5040 | |
| Tricaine | Sigma | E10521 | |
| tRNA | Sigma | R6625 | |
| Tween20 | Sigma | P2287 | |
| Plasmid | |||
| pBLK-fabp10a-il34-sv40 | For Tg (fab10a:il34) transgenic line generation | ||
| pBSK-il34 | For il34 probe preparation | ||
| Fish | |||
| Tg (mpeg1: GFP) | Label macrophages with GFP | ||
| Tg (fabp10a: DsRed) | Label liver cells with DsRed | ||
| Tg (fab10a:il34) | Over-expression IL-34 in liver cells |
Referencias
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Wang, Y., et al. IL-34 is a tissue-restricted ligand of CSF1R required for the development of Langerhans cells and microglia. Nature Immunology. 13 (8), 753-760 (2012).
- Lin, H., et al. Discovery of a cytokine and its receptor by functional screening of the extracellular proteome. Science. 320 (5877), 807-811 (2008).
- Wei, S., et al. Functional overlap but differential expression of CSF-1 and IL-34 in their CSF-1 receptor-mediated regulation of myeloid cells. Journal of leukocyte biology. 88 (3), 495-505 (2010).
- Etienne, D., Foucher, S. B. L. P., Norbert Ifrah, P. G. Y. D. IL-34 Induces the Differentiation of Human Monocytes into Immunosuppressive Macrophages. Antagonistic Effects of GM-CSF and IFNc. PLoS One. 8 (2), e56045(2013).
- Segaliny, A. I., et al. Syndecan-1 regulates the biological activities of interleukin-34. Biochimica et Biophysica Acta. 1853 (5), 1010-1021 (2015).
- Zhou, S. L., et al. miR-28-5p-IL-34-macrophage feedback loop modulates hepatocellular carcinoma metastasis. Hepatology. 63 (5), 1560-1575 (2016).
- Gordon, J. I., et al. Tissue specific expression and developmental regulation of two genes coding for rat fatty acid binding proteins. Journal of Biological Chemistry. 260 (4), 1995-1998 (1985).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio rerio). , University of Oregon. Eugene, OR. (2000).
- Nüsslein-Volhard, C., Dahm, R. Zebrafish: a practical approach. , Oxford University Press. New York. (2002).
- Jiang, Y., Chen, J., Yen, K., Xu, J. Ectopically Expressed IL-34 Can Efficiently Induce Macrophage Migration to the Liver in Zebrafish. Zebrafish. 16 (2), 165-170 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ISSN 2578-2746
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados
Utilizamos cookies para mejorar su experiencia en nuestra página web.
Al continuar usando nuestro sitio web o al hacer clic en 'Continuar', está aceptando nuestras cookies.
