Method Article
In Vivo Monitoraggio dell'espressione genica orologio circadiano nel nucleo soprachiasmatico Mouse mediante fluorescenza reporter
In questo articolo
Riepilogo
Questa nuova tecnologia basata sulla fluorescenza consente il monitoraggio a lungo termine della trascrizione dei geni dell'orologio circadiano nel nucleo soprachiasmatico (SCN) di topi liberi di muoversi in tempo reale e ad alta risoluzione temporale.
Abstract
Questa tecnica combina fibra ottica mediata fluorescenza registrazioni con la consegna precisa dei reporter gene ricombinante virus adeno-associato basato. Questo sistema monitoraggio nuovo e facile da usare in vivo di fluorescenza è stato sviluppato per registrare il ritmo trascrizionale del gene dell'orologio, Cry1, nel nucleo soprachiasmatico (SCN) di topi liberi di muoversi. A tal fine, un reporter di fluorescenza di trascrizione Cry1 è stato progettato e confezionato in virus Adeno-associato. Purificata, concentrato di virus è stato iniettato nel mouse SCN seguita dall'inserimento di una fibra ottica, che è stato poi fissata sulla superficie del cervello. Gli animali sono stati restituiti alle loro gabbie casa e concesso un periodo di recupero post-operatorio 1 mese assicurare sufficiente reporter espressione. Fluorescenza è stato poi registrato in topi liberi di muoversi attraverso un in vivo monitoraggio sistema che è stato costruito alla nostra istituzione. Per l' in vivo sistema di registrazione, un laser a 488 nm è stato accoppiato con un divisore di fascio 1 × 4 che divide la luce in quattro uscite di eccitazione laser di pari potenza. Questa impostazione ci ha permesso di registrare contemporaneamente da quattro animali. Ciascuno dei segnali di fluorescenza emessa è stato raccolto tramite un tubo di fotomoltiplicatore e una scheda di acquisizione dati. Al contrario per la bioluminescenza in vivo registrazione orologio circadiano tecnica precedente, questa fluorescenza in vivo sistema di registrazione ha permesso la registrazione dell'espressione genica orologio circadiano durante il ciclo di luce.
Introduzione
Nei mammiferi, il nucleo soprachiasmatico (SCN) governa il ritmo circadiano del corpo intero per coordinare la risposta di un individuo a cambiamenti ambientali esogeni (ad es., luce, temperatura, stress, ecc.)1. Componenti di base orologio consistono di Per1-3, Cry1-2, orologioe Bmal1e svolgono un ruolo centrale nel regolare l'orologio circadiano di ciascuna cella. Ogni cella in SCN contiene l'attivatore trascrizionale, orologio/BMAL1, che agisce come un eterodimero di indurre l'espressione di PER e piangere. Il PER / CRY complesso quindi inibisce la funzione di orologio/BMAL1 per formare un ciclo di feedback di trascrizione-traduzione che prende circa 24 h a completa2,3.
Gli studi precedenti su SCN principalmente impiegato l'ex vivo SCN fetta cultura metodo4,5,6 e, mentre questo approccio ha fornito preziose informazioni, suoi limiti hanno inibito la nostra capacità di ottenere i dati per quanto riguarda l'influenza di altri nuclei di cervello su SCN, come pure l'effetto di stimoli esogeni (ad es., luce) sulle cellule che risiedono in questa regione critica. Nel 2001, gruppo di Hitoshi Okamura è stato il primo ad utilizzare il sistema di bioluminescenza per in vivo l'espressione genica monitor orologio circadiano in SCN in topi7liberi di muoversi. Gruppo di Ken-ichi Honma ha trascorso alcuni anni sviluppando ulteriormente la bioluminescenza in vivo sistema di registrazione in SCN8,9,10. Insieme, questi studi hanno fornito i ricercatori con la possibilità di monitorare l'orologio circadiano in costante oscurità o dopo un impulso di luce. Tuttavia, poiché la bioluminescenza è troppo debole per consentire il monitoraggio continuo durante il ciclo luce/buio, accoppiato con il fatto che la luce è il predominante segnale necessario per il trascinamento di SCN-mediata di orologi circadiani11, c'è la crescente domanda per lo sviluppo di metodi sperimentali che superare i limiti associati con registrazione di bioluminescenza. Il rapporto corrente descrive un sistema basato su fluorescenza che è stato costruito per monitorare l'orologio circadiano del SCN in vivo in topi liberi di muoversi. Questo metodo facile da usare consente un monitoraggio continuo durante il ciclo luce/buio e per l'osservazione a lungo termine della trascrizione dei geni dell'orologio circadiano in SCN in tempo reale e ad alta risoluzione temporale.
Protocollo
Tutte le procedure in questo protocollo sono state condotte con l'approvazione del istituzionale Animal Care e uso Committee (IACUC) dell'Istituto nazionale di scienze biologiche, Pechino, in conformità con i regolamenti governativi della Cina.
1. costruzione del Cry1 fluorescenza Reporter
Nota: Gli studi circadiani precedenti utilizzando la bioluminescenza sistema2,12,13 fusibile un promotore circadiano con un destabilizzato luciferasi (dLuc) di indurre l'espressione ritmica dLuc . Per sviluppare un reporter di fluorescenza, destabilizzato luciferasi è stato sostituito con la proteina destabilizzato fluorescenza (dVenus) di indurre l'espressione ritmica dVenus . La generazione del P (Cry1)-dVenus reporter è descritto di seguito, il costrutto di cui attualmente sta per essere inviato a Addgene e sarà presto disponibile per uso accademico.
- Amplificare il promotore di Cry1 mouse funzionale (+328 regione-1208 bp del gene di Cry1 , sito di inizio della trascrizione designato come '-1 '). Usano l'enzima PCR (KOD Plus Neo), iniettori F1 e R1 (vedere tabella 2), mouse genoma come un modello e PCR del produttore protocollo per amplificare il frammento di DNA del promotore di Cry1 .
- Amplificare introne 336 del gene Cry1 , che è importante per la funzione di orologio circadiano di Cry114. Usano l'enzima PCR, primer F2 e R2, genoma di topo come un modello e protocollo PCR del produttore per amplificare il frammento di DNA introne 336.
- Amplificare Venus. Usano l'enzima PCR, primer F3 e R3, pVENUS-N1 plasmide come un modello e protocollo PCR del produttore per amplificare il frammento di DNA di Venus .
- Amplificare il frammento del DNA NLS-D2. Usano l'enzima PCR, primer F4 e R4, plasmide pcDNA3.3-d2eGFP come un modello e protocollo PCR del produttore per amplificare il frammento di DNA di NLS-D2.
- Sintetizzare il frammento di DNA 1 come esone 1.
- Sintetizzare il frammento di DNA 2 come il linker tra introne 336 e il frammento di DNA di Venus .
- Tagliare il pAAV-EF1a-double floxed-hChR2 (H134R) - mCherry - WPRE-plasmide vettore HGHpA con gli enzimi MluI ed EcoRI. Utilizzare il Kit di estrazione del Gel per purificare il frammento di DNA più grande. Utilizzare il protocollo del produttore.
- Utilizzando un mix di assemblaggio di Gibson e il suo protocollo standard, assemblare tutti i frammenti di DNA insieme per produrre il reporter-intron336-Venus-NLS-D2-WPRE-HGHpA pAAV-P (Cry1). Utilizzare il protocollo del produttore.
2. produzione di Virus Adeno-associato15
- Preparare il plasmide seguente stock di DNA: 31,25 µ g di pRV1, 31,25 µ g di pH21, 125 µ g di pFdelta6 e 62,5 µ g di pAAV-P (Cry1) - intron336 - Venus-NLS - D2-WPRE-HGHpA.
- Preparare cinque piatti di 15cm di cellule 293T e attendere fino a quando le cellule diventano 90% confluenti.
- Transfect i plasmidi nelle cellule 297T seguendo il protocollo del reagente di transfezione. Per ogni piatto, utilizzare 6,25 µ g di pRV1, 6,25 µ g di pH21, 25 µ g di pFdelta6, 12,5 µ g di pAAV-P (Cry1)-intron336-Venus-NLS-D2-WPRE-HGHpA e 120 µ l di reagente di transfezione.
- 72 ore dopo la trasfezione, staccare le cellule da ogni piatto usando una pistola di pipetta; raccogliere le cellule in due provette da 50 mL.
- Agglomerare le cellule a 800 x g per 10 min e scartare il surnatante. Lavare le cellule mettendo 20 mL di tampone fosfato salino (PBS) in ogni provetta; agglomerare le cellule a 800 x g per 10 min e scartare il surnatante.
- Risospendere il pellet in NaCl 150 mM e 20 mM Tris soluzione a pH 8.0; utilizzare 25 mL di soluzione/tubo. Aggiungere 1,25 mL di 10% sodio desossicolato preparata al momento (in dH2O) ad ogni provetta. Aggiungere benzonase nucleasi a una concentrazione finale di 50 unità per mL e mescolare accuratamente. Incubare a 37 ° C per 1 h.
- Rimuovere detriti cellulari mediante centrifugazione a 3000 x g per 15 min. Garantire che tutti i detriti sono stato rimosso spingendo la miscela attraverso un filtro di 0,45 μm siringa; Questo passaggio vi aiuterà a evitare il blocco delle colonne di eparina.
- Impostare le colonne di eparina mediante una pompa peristaltica; impostare la portata a 1 mL/min, assicurando che bolle d'aria non vengono introdotti nelle colonne.
- Equilibrare le colonne con 10 mL di un 150 mM NaCl, 20 mM Tris soluzione a pH 8.0. Aggiungere la soluzione contenente il virus a ogni colonna. Lavare le colonne con 20 mL di un 100 mM NaCl, 20 mM Tris soluzione a pH 8.0. Poi, usando una siringa da 5 mL, lavare le colonne con 1 mL di NaCl-un 200 mM e 20 mM Tris soluzione pH 8.0, seguite da 1 mL di un 300 mM NaCl e soluzione di 20 mM Tris, pH 8.0.
- Usare una siringa da 5 mL per eluire il virus con 3 mL di un 400 mM NaCl, 20 mM Tris soluzione, pH 8.0, seguito da 4 mL di un 450 mM NaCl, 20 mM Tris soluzione, pH 8.0 e infine da un 3 mL di un 500 mM NaCl , soluzione di 20 mM Tris, pH 8.0. Raccogliere i materiali estratti.
- Per la concentrazione di virus adeno-associato ricombinante (rAAV), caricare i materiali estratti, 4 mL alla volta, in unità di filtraggio centrifugo con un peso molecolare 100.000 cutoff. Centrifugare ciascun campione di 4 mL a 2000 x g per 5 min a temperatura ambiente, scartando il flusso continuo.
- Quando tutti i materiali estratti sono stati eseguiti tramite le unità filtro centrifugo, aggiungere 4 mL di PBS e centrifugare la miscela in circa 250 μL. Rimuovere rAAVs (titolo virale dovrebbe essere di circa 2-8 x 1012 particelle virali per mL) dall'unità di filtraggio centrifugo, aliquotare e conservare a-80 ° C fino all'utilizzo. In questa fase, il virus può essere memorizzato per diversi mesi.
3. rAAV iniezione e ottica fibra inserimento nell'adulto Mouse SCN
- Iniezione di circadiano fluorescenza reporter-trasportare rAAVs in SCN
- Caricare 5 µ l di rAAV (il titolo del virus è di circa 2-8 x 1012 particelle di virus per millilitro) in una micro-siringa.
- Anestetizzare un topo adulto (sfondo di 2 - a 5-month-old, C57BL/6) con Nembutal (80 mg/kg) tramite l'iniezione intraperitoneale. Verificare la mancanza di una risposta di punta-pizzico sufficiente profondità dell'anestesia.
- Usare le forbici per rimuovere il pelo dalla testa del mouse e poi montare il mouse in un apparato stereotassica. Fornire una fonte di calore per animali anestetizzati tramite una circolazione acqua calda coperta o un altro dispositivo di riscaldamento.
Nota: In alternativa, utilizzare il tagliacapelli elettrico o crema depilatoria per rimuovere il pelo. - Pulire il sito chirurgico con gli agenti disinfettanti appropriati. Ad esempio, potrebbe includere acqua sterile o alcool e iodio, betadine, o diluire clorexidina in alternanza scrub. Ripetere almeno 3 volte. Drappo sito chirurgico.
- Utilizzare le forbici per fare un'incisione nel cuoio capelluto ed esporre il cranio.
- Regolare l'apparato stereotassica affinché bregma e lambda sono sullo stesso piano orizzontale; fare due punti simmetrici di bregma sullo stesso piano orizzontale.
- Utilizzare un tampone di cotone sterile imbevuto di 4% H2O2/h2O a corrodere il periostio del cervello. Quindi utilizzare un batuffolo di cotone pulito e sterile per cancellare e asciugare la superficie del cranio.
- Utilizzando una micro punta, fare un foro di circa 1 mm di diametro 0,46 mm posteriormente e 0,25 mm laterale di bregma.
- Fi Utilizzare un tampone sterile di cotone pulito con soluzione fisiologica per cancellare frammenti di ossa dalla superficie esposta del cervello.
- Inserire una micro-siringa nel cervello, assicurando che la punta della micro-siringa è 0,46 mm posteriore e laterale di 0,25 mm di bregma e 5,7 mm di profondità dalla superficie del cranio.
- Iniettare 500 nL di rAAV (50 nL min-1) in SCN, lasciando la micro-siringa in posizione per 10 minuti dopo l'iniezione per garantire completa diffusione di rAAV.
- Estrarre lentamente la micro-siringa.
- Inserire la fibra ottica nell'area SCN.
- Usando un coltello di fibra ottica, tagliare una fibra 17 mm di lunghezza, facendo in modo che ogni lato della fibra ottica è liscia.
- Inserire la fibra ottica ferrula in ceramica e fissare la fibra ottica a una ghiera in ceramica con perossido etilico metilico del chetone (colla di AB), garantendo che le superfici della fibra ottica e puntale di ceramica sono a filo.
- Pulire la fibra con una soluzione o etilene ossido di sterilizzanti a freddo.
- Dopo l'iniezione di rAAV (punto 3.1.11), inserire la fibra ottica ferrula contenenti ceramica SCN.
- Difficoltà il puntale di ceramica e fibra ottica sul cranio usando resina dentale e lasciare per asciugare.
- Spalmare la superficie della resina dentale con smalto posteriore. Ripetere le procedure descritte in 3.1.2 a 3.2.5, omettendo l'effettiva iniezione di rAAV per controllo.
- Cura post-chirurgica
- Fornire per via sottocutanea, topi con analgesici post-operatori, come buprenorfina 2 mg/kg di peso corporeo, due volte al giorno.
- Posizionare i topi in una gabbia di recupero mentre escono anestesia.
- Mouse della casa individualmente in una condizione di luce/buio di 12 h (intensità luminosa lux ~ 100 nella parte inferiore della gabbia) con cibo/acqua disponibile ad libitum. Prima di ulteriori sperimentazioni per consentire tempo sufficiente di recupero e per assicurare ottimale fluorescenza reporter espressione è opportuno attribuire ad otto giorni.
4. in vivo fluorescenza segnale monitoraggio e raccolta dati
- Otto giorni dopo la chirurgia, collegare la fibra sulla testa del mouse con il monitor del segnale di fluorescenza (ad es., Gene Observer). Controllare il segnale di fluorescenza in SCN dei topi di controllo per stabilire il segnale di fondo. Condurre l'osservazione e la misurazione di questo segnale utilizzando monitor di segnale di fluorescenza.
- Valutare il segnale di fluorescenza di sperimentale del topo con la stessa operazione di controllo del mouse. Escludere i topi che hanno ricevuto il vettore virale ma che visualizza solo il segnale di fondo da analisi, come questo probabilmente indica che la punta della fibra ottica ha perso il SCN. Dopo queste prime misure, tornare topi alla loro casa gabbie per altre 3 settimane consentire il segnale di fluorescenza stabilizzare.
- Dopo 3 settimane, è necessario collegare i topi con il monitor del segnale di fluorescenza. Misurare la fluorescenza per 15 s ogni 10 min, ad una frequenza di 100 Hz. Durante queste misurazioni, i topi sono liberamente commoventi nelle loro gabbie con cibo/acqua disponibile ad libitum. Utilizzare un'intensità luminosa nella parte inferiore della gabbia di ~ 100 lux e una potenza del laser sulla punta della fibra ottica tra 15-20 µW.
- Dopo il completamento di tutte le registrazioni, irrorare topi con paraformaldeide al 4% e rimuovere e affettare il cervello per controllare il posizionamento della fibra ottica. Escludere i topi da analisi se la punta della fibra ottica non è correttamente impiantata in SCN.
5. presentazione e analisi di dati
- Segnale di fluorescenza di misura ogni 10 min per 15 s, ad una frequenza di 100 Hz, per produrre 1500 dati punti. La media di questi 1500 punti si traduce il segnale di fluorescenza in qualsiasi dato momento. Stampa più medie nel tempo produce una curva di segnale-a-tempo che corrisponde al ritmo trascrizionale di Cry1 sotto una condizione di luce di controllo in SCN di topi liberi di muoversi.
Risultati
Progettazione di reporter di fluorescenza di Cry1 era spettacolo in Figura 1A. Utilizzando l'approccio descritto sopra, 500 nL di rAAV-P (Cry1) - intron336 - Venus-NLS-D2 con successo è stato iniettato in SCN di un topo adulto ed esposto robusta espressione di Venere (Figura 1B, 1C). I segnali di fluorescenza registrati in condizioni di buio/buio (DD) (Figura 2) e 12h / 12h chiaro/scuro (LD) ha reso robusto ritmo circadiano in entrambe le condizioni. Infine, il segnale di fluorescenza è stato registrato in condizioni di lunga e breve fotoperiodo (Figura 3).
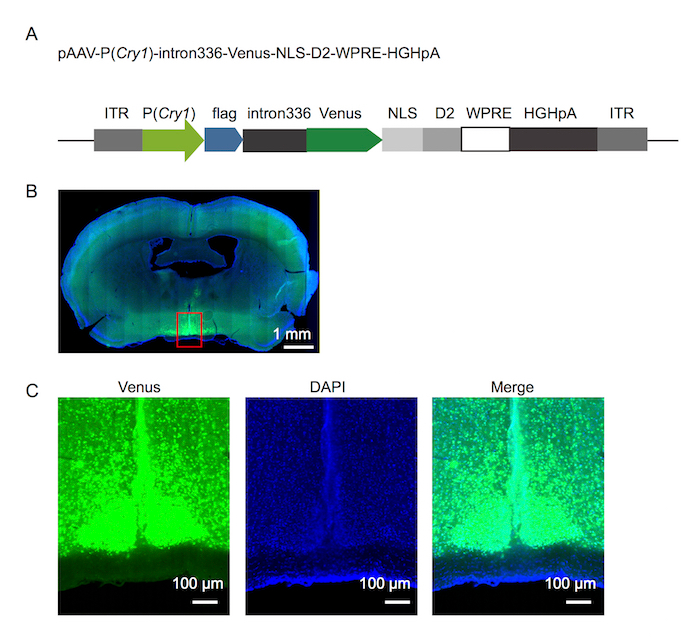
Figura 1. Fluorescenza reporter design ed espressione in SCN
(A) la progettazione di reporter di fluorescenza di Cry1. (B) microfotografia del cervello del mouse mostrano espressione reporter 8 giorni dopo l'iniezione di virus. (C) ingrandimento dell'area inscatolato in rosso la microfotografia illustrata in (B). Clicca qui per visualizzare una versione più grande di questa figura.
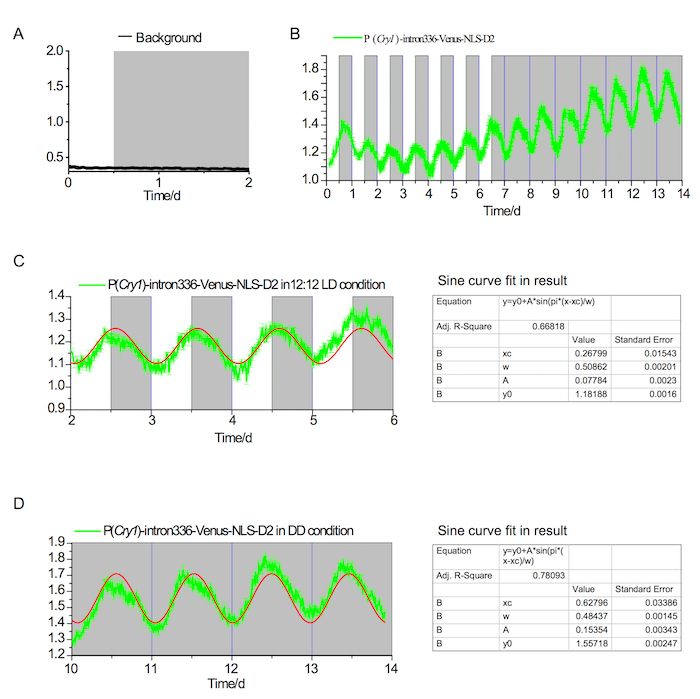
Nella figura 2. Fluorescenza in vivo di registrazione in SCN condizioni LD e DD
(A) Background fluorescenza in SCN non risultati ritmo rilevabile in condizioni LD e DD. Le aree bianche e grigie indicano periodi di luce e luce-fuori, rispettivamente. (B) P (Cry1) - intron336 - Venus-NLS-D2 espressione nel corso di 7 giorni in una condizione di LD 12-12 e 7 giorni in una condizione DD. (C) analisi del ritmo circadiano nella condizione di LD (~24.4 h); la linea rossa indica il convergente adatta nella curva sinusoidale (il risultato della misura viene visualizzato nel pannello di sinistra). (D) analisi del ritmo circadiano nella condizione di DD (~23.25 h); la linea rossa indica il convergente adatta nella curva sinusoidale (il risultato della misura viene visualizzato nel pannello di sinistra). Clicca qui per visualizzare una versione più grande di questa figura.
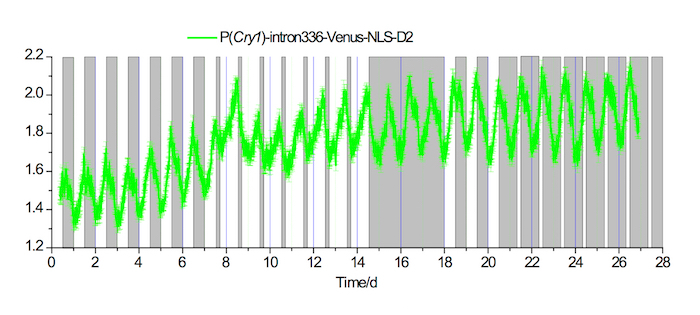
Nella figura 3. In vivo le registrazioni di fluorescenza in SCN sotto lunga 20-4 e 4-20 breve fotoperiodo condizioni
Esempio di una registrazione di fluorescenza di P (Cry1) - intron336 - Venus-NLS-D2 espressione in SCN per 7 giorni in una condizione di LD 12-12, seguita da 7 giorni in una condizione di fotoperiodo lungo 20-4, 3 giorni in una condizione DD, 3 giorni in una condizione di LD 12-12, e quindi 7 giorni in una condizione di breve fotoperiodo 4-20. Aree bianche e grigie indicano i periodi di luce e luce-fuori, rispettivamente. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1. Reagenti specifici necessari per il protocollo (Vedi Tabella materiali)
| nome di primer | sequenza | ||
| Primer F1 | cactaggggttcctgcggccgcACGCGTGTAAAGATGCACATGTG | ||
| Primer R1 | TTGTAACCTTGATACTTACCTACTTAGATCGCAGATCTCGTCCGG | ||
| Primer F2 | GTTATGACACAGTGTAGAAACTATGGCATAGGACAGATGACTGTG | ||
| Primer R2 | CATGGTCTTTGTAGTCCATGGTGGGTACCtCTTGACAGCTCTACC | ||
| Primer F3 | ctgtattttcagggcCCTGCAGGtGTGAGCAAGGGCGAGGAGCTG | ||
| Primer R3 | TACCTTTCTCTTCTTTTTTGGAGGCTTGTACAGCTCGTCCATGCC | ||
| Primer F4 | GCATGGACGAGCTGTACAAGCCTCCAAAAAAGAAGAGAAAGGTAG | ||
| primer R4 | tgatatcgaattcGGATCCCTACACATTGATCCTAGCAGAAGCAC | ||
Tabella 2. Primer utilizzati nel protocollo
Tabella 3. Sequenza di DNA utilizzata nel protocollo Clicca qui per scaricare questo file.
Discussione
Contrariamente ai metodi di ex vivo , come fetta cultura4,5, RT-PCR16e in situ ibridazione17, che richiedono che gli animali abbattuti, l' in vivo registrazione metodo consente ricercatori a studiare l'espressione genica circadiana in un animale vivente. Come tale, questa tecnologia offre la possibilità di valutare l'effetto delle perturbazioni fisiche differenti (ad es., privazione del sonno, lo stress, l'assunzione di cibo, ecc.) sull'orologio circadiano neurale. In contrasto con l'in vivo bioluminescenza7,8,10 metodo che può funzionare solo in condizioni di buio costante, la fluorescenza in vivo di registrazione metodo può essere applicato sotto la luce condizioni, un importante vantaggio della tecnologia come luce segnala ri-trascinamento dell' orologio circadiano SCN18. Anche se relativamente costanti, robusti i ritmi circadiani possono essere rilevati con questo metodo, va ricordato che la tecnologia di reporter non fornisce informazioni quantitative e non può quindi essere utilizzata per misurare i livelli di trascrizione genica e traduzione.
Ci sono molti passaggi critici del presente protocollo. Il titolo di rAAV concentrato dovrebbe essere alto come 2-8 x 1012 particelle virali per mL; i titoli virali più bassi darebbe i segnali di fluorescenza debole o addirittura non rilevabile. Nel fissare la testa del mouse nell'apparecchio stereotassica, assicurarsi che il cranio sia orizzontale per un posizionamento preciso sicuro per l'impianto di iniezione e fibra ottica di virus. Iniezione del virus deve essere fatto lentamente (≤ 50 nL min-1) per garantire che il virus rimane in SCN. Il puntale di ceramica e fibra ottica deve essere fissati saldamente sul cranio e resina dentale non venga a contatto della pelle. Se il puntale di ceramica e la fibra ottica non sono adeguatamente corretti, essi gradualmente si staccherà dalla testa del mouse.
Questa tecnica può essere estesa per includere altri reporter gene (ad es., Per2, Bmal1, c-Fos, Pomc, ecc.) modificando il promotore e il corrispondente introne19. Invertendo la cassetta di bandiera-inton336-Venus-NLS-D2 con due sequenze di lox invertita e combinando il reporter con una linea di topo Cre, questa tecnologia consente di registrare l'attività in neuroni specifici. Ad esempio, combinando questo approccio con una linea di topo Vip-cre consentirebbe per la registrazione di Cry1 ritmi trascrizionale nei neuroni esprimenti polipeptide intestinali vasoattivi in SCN. Questo metodo può anche impiegare GCaMP6 come un indicatore di Ca2 + per registrare il ritmo circadiano di Ca2 + nel cervello.
Divulgazioni
Gli autori non hanno conflitti di interesse a divulgare.
Riconoscimenti
Ringraziamo i membri del laboratorio di Zhang per la fornitura di stimolare discussioni e membri del laboratorio di Zhan per assistenza tecnica. Questa ricerca è stata sostenuta da sovvenzioni 31500860 (a C.Z.) di NSFC, 2012CB837700 (per E.E.Z. e C.Z.) del programma 973 dal M.O.S.T. della Cina e da finanziamenti dal governo municipale di Pechino. E.E.Z. è stato sostenuto dai cinesi "Programma di reclutamento di esperti di gioventù globale".
Materiali
| Name | Company | Catalog Number | Comments |
| KOD Plus Neo | TOYOBO | KOD-401 | Reagent |
| pVENUS-N1 | addgene | #61854 | Plasmid |
| pcDNA3.3_d2eGFP | addgene | #26821 | Plasmid |
| pAAV-EF1a-double floxed-hChR2(H134R)-mCherry-WPRE-HGHpA | addgene | #20297 | Plasmid |
| MluI | Thermo Scientific | FD0564 | Reagent |
| EcoRI | Thermo Scientific | FD0274 | Reagent |
| Gibson Assembly Mix | NEB | E2611s | Reagent |
| Lipofectamine 2000 | Thermo Scientific | 12566014 | Reagent |
| Syringe Filter | EMD Millipore | SLHV033RS | 0.45 µm |
| HiTrap heparin columns | gelifesciences | 17-0406-01 | 1 mL |
| Amicon ultra-4 centrifugal filter | EMD Millipore | UFC810024 | 100,000 MWCO |
| Benzonase nuclease | Sigma-Aldrich | E1014 | Reagent |
| Sodium deoxycholate | Sigma-Aldrich | D5670 | Make fresh solution for each batch |
| mouse stereotaxic apparatus | B&E TEKSYSTEMS LTD | #SR-5M/6M | Equipment |
| pentobarbital | SigmaAldrich | #1507002 | Reagent |
| mouse stereotaxic apparatus | B&E TEKSYSTEMS LTD | #SR-5M/6M | Equipment |
| Hydrogen peroxide solution | SigmaAldrich | #216763 | Reagent |
| Optical Fiber | Thorlabs | FT200EMT | 0.39 NA, Ø200 µm |
| microsyringe pump | Nanoliter 2000 Injector, WPI | Equipment | |
| ceramic ferrule | Shanghai Fiblaser | 230 μm I.D., 2.5 mm O.D. | |
| Gene Observer | BiolinkOptics | Equipment |
Riferimenti
- Welsh, D. K., Takahashi, J. S., Kay, S. A. Suprachiasmatic nucleus: cell autonomy and network properties. Annual Review of Physiology. 72, 551-577 (2010).
- Zhang, E. E., Kay, S. A. Clocks not winding down: unravelling circadian networks. Nature Reviews Molecular Cell Biology. 11, 764-776 (2010).
- Takahashi, J. S. Transcriptional architecture of the mammalian circadian clock. Nature Reviews Genetics. 18, 164-179 (2017).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences U S A. 101, 5339-5346 (2004).
- Davidson, A. J., Castanon-Cervantes, O., Leise, T. L., Molyneux, P. C., Harrington, M. E. Visualizing jet lag in the mouse suprachiasmatic nucleus and peripheral circadian timing system. European Journal of Neuroscience. 29, 171-180 (2009).
- Savelyev, S. A., Larsson, K. C., Johansson, A. S., Lundkvist, G. B. Slice preparation, organotypic tissue culturing and luciferase recording of clock gene activity in the suprachiasmatic nucleus. Journal of Visualized Experiments. (48), (2011).
- Yamaguchi, S., et al. Gene expression: View of a mouse clock gene ticking. Nature. 409, 684-684 (2001).
- Ono, D., Honma, K. I., Honma, S. Circadian and ultradian rhythms of clock gene expression in the suprachiasmatic nucleus of freely moving mice. Science Reports. 5, 12310(2015).
- Ono, D., Honma, S., Honma, K. Circadian PER2::LUC rhythms in the olfactory bulb of freely moving mice depend on the suprachiasmatic nucleus but not on behaviour rhythms. European Journal of Neuroscience. 42, 3128-3137 (2015).
- Ono, D., et al. Dissociation of Per1 and Bmal1 circadian rhythms in the suprachiasmatic nucleus in parallel with behavioral outputs. Proceedings of the National Academy of Sciences U S A. 114, E3699-E3708 (2017).
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Liu, A. C., et al. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genetics. 4, e1000023(2008).
- Maywood, E. S., et al. Analysis of core circadian feedback loop in suprachiasmatic nucleus of mCry1-luc transgenic reporter mouse. Proceedings of the National Academy of Sciences U S A. 110, 9547-9552 (2013).
- Ukai-Tadenuma, M., et al. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- McClure, C., Cole, K. L., Wulff, P., Klugmann, M., Murray, A. J. Production and titering of recombinant adeno-associated viral vectors. Journal of Visualized Experiments. 57, e3348(2011).
- Yamaguchi, Y., et al. Mice genetically deficient in vasopressin V1a and V1b receptors are resistant to jet lag. Science. 342, 85-90 (2013).
- Nagano, M., et al. An abrupt shift in the day/night cycle causes desynchrony in the mammalian circadian center. Journal of Neuroscience. 23, 6141-6151 (2003).
- Golombek, D. A., Rosenstein, R. E. Physiology of Circadian Entrainment. Physiological Reviews. 90, 1063-1102 (2010).
- Mei, L., et al. Long-term in vivo recording of circadian rhythms in brains of freely moving mice. Proceedings of the National Academy of Sciences. 115, 4276-4281 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon