Method Article
Isolamento delle cellule della ganglioma della retina primaria (RGC) mediante citometria a flusso
In questo articolo
Riepilogo
Milioni di persone soffrono di malattie degenerative della retina che provocano cecità irreversibile. Un elemento comune di molte di queste malattie è la perdita delle cellule del ganglio retinico (RGC). Questo protocollo dettagliato descrive l'isolamento di RGC murine primari mediante selezione positiva e negativa con citometria a flusso.
Abstract
Le malattie neurodegenerative spesso hanno un impatto devastante su coloro che sono colpiti. La perdita di cellule della ganglioma retinica (RGC) è implicata in una serie di malattie, tra cui la retinopatia diabetica e il glaucoma, oltre al normale invecchiamento. Nonostante la loro importanza, i RGCs sono stati estremamente difficili da studiare finora, in parte a causa del fatto che essi comprendono solo una piccola percentuale della grande varietà di cellule della retina. Inoltre, i metodi di isolamento attuali utilizzano marcatori intracellulari per identificare RGC, che producono cellule non vitali. Queste tecniche coinvolgono anche lunghi protocolli di isolamento, per cui esiste una mancanza di metodi pratici, standardizzati e affidabili per ottenere e isolare i RGC. Questo lavoro descrive un metodo efficiente, completo e affidabile per isolare i RGC primari dalla retina dei topi usando un protocollo basato sui criteri di selezione sia positivi che negativi. I metodi presentati consentono il futuro studio dei RGC, con l'obiettivo di una migliore comprensione dei principaliDiminuzione dell'acuità visiva che risulta dalla perdita di RGC funzionali nelle malattie neurodegenerative.
Introduzione
I RGC sono terminali differenziati dai neuroni e pertanto sono necessarie le cellule primarie per la sperimentazione. Lo sviluppo di un protocollo per l'isolamento e l'arricchimento delle cellule primarie di ghiandole retiniche murine (RGCs) è fondamentale per rivelare i meccanismi di salute e degenerazione di RGC in vitro . Ciò è particolarmente importante per studi che cercano di generare potenziali terapie per promuovere la funzione RGC e per ridurre al minimo la loro morte. La degenerazione dei RGC è associata a malattie degenerative della retina, come il glaucoma, la retinopatia diabetica e l'invecchiamento normale. Anche se i meccanismi cellulari specifici alla base della perdita di RGC non sono chiari, sono stati individuati una serie di fattori di rischio. La mancanza di ossigenazione alla testa del nervo ottico 1 , 2 , 3 provoca la morte di RGC 4 e funge da disturbo dell'omeostasi tra l'attivazione di eccitatori e iRecettori nhibitory all'interno di singoli RGCs 5 , 6 . Una serie di sfide impediscono il progresso verso l'uso di queste cellule per studi approfonditi. In primo luogo, il numero di RGC presenti in una retina murina è piccolo. RGC rappresentano meno dell'1% delle cellule retiniche totali 7 , 8 , 9 . Secondo, la maggior parte dei marker RGC-specifici sono proteine intracellulari 10 , 11 , 12 . La selezione basata su questi marcatori lascia le cellule inattive, che esclude le analisi funzionali a valle. Infine, i protocolli attualmente disponibili sono lunghi e mancano la standardizzazione 13 , 14 . I primi protocolli di isolamento RGC sono stati basati su metodi di immunopanning. Barres et al. 15 Adattato il classico immunoplayAnnotando la tecnica e ha aggiunto un secondo passo, che escludeva monociti e cellule endoteliali dalla maggior parte delle cellule retiniche prima della selezione positiva basata sull'immunopositività all'antigene anti-timocitico (aka Thy1), un marker di superficie cellulare. Anni dopo, Hong et al. Tecniche di isolamento del tallone magnetico combinato con strategie di selezione delle cellule per isolare RGC con purezza superiore 16 . L'uso di perline magnetiche è ancora utilizzato in molte applicazioni scientifiche. Insieme, i branelli magnetici ei protocolli di citometria di flusso hanno migliorato la purezza delle cellule isolate. Tuttavia, questi sistemi di purificazione non sono ancora stati standardizzati per l'isolamento dei RGC murini da retina dissociata.
La citometria di flusso è un potente metodo analitico che misura le caratteristiche ottiche e fluorescenti delle sospensioni cellulari. Le cellule sono analizzate in modo quantitativo e qualitativo con un elevato livello di sensibilità, fornendo un'analisi multidimensionale del populino cellularezione. La discriminazione cellulare si basa su due principali proprietà fisiche: dimensione delle cellule o superficie e granularità o complessità interna 17 . È possibile eseguire un'analisi multidimensionale combinando anticorpi tagliati con fluorochromi che presentano lunghezze d'onda simili e diverse emissioni. La citometria del flusso è veloce, riproducibile e sensibile. I laser Multitpe permettono di analisi multidimensionali ancora più grandi di singole cellule mediante citometria a flusso. Quindi è una metodologia attraente per lo studio di campioni citologici. La classificazione delle cellule attivata con fluorescenza (FACS) utilizza le differenze fenotipiche multidimensionali identificate dalla citometria di flusso per ordinare singole cellule in sottopopolazioni distinte.
Nell'ultimo decennio, proteine multiple e proteine intracellulari sono state identificate come potenziali biomarcatori per la selezione delle cellule, compresi i neuroni. Gli studi iniziali che cercavano di isolare i RGC dai topi utilizzavano Thy1 come un ganglio celL marker. Purtroppo, Thy1, aka CD90, ha diverse isoforme in altre specie di roditore 18 , 19 e 20 ed è espresso da più tipi di cellule retiniche 19 , 20 , rendendolo un marker non specifico per i RGC. Un altro marcatore di superficie, CD48, si trova sulle popolazioni monocitiche nella retina, tra cui macrofagi e microglia. Usando questi due marcatori di superficie, una delle cellule negate di RGC firmate Thy1 + e CD48 è stata sviluppata 15 , 16 , 21 , 22 . Purtroppo, questi due criteri di selezione non sono sufficienti per selezionare una popolazione RGC altamente arricchita. Per affrontare questa necessità non soddisfatta, è stato sviluppato un protocollo di citometria di flusso 23 basato su criteri di selezione positivi e negativi multi-layer usiI marker di superficie delle cellule noti per arricchire e purificare RGC murine primarie.
Protocollo
Tutte le procedure descritte nel seguente protocollo sono state approvate dal comitato di revisione istituzionale per la cura e l'uso degli animali (IACUC) presso l'Università di Tennessee Health Science Center (UTHSC) e ha seguito l'Associazione per la ricerca in visione e oftalmologia (ARVO) Degli animali in ricerca oftalmica e visiva, in aggiunta alle linee guida per gli esperimenti sugli animali da laboratorio (Istituto di risorse animali da laboratorio, Politica sanitaria pubblica sulla cura umana e uso degli animali da laboratorio).
1. Preparazione di strumenti, soluzioni e supporti
Nota: Tutte le informazioni sui materiali, reagenti, strumenti e strumenti riportati nel protocollo sono specificati nella tabella dei materiali .
- Autoclave tutti gli strumenti di dissezione e conservarli in una zona sterile. Utilizzare i seguenti strumenti: 4 pinze standard (2 lunghe e 2 corte) e 2 forbici,Così come 2 pinze (1 lungo e 1 corto) e 1 forbice per la dissezione; Tenere un set extra come backup.
- Preparare 100 ml di soluzione sterile PBS / 1% FBS da utilizzare durante le lavaggi, le procedure di immunoabilitazione e le fasi di ordinamento delle cellule. Tenere la soluzione refrigerata a 4 ° C.
Nota: non aggiungere sodio azide (NaN 3 ) alla soluzione, in quanto può essere tossico per le cellule vive.- Preparare 100 mL di PBS / 1% FBS con 99 mL di PBS e 1 mL di FBS.
- Preparare 100 ml di mezzo di cellule neurali sterili integrate con il 3% di FBS (vedi tabella dei materiali ) per l'uso come mezzo di raccolta e coltura. Tenere il terreno di coltura cellulare sterile a 4 ° C. Solo caldo a temperatura ambiente (RT) prima dell'uso.
- Preparare 100 ml di mezzo di cellule neurali integrate con il 3% di FBS usando 97 ml di mezzo di cellule neurali e 3 ml di FBS.
- I tubi di raccolta pre-raffreddamento (tubi da 15 ml) pre-rivestiti con 5 ml di mezzo di raccolta posizionando tBordo in un secchio di ghiaccio. Utilizzare solo tubi in polipropilene per evitare che le celle si aderiscano alla superficie del tubo.
- Posizionare piatti da 40 mm, filtri in nylon da 70 μm, siringhe, pestelli per il filtro delle cellule e provette sterili in polipropilene nell'armadio della biosicurezza. Sterilizzare tutti gli oggetti prima delle procedure e mantenerli in modo sterile in tutte le procedure.
Nota: Tutte le fasi successive alla raccolta della retina saranno eseguite nell'armadio della biosicurezza.
2. Enucleazione
Nota: In questo esperimento sono stati utilizzati complessivamente 10 giovani (5-7 settimane) C57BL / 6J topi per isolare RGC da 1,0 x 10 6 con il fenotipo CD90.2 + CD48 neg negativo CD15 neg negativo CD57 neg .
- Eutanizzare i topi usando l'inalazione del CO 2 seguita da dislocazione cervicale. Utilizzare sempre un metodo di eutanasia secondario per assicurare che l'animale eutanizzato sia morto.
Nota: l'isofluorano può essere utilizzato come alternativa alla CO 2 . La Ketamina non è raccomandata, in quanto è associata ad un aumento della pressione intraoculare (IOP) quando usato come induttore di anestesia 24 , 25 . - Inserire le pinze sotto il globo dell'occhio, afferrare il nervo ottico e tirare su; Il globo sarà enucleato, con il nervo ottico intatto. Mettere gli occhi raccolti in un flaconcino contenente PBS e mantenere la fiala sul ghiaccio fino al passo successivo.
Nota: Qualsiasi persona può eseguire questo passaggio dopo aver ricevuto la formazione adeguata dalla LACU.
3. Preparazione della sospensione delle cellule retiniche
- Posizionare l'occhio raccolto sulla piastra di base di un microscopio di dissezione per iniziare la dissezione corneale. Dissecchi ogni occhio singolarmente.
- Tenete premuto il globo sulla base del nervo ottico usando le pinze. Per questo passo, utilizzare un lonG e una pinza standard corta.
- Utilizzare un ago tagliente a 30 gauge (30G) per forare la cornea per consentire l'evacuazione dell'umorismo acquoso dall'occhio, rendendo più facile tenere l'occhio con le pinze.
- Tenere la cornea con le pinze e utilizzare forbici per effettuare una piccola incisione nella cornea. Sbucciate delicatamente la cornea e la sclera usando le pinze. Quando il globo viene sbucciato a metà strada, ruotare la retina e l'obiettivo usando la pinza. Scartare la cornea, la sclera e l'obiettivo.
Nota: Questo metodo assicura che la retina si stacca completamente dal resto dell'occhio. - Mettere la retina in un piccolo piatto da 40 mm di Petri contenente PBS / 1% FBS.
Nota: In alternativa al piatto di Petri, è possibile utilizzare un piatto di coltura cellulare. Assicurarsi di mantenere sempre la retina umida, come in condizioni fisiologiche.- Lavare ogni retina tre volte con PBS fresco sterile / 1% FBS nell'armadietto della biosicurezza.
- Posiziona fino a 12 retinaUn filtro in nylon sterile da 70 μm inumidito con PBS / 1% FBS. Usando la parte posteriore di una siringa da 10 ml, macerare delicatamente la retina utilizzando un movimento circolare per staccare le cellule.
- In alternativa, utilizzare un pestello per la macerazione del filtro delle cellule o utilizzare la digestione enzimatica con una combinazione di 15 UI / ml di papain, 5 mM di L-cisteina e di 200 U / ml di DNasi I per 15 minuti a 37 ° C, seguita da inattivazione con PBS / 10% FBS.
Nota: Nessuna alterazione della percentuale di celle recuperate è stata osservata quando la digestione enzimatica è stata confrontata alla macerazione con la parte posteriore della siringa o il pestello.
- In alternativa, utilizzare un pestello per la macerazione del filtro delle cellule o utilizzare la digestione enzimatica con una combinazione di 15 UI / ml di papain, 5 mM di L-cisteina e di 200 U / ml di DNasi I per 15 minuti a 37 ° C, seguita da inattivazione con PBS / 10% FBS.
- Per trasferire le celle isolate, posizionare il filtro in nylon sterile da 70 μm sopra il tubo di raccolta di polipropilene. Passare le cellule raccolte attraverso il filtro usando una pipetta P1000. Sciacquare il filtro ( punto 3.6 ) con PBS / 1% FBS per rilasciare eventuali celle rimanenti e trasferirle nel tubo di raccolta.
- Aggiungere PBS / 1% FBS a achUn volume finale di 1 ml per la retina. Centrifugare la sospensione cellulare per 7 minuti a 200 xg e RT.
Nota: A seconda del numero di retina macerata (<6 retina), il pellet cellulare può essere troppo piccolo per essere visibile.- Scartare il surnatante cellulare e risospendere il pellet cellulare in PBS / 1% FBS utilizzando un rapporto di 1 mL per 5 retina.
- Conta le cellule usando un emocitometro.
- Pulire un emocitometro di vetro e coprire con alcol al 70%. Infilare delicatamente il tubo contenente le celle per assicurare che la sospensione delle cellule retiniche sia distribuita uniformemente. Posizionare 20 μl di 0,4% di blue trypan in un tubo di microcentrifuga e mescolare con 20 μL della sospensione cellulare.
- Mescolare delicatamente e applicare 10 μL del mix di sospensione di blu / cella di 0.4% di trypan / blu all'emocitometro, riempiendo entrambe le camere; L'azione capillare disegna il mix 0.4% di sospensione blu / cellulare sotto il coperchio. Utilizzando un microscopio, contate tutte le cellule blue-negative trypan; thiIl numero di numeri rappresenta le celle vive.
- Determinare la vitalità cellulare utilizzando la formula seguente: conteggio delle cellule vive / numero totale di cellule = vitalità%.
Nota: Se la vitalità delle cellule è inferiore al 95%, durante il FACS è necessaria l'uso di colorante fluorescente per la discriminazione di vitalità cellulare.
- Utilizzare un colorante fluorescente di vitalità che non è permeabile alle cellule vive. Lavare le cellule con PBS per rimuovere qualsiasi traccia di siero. Aggiungere 1 μl di colorante fluorescente per la discriminazione della vitalità cellulare per 5,0 x 106 cellule in un volume finale di 100 μl. Incubare a RT per 15 minuti, protetto dalla luce. Aggiungere 10 volte il volume di PBS / 1% FBS. Centrifugare per 5 min a 200 xg e RT.
- Incubare la sospensione cellulare durante la notte a 4 ° C, se necessario. Se questo è fatto, riempire il tubo di sospensione cellulare con un mezzo di cellule neurali piuttosto che PBS / 1% FBS. Mettere il tubo orizzontalmente e mantenerlo a 4 ° C.
4. Immunolabelazione del ReCellule tinali
- Utilizzare la seguente conversione per determinare il volume di anticorpo per numero di cellule: 2 μL di anticorpi per 5,0 x 106 cellule in un volume di 100 μL.
- Lavare le cellule e mantenerle in PBS / 1% FBS. Prendi una piccola aliquota di cellule (5.0 x 10 6 cellule) da utilizzare come controllo negativo (non etichettato) al momento della configurazione del tipo.
Nota: questo controllo negativo è fondamentale per la corretta calibrazione del separatore di celle. - Per ridurre al minimo il legame non specifico degli anticorpi alle cellule che esprimono i recettori Fcγ II e III, aggiungere 1 μL di un anticorpo anti-mouse CD16 / 32 per 1,0 x 10 6 cellule in un volume finale di 50 μl usando PBS / 1% FBS. Incubare per 10 minuti a RT.
- Aggiungere il cocktail anticorpale al campione e mescolare delicatamente pipettando. Incubare per 30 minuti in un secchio di ghiaccio. Assicurarsi che il secchio di ghiaccio sia coperto, poiché la luce potrebbe compromettere l'esperimento a causa della foto-blecatura del fluoro taggatophores.
- Preparare il cocktail anticorpale usando i seguenti anticorpi anti-mouse anti-mouse contrassegnati fluorescenti: CD90.2 AF-700, CD48 PE-Cyanine7, CD15 PE e CD57 non tagliato.
Nota: Per ogni anticorpo, utilizzare le seguenti concentrazioni per cellule 5.0 x 10 6 : CD90.2 AF700, 1 μg; CD48 PE-Cyanine7, 0,4 μg; CD15 PE, 0,02 μg; E CD57 non tagliato, 0,4 μg.- Utilizzare i volumi secondo i seguenti calcoli (in base agli anticorpi disponibili in commercio nella tabella dei materiali ): totale di 5.0 x 10 7 cellule da etichettare; 2 μL per 5 x 10 6 cellule = 20 μL di ciascun anticorpo; 4 diversi anticorpi = 80 μL di cocktail anticorpale; Volume finale = [(5,0 x 10 7 cellule totali) /5,0 x 10 6 cellule] x 100 = 1.000 μL; 80 μL Abs + 50 μL di anti-mouse CD16 / 32 + 870 μL PBS / 1% FBS = 1.000 μL.
Nota: Questa combinazione ha fornito ilCombinazione ottimale di fluorochromi per la configurazione dello strumento. I seguenti parametri sintetizzano l'emissione e l'eccitazione: AF-700, emissione di 719 nm quando viene eccitato con un laser diodo a 638 nm; PE-Cyanine7, emissione di 767 nm quando viene eccitato con un laser diodo a 488 nm; E PE, emissione di 575 nm quando eccitato con un laser diodo a 488 nm.
- Utilizzare i volumi secondo i seguenti calcoli (in base agli anticorpi disponibili in commercio nella tabella dei materiali ): totale di 5.0 x 10 7 cellule da etichettare; 2 μL per 5 x 10 6 cellule = 20 μL di ciascun anticorpo; 4 diversi anticorpi = 80 μL di cocktail anticorpale; Volume finale = [(5,0 x 10 7 cellule totali) /5,0 x 10 6 cellule] x 100 = 1.000 μL; 80 μL Abs + 50 μL di anti-mouse CD16 / 32 + 870 μL PBS / 1% FBS = 1.000 μL.
- Preparare il cocktail anticorpale usando i seguenti anticorpi anti-mouse anti-mouse contrassegnati fluorescenti: CD90.2 AF-700, CD48 PE-Cyanine7, CD15 PE e CD57 non tagliato.
- Dopo l'incubazione di 30 minuti, portare il volume fino a 5 ml utilizzando PBS / 1% FBS. Lavare i campioni mediante centrifugazione per 7 minuti a 200 xg e RT. Ripetere la procedura una volta per rimuovere tutti gli anticorpi non legati.
- Aggiungere 2 μL di anticorpo secondario (0,1 μg), che legherà il CD57 di 5,0 x 106 cellule. Incubare per 30 minuti in un secchio di ghiaccio, come nel punto 4.4 . Lavare le cellule due volte, come nel punto 4.5 .
- Etichettare le cellule con anticorpo secondario se un anticorpo primario non tagliato è stato utilizzato nel passaggio 4.4 .
Nota: il secondoAntitumore a scelta di questa configurazione è elencato nella tabella dei materiali ; Ha una lunghezza d'onda di emissione di 421 nm. Usalo con un filtro a banda passante di 450/50 nm quando viene eccitato con il laser a diodo viola a 405 nm. - Utilizzare i volumi secondo i seguenti calcoli (in base all'anticorpo commercialmente disponibile nella tabella dei materiali ): 2 μL per cellule di 5,0 x 10 6 = 20 μL di anticorpo secondario; Volume finale = [(5,0 x 10 7 cellule totali) /5,0 x 10 6 cellule] x 50 = 500 μL; 20 μL di Abs + 480 μl PBS / 1% FBS = 500 μL.
- Etichettare le cellule con anticorpo secondario se un anticorpo primario non tagliato è stato utilizzato nel passaggio 4.4 .
- Tenere le celle etichettate in PBS / 1% FBS. Conte le cellule usando un emocitometro; Utilizzare una concentrazione cellulare finale di 3,0-8,0 x 10 7 cellule / ml.
Nota: non utilizzare mezzi di coltura cellulare per diluire le cellule perché il fenolo rosso può aumentare l'autofluorescenza, riducendo così la risoluzione tra negazioneIve e le cellule positive. La concentrazione cellulare finale per volume dipende in larga misura dalla portata di volume della configurazione dello strumento. - Utilizzare i controlli monocromatici durante l'installazione per ridurre al minimo lo spargimento di fluorescenza.
Nota: Questo passaggio, noto anche come compensazione, viene applicato per correggere il rumore creato dalla combinazione di fluorofori. Le microsfere in polistirene in PBS / 0,1% BSA / 2 mM NaN3 con la capacità di legare isotipi di immunoglobuline (Ig) fluorescenti con diverse specie forniscono controlli positivi per impostare la compensazione.- Posizionare 3 gocce di microsfere in polistirene in tubi FACS sterili, assegnando uno per fluoroforo. Aggiungere 1 μg del rispettivo fluoroforo ad ogni tubo. Incubare per 15 minuti a RT, protetto dalla luce. Aggiungere 3 mL di PBS / 1% FBS ai tubi del campione. Centrifugare per 5 min a 200 xg e RT. Rimuovere con cautela il surnatante e risospendere in 250 μl di PBS / 1% FBS.
- Per impostare il contr negativoOls, utilizzare microsfere di polistirolo che non legano a Ig. Effettuare questo passaggio il giorno prima, se necessario.
5. Strategia di ordinamento delle cellule
Nota: Istruzioni specifiche per l'impostazione dello strumento per FACS a 355 nm, UV; 405 nm, viola; 488 nm, blu; E 640 nm, laser rosso con distribuzione di canali di fluorescenza 2, 2, 5 e 3 rispettivamente. Il software operativo era DIVA versione 8.0.1. La selezione delle cellule è stata effettuata con un ugello di 70 μm, una pressione del tubo da 70 psi, una frequenza di 87,5, una ampiezza di 48,6 con la prima caduta a 333, il posizionamento di gap di 6, la precisione di ordinamento impostata su una purezza a quattro vie con la maschera di purezza (32) Ritardo regolato a 42,98 usando le perline.
- Utilizzare tubi conici da 15 ml di polipropilene come recipienti di raccolta. Aggiungere 5 ml del mezzo di raccolta (mezzo di coltura cellulare) ad ogni tubo e ruotare il tubo per rivestire le pareti.
Nota: questo passaggio impedisce alle cellule di attaccarsi ai lati di tTubo di raccolta. In alternativa, il tubo può essere rivestito con FBS non diluito. - Prendere in considerazione l'efficienza del collettore cellulare per stimare la resa finale delle cellule.
Nota: L'efficienza del tipo varia in base al citometro di flusso utilizzato. RGC con CD90.2 + CD48 neg negativo CD15 neg negativo fenotipo comprendono circa l'1% di tutte le cellule retiniche. - La classificazione delle celle deve essere eseguita da un operatore addestrato (di solito in una struttura principale). Assicurarsi che lo strumento sia pulito e sterilizzato con il 70% di etanolo prima dell'acquisizione del campione. Pulire attentamente l'esterno del tubo di raccolta con il 70% di etanolo.
- Se le cellule si sono stabilite o aggregate, pipettare le cellule su e giù e filtrare la sospensione attraverso un filtro in nylon 40 μm sterile prima dell'acquisizione. Assicurarsi di controllare la temperatura e mantenerla a 4 ° C mentre è in corso l'ordinamento delle celle; La mancata riduzione del celLa resa.
- Discutere i dettagli dell'ordine con l'operatore di selezione delle cellule prima di eseguire gli esperimenti.
Nota: Al momento dell'esperimento, l'operatore del riepilogo di celle avrà cliccato una serie di pulsanti dalla barra degli strumenti Workspace del software di acquisizione per il citometro. Questi sono il Browser , Cytometer , Ispettore , Foglio di lavoro e Dashboard di Acquisizione . La strategia di selezione delle cellule qui utilizzata è conosciuta come un sistema a due vie. Si raccolgono due popolazioni: i RGC arricchiti e tutti gli altri tipi di cellule. Se si devono raccogliere altre popolazioni, si possono isolare fino a due popolazioni aggiuntive come un tipo a 4 vie. Se si raccolgono altre popolazioni, il riutilizzatore di celle richiede diversi tubi di raccolta e configurazione. - Eseguire la compensazione per correggere la sovrapposizione spettrale dei fluorofori.
Nota: questo processo può essere fatto manualmente per regolareOgni impostazione, oppure può essere eseguita automaticamente come parte del software in uso. La maggior parte dei software per le apparecchiature di selezione delle celle hanno la possibilità di una compensazione automatizzata. È considerato il gold standard e il tipo di compensazione più preciso. Il campione negativo (nessun fluoroforo) e i singoli controlli preparati con microsfere di polisterene (perle di compensazione) specificamente fabbricate per associare tutti gli anticorpi monoclonali rilevanti utilizzati nell'esperimento saranno utilizzati in questa fase.- Seleziona Esperimento> Configurazione compensazione> Crea controlli di compensazione . Aggiungere i controlli specifici per fluoroforo dall'elenco visualizzato sullo schermo. Fare clic su OK .
Nota: Verrà visualizzato un tubo di compensazione per ciascuno dei comandi.- Utilizzare un controllo non contrassegnato (nessun fluoroforo, punto 4.2 ) per verificare la luce sparsa in avanti (FSC), la luce sparsa laterale (SSC) e per portare la popolazione iniziale (P1).
- Installare il tubo di controllo negativo sul citometro e fare clic su Carica. Verificare che la popolazione di interesse sia visualizzata e selezionarla. Questa è la popolazione 1 (P1). Fare clic con il pulsante destro del mouse sulla porta P1 e selezionare Applica a tutti i controlli di compensazione . Fare clic su Record Data . Una volta che la registrazione è stata fatta, fare clic su Scarica e rimuovere il tubo.
- Installare il tubo successivo sul citometro, come mostrato sullo schermo. Ripetere la fase 5.6.2 finché non sono stati registrati tutti i dati dai controlli.
- Selezionare Esperimento> Configurazione compensazione> Calcola compensazione . Salvare l'impostazione e denominare l'esperimento. Fare clic su Link & Salva .
Nota: la compensazione è un passo necessario poiché rimuove la sovrapposizione dello spettro tra i rivelatori. - Se è necessaria una compensazione manuale, farlo regolando i mezzi dei segnali positivi in modo che siano uguali ai negativi per l'eacH dei fluorochromi utilizzati.
Nota: questo passaggio è eseguito dall'operazione di selezione delle celle e può richiedere 30 minuti.
- Seleziona Esperimento> Configurazione compensazione> Crea controlli di compensazione . Aggiungere i controlli specifici per fluoroforo dall'elenco visualizzato sullo schermo. Fare clic su OK .
- Strategia di accesso ( Figura 3A ).
- Impostare P1 plottando FSC contro SSC; L'FSC è indicativo della dimensione e il SSC indica la complessità interna delle cellule. Vedere la Figura 3A .
- Disegnare un grafico pseudocolore di SSC-H (altezza) rispetto a SSC-W (larghezza) utilizzando la popolazione selezionata nel punto 5.7.1 (P1); La trama pseudocolore consente la rappresentazione visiva della densità delle cellule relative all'altro.
Nota: una densità delle cellule più basse è rappresentata dal blu e dal verde, mentre le aree rosse e arancioni rappresentano una elevata densità delle cellule.- Eseguire questo passaggio per raccogliere solo le singole celle; La porta singola cellulare è chiamata P2. Vedere la Figura 3A .
- Ripetere la fase 5.7.2 aTrama FSC-H rispetto FSC-W usando P2. Assicurarsi che la selezione di singole celle come "dublets" o cell clumps contiene sia un marker positivo che un negativo, fornendo una falsa positività.
- Disegnare un grafico pseudocolore di CD90.2 contro CD48. Selezionare tutte le cellule negate CD90.2 + CD48 per eliminare monociti dall'arricchimento RGC; Chiamare questa porta CD90.2 + CD48 neg , o P3. Vedere la Figura 3A .
- Utilizzando la pop pop negativa o la porta P3 selezionata CD90.2 + CD48, disegnare una trama pseudocolore di CD57 contro CD15 per eliminare contaminanti delle cellule amacrine dall'arricchimento di RGC. Porta la popolazione CD15 neg negativa CD57. Vedere la Figura 3A .
- Definire le popolazioni da raccogliere per il campione nella finestra "Ordina layout" e procedere con l'ordinamento delle celle; Il campione da raccogliere è CD90.2 + CD48 neg negativo CD15 neg negativo CD57 p>.
Nota: la popolazione da ordinare è selezionata dal menu a discesa Aggiungi nella finestra di ordinamento. Dopo aver aggiunto la popolazione, chiederà gli eventi di destinazione o quanti eventi devono essere raccolti. In qualsiasi momento, il layout di ordinamento può essere modificato facendo clic sul campo Ordina località contenente la popolazione. 0,9% ± 0,3 di RGC con il fenotipo CD90.2 + CD48 neg negativo CD15 neg negativo CD57 neg sono ottenuti da giovani (5-7 settimane) e 0,5% ± 0,3 e vecchi (> 12 mesi) C57BL / 6J topi 23 . - Utilizzando 25.000 celle, eseguire un controllo di purezza 23 . Vedere la Figura 3B -E.
Nota: il controllo della purezza è il processo di riesame delle celle ordinate per verificare l'esattezza del tipo di cella. Una piccola aliquota di cellule ordinate verrà caricata sul citometro per verificare l'efficacia del tipo.
- Etichettatura intracellulare.
- Fissare le cellule ordinate per 1 h e permeabilizzare a 4 ° C per rendere le cellule metaboliche inattive e per consentire la penetrazione di anticorpi intracellulari.
- Diluire i seguenti anticorpi nella soluzione di permeabilizzazione: proteina legante anti-RNA con splicing multiplo (RBPMS) a una diluizione 1: 100 in una quantità di 100 μg / mL; Gamma anti-sinucleina (SNCG), 1: 100 in un dosaggio di 1 mg / ml; Homeobox / POU dominio proteico 3A (BRN3A), 1: 100 in un titolo di 200 μg / mL; E tubulina beta-beta di classe III (TUJ1) specifica anti-neurone, 1: 100 in un titolo di 1 mg / ml. Incubare le cellule e le soluzioni anticorpali per 1 h in un secchio di ghiaccio coperto.
- Lavare due volte i campioni con PBS / 1% FBS.
- Risospendere le cellule nel corrispondente anticorpo secondario contrassegnato con AF488 a una diluizione 1: 200 per 30 minuti in un secchio di ghiaccio.
- Lavare i campioni come nel punto 6.1.3. Mantenere le celle dentroPBS / 1% FBS (250 μL) fino a preparare l'analisi.
7. Convalida della cella Sort by qPCR Analysis
Nota: vedere la Figura 4 .
- RNA estrazione 23 .
- Estrarre l'RNA dalle cellule ordinate di 5,0 x 10 5 mediante la lisi cellulare e l'omogeneizzazione seguita dall'aggiunta di cloroformio.
- Trasferire la fase superiore e incolore a un microtube pulito per la precipitazione di alcool seguita da concentrazione di estratto in una colonna di rotazione. Eseguire la digestione del DNAse sulla colonna.
- Lavare la colonna con acqua RNase-free per l'eluizione di RNA.
- Valutare la concentrazione di RNA per spettrofotometria.
- Sintesi di cDNA e preamplificazione.
- Utilizzare 100 ng di materiale RNA e mescolare con una soluzione contenente enzima inversa della trascrittasi, inibitore della ribonucleasi ricombinante (per evitare il degrado del RNA), cloruro di magnesio (MgCL 2 ) e deossinucleotidi.
- Mescolare il contenuto del tubo e incubare per 10 minuti a 25 ° C seguito da 60 min a 42 ° C. Terminare la reazione con un'incubazione a 5 minuti a 85 ° C. Conservare il cDNA risultante a -20 ° C fino all'uso pronto all'uso.
Nota: il materiale cDNA può essere conservato a -20 ° C per un massimo di un mese se la fase di preamplificazione non può essere eseguita immediatamente.
- Preamplificazione del cDNA.
- Pre-amplificare il materiale cDNA usando una serie di primer specifici per diversi tipi di cellule retiniche, tra cui: cellule del ganglio retinico, cellule amacrine, astrociti, Müller, bipolare, orizzontale, fotorecettori e cellule epiteliali del pigmento retinico, come specificato nel Riferimento 23 e nella Tabella Di materiali .
Nota: La reazione di pre-amplificazione è preparata per aumentare la sensibilità del rilevamento per la quantificazione. La reazione di preamplificazione contiene una miscela del primerMescola dalla tabella dei materiali ; 2,5 μL di cDNA, iniziato a 100 ng di RNA; Enzima trascriptasi inversa; E l'acqua senza nucleasi in un volume finale di 10 μL. - Effettuare la fase di attivazione enzimatica a 95 ° C per 10 minuti, seguita da 14 cicli a 95 ° C per 15 s, seguita da 60 ° per 4 min. Diluire il materiale preamplificato 1:10 in tampone Tris EDTA e mantenerlo a -20 ° C fino a quando è pronto per l'uso.
- Pre-amplificare il materiale cDNA usando una serie di primer specifici per diversi tipi di cellule retiniche, tra cui: cellule del ganglio retinico, cellule amacrine, astrociti, Müller, bipolare, orizzontale, fotorecettori e cellule epiteliali del pigmento retinico, come specificato nel Riferimento 23 e nella Tabella Di materiali .
- 7.4 Reazione qPCR.
- Preparare tutte le reazioni qPCR in un volume finale di 10 μL usando i cDNA preamplificati (2,5 μL), i primer (elencati nella tabella dei materiali ), l'acqua senza nucleasi e un concentrato con DNA polimerasi e deossinucleotidi.
- Utilizzare le seguenti condizioni dello strumento per eseguire il qPCR: una fase di mantenimento di 50 ° C per 2 minuti, seguita da 95 ° C per 10 minuti. Eseguire un totale di 40 cicli a 95 ° C per 15 s seguiti da 60 ° C per 1 minuto. Eseguire tutte le misureIn tre repliche.
- Eseguire la quantificazione relativa usando la soglia comparativa (C T ) dopo aver determinato i valori di C T per il gene di pulizia e i geni bersaglio in ciascun campione. Calcolare il cambiamento relativo della piega (R q ) utilizzando la seguente equazione: R q = 2 T -ΔC , dove ΔC T = C T target gene - C T gene di riferimento 23 .
Nota: il C T è definito come il ciclo PCR in cui il segnale fluorescente del colorante reporter attraversa una soglia arbitrariamente posizionata 26 . Il C T è inversamente correlato alla quantità di amplicone (prodotto PCR) nella reazione.
Risultati
Lo studio approfondito di RGC è ostacolato da molti fattori, tra cui la loro bassa frequenza e la mancanza di una metodologia robusta e standardizzata per il loro isolamento. La figura 1 mostra la metodologia utilizzata per l'isolamento della retina. Variazioni nella procedura di enucleazione esistono in base al tipo di analisi, come se l'enucleazione fa parte della sperimentazione in vivo 27 . L'enucleazione in questo protocollo è eseguita su topi eutanizzati. Come mostrato nella Figura 1A- B , le pinze sono poste sotto l'occhio e tirate su per causare un minimo sanguinamento e rimuovere un globo occhio con un nervo ottico intatto.
Differenze esistono nel numero di RGC in vari ceppi di topo, in particolare nei topi alterati geneticamente 23 , 28 ,La retina dei vecchi topi C57BL / 6J ha meno di cellule retiniche in vivo rispetto alle loro controparti più piccole 23. Pertanto, la dissezione della retina deve essere attentamente eseguita per determinare il numero di topi da utilizzare. Massima la resa delle cellule Una procedura step-by-step per la dissezione della retina è presentata in Figura 1C- J . Le cellule retiniche sono fragili, quindi le retina disciolte vengono collocate in filtri in nylon e macerate con l'estremità posteriore di una siringa Mostrato nella figura 2A o con un pestello per i filtri delle cellule La macerazione delle cellule direttamente nel filtro delle cellule è veloce e riduce i grumi di cellule È rappresentata un'immagine rappresentativa della sospensione cellulare nella figura 2B.Clicole multiple di retina interna possono essere visualizzate. A questo punto, i RGC hanno perso il loro morfolo di firmaGy dovuta all'assotomia durante l'isolamento cellulare e alla preparazione della sospensione cellulare.
Il passo più intenso di questa metodologia è l'impostazione della selezione delle celle. Questa fase è un passo fondamentale durante il FACS multicolore, in quanto massimizza la risoluzione del segnale-rumore. La Figura 3A mostra la strategia di gating utilizzata per l'isolamento di RGCs. Questa strategia ha mirato alla rimozione delle cellule contaminanti dalla sospensione cellulare, che comprendeva le cellule monocitarie, gliali, amacrine e fotorecettori. Come parte della metodologia, marcatori di superficie aggiuntivi sono stati confermati mediante analisi immunohistochemica prima di essere utilizzati come parte della strategia di esclusione. I dati precedenti hanno dimostrato che una piccola percentuale di CD90.2 + sono CD48 + . L'esclusione di queste cellule ha rimosso monociti ed eventualmente microglia dalla piscina delle cellule retiniche. È stato precedentemente dimostrato che il classico Thy1 + CD48 neg fenotipo di superficie non è sufficiente per identificare e isolare RGC murine 23 , poiché queste cellule esprimono geni associati a fotorecettori amacrine, Müller, bipolari, orizzontali e retinici epiteliali ( Figura 4A ). Ciò è ulteriormente affrontato indagando marcatori aggiuntivi per l'esclusione cellulare. CD15 è stato descritto come un marcatore delle cellule amacrine e bipolari 31 , che ha indotto il suo utilizzo come marcatore aggiuntivo per la selezione negativa. Lavoro da Uusitalo et al. 32 descrisse CD57 come marcatore di identificazione per le cellule gliali e fotorecettori. Pertanto, questo anticorpo è stato aggiunto alla strategia di classificazione delle cellule.
Successivamente, queste cellule sono state caratterizzate per convalidare la metodologia per l'isolamento di RGC murine. I fenotipi delle CD90.2 + CD48 neg negativo CD15 negative negativo CD57 cellule ordinate ( Figura 3B) sono stati valutati per l'espressione dei seguenti marcatori intracellulari associati a RGCs 10 , 11 , 12 , 33 : SNCG, BRN3A, TUJ1 e RBPMS. Come mostrato nella Figura 3C , le cellule ordinate hanno espresso tutte le quattro proteine intracellulari associate a RGC. Successivamente, la citometria a flusso di imaging è stata utilizzata in Figura 3D per mostrare la localizzazione intracellulare di RBPMS e l'espressione della superficie cellulare di CD90.2. Questi risultati sono stati testati in sistemi multipli di citometro, confermando la riproducibilità e la standardizzazione. Come mostrato nella figura 3E , alcune delle cellule ordinate hanno iniziato a mostrare la morfologia associata a RGC dopo la coltura cellulare in vitro .
Infine, un confronto delle cellule prima dell'arricchimento e del post-ceL'analisi è stata effettuata mediante analisi qPCR. Il confronto del fenotipo Thy1 + CD48 con il CD90 negativo negativo CD15 negativo CD15 negativo ha determinato che il fenotipo negativo Thy1 + CD48 esprime geni associati a RGC, ma anche con altre cellule retiniche. Tuttavia, la popolazione di cellule ordinate altamente arricchita ( Figura 4B ) ha mostrato un aumento moltiplicato nei geni che codificano i marker intracellulari specifici RGC Sncg (SNCG), Pouf4L (BRN3A), Tubb3 (TUJ1) e Rbpms (RBPMS). Complessivamente, le valutazioni di mRNA e proteine convalidarono la metodologia.
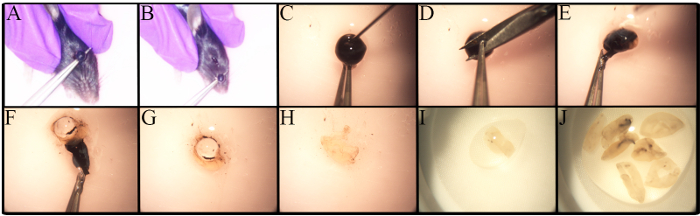
Figura 1 . Enucleazione e dissezione oculare per l'isolamento della retina. I topi giovani C57BL / 6J sono stati eutanizzati primaO rimozione del globo occhio con CO 2 e dislocazione cervicale. A) Posizionare la pinza sotto l'occhio e tirare l'occhio in un solo movimento. B) L'occhio viene rimosso, compreso il nervo ottico. CJ) Guida passo-passo per rimuovere la retina. C) Una puntura viene eseguita usando un ago 30G prima della rimozione corneale per consentire all'omorismo acquoso di uscire dall'occhio. D) La cornea è tenuta con pinze per eseguire una piccola incisione. EF) L'uso della pinza permette di sbucciare la cornea, l'epitelio del pigmento retinico, il coroide e la sclera. La retina è staccata dalla sclera, rotolata e rimossa. G) L'obiettivo viene rimosso e scartato. HJ) La retina raccolta viene posta in un piccolo piatto contenente PBS / 1% FBS per mantenerli umidi in ogni momento. Clicca qui per visualizzare una versione più grande di questofigura.
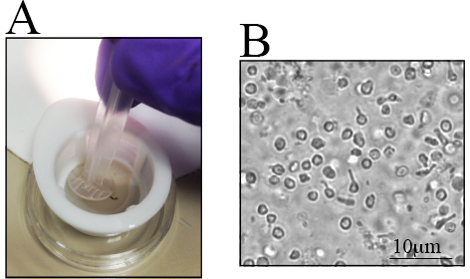
Figura 2 . Sospensione delle cellule retiniche dopo la macerazione della retina raccolta. La retina raccolta viene posta in un piccolo piatto per isolare le cellule. A) Le retine sono poste in un filtro in nylon da 70 μm e macerate utilizzando la parte posteriore di una siringa. B) Immagine rappresentativa della sospensione cellulare, in cui vengono osservate cellule retiniche distinte. La barra di scala è di 10 μm.

Figura 3 . Strategia di classificazione per l'isolamento delle celle con CD57.2 + CD48 neg negativo CD15 negativo CD57 neg Fenotipo e analisi post-sorting. La strategia di classificazione si basa sull'inclusione delle cellule CD90.2 e l'esclusione delle cellule CD48-, CD15- e CD57-positive, che sono cellule contaminanti. A) Come primo passo, dimensione della trama (FSC) e complessità interna (SSC) per ottenere una panoramica della popolazione cellulare. La popolazione di canali iniziali (P1) viene utilizzata per discriminare tra cellule singole e cellule o aggregati clumpati utilizzando l'altezza SSC (H) rispetto alla larghezza (W), P2. La selezione delle singole celle viene utilizzata per scegliere le celle negate CD90.2 + CD48. Per confermare la rimozione di tutti i dublets, viene eseguita una trama di FSC-H rispetto a FSC-W, P3 (pannello centrale ). Le cellule sono state etichettate con CD90.2 anti-mouse coniugato con AF700, CD48 anti-mouse coniugato con PE-Cyanine7, CD15 coniugato con PE e anti-mouse CD57. Come anticorpo secondario per contrassegnare il CD57 anti-mouse, è stato utilizzato anti-mouse BV421. La popolazione 3 (P3) è stata plottata nel quarto </ Em> per selezionare le celle negate CD90.2 + CD48, rimuovendo la maggior parte delle celle contaminanti. Successivamente, viene generata una trama CD57 rispetto a CD15 utilizzando le nozze selezionate CD90.2 + CD48. Viene selezionato il quadrante 4 (Q4), in quanto rappresenta le neg negative di CD90.2 + CD48 che sono negative per CD15 e CD57. Il fenotipo risultante della popolazione gated è CD90.2 + CD48 neg negativo CD15 neg negativo CD57. B) Analisi post-sorta dei marcatori superficiali utilizzati in A). Le celle ordinate sono di dimensioni omogenee, come mostrato nel primo pannello. Gli istogrammi successivi mostrano la percentuale di ciascun marker di superficie utilizzato nella strategia di ordinamento descritta in A). Un totale del 95% delle cellule sono CD90.2 + , come mostrato nella linea nera rispetto al controllo Ig, rappresentato dall'istogramma solido. Queste cellule sono state gated per valutare le percentuali di CD48, CD15 e CD57, rappresentate dal rosso, blu e greeN, rispettivamente. I risultati mostrano un'espressione minima di questi marker di superficie cellulare. C) Conferma del fenotipo RGC utilizzando gli indicatori intracellulari specifici RGC SNCG, BRN3A, TUJ1 e RBPMS. Le righe nere rappresentano la percentuale di cellule che esprimono ogni marcatore intracellulare. D) Immagini rappresentative prese in una sorta di cellule di imaging che mostra la localizzazione intracellulare di RBPMS, un marker intracellulare specifico RGC e il marker di superficie della cellula CD90.2. La barra di scala è di 20 μm. E) Immagine rappresentativa di RGC ordinati dopo 24 ore di coltura utilizzando un microscopio confocale. La barra di scala è di 20 μm. Le immagini BE sono adattate da lavori precedentemente pubblicati con permesso 23 . Le immagini DE sono state prese a 20X. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4 . Analisi mRNA pre- e post-sorting. Le cellule negate e ordinate di Thy1 + CD48 con il fenotipo CD90.2 + CD48 Neg negativo CD15 Neg negativo sono state valutate mediante analisi qPCR usando un pannello di 25 geni espresso da cellule retiniche. I livelli di espressione genica target sono presentati come un cambiamento di Log 2- volte utilizzando Hprt come un gene di pulizia e l'acqua come controllo negativo. Il calcolo è stato effettuato sulla base del metodo ΔC T. Mean ± SEM; N = 3 repliche biologiche sono state eseguite in triplice copia. Figure ottenute da lavori precedentemente pubblicati con permesso 23 .
Discussione
FACS è la tecnica di scelta per purificare le popolazioni cellulari. Altri metodi di isolamento includono immunopanning, perline magnetiche e depletionamento della fissazione del complemento. Il vantaggio di FACS rispetto a queste altre metodologie si basa sull'identificazione simultanea di marcatori a superficie cellulare con gradi di intensità variabili. L'intensità fluorescente della molecola è proporzionale alla quantità di espressione proteica. Fino ad ora l'isolamento dei RGC è basato esclusivamente sulla positività Thy1 (CD90) e sulla negatività CD48 15 , 16 , 22 , 34 , indipendentemente dal metodo di isolamento utilizzato. Recentemente è stato dimostrato che il fenotipo negativo Thy1 + CD48 non è sufficiente ad isolare una popolazione omogenea di cellule che esprimono marcatori intracellulari RGC 23 . L'identificazione della popolazione RGC è essenziale per il loro isolamento, in particolareY perché comprendono una piccola percentuale di cellule retiniche 7 , 8 , 9 . La maggior parte dei RGC si trovano nello strato più interno della retina, mentre un piccolo numero si trova negli strati plexiformi interni (RGC 35 spostati). Pertanto, la ricerca di RGC dai colliculi superiori da iniezioni stereotattiche e tracciamento con idrossistilbamidina (un tracciante retrogrado per delineare i neuroni) è diventato una scelta attraente per molti laboratori 36 , 37 e 38 . Questi sistemi richiedono l'iniezione di traccianti, che, se non correttamente eseguiti, possono portare ad alcune regioni retiniche lasciate inarrestabili. Inoltre, sono più tecnicamente esigenti e, come altre metodologie come l'immunopanning, sono lunghe. L'immunopanizzazione usando gli anticorpi anti-Thy1 e -CD48 richiede 48 ore per completare e non raggiungere più di 95Purezza%. Questo lavoro descrive una metodologia basata su FACS che offre un protocollo veloce e riproducibile per isolare una popolazione omogenea di RGC in vivo con il fenotipo negativo CD57 neg negativo CD15, CD90.2 + CD48, senza l'uso di traccianti, perline magnetiche o tecniche di immunopanizzazione .
I seguenti fattori sono necessari per l'isolamento efficace di RGC pura da FACS: 1) l'efficienza di ordinamento, che dipende in larga misura dall'apparecchiatura utilizzata; 2) combinazione ottimale di fluorochromi contrassegnati con anticorpi, per ridurre al minimo il rumore; E 3) l'impostazione della selezione delle celle. L'efficienza di ordinamento viene calcolata prendendo il numero di eventi target selezionati per l'ordinamento diviso per il numero di eventi target rilevati, espressi in percentuale. L'efficienza di ordinamento è un calcolo fornito dall'apparecchiatura. Questa efficienza dipende dalla configurazione del sistema di ordinamento e dalla modalità di selezione delle celle. La scelta di una combinazione ottimale dei fluorochromi è un processo complesso. Ogni influenzaL'orochrome ha proprietà distinte e si caratterizza per le sue lunghezze d'onda di eccitazione e emissione. Mentre l'eccitazione viene letto con un laser, l'emissione viene letta da tubi fotomoltiplicatori, limitati dai filtri ottici disponibili nell'apparecchiatura di ordinamento FACS. Qui viene fornita una combinazione di fluorochromi PE, PE-Cyanine7, AF700 e BV421 con anticorpo. Questo è stato determinato dopo aver considerato più combinazioni di fluorochrome che hanno fornito la migliore risoluzione riducendo contemporaneamente la sovrapposizione spettrale. Infine, l'impostazione del tipo è fondamentale. In generale, le cellule retiniche sono fragili. Pertanto, è meglio utilizzare una pressione più bassa per eseguire i campioni, per ridurre al minimo lo stress sulle cellule. È fondamentale mantenere il campione a 4 ° C, poiché mantenere i RGC murini per periodi più lunghi a temperatura ambiente può ridurre la resa cellulare, soprattutto quando ordinare un gran numero di cellule.
La classificazione basato su FACS è una metodologia ideale per l'isolamento delle cellule che compongono l'avPiccola percentuale della sospensione cellulare. Il processo, dalla dissezione della retina al completamento della selezione delle cellule, richiede circa 5 - 6 h, rispetto ai sistemi di immunopanning e traccianti, che richiedono giorni per completare. L'analisi multidimensionale di FACS e la capacità dell'apparecchiatura di raccogliere diverse popolazioni vitali consentono ulteriori analisi funzionali delle cellule. Il protocollo qui descritto è un potente strumento per l'isolamento dei RGC primari murini. Nonostante i suoi molteplici vantaggi, tra cui la sua sensibilità, la riproducibilità e l'immediata identificazione delle cellule vitali, esistono alcune limitazioni. In primo luogo, richiede costose strumentazioni e un operatore altamente qualificato. Di solito, l'operatore è un immunologo o un individuo altamente addestrato nel campo, con i quali è necessario incontrare al momento dell'assetto di esperimenti. Oggi le strutture accademiche hanno più strutture centrali, che possono facilitare l'esecuzione di questi tipi di esperimenti. In secondo luogo, i RGC perdono il loro lavoroMorfologia picale dovuta alla atoxomia, rendendole molto piccole. A questo punto, non è noto se alcuni dei loro geni possono essere modulati a causa di atoxomia.
La metodologia qui presentata consente l'analisi a valle della funzione RGC in vitro ed è uno strumento prezioso da utilizzare nei settori delle scienze visive e sanitarie. Il mantenimento della produzione di cellule gangliali al cervello è necessario per la percezione visiva e rischia di essere presentato in molteplici malattie. Queste cellule possono essere usate per la sperimentazione controllata in vitro , sia nei modelli sani che nelle malattie. Su queste cellule si possono eseguire studi elettrofisiologici, farmacologici, biochimici e molecolari, ideali per lo sviluppo di futuri target terapeutici.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori vorrebbero ringraziare il Sig. Tim Higgins, Senior Illustrator del Dipartimento di Microbiologia, Immunologia e Biochimica, per l'assistenza tecnica video; Il dottor Matthew W. Wilson per le discussioni ei membri dei laboratori Jablonski e Morales-Tirado per i loro commenti utili. Questo lavoro è stato sostenuto dall'Istituto di Ricerca Giovani Investigatori dell'Istituto di Ricerca di Alcon (VMM-T), dall'Università della Tennessee Research Foundation (VMM-T), dall'Istituto Nazionale di Occhi EY021200 (MMJ), dalla Gerwin Fellowship (VMM-T); La borsa di studio pre-dottorale di Gerwin (ZKG), la ricerca medica dell'esercito della difesa e il comando dei materiali (VMM-T) e la sovvenzione senza restrizioni dalla ricerca per prevenire la cecità.
Materiali
| Name | Company | Catalog Number | Comments |
| Anti-mouse CD15 PE | BioLegend | 125606 | Clone MC-480 |
| Anti-mouse CD48 PE-Cy7 | BioLegend | 103424 | Clone HM48-1 |
| Anti-mouse CD57 | Sigma Aldrich | C6680-100TST | Clone VC1.1 |
| Anti-mouse CD90.2 AF700 | BioLegend | 105320 | Clone 30-H12 |
| Brilliant Violet 421 Goat Anti-mouse IgG | BioLegend | 405317 | Clone Poly4053 |
| Purified Anti-mouse CD16/32 | BioLegend | 101302 | FcgRII/III block, Clone 93 |
| Zombie Aqua | BioLegend | 423102 | Live cell/ Dead cell discrimination |
| Fetal Bovine Serum | Hyclone | SH30071.03 | U.S. origin |
| AbC Total Antibody Compensation Bead Kit | Thermo Fisher Scientific | A10497 | Multi-species Ig |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | Add serum to media prior to culture. |
| Phosphate-Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | Saline solution |
| Dissection Microscope | Olympus | SZ-PT Model | Stereo Microscope |
| Sorvall Centrifuge | Thermo Scientific | ST 16R | All centrifugation performed at RT |
| Base Plate – Dissection Pan | Fisher Scientific | SB15233FIM | A wax plate can also be used |
| Forceps | Aesculap | 5002-7 | 4 ½ inches |
| Iris Scissors, Straight | Aesculap | 1360 | 5 ½ inches |
| Falcon 15 mL conical tubes | Fisher Scientific | 352097 | Polypropylene tubes |
| Falcon 50 mL conical tubes | Fisher Scientific | 352098 | Polypropylene tubes |
| BD FACS Tubes | Fisher Scientific | 352003 | Polypropylene tubes |
| 40 mm dishes | MidSci | TP93040 | Tissue culture treated |
| 70 μm nylon strainer | MidSci | 70ICS | sterile |
| 40 μm nylon strainer | MidSci | 40ICS | sterile |
| BD 10 mL syringe | Fisher Scientific | 301604 | Disposable Syringe without needle |
| Pestles | MidSci | PEST | sterile |
| Wheaton Vials | Fisher Scientific | 986734 | No Liner |
| BD 30 G needle | Fisher Scientific | 305128 | 1 inch |
| Hausser Scientific Bright-Line Glass Counting Chamber | Fisher Scientific | 0267151B | Hemocytometer |
| Gibco Trypan blue 0.4% Solution | Fisher Scientific | 15250061 | Viability Dye |
| Eppendorf tubes | Fisher Scientific | 05-402-25 | 1.5mL |
| EVOS Floid Cell Imaging | Thermo Fisher Scientific | 447113 | Fluorescence Imaging with a 20X objective |
| 100% Ethanol | Fisher Scientific | 04-355-452 | Used to make 70% Ethanol |
| Pipet-Lite LTS Pipette L-1000XLS+ | Rainin | 17014282 | LTS Pipette |
| Pipet-Lite LTS Pipette L-200XLS+ | Rainin | 17014391 | LTS Pipette |
| Pipet-Lite LTS Pipette L-20XLS+ | Rainin | 17014392 | LTS Pipette |
| Rack LTS 1000 mL – GPS-L1000S | Rainin | 17005088 | Blue Rack Sterile Tips |
| Rack LTS 250 mL – GPS-L250S | Rainin | 17005092 | Green Rack Sterile Tips |
| Rack LTS 20 mL – GPS-L10S | Rainin | 17005090 | Red Rack Sterile Tips |
| FACSAria II Cell Sorter | BD Biosciences | N/A | Custom order |
| LSR II Cytometer | BD Biosciences | N/A | Custom order |
| Abca8a | Thermo Fisher Scientific | Mm00462440_m1 | Müller cells |
| Aldh1al | Thermo Fisher Scientific | Mm00657317_m1 | Müller cells |
| Aqp4 | Thermo Fisher Scientific | Mm00802131_m1 | Astrocytes |
| Calb2 | Thermo Fisher Scientific | Mm00801461_m1 | Amacrine, Horizontal |
| Cd68 | Thermo Fisher Scientific | Mm03047340_m1 | Retinal Pigment Epithelial Cells |
| Gad2 | Thermo Fisher Scientific | Mm00484623_m1 | Amacrine |
| Hprt | Thermo Fisher Scientific | Mm01545399_m1 | House keeping gene |
| Lhx1 | Thermo Fisher Scientific | Mm01297482_m1 | Horizontal |
| Lim2 | Thermo Fisher Scientific | Mm00624623_m1 | Horizontal |
| Nrl | Thermo Fisher Scientific | Mm00476550_m1 | Photoreceptors |
| Ntrk1 | Thermo Fisher Scientific | Mm01219406_m1 | Horizontal |
| Pcp4 | Thermo Fisher Scientific | Mm00500973_m1 | Bipolar, Amacrine |
| Pou4f1 | Thermo Fisher Scientific | Mm02343791_m1 | Retinal Ganglion Cells |
| Prdx6 | Thermo Fisher Scientific | Mm00725435_s1 | Astrocytes |
| Prkca | Thermo Fisher Scientific | Mm00440858_m1 | Bipolar |
| Prox1 | Thermo Fisher Scientific | Mm00435969_m1 | Horizontal |
| Pvalb | Thermo Fisher Scientific | Mm00443100_m1 | Amacrine |
| Rbpms | Thermo Fisher Scientific | Mm02343791_m1 | Retinal Ganglion Cells |
| Rom1 | Thermo Fisher Scientific | Mm00436364_g1 | Photoreceptors |
| Rpe65 | Thermo Fisher Scientific | Mm00504133_m1 | Retinal Pigment Epithelial cells |
| Slc1a3 | Thermo Fisher Scientific | Mm00600697_m1 | Astrocytes |
| Slc6a9 | Thermo Fisher Scientific | Mm00433662_m1 | Amacrine |
| Sncg | Thermo Fisher Scientific | Mm00488345_m1 | Retinal Ganglion Cells |
| Tubb3 | Thermo Fisher Scientific | Mm00727586_s1 | Retinal Ganglion Cells |
| Vim | Thermo Fisher Scientific | Mm01333430_m1 | Müller cells |
| Taqman Universal Master Mix | Thermo Fisher Scientific | 4440047 | qPCR Reagent |
| miRNeasy Mini Kit | Qiagen | 217004 | RNA Isolation |
| SuperScript VILO cDNA Synthesis Kit | Thermo Fisher Scientific | 11754250 | cDNA synthesis |
| Taqman PreAmp Master Mix | Thermo Fisher Scientific | 4391128 | Pre-Amplification step |
| BD Cytofix/ Cytoperm | BD Biosciences | 554714 | Fixation/ Permeabilization Buffer |
| BD Perm/ Wash | BD Biosciences | 554723 | Permeabilization Solution |
| RBPMS | Santa Cruz Biotechnology | sc-86815 | intracellular antibody |
| SNCG | Gene Tex | GTX110483 | intracellular antibody |
| BRN3A | Santa Cruz Biotechnology | sc-8429 | intracellular antibody |
| TUJ1 | BioLegend | 801202 | intracellular antibody |
Riferimenti
- Flammer, J., Orgul, S. Optic nerve blood-flow abnormalities in glaucoma. Prog Retin Eye Res. 17 (2), 267-289 (1998).
- Bathija, R. Optic nerve blood flow in glaucoma. Clin Exp Optom. 83 (3), 180-184 (2000).
- Osborne, N. N., Melena, J., Chidlow, G., Wood, J. P. A hypothesis to explain ganglion cell death caused by vascular insults at the optic nerve head: possible implication for the treatment of glaucoma. Br J Ophthalmol. 85 (10), 1252-1259 (2001).
- Morgan, J. E. Circulation and axonal transport in the optic nerve. Eye (Lond). 18 (11), 1089-1095 (2004).
- Tian, N., Hwang, T. N., Copenhagen, D. R. Analysis of excitatory and inhibitory spontaneous synaptic activity in mouse retinal ganglion cells. J Neurophysiol. 80 (3), 1327-1340 (1998).
- Schmidt, K. G., Bergert, H., Funk, R. H. Neurodegenerative diseases of the retina and potential for protection and recovery. Curr Neuropharmacol. 6 (2), 164-178 (2008).
- Dreher, B., Sefton, A. J., Ni, S. Y., Nisbett, G. The morphology, number, distribution and central projections of Class I retinal ganglion cells in albino and hooded rats. Brain Behav Evol. 26 (1), 10-48 (1985).
- Williams, R. W., Strom, R. C., Rice, D. S., Goldowitz, D. Genetic and environmental control of variation in retinal ganglion cell number in mice. J Neurosci. 16 (22), 7193-7205 (1996).
- Jeon, C. J., Strettoi, E., Masland, R. H. The major cell populations of the mouse retina. J Neurosci. 18 (21), 8936-8946 (1998).
- Surgucheva, I., Weisman, A. D., Goldberg, J. L., Shnyra, A., Surguchov, A. Gamma-synuclein as a marker of retinal ganglion cells. Mol Vis. 14, 1540-1548 (2008).
- Nadal-Nicolas, F. M., et al. Brn3a as a marker of retinal ganglion cells: qualitative and quantitative time course studies in naive and optic nerve-injured retinas. Invest Ophthalmol Vis Sci. 50 (8), 3860-3868 (2009).
- Kwong, J. M., Caprioli, J., Piri, N. RNA binding protein with multiple splicing: a new marker for retinal ganglion cells. Invest Ophthalmol Vis Sci. 51 (2), 1052-1058 (2010).
- Van Bergen, N. J., et al. Recharacterization of the RGC-5 retinal ganglion cell line. Invest Ophthalmol Vis Sci. 50 (9), 4267-4272 (2009).
- Wood, J. P., Chidlow, G., Tran, T., Crowston, J. G., Casson, R. J. A comparison of differentiation protocols for RGC-5 cells. Invest Ophthalmol Vis Sci. 51 (7), 3774-3783 (2010).
- Barres, B. A., Silverstein, B. E., Corey, D. P., Chun, L. L. Immunological, morphological, and electrophysiological variation among retinal ganglion cells purified by panning. Neuron. 1 (9), 791-803 (1998).
- Hong, S., Iizuka, Y., Kim, C. Y., Seong, G. J. Isolation of primary mouse retinal ganglion cells using immunopanning-magnetic separation. Mol Vis. 18, 2922-2930 (2012).
- Julius, R. S. The sensitivity of exponentials and other curves to their parameters. Comput Biomed Res. 5 (5), 473-478 (1972).
- Reif, A. E., Allen, J. M. The Akr Thymic Antigen and Its Distribution in Leukemias and Nervous Tissues. J Exp Med. 120, 413-433 (1964).
- Watanabe, M., Noguchi, T., Tsukada, Y. Regional, cellular, and subcellular distribution of Thy-1 antigen in rat nervous tissues. Neurochem Res. 6 (5), 507-519 (1981).
- Haeryfar, S. M., Hoskin, D. W. Thy-1: more than a mouse pan-T cell marker. J Immunol. 173 (6), 3581-3588 (2004).
- Sahagun, G., Moore, S. A., Fabry, Z., Schelper, R. L., Hart, M. N. Purification of murine endothelial cell cultures by flow cytometry using fluorescein-labeled griffonia simplicifolia agglutinin. Am J Pathol. 134 (6), 1227-1232 (1989).
- Shoge, K., et al. Rat retinal ganglion cells culture enriched with the magnetic cell sorter. Neurosci Lett. 259 (2), 111-114 (1999).
- Chintalapudi, S. R., et al. Isolation and Molecular Profiling of Primary Mouse Retinal Ganglion Cells: Comparison of Phenotypes from Healthy and Glaucomatous Retinas. Front Aging Neurosci. 8, 93 (2016).
- Nagdeve, N. G., Yaddanapudi, S., Pandav, S. S. The effect of different doses of ketamine on intraocular pressure in anesthetized children. J Pediatr Ophthalmol Strabismus. 43 (4), 219-223 (2006).
- Ding, C., Wang, P., Tian, N. Effect of general anesthetics on IOP in elevated IOP mouse model. Exp Eye Res. 92 (6), 512-520 (2011).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Aerts, J., Nys, J., Arckens, L. A highly reproducible and straightforward method to perform in vivo ocular enucleation in the mouse after eye opening. J Vis Exp. (92), e51936 (2014).
- Li, X., et al. Loss of AP-2delta reduces retinal ganglion cell numbers and axonal projections to the superior colliculus. Mol Brain. 9 (1), 62 (2016).
- Moshiri, A., et al. Near complete loss of retinal ganglion cells in the math5/brn3b double knockout elicits severe reductions of other cell types during retinal development. Dev Biol. 316 (2), 214-227 (2008).
- Danias, J., et al. Quantitative analysis of retinal ganglion cell (RGC) loss in aging DBA/2NNia glaucomatous mice: comparison with RGC loss in aging C57/BL6 mice. Invest Ophthalmol Vis Sci. 44 (12), 5151-5162 (2003).
- Jakobs, T. C., Ben, Y., Masland, R. H. CD15 immunoreactive amacrine cells in the mouse retina. J Comp Neurol. 465 (3), 361-371 (2003).
- Uusitalo, M., Schlotzer-Schrehardt, U., Kivela, T. Ultrastructural localization of the HNK-1 carbohydrate epitope to glial and neuronal cells of the human retina. Invest Ophthalmol Vis Sci. 44 (3), 961-964 (2003).
- Jackson, C. J., Garbett, P. K., Nissen, B., Schrieber, L. Binding of human endothelium to Ulex europaeus I-coated Dynabeads: application to the isolation of microvascular endothelium. J Cell Sci. 96 ( Pt 2), 257-262 (1990).
- Pennartz, S., Perraut, M., Pfrieger, F. Purification of retinal ganglion cells from postnatal rats by magnetic cell sorting. MACSmore. 12 (2), 16-18 (2010).
- Nadal-Nicolas, F. M., et al. Displaced retinal ganglion cells in albino and pigmented rats. Front Neuroanat. 8, 99 (2014).
- Nadal-Nicolas, F. M., Salinas-Navarro, M., Vidal-Sanz, M., Agudo-Barriuso, M. Two methods to trace retinal ganglion cells with fluorogold: from the intact optic nerve or by stereotactic injection into the optic tract. Exp Eye Res. 131, 12-19 (2015).
- Barnstable, C. J., Drager, U. C. Thy-1 antigen: a ganglion cell specific marker in rodent retina. Neuroscience. 11 (4), 847-855 (1984).
- Chiu, K., Lau, W. M., Yeung, S. C., Chang, R. C., So, K. F. Retrograde labeling of retinal ganglion cells by application of fluoro-gold on the surface of superior colliculus. J Vis Exp. (16), (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon