Method Article
La quantificazione di espressione della proteina e co-localizzazione Usando multiplex immunoistochimica di colorazione e multispettrale Imaging
In questo articolo
Riepilogo
Immunohistochemistry is a powerful lab technique for evaluating protein localization and expression within tissues. Current semi-automated methods for quantitation introduce subjectivity and often create irreproducible results. Herein, we describe methods for multiplexed immunohistochemistry and objective quantitation of protein expression and co-localization using multispectral imaging.
Abstract
Immunohistochemistry is a commonly used clinical and research lab detection technique for investigating protein expression and localization within tissues. Many semi-quantitative systems have been developed for scoring expression using immunohistochemistry, but inherent subjectivity limits reproducibility and accuracy of results. Furthermore, the investigation of spatially overlapping biomarkers such as nuclear transcription factors is difficult with current immunohistochemistry techniques. We have developed and optimized a system for simultaneous investigation of multiple proteins using high throughput methods of multiplexed immunohistochemistry and multispectral imaging. Multiplexed immunohistochemistry is performed by sequential application of primary antibodies with secondary antibodies conjugated to horseradish peroxidase or alkaline phosphatase. Different chromogens are used to detect each protein of interest. Stained slides are loaded into an automated slide scanner and a protocol is created for automated image acquisition. A spectral library is created by staining a set of slides with a single chromogen on each. A subset of representative stained images are imported into multispectral imaging software and an algorithm for distinguishing tissue type is created by defining tissue compartments on images. Subcellular compartments are segmented by using hematoxylin counterstain and adjusting the intrinsic algorithm. Thresholding is applied to determine positivity and protein co-localization. The final algorithm is then applied to the entire set of tissues. Resulting data allows the user to evaluate protein expression based on tissue type (ex. epithelia vs. stroma) and subcellular compartment (nucleus vs. cytoplasm vs. plasma membrane). Co-localization analysis allows for investigation of double-positive, double-negative, and single-positive cell types. Combining multispectral imaging with multiplexed immunohistochemistry and automated image acquisition is an objective, high-throughput method for investigation of biomarkers within tissues.
Introduzione
Immunoistochimica (IHC) è una tecnica di laboratorio standard per il rilevamento di proteine nei tessuti, e IHC è ancora ampiamente utilizzato sia nella ricerca e patologia diagnostica. La valutazione di IHC colorazione è spesso semi-quantitativa, l'introduzione di potenziali bias in interpretazione dei risultati. Molti approcci semi-quantitativi sono stati sviluppati che incorporano sia l'intensità di colorazione e misura colorazione in diagnosi finale 1-4. Altri sistemi includono intensità punteggio e posizione subcellulare al fine di localizzare meglio l'espressione 5. Incorporazione di punteggi medi da più spettatori è spesso utilizzato per ridurre al minimo gli effetti di singolo spettatore pregiudizi 6. Nonostante questi sforzi, la soggettività nell'analisi rimane, in particolare al momento di valutare il grado di colorazione 7. Protocollo di standardizzazione e la soggettività riducendo al minimo da input umano è fondamentale per la creazione di accurati, riproducibili risultati IHC.
content "> Ci sono altre opzioni oltre IHC per determinare l'espressione della proteina all'interno dei tessuti. All'interno del contesto della ricerca, l'immunoistochimica è stata tradizionalmente vista come un mezzo per esaminare la proteina localizzazione 8, mentre altre tecniche come immunoblotting sono visti come gold standard per indagare l'espressione della proteina . Determinare il tessuto o l'espressione specifici vano cella è difficile senza incorporare tecniche avanzate come il frazionamento delle cellule o microdissezione laser 9,10. L'uso di anticorpi fluorescenti su slitte di tessuto offre un compromesso ragionevole, ma sfondo autofluorescenza a causa di NADPH, lipofuscine, reticolare fibre, collagene, elastina e possono rendere difficile la quantificazione precisa 11.Piattaforme patologia di calcolo automatici sono una direzione promettente per più quantificazione oggettiva della patologia colorazione 12-15. La combinazione di imaging multispettrale con microarray di tessutifacilita l'analisi ad alta produttività di espressione della proteina in campioni di grandi dimensioni. Con queste tecniche, analisi di proteine co-localizzazione, eterogeneità colorazione, e tessuti e localizzazione subcellulare è possibile riducendo sostanzialmente due polarizzazioni e il tempo necessario per l'analisi intrinseche, mentre restituzione dei dati in modo continuo piuttosto che in formato categorica 16. Pertanto, lo scopo di questo studio era quello di dimostrare l'utilità di e metodologia per l'esecuzione di immunoistochimica multiplex con l'analisi, utilizzando software di imaging multispettrale.
Questo protocollo è stato scritto per il manuale, multiplex colorazione immunoistochimica di una singola diapositiva sezione di tessuto con quattro anticorpi monoclonali ottimizzati. Come un esperimento rappresentativo, nucleare anti-coniglio recettore degli estrogeni alfa (ERα) e del recettore degli androgeni (AR) sono multiplexati con legato alla membrana anti-topo CD147 e legata alla membrana anti-topo E-caderina. Qualsiasi anticorpo di scelta può essere substitutEd per gli anticorpi elencati qui, ma ogni combinazione di anticorpi richiede l'ottimizzazione separata. Pre-trattamento per tutti gli anticorpi devono essere identici. Gli anticorpi AR e CD147 dovrebbe essere ottimizzato individualmente e poi come un cocktail. Ogni anticorpo viene rilevato utilizzando un sistema polimerico privo di biotina e uno dei 4 cromogeni unici.
Protocollo
NOTA: Il protocollo descrive qui colorazione e analisi di un microarray tissutale (TMA), precedentemente descritto 12,17,18. La spessa sezione TMA 4 micron è stato ottenuto da un blocco di paraffina utilizzando un microtomo standard.
NOTA: Una libreria spettrale per i 4 cromogeni e di contrasto deve essere creato per un'immagine quantificazione. Per fare questo, il protocollo ottimizzato per ciascun anticorpo deve essere eseguito con un anticorpo per vetrino, meno la controcolorazione finale. Un quinto diapositiva deve essere colorato con ematossilina per generare le 5 immagini necessarie per creare la libreria spettrale.
1. Multiplex immunoistochimica
- diapositive Cuocere in forno a 60 ° C per 20 min. Usando una cappa, immergere scivolo in 100% xilene. Ripetere per 2 cambi di xilene. Immergere scorrevole in 100% di etanolo per 5 min. Ripetere con fresco al 100% di etanolo.
- Immergere vetrino in etanolo al 95% per 3 min. Ripetere con fresco95% di etanolo. Immergere vetrino in etanolo al 70% per 3 min. Immergere vetrino in acqua distillata per 3 min. Ripetere con acqua fresca distillata. Toccare scorrere verticalmente per drenare l'acqua dalla diapositiva. Applicare 200 ml di ready-to-use endogena bloccanti perossidasi per 5 min.
- Drain perossidasi bloccante dal vetrino ed immergerlo in 45 ml di tampone di recupero ready-to-use (pH inferiore a 7,0). Collocare il contenitore di diapositive in una pentola a pressione e avviare il programma impostato a 124 ° C per 4 min. Lasciare pentola a pressione raffreddare per 20 minuti. prima dell'apertura.
- Rimuovere il contenitore di tampone recupero e lasciare raffreddare altri 10 min. Sciacquarlo in acqua distillata per 10 min. Scaricare l'acqua dal vetrino e delimitare accuratamente il tessuto sul vetrino con una penna barriera idrofobica.
- Aggiungere 200 ml di tampone fosfato di scorrimento (PBS) .Transfer ad una presa piatta con coperchio (camera di incubazione) contenente una garza umida.
- Preparare 1 litro di soluzione salina 1x Tris tamponata con Tween-20 (TBST) diluendo 50 ml di 20x Tris tamponata salina contenente 1% Tween-20 e 950 ml di acqua distillata.
- Scolare PBS dal vetrino e applicare 200 ml di 1x TBST. Incubare 2 min. Scolare TBST dalla presentazione e tornare scorrevole nella camera di incubazione. Applicare 200 ml di blocco della proteina universale pronta per l'uso, nei pressi della camera e incubare per 7 min.
- Scolate blocco di proteine e tornare scorrevole nella camera di incubazione. Diluire anti-ERα anticorpi 1: 400 con l'aggiunta di 0,6 ml di anticorpi a 239,4 ml di diluente. Applicare 200 ml di anticorpo diluito a scivolare e vicino da camera.
- Incubare il vetrino a temperatura ambiente per 2 ore. Drain anticorpo vetrino e sciacquare con TBST seguito da immersione per 5 min in TBST.
- Scolare TBST dalla presentazione e tornare scorrevole nella camera di incubazione. Applicare 200 ml di capra anti-coniglio perossidasi polimero da usare pronto e vicino da camera. Incubare per 30 min. a temperatura ambiente. vetrino Risciacquare con TBST e posto in un contenitore di diapositive TBST per 5 min.
- Preparare marrone perossidasi-3,3'-diaminobenzidina (HRP-DAB) cromogeno con l'aggiunta di 8 ml di cromogeno DAB a 250 microlitri tampone substrato DAB e mescolando bene.
- Scolare TBST da slitta, slitta ritorno alla camera di incubazione e applicare 200 ml di marrone cromogeno HRP-DAB per 6 min. Risciacquare il vetrino con acqua distillata e posto vetrino in un recipiente di acqua distillata.
- Miscelare la soluzione di denaturazione combinando 50 microlitri del primo reagente eluizione con 150 ml di secondo reattivo tampone. Scolare l'acqua dal vetrino e applicare la soluzione di denaturazione diluita alla diapositiva per 3 min. a temperatura ambiente. Decantare e risciacquare denaturazione reagente con TBST. Immergere scivolo in TBST per 5 min.
- Preparare AR / CD147 anticorpo cocktail diluendo 4,2 ml di anticorpo anti-AR e 2,8 ml di anticorpo anti-CD147 con 203 ml di diluente. Scolare TBST dalla diapositiva, tornare scorrevole alla camera di incubazione e applicare 200 ml di AR / CD147 cocktail alla diapositiva. Chiudere la camera e incubare per 1 ora a temperatura ambiente. vetrino Risciacquare con TBST e immergersi in TBST per 5 min.
- Scolare TBST, scivolo ritorno alla camera di incubazione e applicare 200 ml di ready-to-use di capra anti-coniglio fosfatasi alcalina polimero. Chiudere la camera e scivolo incubare per 30 min. a temperatura ambiente. vetrino Risciacquare con TBST e immergersi in TBST per 5 min.
- Preparare fosfatasi alcalina rosso cromogeno con l'aggiunta di 3,2 ml di cromogeno rosso a 250 microlitri di buffer cromogeno rosso e mescolare bene.
- Scolare TBST dalla presentazione e tornare scorrevole nella camera di incubazione. Applicare 200 ml di diluito alcalina rosso fosfatasi cromogeno alla diapositiva, chiudere da camera e incubare per 7 min.
- Scolare cromogeno e sciacquare scivolo con acqua distillata. Posizionare vetrino in un recipiente di acqua distillata per 5 min. Scolare l'acqua dal vetrino, sciacquare con TBST e tornare alla camera di incubazione. Applicare 200 microlitri di capra anti-topo-HRP polimero alla slitta e close da camera. Incubare per 30 min. a temperatura ambiente.
- Scolare polimero dalla diapositiva e risciacquare con TBST. Porre il vetrino in TBST per 5 min. Preparare 200 ml di viola cromogeno HRP-linked con l'aggiunta di 3,2 ml di reagente di stabilizzazione a 250 microlitri di buffer e mescolare bene. Aggiungere 3,2 ml di cromogeno viola e mescolate bene, poi 3,2 ml di reagente perossido di idrogeno e mescolare bene.
- Scolate TBST off vetrino e applicare 200 ml di cromogeno viola pronti a scivolare e incubare per 7 min. a temperatura ambiente. scivolo Risciacquare con acqua e posto distillata diapositiva in un contenitore di acqua distillata per 5 minuti.
- Miscelare la soluzione di denaturazione miscelando 50 ml di primo reattivo eluizione con 150 ml di secondo reattivo tampone. Scolare l'acqua dal vetrino e applicare la soluzione di denaturazione diluita alla diapositiva per 3 min. a temperatura ambiente.
- Risciacquare denaturazione reagente via con TBST. Porre il vetrino in un contenitore di TBST per 5 min. Diluire anti- E-caderina-anticorpia 1: 200 con l'aggiunta di anticorpi 1,1 ml a 218,9 ml diluente. Scolare la TBST dalla diapositiva, scorrevole ritorno alla camera di incubazione e applicare 200 ml di E-caderina alla diapositiva. Chiudere la camera e incubare 30 min. a temperatura ambiente.
- vetrino Risciacquare con TBST e posto in un contenitore di diapositive TBST per 5 min. Scolare la TBST da slitta, slitta tornare alla camera di incubazione e applicare 200 pl di capra anti-topo HRP polimero. Incubare a temperatura ambiente per 30 min. vetrino Risciacquare con TBST e posto in un contenitore di diapositive TBST per 5 min.
- Preparare nero HRP-linked cromogeno con l'aggiunta di 8 ml di cromogeno a 250 microlitri di buffer. Applicare 200 microlitri cromogeno a scivolare e sviluppare per 2 min. Sciacquare accuratamente scivolo con acqua distillata e immergere in un contenitore di acqua distillata.
- Diluire ematossilina con l'aggiunta di 40 microlitri ematossilina a 200 ml di acqua distillata. Scolare l'acqua dal vetrino e applicare 200 ml di ematossilina diluita a scivolare per 30 secondi. scivolo Rinseaccuratamente in acqua corrente per 2 min., quindi risciacquare in acqua distillata per 30 secondi. Porre il vetrino in un forno a 60 ° C per 15-30 minuti.
- Immergere diapositiva appena preparato al 100% xilene per 30 secondi, eliminare l'eccesso xilene fuori e aggiungere una goccia di mezzo di montaggio permanente. Coprioggetti con un No. 1.5 vetrino.
2. Automated Image Acquisition e analisi
- Caricare i vetrini colorati su uno scanner automatico scorrevole. Sulla base del diametro, la separazione, e il numero di core TMA, creare un protocollo di scansione automatica in funzione manuale di istruzioni del produttore.
- Aprire il software di imaging multispettrale per costruire una libreria spettrale da vetrini di controllo colorati con i singoli cromogeni e la slitta ematossilina macchiato. Aprire un cubo di immagine acquisita da un vetrino di controllo e selezionare quattro o cinque aree colorate positivamente per definire otticamente che cromogeno. Ripetere con cubetti di immagini provenienti da altri vetrini di controllo fino a quando un completo representi biblioteca spettraleng tutti i cromogeni è stato creato, e quindi salvare la libreria spettrale.
- Iniziare un nuovo progetto all'interno di software di imaging multispettrale. Selezionare "multispettrale (.im3)" per l'opzione formato di immagine e "campo chiaro" per il formato campione. In primo luogo, configurare il progetto scegliendo opzioni desiderate: "Segmento Tissue", "Trova Funzioni", "Fenotipizzazione", "punteggio" e "Esporta". A questo punto, la risoluzione dell'immagine può essere modificata per accelerare i tempi di analisi, se desiderato.
3. Tessuto Segmentazione
- Importare la libreria spettrale creato in precedenza e selezionare tutti i cromogeni da includere nell'analisi.
- Aperte cubetti di immagini da includere nei dati di formazione di selezionando l'opzione "Apri immagine Cube". Per garantire la precisione di formazione, selezionare almeno il 18% del numero totale di immagini da analizzare. Scegliere le immagini che rappresentano tutti gli stati di malattia per aumentare la precisione di segmentazione. Questa porzione di immagini is chiamato il training set di immagini.
- immagini di bilanciamento del bianco nella formazione impostata selezionando lo strumento contagocce e selezionate un territorio in una sola immagine che è bianco.
- Selezionare il pulsante "advance" per passare alla segmentazione dei tessuti. Utilizzare il pannello "Tissue Categorie" per scegliere i tipi di tessuto da analizzare (cioè "tessuto" e "non-tessuto"). Per più precisa localizzazione dei tessuti di proteine, più categorie di tessuti possono essere utilizzati (es. "Epiteli," "stroma," e "non-tessuto").
- Iniziare a creare l'algoritmo e definizione delle categorie di tessuto disegnando intorno a gruppi di cellule all'interno di immagini di addestramento. Una volta terminato con una categoria di tessuto, ripetere per altre categorie di tessuto. Siate sicuri di scegliere gruppi di cellule all'interno delle immagini che sono caratteristici di questo tipo di tessuto categoria.
- Ripetere questa procedura per tutte le immagini all'interno del training set di immagini.
- Selezionare i componenti (cromogeni) da inserire nel Training per il "Tissue segmenter". Quando si esegue l'analisi di immagini in campo chiaro immunoistochimica, tutti i componenti sono normalmente inclusi nella formazione. Includi abbondanti immagini colorazione negativa nella formazione impostata per evitare distorsioni durante questa fase.
- Scegliere un adeguato "Scala del modello" per la formazione del tessuto segmenter. Sotto 20X di ingrandimento, una scala modello di grandi dimensioni è in genere appropriato. Quando si lavora con i tessuti con una raffinata architettura sotto ingrandimento più elevato, una scala modello più piccolo è più appropriato.
- Selezionare il pulsante "Treno Tissue segmenter" per avviare la formazione del tessuto segmenter. Osservare una precisione finestra pop-up che visualizza riflette la percentuale di pixel all'interno di regioni di formazione che sono correttamente classificate.
NOTA: Il software tenta continuamente di migliorare la precisione degli algoritmi fino all'arresto manuale. Abbiamo precedentemente dimostrato che la formazione, il 18% delle immagini risultati nel 97% di precisione di formazione 12. - Dopo il segmenter tessuto è formato, scegliere una risoluzione segmento appropriato. risoluzione segmento corrisponde al tempo necessario per le immagini del segmento, con una risoluzione grossolana che richiede meno tempo e risoluzione fine che richiede più tempo.
- Segmento l'intero set di formazione di immagini facendo clic su "segmento immagini". Lasciare il software tempo sufficiente per applicare l'algoritmo a tutte le immagini di addestramento. Al termine, rivedere l'allenamento impostato per trovare qualsiasi tessuto classificato erroneamente con l'algoritmo di formazione corrente.
- Per mettere a punto il processo di segmentazione dei tessuti, aggiungere, modificare o rimuovere le regioni di formazione di tessuto. Utilizzare l'opzione "bordi di taglio", se si estendono pixel sui bordi di una categoria di tessuto in un altro. Se piccoli gruppi di pixel sono classificate erroneamente, regolare la soglia della dimensione minima dei segmenti.
- Quando le modifiche sono completate, selezionare "Immagini del settore", questo sarà ri-formare il segmenter tessuti per creare un nuovo algoritmo per la segmentazione dei tessuti. L'algoritmo precedenteviene salvato automaticamente e possono essere restituiti a se necessario.
- Quando fiducioso con risultati algoritmo di segmentazione dei tessuti, avanzare alla segmentazione delle cellule selezionando il pulsante "Advance".
4. cellulare Segmentazione
- Scegli i compartimenti subcellulari da includere nella segmentazione delle cellule. Assicurarsi che "nuclei" è già selezionato per scegliere "citoplasma" o "a membrana".
NOTA: "a membrana" segmentazione dovrebbe essere scelto soltanto quando un marcatore specifica proteina-membrana è stata inclusa nel IHC, ad esempio E-caderina. - All'interno della scheda "Nuclei", ci sono diverse opzioni. Inizia scegliendo impostazioni appropriate per la segmentazione nucleare (vedi punto 4.3). Scegliere se singole o tutte le categorie di tessuto saranno incluse nella segmentazione.
- Selezionare un metodo per la segmentazione nucleare
- Selezionare la di contrasto "basato su pixel (soglia)" approccio per un metodo semplificato per l'ottenimento di un buonrisultati senza sapere molto sulle impostazioni di elaborazione delle immagini.
NOTA: Questo rende spesso una buona scelta. Quando si seleziona un approccio "basato su pixel (soglia)" è più appropriato quando vi è una contro-colorazione nucleare affidabile, mentre "object-based" dovrebbe essere usato se vi è una mancanza di di contrasto nucleare coerente. - Selezionare il "Pixel-Based (soglia)" approccio quando vi è un di contrasto nucleare affidabile.
NOTA: Questo approccio è puramente basato su pixel. Questo approccio può essere utilizzato anche per altre esigenze di analisi delle immagini che possono essere soddisfatte con un semplice soglia, ad esempio rilevare tutti i pixel all'interno di una categoria tessuto che macchia positivo per una macchia IHC. - Selezionare l'approccio basato su oggetti se il nucleare di contrasto non fornisce colorazione coerente e specifico di oggetti nucleari, e sono necessari approcci morfometria basata più avanzati per rilevare nuclei.
- Selezionare "Componenti per la segmentazione Nucleare", "Nuclorecchio Size "e" Clean-up "se l'utente desidera definire ulteriormente le impostazioni di segmentazione nuclei.
- Selezionare la di contrasto "basato su pixel (soglia)" approccio per un metodo semplificato per l'ottenimento di un buonrisultati senza sapere molto sulle impostazioni di elaborazione delle immagini.
- Selezionare parametri di forma citoplasma
- Selezionare il pulsante "advance" per passare alla segmentazione del citoplasma.
NOTA: Ci sono diverse opzioni all'interno di citoplasma segmentazione tra cui scegliere. L'opzione chiamata "distanza interno al nucleo" è pixel in base. Essa riflette la distanza tra l'esterno del nucleo e l'interno del citoplasma. Aumentando questo valore diminuisce la probabilità che il segnale nucleare attraversa nel vano citoplasma. - Avanti, selezionare, "distanza esterno per nucleo".
NOTA: Questo è anche pixel in base. La distanza esterna al nucleo riflette la distanza tra l'esterno del nucleo e l'esterno della cellula. Questo valore può essere impostato o grande abbastanza piccolo da includere o escludere segnali di membrana se desiderato. - Avanti, selezionare, "dimensione minima".
NOTA: La dimensione minima, PIXEl basato, riflette la dimensione minima del campione citoplasma da includere nell'analisi. Se la dimensione citoplasma è più piccolo in una cella particolare come quando le cellule sono strettamente imballati insieme, questo citoplasma segmentazione è escluso dall'analisi. - Selezionare l'opzione successiva, "Component" con "Primario", "secondario", e selezionare "Terziario" come opzioni secondarie.
NOTA: Qui si può scegliere un marcatore citoplasma specifica. Simile alla segmentazione nucleare, se è stato usato un marcatore IHC citoplasmatica specifica, questo componente può essere utilizzato per definire citoplasma trovando un valore minimo di soglia. Questo aiuta nel definire citoplasma, ma non è necessario per la precisione accettabile. - Spostare l'ultima delle tre opzioni a schede è "a membrana". Qui, l'utente definisce il vano membrana con proteina marker. Scegliere "Primario", "Secondario" e / o "terziario" per ogni marker specifico membrana utilizzata. Regolare "Full Scale OD "per trovare una soglia minima o positiva per ogni marcatore sulle membrane cellulari.
- Proseguendo nella scheda "a membrana" e selezionare l'opzione "Cell Dimensione massima".
NOTA: Questa è la distanza al valore di membrana, in pixel, che specifica la dimensione massima delle celle. Un valore di 12 è normalmente adeguata per le immagini scattate a 20X di ingrandimento. - Dopo aver selezionato tutte le opzioni, selezionare "Segment immagine" o "Segmento All". Applicare le impostazioni per immagini e osservarle "singolarmente" o in modalità "Galleria".
NOTA: regolare le impostazioni e le soglie, se necessario, e ri-segmento fino soddisfatti con i risultati di segmentazione delle cellule del training set di immagini.
- Selezionare il pulsante "advance" per passare alla segmentazione del citoplasma.
5. Fenotipizzazione di celle
NOTA: accurata segmentazione delle cellule è necessaria al fine di ottenere precise fenotipizzazione delle cellule, e la funzione di fenotipizzazione è addestrabile.
- Utilizzare il "Aggiungi &# 34; pulsante per aggiungere fenotipi alla lista "fenotipi". Gli utenti possono selezionare un colore per il fenotipo.
- Clicca su "Modifica fenotipi" per assegnare un fenotipo a una cella. Clicca su una particolare cella per visualizzare un menu a discesa, che permette all'utente di scegliere il fenotipo e poi passare. Identificare almeno cinque (5) cellule in ciascun fenotipo per procedere. La scelta di 25 o più celle di ogni fenotipo è necessaria per ottenere risultati ottimali.
6. punteggio IHC e co-localizzazione
- Scegliere "Advance" per passare al "punteggio IHC o IF" step. Scegliere un "tessuto categoria" per segnare.
NOTA: solo una categoria di tessuto può essere segnata nel corso di una particolare analisi, ma se i risultati di punteggio in un'altra categoria dei tessuti sono desiderati, l'analisi può essere ripetuta più tardi con impostazioni diverse. - Scegliere un tipo di "scoring" desiderato.
NOTA: positività crea due scomparti (positivo e negativo) per un componente di interesse, Mentre doppia positività è utilizzato per l'analisi co-localizzazione. Altre opzioni includono 4-bin, 10-bin, e 50-bin analisi per un marker di scelta. - Scegliere la cella "scomparto" da utilizzare nel notare analisi.
- Simile alla ricerca di una soglia minima per la componente nucleare primaria, selezionare "View componente dati" e spostare il cursore sopra le immagini di addestramento per trovare appropriate densità ottica soglie minime di colorazione per le cellule positive per il componente (s) di interesse.
- Se le cellule intensamente colorate devono essere esclusi, abbassare la soglia massima ad un livello appropriato. In caso contrario, utilizzare il tasto AUTO per scegliere una soglia massima per l'analisi.
- Scegliere la formazione immagini di segnare al fine di convalidare i valori thresholding.
NOTA: La percentuale di cellule all'interno di un particolare bin verrà restituito e può essere confrontato mediante ispezione visiva o conteggio manuale. Una volta completato, selezionare "Advance" per passare al punto "Esporta".
7. Applicando l'algoritmo e Batch Analysis
- Testare l'algoritmo creato attraverso la segmentazione dei tessuti, la segmentazione delle cellule, e dei valori di punteggio esportando i dati per il training set. Seguire le istruzioni per creare una nuova cartella per la directory di esportazione. Selezionare le immagini e le tabelle da creare e inclusi nell'analisi. Eseguire l'analisi selezionando "Esporta per tutti".
- Le tabelle vengono esportati in file di testo separati da tabulazioni e possono essere aperti nella maggior parte dei programmi di analisi dei dati.
- Quando l'analisi è completa, visualizzare i dati di segmentazione delle cellule e di punteggio per le immagini con alta e bassa colorazione per valutare l'accuratezza delle impostazioni.
- Quando si è soddisfatti con le impostazioni, applicare l'algoritmo creato dal training set di immagini per l'intero set di immagini attraverso l'analisi dei lotti. Fare clic sulla scheda "Batch Analysis" e l'algoritmo aperto verrà copiato dal progetto attivo.
- Scegli una nuova directory di esportazione e selezionare quali immagini e tables devono essere inclusi nell'analisi. Sotto l'opzione file di input, selezionare "Aggiungi immagini" e scegliere tutte le immagini da includere nell'analisi batch.
- Eseguire l'analisi selezionando l'opzione "Esegui". Come con l'analisi del training set, questo passaggio richiede un tempo variabile, a seconda delle particolari impostazioni e numero di immagini incluse nell'analisi.
- Una volta completato, passare alla scheda "Review / Merge". Le impostazioni predefinite della directory in batch nella directory di esportazione dall'analisi dei lotti e possono essere modificati se lo si desidera. Selezionare "Includi tutto" e selezionare "Merge" per creare schede tecniche con i dati di riepilogo per l'analisi.
8. Analisi dei dati esportati
- Aprire tavoli esportati software di analisi dei dati e rimuovere o nascondere le colonne di dati che sono irrilevanti per l'analisi, come ad esempio i valori minimo e massimo per ciascun componente. I dati primario utilizzato in analisi è la densità ottica c medioolumn per ogni componente.
- Recensione esportato mappe di segmentazione e le immagini RAW per determinare quali campioni o per anime TMA devono essere esclusi dall'analisi. valori percentuali tessuto aiutano a determinare se un campione deve essere inclusa o meno, con un cut-off arbitrario di <5% utilizzato più spesso per criteri di esclusione.
- Rimuovere i dati irrilevanti o le righe contenenti i campioni che devono essere esclusi dall'analisi dei dati.
- Utilizzare i dati di proteine di quantificazione per indagare cambiamenti di stato di malattia o il rapporto con le caratteristiche clinico-patologici della malattia 12,18-21.
Risultati
In figura 1, la formazione viene eseguito sui tessuti della prostata per immagini segmento in porzioni epiteliali e stromali, insieme con il vano non tessuto. Utilizzando il marcatore epiteliale di membrana E-caderina, la segmentazione delle cellule è stata eseguita per separare il nucleo, il citoplasma, e porzioni di membrana, mostrate nella Figura 2.
In un esperimento, abbiamo usato multiplato IHC per studiare l'espressione e la localizzazione di AR, ERα, E-caderina, e CD147, come mostrato in figura 3. Utilizzando queste tecniche, siamo in grado di identificare le cellule positive per l'espressione nucleare sia ERα e AR (Figura 3B - 3C), nonostante la sovrapposizione di segnali colorimetriche, come indicato con le frecce verdi in Figura 3A. Abbiamo trovato marcate differenze nella proporzione di doppi cellule stromali positive all'interno di diversi stati di prmalattia ostate (Figura 3D). Abbiamo quantificato-specifico espressione membrana cellulare dei CD147 utilizzando E-caderina come una proteina marcatore (frecce rosse in figura 3A), e siamo stati il primo gruppo di indagare specifica espressione CD147 membrana nei tessuti della prostata. Abbiamo trovato una significativa diminuzione dell'espressione CD147 in associazione con la progressione del cancro alla prostata (Figura 3E) e abbiamo trovato una importante associazione con la prognosi post-chirurgica 19.
Ci sono casi in cui cattiva progettazione esperimento può portare alla segmentazione dei tessuti impreciso. Nella Figura 4, l'algoritmo applicato a un insieme di tessuti non ha accuratamente segmento epiteliale e scomparti stromali (Figura 4C). In questo esperimento, α-actina del muscolo liscio (α-SMA) è stato utilizzato per contrassegnare il compartimento stromale. Perché α-SMA e della muscolatura liscia è diminuita o persi in alcuni tumori (Figure 4A), l'algoritmo creato attraverso la segmentazione dei tessuti (Figura 4B) è stato in grado di distinguere con precisione tra le epiteliali e stromali scomparti a partire da soli dati morfometrici. Si deve prestare attenzione quando si sceglie marcatori proteici per compartimenti o cellulari.

Figura 1: Tissue Segmentazione Utilizzando software di imaging multispettrale. Un microarray tessuto prostatico era macchiato con immunoistochimica multiplex per il recettore degli androgeni (AR), il recettore degli estrogeni alfa (ERα), E-caderina, e CD147. Una serie di immagini di addestramento sono stati importati in un software di imaging multispettrale che rappresentano tipi di tessuto e stati di malattia di tutta la serie di immagini (A). categorie tessuto sono stati creati compresa stroma (verde), epiteli (rosso) e non-tessuto (blu), e categorie sono state definite disegnando manualmente in cimadi formazione immagini (B). Dopo aver disegnato sulle immagini di formazione, un algoritmo per la segmentazione dei tessuti è stato creato e applicato al training set di immagini (C). Si prega di cliccare qui per vedere una versione più grande di questa figura.

Figura 2: Cell segmentazione in nucleare, citoplasmatica, e membrana scomparti. Il compartimento nucleare è stato definito per la segmentazione delle cellule fissando una soglia minima per Ematossilina di contrasto (A). Il citoplasma è stata definita in relazione al nucleo utilizzando algoritmi pre-impostati all'interno del software (B). E-caderina è stato utilizzato come marcatore di membrana e significa una soglia minima OD è stato applicato per definire il vano membrana (C). Questa tecnica tuttoOWS quantificazione simultanea di espressione della proteina in tutti i compartimenti subcellulari (D). Clicca qui per vedere una versione più grande di questa figura.
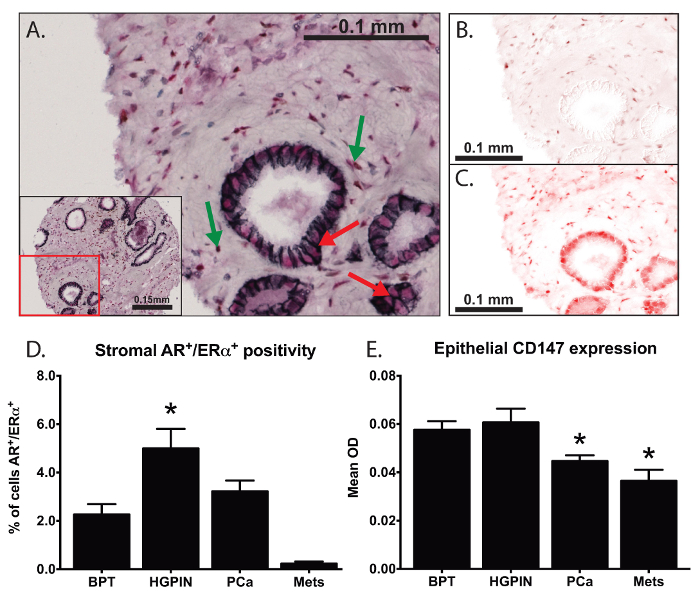
Figura 3: Analisi di co-localizzazione e membrana specifica espressione della proteina. Multiplex IHC è stato utilizzato per studiare l'espressione e la localizzazione di recettore degli androgeni (AR), il recettore degli estrogeni alfa (ERα), E-caderina, e CD147 (A). DAB (marrone cromogeno) è stato usato per marcare AR (B) e un cromogeno rosso usato per marcare ERα (C). Siamo stati in grado di identificare biomarcatori spazialmente sovrapposte (frecce verdi in A) e quantificare (D) la proporzione di cellule con nucleare co-localizzazione di ERα e ARall'interno stroma prostatico. Usando E-caderina (cromogeno nero) per definire la membrana plasmatica (frecce rosse in A), abbiamo quantificato specifica espressione-membrana CD147 all'interno dei tessuti della prostata (E) 19. Unidirezionale analisi della varianza (ANOVA) è stato utilizzato per l'analisi statistica, le barre di errore riflettono l'errore standard della media, e asterischi rappresentano p <0.05. Clicca qui per vedere una versione più grande di questa figura.
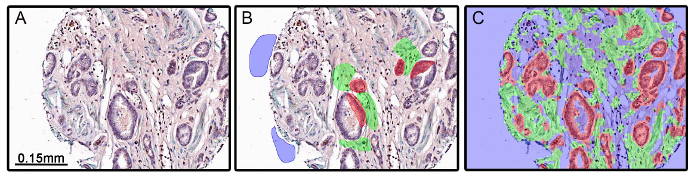
Figura 4: inadeguata Tissue segmentazione risultante dal disegno sperimentale. tessuti prostatici benigni e maligni sono state colorate per actina α-muscolo liscio (α-SMA; cromogeno verde), recettore degli androgeni (cromogeno rosso) e del recettore degli androgeni variante 7 (cromogeno DAB). alfa-SMA è stato utilizzato comeun marker di stroma ed è diminuita in alcuni tumori, compreso la prostata (A). Quando la formazione è stata eseguita (B) e l'algoritmo di segmentazione del tessuto è stato applicato, il stromali ed epiteliali scompartimenti erano adeguatamente segmentata per l'assenza di α-SMA colorazione (C). Cliccate qui per vedere una versione più grande di questa figura.
Discussione
L'uso di immunoistochimica tradizionale per valutare l'espressione della proteina è limitata dalla metodi semiquantitativi soggettivi di analisi 22,23. piattaforme Advance sono stati creati per l'analisi high-throughput di espressione biomarker e la localizzazione. la segmentazione dettagliata di entrambi i tessuti e compartimenti subcellulari consente agli utenti di studiare l'espressione biomarker, la localizzazione, e co-localizzazione con altri marcatori di interesse. In studi precedenti, abbiamo dimostrato l'utilità di IHC e di imaging multispettrale, in particolare quando usato per studiare le proteine localizzate nella stessa 18,20,21 compartimento cellulare. Quando combinato con microarrays tissutali 24, queste tecniche consentono quantificazione più veloce e più obiettivo dell'espressione proteica di quanto consentito dalle analisi manuale da un patologo.
Un problema con l'uso di immunoistochimica per la quantificazione della proteina è irriproducibilità dei risultati a causa della subjecla natura tiva di analisi e differenze insite nella tecnica e reagenti. Un significativo vantaggio di utilizzare piattaforme come l'imaging multispettrale per misurare l'espressione proteica è riproducibilità dei risultati. I dati provenienti da piattaforme di patologia computerizzati correlano altamente con l'analisi manuale da un patologo mentre tornava dati in un formato continuo e riducendo notevolmente il tempo di lavoro, soprattutto quando si lavora con ampio campione dimensioni 12,16,25. Gli studi hanno dimostrato elevata concordanza complessiva nei risultati tra diverse piattaforme multispettrali 14. Inoltre, abbiamo precedentemente dimostrato che i risultati di colorazione sono altamente riproducibili anche quando c'è variabilità nel numero di proteine indagato 12. L'uso di piattaforme patologia automatizzati sia per la colorazione e analisi non eliminerà tutte le discrepanze nei risultati, come quelli derivanti da anticorpi creati verso diversi epitopi, ma queste piattaforme non sostanzialmente ridurre i pregiudizi e irriproducibilità comunemente associato con immunoistochimica.
Ci sono particolari passaggi all'interno del protocollo che sono essenziali per la restituzione risultati accurati e riproducibili. disegno sperimentale appropriato attraverso la selezione di proteine da inserire come tessuto o marcatori subcellulari è importante per la segmentazione accurata. Quando si esegue l'analisi positività, la selezione di una soglia inferiore per colorazione positiva ha un grande effetto sui risultati finali. Mentre la scelta di una soglia per "on / off" proteine come fattori di trascrizione nucleare è piuttosto semplice, trovando una soglia per le proteine più eterogeneo espresse è difficile. Questo dovrebbe idealmente essere eseguita in collaborazione con un patologo genitourinario bordo certificata o come un punteggio di media tra più osservatori di trovare la soglia ideale per l'analisi.
È importante riconoscere alcune limitazioni delle attuali versioni di questa tecnologia. quando deaffinamento vano citoplasma, tre approcci possono essere utilizzati: (1) colorazione con un marcatore specifico citoplasma (2) colorazione con un marcatore specifico membrana e utilizzando il nucleo a membrana distanza marcatore come il citoplasma, e (3) usando un approccio disegno per definire manualmente i confini del citoplasma in relazione al nucleo. Dalla nostra esperienza, utilizzando un marcatore specifico membrana è la tecnica più accurata. L'approccio disegno manuale è generalmente precisi se i nuclei sono in posizione centrale o se il biomarker di interesse è distribuito in modo uniforme in tutto il citoplasma. Accuratamente definire il citoplasma delle cellule stromali come fibroblasti e cellule muscolari lisce resta difficile e dovrebbe essere preso in considerazione quando si progetta un esperimento.
Un'altra limitazione della tecnologia è la dipendenza da nuclei per la segmentazione delle cellule. Se piano di sezione esclude il nucleo di una cella particolare, questa cella non sarà incluso nell'analisi. Se non c'ècitoplasma visibile tra i nuclei o ciuffi di nuclei adiacenti, questi sono spesso riconosciuto come una grossa protuberanza nucleare, piuttosto che i nuclei distinti. In generale, ematossilina di contrasto è normalmente sufficiente per la segmentazione nucleari accettabili, e la manipolazione delle impostazioni del software, come soglia massima per le dimensioni nucleo può risolvere la maggior parte dei problemi con la segmentazione nucleare.
Una limitazione finale di piattaforme automatizzate patologia è la segmentazione affidabile di tessuti derivanti da cattiva progettazione sperimentale. L'importanza di scegliere appropriati biomarcatori di tessuti e cellule per rispondere alla domanda di interessi non può essere sopravvalutata. Come abbiamo dimostrato nei nostri risultati rappresentativi, un indicatore epiteliale o stromale che cambia in maniera significativa espressione tra gli stati di malattia può porre problemi durante la creazione di un algoritmo per la segmentazione dei tessuti. Vale la pena di notare che ci sono opzioni alternative quando difficile analisi come questo si pone. Ad esempio, imag individualees possono essere analizzati disegnando manualmente tutti compartimenti, piuttosto che applicando un algoritmo da un insieme di formazione di immagini. Ciò fornisce il vantaggio di segmentazione è perfettamente in linea con quanto desidera l'utente, ma introduce anche soggettività e può aumentare significativamente il tempo richiesto per completare l'analisi. disegno sperimentale appropriato è il modo più semplice per accelerare l'analisi e massimizzare l'accuratezza dei tessuti e delle cellule di segmentazione.
Questa tecnologia e protocollo hanno molte applicazioni future. Numerosi biomarcatori per particolari stati di malattia sono stati identificati, ma non convalidato. Alta produttività analisi oggettiva con le piattaforme multispettrale facilita la validazione di questi marcatori. Inoltre, la valutazione di espressione e di co-localizzazione di più proteine in grado di fornire approfondimenti di vie di segnalazione mal comprese. Indubbiamente, riducendo la soggettività intrinseca associata con l'analisi di immunoistochimica staining è utile per comprendere l'espressione e la localizzazione di una vasta gamma di marcatori proteici.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori ringraziano l'Università del Wisconsin traslazionale Initiative di ricerca nel laboratorio di patologia, in parte sostenuto dal Dipartimento UW di Patologia e Medicina di Laboratorio e UWCCC concessione P30 CA014520, per l'utilizzo delle sue strutture e servizi.
Materiali
| Name | Company | Catalog Number | Comments |
| Xylene | Fisher Chemical | X3F1GAL | NFPA rating: Health – 2, Fire – 3 , Reactivity – 0 |
| Ethyl Alcohol-200 proof | Fisher Scientific | 4355223 | NFPA rating: Health – 0, Fire – 3 , Reactivity – 0 |
| Tris Base | Fisher Scientific | BP152-500 | NFPA rating: Health – 2, Fire – 0 , Reactivity – 0 |
| Tris Hydroxymethyl aminomethane HCl | Fisher Scientific | BP153-1 | NFPA rating: Health – 2, Fire – 0 , Reactivity – 0 |
| Tween 20 | Chem-Impex | 1512 | NFPA rating: Health – 0, Fire – 1 , Reactivity – 0 |
| Phosphate-buffered saline | Fisher Scientific | BP2944-100 | NFPA rating: Health – 1, Fire – 0 , Reactivity – 0 |
| Peroxidazed | Biocare Medical | PX968 | Avoid contact with skin and eyes. May cause skin irritation and eye damage. |
| Estrogen Receptor alpha | Thermo Fisher Scientific-Labvision | RM9101 | Not classified as hazardous |
| Androgen Receptor | SCBT | sc-816 | Not classified as hazardous |
| CD147 | Biodesign | P87535M | Not classified as hazardous |
| E-cadherin | Dako | M3612 | Not classified as hazardous |
| Renoir Red Andibody Diluent | Biocare Medical | PD904 | It is specially designed to work with Tris-based antibodies |
| DeCloaking Chamber | Biocare Medical | Model DC2002 | Take normal precautions for using a pressure cooker |
| Barrier pen-Immuno Edge | Vector Labs | H-4000 | |
| Denaturing Kit-Elution step | Biocare Medical | DNS001H | Not classified as hazardous |
| Mach 2 Goat anti-Rabbit HRP Polymer | Biocare Medical | RHRP520 | Not classified as hazardous |
| Mach 2 Goat anti-Rabbit AP Polymer | Biocare Medical | RALP525 | Not classified as hazardous |
| Mach 2 Goat anti-Mouse HRP Polymer | Biocare Medical | M3M530 | Not classified as hazardous |
| Betazoid DAB Chromogen Kit | Biocare Medical | BDB2004 | 1. DAB is known to be a suspected carcinogen. 2. Do not expose DAB components to strong light or direct sunlight. 3. Wear appropriate personal protective equipment and clothing. 4. DAB may cause sensitization of skin. Avoid contact with skin and eyes. 5. Observe all federal, state and local environmental regarding disposal |
| Warp Red Chromogen Kit | Biocare Medical | WR806 | Corrosive. Acid that may cause skin irritation or eye damage. |
| Vina Green Chromogen Kit | Biocare Medical | BRR807 | Harmful if swallowed |
| Bajoran Purple Chromogen Kit | Biocare Medical | BJP807 | Flammable liquid. Keep away from heat, flames and sparks. Harmful by ingestion or absorption. Avoid contact with skin or eyes, and avoid inhalation. |
| Cat Hematoxylin | Biocare Medical | CATHE | Purple solution with a mild acetic acid (vinegar) scent. May be irritating to skin or eyes. Avoid contact with skin and eyes. Avoid ingestion. |
| XYL Mounting Media | Richard Allen | 8312-4 | NFPA rating: Health – 2, Fire – 3 , Reactivity – 0 |
| 1.5 Coverslips | Fisher Brand | 22266858 | Sharp edges |
| Incubation (Humidity)Chamber | obsolete | obsolete | Multiple vendors available |
| Convection Oven | Stabil- Therm | C-4008-Q | |
| Background Punisher Blocking Reagent | Biocare Medical | BP974 | This product is not classified as hazardous. |
| inForm software | PerkinElmer | CLS135781 | Primary multispectral imaging software used in manuscript |
| Nuance software | PerkinElmer | Nuance EX | Software used for making spectral libraries within manuscript |
| Vectra microscope and slide scanner | PerkinElmer | VECTRA | Automated slide scanner and microscope for obtaining IM3 image cubes |
Riferimenti
- Valdman, A., et al. Expression of redox pathway enzymes in human prostatic tissue. Anal Quant Cytol Histol. 31 (6), 367-374 (2009).
- Rimm, D. L., Camp, R. L., Charette, L. A., Olsen, D. A., Provost, E. Amplification of tissue by construction of tissue microarrays. Exp Mol Pathol. 70 (3), 255-264 (2001).
- Jonmarker, S., et al. Expression of PDX-1 in prostate cancer, prostatic intraepithelial neoplasia and benign prostatic tissue. APMIS. 116 (6), 491-498 (2008).
- McCarty, K. S., Miller, L. S., Cox, E. B., Konrath, J., McCarty, K. S. Sr Estrogen receptor analyses. Correlation of biochemical and immunohistochemical methods using monoclonal antireceptor antibodies. Arch Pathol Lab Med. 109 (8), 716-721 (1985).
- Volante, M., et al. Somatostatin receptor type 2A immunohistochemistry in neuroendocrine tumors: a proposal of scoring system correlated with somatostatin receptor scintigraphy. Mod Pathol. 20 (11), 1172-1182 (2007).
- Muris, J. J., et al. Immunohistochemical profiling of caspase signaling pathways predicts clinical response to chemotherapy in primary nodal diffuse large B-cell lymphomas. Blood. 105 (7), 2916-2923 (2005).
- Jaraj, S. J., et al. Intra- and interobserver reproducibility of interpretation of immunohistochemical stains of prostate cancer. Virchows Arch. 455 (4), 375-381 (2009).
- Nakane, P. K., Pierce, G. B. Enzyme-labeled antibodies: preparation and application for the localization of antigens. J Histochem Cytochem. 14 (12), 929-931 (1966).
- Peters, T. J. Investigation of tissue organelles by a combination of analytical subcellular fractionation and enzymic microanalysis: a new approach to pathology. J Clin Pathol. 34 (1), 1-12 (1981).
- Emmert-Buck, M. R., et al. Laser Capture Microdissection. Science. 274 (5289), 998-1001 (1996).
- Waters, J. C. Accuracy and precision in quantitative fluorescence microscopy. J Cell Biol. 185 (7), 1135-1148 (2009).
- Huang, W., Hennrick, K., Drew, S. A colorful future of quantitative pathology: validation of Vectra technology using chromogenic multiplexed immunohistochemistry and prostate tissue microarrays. Hum Pathol. 44 (1), 29-38 (2013).
- Rimm, D. L. C-Path: A Watson-Like Visit to the Pathology Lab. Science Translational Medicine. 3 (108), (2011).
- Fiore, C., et al. Utility of multispectral imaging in automated quantitative scoring of immunohistochemistry. J Clin Pathol. 65 (6), 496-502 (2012).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: A review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Rizzardi, A. E., et al. Quantitative comparison of immunohistochemical staining measured by digital image analysis versus pathologist visual scoring. Diagn Pathol. 7, 42(2012).
- Bauman, T. M., et al. Characterization of fibrillar collagens and extracellular matrix of glandular benign prostatic hyperplasia nodules. PLoS One. 9 (10), e109102(2014).
- Bauman, T. M., et al. Beta-catenin is elevated in human benign prostatic hyperplasia specimens compared to histologically normal prostate tissue. Am J Clin Exp Urol. 2 (4), 313-322 (2014).
- Bauman, T. M., Ewald, J. A., Huang, W., Ricke, W. A. CD147 expression predicts biochemical recurrence after prostatectomy independent of histologic and pathologic features. BMC Cancer. 15 (1), 549(2015).
- Bauman, T. M., et al. Finasteride treatment alters tissue specific androgen receptor expression in prostate tissues. Prostate. 74 (9), 923-932 (2014).
- Nicholson, T. M., Sehgal, P. D., Drew, S. A., Huang, W., Ricke, W. A. Sex steroid receptor expression and localization in benign prostatic hyperplasia varies with tissue compartment. Differentiation. 85 (4-5), 140-149 (2013).
- Taylor, C. R., Levenson, R. M. Quantification of immunohistochemistry--issues concerning methods, utility and semiquantitative assessment II. Histopathology. 49 (4), 411-424 (2006).
- Matos, L. L., Trufelli, D. C., de Matos, M. G., da Silva Pinhal, M. A. Immunohistochemistry as an important tool in biomarkers detection and clinical practice. Biomark Insights. 5, 9-20 (2010).
- Kononen, J., et al. Tissue microarrays for high-throughput molecular profiling of tumor specimens. Nat Med. 4 (7), 844-847 (1998).
- Ong, C. W., et al. Computer-assisted pathological immunohistochemistry scoring is more time-effective than conventional scoring, but provides no analytical advantage. Histopathology. 56 (4), 523-529 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon