Method Article
Die Quantifizierung der Proteinexpression und Co-Lokalisation Mit Multiplexed immunhistochemischen Anfärbung und Multispektraltechnik
In diesem Artikel
Zusammenfassung
Immunohistochemistry is a powerful lab technique for evaluating protein localization and expression within tissues. Current semi-automated methods for quantitation introduce subjectivity and often create irreproducible results. Herein, we describe methods for multiplexed immunohistochemistry and objective quantitation of protein expression and co-localization using multispectral imaging.
Zusammenfassung
Immunohistochemistry is a commonly used clinical and research lab detection technique for investigating protein expression and localization within tissues. Many semi-quantitative systems have been developed for scoring expression using immunohistochemistry, but inherent subjectivity limits reproducibility and accuracy of results. Furthermore, the investigation of spatially overlapping biomarkers such as nuclear transcription factors is difficult with current immunohistochemistry techniques. We have developed and optimized a system for simultaneous investigation of multiple proteins using high throughput methods of multiplexed immunohistochemistry and multispectral imaging. Multiplexed immunohistochemistry is performed by sequential application of primary antibodies with secondary antibodies conjugated to horseradish peroxidase or alkaline phosphatase. Different chromogens are used to detect each protein of interest. Stained slides are loaded into an automated slide scanner and a protocol is created for automated image acquisition. A spectral library is created by staining a set of slides with a single chromogen on each. A subset of representative stained images are imported into multispectral imaging software and an algorithm for distinguishing tissue type is created by defining tissue compartments on images. Subcellular compartments are segmented by using hematoxylin counterstain and adjusting the intrinsic algorithm. Thresholding is applied to determine positivity and protein co-localization. The final algorithm is then applied to the entire set of tissues. Resulting data allows the user to evaluate protein expression based on tissue type (ex. epithelia vs. stroma) and subcellular compartment (nucleus vs. cytoplasm vs. plasma membrane). Co-localization analysis allows for investigation of double-positive, double-negative, and single-positive cell types. Combining multispectral imaging with multiplexed immunohistochemistry and automated image acquisition is an objective, high-throughput method for investigation of biomarkers within tissues.
Einleitung
Immunhistochemie (IHC) ist eine Standardlaborverfahren zum Nachweis von Protein in Gewebe und IHC ist immer noch weit verbreitet in Forschung und Diagnose der Pathologie verwendet. Die Auswertung der IHC-Färbung ist oft semi-quantitative, Potentialvorspannung in die Interpretation der Ergebnisse einzuführen. Viele semi-quantitative Ansätze wurden die 1-4 sowohl Färbeintensität und Färbung Ausmaß in die endgültige Diagnose integrieren entwickelt. Andere Systeme sind Scoring - Intensität und subzellulären Ort, um eine bessere Ausdruck 5 lokalisieren. Der Einbau von Durchschnittswerte von mehreren Zuschauern wird häufig genutzt , um die Auswirkungen der einzelnen Betrachter Bias 6 zu minimieren. Trotz dieser Bemühungen bei der Analyse, Subjektivität noch bleibt, vor allem bei der Bewertung des Ausmaßes der 7 - Färbung. Protokoll Standardisierung und zur Minimierung der Subjektivität von menschlichen Input ist von größter Bedeutung genaue, reproduzierbare IHC Ergebnisse zu schaffen.
Inhalt "> Es gibt andere Optionen neben IHC für die Proteinexpression in Geweben zu bestimmen. Im Rahmen des Forschungs Einstellung hat Immunhistochemie traditionell als Mittel angesehen Proteinlokalisierung 8, während andere Techniken wie Immunoblotting angesehen werden als Goldstandard für die Untersuchung von Protein - Expression zu untersuchen , . Bestimmung Gewebe oder Zellkompartiment spezifische Expression ist schwierig , ohne fortgeschrittene Techniken wie Zell Fraktionierung oder Laser Mikrodissektion 9,10 enthält. Die Verwendung von fluoreszierenden Antikörpern auf Gewebeschnitten einen vernünftigen Kompromiß bietet, aber aufgrund NADPH Hintergrund - Autofluoreszenz, Lipofuscine, retikuläre Fasern, Kollagen und Elastin kann 11 genaue Quantifizierung erschweren.Automatisierte Rechen Pathologie Plattformen sind eine vielversprechende Richtung für mehr objektive Quantifizierung der Pathologie Färbung 12-15. Die Kombination von multispektrale Bild mit Gewebe-Mikroarrayserleichtert Hochdurchsatz-Analyse der Proteinexpression in großen Stichprobengrößen. Mit diesen Techniken Analyse von Protein - Co-Lokalisation, Färbungs Heterogenität und Gewebe und subzelluläre Lokalisierung ist möglich , während im Wesentlichen sowohl inhärent vorspannt und die Zeit , die zur Analyse zu reduzieren, während die Daten in einem kontinuierlichen und nicht kategorische Format 16 zurückkehrt. Daher war das Ziel dieser Studie zur Durchführung einer Multiplex-Immunhistochemie mit Analyse die Nützlichkeit und die Methodik zu veranschaulichen, multispektrale Abbildungssoftware.
Dieses Protokoll wird für die manuelle, multiplex immunhistochemische Färbung eines einzigen Gewebeschnitt Schlitten mit vier optimierte monoklonalen Antikörpern geschrieben. Als ein repräsentatives Experiment werden Kern anti-Kaninchen-Östrogenrezeptor alpha (ER & agr;) und Androgen-Rezeptor (AR) gemultiplext mit membrangebundenen anti-Maus-CD147 und membrangebundenen anti-Maus-E-Cadherin. Jeder Antikörper der Wahl kann Substituted für die aufgeführten Antikörper hier, aber jede Kombination von Antikörpern erfordert getrennte Optimierung. Vorbehandlung für alle Antikörper müssen identisch sein. Die AR und CD147-Antikörper sollten einzeln und dann als Cocktail optimiert werden. Jeder Antikörper erkannt wird ein Biotin freie Polymersystems und eines von 4 einzigartigen Chromogene verwendet wird.
Protokoll
HINWEIS: Das Protokoll beschreibt hierin Färbung und Analyse eines Gewebemikroarray (TMA), die zuvor 12,17,18 beschrieben. Das 4 um dicke TMA Abschnitt wurde aus einem Paraffinblock mit einem Standard-Mikrotom erhalten.
HINWEIS: Eine spektrale Bibliothek für die 4 Chromogene und counterstain sollte für die Bild Quantifizierung erstellt werden. Um dies zu tun, sollte das optimierte Protokoll für jeden einzelnen Antikörper mit einem Antikörper pro Folie ausgeführt werden, abzüglich der letzten counterstain. Eine fünfte Folie sollte mit Hämatoxylin gefärbt werden, um die 5 Bilder zu erzeugen, benötigt, um die spektrale Bibliothek zu erstellen.
1. Multiplex Immunhistochemie
- Bake Dias in einem 60 ° C Ofen für 20 min. Mit Hilfe einer chemischen Abzugshaube, tauchen Folie in 100% Xylol. Wiederholen Sie dies für zwei Änderungen Xylol. Tauchen slide in 100% Ethanol für 5 min. Wiederholen Sie mit frischem 100% Ethanol.
- Tauchen slide in 95% Ethanol für 3 min. Wiederholen Sie mit frischem95% Ethanol. Tauchen slide in 70% Ethanol für 3 min. Tauchen Folie in destilliertem Wasser für 3 min. Wiederholen Sie mit frischem destilliertem Wasser. Tippen Schlitten vertikal Wasser aus dem Schlitten zu entleeren. Bewerben 200 ul ready-to-use endogenen Peroxidase-Blocker für 5 min.
- Abfluss-Peroxidase-Blocker von Folie und tauchen in 45 ml ready-to-use Abrufpuffer (pH-Wert von weniger als 7,0). Den Objektträger Behälter in einem Dampfkochtopf und Startprogramm auf 124 ° C für 4 min. Erlauben Dampfkochtopf 20 min abkühlen. vor dem Öffnen.
- Entfernen Sie Behälter des Abrufs Puffer und ermöglichen weitere 10 Minuten abkühlen lassen. Spülen Folie in destilliertem Wasser für 10 min. Lassen Sie das Wasser aus dem Schlitten und beschreiben sorgfältig das Gewebe auf dem Objektträger mit einer hydrophoben Barriere Stift.
- In 200 ul phosphatgepufferter Salzlösung (PBS) .Transfer Folie in eine flache Schale mit Deckel (Inkubationskammer) mit feuchter Gaze.
- Bereiten Sie 1 Liter 1x Tris-Kochsalzlösung mit Tween-20 (TBST) um 50 Verdünnung ml 20x Tris-gepufferter Salzlösung, enthaltend 1% Tween-20 und 950 ml destilliertes Wasser.
- Ablassen PBS von Folie und anwenden 200 ul 1x TBST. Inkubieren 2 min. Ablassen TBST von Folie und Rück Folie Inkubationskammer. Bewerben 200 ul ready-to-use universellen Protein Block, in der Nähe Kammer und Inkubation für 7 min.
- Ablassen Protein blockieren und Rück Folie Inkubationskammer. Verdünnte Anti-ER & agr; Antikörper 1: 400 durch Zugabe von 0,6 & mgr; l Antikörper zu 239,4 & mgr; l Antikörperverdünnungsmittel. Bewerben 200 ul verdünnten Antikörpers und in der Nähe Kammer zu gleiten.
- Inkubieren Rutsche bei Raumtemperatur für 2 Stunden. Lassen Sie Antikörper von Folie und Spülen mit TBST gefolgt von Eintauchen für 5 min in TBST.
- Ablassen TBST von Folie und Rück Folie Inkubationskammer. Bewerben 200 ul ready-to-use Ziegen-Anti-Kaninchen-Meerrettich-Peroxidase-Polymer und in der Nähe Kammer. Inkubieren für 30 min. bei Raumtemperatur. Rinse Rutsche mit TBST und Platz Rutsche in Behälter von TBST für 5 min.
- Bereiten braun Meerrettich-Peroxidase-3,3'-Diaminobenzidin (HRP-DAB) Chromogen durch Zugabe von 8 & mgr; l von DAB Chromogen zu 250 ul DAB Substratpuffer und gut mischen.
- Ablassen TBST von Folie, Rückkehr Folie Inkubationskammer und anwenden 200 ul braun HRP-DAB Chromogen für 6 min. Spülen Sie die Folie mit destilliertem Wasser und Platz Rutsche in einen Behälter mit destilliertem Wasser.
- Mischen Sie die Denaturierungslösung durch Kombinieren von 50 & mgr; l des ersten Reagens Elution mit 150 & mgr; l des zweiten Reagens Puffer. Lassen Sie das Wasser aus dem Schieber und gelten verdünnte Denaturierungslösung auf den Objektträger für 3 min. bei Raumtemperatur. Dekantieren und Spülreagens mit TBST denaturieren. Tauchen Folie in TBST für 5 min.
- Bereiten Sie AR / CD147-Antikörper-Cocktail um 4,2 ul anti-AR-Antikörper und 2,8 ul anti-CD147-Antikörper mit 203 & mgr; l Antikörperverdünnung verdünnt wird. Ablassen TBST aus der Folie zurückkehren Folie Inkubationskammer und anwenden 200 ul AR / CD147 cocktail auf der Folie. Schließen Kammer und für 1 Stunde bei Raumtemperatur inkubieren. Rinse Objektträger mit TBST und für 5 min in TBST eintauchen.
- Ablassen TBST, Rückkehr Folie Inkubationskammer und anwenden 200 ul ready-to-use Ziegen-Anti-Kaninchen-alkalische Phosphatase-Polymer. Schließen Kammer und incubate Rutsche für 30 min. bei Raumtemperatur. Rinse Objektträger mit TBST und für 5 min in TBST eintauchen.
- Bereiten rote alkalische Phosphatase Chromogen durch Zugabe von 3,2 ul roten Chromogen bis 250 & mgr; l roten Chromogen-Puffer und gut mischen.
- Ablassen TBST von Folie und Rück Folie Inkubationskammer. Bewerben 200 ul verdünnt rot alkalische Phosphatase Chromogen zu der Folie, in der Nähe Kammer und Inkubation für 7 min.
- Ablassen Chromogen und gründlich spülen Objektträger mit destilliertem Wasser. Den Objektträger in einen Behälter mit destilliertem Wasser für 5 min. Lassen Sie das Wasser aus der Folie gründlich mit TBST und zurück zur Inkubationskammer. Gelten 200 ul Ziegen-Anti-Maus-HRP-Polymer auf den Objektträger und close Kammer. Inkubieren für 30 min. bei Raumtemperatur.
- Ablassen Polymer aus dem Schlitten und Spülen mit TBST. Den Objektträger in TBST für 5 min. Bereiten 200 ul purple HRP-verknüpften Chromogen durch Zugabe von 3,2 & mgr; l des stabilisierenden Reagenzes zu 250 ul des Puffers und gut mischen. In 3,2 ul lila Chromogen und gut mischen, dann 3,2 ul des Wasserstoffperoxid-Reagens und gut mischen.
- Ablassen TBST abrutschen und anwenden 200 ul vorbereitet lila Chromogen gleiten und für 7 min inkubiert. bei Raumtemperatur. Spülen Sie Objektträger mit destilliertem Wasser und Platz Rutsche in einen Behälter mit destilliertem Wasser für 5 min.
- Mischen Sie die Denaturierungslösung durch Mischen von 50 & mgr; l des ersten Reagens Elution mit 150 & mgr; l des zweiten Reagens Puffer. Lassen Sie das Wasser aus dem Schieber und gelten verdünnte Denaturierungslösung auf den Objektträger für 3 min. bei Raumtemperatur.
- Spülreagens Denaturierung off mit TBST. Den Objektträger in Behälter mit TBST für 5 min. Verdünnte Anti-E-Cadherin-Antikörperbis 1: 200 1,1 ul Antikörper zu 218,9 ul Antikörperverdünnung durch Zugabe. Lassen Sie das TBST aus der Folie Rückkehr Folie Inkubationskammer und anwenden 200 ul von E-Cadherin auf der Folie. Schließen Kammer und 30 min inkubiert. bei Raumtemperatur.
- Rinse Rutsche mit TBST und Platz Rutsche in Behälter von TBST für 5 min. Lassen Sie das TBST von Folie, kehren Folie Inkubationskammer und anwenden 200 ul Ziegen-Anti-Maus-HRP-Polymer. für 30 min bei Raumtemperatur inkubieren. Rinse Rutsche mit TBST und Platz Rutsche in Behälter von TBST für 5 min.
- Bereiten schwarz HRP-verknüpften Chromogen durch Zugabe von 8 ul Chromogen zu 250 & mgr; l Puffer. Bewerben 200 ul Chromogen gleiten und für 2 min zu entwickeln. Spülen Sie Objektträger mit destilliertem Wasser und tauchen Sie in Behälter mit destilliertem Wasser.
- Verdünnen Sie Hämatoxylin durch Zugabe von 40 & mgr; l Hämatoxylin bis 200 & mgr; l destilliertes Wasser. Lassen Sie das Wasser aus Rutsche und anwenden 200 ul verdünnt Hämatoxylin für 30 Sekunden zu gleiten. Rinse Rutschegründlich fließendem Leitungswasser für 2 min., spülen Sie dann in destilliertem Wasser für 30 Sekunden. Den Objektträger in einem 60 ° C Ofen für 15-30 min.
- Tauchen Folie in frisch 100% Xylol vorbereitet für 30 Sekunden, wischen überschüssiges Xylol aus und gibt einen Tropfen festen Aufschrauben Medien. Deckglas mit einem Nr 1.5 Deck.
2. Automatische Bildaufnahme und Analyse
- Legen gefärbten Folien auf einem automatisierten Dia-Scanner. Basierend auf dem Durchmesser, Trennung, und die Anzahl der TMA-Kerne, schaffen den Hersteller ein automatisiertes Scanprotokoll Bedienungsanleitung nach.
- Offene Multispektral-Imaging-Software mit einer spektralen Bibliothek von Steuerschiebern gefärbt mit einzelnen Chromogene und der Hämatoxylin gefärbt Schlitten zu bauen. Öffnen Sie ein Bild Würfel von einem Steuerschieber erworben und wählen Sie vier vor fünf positiv gefärbte Bereiche optisch dass Chromogen definieren. Wiederholen Sie mit Bild Würfel von anderen Steuerschieber bis eine vollständige Spektralbibliothek representing alle Chromogene erstellt wird, und dann die spektrale Bibliothek speichern.
- Beginnen Sie ein neues Projekt in Multispektral-Imaging-Software. Wählen Sie "Multispektrale (.im3)" für das Bildformat-Option und "Hellfeld" für die Probe-Format. Konfigurieren Sie zunächst das Projekt, indem die gewünschten Optionen wählen: "Segment Tissue", "Finden Eigenschaften", "Phänotypisierung", "Score" und "Export". An diesem Punkt kann die Bildauflösung geändert werden die Analysezeit zu beschleunigen, falls gewünscht.
3. Die Gewebesegmentierung
- Importieren Sie die zuvor Spektralbibliothek erstellt und wählen Sie alle Chromogene werden in der Analyse berücksichtigt.
- Bild öffnen Würfel in den Trainingsdaten enthalten sein, nach der "Bild öffnen Cube" Option auswählen. Zur Ausbildung Genauigkeit, wählen Sie mindestens 18% der Gesamtzahl der Bilder zu analysiere gewährleisten. Wählen Sie die Bilder, die alle Krankheitszustände darstellen Segmentierung Genauigkeit zu erhöhen. Dieser Teil der Bilder is rief den Trainingssatz von Bildern.
- Weißabgleich Bilder in der Trainingssatz durch die Pipette Werkzeug auswählen und einen Bereich in einem Bild auswählen, die weiß ist.
- Wählen Sie "Erweitert", um Gewebe Segmentierung zu bewegen. Das "Tissue Kategorien" Panel verwenden Gewebearten zu wählen , analysiert (dh "Gewebe" und "Nicht-Gewebe") werden. Für eine genauere Protein Gewebelokalisierung können mehrere Gewebe Kategorien verwendet werden (dh. "Epithelien", "Stroma" und "Nicht-Gewebe").
- Beginnen Sie den Algorithmus und die Definition Gewebe Kategorien Schaffung von Gruppen von Zellen innerhalb Trainingsbilder zeichnen sich um. Wenn sie mit einer Gewebe Kategorie fertig sind, wiederholen Sie für andere Gewebekategorien. Seien Sie sicher, dass Gruppen von Zellen innerhalb von Bildern zu wählen, die von diesem Gewebe Kategorie Typ charakteristisch sind.
- Wiederholen Sie diesen Vorgang für alle Bilder im Trainingssatz von Bildern.
- Wählen Sie Komponenten (Chromogene) werden in trai enthaltenning für das "Tissue Segmentierer". Wenn die Analyse von Hell- Immunhistochemie Bilder durchgeführt wird, werden alle Komponenten, die normalerweise in der Ausbildung enthalten. Fügen Sie reichlich negative Färbung Bilder in der Ausbildung gesetzt Bias bei diesem Schritt zu vermeiden.
- Wählen Sie einen geeigneten "Pattern Scale" für das Gewebe segmenter trainieren. Unter 20-facher Vergrößerung, ist eine große Muster Maßstab typischerweise geeignet. Wenn sie mit Gewebe mit einer feinen Architektur bei starker Vergrößerung arbeiten, ist eine kleinere Muster Maßstab besser geeignet.
- Wählen Sie den "Train Tissue Segmentierer" Taste zum Starten des Gewebes segmenter trainieren. Beachten Sie ein Pop-up-Fenster angezeigt wird Genauigkeit reflektieren das Verhältnis von Pixeln in Ausbildung Regionen, die richtig klassifiziert werden.
HINWEIS: Die Software kontinuierlich die Genauigkeit des Algorithmus zu verbessern versucht, bis sie manuell gestoppt wird. Wir haben früher , dass die Ausbildung auf 18% der Bilder ergibt in 97% Training Genauigkeit 12 demonstriert. - Nachdem das Gewebe segmenter trainiert wird, ein entsprechendes Segment Auflösung wählen. Segmentauflösung entspricht der Zeit zu segmentieren Bilder erforderlich, mit grober Auflösung erfordern weniger Zeit und feine Auflösung mehr Zeit erfordern.
- Segment die gesamte Trainingssatz von Bildern durch "Segment Bilder" klicken. Lassen Sie die Software ausreichend Zeit, um den Algorithmus für alle Trainingsbilder anzuwenden. Wenn Sie fertig sind, überprüfen Sie die Ausbildung so einstellen, dass falsch klassifizierte Gewebe mit dem aktuellen Trainingsalgorithmus zu finden.
- Zur Feinabstimmung der Gewebesegmentierungsprozess, hinzufügen, bearbeiten oder Gewebetrainingsbereiche zu entfernen. Verwenden Sie die "Beschnitträndern" Option, wenn Pixel an den Rändern einer Gewebe Kategorie in die andere erstrecken. Wenn kleine Gruppen von Pixeln jedoch nicht korrekt angezeigt werden, stellen Sie die Schwelle auf die minimale Segmentgröße.
- Wenn die Änderungen abgeschlossen sind, wählen Sie "Segment Images", wird dies das Gewebe segmenter umschulen einen neuen Algorithmus für die Gewebe Segmentierung zu schaffen. Der bisherige Algorithmusautomatisch gespeichert und kann zurückgegeben werden, wenn nötig.
- Wenn zuversichtlich, mit Gewebesegmentierungsalgorithmus Ergebnisse, vorab zu Zelle Segmentierung durch den "Erweiterte" Taste auswählen.
4. Zellsegmentierung
- Wählen Sie die subzellulärer Kompartimente werden in Zelle Segmentierung enthalten. Stellen Sie sicher, dass "Nuclei" ist bereits ausgewählt, um "Zytoplasma" oder "Membrane".
ANMERKUNG: "Membran" Segmentierung sollte nur gewählt werden, wenn ein membranspezifische Proteinmarker in IHC enthalten war, wie beispielsweise E-Cadherin. - Im Rahmen der "Nuclei" Registerkarte, gibt es mehrere Optionen. Beginnen Sie mit der entsprechenden Einstellungen für die nukleare Segmentierung Auswahl (siehe Schritt 4.3). Wählen Sie, ob einzelne oder alle Gewebe Kategorien werden bei der Segmentierung einbezogen werden.
- Wählen Sie einen Ansatz für die nukleare Segmentierung
- Wählen Sie die counterstain "Pixel-Based (Threshold)" Ansatz für ein vereinfachtes Verfahren zum Erhalt von gutenErgebnisse ohne viel über Bildverarbeitungseinstellungen zu kennen.
HINWEIS: Dies ist oft eine gute erste Wahl. Wenn ein Ansatz der Auswahl "Pixel-Based (Threshold)" ist besser geeignet, wenn es eine zuverlässige Kerngegenfärbung ist, während "objektbasierte" verwendet werden sollte, wenn es einen Mangel an einheitlichen Kern counterstain ist. - Wählen Sie die "Pixel-basierte (Threshold)" Ansatz, wenn es eine zuverlässige Kern counterstain ist.
HINWEIS: Dieser Ansatz ist rein pixelbasierte. Dieser Ansatz kann auch für andere Bildanalyseanforderungen verwendet werden, die mit einem einfachen Schwellwert, wie beispielsweise Erfassen aller Pixel innerhalb eines Gewebes Kategorie, die positive Färbung für eine IHC stain befriedigt werden kann. - Wählen Sie das Objekt-basierte Ansatz, wenn die Kern counterstain nicht konsistent und spezifische Färbung der Kern Objekte liefert, und erweiterte morphometry basierte Ansätze werden benötigt, um Kerne zu erkennen.
- Wählen Sie "Komponenten für Kern Segmentation", "NuclOhrgröße "und" Clean-up ", wenn der Benutzer wünscht, die weitere Kerne Segmentierungseinstellungen definieren.
- Wählen Sie die counterstain "Pixel-Based (Threshold)" Ansatz für ein vereinfachtes Verfahren zum Erhalt von gutenErgebnisse ohne viel über Bildverarbeitungseinstellungen zu kennen.
- Wählen Sie Zytoplasma Formparameter
- Wählen Sie den "Vorwärts" Taste, um Zytoplasma Segmentierung zu bewegen.
HINWEIS: Es gibt mehrere Optionen innerhalb Zytoplasma Segmentierung sind zur Auswahl. Die Option als "innere Distanz zu Keimzelle" ist Pixel basiert. Es spiegelt die Entfernung zwischen der Außenseite des Kerns und der Innenseite des Zytoplasmas. Eine Erhöhung dieses Wertes verringert die Wahrscheinlichkeit, dass die Kern Signal über in das Zytoplasma Fach kreuzt. - Als nächstes wählen Sie "äußere Abstand zum Nukleus".
Hinweis: Dies ist auch Pixel basiert. Der äußere Abstand zum Kern reflektiert den Abstand zwischen der Außenseite des Kerns und der Außenseite der Zelle. Dieser Wert kann groß oder klein genug eingestellt werden, enthalten oder Membran Signale auszuschließen, falls gewünscht. - Als nächstes wählen Sie "Mindestgröße".
HINWEIS: Die Mindestgröße, pixel basiert, reflektiert die minimale Zytoplasma Probengrße in Analyse einbezogen werden. Wenn das Zytoplasma Größe kleiner in einer bestimmten Zelle ist, wie beispielsweise, wenn die Zellen miteinander dicht gepackt sind, wird diese Zytoplasma Segmentierung von der Analyse ausgeschlossen. - Wählen Sie die nächste Option, "Component" mit "Primär", "Sekundär", und wählen Sie "Tertiär" als sekundäre Optionen.
HINWEIS: Hier ein ein bestimmtes Zytoplasma Marker können. Ähnlich wie bei der Kernsegmentierung, wenn eine bestimmte cytoplasmatische IHC Marker verwendet wurde, kann diese Komponente verwendet werden, um das Zytoplasma zu definieren, durch einen minimalen Schwellenwert zu finden. Dies hilft in das Zytoplasma zu definieren, sondern ist für eine akzeptable Genauigkeit nicht erforderlich. - Der Umzug in die letzte der drei Registerkarten Optionen ist "Membrane". Hier definiert der Benutzer das Membrankompartiment mit einem Protein-Marker. Wählen Sie "Primär", "Sekundär" und / oder "Tertiär" für jede Membran spezifischer Marker verwendet. Stellen Sie "Full Scale OD "eine Mindestschwelle oder eine positive für jede Markierung auf Zellmembranen zu finden.
- Fortsetzung unter der "Membrane" und wählen Sie die Option "Maximale Zellengröße".
Hinweis: Dies ist der Abstand zur Membran Wert in Pixeln, die die maximale Größe der Zellen angibt. Ein Wert von 12 ist in der Regel geeignet für Bilder bei 20facher Vergrößerung. - alle Optionen Nach der Auswahl, wählen Sie "Segment Bild" oder "Segment All". Übernehmen Sie die Einstellungen, um Bilder und beobachten sie "individuell" oder in "Galerie" Modus.
HINWEIS: Passen Sie die Einstellungen und Schwellenwerte bei Bedarf und neu Segment bis zufrieden mit Zellsegmentierungsergebnisse im Trainingssatz von Bildern.
- Wählen Sie den "Vorwärts" Taste, um Zytoplasma Segmentierung zu bewegen.
5. Phänotypisierung von Zellen
HINWEIS: Die genaue Zell Segmentierung ist erforderlich, um eine genaue Zelle Phänotypisierung zu erhalten, und die Phänotypisierung Funktion ist trainierbar.
- Verwenden Sie die Schaltfläche "Hinzufügen &# 34; Taste Phänotypen der "Phänotypen" Liste hinzuzufügen. Benutzer können eine Farbe für den Phänotyp wählen.
- Klicken Sie auf "Bearbeiten Phänotypen" einen Phänotyp einer Zelle zuzuordnen. Klicken Sie auf eine bestimmte Zelle ein Drop-Down-Menü aufzurufen, Benutzer zu ermöglichen, den Phänotyp zu wählen und dann weiterziehen. Identifizieren mindestens fünf (5) Zellen in jedem Phänotyp, um fortzufahren. Auswahl 25 oder mehr Zellen jedes Phänotyps erforderlich optimale Ergebnisse zu erzielen.
6. Scoring IHC und Co-Lokalisierung
- Wählen Sie "Erweitert", um die "Score IHC oder IF" Schritt zu bewegen. Wählen Sie eine "Tissue-Kategorie" punkten.
HINWEIS: Nur eine Gewebekategorie kann während einer bestimmten Analyse erzielt werden, aber wenn scoring Ergebnisse in einem anderen Gewebe Kategorie gewünscht sind, kann die Analyse später mit unterschiedlichen Einstellungen wiederholt werden. - Wählen Sie die gewünschte "Scoring" -Typ.
HINWEIS: Positivität erzeugt zwei Bins (positive und negative) für eine Komponente von Interessefür die Co-Lokalisierung Analyse, während doppelte Positivität verwendet. Weitere Optionen sind 4-Fach, 10-bin, und 50-bin für einen Marker der Wahl analysiert. - Wählen Sie die Zelle "Fach", um in Scoring-Analyse verwendet werden.
- Ähnliche eine Mindestschwelle für die primäre Kernkomponente zu finden, wählen Sie "View Component Data" und Cursor über die Trainingsbilder bewegen, um geeignete optische Dichte Mindestschwellen der Färbung für positive Zellen für die Komponente (n) von Interesse.
- Wenn intensiv gefärbten Zellen ausgeschlossen werden sollen, die Schwelle max auf ein angemessenes Niveau zu senken. Andernfalls verwenden Sie die Auto-Taste, um einen Schwellenwert Maximum für die Analyse zu wählen.
- Wählen Sie Bilder trainieren, um zu punkten Thresholding Werte zu bestätigen.
HINWEIS: Der Prozentsatz der Zellen in einem bestimmten Behälter zurückgeführt werden und kann durch visuelle Inspektion oder manuelle Zählung verglichen werden. Wenn Sie fertig sind, wählen Sie "Erweitert" auf "Export" Schritt fortzufahren.
7. Die Anwendung des Algorithmus und Batch-Analyse
- Testen Sie den Algorithmus durch das Gewebe Segmentierung, Zell Segmentierung erstellt und Scoring-Werte, die durch die Daten für den Trainingssatz zu exportieren. Folgen Sie Anweisungen, um einen neuen Ordner für das Exportverzeichnis zu erstellen. Wählen Sie Bilder und Tabellen erstellt werden und in der Analyse berücksichtigt. Führen Sie die Analyse von "Export für alle" auswählen.
- Die Tische sind in Tab-separierte Textdateien exportiert und können in den meisten Datenanalyseprogrammen geöffnet werden.
- Wenn die Analyse abgeschlossen ist, sehen Zelle Segmentierung und Scoring-Daten für Bilder mit hoher und niedriger Färbung Genauigkeit der Einstellungen zu bewerten.
- Wenn mit den Einstellungen zufrieden sind, wenden Sie den Algorithmus aus dem Trainingssatz von Bildern auf den gesamten Satz von Bildern durch Batch-Analyse erstellt. Klicken Sie auf das "Batch-Analyse" Registerkarte und der geöffnete Algorithmus aus dem aktiven Projekt kopiert werden.
- Wählen Sie einen neuen Exportverzeichnis und auswählen, welche Bilder und tables sind in die Analyse einbezogen werden. Unter der Eingabedateien Option, wählen Sie "Bilder hinzufügen" und wählen Sie alle Bilder im Batch-Analyse einbezogen werden.
- Führen Sie die Analyse durch die Option "Ausführen" auswählen. Wie bei der Analyse des Trainingssatzes wird dieser Schritt eine variable Menge an Zeit erfordern, abhängig von bestimmten Einstellungen für die Anzahl der Bilder in die Analyse einbezogen.
- Wenn Sie fertig sind, vorab auf die "Überprüfung / Merge" aus. Die Batch-Verzeichnis standardmäßig auf das Exportverzeichnis aus der Batch-Analyse und kann geändert werden, falls gewünscht. Wählen Sie "Alle einschließen" und wählen Sie "Merge", um Datenblätter mit zusammengefassten Daten für die Analyse erstellen.
8. Analyse der exportierten Daten
- Öffnen exportierten Tabellen in Datenanalyse-Software und entfernen oder Daten Spalten verbergen, die auf die Analyse irrelevant sind, beispielsweise Minimal- und Maximalwerte für jede Komponente. Die Primärdaten in der Analyse verwendet wird, ist in der mittleren optischen Dichte cPALTE für jede Komponente.
- Bewertung exportiert Segmentierung Karten und RAW-Bilder zu bestimmen, von der Analyse ausgeschlossen, welche Proben oder TMA-Kerne werden sollen. Tissue Prozentwerte bei der Ermittlung, ob eine Probe aufgenommen werden soll oder nicht, mit einer beliebigen Cutoff von <5% verwendet, am häufigsten Ausschlusskriterien.
- Entfernen irrelevante Daten oder Zeilen Proben, die aus der Datenanalyse ausgeschlossen werden.
- Verwenden Proteinquantifizierung Datenänderungen in Krankheitszustand zu untersuchen , oder die Beziehung mit klinisch-pathologischen Merkmale der Krankheit 12,18-21.
Ergebnisse
In Figur 1 ist die Ausbildung auf Prostatagewebe zu segmentieren Bilder in Epithel- und Stroma - Abschnitte durchgeführt, zusammen mit dem nicht-Kompartiment. Unter Verwendung der epithelialen Membranmarker E-Cadherin, Zellsegmentierung durchgeführt , den Zellkern, Zytoplasma und Membranabschnitte zu trennen, die in 2 gezeigt.
In einem Experiment haben wir gemultiplexten IHC verwendet , um die Expression und Lokalisation von AR, ER & agr;, E-Cadherin und CD147 zu untersuchen, wie in Figur 3 gezeigt ist . Diese Techniken zu benutzen, können wir Zellen positiv für nukleare Expression sowohl ER & agr; zu erkennen und AR (3B - 3C) trotz überlappende kolorimetrische Signale, wie mit grünen Pfeilen in 3A gezeigt. Wir fanden deutliche Unterschiede im Verhältnis von Doppel positive Stromazellen in verschiedenen Zuständen der prostate Erkrankung (Figur 3D). Wir quantifiziert Zellmembran-spezifische Expression von CD147 von E-Cadherin als Markerprotein (rote Pfeile in 3A) mit, und wir waren die erste Gruppe Membran spezifische CD147 - Expression in Prostatagewebe zu untersuchen. Wir fanden eine signifikante Abnahme der CD147 Expression in Assoziation mit Prostatakrebs Progression (3E) und fand eine wichtige Verbindung mit postoperativen Prognose 19.
Es gibt Fälle, in denen schlechte Experimentdesign zu ungenauen Gewebe Segmentierung führen kann. In 4 ist der Algorithmus auf einen Satz von Geweben angewendet hat nicht exakt Segment Epithel- und Stroma - Kompartimente (4C). In diesem Experiment α-smooth muscle actin (α-SMA) wurde verwendet, um das stromale Kompartiment zu markieren. Da α-SMA und der glatten Muskulatur wird in einigen Tumoren verringert oder verloren (Figure 4A), wurde der Algorithmus durch Gewebe Segmentierung erzeugt (4B) nicht in der Lage genau allein zwischen Epithel- und Stroma - Kompartimente von morphometrischen Daten zu unterscheiden. Es sollte darauf geachtet werden, wenn Protein-Marker für Gewebe oder Zellkompartimenten wählen.

Abbildung 1: Gewebe Segmentation Multispektraltechnik Software. Eine Prostatagewebe-Mikroarray wurde mit Multiplex-Immunhistochemie für den Androgenrezeptor (AR), Östrogen-Rezeptor-alpha (ERa), E-Cadherin und CD147 gefärbt. Eine Reihe von Trainingsbildern wurden in multispektrale Imaging - Software Typen , die Gewebe eingeführt und Krankheitszustände des gesamten Satzes von Bildern (A). Tissue Kategorien wurden einschließlich Stroma (grün) erstellt, Epithelien (rot), und Nicht-Gewebe (blau) und Kategorien von manuell Zeichnung oben definiert wurden,von Trainingsbildern (B). Nach dem Zeichnen auf Trainingsbildern wurde ein Algorithmus für die Gewebe Segmentierung erstellt und auf dem Trainingssatz von Bildern (C) angewendet. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.

Abbildung 2: Zell Segmentierung in Nuclear, Zytoplasmatischen und Membrankompartimente. Das Kernfach wurde für die Zell Segmentierung definiert durch eine Mindestschwelle für Hämatoxylin Gegenfärbelösung (A) einstellen. Das Zytoplasma wurde in Bezug auf den Kern definiert durch voreingestellten Algorithmen in der Software (B) verwendet wird . E-Cadherin wurde als Membranmarker verwendet wird und ein Minimum bedeuten OD Schwelle der Membranraum (C) zu definieren , angewendet wurde. Diese Technik alleows simultane Quantifizierung von Proteinexpression in allen Zellkompartimenten (D). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
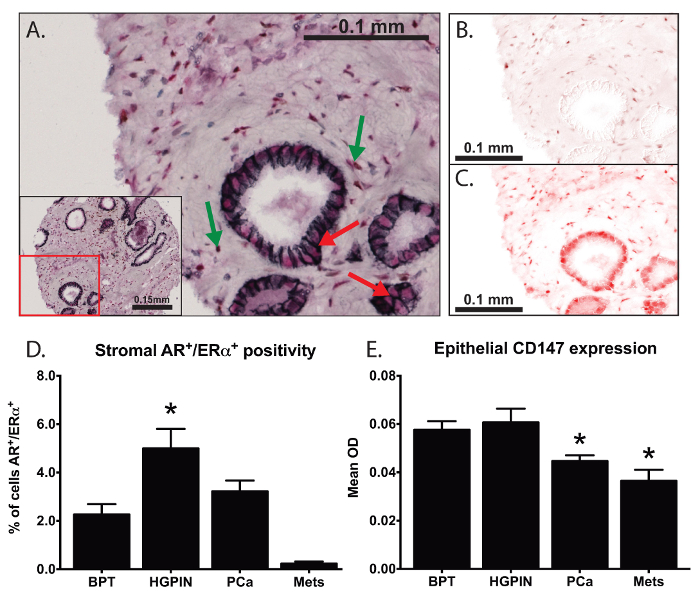
Abbildung 3: Analyse von Co-Lokalisation und Membran-spezifische Protein Expression. Multiplexed IHC wurde verwendet , um die Expression und Lokalisierung von Androgen - Rezeptor (AR), den Östrogenrezeptor-alpha (ER & agr;), E-Cadherin und CD147 (A) zu untersuchen. DAB (brown Chromogen) wurde verwendet , AR (B) und ein rotes Chromogen zu markieren verwendet ERa (C) zu markieren. Wir waren in der Lage , räumlich überlappende Biomarker (grüne Pfeile in A) zu identifizieren und zu quantifizieren (D) den Anteil an Zellen mit Kern-Co-Lokalisation von ERa und ARinnerhalb der Prostata Stroma. Durch die Verwendung von E-Cadherin (black Chromogen) , um die Plasmamembran (rote Pfeile in A) zu definieren, quantifizierten wir membranspezifische Expression von CD147 in Prostatagewebe (E) 19. Einweg-Varianzanalyse (ANOVA) wurde für die statistische Analyse verwendet, Fehlerbalken reflektieren Standardfehler des Mittelwerts und Sternchen repräsentieren p <0.05. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
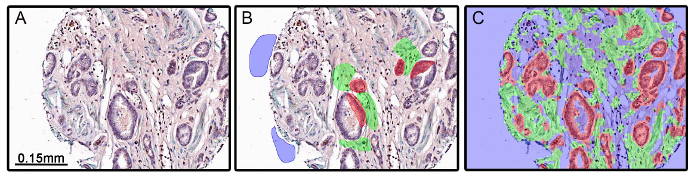
Abbildung 4: Unzureichende Tissue Segmentation Resultierend aus Experimentelles Design. , Androgen-Rezeptor (rot Chromogen) und Androgen-Rezeptor-Variante 7 (DAB Chromogen); Gutartige und bösartige Prostatagewebe wurden für α-Glattmuskelaktin (grün Chromogen α-SMA) gefärbt. a-SMA wurde verwendet alsein Marker von Stroma und in einigen Tumoren, einschließlich Prostata (A) verringert. Beim Training durchgeführt wurde (B) und dem Segmentierungsalgorithmus Gewebe angelegt wurde, waren die Stromazellen und Epithelzellen Abteile unzureichend segmentierten aufgrund des Fehlens von α-SMA - Färbung (C). Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Diskussion
Die Verwendung von traditionellen Immunhistochemie zur Bewertung der Proteinexpression durch subjektive, semi-quantitative Methoden der Analyse 22,23 begrenzt. Advance-Plattformen sind für Hochdurchsatz-Analyse von Biomarkern Expression und Lokalisation erstellt. Detaillierte Segmentierung der beiden Gewebe und subzellulärer Kompartimente ermöglicht es Benutzern, Biomarker Ausdruck zu studieren, Lokalisierung und Co-Lokalisation mit anderen Markern von Interesse. In früheren Studien haben wir die Brauchbarkeit von IHC und multispektralen Abbildungs gezeigt, insbesondere , wenn verwendet , um Proteine an der gleichen Zellkompartiment 18,20,21 lokalisiert zu studieren. Bei 24 mit dem Gewebe - Mikroarrays kombiniert ermöglichen diese Techniken für eine schnellere und objektiven Quantifizierung der Proteinexpression als durch einen Pathologen durch manuelle Analyse erlaubt.
Ein Problem bei der Verwendung von Immunhistochemie für Proteinquantifizierung ist Irreproduzibilität der Ergebnisse aufgrund der subjective Art der Analyse und der inhärenten Unterschiede in Technik und Reagenzien. Ein wesentlicher Vorteil der Plattformen wie multispektrale Bildgebung unter Verwendung von Protein-Expression zur Messung der Reproduzierbarkeit der Ergebnisse. Die Daten aus EDV - Pathologie - Plattformen weisen eine hohe Korrelation mit manueller Analyse durch einen Pathologen , während die Daten in einem kontinuierlichen Format zurückkehrt und sich im wesentlichen Arbeitszeit zu reduzieren, vor allem , wenn sie mit großen Probenarbeitsgrößen 12,16,25. Studien haben in den Ergebnissen zwischen verschiedenen Plattformen Multispektraldaten 14 insgesamt hohe Übereinstimmung gezeigt. Des Weiteren haben wir bereits gezeigt , dass Färbungsergebnisse auch in hohem Maße reproduzierbar sind , wenn es Schwankungen in der Anzahl von Proteinen 12 untersucht. Die Verwendung von automatischen Pathologie Plattformen sowohl für Färbung und Analyse nicht alle Unstimmigkeiten in den Ergebnissen, wie sie beseitigen von Antikörpern stammen gegen verschiedene Epitope erstellt, aber diese Plattformen verringern können wesentlich Voreingenommenheit und irReproduzierbarkeit häufig im Zusammenhang mit der Immunhistochemie in Verbindung gebracht.
Es gibt bestimmte Schritte im Protokoll, das für die Rückkehr genaue, reproduzierbare Ergebnisse wesentlich sind. Entsprechende Versuchsdesign durch Selektion von Proteinen eingeschlossen sein, wie Gewebe oder subzellulärer Marker wichtig für eine genaue Segmentierung ist. Wenn Positivität Analyse durchgeführt wird, hat die Auswahl einer unteren Schwelle für die positive Färbung eine große Wirkung auf die endgültigen Ergebnisse. Während eine Schwelle für "on / off" Proteine wie Kerntranskriptionsfaktoren der Auswahl ist ziemlich unkompliziert, schwierig ist, einen Schwellwert für mehr heterogen exprimierten Proteine zu finden. Dies sollte idealerweise mit einem Board-zertifizierten urogenitalen Pathologe oder als gemittelte Punktzahl über mehrere Beobachter finden die ideale Schwelle für die Analyse in Zusammenarbeit durchgeführt werden.
Es ist wichtig, einige Einschränkungen der aktuellen Versionen dieser Technologie zu erkennen. Nach AbwahlLäuterung kann in das Zytoplasma Fach, drei Ansätze verwendet werden: (1) mit einem Zytoplasma-spezifischen Marker Anfärben, (2) mit einer Membran-spezifischen Marker Färbung und den Kern-zu-Membran als Zytoplasma unter Verwendung Markierungsabstand, und (3) unter Verwendung von ein Ziehansatz manuell die Grenzen des Zytoplasmas in Bezug auf den Kern definieren. Aus unserer Erfahrung eine Membran spezifische Marker ist die genaueste Methode. Die manuelle Zeichnung Ansatz ist in der Regel genau, wenn Kerne zentral gelegen sind oder wenn die Biomarker von Interesse wird gleichmäßig über das Zytoplasma verteilt. Genau zu definieren, in das Zytoplasma von Stromazellen wie Fibroblasten und glatten Muskelzellen wie vor schwierig und sollte in Betracht gezogen werden, wenn ein Experiment zu entwerfen.
Eine weitere Einschränkung der Technik ist die Abhängigkeit von Kernen für die Zellsegmentierung. Wenn Schnittebene den Kern einer bestimmten Zelle schließt, wird diese Zelle nicht in Analyse einbezogen werden. Wenn es keinensichtbar Zytoplasma zwischen benachbarten Kernen oder Klumpen der Kerne, werden diese oft als ein großes Atom Klumpen erkannt, anstatt verschiedene Kerne. Generell ist Hämatoxylin counterstain normalerweise ausreichend für eine akzeptable Kernsegmentierung und Manipulation von Software-Einstellungen wie maximale Schwelle für Kern Größe können die meisten Probleme mit der nuklearen Segmentierung beheben.
Eine letzte Einschränkung von automatisierten Pathologie-Plattformen ist unzuverlässig Segmentierung von Geweben aus armen experimentellen Design führt. Die Bedeutung der Wahl der geeigneten Gewebe und Zellen Biomarker für die Beantwortung der Frage von Interesse kann nicht hoch genug eingeschätzt werden. Wie wir in unserer repräsentativen Ergebnisse zeigten, Marker eine epitheliale oder Stromazellen, welche die Expression zwischen Krankheitszuständen signifikant ändert kann Probleme bereiten, wenn ein Algorithmus zur Segmentierung Gewebe zu schaffen. Es ist erwähnenswert, dass es alternative Möglichkeiten, wenn schwierig wie diese Analysen ergeben. Beispielsweise einzelne images kann durch manuelles Ziehen alle Gewebegruppen analysiert werden, und nicht durch einen Algorithmus aus einem Trainingssatz von Bildern anwenden. Dies bietet den Vorteil der Segmentierung, die perfekt im Einklang mit dem, was der Benutzer wünscht, aber es führt auch Subjektivität und die Zeit erheblich erhöhen können erforderlich, um eine Analyse zu beenden. Geeignete Versuchsanordnung ist der einfachste Weg Analyse zu beschleunigen und um die Genauigkeit der Gewebe- und Zellsegmentierung maximieren.
Diese Technologie und Protokoll haben viele zukünftige Anwendungen. Zahlreiche Biomarker für bestimmte Krankheitszustände wurden identifiziert, aber nicht validiert. Hoher Durchsatz objektive Analyse mit Multispektraldaten Plattformen erleichtert die Validierung dieser Biomarker. Ferner kann die Bewertung der Expression und Co-Lokalisierung mehrerer Proteine Einblicke in schlecht verstandene Signalwege. Zweifellos, die Verringerung der inhärenten Subjektivität mit der Analyse von immunhistochemischen staini assoziiertng ist wertvoll, um die Expression und Lokalisation von einer Vielzahl von Proteinmarkern für das Verständnis.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen haben.
Danksagungen
Die Autoren danken der University of Wisconsin Translational Forschungsinitiativen in der Pathologie Labor, teilweise durch die UW Abteilung für Pathologie und Labormedizin und UWCCC Zuschuss P30 CA014520 unterstützt, für die Benutzung ihrer Einrichtungen und Dienstleistungen.
Materialien
| Name | Company | Catalog Number | Comments |
| Xylene | Fisher Chemical | X3F1GAL | NFPA rating: Health – 2, Fire – 3 , Reactivity – 0 |
| Ethyl Alcohol-200 proof | Fisher Scientific | 4355223 | NFPA rating: Health – 0, Fire – 3 , Reactivity – 0 |
| Tris Base | Fisher Scientific | BP152-500 | NFPA rating: Health – 2, Fire – 0 , Reactivity – 0 |
| Tris Hydroxymethyl aminomethane HCl | Fisher Scientific | BP153-1 | NFPA rating: Health – 2, Fire – 0 , Reactivity – 0 |
| Tween 20 | Chem-Impex | 1512 | NFPA rating: Health – 0, Fire – 1 , Reactivity – 0 |
| Phosphate-buffered saline | Fisher Scientific | BP2944-100 | NFPA rating: Health – 1, Fire – 0 , Reactivity – 0 |
| Peroxidazed | Biocare Medical | PX968 | Avoid contact with skin and eyes. May cause skin irritation and eye damage. |
| Estrogen Receptor alpha | Thermo Fisher Scientific-Labvision | RM9101 | Not classified as hazardous |
| Androgen Receptor | SCBT | sc-816 | Not classified as hazardous |
| CD147 | Biodesign | P87535M | Not classified as hazardous |
| E-cadherin | Dako | M3612 | Not classified as hazardous |
| Renoir Red Andibody Diluent | Biocare Medical | PD904 | It is specially designed to work with Tris-based antibodies |
| DeCloaking Chamber | Biocare Medical | Model DC2002 | Take normal precautions for using a pressure cooker |
| Barrier pen-Immuno Edge | Vector Labs | H-4000 | |
| Denaturing Kit-Elution step | Biocare Medical | DNS001H | Not classified as hazardous |
| Mach 2 Goat anti-Rabbit HRP Polymer | Biocare Medical | RHRP520 | Not classified as hazardous |
| Mach 2 Goat anti-Rabbit AP Polymer | Biocare Medical | RALP525 | Not classified as hazardous |
| Mach 2 Goat anti-Mouse HRP Polymer | Biocare Medical | M3M530 | Not classified as hazardous |
| Betazoid DAB Chromogen Kit | Biocare Medical | BDB2004 | 1. DAB is known to be a suspected carcinogen. 2. Do not expose DAB components to strong light or direct sunlight. 3. Wear appropriate personal protective equipment and clothing. 4. DAB may cause sensitization of skin. Avoid contact with skin and eyes. 5. Observe all federal, state and local environmental regarding disposal |
| Warp Red Chromogen Kit | Biocare Medical | WR806 | Corrosive. Acid that may cause skin irritation or eye damage. |
| Vina Green Chromogen Kit | Biocare Medical | BRR807 | Harmful if swallowed |
| Bajoran Purple Chromogen Kit | Biocare Medical | BJP807 | Flammable liquid. Keep away from heat, flames and sparks. Harmful by ingestion or absorption. Avoid contact with skin or eyes, and avoid inhalation. |
| Cat Hematoxylin | Biocare Medical | CATHE | Purple solution with a mild acetic acid (vinegar) scent. May be irritating to skin or eyes. Avoid contact with skin and eyes. Avoid ingestion. |
| XYL Mounting Media | Richard Allen | 8312-4 | NFPA rating: Health – 2, Fire – 3 , Reactivity – 0 |
| 1.5 Coverslips | Fisher Brand | 22266858 | Sharp edges |
| Incubation (Humidity)Chamber | obsolete | obsolete | Multiple vendors available |
| Convection Oven | Stabil- Therm | C-4008-Q | |
| Background Punisher Blocking Reagent | Biocare Medical | BP974 | This product is not classified as hazardous. |
| inForm software | PerkinElmer | CLS135781 | Primary multispectral imaging software used in manuscript |
| Nuance software | PerkinElmer | Nuance EX | Software used for making spectral libraries within manuscript |
| Vectra microscope and slide scanner | PerkinElmer | VECTRA | Automated slide scanner and microscope for obtaining IM3 image cubes |
Referenzen
- Valdman, A., et al. Expression of redox pathway enzymes in human prostatic tissue. Anal Quant Cytol Histol. 31 (6), 367-374 (2009).
- Rimm, D. L., Camp, R. L., Charette, L. A., Olsen, D. A., Provost, E. Amplification of tissue by construction of tissue microarrays. Exp Mol Pathol. 70 (3), 255-264 (2001).
- Jonmarker, S., et al. Expression of PDX-1 in prostate cancer, prostatic intraepithelial neoplasia and benign prostatic tissue. APMIS. 116 (6), 491-498 (2008).
- McCarty, K. S., Miller, L. S., Cox, E. B., Konrath, J., McCarty, K. S. Sr Estrogen receptor analyses. Correlation of biochemical and immunohistochemical methods using monoclonal antireceptor antibodies. Arch Pathol Lab Med. 109 (8), 716-721 (1985).
- Volante, M., et al. Somatostatin receptor type 2A immunohistochemistry in neuroendocrine tumors: a proposal of scoring system correlated with somatostatin receptor scintigraphy. Mod Pathol. 20 (11), 1172-1182 (2007).
- Muris, J. J., et al. Immunohistochemical profiling of caspase signaling pathways predicts clinical response to chemotherapy in primary nodal diffuse large B-cell lymphomas. Blood. 105 (7), 2916-2923 (2005).
- Jaraj, S. J., et al. Intra- and interobserver reproducibility of interpretation of immunohistochemical stains of prostate cancer. Virchows Arch. 455 (4), 375-381 (2009).
- Nakane, P. K., Pierce, G. B. Enzyme-labeled antibodies: preparation and application for the localization of antigens. J Histochem Cytochem. 14 (12), 929-931 (1966).
- Peters, T. J. Investigation of tissue organelles by a combination of analytical subcellular fractionation and enzymic microanalysis: a new approach to pathology. J Clin Pathol. 34 (1), 1-12 (1981).
- Emmert-Buck, M. R., et al. Laser Capture Microdissection. Science. 274 (5289), 998-1001 (1996).
- Waters, J. C. Accuracy and precision in quantitative fluorescence microscopy. J Cell Biol. 185 (7), 1135-1148 (2009).
- Huang, W., Hennrick, K., Drew, S. A colorful future of quantitative pathology: validation of Vectra technology using chromogenic multiplexed immunohistochemistry and prostate tissue microarrays. Hum Pathol. 44 (1), 29-38 (2013).
- Rimm, D. L. C-Path: A Watson-Like Visit to the Pathology Lab. Science Translational Medicine. 3 (108), (2011).
- Fiore, C., et al. Utility of multispectral imaging in automated quantitative scoring of immunohistochemistry. J Clin Pathol. 65 (6), 496-502 (2012).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: A review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Rizzardi, A. E., et al. Quantitative comparison of immunohistochemical staining measured by digital image analysis versus pathologist visual scoring. Diagn Pathol. 7, 42(2012).
- Bauman, T. M., et al. Characterization of fibrillar collagens and extracellular matrix of glandular benign prostatic hyperplasia nodules. PLoS One. 9 (10), e109102(2014).
- Bauman, T. M., et al. Beta-catenin is elevated in human benign prostatic hyperplasia specimens compared to histologically normal prostate tissue. Am J Clin Exp Urol. 2 (4), 313-322 (2014).
- Bauman, T. M., Ewald, J. A., Huang, W., Ricke, W. A. CD147 expression predicts biochemical recurrence after prostatectomy independent of histologic and pathologic features. BMC Cancer. 15 (1), 549(2015).
- Bauman, T. M., et al. Finasteride treatment alters tissue specific androgen receptor expression in prostate tissues. Prostate. 74 (9), 923-932 (2014).
- Nicholson, T. M., Sehgal, P. D., Drew, S. A., Huang, W., Ricke, W. A. Sex steroid receptor expression and localization in benign prostatic hyperplasia varies with tissue compartment. Differentiation. 85 (4-5), 140-149 (2013).
- Taylor, C. R., Levenson, R. M. Quantification of immunohistochemistry--issues concerning methods, utility and semiquantitative assessment II. Histopathology. 49 (4), 411-424 (2006).
- Matos, L. L., Trufelli, D. C., de Matos, M. G., da Silva Pinhal, M. A. Immunohistochemistry as an important tool in biomarkers detection and clinical practice. Biomark Insights. 5, 9-20 (2010).
- Kononen, J., et al. Tissue microarrays for high-throughput molecular profiling of tumor specimens. Nat Med. 4 (7), 844-847 (1998).
- Ong, C. W., et al. Computer-assisted pathological immunohistochemistry scoring is more time-effective than conventional scoring, but provides no analytical advantage. Histopathology. 56 (4), 523-529 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten