Method Article
La cuantificación de la expresión de la proteína y Co-localización Uso de multiplexado inmunohistoquímica y tinción de imágenes multiespectrales
En este artículo
Resumen
Immunohistochemistry is a powerful lab technique for evaluating protein localization and expression within tissues. Current semi-automated methods for quantitation introduce subjectivity and often create irreproducible results. Herein, we describe methods for multiplexed immunohistochemistry and objective quantitation of protein expression and co-localization using multispectral imaging.
Resumen
Immunohistochemistry is a commonly used clinical and research lab detection technique for investigating protein expression and localization within tissues. Many semi-quantitative systems have been developed for scoring expression using immunohistochemistry, but inherent subjectivity limits reproducibility and accuracy of results. Furthermore, the investigation of spatially overlapping biomarkers such as nuclear transcription factors is difficult with current immunohistochemistry techniques. We have developed and optimized a system for simultaneous investigation of multiple proteins using high throughput methods of multiplexed immunohistochemistry and multispectral imaging. Multiplexed immunohistochemistry is performed by sequential application of primary antibodies with secondary antibodies conjugated to horseradish peroxidase or alkaline phosphatase. Different chromogens are used to detect each protein of interest. Stained slides are loaded into an automated slide scanner and a protocol is created for automated image acquisition. A spectral library is created by staining a set of slides with a single chromogen on each. A subset of representative stained images are imported into multispectral imaging software and an algorithm for distinguishing tissue type is created by defining tissue compartments on images. Subcellular compartments are segmented by using hematoxylin counterstain and adjusting the intrinsic algorithm. Thresholding is applied to determine positivity and protein co-localization. The final algorithm is then applied to the entire set of tissues. Resulting data allows the user to evaluate protein expression based on tissue type (ex. epithelia vs. stroma) and subcellular compartment (nucleus vs. cytoplasm vs. plasma membrane). Co-localization analysis allows for investigation of double-positive, double-negative, and single-positive cell types. Combining multispectral imaging with multiplexed immunohistochemistry and automated image acquisition is an objective, high-throughput method for investigation of biomarkers within tissues.
Introducción
La inmunohistoquímica (IHC) es una técnica de laboratorio estándar para la detección de la proteína en el tejido, y IHC es aún ampliamente utilizado en la investigación y diagnóstico de la patología. La evaluación de la tinción IHC es a menudo semi-cuantitativa, la introducción de sesgo potencial en la interpretación de los resultados. Muchos enfoques semi-cuantitativos se han desarrollado que incorporan tanto intensidad de la tinción y el grado de tinción en diagnóstico final 1-4. Otros sistemas incluyen la intensidad de puntuación y la ubicación subcelular con el fin de localizar mejor expresión 5. La incorporación de las puntuaciones medias de varios espectadores se utiliza a menudo con el fin de minimizar los efectos de un solo espectador sesgo 6. A pesar de estos esfuerzos, la subjetividad en el análisis sigue siendo, sobre todo cuando se evalúa el grado de tinción 7. la estandarización del protocolo y de la subjetividad de minimizar la intervención humana es de suma importancia para la creación de resultados precisos y reproducibles IHC.
contenido "> Hay otras opciones además de IHC para determinar la expresión de la proteína dentro de los tejidos. En el marco de la investigación, la inmunohistoquímica ha sido considerado tradicionalmente como un medio para examinar la localización de proteínas 8, mientras que otras técnicas como la inmunotransferencia son vistos como estándar de oro para la investigación de la expresión de proteínas . La determinación de los tejidos o expresión específica para el compartimento celular es difícil sin la incorporación de técnicas avanzadas tales como el fraccionamiento celular o captura por láser microdisección 9,10. El uso de anticuerpos fluorescentes en portaobjetos de tejidos ofrece un compromiso razonable, pero autofluorescencia de fondo debido a NADPH, la lipofuscina, reticular fibras de colágeno, elastina, y pueden hacer difícil cuantificación precisa 11.Plataformas computacionales de patología automatizados son una dirección prometedora para más cuantificación objetiva de la tinción de la patología 12-15. La combinación de imágenes multiespectrales con microarrays de tejidosfacilita el análisis de alto rendimiento de expresión de proteínas en muestras de gran tamaño. Con estas técnicas, el análisis de la proteína co-localización, la heterogeneidad de la tinción, y el tejido y localización subcelular es posible al tiempo que reduce sustancialmente tanto los sesgos y tiempo necesario para el análisis inherentes, mientras que la devolución de datos en un formato continuo en lugar de categórica 16. Por lo tanto, el propósito de este estudio fue demostrar la utilidad de la metodología y para llevar a cabo la inmunohistoquímica multiplexado con el análisis, utilizando software de imagen multiespectral.
Este protocolo está escrito para manual de tinción, múltiplex inmunohistoquímica de una sola sección de tejido de diapositivas con cuatro anticuerpos monoclonales optimizados. Como un experimento representativo, anti-conejo nuclear alfa del receptor de estrógeno (ER) y receptor de andrógenos (AR) se multiplexan con membrana anti-ratón de CD147 y unida a la membrana anti-ratón de E-cadherina. Cualquier anticuerpo de elección puede ser substituted de los anticuerpos mencionados en el presente documento, pero cada combinación de anticuerpos requiere la optimización separada. Pre-tratamiento para todos los anticuerpos debe ser idéntico. Los anticuerpos AR y CD147 deben optimizarse individualmente y luego como un cóctel. Cada anticuerpo se detecta utilizando un sistema de polímero libre de biotina y una de las 4 cromógenos únicas.
Protocolo
NOTA: El protocolo en la presente memoria describe la tinción y análisis de un microarray de tejido (TMA), descrita anteriormente 12,17,18. La sección de TMA de espesor 4 m se obtuvo de un bloque de parafina usando un microtomo estándar.
NOTA: Una biblioteca espectral para las 4 cromógenos y de contraste debe ser creado para una imagen cuantificación. Con el fin de hacer esto, el protocolo optimizado para cada anticuerpo individual debe ser ejecutado con un anticuerpo por diapositiva, menos la contratinción final. Una quinta diapositiva debe ser teñidas con hematoxilina para generar los 5 imágenes necesarias para crear la biblioteca espectral.
1. La inmunohistoquímica Multiplex
- diapositivas Hornear en un horno a 60 ° durante 20 minutos. Usando una campana de humos química, sumerja la diapositiva en el 100% de xileno. Repita para 2 cambios de xileno. Sumergir diapositiva en 100% de etanol durante 5 min. Repita con frescos etanol al 100%.
- Sumergir diapositiva en 95% de etanol durante 3 min. Repita con frescos95% de etanol. Sumergir diapositiva en 70% de etanol durante 3 min. Sumergir portaobjetos en agua destilada durante 3 min. Repita con agua destilada fresca. Toque de diapositivas en vertical para drenar el agua de la diapositiva. Aplicar 200 l de bloqueador de peroxidasa endógena lista para uso para 5 min.
- Escurrir bloqueador de peroxidasa de diapositiva y sumergirse en 45 ml tampón de recuperación lista para el uso (pH inferior a 7,0). Coloque el recipiente de diapositivas en una olla a presión y el programa de arranque ajustado a 124 ° C durante 4 minutos. Deje que se enfríe la olla a presión durante 20 minutos. antes de abrir.
- Separar el recipiente de tampón de recuperación y dejar enfriar 10 minutos adicionales. Enjuague de diapositivas en agua destilada durante 10 min. Drenar el agua de la diapositiva y delimitar cuidadosamente el tejido en la diapositiva con una pluma barrera hidrofóbica.
- Añadir 200 l de solución salina tamponada con fosfato (PBS) .Transfer deslizante a un receptáculo plana con tapa (cámara de incubación) que contiene gasa húmeda.
- Preparar 1 litro de 1x solución salina tamponada con Tris con Tween-20 (TBST) por dilución de 50 ml de 20x Tris solución salina tamponada que contiene 1% de Tween-20 y 950 ml de agua destilada.
- Escurrir PBS a partir de portaobjetos y aplique 200 l de 1x TBST. Incubar 2 min. Escurrir TBST de diapositiva y diapositiva volver a la cámara de incubación. Aplicar 200 l de bloque de proteína universales listos para el uso, la cámara de cierre y se incuba durante 7 min.
- Drenaje del bloque de la proteína y el tobogán volver a la cámara de incubación. Diluir anticuerpo anti-ER 1: 400 añadiendo 0,6 l de anticuerpo a 239,4 l de diluyente de anticuerpo. Aplicar 200 l de anticuerpo diluido a deslizar y la cámara de cierre.
- Incubar diapositiva a temperatura ambiente durante 2 horas. Escurrir anticuerpo de la diapositiva y enjuague con TBST, seguido de inmersión durante 5 min en TBST.
- Escurrir TBST de diapositiva y diapositiva volver a la cámara de incubación. Aplicar 200 l de cabra anti-conejo de polímero con peroxidasa de rábano listo para usar y la cámara de cierre. Incubar durante 30 minutos. a temperatura ambiente. diapositiva enjuague con TBST y lugar de diapositivas en un recipiente de TBST durante 5 min.
- Preparar marrón peroxidasa de rábano-3,3'-diaminobencidina (HRP-DAB) cromógeno mediante la adición de 8 l de cromógeno DAB a 250 l de tampón de sustrato DAB y mezclando bien.
- Escurrir TBST de diapositivas, diapositivas retorno a la cámara de incubación y se aplican 200 l de cromógeno marrón HRP-DAB durante 6 min. Enjuague bien el portaobjetos con agua destilada y el lugar de diapositivas en un recipiente con agua destilada.
- Mezclar la solución desnaturalizante mediante la combinación de 50 l de la primera reactivo de elución con 150 l de la segunda reactivo tampón. Drenar el agua fuera de la diapositiva y aplique la solución diluida de desnaturalización a la diapositiva durante 3 min. a temperatura ambiente. Decantar y enjuague reactivo desnaturalizante con TBST. Sumergir la diapositiva en TBST durante 5 minutos.
- Preparar cóctel de anticuerpos AR / CD147 mediante la dilución de 4.2 l de anticuerpo anti-AR y 2,8 l de anticuerpo anti-CD147 con 203 l de diluyente de anticuerpo. Escurrir TBST de la diapositiva, tobogán volver a aplicar cámara de incubación y 200 l de AR / c CD147ocktail a la diapositiva. Cerrar la cámara e incubar durante 1 hora a temperatura ambiente. diapositiva enjuague con TBST y se sumerja en TBST durante 5 minutos.
- Escurrir TBST, el retorno de diapositivas para cámara de incubación y se aplican 200 l de anti-conejo de polímero de fosfatasa alcalina de cabra lista para su uso. Cerrar la cámara y deslice incubado durante 30 min. a temperatura ambiente. diapositiva enjuague con TBST y se sumerja en TBST durante 5 minutos.
- Preparar la fosfatasa alcalina roja cromógeno mediante la adición de 3,2 l de cromógeno rojo de 250 l de tampón de cromógeno rojo y mezclar bien.
- Escurrir TBST de diapositiva y diapositiva volver a la cámara de incubación. Aplicar 200 l de cromógeno de la fosfatasa alcalina diluida roja a la diapositiva, cámara de cierre y se incuba durante 7 minutos.
- Escurrir y enjuagar bien cromógeno portaobjetos con agua destilada. Coloque los portaobjetos en un recipiente con agua destilada durante 5 minutos. Drenar el agua de la diapositiva, lavar con TBST y volver a la cámara de incubación. Aplicar 200 l de cabra polímero anti-ratón-HRP a la diapositiva y close cámara. Incubar durante 30 minutos. a temperatura ambiente.
- Escurrir polímero a partir de la diapositiva y enjuague con TBST. Colocar el portaobjetos en TBST durante 5 minutos. Preparar 200 l de púrpura cromógeno HRP-vinculada mediante la adición de 3,2 l de reactivo de estabilización a 250 l de la memoria intermedia y mezclar bien. Añadir 3,2 l de cromógeno púrpura y mezclar bien, a continuación, 3,2 l de reactivo de peróxido de hidrógeno y mezclar bien.
- Escurrir TBST fuera portaobjetos y aplique 200 l de cromógeno púrpura preparado para porta e incubar durante 7 minutos. a temperatura ambiente. diapositiva enjuague con agua destilada tobogán de agua y colocar en un recipiente con agua destilada durante 5 minutos.
- Mezclar la solución desnaturalizante mediante la mezcla de 50 l de la primera reactivo de elución con 150 l de la segunda reactivo tampón. Drenar el agua fuera de la diapositiva y aplique la solución diluida de desnaturalización a la diapositiva durante 3 min. a temperatura ambiente.
- Enjuague reactivo desnaturalizante con TBST. Coloque los portaobjetos en un recipiente de TBST durante 5 min. Diluir contra la E-cadherina de anticuerposa 1: 200 añadiendo anticuerpo 1,1 l a 218,9 l de diluyente de anticuerpo. Escurrir el TBST de la diapositiva, diapositiva de retorno a la cámara de incubación y se aplican 200 l de E-cadherina a la diapositiva. Cerrar la cámara e incubar 30 min. a temperatura ambiente.
- diapositiva enjuague con TBST y lugar de diapositivas en un recipiente de TBST durante 5 min. Escurrir el TBST de diapositivas, diapositivas regresar a cámara de incubación y aplicar polímero HRP anti-ratón de cabra 200 l. Incubar a temperatura ambiente durante 30 min. diapositiva enjuague con TBST y lugar de diapositivas en un recipiente de TBST durante 5 min.
- Prepare negro HRP-vinculada cromógeno mediante la adición de 8 l de cromógeno a 250 l de tampón. Aplicar 200 l de cromógeno para deslizarse y desarrollar durante 2 min. Enjuague bien portaobjetos con agua destilada y se sumerja en un recipiente de agua destilada.
- Diluir hematoxilina mediante la adición de hematoxilina 40 l 200 l de agua destilada. Drenar el agua del portaobjetos y aplique 200 l de hematoxilina diluida para deslizarse durante 30 segundos. enjuague de diapositivascorrer el agua del grifo a fondo durante 2 min., luego enjuague en agua destilada durante 30 segundos. Lugar de diapositivas en un horno de 60 ° C durante 15-30 min.
- Sumergir diapositiva en recién preparada xileno al 100% durante 30 segundos, limpie el exceso de xileno fuera y añadir una gota de medio de montaje permanente. Cubreobjetos con un No. 1.5 cubreobjetos.
2. Adquisición de imágenes automatizado y análisis
- Cargar muestras teñidas sobre un escáner de diapositivas automatizada. Con base en el diámetro, la separación, y el número de núcleos de TMA, crear un protocolo de análisis automatizado según el manual de instrucciones del fabricante.
- software de imágenes multiespectrales abierta para construir una biblioteca espectral a partir de diapositivas control teñidas con cromógenos individuales y la hematoxilina manchado diapositivas. Abrir un cubo de imagen adquirida a partir de una corredera de control y seleccione entre cuatro y cinco áreas teñidas positivamente para definir ópticamente que cromógeno. Repita con cubos de imágenes a otros portaobjetos de control hasta que una completa biblioteca espectral representing todos los cromógenos se crea, a continuación, guardar la biblioteca espectral.
- Comenzar un nuevo proyecto dentro de software de imágenes multiespectrales. Seleccione "multiespectral (.im3)" para la opción de formato de imagen y "Campo claro" para el formato de muestra. En primer lugar, configurar el proyecto eligiendo opciones deseadas: "segmento de tejido", "Buscar características", "Fenotipificación", "Score" y "Exportar". En este punto, la resolución de la imagen se puede cambiar a acelerar el tiempo de análisis si se desea.
Segmentación 3. Tejido
- Importar la biblioteca espectral creada anteriormente y seleccione todos los cromógenos que se incluirán en el análisis.
- cubos de imagen abiertas que deben incluirse en los datos de entrenamiento seleccionando la opción "Abrir Cubo de imagen". Para asegurar la precisión de entrenamiento, seleccione al menos un 18% del número total de imágenes para ser analizada. Seleccionar las imágenes que representan a todos los estados de la enfermedad para aumentar la precisión de segmentación. Esta porción de imágenes is llama el conjunto de formación de imágenes.
- imágenes de balance de blancos en el conjunto de entrenamiento mediante la selección de la herramienta cuentagotas y la elección de un área de una imagen que es de color blanco.
- Seleccione el botón de "Avance" para pasar a la segmentación del tejido. Utilice el panel "Tejido Categorías" para elegir los tipos de tejido que va a analizarse (es decir, "tejido" y "no tejido"). Para la localización de tejido de proteínas más precisa, múltiples categorías de tejido se pueden utilizar (es decir. "Epitelio", "estroma", y "no tejido").
- Comience a crear el algoritmo y la definición de las categorías de tejidos mediante la elaboración en torno a grupos de células dentro de imágenes de entrenamiento. Cuando haya terminado con una categoría de tejidos, repetir para otras categorías de tejidos. Asegúrese de elegir los grupos de células dentro de las imágenes que son característicos de ese tipo de categoría de tejidos.
- Repita este proceso para todas las imágenes dentro del conjunto de entrenamiento de las imágenes.
- Seleccione los componentes (cromógenos) que se incluirán en traiNing para el "Tejido Segmenter". Al realizar el análisis de imágenes de campo claro de inmunohistoquímica, todos los componentes se incluyen normalmente en la formación. Incluya abundantes imágenes tinción negativa en el conjunto de entrenamiento para evitar el sesgo durante este paso.
- Elija una "escala del patrón de" apropiado para la formación del tejido segmenter. Bajo 20 aumentos, a gran escala patrón es normalmente adecuada. Cuando se trabaja con tejidos con una arquitectura muy bien debajo de un aumento mayor, una escala modelo más pequeño es más apropiado.
- Seleccione el botón "Tren de tejidos Segmenter" para comenzar a entrenar a la segmenter tejido. Observar una precisión de cuadro emergente que muestra que refleja la proporción de píxeles dentro de las regiones de formación que se clasifican correctamente.
NOTA: El software intenta continuamente para mejorar la precisión del algoritmo hasta que se detiene manualmente. Hemos demostrado previamente que el entrenamiento el 18% de los resultados de imágenes en 97% de precisión de entrenamiento de 12. - Después de que el tejido se entrenó segmenter, elegir una resolución de segmento apropiado. resolución del segmento corresponde al tiempo requerido para las imágenes del segmento, con baja resolución que requiere menos tiempo y resolución fina que requiere más tiempo.
- Segmento de todo el conjunto de entrenamiento de las imágenes haciendo clic en "imágenes" del segmento. Deje que el software de tiempo suficiente para aplicar el algoritmo para todas las imágenes de entrenamiento. Cuando haya terminado, revisar el conjunto de entrenamiento para encontrar cualquier tejido mal clasificados con el algoritmo de entrenamiento actual.
- Para afinar el proceso de segmentación de tejidos, añadir, editar o eliminar regiones de formación de tejido. Utilice la opción "bordes de recorte" Si los pixels se extienden en los bordes de una categoría de tejidos en otra. Si están mal clasificados pequeños grupos de píxeles, ajustar el umbral del tamaño mínimo del segmento.
- Cuando se hayan completado las modificaciones, seleccione "Segmento de Imágenes", esta se volverá a entrenar el segmenter tejido para crear un nuevo algoritmo para la segmentación de tejidos. El algoritmo anteriorse guarda automáticamente y pueden ser devueltos a si es necesario.
- Cuando confiado con los resultados algoritmo de segmentación de tejidos, avanzar a la segmentación de célula, seleccione el botón de "Avance".
Segmentación 4. celular
- Elegir los compartimentos subcelulares que se incluirán en la segmentación celular. Asegúrese de que "núcleos" ya está seleccionada para elegir "citoplasma" o "membrana".
NOTA: la segmentación "membrana" sólo debe ser elegido cuando un marcador de proteína de membrana específico se incluyó en IHC, tales como E-cadherina. - Dentro de la pestaña "núcleos", hay varias opciones. Comience eligiendo valores apropiados para la segmentación nuclear (véase el paso 4.3). Elija si las categorías de tejidos individuales o todos serán incluidos en la segmentación.
- Seleccionar un método para la segmentación nuclear
- Seleccione el colorante de contraste "basado en píxeles (Umbral)" enfoque de un método simplificado para la obtención de buenosresultados sin saber mucho acerca de la configuración de procesamiento de imágenes.
NOTA: Esto a menudo hace una buena primera opción. Al seleccionar un enfoque "basado en píxeles (Umbral)" es más apropiado cuando hay una contra-tinción nuclear fiable, mientras que "objeto-base" se debe utilizar si hay una falta de contratinción nuclear consistente. - Seleccione la opción "basado en píxeles (Umbral)" enfoque cuando hay una contratinción nuclear fiable.
NOTA: Este enfoque es puramente basado en píxeles. Este enfoque también se puede utilizar para otras necesidades de análisis de imágenes que pueden ser satisfechos con un simple umbral, tales como la detección de todos los píxeles dentro de una categoría tejido que tiñe positivo para una mancha IHC. - Seleccionar el enfoque basado en objetos si el contratinción nuclear no proporciona tinción coherente y específica de objetos nucleares, y se necesitan enfoques más avanzados basados en la morfometría para detectar los núcleos.
- Seleccione "Componentes para la segmentación nuclear", "NuclTamaño del oído ", y" limpieza "si el usuario desea definir aún más opciones de segmentación núcleos.
- Seleccione el colorante de contraste "basado en píxeles (Umbral)" enfoque de un método simplificado para la obtención de buenosresultados sin saber mucho acerca de la configuración de procesamiento de imágenes.
- Seleccionar parámetros de forma citoplasma
- Seleccione el botón de "Avance" para pasar a la segmentación del citoplasma.
NOTA: Hay varias opciones dentro de la segmentación del citoplasma para elegir. La opción denominada "distancia al núcleo interno" está basado en píxeles. Refleja la distancia entre el exterior del núcleo y el interior del citoplasma. El aumento de este valor disminuye la probabilidad de que la señal nuclear cruza en el compartimiento citoplasma. - A continuación, seleccione "distancia al núcleo externo".
NOTA: Este es también basada en píxeles. La distancia exterior de núcleo refleja la distancia entre el exterior del núcleo y el exterior de la célula. Este valor se puede establecer grande o lo suficientemente pequeño como para incluir o excluir señales de membrana, si se desea. - A continuación, seleccione "mínimo".
NOTA: El tamaño mínimo, pixel basado, refleja el tamaño mínimo de la muestra citoplasma para ser incluido en el análisis. Si el tamaño del citoplasma es más pequeño en una célula particular, tal como cuando las células están estrechamente empaquetadas, esta segmentación citoplasma se excluye del análisis. - Seleccione la siguiente opción, "Componente" a "Principal", "Secundario", y seleccione "terciario" como opciones secundarias.
NOTA: Aquí se puede elegir un marcador específico citoplasma. Similar a la segmentación nuclear, si un marcador IHC citoplasmática específica se ha utilizado, este componente se puede utilizar para definir el citoplasma mediante la búsqueda de un valor de umbral mínimo. Esto ayuda a definir el citoplasma, pero no es necesario para una precisión aceptable. - Pasando a la última de las tres opciones de las fichas es "membrana". Aquí, el usuario define el compartimento de membrana con un marcador de proteína. Elija "primaria", "Secundario" y / o "terciario" para cada marcador específico de membrana utilizado. Ajuste "Escala O completaD "para encontrar un umbral mínimo o un punto positivo para cada marcador en las membranas celulares.
- Continuando bajo la etiqueta de "membrana" y seleccione la opción "Tamaño máximo de la célula".
NOTA: Esta es la distancia al valor de la membrana, en píxeles, que especifica el tamaño máximo de las células. Un valor de 12 es normalmente adecuada para imágenes tomadas con 20 aumentos. - Después de seleccionar todas las opciones, seleccione "Segmento de imagen" o "Segmento de todos". Aplicar los ajustes a las imágenes y observarlos "individual" o en modo "Galería".
NOTA: Ajustar la configuración de umbrales y si es necesario, y re-segmento hasta que esté satisfecho con los resultados de la segmentación de células en el conjunto de formación de imágenes.
- Seleccione el botón de "Avance" para pasar a la segmentación del citoplasma.
5. El fenotipado de células
Se requiere Accurate segmentación de células con el fin de obtener el fenotipo celular exacto, y la función de fenotipificación es entrenable: NOTA.
- Utilice la opción "Add &# 34; botón para añadir a la lista de fenotipos "fenotipos". Los usuarios pueden seleccionar un color para el fenotipo.
- Haga clic en "Editar fenotipos" para asignar un fenotipo de una célula. Haga clic en una celda en particular para que aparezca un menú desplegable, que permite al usuario elegir el fenotipo y luego seguir adelante. Identificar al menos cinco (5) células en cada fenotipo con el fin de proceder. La elección de 25 o más células de cada fenotipo se necesita para lograr resultados óptimos.
6. alcanzaron el IHC y co-localización
- Seleccione la opción "Avance" para pasar a la "Puntuación IHC o SI" paso. Elija una "categoría de tejidos" de marcar.
NOTA: Sólo una categoría de tejido puede ser anotado durante un análisis particular, pero si se desean resultados de la puntuación en otra categoría de tejidos, el análisis se puede repetir más tarde con diferentes configuraciones. - Elija un tipo de "Scoring" deseada.
NOTA: La positividad crea dos contenedores (positivos y negativos) para un componente de interés, Mientras que el doble positividad se utiliza para el análisis de co-localización. Otras opciones incluyen 4-bin, de 10 bandejas, y el 50-bin análisis para un marcador de elección. - Elige la célula "Compartimiento" para ser utilizado en la puntuación de análisis.
- Al igual que en la búsqueda de un umbral mínimo del componente nuclear principal, seleccione "Ver el componente de datos" y mover el cursor sobre las imágenes de entrenamiento para encontrar los umbrales mínimos de densidad óptica apropiadas de tinción de células positivas para el componente (s) de interés.
- Si las células intensamente teñidas deben ser excluidos, bajar el umbral máximo a un nivel apropiado. De lo contrario, utilice el botón de auto para elegir un umbral máximo para el análisis.
- Elige la formación de imágenes para marcar el fin de validar los valores de umbral.
NOTA: Se devolverá el porcentaje de células dentro de un contenedor particular y puede ser comparado mediante inspección visual o conteo manual. Una vez terminado, seleccione "Avance" para proceder con la etapa "Exportar".
7. Aplicando el algoritmo de análisis y Batch
- Probar el algoritmo creado a través de la segmentación del tejido, la segmentación celular, y los valores de puntuación mediante la exportación de los datos para el conjunto de entrenamiento. Siga las indicaciones para crear una nueva carpeta para el directorio de exportación. Seleccione las imágenes y tablas que se creen y se incluyeron en el análisis. Realizar el análisis mediante la opción "Exportar para todos".
- Las tablas se exportan en archivos de texto separados por tabulaciones y se pueden abrir en la mayoría de los programas de análisis de datos.
- Cuando el análisis se ha completado, ver los datos de segmentación celular y de puntuación para las imágenes con alta y baja tinción para evaluar la exactitud de los ajustes.
- Cuando esté satisfecho con los ajustes, aplicar el algoritmo creado a partir de la formación conjunto de imágenes a todo el conjunto de imágenes a través del análisis por lotes. Haga clic en la pestaña "Análisis de lote" y el algoritmo abierto se copiará desde el proyecto activo.
- Elegir un nuevo directorio de exportación y seleccionar las imágenes y tablES son para ser incluidos en el análisis. Bajo la opción de archivos de entrada, seleccionar "Añadir imágenes" y elija todas las imágenes que se incluirán en el análisis por lotes.
- Realizar el análisis seleccionando la opción "Ejecutar". Al igual que con el análisis del conjunto de entrenamiento, este paso va a requerir una cantidad de tiempo variable, dependiendo de la configuración y el número de imágenes incluidas en el análisis particulares.
- Cuando se haya completado, avanzar a la pestaña "Revisión / fusión". Los lotes de directorio por defecto en el directorio de exportación a partir del análisis por lotes y se pueden cambiar si lo desea. Seleccione "Incluir todos" y seleccione "Combinar" para crear hojas de datos con los datos de resumen para el análisis.
8. Análisis de los datos exportados
- Abrir tablas exportadas en el software de análisis de datos y eliminar u ocultar columnas de datos que son irrelevantes para el análisis, como los valores mínimos y máximos para cada componente. Los datos primarios utilizado en el análisis es en la densidad media c ópticoOLUMNA para cada componente.
- Revisión exportado mapas de segmentación de imágenes en bruto y para determinar qué muestras o núcleos de TMA han de ser excluidos del análisis. los valores de porcentaje de tejido ayudan a determinar si una muestra se debe incluir o no, con un punto de corte arbitrario de <5% utilizado más a menudo por criterios de exclusión.
- Eliminar datos o filas que contienen muestras irrelevantes para ser excluidos del análisis de datos.
- Utilizar los datos de cuantificación de proteínas para investigar los cambios en el estado de la enfermedad o la relación con las características clínico-patológicas de la enfermedad 12,18-21.
Resultados
En la Figura 1, la formación se realiza en tejidos de la próstata de las imágenes del segmento en porciones epiteliales y del estroma, junto con el compartimiento de no tejido. Mediante el uso de la membrana epitelial marcador de E-cadherina, la segmentación de las células se realizó para separar el núcleo, citoplasma, y las porciones de membrana, que se muestran en la Figura 2.
En un experimento, se utilizó multiplexada IHC para investigar la expresión y localización de AR, ER, E-cadherina, y CD147, como se muestra en la Figura 3. Utilizando estas técnicas, que son capaces de identificar las células positivas para la expresión nuclear de ambos ER y AR (Figura 3B - 3C) a pesar de la superposición de señales colorimétricos, como se muestra con flechas verdes en la figura 3A. Nos encontramos marcadas diferencias en la proporción de células estromales doble positivas dentro de los diferentes estados de prostate la enfermedad (Figura 3D). Se cuantificó la expresión de células de la membrana específico de CD147 mediante el uso de E-cadherina como una proteína marcadora (flechas rojas en la figura 3A), y fuimos el primer grupo para investigar la expresión de CD147 específico de membrana en los tejidos de la próstata. Se encontró una disminución significativa en la expresión de CD147 en asociación con la progresión del cáncer de próstata (Figura 3E) y encontramos una importante asociación con el pronóstico post-quirúrgico 19.
Hay casos en que un mal diseño experimento puede conducir a la segmentación del tejido incorrecto. En la figura 4, el algoritmo aplicado a un conjunto de tejidos hizo no con precisión epitelial segmento y compartimentos del estroma (Figura 4C). En este experimento, se utilizó actina de músculo liso α-(α-SMA) para marcar el compartimiento del estroma. Debido a α-SMA y el músculo liso se disminuye o se pierde en algunos tumores (Figure 4A), el algoritmo creado a través de la segmentación del tejido (Figura 4B) era incapaz de distinguir con precisión entre epiteliales y del estroma compartimentos de datos morfométricos solo. Se debe tener cuidado al elegir los marcadores de proteínas para los compartimentos de tejido o células.

Figura 1: Segmentación de tejido utilizando software de imágenes multiespectrales. Un microarray de tejido de próstata se tiñó mediante inmunohistoquímica multiplexado para el receptor de andrógenos (AR), receptores de estrógenos alfa (ER), E-cadherina, y CD147. Un conjunto de imágenes de entrenamiento se importaron en el software de imágenes multiespectrales que representa los tipos de tejidos y estados de enfermedad de todo el conjunto de imágenes (A). categorías de tejido fueron creados incluyendo estroma (verde), epitelios (rojo), y no tejidos (azul), y las categorías fueron definidas por el dibujo manualmente en la parte superiorde formación de imágenes (B). Después de dibujar en las imágenes de entrenamiento, un algoritmo para la segmentación del tejido se crea y se aplica al conjunto de formación de imágenes (C). Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Segmentación de la célula en nuclear, citoplasmática y la membrana compartimentos. El compartimiento nuclear se definió para la segmentación de las células mediante el establecimiento de un umbral mínimo para la contratinción de hematoxilina (A). El citoplasma se define en relación con el núcleo mediante el uso de algoritmos preestablecidos dentro del software (B). E-cadherina fue utilizado como un marcador de membrana y un mínimo significa umbral OD se aplicó para definir el compartimiento de membrana (C). Esta técnica todoOWS cuantificación simultánea de la expresión de proteínas en todos los compartimentos subcelulares (D). Haga clic aquí para ver una versión más grande de esta figura.
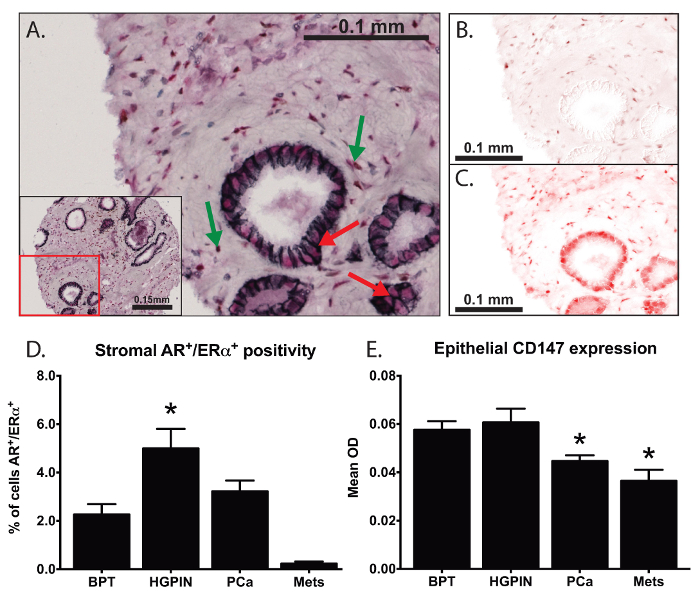
Figura 3: Análisis de Co-localización y de membrana específico de la expresión proteica. Multiplexado IHC se utilizó para investigar la expresión y localización de los receptores de andrógenos (AR), receptores de estrógenos alfa (ER), E-cadherina, y CD147 (A). Se usó DAB (cromógeno marrón) para marcar AR (B) y un cromógeno rojo utilizado para marcar ER (C). Hemos sido capaces de identificar biomarcadores superpuestas espacialmente (flechas verdes en A) y cuantificar (D) la proporción de células con nuclear co-localización de ER y ARdentro de estroma de próstata. Mediante el uso de E-cadherina (cromógeno negro) para definir la membrana plasmática (flechas rojas en A), se cuantificó la expresión de membrana específico de CD147 dentro de los tejidos de la próstata (E) 19. Unidireccional se utilizó el análisis de varianza (ANOVA) para el análisis estadístico, las barras de error reflejan el error estándar de la media, y los asteriscos representan p <0,05. Por favor, haga clic aquí para ver una versión más grande de esta figura.
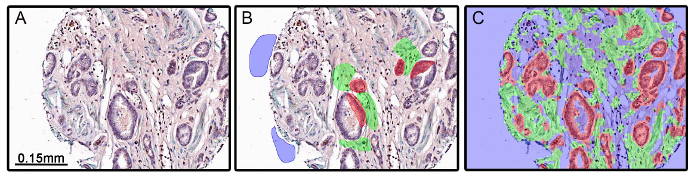
Figura 4: Segmentación inadecuada de tejido resultante de Diseño Experimental. tejidos de próstata benignos y malignos se tiñeron para la actina α-músculo liso (α-SMA; cromógeno verde), receptor de andrógenos (cromógeno rojo), y del receptor de andrógenos variante 7 (cromógeno DAB). a-SMA se utilizó comoun marcador de estroma y se reduce en algunos tumores, incluyendo el de próstata (A). Cuando se realizó el entrenamiento (B) y se aplicó el algoritmo de segmentación del tejido, el estroma y compartimentos epiteliales fueron inadecuadamente segmentado debido a la ausencia de tinción de α-SMA (C). Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
El uso de inmunohistoquímica tradicional para evaluar la expresión de proteínas está limitada por métodos subjetivos, semi-cuantitativos de análisis 22,23. plataformas de avance se han creado para el análisis de alto rendimiento de expresión de biomarcadores y la localización. la segmentación detallada de ambos tejidos y compartimentos subcelulares permite a los usuarios estudiar la expresión de biomarcadores, localización y co-localización con otros marcadores de interés. En estudios previos, se ha demostrado la utilidad de IHC y de imágenes multiespectrales, especialmente cuando se utiliza para estudiar las proteínas localizadas en el mismo compartimento celular 18,20,21. Cuando se combina con microarrays de tejidos 24, estas técnicas permiten la cuantificación más rápido y más objetiva de la expresión de proteínas que los permitidos por el análisis manual por un patólogo.
Un problema con el uso de inmunohistoquímica para la cuantificación de proteínas es irreproducibilidad de los resultados debido a la subjectiva la naturaleza del análisis y las diferencias inherentes en la técnica y reactivos. Una ventaja significativa de la utilización de plataformas como imágenes multiespectrales para la medición de la expresión de proteínas es la reproducibilidad de los resultados. Los datos de las plataformas de patología computarizados se correlacionan altamente con el análisis manual por un patólogo, mientras que la devolución de datos en un formato continuo y reducir sustancialmente el tiempo de trabajo, sobre todo cuando se trabaja con grandes tamaños de muestra 12,16,25. Los estudios han demostrado una alta concordancia general en los resultados entre diferentes plataformas multiespectrales 14. Además, hemos demostrado anteriormente que los resultados de tinción son altamente reproducibles incluso cuando no hay variabilidad en el número de proteínas investigó 12. El uso de plataformas de patología automatizados, tanto para la tinción y el análisis no eliminará todas las discrepancias en los resultados, tales como los derivados de anticuerpos creados hacia diferentes epítopos, pero estas plataformas reducen sustancialmente el sesgo y irreproducibilidad comúnmente asociado con inmunohistoquímica.
Hay medidas particulares dentro del protocolo que son esenciales para el retorno de resultados precisos y reproducibles. el diseño experimental apropiado a través de la selección de las proteínas que se incluirá como tejido o marcadores subcelulares es importante para la segmentación precisa. Al realizar el análisis de la positividad, la selección de un umbral inferior para la tinción positiva tiene un gran efecto en los resultados finales. Si bien la elección de un umbral de "On / Off" proteínas tales como factores de transcripción nuclear es bastante sencillo, la búsqueda de un umbral para las proteínas expresadas más heterogénea es difícil. Idealmente, esto debería llevarse a cabo en colaboración con un patólogo certificado por el consejo genitourinario o como una puntuación promedio a través de múltiples observadores para encontrar el umbral ideal para el análisis.
Es importante reconocer algunas limitaciones de las versiones actuales de esta tecnología. Si se anulaafinado el compartimiento citoplasma, tres enfoques pueden ser utilizados: (1) la tinción con un marcador-citoplasma específico, (2) de la tinción con un marcador específico de membrana y con el núcleo a membrana distancia marcador como citoplasma, y (3) usando un enfoque de dibujo para definir manualmente los límites del citoplasma en relación con el núcleo. A partir de nuestra experiencia, el uso de un marcador específico de membrana es la técnica más precisa. El enfoque de dibujo manual es normalmente precisa si los núcleos están situados en el centro o si el biomarcador de interés se distribuye uniformemente por todo el citoplasma. definir con precisión el citoplasma de las células del estroma como fibroblastos y células de músculo liso sigue siendo difícil y se debe tomar en consideración en el diseño de un experimento.
Otra limitación de esta tecnología es la dependencia de los núcleos para la segmentación de la célula. Si plano de la sección excluye el núcleo de una célula en particular, esta célula no se incluirá en el análisis. Si no haycitoplasma visible entre núcleos o grupos de núcleos adyacentes, estos son a menudo reconocido como un abultamiento grande nuclear, en lugar de núcleos distintos. En términos generales, contratinción de hematoxilina es normalmente suficiente para la segmentación nuclear aceptable, y la manipulación de la configuración de software, tales como umbral máximo para el tamaño de núcleo puede solucionar la mayoría de los problemas con la segmentación nuclear.
Una última limitación de plataformas automatizadas patología es poco fiable la segmentación de los tejidos resultantes de un mal diseño experimental. La importancia de la elección de los biomarcadores de tejidos y células apropiadas para responder a la pregunta de interés no puede ser exagerada. Como hemos demostrado en nuestros resultados representativos, un marcador epitelial o estromal que cambia significativamente la expresión entre los estados de la enfermedad puede plantear problemas al crear un algoritmo para la segmentación de tejidos. Vale la pena señalar que hay opciones alternativas cuando se analiza difícil como éste plantea. Por ejemplo, imag individuoES se pueden analizar mediante la elaboración manualmente todos los compartimentos de tejido, en lugar de mediante la aplicación de un algoritmo a partir de un conjunto de formación de imágenes. Esto proporciona la ventaja de segmentación que está perfectamente en línea con lo que el usuario desea, pero también introduce la subjetividad y puede aumentar significativamente el tiempo requerido para terminar un análisis. el diseño experimental apropiado es la forma más fácil de acelerar el análisis y maximizar la precisión de la segmentación del tejido y célula.
Esta tecnología y el protocolo tienen muchas aplicaciones futuras. Numerosos biomarcadores para determinados estados de la enfermedad han sido identificados pero no validado. análisis objetivo de alto rendimiento con plataformas multiespectrales facilita la validación de estos biomarcadores. Además, la evaluación de la expresión y co-localización de múltiples proteínas puede proporcionar conocimientos sobre las vías de señalización mal entendido. Sin lugar a dudas, lo que reduce la subjetividad inherente asociado con el análisis inmunohistoquímico de staining es valiosa para comprender la expresión y localización de una amplia gama de marcadores de proteínas.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Los autores agradecen a la Universidad de Wisconsin Iniciativas de Investigación Traslacional en el laboratorio de patología, en parte apoyado por el Departamento de Patología y Medicina de Laboratorio de la Universidad de Washington y la subvención CA014520 UWCCC P30, para el uso de sus instalaciones y servicios.
Materiales
| Name | Company | Catalog Number | Comments |
| Xylene | Fisher Chemical | X3F1GAL | NFPA rating: Health – 2, Fire – 3 , Reactivity – 0 |
| Ethyl Alcohol-200 proof | Fisher Scientific | 4355223 | NFPA rating: Health – 0, Fire – 3 , Reactivity – 0 |
| Tris Base | Fisher Scientific | BP152-500 | NFPA rating: Health – 2, Fire – 0 , Reactivity – 0 |
| Tris Hydroxymethyl aminomethane HCl | Fisher Scientific | BP153-1 | NFPA rating: Health – 2, Fire – 0 , Reactivity – 0 |
| Tween 20 | Chem-Impex | 1512 | NFPA rating: Health – 0, Fire – 1 , Reactivity – 0 |
| Phosphate-buffered saline | Fisher Scientific | BP2944-100 | NFPA rating: Health – 1, Fire – 0 , Reactivity – 0 |
| Peroxidazed | Biocare Medical | PX968 | Avoid contact with skin and eyes. May cause skin irritation and eye damage. |
| Estrogen Receptor alpha | Thermo Fisher Scientific-Labvision | RM9101 | Not classified as hazardous |
| Androgen Receptor | SCBT | sc-816 | Not classified as hazardous |
| CD147 | Biodesign | P87535M | Not classified as hazardous |
| E-cadherin | Dako | M3612 | Not classified as hazardous |
| Renoir Red Andibody Diluent | Biocare Medical | PD904 | It is specially designed to work with Tris-based antibodies |
| DeCloaking Chamber | Biocare Medical | Model DC2002 | Take normal precautions for using a pressure cooker |
| Barrier pen-Immuno Edge | Vector Labs | H-4000 | |
| Denaturing Kit-Elution step | Biocare Medical | DNS001H | Not classified as hazardous |
| Mach 2 Goat anti-Rabbit HRP Polymer | Biocare Medical | RHRP520 | Not classified as hazardous |
| Mach 2 Goat anti-Rabbit AP Polymer | Biocare Medical | RALP525 | Not classified as hazardous |
| Mach 2 Goat anti-Mouse HRP Polymer | Biocare Medical | M3M530 | Not classified as hazardous |
| Betazoid DAB Chromogen Kit | Biocare Medical | BDB2004 | 1. DAB is known to be a suspected carcinogen. 2. Do not expose DAB components to strong light or direct sunlight. 3. Wear appropriate personal protective equipment and clothing. 4. DAB may cause sensitization of skin. Avoid contact with skin and eyes. 5. Observe all federal, state and local environmental regarding disposal |
| Warp Red Chromogen Kit | Biocare Medical | WR806 | Corrosive. Acid that may cause skin irritation or eye damage. |
| Vina Green Chromogen Kit | Biocare Medical | BRR807 | Harmful if swallowed |
| Bajoran Purple Chromogen Kit | Biocare Medical | BJP807 | Flammable liquid. Keep away from heat, flames and sparks. Harmful by ingestion or absorption. Avoid contact with skin or eyes, and avoid inhalation. |
| Cat Hematoxylin | Biocare Medical | CATHE | Purple solution with a mild acetic acid (vinegar) scent. May be irritating to skin or eyes. Avoid contact with skin and eyes. Avoid ingestion. |
| XYL Mounting Media | Richard Allen | 8312-4 | NFPA rating: Health – 2, Fire – 3 , Reactivity – 0 |
| 1.5 Coverslips | Fisher Brand | 22266858 | Sharp edges |
| Incubation (Humidity)Chamber | obsolete | obsolete | Multiple vendors available |
| Convection Oven | Stabil- Therm | C-4008-Q | |
| Background Punisher Blocking Reagent | Biocare Medical | BP974 | This product is not classified as hazardous. |
| inForm software | PerkinElmer | CLS135781 | Primary multispectral imaging software used in manuscript |
| Nuance software | PerkinElmer | Nuance EX | Software used for making spectral libraries within manuscript |
| Vectra microscope and slide scanner | PerkinElmer | VECTRA | Automated slide scanner and microscope for obtaining IM3 image cubes |
Referencias
- Valdman, A., et al. Expression of redox pathway enzymes in human prostatic tissue. Anal Quant Cytol Histol. 31 (6), 367-374 (2009).
- Rimm, D. L., Camp, R. L., Charette, L. A., Olsen, D. A., Provost, E. Amplification of tissue by construction of tissue microarrays. Exp Mol Pathol. 70 (3), 255-264 (2001).
- Jonmarker, S., et al. Expression of PDX-1 in prostate cancer, prostatic intraepithelial neoplasia and benign prostatic tissue. APMIS. 116 (6), 491-498 (2008).
- McCarty, K. S., Miller, L. S., Cox, E. B., Konrath, J., McCarty, K. S. Sr Estrogen receptor analyses. Correlation of biochemical and immunohistochemical methods using monoclonal antireceptor antibodies. Arch Pathol Lab Med. 109 (8), 716-721 (1985).
- Volante, M., et al. Somatostatin receptor type 2A immunohistochemistry in neuroendocrine tumors: a proposal of scoring system correlated with somatostatin receptor scintigraphy. Mod Pathol. 20 (11), 1172-1182 (2007).
- Muris, J. J., et al. Immunohistochemical profiling of caspase signaling pathways predicts clinical response to chemotherapy in primary nodal diffuse large B-cell lymphomas. Blood. 105 (7), 2916-2923 (2005).
- Jaraj, S. J., et al. Intra- and interobserver reproducibility of interpretation of immunohistochemical stains of prostate cancer. Virchows Arch. 455 (4), 375-381 (2009).
- Nakane, P. K., Pierce, G. B. Enzyme-labeled antibodies: preparation and application for the localization of antigens. J Histochem Cytochem. 14 (12), 929-931 (1966).
- Peters, T. J. Investigation of tissue organelles by a combination of analytical subcellular fractionation and enzymic microanalysis: a new approach to pathology. J Clin Pathol. 34 (1), 1-12 (1981).
- Emmert-Buck, M. R., et al. Laser Capture Microdissection. Science. 274 (5289), 998-1001 (1996).
- Waters, J. C. Accuracy and precision in quantitative fluorescence microscopy. J Cell Biol. 185 (7), 1135-1148 (2009).
- Huang, W., Hennrick, K., Drew, S. A colorful future of quantitative pathology: validation of Vectra technology using chromogenic multiplexed immunohistochemistry and prostate tissue microarrays. Hum Pathol. 44 (1), 29-38 (2013).
- Rimm, D. L. C-Path: A Watson-Like Visit to the Pathology Lab. Science Translational Medicine. 3 (108), (2011).
- Fiore, C., et al. Utility of multispectral imaging in automated quantitative scoring of immunohistochemistry. J Clin Pathol. 65 (6), 496-502 (2012).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: A review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Rizzardi, A. E., et al. Quantitative comparison of immunohistochemical staining measured by digital image analysis versus pathologist visual scoring. Diagn Pathol. 7, 42(2012).
- Bauman, T. M., et al. Characterization of fibrillar collagens and extracellular matrix of glandular benign prostatic hyperplasia nodules. PLoS One. 9 (10), e109102(2014).
- Bauman, T. M., et al. Beta-catenin is elevated in human benign prostatic hyperplasia specimens compared to histologically normal prostate tissue. Am J Clin Exp Urol. 2 (4), 313-322 (2014).
- Bauman, T. M., Ewald, J. A., Huang, W., Ricke, W. A. CD147 expression predicts biochemical recurrence after prostatectomy independent of histologic and pathologic features. BMC Cancer. 15 (1), 549(2015).
- Bauman, T. M., et al. Finasteride treatment alters tissue specific androgen receptor expression in prostate tissues. Prostate. 74 (9), 923-932 (2014).
- Nicholson, T. M., Sehgal, P. D., Drew, S. A., Huang, W., Ricke, W. A. Sex steroid receptor expression and localization in benign prostatic hyperplasia varies with tissue compartment. Differentiation. 85 (4-5), 140-149 (2013).
- Taylor, C. R., Levenson, R. M. Quantification of immunohistochemistry--issues concerning methods, utility and semiquantitative assessment II. Histopathology. 49 (4), 411-424 (2006).
- Matos, L. L., Trufelli, D. C., de Matos, M. G., da Silva Pinhal, M. A. Immunohistochemistry as an important tool in biomarkers detection and clinical practice. Biomark Insights. 5, 9-20 (2010).
- Kononen, J., et al. Tissue microarrays for high-throughput molecular profiling of tumor specimens. Nat Med. 4 (7), 844-847 (1998).
- Ong, C. W., et al. Computer-assisted pathological immunohistochemistry scoring is more time-effective than conventional scoring, but provides no analytical advantage. Histopathology. 56 (4), 523-529 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados