Method Article
Generazione di cellule staminali Integrazione-libero umano pluripotenti indotte Utilizzando capelli derivati da cheratinociti
In questo articolo
Riepilogo
This manuscript provides a step-by-step procedure for the derivation and maintenance of human keratinocytes from plucked hair and subsequent generation of integration-free human induced pluripotent stem cells (hiPSCs) by episomal vectors.
Abstract
Recent advances in reprogramming allow us to turn somatic cells into human induced pluripotent stem cells (hiPSCs). Disease modeling using patient-specific hiPSCs allows the study of the underlying mechanism for pathogenesis, also providing a platform for the development of in vitro drug screening and gene therapy to improve treatment options. The promising potential of hiPSCs for regenerative medicine is also evident from the increasing number of publications (>7000) on iPSCs in recent years. Various cell types from distinct lineages have been successfully used for hiPSC generation, including skin fibroblasts, hematopoietic cells and epidermal keratinocytes. While skin biopsies and blood collection are routinely performed in many labs as a source of somatic cells for the generation of hiPSCs, the collection and subsequent derivation of hair keratinocytes are less commonly used. Hair-derived keratinocytes represent a non-invasive approach to obtain cell samples from patients. Here we outline a simple non-invasive method for the derivation of keratinocytes from plucked hair. We also provide instructions for maintenance of keratinocytes and subsequent reprogramming to generate integration-free hiPSC using episomal vectors.
Introduzione
La scoperta di umani cellule staminali pluripotenti indotte (hiPSCs) ha rivoluzionato il campo della medicina rigenerativa, che fornisce un metodo fattibile per la generazione di cellule staminali paziente-specifici 1-3. hiPSCs sono stati generati con successo da vari tipi di cellule somatiche, inclusi i fibroblasti, cellule ematopoietiche 4,5 6,7, cellule epiteliali renali di urina 8 e cheratinociti 9,10. Fino ad oggi, i fibroblasti della pelle e cellule ematopoietiche rappresentano le fonti di cellule più comunemente usati per la generazione iPSCs paziente-specifici. Probabilmente, questo è dovuto al fatto che le biopsie cutanee e raccolta del sangue sono procedure mediche di routine e grandi biobanche di sangue o di pelle campioni di pazienti sono stati stabiliti in molti paesi.
A differenza di cellule del sangue e fibroblasti cutanei che richiedono metodi di estrazione invasivi, cheratinociti rappresentano un tipo di cellula facilmente accessibile per la generazione hiPSC. Keratinocytes sono cellule epiteliali ricchi di cheratina che formano la barriera epidermica esterno della pelle e si trovano anche in unghie e capelli 11. In particolare, cheratinociti possono essere trovati sulla guaina esterna della radice (ORS) dei follicoli piliferi, uno strato cellulare esterno che copriva il fusto del capello insieme con la guaina interna della radice (IRS) celle (12, figura 1). Come la raccolta dei capelli è una procedura semplice che non richiede l'assistenza di personale medico, si prevede la possibilità per i pazienti di recuperare ed inviare i propri campioni di capelli ai laboratori, il che faciliterà enormemente la raccolta dei campioni dei pazienti per la generazione hiPSC. Cheratinociti epidermici hanno una maggiore efficienza riprogrammazione e cinetica riprogrammazione più veloci rispetto ai fibroblasti, aggiungendo i vantaggi di utilizzare cheratinociti come le cellule di partenza per hiPSC generazione 9,13. Inoltre, hiPSCs può essere generato anche utilizzando altre popolazioni di cellule all'interno del follicolo pilifero,comprese le cellule della papilla dermica situate alla base del 14,15 follicolo pilifero.
Precedenti rapporti di generazione IPSC utilizzando cellule dei capelli derivate utilizzano spesso metodi di riprogrammazione retrovirali o basati lentivirali-9,14,15. Tuttavia, questi metodi virali introducono integrazione genomica indesiderabile dei transgeni esteri durante la riprogrammazione. In confronto, l'uso di vettori episomiale rappresenta un metodo di riprogrammazione fattibile, non virale per generare iPSCs senza integrazione 4. Abbiamo già messo a punto un metodo semplice, economico e non virale per riprogrammare efficientemente cheratinociti in hiPSCs utilizzando vettori episomiale 13. Qui forniamo un protocollo dettagliato per la generazione di hiPSCs cheratinociti derivate, compresa la derivazione di cheratinociti da capelli strappato, espansione e manutenzione dei cheratinociti e successiva riprogrammazione per generare hiPSCs.
Protocollo
La raccolta del campione di capelli umani da individui richiede l'approvazione etica dal comitato etico della ricerca umana nelle istituzioni ospitanti e deve essere fatto in conformità con le linee guida istituzionali.
1. Isolamento dei cheratinociti da capelli a pizzico
- Disgelo matrice extracellulare soluzione (ECM) (cioè, Matrigel) su ghiaccio O / N.
- Utilizzando puntali delle pipette pre-refrigerati, aggiungere 200 ml soluzione ECM per 12 ml di acqua refrigerata a medio DMEM / F12. Coat un 12-pozzetti con 1 ml di soluzione di ECM diluita in ciascun pozzetto. Incubare la piastra O / N rivestito a 37 ° C. Lavare la piastra ricoperta di pre-riscaldato DMEM / F12 medio prima dell'uso.
- Raccogliere i capelli umani primaria dalla testa del soggetto posizionando le dita vicino alla radice dei capelli e spennare i capelli in un movimento veloce e regolare. Controllare per confermare che ogni capelli raccolti contiene un follicolo pilifero intatta e determinare la fase di crescita delle Nazioni Unite capellider un microscopio da dissezione (Figura 1).
- Raccogliere 5-10 peli anagen da ogni individuo per estrarre cheratinociti. Processo pizzicato peli più presto possibile garantire la corretta derivazione dei cheratinociti.
Nota: La Figura 1 illustra la morfologia di capelli in diverse fasi del ciclo di crescita. Capelli anagen è in fase di crescita, con uno strato esterno visibile dell'epitelio circonda la lunghezza dei capelli, noto come ORS. D'altra parte, i capelli telogen rappresenta capelli nella fase di riposo e non ha un ORS visibile, ma ha una chiara palla di cellule che ricoprono la radice dei capelli, noto come il rigonfiamento telogen.
- Raccogliere 5-10 peli anagen da ogni individuo per estrarre cheratinociti. Processo pizzicato peli più presto possibile garantire la corretta derivazione dei cheratinociti.
- Porre i campioni di capelli in una capsula petri contenente 10 ml di antibiotico Mix per lavare per 5 min a temperatura ambiente.
- Utilizzando forbici e pinze sterilizzate, tagliare eccessiva fusto del capello, lasciando un frammento di capelli con un follicolo pilifero intatto e dintorni 0,5-1 cm di fusto del capello.
- Attentamente TRAnsfer frammento 1 capelli in ciascun pozzetto della piastra ECM rivestite con pinze.
- Con una pipetta 1 ml, aggiungere circa 3 gocce di Knockout siero sostituzione (KSR) medio (~ 100-200 mL) ai campioni di capelli.
Nota: Mantenere i frammenti di capelli a stretto contatto con la superficie della piastra per consentire l'attaccamento e la successiva escrescenza cheratinociti. L'aggiunta eccessiva della carta in questa fase può provoca i frammenti di capelli di galleggiare e diminuisce il rischio di attacco di capelli di successo. - Incubare i campioni di capelli in un incubatore a 37 ° con il 5% di CO 2 e consentire i follicoli dei capelli per fissare O / N. Il giorno dopo, aggiungere delicatamente altri 3 gocce di KSR media (~ 100-200 ml) per mantenere i campioni di capelli umidi.
- Osservare i campioni di capelli ogni giorno per confermare che i follicoli dei capelli hanno attaccato con successo. Generalmente attaccamento dei follicoli dei capelli è evidente entro 1-3 giorni. Capelli spessi sono di solito più facili da montare rispetto a peli fini, come peli sottili hanno una maggioretendenza a galleggiare che ostacolano il processo di attaccamento.
- Una volta che i capelli sono attaccato alla piastra rivestita, aggiungere 500 ml di KSR mezzo al pozzo. Prestare particolare attenzione quando si aggiunge il mezzo come campioni di capelli potrebbero non essere montato saldamente. Da questo punto, cambiare supporto ogni 2 giorni.
Nota: escrescenze cheratinociti devono essere visibili dalla regione ORS del frammento capelli dopo 3-7 giorni (Figura 2A-B). Lasciare i cheratinociti a crescere fino a 14 giorni prima passaging le cellule come descritto nella sezione successiva.
- Una volta che i capelli sono attaccato alla piastra rivestita, aggiungere 500 ml di KSR mezzo al pozzo. Prestare particolare attenzione quando si aggiunge il mezzo come campioni di capelli potrebbero non essere montato saldamente. Da questo punto, cambiare supporto ogni 2 giorni.
2. Manutenzione e Passaging dei cheratinociti
- Pre-coat un 6-pozzetti utilizzando il kit Coating Matrix. Aggiungere 680 pl di mezzo di diluizione dal kit a ciascun pozzetto, seguito da 6,8 ml di soluzione di rivestimento Matrix. Agitare la piastra per assicurare una distribuzione uniforme della soluzione di rivestimento Matrix sulla piastra.
- Il lamierino rivestito per incubare per 30 minuti a RT. Rimuoverela soluzione di rivestimento Matrix prima placcatura dei cheratinociti.
- Dissociarsi cheratinociti in una sospensione di cellule singole con 500 ml di 0,25% tripsina per pozzetto per 2-5 minuti a 37 ° C. Disattivare la tripsina con 500 ml di mezzo contenente siero (ad esempio, siero fetale bovino (FBS) medium) e raccogliere in una provetta sterile da 15 ml. Rimuovere il frammento capelli con pinze sterilizzate.
- Determinare il numero di cellule raccolte utilizzando un emocitometro.
- Centrifugare le cellule per 3 min a 200 x g. Lavare le cellule con PBS e centrifugazione ripetere. Aspirare il PBS e risospendere il pellet cellulare in fresco medio dei cheratinociti.
- Piatto giù 4 × 10 4 cheratinociti in un pozzetto della piastra ECM rivestite in presenza di cheratinociti media. Mantenere cheratinociti in un incubatore a 37 ° con il 5% di CO 2 (Figura 2C). Modificare i media ogni giorno e di passaggio celle quando la cultura è ~ 80% confluenti, WHIch può richiedere circa 2-7 giorni a seconda del numero di passaggio e la proliferazione tasso dei cheratinociti. I cheratinociti derivati dovrebbero essere estesi per generare scorte congelate sufficienti utilizzando metodi di crioconservazione lento congelamento standard di 16.
Nota: Le condizioni di coltura descritte supportano l'espansione della popolazione omogenea dei cheratinociti con la tipica morfologia ciottoli (Figura 2C). Di tanto in tanto, qualche cheratinociti differenziati, con grande e piatta morfologia e hanno perso potenziale replicativo, possono essere osservati in cultura. Caratterizzazione dei cheratinociti può essere eseguita mediante immunocolorazione utilizzando anticorpi contro citocheratina 14 come descritto in precedenza 17. Linee cellulari di cheratinociti Fondata dovrebbero essere testati per micoplasma dopo 1-3 passaggi utilizzando il kit disponibile in commercio.
3. Generazione di hiPSCs da cheratinociti Utilizzando episomiale Vettori
- Pre-coat un 6-pozzetti con0,1% di gelatina (2 ml / pozzetto). Consentire la gelatina per rivestire per almeno 20 minuti a temperatura ambiente.
- Per esperimento riprogrammazione, uso cheratinociti non oltre il passaggio 6. Dissociate cheratinociti in una sospensione di cellule singole con 1 ml di 0,25% tripsina per bene per 2-5 minuti a 37 ° C. Inattivare tripsina con 1 ml di supporti contenenti siero (ad esempio, FBS media) e raccogliere in una provetta da 15 ml.
- Celle di centrifugazione per 3 min a 200 xg e risospendere le cellule in cheratinociti media. Piastra 1 × 10 5 -1.5 × 10 5 cheratinociti per pozzetto in una piastra O / N gelatinizzato.
- Il giorno successivo (giorno 0), controllare la piastra per garantire cellule sono il 50-60% confluenti.
Nota: Se cheratinociti sono meno di ~ 50% confluenti potrebbero non crescono bene dopo la trasfezione. - Il giorno 0, effettuare trasfezione dei vettori episomiale riprogrammazione sui cheratinociti, utilizzando un rapporto di 8 microlitri trasfezione reagente 2 ug di DNA plasmidico totale.
- Prendete 0,5 mg per ciascuno dei 4 plasmidi (pCXLE-eGFP, pCXLE-hOct3 / 4-shP53F, pCXLE-HSK, pCXLE-Hul) e diluire in 100 ml di media Serum ridotta (per esempio, OPTI-MEM).
Nota: Il plasmide pCXLE-eGFP è utilizzato per monitorare l'efficienza di trasfezione di cheratinociti e non è necessaria per la riprogrammazione successo. - Aggiungere 8 ml di reagente di trasfezione di DNA diluito. Mescolare muovendo il tubo. Lasciare la miscela di reazione DNA riposare indisturbati a temperatura ambiente per 15 min.
- Sostituire il supporto dei cheratinociti con terreno fresco.
- Aggiungere delicatamente la miscela di reazione del DNA di coltura di cheratinociti e incubare a 37 ° C incubatore con 5% di CO 2 per 4 ore.
Nota: non è consigliabile eseguire prolungata trasfezione O / N nei cheratinociti in quanto presentano bassa sopravvivenza dopo la trasfezione. - Dopo 4 ore dopo la trasfezione, sostituire il mezzo di cheratinociti con terreno fresco.
- Prendete 0,5 mg per ciascuno dei 4 plasmidi (pCXLE-eGFP, pCXLE-hOct3 / 4-shP53F, pCXLE-HSK, pCXLE-Hul) e diluire in 100 ml di media Serum ridotta (per esempio, OPTI-MEM).
- Il giorno 1 (1 giorno post-transfection), cambiare il mezzo cheratinociti e verificare l'efficienza di trasfezione stimando la percentuale di cellule positive eGFP sotto un microscopio a fluorescenza. Tipicamente> 60-70% efficienza di trasfezione si osserva per cheratinociti primari che utilizzano questo protocollo.
- Il giorno 2, ripetere trasfezione come descritto al punto 3.5. Due trasfezioni consecutivi sarebbero tipicamente raggiungere il> 90% efficienza di trasfezione. Coltura di cheratinociti solito raggiunge la confluenza prima della fine del secondo trasfezione.
- Preparare un piatto 10 centimetri di topo alimentatore fibroblasti embrionali (MEF) per ogni reazione riprogrammazione. Preparare alimentatori MEF mitoticamente inattivati come descritto in precedenza 18.
- Pre-coat un piatto 10 centimetri con 5 ml di 0,1% di gelatina per almeno 20 min. Tavola 4 × 10 6 MEF mitoticamente inattivato per 10 cm piatto in FBS medio. Permettono alle cellule di stabilirsi e di allegare O / N in un incubatore a 37 ° con il 5% di CO 2.
- Il giorno 3, harconferire cheratinociti con 0,25% tripsina come descritto al punto 3.2 e 3.3. Risospendere cheratinociti in KSR medio e piastra 90% dei cheratinociti da 1 bene di un 6-pozzetti in un piatto alimentatore MEF 10 centimetri con KSR media.
- Dal 4 ° giorno in poi, cambia il mezzo KSR ogni giorno.
- In alternativa, dopo il giorno 18, sostituire terreno di coltura ogni due giorni.
Nota: cheratinociti non-riprogrammato cesseranno di proliferare, mentre le colonie hiPSC con contorno definito che assomigliano cellule staminali embrionali umane (hESC) emergeranno dal giorno 14-21.
- In alternativa, dopo il giorno 18, sostituire terreno di coltura ogni due giorni.
- Isolare colonie hiPSC da dissezione manuale dal giorno 21 in poi. Evitare colonie hiPSC che sono strettamente raggruppati insieme e solo raccolgono le colonie hiPSC che sono nettamente separati dagli altri.
Nota: le colonie hiPSC possono essere presi in precedenza, ma la colonia con confini definiti potrebbero non essere evidente in precedenti momenti viste le ridotte dimensioni della colonia. Efficienze Riprogrammazione possono variare tra diffcheratinociti di erenti paziente, ed è influenzato anche da altri fattori, quali il numero di passaggio o il tasso di proliferazione dei cheratinociti.- Per la dissezione manuale di colonie hiPSC, utilizzare un ago 26G per tagliare colonie hiPSC sotto un microscopio da dissezione. Tagliare una colonia hiPSC in piccoli pezzi intorno a 300-600 micron di lunghezza. Trasferire i pezzi da una colonia hiPSC in una nuova piastra alimentatore MEF (2,5 × 10 4 MEF / cm 2) per stabilire una linea clonale di hiPSCs. Inizialmente, la cultura ogni linea clonale hiPSC in un pozzetto di una piastra a 12 o coltura di organi e piatti, quindi espandere a 6 formato pozzetti.
- Mantenere linee clonali stabilite di hiPSCs in KSR medio su alimentatori MEF. Una volta che la cultura raggiungere il 70 - 80% di confluenza con le colonie hiPSC ~ 1,5 millimetri di diametro, hiPSCs passaggio utilizzando metodi standard enzimatici passaging (Accutase, dispasi o Collagenase IV) secondo le istruzioni del produttore.
- Caratterizzare stabilito linea hiPSCs per confermare la pluripotenza e la loro capacità di differenziarsi in cellule rappresentante dei tre foglietti embrionali in vitro e / o in vivo 13,19. Inoltre, le linee hiPSC routine test stabilite per micoplasma utilizzando kit disponibili in commercio per garantire che siano privi di agenti patogeni. Ordinariamente monitorare la stabilità del genoma di linee hiPSC da cariotipo o numero di copie variazione basata su array (CNV) analisi.
Risultati
I capelli passa attraverso 3 diverse fasi del ciclo di sviluppo: anagen (fase di crescita), catagen (la fase di regressione) e telogen (fase di riposo) il 20,21. Il follicolo pilifero anagen contiene più strati di epitelio; questi strati comprendono i ORS, IRS e il fusto del capello (Figura 1). Capelli Anagen alla fine subisce la transizione alla fase catagen, che è caratterizzato da apoptosi delle ORS e alla conclusione della differenziazione fusto del capello. Infine, la transizione capelli catagen alla fase telogen, dove cessa apoptosi e il follicolo diventa telogen di riposo con un telogen caratteristico rigonfiamento 20,21.
La Figura 1 illustra la morfologia di un capello telogen e un pelo anagen. Noi di solito utilizziamo capelli anagen per cheratinociti derivazione. A seguito di tale protocollo, escrescenze cheratinociti si possono osservare già dopo tre giorni dopo il collegamento dei capelli (Figura 2A) e continueranno a proliferate (Figura 2B). Nella nostra esperienza, alcuni peli anagen possono non riuscire a collegare o non osservare cheratinociti escrescenza; quindi raccogliere almeno 5-10 anageni di ogni individuo al fine di garantire il successo isolamento dei cheratinociti. Successivamente, i cheratinociti possono essere diversi passaggi su una nuova piastra e mantenuti per passaggi multipli (Figura 2C). È importante notare che non vi è una migliore crescita dei cheratinociti su una matrice di rivestimento con cheratinociti terreno come descritto nella sezione 2, rispetto al Matrigel con KSR media.
Dopo l'espansione, i cheratinociti possono essere riprogrammate per generare hiPSCs come descritto nel capitolo 3. Figura 3A mostra un cheratinociti derivati dal tipico hiPSC colonia dopo 32 giorni di riprogrammazione. È comune osservare alcuni differenziazione al centro della colonia hiPSC. Una volta raccolte manualmente, i hiPSCs derivati tipicamente presentino un alto rapporto nucleo citoplasma e una colonia definito limiteary (Figura 3B). Linee cellulari stabilizzate di hiPSCs possono essere caratterizzati per pluripotency come descritto in precedenza 13,19, come l'espressione di marcatori pluripotenti OCT4, Nanog e TRA-160 (Figura 3C-E).
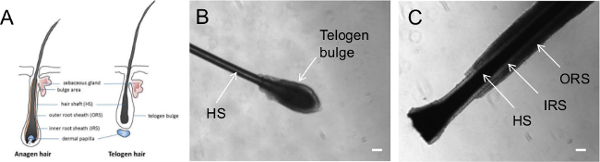
Figura 1. Immagini rappresentative della peli pizzicate in diverse fasi di crescita. (A) Diagramma capelli in fase anagen o telogen. Immagini a contrasto di fase che mostrano un capello strappato a (B) fase telogen e (C) anagen di fase. HS albero = capelli; IRS = interno Root guaina; ORS = esterna Root guaina. Scala bar = 100 micron. Cliccate qui per vedere una versione più grande di questa figura.

Immagini Figura 2. Immagini rappresentative della cheratinociti-capelli derivati. Contrasto di fase di un capello strappato placcato giù per (A) 3 giorni e (B) 10 giorni. Frecce bianche segnano la conseguenza di cheratinociti da un capello strappato. Immagine contrasto (C) Fase del Day 3 coltura di cheratinociti con una morfologia tipica di ciottoli dopo passaging. Scala bar = 100 micron. Cliccate qui per vedere una versione più grande di questa figura.

Figura 3. Immagini rappresentative della hiPSCs cheratinociti di derivazione. (A) contrasto di fase immagine di una cultura riprogrammazione Giorno 32 mostra una cheratinociti derivati da hiPSC coLony. (B) selezionato manualmente hiPSCs cheratinociti derivati da con morfologia simile a hESC. Immunostaining di hiPSCs con pennarelli pluripotenti (C) Nanog e (D) OCT4 e (E) TRA-160 in hiPSCs cheratinociti-derivati. Scala bar = 100 micron. Cliccate qui per vedere una versione più grande di questa figura.
Discussione
Generazione di hiPSCs paziente-specifici offre un approccio unico per lo studio patogenesi nei tipi di cellule malate in vitro, e fornisce anche una piattaforma per lo screening di stupefacenti per identificare nuove molecole in grado di salvare i fenotipi di malattia. Questo approccio di modellazione malattia utilizzando hiPSCs ha dato risultati promettenti per una varietà di malattie, tra cui la sindrome del QT lungo, malattia di Huntington, il morbo di Parkinson e la sclerosi laterale amiotrofica 22. Diverse iniziative sono già in corso per stabilire le librerie su larga scala di hiPSCs paziente-specifici, tra cui consorzi in USA, Europa, Australia, Cina, Corea del Sud e Giappone 23,24.
Qui un protocollo per stabilire hiPSCs da cheratinociti-capelli derivata viene descritto, che ha il potenziale di facilitare e accelerate la creazione di librerie di larga scala di hiPSCs malattie specifiche. Questo protocollo offre due vantaggi: in primo luogo, il episomialesistema utilizzato in questo protocollo genera hiPSCs senza integrazione utilizzando un cocktail di sei fattori di riprogrammazione (OCT4, SOX2, Klf4, L-MYC, LIN28, shRNA per p53) relativamente alta efficienza 4. Rispetto ai metodi di riprogrammazione retrovirali o lentivirali-mediata, l'uso di episomiale vettore evita l'integrazione genomica indesiderabile dei transgeni stranieri durante la riprogrammazione., Pertanto, molte iniziative favoriscono l'uso di metodi di riprogrammazione senza integrazione per l'istituzione di biblioteche hiPSC su larga scala, come ad esempio vettori episomiale, virus Sendai o mRNA, rispetto ai metodi di riprogrammazione integrative 23.
In secondo luogo, i capelli è facilmente accessibile e può essere raccolto dai pazienti stessi, senza l'utilizzo di procedure invasive o la presenza di personale medico. Questo offre un'opportunità unica per i pazienti provenienti da diverse regioni per la raccolta e la posta nei propri campioni di capelli al laboratorio per cheratinociti derivazione e la successiva riprogrammazione. A sostegno della fattibilità di questa strategia, i nostri dati non pubblicati indicano che i cheratinociti possono essere isolati con successo da capelli pizzico che è stato conservato a 4 ° C per un massimo di 10 giorni.
La metodologia qui descritta permette la derivazione di cheratinociti da capelli spennati. Mentre cheratinociti possono derivare da un solo capello, la nostra raccomandazione è di raccogliere almeno 5 anageni per cheratinociti derivazione. Una limitazione di questo protocollo è che alcuni peli non possono allegare bene e cheratinociti potrebbe non riuscire a crescere. Capelli più spesso tende a collegare meglio, mentre i capelli bene richiede una ridotta quantità di terreno di coltura durante la placcatura per evitare galleggiante e migliorare l'attaccamento. Una volta che i cheratinociti sono derivati, si consiglia di ampliare e congelare giù scorte con metodi di crioconservazione lento congelamento standard di 16. Riprogrammazione successiva di cheratinociti può essere effettuata seguenti passi descritti in questo protocollo. È importantenotare che il tasso di proliferazione delle cellule di partenza può influenzare l'efficienza riprogrammazione, con una diminuzione di efficienza riprogrammazione osservato nel tardo passaggi come cellule raggiungono la senescenza cellulare 25-27. A questo proposito, utilizzare cheratinociti entro passaggio 6 per esperimenti di riprogrammazione. Inoltre, l'efficienza di riprogrammazione possono variare tra i cheratinociti diverso dell'individuo.
Studi recenti suggeriscono diffusa mosaicismo genetico in diversi tipi di tessuti somatici 28, con ~ 30% di fibroblasti cutanei segnalati per avere somatica CNVs nel genoma 29. Questi CNVs possono essere causati da errori nella replicazione del DNA, riparazione del DNA o trasposone mobilitazione. È anche possibile che l'esposizione UV prolungata alla pelle può causare CNVs aggiuntivi in cellule epidermiche inclusi i fibroblasti e cheratinociti, ma la portata di questo non è ben compreso. Come CNVs in cheratinociti saranno riportati durante il processo di riprogrammazione, è important per eseguire cariotipo di routine o analisi CNV per garantire che le hiPSCs stabiliti mantenere la stabilità genomica. In sintesi, qui abbiamo descritto le procedure per la generazione di hiPSCs da cheratinociti-capelli derivati, che potrebbero essere utilizzati per la modellazione della malattia e della medicina rigenerativa.
Divulgazioni
The authors have no conflict of interest.
Riconoscimenti
The authors wish to thank Harene Ranjithakumaran and Stacey Jackson for technical support. This work was supported in part by grants from the National Health and Medical Research Council (R.C.B. Wong, A. Pébay), the University of Melbourne (R.C.B. Wong), Retina Australia (R.C.B. Wong, S.S.C. Hung, A. Pébay) and the Ophthalmic Research Institute of Australia (R.C.B. Wong, S.S.C. Hung, A. Pébay); Australian Research Council Future Fellowship (A. Pébay, FT140100047), Cranbourne Foundation Fellowship (R.C.B. Wong); intramural funding from the National Institutes for Health (R.C.B. Wong, S.S.C. Hung) and operational infrastructure support from the Victorian Government.
Materiali
| Name | Company | Catalog Number | Comments |
| Antibiotic Mix: | |||
| 250 ng/ml Antimycotic amphotericin B | Sigma | A2942-20ml | Antibiotic mix is made up in PBS. |
| 1X Penicillin/Streptomycin | Invitrogen | 15140-122 | |
| PBS (-) | Invitrogen | 14190-144 | |
| Knockout Serum Replacement (KSR) medium: | KSR medium is filtered using Stericup (Millipore, #SCGPU05RE) before use. bFGF is added fresh to the media before use. | ||
| 20% knockout serum replacement (KSR) | Invitrogen | 10828-028 | |
| DMEM/F12 with glutamax | Invitrogen | 10565-042 | |
| 1× MEM non-essential amino acid | Invitrogen | 11140-050 | |
| 0.5× Penicillin/Streptomycin | Invitrogen | 15140-122 | |
| 0.1 mM β-mercaptoethanol | Invitrogen | 21985 | |
| bFGF (10 ng/ml, added fresh) | Millipore | GF003 | |
| Keratinocyte medium: | |||
| EpiLife with 60 µM Calcium | Invitrogen | M-EPI-500-CA | |
| 1× Human keratinocyte growth supplement (HKGS) | Invitrogen | S-001-5 | |
| Fetal Bovine Serum (FBS) medium: | FBS medium is filtered using Stericup (Millipore, #SCGPU05RE) before use. | ||
| 10% fetal bovine serum (FBS) | Invitrogen | 26140079 | |
| DMEM | Invitrogen | 11995-073 | |
| 0.5x Penicillin/Streptomycin | Invitrogen | 15140-122 | |
| 2 mM L-glutamine | Invitrogen | 25030 | |
| 0.25% trypsin-EDTA | Invitrogen | 25200-056 | |
| Extracellular Matrix (ECM): | |||
| Matrigel | Corning | 354234 | Aliquot Matrigel stock and store in -80°C following manufacturer’s instructions. Stock concentration of Matrigel varies slightly from batch to batch (~9mg/ml). We recommend to use 200µl matrigel for coating a 12-well plate (~150µg/well). |
| Coating Matrix Kit | Invitrogen | R-011-K | |
| Plasmids: | Note that pCXLE-eGFP is only used for monitoring transfection efficiency and is not required for reprogramming. | ||
| - pCXLE-eGFP | Addgene | 27082 | |
| - pCXLE-hOct3/4-shP53F | Addgene | 27077 | |
| - pCXLE-hSK | Addgene | 27078 | |
| - pCXLE-hUL | Addgene | 27080 | |
| Transfection reagent Fugene HD | Promega | E231B | |
| Gelatin (from porcine skin) | Sigma | G1890 | Make up 0.1% gelatin in distilled water. Autoclave before use. |
| Reduced Serum medium: OPTI-MEM | Invitrogen | 31985062 | |
| Accutase | Sigma | A6964-100ml | |
| Mouse embryonic fibroblast (MEF) feeder | MEF can be inactivated by mitomycin C treatment or irradiation as described previously 16. | ||
| 26G needle | Terumo | NN2613R | |
| 6-well plate (tissue culture treated) | BD Biosciences | 353046 | |
| 12-well plate (tissue culture treated) | BD Biosciences | 353043 | |
| 10 cm dish (tissue culture treated) | BD Biosciences | 353003 | |
| Dispase | Invitrogen | 17105-041 | Use at 10 mg/ml |
| Collagenase IV | Invitrogen | 17104-019 | Use at 1 mg/ml |
| TRA-160 antibody | Millipore | MAB4360 | Use at 5 µg/ml |
| OCT4 antibody | Santa Cruz | SC-5279 | Use at 5 µg/ml |
| NANOG antibody | R&D Systems | AF1997 | Use at 10 µg/ml |
| MycoAlert Detection kit | Lonza | LT07-418 |
Riferimenti
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126, 663-676 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131, 861-872 (2007).
- Yu, J., et al. Induced pluripotent stem cell lines derived from human somatic cells. Science. 318, 1917-1920 (2007).
- Okita, K., et al. A more efficient method to generate integration-free human iPS cells. Nat Methods. 8, 409-412 (2011).
- Park, I. H., et al. Reprogramming of human somatic cells to pluripotency with defined factors. Nature. 451, U141-U141 (2008).
- Okita, K., et al. An efficient nonviral method to generate integration-free human-induced pluripotent stem cells from cord blood and peripheral blood cells. Stem Cells. 31, 458-466 (2013).
- Dowey, S. N., Huang, X., Chou, B. K., Ye, Z., Cheng, L. Generation of integration-free human induced pluripotent stem cells from postnatal blood mononuclear cells by plasmid vector expression. Nat Protoc. 7, 2013-2021 (2012).
- Zhou, T., et al. Generation of induced pluripotent stem cells from urine. J Am Soc Nephrol. 22, 1221-1228 (2011).
- Aasen, T., et al. Efficient and rapid generation of induced pluripotent stem cells from human keratinocytes. Nat Biotechnol. 26, 1276-1284 (2008).
- Peters, A., Zambidis, E. Chapter 16. Generation of nonviral integration-free induced pluripotent stem cells from plucked human hair follicles. Human Embryonic and Induced Pluripotent Stem Cells: Lineage-Specific Differentiation Protocols.Springer Protocols Handbooks. Ye, K., Jin, S. , Springer. 203-227 (2012).
- Fuchs, E. Scratching the surface of skin development. Nature. 445, 834-842 (2007).
- Limat, A., Noser, F. K. Serial cultivation of single keratinocytes from the outer root sheath of human scalp hair follicles. J Invest Dermatol. 87, 485-488 (1986).
- Piao, Y., Hung, S. S., Lim, S. Y., Wong, R. C., Ko, M. S. Efficient generation of integration-free human induced pluripotent stem cells from keratinocytes by simple transfection of episomal vectors. Stem Cells Transl Med. 3, 787-791 (2014).
- Higgins, C. A., et al. Reprogramming of human hair follicle dermal papilla cells into induced pluripotent stem cells. J Invest Dermatol. 132, 1725-1727 (2012).
- Muchkaeva, I. A., et al. Generation of iPS Cells from Human Hair Follice Dermal Papilla Cells. Acta naturae. 6, 45-53 (2014).
- Naaldijk, Y., Friedrich-Stockigt, A., Sethe, S., Stolzing, A. Comparison of different cooling rates for fibroblast and keratinocyte cryopreservation. J Tissue Eng Regen. , (2013).
- Sporl, F., et al. Real-time monitoring of membrane cholesterol reveals new insights into epidermal differentiation. J Invest Dermatol. 130, 1268-1278 (2010).
- Conner, D. A., et al. Mouse embryo fibroblast (MEF) feeder cell preparation. Current protocols in molecular biology. Ausubel, F. N., et al. 23, Unit 23 22(2001).
- Pebay, A., et al. Essential roles of sphingosine-1-phosphate and platelet-derived growth factor in the maintenance of human embryonic stem cells. Stem Cells. 23, 1541-1548 (2005).
- Myung, P., Ito, M. Dissecting the bulge in hair regeneration. J Clin Invest. 122, 448-454 (2012).
- Alonso, L., Fuchs, E. The hair cycle. J Cell Sci. 119, 391-393 (2006).
- Robinton, D. A., Daley, G. Q. The promise of induced pluripotent stem cells in research and therapy. Nature. 481, 295-305 (2012).
- Soares, F. A., Sheldon, M., Rao, M., Mummery, C., Vallier, L. International coordination of large-scale human induced pluripotent stem cell initiatives: Wellcome Trust and ISSCR workshops white paper. Stem cell reports. 3, 931-939 (2014).
- McKernan, R., Watt, F. M. What is the point of large-scale collections of human induced pluripotent stem cells. Nat Biotechnol. 31, 875-877 (2013).
- Utikal, J., et al. Immortalization eliminates a roadblock during cellular reprogramming into iPS cells. Nature. 460, U1145-U1112 (2009).
- Xu, Y., et al. Proliferation rate of somatic cells affects reprogramming efficiency. J Biol Chem. 288, 9767-9778 (2013).
- Liu, J., et al. Late passage human fibroblasts induced to pluripotency are capable of directed neuronal differentiation. Cell Transplant. 20, 193-203 (2011).
- Huallachain, M., Karczewski, K. J., Weissman, S. M., Urban, A. E., Snyder, M. P. Extensive genetic variation in somatic human tissues. Proc Natl Acad Sci U S A. 109, 18018-18023 (2012).
- Abyzov, A., et al. Somatic copy number mosaicism in human skin revealed by induced pluripotent stem cells. Nature. 492, 438-442 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon