Method Article
Selezione e isolamento di colonie di cellule pluripotenti indotte umane staminali riprogrammato da fibroblasti adulti
In questo articolo
Riepilogo
Vi presentiamo un protocollo efficace per la riprogrammazione di cellule somatiche umane in cellule staminali pluripotenti indotte umane (hiPSC) che utilizzano vettori retrovirali codificanti Oct3 / 4, Sox2, Klf4 e c-myc (OSKM) e l'individuazione di hiPSC correttamente riprogrammata mediante colorazione dal vivo con Tra- 1-81 anticorpo.
Abstract
Qui presentiamo un protocollo di riprogrammazione fibroblasti umani adulti in cellule pluripotenti indotte umane staminali (hiPSC) utilizzando vettori retrovirali codificanti Oct3 / 4, Sox2, Klf4 e c-myc (OSKM) in presenza di sodio butirrato 1-3. Abbiamo usato questo metodo per riprogrammare il passaggio in ritardo (> p10) fibroblasti umani adulti ottenuti da paziente l'atassia di Friedreich (GM03665, Coriell Repository). L'approccio riprogrammazione comprende protocollo trasduzione altamente efficiente mediante centrifugazione ripetitivo di fibroblasti in presenza di virus contenenti supporti. Le colonie hiPSC riprogrammate sono stati identificati usando immunostaining dal vivo per Tra-1-81, un marcatore di superficie delle cellule pluripotenti, separato da fibroblasti non riprogrammate e manualmente diversi passaggi 4,5. Questi hiPSC state poi trasferite in piastre Matrigel e coltivate in condizioni prive di alimentazione, direttamente dalla piastra riprogrammazione. A partire dal primo passaggio, colonie hiPSC dimostrare caratteristica hES-lmorfologia ike. Usando questo protocollo più del 70% delle colonie selezionati può essere espansa con successo e stabilito in linee cellulari. Le linee stabilite hiPSC visualizzata caratteristici marcatori pluripotenziali tra cui marcatori di superficie TRA-1-60 e SSEA-4, così come marcatori nucleari Oct3 / 4, Sox2 e Nanog. Il protocollo qui presentato è stato stabilito e testato utilizzando fibroblasti ottenuti da pazienti adulti Friedreich atassia e individui di controllo 6, fibroblasti umani neonati, nonché cheratinociti umani.
Protocollo
1. Virus produzione e trasduzione
- Plate Phoenix Ampho cellule ad una densità di ~ 7-8x10 6 per piastra 10 cm in 10 ml di terreno DMEM (DMEM elevato glucosio, 10% FBS inattivato al calore, 2 mM L-glutammina, senza antibiotici). Posto nell'incubatrice e coltura di una notte a 37 ° C, 5% CO 2.
- Il giorno seguente trasfezione delle cellule Phoenix con 12 mcg di un vettore codificante sia Oct3 / 4, Sox2, Klf4, c-myc, o gene GFP (Addgene plasmidi 17.217, 17.218, 17.219, 17.220) e 35 microlitri Fugene 6. Preparare miscela di trasfezione in 500 pl di mezzo DMEM (glucosio DMEM alto, 2 mM L-glutammina, antibiotici e non FBS). Incubare 20 min. Pipettare delicatamente complessi di DNA nei 10 piatti cm contenenti 70-80% di cellule confluenti Phoenix.
- Sostituire supporti 6 - 8 h post-trasfezione con mezzo DMEM (DMEM elevato glucosio, 10% FBS inattivato al calore, 2 mM L-glutammina) contenente penicillina e streptomicina.
- Successivamente, la raccolta contenenti virus dei media 4 volte in 12 hintervalli, unire tutte le parti e filtrato utilizzando un filtro da 0,45 micron per rimuovere le cellule staccate e detriti (supporti contenenti particelle virali può essere conservato in frigorifero per 2 settimane senza perdere l'attività infettiva, tuttavia il congelamento non è raccomandato).
- Per retrovirali trasduzione, fibroblasti umani adulti piastra (passaggio 10, Coriell Laboratories) da un magazzino congelato 24 h precedenti l'ultimo giorno di retrovirale raccolta multimediale. Seme le celle coperte piastre da 6 pozzetti gelatina alla densità 1x10 5 cellule per pozzetto in DMEM elevato glucosio, 10% FBS, 2 mM L-glutammina, penicillina, streptomicina e amminoacidi non essenziali.
- Il sostituto giorno successivo DMEM media con i supporti contenenti le particelle virali ottenuto da cellule di Phoenix. Aggiungi media virali (1 ml di ogni Oct4, Sox2, Klf4, e c-myc media, 4 in totale ml) integrati con 6 mg / ml di polibrene in ciascun pozzetto della piastra da 6 pozzetti delle colture di fibroblasti e centrifugare a 1600g per 1h a 20 ° C. Circa 12 h after la trasduzione, sostituire i media virali con mezzo di coltura di fibroblasti. Eseguire l'infezione virale e centrifugazione 3 volte in intervalli di 24 h.
- Fibroblasti cultura in DMEM media per 48 ore dopo l'infezione scorso. Controllare l'efficienza della infezione da trasduzioni parallele con un supporto retrovirali esprimenti GFP. La Figura 1 illustra l'efficienza di centrifugazione-facilitato infezione retrovirale di fibroblasti umani.
2. Riprogrammazione
- Preparare piastre da 6 pozzetti con strati MEF γ-irradiati mediante inseminazione di cellule feeder MEF ad una densità di ~ 2x10 5 cellule per pozzetto della gelatina trattata 6 pozzetti in terreno di crescita dei fibroblasti. Il giorno seguente suddiviso infettate fibroblasti umani usando 0,05% di tripsina / EDTA e sementi loro alla densità di ~ 1.2x10 4 per pozzetto in mezzo di crescita dei fibroblasti.
- Il giorno dopo sostituzione del supporto con HES media (DMEM/F12, 20% sostituzione Serum Knockout, aminoacidi non essenziali, penicillin / streptomicina, 2 mM L-glutammina, 0,1 mM β-mercaptoetanolo, 20 ng / ml basale fattore di crescita dei fibroblasti (bFGF), supplementato con 0,5 mM sodio butirrato per i primi 7 giorni di riprogrammazione. Modificare i media giornaliera.
- Monitorare i cambiamenti morfologici nelle cellule trasdotte quotidiana. Le colonie di HIPS-come le cellule inizieranno ad emergere circa 5 - 10 giorni dopo il trasferimento dei fibroblasti trasdotte su cellule di alimentazione. Le colonie hiPSC sono pronti per essere isolati circa 14 - 28 giorni dopo la placcatura di loro sulle cellule feeder le MEF.
3. Isolamento di colonie hiPSC
- Un giorno prima di prendere le colonie hiPSC preparare piastre da 24 pozzetti con cellule MEF γ-irradiati feeder (circa 4x10 4 cellule per pozzetto) in crescita media fibroblasti. In questa fase le colonie hiPSC possono essere trasferiti anche alla cultura alimentatore gratuitamente utilizzando Matrigel e medie mTeSR1.
- Prima di isolamento di colonie hiPSC preparare lastre MEF rimuovendo fibroblasti crescita Medium MEF e risciacquo con PBS per rimuovere tracce di FBS. Successivamente si aggiungono 0,5 ml hES medio (o mTeSR1 media nel caso di Matrigel pozzetti) con 10 pM inibitore ROCK Y27632 a ciascun pozzetto 7,8.
- Se necessario, rimuovere i fibroblasti che circondano le colonie hiPSC con un ago calibro 21 sotto un microscopio montato in una cappa a flusso laminare (Figura 2A - C). Lavare le piastre con PBS e aggiungere nuovi mezzi di comunicazione hES contenente l'anticorpo Tra-1-81 StainAlive specifico (1:200, Stemgent). Dopo 30 min., Sostituire i supporti contenenti l'anticorpo con nuovi mezzi di comunicazione integrato con il 10 hES inibitore ROCK pM. Esaminare le piastre al microscopio a fluorescenza e mark Tra-1-81 colonie positive con un pennarello obiettivo (Figura 2D).
- Tagliare i Tra-1-81 colonie positive hiPSC sotto un microscopio in una cappa laminare, in alcuni piccoli pezzi utilizzando un ago da 21 gauge, come mostrato nella Figura 2E e F.
- Utilizzando una pipetta automatica (P200) frammenti di trasferimento di colonie hiPSC in indpozzi ividual della piastra da 24 pozzetti con il MEF o Matrigel. Evitare di trasferire non HIPS cellule. Porre le piastre a 37 ° C, 5% CO 2 incubatore e consentire le colonie hiPSC attaccare per 24-36 h.
- Cambia HES o mTeSR1 (per le colonie coltivate su Matrigel) media giornaliera. Le colonie hiPSC con corretto HES-morfologia simile sarà visibile 48 h dopo il trasferimento iniziale su una piastra da 24 pozzetti.
- Passaggio manuale delle colonie hiPSC ogni 6 - 8 giorni su un 12-well e successivamente su un piastra da 6 pozzetti.
- Dopo l'espansione clonale e stabilire le linee hiPSC, analizzare l'espressione dei marcatori pluripotenziali TRA-1-60, SSEA-4, Oct3 / 4, Sox2 e Nanog immunocitochimica utilizzando come mostrato nella Figura 3. Valutare l'integrità genomica e potenziale di differenziazione delle linee ottenute con cariotipo e analisi di formazione teratoma 6,9.
4. Risultati rappresentativi
Trasduzione efficiente con retrovirus contenenti i supportis critico per la riprogrammazione di successo. Si raccomanda di condurre l'intero trasfezione / infezione procedura utilizzando un virus che esprime GFP ogni singolo esperimento riprogrammazione per monitorare l'efficienza, come mostrato nella Figura 1. Il titolo del virus GFP esprimere determinato, come descritto in 10, attraverso la trasduzione di fibroblasti umani utilizzando media virali non-concentrata è tipicamente nell'intervallo di 0,5-5 x 10 7 particelle virali per ml (vp / ml).
I fibroblasti cambiano morfologia già 2 giorni dopo l'infezione precedente. Tripsinizzate fibroblasti umani devono essere attentamente considerato prima di semina loro su cellule di alimentazione del MEF. Si raccomanda di Piastrare 3 a diverse densità (6x10 3, 4 1.2x10, 2.5x10 4 per singolo pozzetto di una piastra da 6 pozzetti) poiché ciascuna linea cellulare viene illustrato diversa caratteristica crescita e densità di semina è critico per l'efficienza di riprogrammazione. Butirrato di sodio utilizzato per lainiziale 7 - 14 giorni di riprogrammazione aumenta l'efficienza di formazione hiPSC circa 5 volte. Spesso, specialmente quando fibroblasti infettati sono state seminate a maggiore densità, i fibroblasti cellule simili possono proliferare un piatto di coltura e coprire colonie hiPSC come mostrato nella Figura 2A. In questo caso, lo strato di fibroblasti può essere alzato attentamente e rimossa per scoprire colonie hiPSC (Figura 2B). Successivamente, le colonie hiPSC devono essere risciacquati con hES media e colorati con Tra-1-81 anticorpo. A seconda dei fibroblasti, circa il 20 - 40% delle colonie che dimostrano con IPSC-come morfologia non si macchia con Tra-1-81 anticorpo. Identificati hiPSC colonie possono essere trasferiti da una piastra all'interno prossima 12 - 24h. Incubazione prolungata si tradurrà in rapida differenziazione di hiPSCs. Le colonie possono essere diversi passaggi manualmente sia su cellule di alimentazione del MEF o Matrigel rivestite con lastre. Dopo l'espansione stabilito cloni hiPSC deve essere testato utilizzando immunocitochimica (ICC) per l'espressione di pluripotency marcatori come mostrato in Figura 3. Inoltre, una dettagliata caratterizzazione molecolare dei generate linee cellulari iPS dovrebbe comprendere: analisi di espressione genica pluripotenza usando RT-PCR, dimostrazione della demetilazione DNA ai promotori di geni pluripotenziali e analisi dei transgeni silenziamento 9.

Figura 1. L'efficienza di trasduzione virale determinata con retrovirus che esprime GFP. (A, B) fibroblasti umani adulti ottenuti da paziente l'atassia di Friedreich (GM03665, Coriell Repository) sono stati visualizzati dopo due infezioni consecutivi con i media GFP retrovirali. (C, D) fibroblasti umani sono stati infettati con lo stesso lotto dei mezzi di GFP retrovirali seguita da centrifugazione delle cellule direttamente sulle piastre da 6 pozzetti per 1h a 1600g. Le immagini sono state catturate 48 ore dopo l'infezione.

Figura 2. Identificazione e isolamento di colonie hiPSC. (A) contrasto di fase di una piastra contenente hiPSCs circondati da fibroblasti. Le cellule sono state coltivate per 21 giorni in hES supporto. (B, C) La colonia stessa hiPSC dopo la rimozione dello strato di fibroblasti circostante. (D) correttamente riprogrammate colonie hiPSC sono identificati dalla colorazione dal vivo con Tra-1-81 anticorpi marker di superficie, tagliati con un ago sterile (E, F) e trasferiti in pozzetti separati di una piastra da 24 pozzetti.
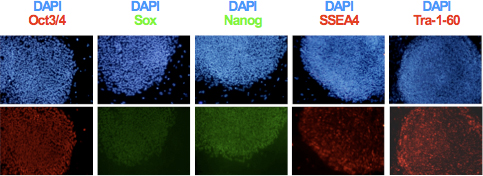
Figura 3. Espressione dei markers specifici pluripotenziali Oct3 / 4, Nanog, Sox2, SSEA4 e Tra-1-60 in hiPSCs è stata determinata mediante immunocitochimica.
Discussione
Studiare le malattie umane, in particolare neurologiche e neurodegenerative, è stata particolarmente difficile a causa l'inaccessibilità di adeguati modelli cellulari umani. La capacità di riprogrammare facilmente ottenibili cellule somatiche in cellule staminali pluripotenti indotte e la possibilità di differenziarle in tipi di cellule diverse ha aperto la possibilità di creare modelli cellulari di malattie genetiche. Inoltre, iPSCs tenere una grande promessa per il futuro della medicina rigenerativa. Pertanto, è essenziale sviluppare e ottimizzare i metodi affidabili, efficienti e sicuri di riprogrammazione delle cellule somatiche di pluripotenza.
Dal punto di vista di applicazioni terapeutiche, è cruciale per sviluppare approcci sicuri di generazione IPSC privi impronta mutazioni nel genoma ospite. Metodi di cellule somatiche riprogrammazione senza introdurre modifiche permanenti nel genoma di cellule riprogrammate come la transfezione di vettori episomale, uso di prodotti soggetti ad accisa TRAnsposons, infezioni adenovirali, e la consegna diretta di mRNA o proteine sono già state stabilite 11-15. Finora l'efficienza riprogrammazione utilizzando questi metodi transgene-free è basso e dipende dal tipo di carattere, e l'età di cellule somatiche riprogrammate. Pertanto, questi protocolli non sono adatti per la riprogrammazione passaggio ritardo, cellule somatiche adulte spesso depositati in banche di cellule. Inoltre, per consentire caratterizzazione dettagliata delle linee IPSC multipli e di stabilire nuovi modelli di malattie umane e per condurre una high-throughput schermi di droga, la riprogrammazione di cellule somatiche utilizzando l'invio di fattori di trascrizione virale è una strategia adeguata. Ad oggi più di 20 studi hanno riportato la generazione del paziente-specifici iPSCs al modello malattie umane neurologiche e neuromuscolari. In tutti i casi, ma uno dei iPSCs sono stati generati utilizzando lentivirale trasduzione retrovirale o 16.
I nostri dati dimostrano che un semplice protocollo basato su maggioconsegna e retrovirale di fattori di trascrizione OSKM può essere usato per riprogrammare fibroblasti umani adulti efficiente, anche di un passaggio alto. La quantità di virus ottenuto da un singolo trasfezione delle cellule Phoenix sul piatto 10 cm è sufficiente per più di 10 esperimenti di riprogrammazione. Ci sono tre fasi critiche nostro protocollo riprogrammazione di fibroblasti adulti: (i) trasduzione efficiente virale facilitato da una fase di centrifugazione aggiuntivo durante la trasduzione che aumenta enormemente l'efficienza di trasduzione, (ii) densità di semina del fibroblasto trasdotto sul MEF e ( iii) l'uso di colorazione dal vivo con l'anticorpo Tra-1-81 marker per facilitare la selezione delle colonie potenzialmente pienamente riprogrammate che possono essere trasferiti direttamente alla rinfusa, senza culture. L'efficienza della riprogrammazione è significativamente migliorata utilizzando butirrato di sodio durante la seconda settimana della riprogrammazione. Inoltre, abbiamo osservato che una frazione maggiore delle colonie IPSC coltivate nellala presenza della droga può essere ampliata e stabiliti in piena riprogrammate linee IPSC.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato da Alliance L'Atassia di Friedreich ricerca e una borsa di studio pilota da Arnold Family Foundation e il Centro per le cellule staminali e biologia dello sviluppo presso MD Anderson Cancer Center.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagente | Azienda | Numero di catalogo | |
| DMEM | Invitrogen | 11965 | |
| DMEM/F12 | Invitrogen | 11330 | |
| KSR | Invitrogen | 10828 | |
| Aminoacidi non essenziali | Invitrogen | 11140 | |
| Butirrato di sodio | Sigma | B5887 | |
| Y27632 | Stemgent | 04-0012 | |
| bFGF | Stemgent | 03-0002 | |
| Tra-1-81 anticorpo | Stemgent | 09-0069 | |
| Oct3 / 4 anticorpo | Santa Cruz | sc-8628 | |
| Nanog anticorpi | Tecnologia delle celle di segnalazione | 4903S | |
| Tra-1-60 anticorpo | Millipore | MAB4360 | |
| Sox2 anticorpi | Tecnologia delle celle di segnalazione | 3579S | |
| SSEA4 | Millipore | MAB4304 | |
| CF1 MEF | Globalstem | GSC-6201G | |
| Obiettivo marker | Nikon | MBW10010 | |
| Matrigel | BD Biosciences | 354277 | |
| mTeSR1 | StemCell Technologies | 05850 | |
| β-mercaptoetanolo | Sigma | M7522 | |
| Fugene 6 | Roche | 11814443001 | |
| polibrene | Sigma | H9268 | |
| Oggetto marker | Nikon | MBW10010 |
Riferimenti
- Takahashi, K., Tanabe, K., Ohnuki, M., Narita, M., Ichisaka, T., Tomoda, K., Yamanaka, S. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131, 861-872 (2007).
- Mali, P., Chou, B. K., Yen, J., Ye, Z., Zou, J., Dowey, S., Brodsky, R. A., Ohm, J. E., Yu, W., Baylin, S. B., Yusa, K., Bradley, A., Meyers, D. J., Mukherjee, C., Cole, P. A., Cheng, L. Butyrate greatly enhances derivation of human induced pluripotent stem cells by promoting epigenetic remodeling and the expression of pluripotency-associated genes. Stem Cells. 28, 713-720 (2011).
- Wernig, M., Meissner, A., Foreman, R., Brambrink, T., Ku, M., Hochedlinger, K., Bernstein, B. E., Jaenisch, R. In vitro reprogramming of fibroblasts into a pluripotent ES-cell-like state. Nature. 448, 318-324 (2007).
- Lowry, W. E., Richter, L., Yachechko, R., Pyle, A. D., Tchieu, J., Sridharan, R., Clark, A. T., Plath, K. Generation of human induced pluripotent stem cells from dermal fibroblasts. Proc. Natl. Acad. Sci. U.S.A. 105, 2883-2888 (2008).
- Dick, E., Matsa, E., Bispham, J., Reza, M., Guglieri, M., Staniforth, A., Watson, S., Kumari, R., Lochmuller, H., Young, L., Darling, D., Denning, C. Two new protocols to enhance the production and isolation of human induced pluripotent stem cell lines. Stem Cell Res. 6, 158-167 (2011).
- Ku, S., Soragni, E., Campau, E., Thomas, E. A., Altun, G., Laurent, L. C., Loring, J. F., Napierala, M., Gottesfeld, J. M. Friedreich's ataxia induced pluripotent stem cells model intergenerational GAATTC triplet repeat instability. Cell Stem Cell. 7, 631-637 (2010).
- Emre, N., Vidal, J. G., Elia, J., O'Connor, E. D., Paramban, R. I., Hefferan, M. P., Navarro, R., Goldberg, D. S., Varki, N. M., Marsala, M., Carson, C. T. The ROCK inhibitor Y-27632 improves recovery of human embryonic stem cells after fluorescence-activated cell sorting with multiple cell surface markers. PLoS One. 5, e12148-e12148 (2011).
- Watanabe, K., Ueno, M., Kamiya, D., Nishiyama, A., Matsumura, M., Wataya, T., Takahashi, J. B., Nishikawa, S., Muguruma, K., Sasai, Y. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat. Biotechnol. 25, 681-686 (2007).
- Maherali, N., Hochedlinger, K. Guidelines and techniques for the generation of induced pluripotent stem cells. Cell Stem Cell. 3, 595-605 (2008).
- Yu, J., Thomson, J. A. Induced Pluripotent Stem Cell Derivation. Essentials of Stem Cell Biology. Lanza, R. , Elsevier Inc. 331-337 (2009).
- Yu, J., Hu, K., Smuga-Otto, K., Tian, S., Stewart, R., Slukvin, I. I., Thomson, J. A. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324, 797-801 (2009).
- Woltjen, K., Michael, I. P., Mohseni, P., Desai, R., Mileikovsky, M., Hamalainen, R., Cowling, R., Wang, W., Liu, P., Gertsenstein, M., Kaji, K., Sung, H. K., Nagy, A. piggyBac transposition reprograms fibroblasts to induced pluripotent stem cells. Nature. 458, 766-770 (2009).
- Zhou, W., Freed, C. R. Adenoviral gene delivery can reprogram human fibroblasts to induced pluripotent stem cells. Stem Cells. 27, 2667-2674 (2009).
- Warren, L., Manos, P. D., Ahfeldt, T., Loh, Y. H., Li, H., Lau, F., Ebina, W., Mandal, P. K., Smith, Z. D., Meissner, A., Daley, G. Q., Brack, A. S., Collins, J. J., Cowan, C., Schlaeger, T. M., Rossi, D. J. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7, 618-630 (2010).
- Kim, D., Kim, C. H., Moon, J. I., Chung, Y. G., Chang, M. Y., Han, B. S., Ko, S., Yang, E., Cha, K. Y., Lanza, R., Kim, K. S. Generation of human induced pluripotent stem cells by direct delivery of reprogramming proteins. Cell Stem Cell. 4, 472-476 (2009).
- Han, S. S., Williams, L. A., Eggan, K. C. Constructing and deconstructing stem cell models of neurological disease. Neuron. 70, 626-644 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon