Method Article
Test rapide de sensibilité aux antimicrobiens par imagerie par diffusion Raman stimulée de l’incorporation de deutérium dans une seule bactérie
Dans cet article
Résumé
Ce protocole présente un test rapide de sensibilité aux antimicrobiens (AST) en 2,5 heures par imagerie de diffusion Raman stimulée par une cellule unique du métabolisme D2O. Cette méthode s’applique aux bactéries dans l’urine ou l’environnement sanguin total, ce qui est transformateur pour l’AST phénotypique rapide à cellule unique en clinique.
Résumé
Pour ralentir et prévenir la propagation des infections résistantes aux antimicrobiens, il est urgent de procéder à des tests rapides de sensibilité aux antimicrobiens (AST) pour déterminer quantitativement les effets antimicrobiens sur les agents pathogènes. Il faut généralement des jours pour compléter l’AST par des méthodes conventionnelles basées sur la culture à long terme, et elles ne fonctionnent pas directement pour les échantillons cliniques. Ici, nous rapportons une méthode AST rapide rendue possible par l’imagerie par diffusion Raman stimulée (SRS) de l’incorporation métabolique d’oxyde de deutérium (D2O). L’incorporation métabolique deD2Odans la biomasse et l’inhibition de l’activité métabolique lors de l’exposition aux antibiotiques au niveau de la bactérie unique sont surveillées par imagerie SRS. La concentration d’inactivation du métabolisme unicellulaire (SC-MIC) des bactéries lors de l’exposition aux antibiotiques peut être obtenue après un total de 2,5 heures de préparation et de détection des échantillons. De plus, cette méthode AST rapide est directement applicable aux échantillons bactériens dans des environnements biologiques complexes, tels que l’urine ou le sang total. L’imagerie métabolique SRS de l’incorporation de deutérium est transformatrice pour l’AST phénotypique rapide à cellule unique en clinique.
Introduction
La résistance aux antimicrobiens (RAM) est une menace mondiale croissante pour le traitement efficace des maladies infectieuses1. On prévoit que la RAM causera 10 millions de décès supplémentaires par an et une perte de PIB mondial de 100 000 milliards de dollars d’ici 2050 si aucune mesure n’est prise pour lutter contre les bactéries résistantes auxantibiotiques1,2. Cela souligne le besoin urgent de méthodes de diagnostic rapides et innovantes pour les tests de sensibilité aux antibiotiques (AST) des bactéries infectieuses afin de ralentir l’émergence de bactéries résistantes aux antibiotiques et de réduire le taux de mortalité associé3. Pour assurer le meilleur résultat clinique possible, il est crucial d’introduire un traitement efficace dans les 24 heures. Cependant, la méthode de référence actuelle, comme la méthode de diffusion sur disque ou de dilution du bouillon, nécessite généralement au moins 24 heures pour la procédure de préincubation des échantillons cliniques et 16 à 24 heures supplémentaires pour obtenir les résultats de la concentration minimale inhibitrice (CMI). Dans l’ensemble, ces méthodes prennent trop de temps pour guider une décision immédiate pour le traitement des maladies infectieuses en clinique, ce qui conduit à l’émergence et à la propagation de la résistance aux antimicrobiens4.
Des méthodes AST génotypiques, telles que les techniques basées sur la réaction en chaîne par polymérase (PCR)5, ont été développées pour une détection rapide. Ces techniques mesurent les séquences génétiques de résistance spécifiques afin de fournir des résultats AST rapides. Ils ne dépendent pas de la culture cellulaire qui prend beaucoup de temps; Cependant, seules des séquences génétiques spécifiques connues avec résistance sont testées. Par conséquent, son application est limitée à diverses espèces bactériennes ou à différents mécanismes de résistance. De plus, ils ne peuvent pas fournir de résultats de CMI pour les décisions thérapeutiques 6,7. En outre, de nouvelles méthodes phénotypiques pour l’AST rapide sont en cours de développement pour surmonter ces limitations8, y compris les dispositifs microfluidiques 9,10,11,12,13, les dispositifs optiques14,15,16, l’AST phénotypique quantifiant les acides nucléiques numéro de copie17,18, et les méthodes spectroscopiques Raman 19, 20,21,22,23,24. Ces méthodes réduisent le temps nécessaire pour guider les résultats de l’AST, mais la plupart d’entre elles ne s’appliquent qu’aux isolats bactériens, et non directement aux échantillons cliniques, et nécessitent toujours une préincubation prolongée.
Dans ce travail, nous présentons une méthode de détermination rapide de la sensibilité des bactéries dans l’urine et le sang total via le suivi de l’activité métabolique cellulaire par imagerie SRS. L’eau (H2O) participe à la grande majorité des processus essentiels de synthèse biomoléculaire dans les cellules vivantes. En tant qu’isotopologue de l’eau, par réaction d’échange H/D catalysée par enzyme entre l’atome d’hydrogène redox-actif dans le NADPH et l’atome D dansD2O, le deutérium peut être incorporé dans la biomasse à l’intérieur d’une cellule25,26. Une réaction de synthèse d’acides gras deutérés est médiée par le deutérium marqué NADPH. L’incorporation deD2Odans les réactions des acides aminés (AA) entraîne la production de protéines deutérées26 (Figure 1). De cette façon, les biomolécules nouvellement synthétisées contenant des liaisons C-D dans des cellules microbiennes simples peuvent être utilisées comme marqueur d’activité métabolique générale à détecter. Pour lire les liaisons C-D synthétisées de novo, la spectroscopie Raman, un outil analytique polyvalent fournissant des informations chimiques spécifiques et quantitatives sur les biomolécules, est largement utilisée pour déterminer la sensibilité aux antimicrobiens et réduire considérablement le temps de test à quelques heures27,28,29,30 . Cependant, en raison de la faible efficacité inhérente au processus de diffusion Raman, la spectroscopie Raman spontanée est de faible sensibilité de détection. Par conséquent, il est difficile d’obtenir des résultats d’image en temps réel en utilisant la spectroscopie Raman spontanée. La diffusion Raman cohérente (CRS), y compris la diffusion Raman cohérente anti-Stokes (CARS) et la diffusion Raman stimulée (SRS), a atteint une sensibilité de détection élevée en raison du champ lumineux cohérent pour générer des ordres de grandeur plus grands que celui de la spectroscopie Raman spontanée, rendant ainsi l’imagerie chimique à grande vitesse, spécifique et quantitative au niveau de la cellule unique 31,32,33,34,35 ,36,37,38,39.
Ici, sur la base de nos travaux les plus récents40, nous présentons un protocole pour la détermination rapide de l’activité métabolique et de la sensibilité aux antimicrobiens par imagerie femtoseconde SRS C-D de l’incorporation de bactéries D2O dans le milieu normal, l’urine et l’environnement sanguin total au niveau unicellulaire. L’imagerie femtoseconde SRS facilite la surveillance de la concentration d’inactivation du métabolisme unicellulaire (SC-MIC) contre les antibiotiques au niveau de la bactérie unique en 2,5 heures. Les résultats SC-MIC sont validés par test MIC standard par microdilution du bouillon. Notre méthode est applicable pour déterminer la sensibilité aux antimicrobiens des bactéries pathogènes des infections des voies urinaires (IVU) et des infections sanguines (BSI) avec un temps de test beaucoup plus court par rapport à la méthode conventionnelle, ce qui ouvre la possibilité d’une AST phénotypique rapide en clinique au niveau unicellulaire.
Protocole
L’utilisation d’échantillons de sang humain est conforme aux directives de l’IRB de l’Université de Boston et des National Institutes of Health (NIH). Plus précisément, les spécimens proviennent d’une banque et sont complètement dépersonnalisés. Ces spécimens ne sont pas considérés comme des sujets humains par le bureau du comité d’examen institutionnel (IRB) de l’Université de Boston.
1. Préparation de la solution mère de bactéries et d’antibiotiques
- Préparer la solution mère d’antibiotiques (sulfate de gentamicine ou amoxicilline) à une concentration de 1 mg/mL dissous dans une solution saline tamponnée au phosphate (PBS) stérile1x ou un solvant de diméthylsulfoxyde (DMSO) dans des microtubes de 1,5 mL. Dissoudre le sulfate de gentamicine dans une solution stérile de PBS et l’amoxicilline dans un solvant DMSO stérile. Par la suite, conserver la solution antibiotique entre 2 et 8 °C comme suggéré.
- Pour fabriquer un milieu contenant du bouillon Mueller-Hinton ajusté en D2 O (MHB), ajouter 220 mg de base de bouillon MHB à 10 mL deD2Opour obtenir unmilieu contenant 100 % deD2O. Stériliser la solution en filtrant avec des filtres de 200 nm de taille de pores.
REMARQUE: Utilisez toujours ce protocole pour la fabrication et la stérilisation des solutions de milieu dans les étapes suivantes. - Pour préparer des échantillons bactériens pour l’imagerie SRS, ajouter 2 mL de milieu MHB normal, qui ne contient pas de deutérium, à un tube de culture stérile à fond rond, puis le préchauffer à 37 °C.
- Utilisez une boucle stérile pour sélectionner une colonie bactérienne (Escherichia coli BW 25113 ou Pseudomonas aeruginosa ATCC 47085) de la culture fraîche sur une plaque de gélose tryptique au soja. Ensuite, suspendez-le dans le milieu de culture préchauffé et tourbillonnez doucement pour préparer la suspension bactérienne.
- Incuber les bactéries à 37 °C dans un agitateur à 200 tours par minute (tr/min) jusqu’à ce qu’elles atteignent la phase logarithmique.
2. D2O traitement d’incorporation en présence d’antibiotiques (Figure 2a)
- Vérifiez la concentration bactérienne en mesurant la densité optique (DO) avec un photomètre à une longueur d’onde de 600 nm.
- Diluer la solution bactérienne en utilisant le milieu MHB normal, qui ne contient pas de deutérium, pour atteindre une concentration cellulaire finale de 8 x 105 UFC/mL. Vortex doucement pour mélanger les cellules bactériennes.
- Préparer 300 μL d’aliquotes de la solution bactérienne dans sept microtubes de 1,5 mL et 600 μL d’aliquotes de la solution bactérienne dans un microtube de 1,5 mL.
- Ajouter 4,8 μL de solution mère d’antibiotique (gentamicine ou amoxicilline) (1 mg/mL) dans le microtube contenant 600 μL de la solution bactérienne, pour porter la concentration finale d’antibiotique à 8 μg/mL.
- Prélever 300 μL de solution sur les 8 μg/mL de solution bactérienne contenant des antibiotiques et ajouter à 300 μL de solution bactérienne pour obtenir une solution bactérienne contenant deux fois un antibiotique dilué (4 μg/mL).
- Répéter la double dilution en série des antibiotiques d’essai, la gentamicine ou l’amoxicilline, jusqu’à ce que le microtube ayant la concentration la plus faible (0,25 μg/mL) soit atteint, et éliminer 300 μL du tube. Pour la gentamicine et l’amoxicilline, les concentrations en série varient de 0,25 μg/mL à 8 μg/mL.
- Laissez un tube sans antibiotiques pour le contrôle à blanc. Ce sera le contrôle positif pour inspecter l’activité métabolique bactérienne sans traitement antibiotique mais avec un traitementD2O.
- Laissez un tube sans antibiotiques et sans D2O pour le témoin négatif.
- Incuber l’aliquote bactérienne avec le certain antibiotique (gentamicine ou amoxicilline) contenant le milieu MHB pendant 1 h.
- Pendant l’incubation, préparer une dilution en série des antibiotiques avec un milieu contenant 100 %D2Oavec le même gradient de concentration que les antibiotiques préparés à l’étape 2.6. Pour la gentamicine et l’amoxicilline, les concentrations en série varient de 0,25 μg/mL à 8 μg/mL.
- Après 1 h de traitement antibiotique, ajouter 700 μL d’antibiotique dilué en série et un milieu MHB contenant 100 % de D 2 O aux 300 μL de bactéries prétraitées par antibiotique à la même concentration d’antibiotique (préparé à l’étape2.6), respectivement.
- Par exemple, ajoutez 700 μL de milieu MHB contenant 100 % D2O (contenant 8 μg/mL d’antibiotique) aux 300 μL de bactéries prétraitées aux antibiotiques à 8 μg/mL de bactéries prétraitées par les antibiotiques. De la même manière, transférer dans les tubes correspondants de la concentration suivante, et homogénéiser en pipetant de haut en bas plusieurs fois.
- Ajouter 700 μL de milieu MHB 100 % D 2 O sansantibiotique à 300 μL de bactéries sans antibiotiques (préparé à l’étape 2.6.1) comme témoin à blanc.
- Incuber à 37 °C dans un agitateur d’incubation à 200 tr/min pendant 30 minutes supplémentaires.
NOTA: Dans cette étape, la concentration finale deD2Odans le milieu pour l’essai est de 70%.
- Centrifuger d’abord l’échantillon bactérien traité à 1 mL d’antibiotique et D2O à 6200 x g pendant 5 min à 4 °C, puis laver deux fois à l’eau purifiée. Enfin, fixez les échantillons dans une solution de formol à 10% et conservez-les à 4 °C.
3. Préparation des bactéries dans l’environnement urinaire (figure 2b)
- Pour préparer E. coli BW 25113 à la phase logarithmique, suivez les étapes indiquées aux points 1.4 et 1.5.
- Vérifiez la concentration bactérienne en mesurant la DO avec un photomètre à une longueur d’onde de 600 nm.
- Pour imiter les échantillons cliniques d’infection urinaire 14,18,41, augmenter la solution d’E. coli dans 10 mL d’urine déidentifiée pour atteindre une concentration cellulaire finale de10 6 UFC/mL.
- Filtrer l’urine enrichie d’E. coli à l’aide d’un filtre de 5 μm, puis diviser la solution bactérienne en aliquotes de 300 μL en sept microtubes de 1,5 mL et 600 μL aliquotes de la solution bactérienne dans un microtube de 1,5 mL.
- Effectuer le traitement d’incorporationD2Oen présence d’antibiotiques et le prélèvement d’échantillons comme décrit dans les étapes précédentes de 2.4 à 2.10.
4. Préparation des bactéries dans l’environnement sanguin (figure 2c)
- Pour préparer Pseudomonas aeruginosa ATCC 47085 à la phase logarithmique, suivez les étapes indiquées aux points 1.4 et 1.5.
- Pour imiter les échantillons cliniques d’infections sanguines42,43, augmenter P. aeruginosa dans 1 mL de sang humain déidentifié pour atteindre une concentration de 107 UFC/mL.
- Ajouter 9 mL d’eau purifiée stérile pour lyser le sang.
- Filtrer le sang enrichi de P. aeruginosa à l’aide d’un filtre de 5 μm. Récoltez ensuite les bactéries à 1 mL de volume par centrifugation à 6200 x g pendant 5 min à 4 °C. Ajouter 9 mL de MHB normal préchauffé dans la solution bactérienne et vortex doucement. La concentration finale de bactéries est de 106 UFC/mL
- Diviser la solution sanguine enrichie de P. aeruginosa en aliquotes de 300 μL en sept microtubes de 1,5 mL et 600 μL aliquotes de la solution bactérienne dans un microtube de 1,5 mL.
- Effectuer le traitement d’incorporationD2Oen présence d’antibiotiques et le prélèvement d’échantillons comme décrit dans les étapes précédentes de 2.4 à 2.10.
5. Imagerie SRS de l’incorporation métabolique D2O dans une seule bactérie
- Laver 1 mL de solution bactérienne fixe avec de l’eau purifiée, puis centrifuger à 6200 x g pendant 5 min à 4 °C. Retirez le surnageant. Enrichir la solution bactérienne à environ 20 μL.
- Déposer la solution bactérienne sur un verre revêtu de poly-L-lysine. Sandwichiser et sceller l’échantillon pour l’imagerie SRS.
- Image des bactéries à la fréquence vibratoire C-D à 2168 cm-1 à l’aide d’un microscope SRS.
- Entrez et réglez la longueur d’onde de la pompe à 852 nm à l’aide du logiciel de contrôle d’un ordinateur.
- Mesurez la puissance laser à l’aide d’un capteur de puissance. Réglez la puissance du laser de la pompe à l’échantillon à ~8 mW et la puissance du laser Stokes à l’échantillon à ~40 mW en ajustant la plaque demi-onde devant la sortie laser.
REMARQUE: Dans le microscope SRS, un laser femtoseconde accordable avec un taux de répétition de 80 MHz fournit les lasers d’excitation de la pompe (680 à 1300 nm) et de Stokes (1045 nm).
- En ajustant les vis des miroirs de réflexion, alignez spatialement la pompe et les faisceaux de Stokes et dirigez les deux faisceaux dans un microscope vertical équipé d’un système de miroir galvo 2D pour le balayage laser.
- Utilisez un objectif d’immersion dans l’eau 60x pour focaliser la pompe et les lasers Stokes sur l’échantillon.
- Utilisez un condenseur d’huile pour recueillir les signaux de l’échantillon vers l’avant.
- Utilisez un filtre passe-bande pour filtrer le laser Stokes avant de le diriger dans une photodiode.
- Extrayez le signal Raman stimulé par un amplificateur verrouillé et détectez les signaux par la photodiode.
- Définissez chaque image SRS pour qu’elle contienne 200 x 200 pixels et le temps de séjour des pixels pendant 30 μs dans le panneau de commande du logiciel. Le temps total d’acquisition d’une image est de ~1,2 s. Définissez la taille de l’étape sur 150 nm, de sorte que la taille de l’image soit d’environ 30 x 30 μm2. Image d’au moins trois champs de vision pour chaque échantillon.
6. Traitement d’images et analyse de données (Figure 3)
- Pour obtenir l’intensité moyenne du signal C-D, ouvrez et traitez les images SRS avec le logiciel ImageJ.
- Tout d’abord, convertissez les images SRS en images de type 8 bits avec couleur inversée en cliquant sur Image | Type | 8 bits, puis Modifier | Inverser les boutons dans le logiciel ImageJ.
- Ensuite, filtrez les images avec le flou gaussien en cliquant sur Traiter | Filtres | Boutons de flou gaussien et définissez le Sigma (rayon) sur 1.
- Utilisez le réglage du seuil d’image pour sélectionner la zone bactérienne. Cliquez sur Image | Ajuster | Seuil pour s’assurer que les tailles bactériennes sélectionnées correspondent à celles des images SRS originales. Éliminez les petites particules en ajustant le seuil de taille pour déterminer les particules. Cliquez sur Appliquer.
- Appliquer Analyser | Boutons d’analyse des particules pour étiqueter et déterminer la zone des bactéries.
- En cliquant sur le bouton Afficher tout dans le gestionnaire de retour sur investissement de l’image SRS non traitée d’origine, étiquetez la même zone de bactéries, déterminez l’intensité moyenne de chaque point de données en cliquant sur le bouton Mesure dans le gestionnaire de retour sur investissement.
- Encerclez la zone d’arrière-plan dans l’image SRS d’origine et mesurez l’intensité moyenne de l’arrière-plan. Les intensités C-D moyennes de chaque bactérie sont obtenues en déduisant l’intensité du signal de fond.
7. Quantification de la sensibilité aux antimicrobiens via SC-MIC
NOTA : La valeur seuil à 0,60 pour déterminer la CMI-SC est établie selon l’analyse statistique des intensités SRS C-D des conditions métaboliquement actives et inhibées par le métabolisme des bactéries à diverses concentrations d’exposition au médicament40. Les intensités C-D pour les groupes sensibles aux antibiotiques et résistants aux antibiotiques ont été ajustées avec une distribution normale.
- Tracez la courbe des caractéristiques de fonctionnement du récepteur (ROC) et évaluez le seuil de coupure à 0,60. Sur la base de cette valeur seuil, le SC-MIC en tant qu’indicateur de l’efficacité des antibiotiques peut être défini pour déterminer le groupe métaboliquement inactif et métaboliquement actif.
- Pour analyser quantitativement les données d’imagerie SRS, tracer les histogrammes des intensités du signal C-D pour chaque groupe de bactéries traitées avec la concentration d’antibiotique diluée en série. Les points de données colorés représentent différentes bactéries individuelles.
- Normaliser les intensités C-D du groupe traité aux antibiotiques à l’intensité moyenne du groupe témoin sans traitement antibiotique. Déterminer les résultats SC-MIC de différentes combinaisons de bactéries et d’antibiotiques en quantifiant les intensités du signal SRS dans la région C-D par rapport à diverses concentrations d’antibiotiques en utilisant la valeur seuil à 0,60.
- Valider et comparer la lecture de la CMI-SC avec la CMI déterminée à l’aide du test de microdilution conventionnel du bouillon.
- Selon le Clinical and Laboratory Standards Institute (CLSI), la catégorie de sensibilité basée sur les résultats de l’imagerie métabolique SRS pour chaque souche bactérienne testée est interprétée comme « sensible », « résistante » ou « intermédiaire ».
Résultats
L’effet du temps d’incubation sur l’incorporation du deutérium est mesuré par microspectroscopie Raman spontanée dans les régions C-D (2070 à 2250 cm-1) et C-H (2 800 à 3 100 cm-1) (Figure 4a). Les spectres Raman monocellulaires en accéléré de P. aeruginosa cultivés dans un milieu contenant du D2O à 70 % montrent une augmentation de l’intensité CD/CH sur le temps d’incubation de 0 à 180 min. (Figure 4b) L’abondance croissante de C-D dans les cellules microbiennes simples révèle queD2Oest incorporé dans des biomolécules deutérées à l’intérieur de la cellule.
D2O un marquage supérieur à 50% affecte significativement le métabolisme bactérien pendant une période d’incubation de 23 h27. Nous avons observé une inhibition de la croissance bactérienne lorsque la concentration de marquageD2Oest supérieure à 70% pendant une période d’incubation de 25 heures (Figure S1). Nous avons effectué la CMI par dilution du bouillon de référence et obtenu les résultats SC-MIC dans deux souches de P. aeruginosa (P. aeruginosa ATCC 47085 et P. aeruginosa 1133) (tableau S1). Nos résultats actuels montrent que 70%D2On’affecte pas les performances de notre méthode chez P. aeruginosa. L’accord de catégorie de notre méthode SC-MIC avec la méthode conventionnelle basée sur la culture est de 100% pour toutes les combinaisons testées de P. aeruginosa et d’antibiotiques, comme indiqué dans le tableau S1. Nous attribuons ce bon accord à la toxicité minimale de 70% D2O sur P. aeruginosa pendant la période d’incubation de 30 minutes dans la détermination SC-MIC.
Après le protocole, P. aeruginosa a été incubé avec de la gentamicine diluée en série pendant 1 h, puis à 70% D2O pendant 30 minutes supplémentaires. L’imagerie métabolique SRS à ~2168 cm-1 (Figure 5a) a été réalisée. Les intensités C-D lors du traitement antibiotique sont divisées par la valeur moyenne du groupe témoin, qui est sans traitement antibiotique. L’analyse statistique quantitative (figure 5b) a montré que les signaux C-D de P. aeruginosa étaient significativement plus faibles à une concentration de gentamicine de 2 μg/mL ou plus élevée que ceux sans traitement à la gentamicine (0 μg/mL). En utilisant le seuil seuil à 0,60, P. aeruginosa a été inhibé métaboliquement à 2 μg/mL et à des concentrations plus élevées de gentamicine. La ligne pointillée montre la valeur seuil définie à 0,60 dans la figure 5b. De cette façon, la CMI-SC pour P. aeruginosa contre la gentamicine dans un milieu MHB normal a été déterminée à 2 μg/mL. Il est vérifié que cette valeur de CMI-SC se situe dans la plage de différence d’un seul facteur avec la CMI (4 μg/mL) déterminée par la méthode de microdilution du bouillon (figure 5c). Pris ensemble, SC-MIC déterminé par notre technologie permet de quantifier la sensibilité aux antimicrobiens.
Pour explorer le potentiel de l’AST rapide par imagerie SRS de l’incorporation métabolique du deutérium pour des applications cliniques, en particulier pour l’infection urinaire la plus répandue, nous avons testé un échantillon d’urine enrichi de bactéries en utilisant E. coli, l’agent pathogène le plus courant pour causer l’infection urinaire44. Pour imiter les échantillons cliniques d’infection urinaire à une concentration bactérienne relative, E. coli est ajouté à l’urine anonymisée jusqu’à une concentration finale de 106 UFC/mL. Après purification de l’échantillon, les échantillons d’urine ont été incubés avec de l’amoxicilline et du D2O. L’arrière-plan propre dans les images SRS a montré que le protocole de préparation des échantillons était applicable à la mesure rapide de l’AST (figure 5d). La CMI-SC de l’échantillon d’urine enrichi d’E. coli contre l’amoxicilline a été établie à 4 μg/mL (figure 5e), ce qui a la même lecture de sensibilité que la CMI (8 μg/mL) par la méthode conventionnelle de dilution du bouillon pour E. coli pur dans un milieu MHB normal (figure 5f). Ces résultats ont montré collectivement que l’AST rapide par imagerie SRS de l’incorporation métabolique du deutérium est d’un grand potentiel pour le diagnostic clinique des agents pathogènes infectieux UTI.
Par rapport à l’infection urinaire, l’AST rapide pour les agents pathogènes BSI est beaucoup plus difficile pour l’étude in situ de l’activité métabolique bactérienne, car beaucoup de cellules sanguines se présentent dans le sang. Pour étudier l’applicabilité de l’AST rapide par imagerie SRS de l’incorporation métabolique D2O pour les échantillons cliniques de BSI, P. aeruginosa enrichi dans le sang humain déidentifié a été détecté. Comme le montre la figure 5g, l’intensité C-D de l’image SRS à 2168 cm-1 était dominée par les signaux bactériens. Étant donné que les globules rouges n’ont pas d’activité métabolique pour absorber D2O pour une biosynthèse ultérieure, les signaux C-D proviennent de l’incorporation métabolique de deutérium de bactéries vivantes. La modulation de phase croisée ou le signal photothermique des espèces de débris ou de globules rouges a contribué à la faiblesse des signaux de fond, sans affecter l’analyse quantitative du SC-MIC. Le résultat de la CMI SC pour P. aeruginosa dans le sang a été déterminé à 2 μg/mL (figure 5h), ce qui concorde bien avec le résultat standard conventionnel de CMI pour P. aeruginosa dans un milieu de croissance normal (figure 5i). Pris ensemble, ces résultats ont montré que l’imagerie métabolique SRS de l’incorporation métabolique du deutérium peut être une méthode AST rapide pour déterminer le SC-MIC pour les bactéries dans les infections BIS.
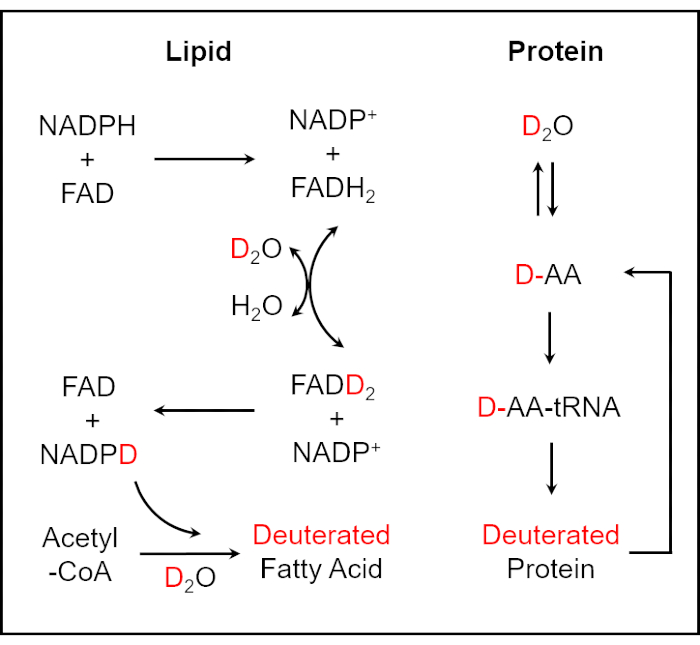
Figure 1 : Schéma d’incorporation deD2Odans les lipides et protéines deutérés25,26. Le deutérium peut être incorporé dans la biomasse à l’intérieur d’une cellule par une réaction d’échange H / D catalysée par enzyme entre l’atome d’hydrogène redox-actif dans le NADPH et l’atome D dans D2O. La réaction de synthèse des acides gras deutérés est médiée par le deutérium marqué NADPH. L’incorporation deD2Odans les réactions des acides aminés entraîne la production de protéines deutérées. Cette figure a été modifiée par rapport à la réf.40. Veuillez cliquer ici pour voir une version agrandie de cette figure.
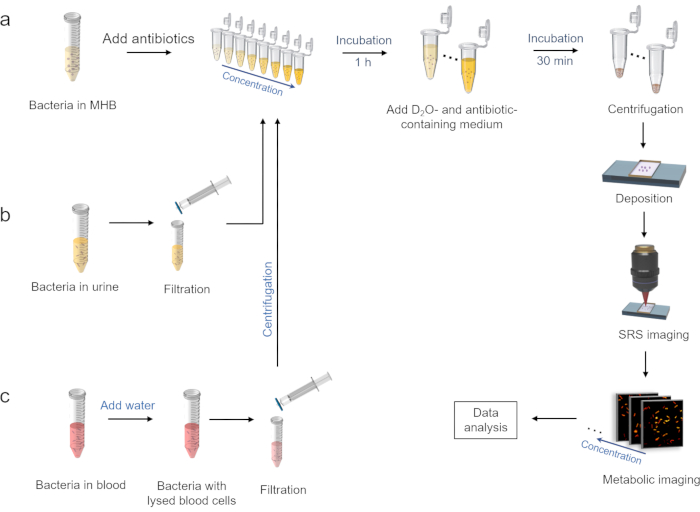
Figure 2 : Flux de travail de l’imagerie métabolique rapide AST par SRS de l’incorporation de deutérium. a) Traitement d’incorporation D2O en présence d’antibiotiques dans le milieu MHB, et les procédures d’imagerie SRS suivantes. b) Préparation de bactéries dans l’environnement urinaire. c) Préparation de bactéries dans l’environnement sanguin. Cette figure a été modifiée par rapport à la réf.40. Veuillez cliquer ici pour voir une version agrandie de cette figure.
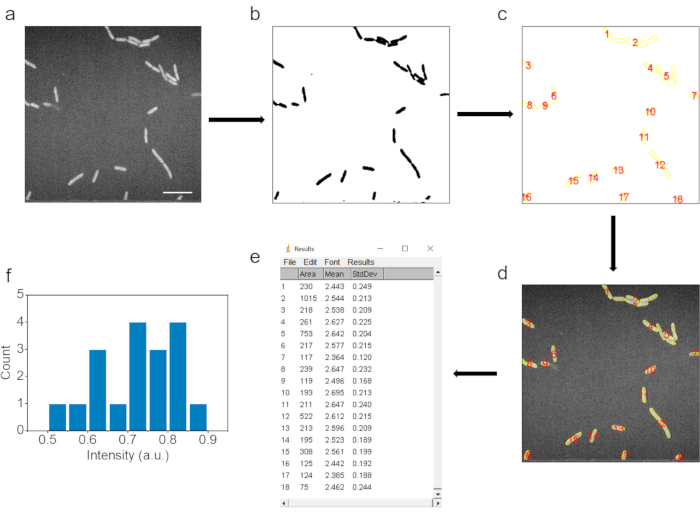
Graphique 3. Traitement automatisé des images et interprétation des données. (a) Image SRS brute. (b) Image après ajustement du seuil d’intensité pour déterminer la surface des cellules bactériennes. c) Points de données sélectionnés après l’étape de l’analyse des particules. (d) Les points de données correspondants sont sélectionnés dans l’image brute. e) Résultats des points de données correspondants dans l’image brute. f) Résultats statistiques de l’intensité moyenne des points de données après soustraction du fond. Barre d’échelle: 10 μm. Cette figure a été modifiée par rapport à la réf.40. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 4. Effet du temps d’incubation sur l’incorporation du deutérium dans les bactéries. a) Mesure accélérée dans les régions C-D (2070 à 2250 cm-1) et C-H (2 800 à 3 100 cm-1) par microspectroscopie Raman spontanée (moyenne calculée à partir de 20 spectres). (b) Histogramme du diagramme du rapport d’intensité CD/CH sur le temps d’incubation D2O pour les bactéries en (a). Chaque point coloré représente une mesure à partir d’une seule bactérie. Les barres d’erreur représentent l’erreur-type de la moyenne (MEB). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 5. Détermination de la CMI-SC à l’aide de l’imagerie SRS de l’incorporation métabolique bactérienne deD2Ocontre des antibiotiques dans un milieu, une urine et un environnement sanguin normaux. a) Imagerie SRS aux vibrations C-D (2168 cm-1) et images de transmission correspondantes de P. aeruginosa en présence deD2Oavec addition de gentamicine diluée en série dans un milieu MHB normal. b) Analyse quantitative de l’intensité du SRS C-D de P. aeruginosa en a). Les points de données colorés dans l’histogramme représentent les différentes bactéries individuelles. La ligne pointillée indique la valeur seuil à 0,60. c) La comparaison de la lecture de la CMI SC avec la CMI par la méthode de microdilution du bouillon et la catégorie de sensibilité pour P. aeruginosa selon le CLSI. d) Imagerie SRS aux vibrations C-D (2168 cm-1) et images de transmission correspondantes d’E. coli dans l’urine après incubation dansD2Oavec l’amoxicilline diluée en série. e) Analyse quantitative de l’intensité du SRS C-D en d). f) La comparaison de la lecture et de la catégorie de sensibilité à E. coli dans le MHB normal et dans l’urine. g) Imagerie SRS à vibration C-D (2168 cm-1) et images de transmission correspondantes de P. aeruginosa dans le sang après incubation dansD2Oavec la gentamicine diluée en série. h) Analyse quantitative de l’intensité du SRS C-D en g). i) La comparaison de la lecture SC-MIC et de la catégorie de sensibilité pour P. aeruginosa dans le MHB normal et dans le sang. S : sensible. Nombre de cellules N ≥ 10 par groupe. Les barres d’erreur représentent le MEB. Barre d’échelle : 10 μm. Cette figure a été modifiée par rapport à la réf.40. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Problème | Raison possible | Solution |
| Peu ou pas de nombre de cellules bactériennes dans le champ de vision de l’imagerie | La densité bactérienne dans la solution est trop faible | Centrifuger plus longtemps pour enrichir davantage les bactéries |
| Photodommages des cellules bactériennes lors de l’imagerie SRS | La puissance laser utilisée est trop élevée | Réglez la puissance laser à une valeur appropriée |
| Aucun signal SRS n’est détectable | Le chevauchement spatial et temporel de la pompe et du faisceau de Stokes n’est pas optimisé | Aligner la pompe et les faisceaux de Stokes à l’aide d’un échantillon standard de diméthylsulfoxyde deutéré |
Tableau 1 : Tableau de dépannage.
Figure S1 : Essai de toxicitéD2Osur P. aeruginosa cultivé dans un milieu Lauria-Bertani (LB) avec différentes concentrations enD2O. Les barres d’erreur indiquent les valeurs de l’écart-type (nombre de mesures = 5). Veuillez cliquer ici pour télécharger cette figure.
Tableau S1. Comparaison des CMI-SC et des CMI dans le MHB normal de P. aeruginosa après traitement antibiotique. S : sensible; R: résistant; I : Intermédiaire. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
L’AST rapide peut être obtenue en évaluant la réponse de l’activité métabolique bactérienne au traitement antibiotique à l’aide de l’imagerie métabolique SRS unicellulaire dans les 2,5 heures suivant l’échantillon aux résultats SC-MIC. La réponse de l’activité métabolique bactérienne et la sensibilité aux antimicrobiens peuvent être détectées en surveillant l’incorporation métabolique de D2O pour la synthèse de biomolécules à l’aide de l’imagerie SRS des liaisons C-D. Étant donné que l’eau est omniprésente dans les cellules vivantes, l’imagerie métabolique SRS fournit une méthode universelle pour l’ASAT rapide. La méthode AST rapide est applicable pour détecter les bactéries dans des environnements biologiques complexes, tels que l’urine ou le sang total à un seul niveau de bactérie. Le SC-MIC peut être déterminé après une culture de bactéries dans l’urine et le sang de 1,5 h, ce qui est considéré comme transformateur pour faire passer le paradigme du diagnostic des infections urinaires et des BSI d’une procédure longue dépendante de la culture à une approche in situ indépendante de la culture. Par conséquent, cela signifie une réduction considérable du temps de diagnostic par rapport à la méthode conventionnelle de microdilution du bouillon, ce qui ouvre la voie à une traduction clinique permettant l’identification à temps des agents antimicrobiens appropriés pour un traitement précis.
Les protocoles de traitement antibiotique décrits ici suivent les lignes directrices du CLSI, dans lequel le milieu MHB suggéré peut généralement être utilisé pour la culture d’une grande variété de micro-organismes. Un paramètre clé est que le nombre de cellules bactériennes utilisées pour les tests de sensibilité aux antimicrobiens est maintenu à environ 5 x 105 UFC /mL, comme le recommande l’ICLS. Ceci est d’une importance cruciale pour obtenir des résultats précis et reproductibles. Dans les expériences de traitement antibiotique, la concentration de bactéries est fixée à 8 x 105 UFC / mL. Une concentration plus élevée de bactéries peut entraîner une augmentation des résultats de la CMI. Une fois la suspension bactérienne ajustée, elle doit être utilisée dans les 30 minutes pour éviter les changements dans la concentration de cellules bactériennes.
Pour les tests de sensibilité à un antibiotique tel que la daptomycine, il est recommandé de compléter 50 mg / L de calcium dans le milieu. Le milieu MHB ajusté aux cations contient 20-25 mg/L de Ca2+. Par conséquent, s’assurer que le milieu est complété par du Ca2+ supplémentaire (solubilisé dans l’eau et stérilisé par filtre) à une concentration de 30 mg/L.
Une autre étape critique de la méthode présentée est le temps d’incubation des bactéries lors de l’exposition aux antibiotiques et de l’incorporation deD2O. Parce que le temps de génération du cycle de vie bactérien est d’environ 30 à 60 min, il est important d’influencer l’activité métabolique bactérienne lors de l’exposition aux antibiotiques pendant un certain temps. Ce test a été évalué pour une variété de combinaisons bactéries-antibiotiques à l’exposition aux antibiotiques pendant 1 h avant 0,5 h D2O traitement. La première étape de traitement antibiotique de 1 h est essentielle pour influencer l’activité métabolique bactérienne. Ensuite, les bactéries sont incubées avec un milieu contenant du D2O et un milieu contenant des antibiotiques pendant 30 minutes supplémentaires. Les concentrations finales d’antibiotiques sont maintenues au même niveau et la concentration finale deD2Oest ajustée à 70%. Globalement, après 1 h de préculture antibiotique et après 0,5 h deJ2Oet d’incorporation d’antibiotiques, les résultats SC-MIC sont ensuite déterminés par imagerie métabolique SRS de l’activité métabolique bactérienne. Cette conception minimise l’impact de l’influence de D2O de l’activité antimicrobienne sur les bactéries et conduit également à une lecture comparable des résultats SC-MIC avec les CMI par la méthode conventionnelle.
Dans les mesures SC-MIC, nous préparons en parallèle 40 échantillons, dont 5 antibiotiques différents, chacun avec 8 concentrations en même temps. Cependant, comme il existe de nombreuses procédures manuelles, le temps total de dosage pour détecter l’AST pour cinq combinaisons bactéries-antibiotiques différentes est supérieur à 2,5 h. Dans notre méthode, chaque image SRS contenant ~20 cellules bactériennes individuelles a été obtenue en ~1,0 s en une seule prise de vue à un temps de séjour de 30 μs pixels. Nous estimons que le temps total de dosage AST pour étudier 10 antibiotiques pour une souche bactérienne serait inférieur à 2,5 heures entre l’échantillon et la lecture SC-MIC, ce qui offre une énorme possibilité d’effectuer des mesures à haut débit. Dans les travaux futurs, une méthode automatisée de préparation des échantillons et d’acquisition des données d’imagerie sera utilisée pour améliorer encore le débit. Les détails de dépannage sont fournis dans le Tableau 1.
Dans les méthodes AST conventionnelles basées sur la culture, pour obtenir des isolats bactériens pour des mesures ultérieures, il est nécessaire de pré-incuber les échantillons cliniques pendant des heures. Des méthodes AST avancées pour les échantillons cliniques d’infection urinaire, telles que la spectroscopie Raman29, le réseau de nanolitres6 et la quantification numérique des acides nucléiques18, ont été développées pour éliminer la préincubation de longue durée. Par rapport à l’infection urinaire, le BSI ou septicémie est beaucoup plus mortel18,45, où une AST rapide est nécessaire de toute urgence pour des diagnostics précis en clinique. Une méthode d’imagerie microscopique pour mesurer la formation de colonies bactériennes à partir d’hémocultures positives a fourni des résultats de CMI46. Cependant, il faut au moins 6 heures pour cultiver des bactéries afin d’effectuer le test AST. De plus, les systèmes automatiques commerciaux 47 et les stratégies de spectrométriede masse 48,49 peuvent fournir une lecture AST à partir d’hémocultures positives. Cependant, les résultats de la CMI pour la décision clinique ne peuvent pas être fournis. Les résultats de l’AST et la lecture de la CMI sont importants pour éviter une dose excessive d’antibiotiques aux patients afin de provoquer des effets secondaires potentiels dans les cliniques, de ralentir et de prévenir la propagation des infections résistantes aux antimicrobiens50,51. Par rapport aux méthodes AST basées sur la microscopie Raman spontanée existantes, notre technologie réduit considérablement le temps d’acquisition de données (environ 600 fois moins) grâce à l’amélioration du signal de plusieurs ordres de grandeur. Dans ce travail, nous démontrons une AST rapide par imagerie SRS du métabolisme du deutérium dans une seule bactérie à une concentration bactérienne cliniquement pertinente (105 ~ 106 UFC / ml) dans l’urine ou le sang total. Comme indiqué dans les résultats précédents, les résultats de la CMI sont déterminés après 1 h de traitement antibiotique et 30 min de mélange deD2Oet d’incubation d’antibiotiques dans les bactéries dans l’urine et le sang. Notre méthode peut fournir des CMI et une classification de sensibilité pour chaque combinaison souche-antibiotique en 2,5 heures et, par conséquent, ouvre une nouvelle voie à la traduction clinique. Pour résumer, sans avoir besoin de préculture et de division bactérienne, notre méthode a un énorme potentiel dans le domaine de l’AST rapide et à haut débit dans les maladies infectieuses.
Notre méthode SC-MIC par imagerie métabolique SRS est applicable pour détecter les CMI et fournir une classification de sensibilité aux agents pathogènes infectieux lorsqu’il s’agit de variétés abondantes de combinaisons souche-antibiotique à usage clinique. Le SC-MIC est déterminé après 30 minutes d’incorporation deJ2Odans les bactéries dans l’urine et le sang, ce qui signifie une réduction considérable du temps de diagnostic par rapport à la méthode conventionnelle de dilution du bouillon coûtant 16 à 24 heures pour la préincubation. Pour fournir des informations d’identification des agents pathogènes pour la prise de décision clinique, la technologie d’imagerie métabolique SRS peut être intégrée davantage à des plateformes de diagnostic capables d’identifier rapidement les agents pathogènes, telles que la spectrométrie de masse à temps de vol par ionisation par désorption laser assistée par matrice 49,52,53. La combinaison de l’identification in situ des agents pathogènes et du diagnostic rapide de l’AST pourrait être d’un grand potentiel de traduction en clinique qui permet d’identifier à temps les agents antimicrobiens appropriés pour un traitement précis.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ce travail a été soutenu par NIH R01AI141439 à J.-X.C et M.S, et R35GM136223 à J.-X.C.
matériels
| Name | Company | Catalog Number | Comments |
| Acousto-optic modulation | Gooch&Housego | R15180-1.06-LTD | Modulating stokes laser beam |
| Amoxicillin | Sigma Aldrich | A8523-5G | |
| Bandpass filter | Chroma | HQ825/150m | Block the stokes laser beam before the photodiode |
| Calcium chloride | Sigma Aldrich | C1016-100G | Cation adjustment |
| Cation-adjusted Mueller-Hinton Broth | Fisher Scientific | B12322 | Antimicrobial susceptibility testing of microorganisms by broth dilution methods |
| Centrifuge | Thermo Scientific | 75002542 | |
| Cover Glasses | VWR | 16004-318 | |
| Culture tube with snap cap | Fisher brand | 149569B | |
| Daptomycin | Acros | A0386346 | |
| Deuterium oxide | 151882 | Organic solvent to dissolve antibiotics | |
| Deuterium oxide-d6 | Sigma Aldrich | 156914 | Organic solvent as a standard to calibrate SRS imaging system |
| Escherichia coli BW 25113 | The Coli Genetic Stock Center | 7636 | |
| Eppendorf polypropylene microcentrifuge tubes 1.5 mL | Fisher brand | 05-408-129 | |
| Gentamicin sulfate | Sigma Aldrich | G4918 | |
| Hydrophilic Polyvinylidene Fluoride filters | Millipore-Sigma | SLSV025NB | pore size 5 µm |
| ImageJ software | NIH | Version: 2.0.0-rc-69/1.52t | Image processing and analysis |
| Incubating orbital shaker set at 37 °C | VWR | 97009-890 | |
| Inoculation loop | Sigma | BR452201-1000EA | |
| InSight DeepSee femtosecond pulsed laser | Spectra-Physics | Model: insight X3 | Tunable laser source and fixed laser source at 1045 nm for SRS imaging |
| Lock-in amplifier | Zurich Instrument | HF2LI | Demodulate the SRS signals |
| Oil condenser | Olympus | U-AAC | NA 1.4 |
| Pseudomonas aeruginosa ATCC 47085 (PAO1) | American Type Culture Collection | ATCC 47085 | |
| Photodiode | Hamamatsu | S3994-01 | Detector |
| Polypropylene conical tube 15 mL | Falcon | 14-959-53A | |
| Polypropylene filters | Thermo Scientific | 726-2520 | pore size 0.2 µm |
| Sterile petri dishes | Corning | 07-202-031 | |
| Syringe 10 mL | Fisher brand | 14955459 | |
| UV/Vis Spectrophotometer | Beckman Coulter | Model: DU 530 | Measuring optical density at wavelength of 600 nm |
| Vortex mixer | VWR | 97043-562 | |
| Water objective | Olympus | UPLANAPO/IR | 60×, NA 1.2 |
Références
- O'Neill, J. Tackling drug-resistant infections globally: final report and recommendations. The review on Antimicrobial Resistance. , (2016).
- Sugden, R., Kelly, R., Davies, S. Combatting antimicrobial resistance globally. Nature Microbiology. 1 (10), 16187 (2016).
- Kumar, A., et al. Duration of hypotension before initiation of effective antimicrobial therapy is the critical determinant of survival in human septic shock. Critical Care Medicine. 34 (6), 1589-1596 (2006).
- Reller, L. B., Weinstein, M., Jorgensen, J. H., Ferraro, M. J. Antimicrobial susceptibility testing: a review of general principles and contemporary practices. Clinical Infectious Diseases. 49 (11), 1749-1755 (2009).
- Frickmann, H., Masanta, W. O., Zautner, A. E. Emerging rapid resistance testing methods for clinical microbiology laboratories and their potential impact on patient management. BioMed Research International. 2014, 375681 (2014).
- Avesar, J., et al. Rapid phenotypic antimicrobial susceptibility testing using nanoliter arrays. Proceedings of the National Academy of Sciences. 114 (29), 5787-5795 (2017).
- Schoepp, N. G., et al. Digital quantification of DNA replication and chromosome segregation enables determination of antimicrobial susceptibility after only 15 minutes of antibiotic exposure. Angewandte Chemie International Edition. 55 (33), 9557-9561 (2016).
- van Belkum, A., et al. Innovative and rapid antimicrobial susceptibility testing systems. Nature Reviews Microbiology. 18 (5), 299-311 (2020).
- Hou, Z., An, Y., Hjort, K., Sandegren, L., Wu, Z. Time lapse investigation of antibiotic susceptibility using a microfluidic linear gradient 3D culture device. Lab on a Chip. 14 (17), 3409-3418 (2014).
- Choi, J., et al. Rapid antibiotic susceptibility testing by tracking single cell growth in a microfluidic agarose channel system. Lab on a Chip. 13 (2), 280-287 (2013).
- Lu, Y., et al. Single cell antimicrobial susceptibility testing by confined microchannels and electrokinetic loading. Analytical Chemistry. 85 (8), 3971-3976 (2013).
- Kim, S. C., Cestellosblanco, S., Inoue, K., Zare, R. N. Miniaturized antimicrobial susceptibility test by combining concentration gradient generation and rapid cell culturing. Antibiotics. 4 (4), 455-466 (2015).
- Choi, J., et al. A rapid antimicrobial susceptibility test based on single-cell morphological analysis. Science Translational Medicine. 6 (267), (2014).
- Baltekin, &. #. 2. 1. 4. ;., Boucharin, A., Tano, E., Andersson, D. I., Elf, J. Antibiotic susceptibility testing in less than 30 min using direct single-cell imaging. Proceedings of the National Academy of Sciences. 114 (34), 9170-9175 (2017).
- Fredborg, M., et al. Real-time optical antimicrobial susceptibility testing. Journal of Clinical Microbiology. 51 (7), 2047-2053 (2013).
- Choi, J., et al. A rapid antimicrobial susceptibility test based on single-cell morphological analysis. Science Translational Medicine. 6 (267), (2014).
- Barczak, A. K., Hung, D. T. RNA signatures allow rapid identification of pathogens and antibiotic susceptibilities. Proceedings of the National Academy of Sciences. 109 (16), 6217-6222 (2012).
- Schoepp, N. G., et al. Rapid pathogen-specific phenotypic antibiotic susceptibility testing using digital LAMP quantification in clinical samples. Science Translational Medicine. 9 (410), (2017).
- Novelli-Rousseau, A., et al. Culture-free antibiotic-susceptibility determination from single-bacterium Raman spectra. Scientific Reports. 8 (1), 1-12 (2018).
- Schröder, U. -. C., et al. Detection of vancomycin resistances in enterococci within 3 1/2 hours. Scientific Reports. 5, 8217 (2015).
- Liu, C. -. Y., et al. Rapid bacterial antibiotic susceptibility test based on simple surface-enhanced Raman spectroscopic biomarkers. Scientific Reports. 6 (1), 1-15 (2016).
- Chang, K. -. W., et al. Antibiotic susceptibility test with surface-enhanced raman scattering in a microfluidic system. Analytical Chemistry. 91 (17), 10988-10995 (2019).
- Galvan, D. D., Yu, Q. surface-enhanced raman scattering for rapid detection and characterization of antibiotic-resistant bacteria. Advanced Healthcare Materials. 7 (13), 1701335 (2018).
- Kirchhoff, J., et al. Simple ciprofloxacin resistance test and determination of minimal inhibitory concentration within 2 h using raman spectroscopy. Analytical Chemistry. 90 (3), 1811-1818 (2018).
- Zhang, Z., Chen, L., Liu, L., Su, X., Rabinowitz, J. D. Chemical basis for deuterium labeling of fat and NADPH. Journal of the American Chemical Society. 139 (41), 14368-14371 (2017).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nature Communications. 9 (1), 2995 (2018).
- Berry, D., et al. Tracking heavy water (D2O) incorporation for identifying and sorting active microbial cells. Proceedings of the National Academy of Sciences. 112 (2), 194-203 (2015).
- Tao, Y., et al. Metabolic-activity-based assessment of antimicrobial effects by D2O-labeled single-cell raman microspectroscopy. Analytical Chemistry. 89 (7), 4108-4115 (2017).
- Yang, K., et al. Rapid antibiotic susceptibility testing of pathogenic bacteria using heavy water-labeled single-cell raman spectroscopy in clinical samples. Analytical Chemistry. 91 (9), 6296-6303 (2019).
- Song, Y., et al. Raman-Deuterium Isotope Probing for in-situ identification of antimicrobial resistant bacteria in Thames River. Scientific reports. 7 (1), 16648 (2017).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Cheng, J. -. X., Xie, X. S. Vibrational spectroscopic imaging of living systems: An emerging platform for biology and medicine. Science. 350 (6264), (2015).
- Zhang, C., Zhang, D., Cheng, J. -. X. Coherent Raman scattering microscopy in biology and medicine. Annual Review of Biomedical Engineering. 17, 415-445 (2015).
- Yue, S., Cheng, J. -. X. Deciphering single cell metabolism by coherent Raman scattering microscopy. Current Opinion in Chemical Biology. 33, 46-57 (2016).
- Hu, F., Shi, L., Min, W. Biological imaging of chemical bonds by stimulated Raman scattering microscopy. Nature Methods. 16 (9), 830-842 (2019).
- Ji, M., et al. Rapid, Label-free detection of brain tumors with stimulated Raman scattering microscopy. Science Translational Medicine. 5 (201), (2013).
- He, R., Liu, Z., Xu, Y., Huang, W., Ma, H., Ji, M. Stimulated Raman scattering microscopy and spectroscopy with a rapid scanning optical delay line. Optics Letters. 42 (4), 659-662 (2017).
- Suzuki, Y., et al. Label-free chemical imaging flow cytometry by high-speed multicolor stimulated Raman scattering. Proceedings of the National Academy of Sciences. 116 (32), 15842-15848 (2019).
- Camp, C. H., et al. High-Speed Coherent Raman Fingerprint Imaging of Biological Tissues. Nature Photonics. 8, 627-634 (2014).
- Zhang, M., et al. Rapid determination of antimicrobial susceptibility by stimulated raman scattering imaging of D2O metabolic incorporation in a single bacterium. Advanced Science. 7 (19), 2001452 (2020).
- Michael, I., et al. A fidget spinner for the point-of-care diagnosis of urinary tract infection. Nature Biomedical Engineering. 4 (6), 591-600 (2020).
- Bhattacharyya, R. P., et al. Simultaneous detection of genotype and phenotype enables rapid and accurate antibiotic susceptibility determination. Nature Medicine. 25 (12), 1858-1864 (2019).
- Stupar, P., et al. Nanomechanical sensor applied to blood culture pellets: a fast approach to determine the antibiotic susceptibility against agents of bloodstream infections. Clinical Microbiology and Infection. 23 (6), 400-405 (2017).
- Barber, A. E., Norton, J. P., Spivak, A. M., Mulvey, M. A. Urinary Tract Infections: Current and Emerging Management Strategies. Clinical Infectious Diseases. 57 (5), 719-724 (2013).
- Cohen, J., et al. Sepsis: a roadmap for future research. The Lancet Infectious Diseases. 15 (5), 581-614 (2015).
- Choi, J., et al. rapid antimicrobial susceptibility test from positive blood cultures based on microscopic imaging analysis. Scientific Reports. 7 (1), 1148 (2017).
- Gherardi, G., et al. Comparative evaluation of the Vitek-2 Compact and Phoenix systems for rapid identification and antibiotic susceptibility testing directly from blood cultures of Gram-negative and Gram-positive isolates. Diagnostic Microbiology and Infectious Disease. 72 (1), 20-31 (2012).
- Machen, A., Drake, T., Wang, Y. F. Same day identification and full panel antimicrobial susceptibility testing of bacteria from positive blood culture bottles made possible by a combined lysis-filtration method with MALDI-TOF VITEK mass spectrometry and the VITEK2 system. Plos One. 9, 87870 (2014).
- Simon, L., et al. Direct identification of 80 percent of bacteria from blood culture bottles by matrix-assisted laser desorption ionization-time of flight mass spectrometry using a 10-minute extraction protocol. Journal of Clinical Microbiology. 57 (2), 01278 (2019).
- Leekha, S., Terrell, C. L., Edson, R. S. General principles of antimicrobial therapy. Mayo Clinic Proceedings. 86 (2), 156-167 (2011).
- Johnson, L., et al. Emergence of fluoroquinolone resistance in outpatient urinary Escherichia coli isolates. The American Journal of Medicine. 121 (10), 876-884 (2008).
- Van Belkum, A., et al. Developmental roadmap for antimicrobial susceptibility testing systems. Nature Reviews Microbiology. 17 (1), 51-62 (2019).
- Dubourg, G., Lamy, B., Ruimy, R. Rapid phenotypic methods to improve the diagnosis of bacterial bloodstream infections: meeting the challenge to reduce the time to result. Clinical Microbiology and Infection. 24 (9), 935-943 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon