Method Article
Génération de cellules souches pluripotentes induites à partir de cellules fœtales du syndrome de Turner (45XO) pour la modélisation en aval des déficits neurologiques associés au syndrome
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit la génération de CSI sans intégration à partir de fibroblastes de tissu fœtal par administration de plasmides épisomiques par nucléofection suivie d’une description des méthodes utilisées pour la caractérisation de l’iPSC et la différenciation neuronale.
Résumé
Les aneuploïdies chromosomiques provoquent de graves malformations congénitales, notamment des malformations du système nerveux central et la mort fœtale. Le dépistage génétique prénatal est purement diagnostique et n’élucide pas le mécanisme de la maladie. Bien que les cellules des fœtus aneuploïdes soient du matériel biologique précieux portant l’aneuploïdie chromosomique, ces cellules sont de courte durée, ce qui limite leur utilisation pour les expériences de recherche en aval. La génération de modèles de cellules souches pluripotentes induites (CSIp) est une méthode efficace de préparation cellulaire pour la conservation perpétuelle des caractères aneuploïdes. Ils se renouvellent d’eux-mêmes et se différencient en cellules spécialisées qui rappellent le développement embryonnaire. Ainsi, les CSPe constituent d’excellents outils pour étudier les événements de développement précoces. Le syndrome de Turner (TS) est une affection rare associée à un chromosome X complètement ou partiellement manquant. Le syndrome est caractérisé par l’infertilité, la petite taille, les troubles endocriniens, métaboliques, auto-immuns et cardiovasculaires et les défauts neurocognitifs. Le protocole suivant décrit l’isolement et la culture de fibroblastes à partir de tissu fœtal TS (45XO), la génération de TSiPSC sans intégration par l’administration de plasmides de reprogrammation épisomique par nucléofection suivie d’une caractérisation. Les TSiPSC de reprogrammation ont d’abord été examinés par coloration de la phosphatase alcaline des cellules vivantes, suivie d’un sondage approfondi pour les biomarqueurs de pluripotence. Les colonies sélectionnées ont été disséquées mécaniquement, ont été croisées plusieurs fois et des cellules stables auto-renouvelées ont été utilisées pour d’autres expériences. Les cellules ont exprimé les facteurs de transcription de pluripotence OCT4, NANOG, SOX2, les marqueurs de surface cellulaire SSEA 4 et TRA1-81 typiques des cellules souches pluripotentes. Le caryotype 45XO original a été conservé après la reprogrammation. Les TSiPSC ont pu former des corps embryoïdes et se différencier en cellules d’endoderme, de mésoderme et d’ectoderme exprimant des biomarqueurs spécifiques à la lignée ((SRY BOX17), (MYOSIN VENTRICULAR HEAVY CHAINα/β), (βIII TUBULIN)). Les plasmides épisomiques exogènes ont été perdus spontanément et non détectés après le passage 15 dans les cellules. Ces TSiPSC sont une ressource cellulaire précieuse pour modéliser le neurodéveloppement moléculaire et cellulaire défectueux provoquant des déficits neurocognitifs associés au syndrome de Turner.
Introduction
Les aneuploïdiens entraînent des malformations congénitales et des pertes de grossesse chez l’homme. ~ 50% à 70% des échantillons de pertes de grossesse présentent des anomalies cytogénétiques. Les embryons aneuploïdes perdus au début de la grossesse ne peuvent pas être facilement obtenus pour une analyse expérimentale, ce qui soulève la nécessité de développer d’autres modèles représentant étroitement l’embryogenèse humaine. Des cellules souches pluripotentes induites (CSI) dérivées de cellules diagnostiquées avec des troubles génétiques ont été utilisées pour modéliser les irrégularités génétiques représentatives et leurs conséquences sur le développement du fœtus1,2,3,4. Ces CSPi ressemblent à des cellules épiblastiques de l’embryon en développement et peuvent récapituler les événements précoces de la formation de l’embryon. Ils permettent de comprendre et de caractériser le programme de développement des lignées cellulaires et des modèles chez les embryons de mammifères précoces. CSPi dérivées précédemment de fibroblastes cutanés et d’amniocytes provenant de tests de diagnostic prénatal de syndromes d’aneuploïdie comme la monosomie X (syndrome de Turner), la trisomie 8 (syndrome de Warkany 2), la trisomie 13 (syndrome de Patau) et la trisomie partielle 11; 22 (syndrome d’Emanuel) ont fourni des informations précieuses concernant l’échec du développement4.
Le syndrome de Turner (TS) est une maladie rare caractérisée par l’infertilité féminine, la petite taille, des troubles endocriniens et métaboliques, un risque accru de maladie auto-immune et une prédisposition aux maladiescardiovasculaires 5. Bien qu’il s’agit du seul syndrome de monosomie survivable, il est également mortel pour l’embryon en développement provoquant des avortements spontanés6. Les individus survivants atteints de TS présentent des degrés d’altération du matériel chromosomique X dans leurs cellules. Les caryotypes vont de la perte complète d’un chromosome X (45,XO) à des mosaïques comme 45,XO/46,XX; 45,XO/47,XXX, la présence de chromosomes annulaires, la présence de matériel chromosomique Y,etc. 5.
Le diagnostic du syndrome se fait généralement par caryotypage du sang des individus symptomatiques et prélèvement de villosités chorioniques (CVS) pour détecter les syndromes d’aneuploïdie précoce. Étant donné que les syndromes d’aneuploïdie représentent environ 30% des avortements spontanés, il est courant de caryotyper le produit de la conception (POC) lors d’un avortement spontané. Ces cellules fœtales, y compris les villosités chorioniques possédant l’anomalie cytogénétique et les CSPi qui en sont dérivées, constituent une source précieuse de matériel biologique pour étudier les syndromes d’aneuploïdie4,6. Les CSI TS ont déjà été établies à partir d’amniocytes via la reprogrammation rétrovirale4,de fibroblastes de villosités chorioniques (obtenus par diagnostic prénatal) via une reprogrammation rétrovirale6,de cellules mononucléaires sanguines7 via la reprogrammation du virus Sendai et de fibroblastes cutanés d’individus TS via une reprogrammation lentivirale4 . Étant donné que l’objectif principal de notre laboratoire est de comprendre l’échec du développement, nous avons généré des CST iPSC à partir de POC, en particulier la composante des villosités chorioniques d’un avortement spontané8. Toutes les cellules isolées de ce tissu fœtal avaient un caryotype 45XO et produisaient des CS IPC avec le même caryotype. Ces CSPe sont uniques car elles sont les premières à être générées à partir d’un fœtus avorté et constituent une ressource précieuse pour étudier les échecs de grossesse liés à l’aneuploïdie. Cet article fournit une méthodologie détaillée de la génération de CSPic à partir de cette source cellulaire unique via la reprogrammation épisomique.
Les premières méthodes de génération d’iPSC utilisaient la transduction virale et les transposons pour fournir les facteurs de reprogrammation. Les méthodes d’induction des cellules à la pluripotence ont évolué de l’intégration des vecteurs rétroviraux9,des vecteurs lentiviraux excisables10,11 et des méthodes basées sur les transposons12 aux vecteurs adénoviraux non intégrateurs13 et aux vecteurs à base du virus Sendai14. La reprogrammation rétrovirale et lentivirale, bien qu’efficace, implique l’intégration des facteurs de reprogrammation dans les chromosomes de l’hôte, provoquant des mutations d’insertion qui ont des effets imprévus dans les CSI. De plus, la reprogrammation virale empêche l’application translationnelle des CSI. Les systèmes à base d’ARN15 et d’administration directe de protéines16 ont été explorés pour éliminer complètement les risques potentiels associés à l’utilisation de virus et de transfections d’ADN. Cependant, ces méthodes se sont avérées inefficaces.
En 2011, Okita et al. ont rapporté une amélioration de l’efficacité de la reprogrammation par des plasmides épisomiques augmentés par la suppression de TP53 via shRNA. Ils ont également remplacé le cMYC par un LMYC (MYC associé au carcinome pulmonaire à petites cellules) non transformant pour améliorer la sécurité des hiPSC. Ces plasmides épisomiques expriment 5 facteurs de reprogrammation : OCT4, LIN28, SOX2, KLF4, LMYC et shRNA pour TP5317,18. Ces vecteurs sont maintenus extra-chromosomiquement et perdus des cellules reprogrammées lors d’une culture continue, rendant ainsi les lignes sans transgène dans 10-15 passages. La nucléofection est une forme spécialisée d’électroporation qui délivre des acides nucléiques directement dans le noyau des cellules hôtes. C’est une méthode efficace pour l’administration des plasmides de reprogrammation dans divers types de cellules. Les plasmides épisomiques sont rentables et compensent les coûts élevés de la nucléofection. Cette méthode est efficace et reproductible dans des conditions optimisées, produisant des CSP stables à partir d’une variété de cellules somatiques. Dans ce protocole, nous décrivons la méthode de génération de CSI i à partir de fibroblastes isolés du tissu fœtal par nucléofection de plasmides de reprogrammation épisomique. Voici les protocoles détaillés pour l’isolement des fibroblastes des villosités chorioniques fœtales, la purification des plasmides, la nucléofection, la cueillette des colonies à partir de la plaque de reprogrammation et l’établissement de CSI stables.
Il est essentiel de confirmer la présence de traits de pluripotence dans les IPSC nouvellement générés. Cela comprend la démonstration de facteurs liés à la pluripotence (par exemple, expression de la phosphatase alcaline, NANOG, SSEA4, Tra 1-80, Tra 1-81, E-cadhérine; généralement montré avec des tests d’immunofluorescence ou d’expression génique), l’identification des trois couches germinales par des tests de différenciation in vitro pour valider leurs potentiels de différenciation, le caryotypage pour déterminer le contenu chromosomique, le typage STR pour établir l’identité avec les cellules mères, vérifier la perte de gènes exogènes, et des essais in vivo plus rigoureux tels que la formation de tératome et la complémentation tétraploïde. Nous décrivons ici les protocoles de caractérisation du caryotypage, la coloration de la phosphatase alcaline des cellules vivantes, la détection de biomarqueurs liés à la pluripotence par immunofluorescence, les tests de différenciation in vitro et la méthode pour démontrer la perte de gènes exogènes19.
Protocole
Les FCV ont été obtenus de l’hôpital Manipal, à Bangalore, sous l’approbation du comité d’éthique des hôpitaux Manipal.
REMARQUE : Voir le tableau 1 pour la composition de tous les tampons et solutions.
1. Isolement des fibroblastes des villosités chorioniques fœtales (FCV)
-
Prélèvement d’échantillons et désintégration tissulaire dans la collagénase
- Recueillir le FCV dans des conditions stériles dans une solution saline tamponnée au phosphate (PBS) et le transporter (à température ambiante) vers l’installation de culture cellulaire.
- Transférer les villosités dans une boîte de Petri de 60 mm et laver plusieurs fois (minimum 4 fois) dans du PBS contenant 1x solution antibiotique antimycotique (PBS-AA). Retirez complètement le PBS-AA par pipetage.
- Traiter les villosités chorioniques avec 1 mL de mélange de collagénase (5 mg/mL) pendant 5 min à 37 °C.
- Neutraliser avec un milieu de culture cellulaire contenant 10% de sérum fœtal bovin (FBS), transférer la digestion dans un tube de 15 mL et centrifuger à 225 x g pendant 5 minutes pour recueillir les villosités désintégrées et les cellules libérées sous forme de granulés.
-
Sous-culture et expansion des stocks
- Les villosités désintégrées sur plaque ainsi que les cellules libérées dans une fiole de culture T25 contenant 5 mL de milieu complet (par exemple, AmnioMAX) et se développent jusqu’à l’obtention d’une culture de fibroblastes confluents.
- Développer les fibroblastes en culture pour préparer les stocks en pour les utiliser dans des expériences ultérieures de transfection et de caractérisation comme suit:
- Ajouter 2 mL de trypsine à 0,05 % dans une fiole de T25 contenant des fibroblastes FCV et incuber à 37 °C pendant 3 à 5 min pour dissocier les cellules.
- Après l’incubation, neutraliser la trypsine en ajoutant fbS (au même volume que la trypsine).
REMARQUE: Le milieu de culture contenant du FBS peut également être utilisé pour neutraliser la trypsine, lorsqu’il est ajouté à un rapport trypsine: média de 1: 3. - Prélever les cellules dissociées dans un tube de 15 mL et centrifuger à 225 x g pendant 5 min pour obtenir une pastille de cellule.
- Décant surnageant et resuspendé la pastille de cellule dans 1 mL de milieu complet.
- Transférer 500 μL chacun dans des boîtes de 60 mm traitées par culture tissulaire et porter le volume à 5 mL. Ce rapport de division de 1:2 a également été utilisé pour les passages suivants.
-
Cryoconservation
- Effectuer une dissociation enzymatique à l’aide de 0,05% de trypsine comme décrit aux étapes 1.2.2.1 - 1.2.2.3 et obtenir une pastille cellulaire.
- Jeter le surnageant et ressusser la pastille cellulaire dans 1 mL de mélange de congélation, comprenant du sulfoxyde de diméthyle (DMSO) 1:9: FBS.
- Transférer le contenu dans un cryovial stérile et placer le flacon dans un récipient de congélation.
- Congeler pendant la nuit à -80 °C, puis transférer les flacons dans de l’azote liquide (-196 °C) le lendemain.
2. Isolement et vérification de l’ADN des plasmides
-
Préparation de cellules bactériennes
- Strier les stocks de glycérol d’E. coli contenant les trois plasmides individuels pCXLE-hOCT3/4-shp53-F, pCXLE-hSK et pCXLE-hUL (d’Addgene) sur des plaques de gélose Luria Bertani-ampicilline séparées.
- Inoculer des colonies uniques dans des cultures de démarrage de 5 mL de milieu Luria Bertani-ampicilline. Incuber pendant 8 heures à 37 °C en agitant (10 x g).
- Inoculer 200 μL de cette culture de départ dans 100 mL de milieu Luria Bertani-ampicilline. Incuber toute la nuit à 37 °C en agitant.
- Récolter la culture bactérienne pendant la nuit en centrifugant à 6000 x g pendant 15 min à 4 °C.
-
Isolation plasmidique avec kit de purification plasmidique Midi
- Resuspendez la pastille bactérienne dans un tampon de resuspension de 4 mL.
- Ajouter 4 mL de tampon de lyse et bien mélanger en inversant vigoureusement 4 à 6 fois et incuber à température ambiante pendant 5 min.
- Ajouter 4 mL de tampon de neutralisation pré-refroidi et inverser le tube 4 à 6 fois pour bien mélanger. Incuber sur de la glace pendant 15 min.
- Centrifuger à ≥20 000 x g pendant 30 min à 4 °C. Recueillir le surnageant dans un tube frais et recentrififer à ≥20 000 x g pendant 15 min à 4 °C.
- Équilibrez la colonne en appliquant 4 mL de tampon d’équilibrage.
- Appliquez le surnageant à la colonne.
- Lavez la colonne deux fois avec 10 mL de tampon de lavage.
- Elute ADN avec 5 mL de tampon d’élution chaud (65 °C).
- Précipiter l’ADN en ajoutant 3,5 mL d’isopropanol à l’ADN élué. Bien mélanger. Centrifuger à ≥15 000 x g pendant 30 min à 4 °C. Décantantantant soigneusement.
- Laver la pastille d’ADN avec 2 mL d’éthanol à 70 % et centrifuger à ≥15 000 x g pendant 10 min. Décantantantant soigneusement.
- Sécher à l’air les granulés pendant 5 à 10 min et dissoudre l’ADN dans un volume approprié d’eau de qualité PCR pour obtenir une concentration finale de 1 μg/mL.
REMARQUE: Ne dissolvez pas l’ADN dans les tampons car il ne convient pas à l’électroporation. Les anciennes préparations d’ADN plasmidique ne produisent pas de colonies reprogrammées.
-
Vérification des plasmides par digestion de restriction EcoRI
- Combiner 15 μL d’eau sans nucléase, 2 μL de tampon 10x, 1 μg d’ADN plasmidique et 1 μL d’enzyme EcoRI. Mélanger doucement.
- Incuber le mélange à 37 °C pendant 15 min dans un bloc chauffant.
- Mélanger les échantillons de plasmides digérés avec 6x colorant de chargement de gel d’ADN et électrophorèse sur du gel d’agarose à 1% dans 1x tampon TAE avec 0,5 μg / mL de bromure d’éthidium. Inclure une échelle d’ADN standard. Imagez le gel après que l’ADN se soit résolu de manière appropriée. Les tailles de bande EcoRI attendues de pCXLE-hOCT3 / 4-shp53-F sont de 6 834 pb, 3 758 pb et 1 108 pb; pCXLE-hUL sont de 10 200 pb et 1 900 pb; pCXLE-hSK sont de 10 200 bp et 2 500 bp.
3. Nucléofection
-
Granulation de cellules
- Cultiver les fibroblastes de villosités chorioniques fœtales isolés dans une fiole de T25 dans 5 mL de milieu complet jusqu’à 80-90% de confluence.
- Lavez les cellules deux fois avec du PBS et essayez-les comme décrit aux étapes 1.2.2.1 - 1.2.2.3.
- Retirer le surnageant, resussuspender la pastille dans 5 mL de milieu de sérum réduit (p. ex. Opti-MEM). Compter les cellules avec hémocytomètre et prendre 106 cellules pour la nucléofection. Centrifuger à 225 x g pendant 5 min. Retirez complètement le surnageant.
-
Préparation et nucléofection de réactifs
- Préparer le réactif du nucléofectoriel en mélangeant 0,5 mL de supplément et 2,25 mL de solution de nucléofector (tous deux fournis dans le kit).
- Ajouter 100 μL de solution de nucléofectoriel dans un tube de 1,5 mL. Ajouter 1μg de pCXLE-hOCT3/4-shp53-F, pCXLE-hSK et pCXLE-hUL au tube. Remettez doucement en question 106 cellules (à partir de l’étape 3.1.2) dans ce mélange.
- Transférer la suspension d’ADN cellulaire dans une cuvette, en veillant à ce que l’échantillon recouvre le fond de la cuvette (fournie en kit) sans bulles d’air. Coiuchez la cuvette et insérez-la dans le support. Sélectionnez le programme de nucléofectoriel U-23 (pour une efficacité élevée) et appliquez.
- Retirer la cuvette du support et ajouter 1 mL de support complet. Transférer doucement le contenu dans une boîte de Petri traitée par culture tissulaire de 60 mm remplie de 4 mL de milieu complet (pour un total de 5 mL de milieu). Incuber les cellules dans un incubateur à CO2 humidifié à 37 °C.
- Après 24 h, vérifiez si les cellules se sont attachées. Remplacez complètement le support.
REMARQUE: Le taux de mort cellulaire est élevé en nucléofection, laissant peu de cellules viables qui se fixent. - Maintenez les cellules dans un milieu complet pendant 10 jours et passez au milieu de pluripotence pendant les 20 prochains jours.
REMARQUE: Visualisez régulièrement les cellules pour suivre les changements morphologiques qui se produisent dans les cellules de reprogrammation (comme la morphologie épithéliale et la formation de colonies compactes) pour confirmer si l’expérience fonctionne. Environ 25 colonies d’iPSC peuvent être observées après 20 jours de culture dans des milieux de pluripotence.
4. Cueillette et propagation des colonies de CS IPC
-
Cueillette de la colonie à partir de la plaque de reprogrammation
- Disséquez manuellement les colonies embryonnaires ressemblant à des cellules souches formées dans le plat de reprogrammation à l’aide de pipettes en verre tiré ou d’aiguilles de seringue de 1 mL et transférez-les sur une plaque préalablement préparée avec des mangeoires de fibroblastes embryonnaires de souris inactivées avec milieu de pluripotence ou établissez des cultures sans mangeoire sur des plaques revêtues de Matrigel avec un milieu mTESR.
REMARQUE: Les fibroblastes embryonnaires de souris (MEF) ont été dérivés en utilisant l’isolement enzymatique d’embryons de souris (disséqués à partir de souris femelles enceintes de 13 à 14 jours) et ont été inactivés mitotiquement par le traitement à la mitomycine C. Établir des populations de clones uniques en cultivant des colonies uniques à partir de plaques de reprogrammation dans des plats séparés ou des populations de clones mixtes en transférant de nombreuses colonies de la plaque de reprogrammation à un seul plat.
- Disséquez manuellement les colonies embryonnaires ressemblant à des cellules souches formées dans le plat de reprogrammation à l’aide de pipettes en verre tiré ou d’aiguilles de seringue de 1 mL et transférez-les sur une plaque préalablement préparée avec des mangeoires de fibroblastes embryonnaires de souris inactivées avec milieu de pluripotence ou établissez des cultures sans mangeoire sur des plaques revêtues de Matrigel avec un milieu mTESR.
-
Transfert mécanique des colonies émergentes vers des mangeoires fraîches et passaging pour établir des CSPe stables
- Propager les CSPi dans un milieu de pluripotence en les nourrissant tous les deux jours et fractionner 1:3 tous les 5 à 7 jours. Préparer les stocks en cryoconservant dans un mélange de congélation de KnockOut Serum Replacement et de DMSO dans un rapport de 9:1.
REMARQUE: KnockOut Serum Replacement est utilisé dans le mélange de congélation pour la cryoconservation des CSI au lieu de FBS, car les composants du FBS pourraient induire une différenciation des cellules pluripotentes pendant la conservation à long terme.
- Propager les CSPi dans un milieu de pluripotence en les nourrissant tous les deux jours et fractionner 1:3 tous les 5 à 7 jours. Préparer les stocks en cryoconservant dans un mélange de congélation de KnockOut Serum Replacement et de DMSO dans un rapport de 9:1.
5. Caractérisation des CS IPC
NOTE: Des études de caractérisation, y compris la PCR et l’immunocoloration pour le biomarqueur de pluripotence, ont été effectuées après le cinquième numéro de passage. Le caryotypage a été effectué à un numéro de passage ultérieur.
- Caryotypage
- Traiter une boîte de Petri confluente de 60 mm de CSI avec du colcémide pendant 45 min dans un incubateur à CO2 humidifié à 37 °C.
- Récolte par 0,05% de traitement à la trypsine et centrifugeuse. Retirez le surnageant et pipetez les traces restantes de milieu pour desserrer la pastille de cellule.
- Ajouter 5 mL de solution hypotonique. Mélanger par tube inversé et incuber pendant 20 minutes à 37 °C. Centrifuger à 225 x g pendant 5 min.
REMARQUE: Le granulé obtenu doit sembler moelleux. - Ajouter lentement 2,5 mL de solution fixatrice de Carnoy, tout en tapotant pour desserrer la pastille.
- Préparez les tartinades pour le caryotypage en déposant la suspension cellulaire sur des lames de verre propres.
- Traiter les lames avec 0,15% de trypsine pendant 1 minute et laver une fois avec du PBS. Ensuite, tachez avec la solution de Giemsa pendant 4 min et terminez par un lavage à l’eau distillée. Acquérir et traiter avec un logiciel approprié.
- Démonstration du statut de transgène libre
-
Isolement génomique de l’ADN
- Pipette 20 μL de protéase dans le fond d’un tube de microcentrifugation de 1,5 mL.
- Ajouter 200 μL de TSiPSCs mis enssais dans le PBS au tube de microcentrifugation.
- Ajouter 200 μL de Buffer AL à l’échantillon et mélanger pendant 15 s par vortex d’impulsions.
- Incuber pendant 10 min à 56 °C.
- Centrifugez brièvement le tube de microcentrifugation pour éliminer les gouttes de l’intérieur du couvercle.
- Ajouter 200 μL d’éthanol (96-100%) à l’échantillon, et mélanger à nouveau pendant 15 s par vortex d’impulsions. Après le mélange, centrifugez brièvement le tube pour éliminer les gouttes de l’intérieur du couvercle.
- Appliquez soigneusement le mélange de l’étape précédente sur la mini colonne de rotation (dans un tube de collecte de 2 mL) sans mouiller la jante. Fermez le bouchon et centrifugez à 6000 x g pendant 1 min. Jetez le tube contenant le filtrat et placez la mini colonne de spin dans un tube de collecte propre de 2 mL.
- Ouvrez soigneusement la mini colonne de rotation et sans mouiller la jante, ajoutez 500 μL de Buffer AW1. Fermer le bouchon et centrifuger à 6000 x g pendant 1 min. Placez la mini colonne de rotation dans un tube de collecte propre de 2 mL et jetez le tube de collecte contenant le filtrat.
- Ouvrez soigneusement la mini colonne de rotation et ajoutez 500 μL de Buffer AW2 sans mouiller la jante. Fermez le bouchon et centrifugez à pleine vitesse (20 000 x g) pendant 3 min.
- Placez la mini colonne de rotation dans un nouveau tube de collecte de 2 mL et jetez l’ancien tube de collecte avec le filtrat. Centrifugez à pleine vitesse pendant 1 min pour éliminer le risque de transfert possible du tampon AW2.
- Placez la mini colonne de rotation dans un tube de microcentrifugation propre de 1,5 mL et jetez le tube de collecte contenant le filtrat. Ouvrez soigneusement la mini colonne de rotation et ajoutez 200 μL de Buffer AE ou d’eau distillée. Incuber à température ambiante (15-25 °C) pendant 5 min, puis centrifuger à 6000 x g pendant 1 min.
-
PCR d’état sans transgène (à l’aide du kit de PCR HiFi KAPA KR0368)
- Assurez-vous que tous les réactifs sont correctement décongelés et mélangés.
- Préparer un mélange maître PCR contenant le volume approprié de tous les composants de la réaction en fonction du tableau 2 (mettre en place des réactions sur la glace).
- Transférez les volumes appropriés de mélange maître, de gabarit et d’amorce de PCR dans des tubes pcR individuels.
- Capuchons les réactions individuelles, mélangez et centrifugez brièvement.
- Effectuez la PCR en suivant le tableau 3.
-
Isolement génomique de l’ADN
- Identification des biomarqueurs de pluripotence
-
Coloration à la phosphatase alcaline (AP)
- Préparer une solution de travail de teinture vivante 1x AP en diluant 3 μL de solution mère 500x dans 1,5 mL de DMEM/F-12 pour chaque 10 cm2 de surface de culture.
- Retirez le milieu de la boîte de culture iPSC. Lavez la culture avec DMEM/F-12 une fois. Ajoutez la solution de coloration sous vie 1x AP sur les iPSC. Incuber à 37 °C pendant 45 min.
- Retirez la tache AP et lavez deux fois avec DMEM/F-12. Ajoutez du DMEM/F-12 frais et une image au microscope fluorescent à l’aide d’un filtre FITC standard dans les 30 à 90 minutes de coloration.
-
Immunocoloration pour les biomarqueurs de pluripotence
- Fixer des cultures iPSC confluentes avec 4% de paraformaldéhyde pendant la nuit à 4 °C. Laver trois fois avec PBS Tween 20 (PBST), chaque lavage pendant 5 min.
- Perméabiliser les cellules avec 0,3% de Triton X-100 dans PBST pendant 15 minutes à température ambiante. Laver trois fois avec PBST.
REMARQUE: La perméabilisation ne doit être effectuée que pour les antigènes intracellulaires. - Bloquer les cellules avec 3% d’albumine sérique bovine (BSA) dans le PBST pendant 30 min à température ambiante. Colorez les cellules avec des anticorps primaires (dilués 1:1000 dans 1% de BSA) pendant la nuit. Après l’incubation primaire des anticorps, laver trois fois avec pbST.
- Incuber les cellules avec l’anticorps secondaire (dilué 1:1000 dans 1% de BSA) pendant 5 h à température ambiante. Laver trois fois avec PBST.
- Étiqueter les noyaux avec 0,5 μg/mL de 4',6-diamidino-2-phénylindole (DAPI) pendant 1 minute. Lavez les cellules une fois avec PBST.
- Capturez des images au microscope fluorescent.
-
Coloration à la phosphatase alcaline (AP)
6. Essais de différenciationin vitro
- Différenciation du corps embryoïde (EB)
- Couper les colonies d’iPSC en petits morceaux, recueillir et plaquer dans des boîtes de Petri à faible attachement dans un milieu de corps embryoïde. Cultivez les cellules pendant 15 jours en remplaçant le milieu tous les 3 jours.
REMARQUE: Le jour 15 EBs peut être utilisé directement pour la détection des trois biomarqueurs de la couche germinale. Alternativement, des lignées cellulaires spécifiques peuvent être induites par des facteurs de croissance, suivies de la détection de biomarqueurs.
- Couper les colonies d’iPSC en petits morceaux, recueillir et plaquer dans des boîtes de Petri à faible attachement dans un milieu de corps embryoïde. Cultivez les cellules pendant 15 jours en remplaçant le milieu tous les 3 jours.
- Différenciation de l’endoderme (hépatocyte)
- Cultivez les iPSC dans des cultures monocouches en milieu de pluripotence.
- Une fois confluent, passez au milieu RPMI 1640 avec 1x sélénite de transferrine d’insuline et 100 ng / mL d’activine A pendant 2 jours, suivi d’une croissance du milieu RPMI 1640 avec 30 ng / mL bFGF et 20 ng / mL BMP4 pendant 9 jours. Remplacer le moyen tous les 2 jours.
- À partir du jour 10, complétez le milieu avec 0,1 μM de dexaméthasone. Terminez l’expérience le jour 20.
- Différenciation du mésoderme (cardiomyocyte)
- Plaque jour 8 EBs sur des plaques enduites de Matrigel à 0,5% dans un milieu de corps embryoïde. Autorisez les ABE à s’attacher et à se réduire.
- Complétez les milieux avec 20 ng / mL BMP4 et cultivez pendant 20 jours. Remplacer le milieu tous les 2-3 jours. Terminez l’expérience le jour 20.
- Différenciation ectodermique (neuronale)
- Plaque jour 4 EBs sur 2 μg/cm2 plaques enduites de collagène de type IV dans un milieu corporel embryoïde. Autorisez les ABE à s’attacher et à se réduire.
- Le lendemain, déplacez le milieu vers DMEM F-12 avec 2 mg / mL de glucose, 1x sélénite de transferrine d’insuline et 2,5 μg / mL de fibronectine. Terminez l’expérience le jour 15.
- Formation d’organoïdes cérébraux
- Cultivez des TSiPSC dans une boîte de culture tissulaire de 35 mm sur des MEF jusqu’à 70-80% confluent. Coupez les colonies et collectez dans un tube de 15 mL. Centrifuger les cellules à 225 x g pendant 5 min. Jetez le surnageant.
- Laver les morceaux de colonies en les remettant en jubasse dans 2 mL de PBS et centrifuger pour enlever le surnageant.
- Ajouter 1 mL de trypsine à 0,05 % et tapoter le tube pour déloger les cellules. Incuber le tube à 37 °C pendant 3-4 min pour dissocier les morceaux de colonie en une suspension à cellule unique.
- Neutraliser la trypsine par dilution avec 4 mL de milieux de pluripotence contenant 10 μg/mL d’inhibiteur de la protéine kinase rho-associée (ROCK) Y-27632 dichlorhydrate (ROCKi) pour prévenir la mort cellulaire induite par la dissociation.
- Centrifuger pour obtenir une pastille. Jeter le surnageant et ressusser les cellules dans un milieu de corps embryoïde de 2 mL contenant 10 μg/mL de ROCKi.
- Retirer 10 μL de suspension cellulaire pour le comptage cellulaire. Ajouter 10 μL de bleu trypan pour détecter les cellules mortes. Comptez les cellules à l’aide d’un hémocytomètre.
- Ajouter un volume approprié de milieu corporel embryoïde avec ROCKi à la suspension cellulaire pour obtenir 9 000 cellules vivantes par 150 μL.
- Plaque 150 μL dans chaque puits d’une plaque à 96 puits à faible fixation et incuber dans un incubateur à CO2 humidifié à 37 °C. Vérifiez l’agrégation des plaques après 24 heures. Le jour 2, retirez doucement le milieu et remplacez-le par un milieu embryside frais sans ROCKi.
- Le jour 6, transférer les OE vers des puits d’une plaque de 24 puits à faible fixation contenant 500 μL de milieu d’induction neuronal composé de DMEM-F12 avec supplément de N2 à 1%, supplément de GlutaMAX à 2 mM et 1 mM d’acides aminés non essentiels et 1 μg/mL d’héparine. Changez le support tous les 2 jours.
- Après 5 jours dans un milieu d’induction neuronale, incorporez les agrégats neuroépithéliaux dans Matrigel en superposant un carré de parafilm de 2 cm x 2 cm sur un plateau de pointe vide de 200 μL. Appuyez sur le parafilm avec les doigts gantés sur chaque trou du plateau de pointe pour faire de petites bosses. Nettoyez le parafilm avec 70% d’éthanol à stériliser.
- Transférer le carré de parafilm dans un plat de 60 mm. Utilisez des pointes coupées de 200 μL pour transférer les agrégats neuroépithéliaux sur les bosses dans le parafilm. Enlever l’excès de milieu par pipetage.
- Ajouter 30 μL de Matrigel décongelé sur les agrégats neuroépithliens et positionner l’agrégat au centre du Matrigel à l’aide d’une pointe de pipette. Placez le plat de 60 mm pendant 20 à 30 min dans un incubateur à 37 °C pour que le Matrigel se polymérise.
- Ajouter 5 mL de milieu de différenciation organoïde cérébrale composé de 1:1 DMEM-F12: milieu neurobasal, supplément de N2 à 0,5%, 2,5 μg / mL d’insuline, supplément de GlutaMAX de 2 mM, 0,5 mM de NEAA, supplément de B27 à 1% et 2,5 mL de pénicilline-streptomycine.
- À l’aide d’une pince stérile, retournez la feuille de parafilm et agitez le plat jusqu’à ce que les agrégats incorporés par Matrigel tombent de la feuille dans le milieu. Cultivez les agrégats incorporés dans un incubateur à CO2 humidifié à 37 °C pendant 4 jours en donnant des changements de média un jour sur deux.
- Après 4 jours de culture statique, placez les plats de 60 mm sur un agitateur orbital installé à l’intérieur de l’incubateur à 50 tr / min. Culture des organoïdes pendant 3 mois en donnant des changements complets du milieu de milieu de différenciation organoïde cérébrale tous les 3 jours.
Résultats
Génération de CSPe sans intégration à partir d’un fœtus avorté spontanément avec un caryotype 45XO
Nous avons isolé des fibroblastes de FCV avec un caryotype 45XO spécifique du syndrome de Turner (TS) et les avons nucléofectés avec des plasmides de reprogrammation épisomique pour générer des TSiPSC qui peuvent être utilisés pour la modélisation en aval du syndrome, en particulier les déficits neurologiques associés(Figure 1a& b). Nous avons utilisé des vecteurs épisomiques non intégrateurs et la nucléofection pour les expériences de transfection(Figure 1 c&d). Nous avons suivi les changements morphologiques des cellules pour surveiller le succès de la reprogrammation. Le passage du fibroblaste à la morphologie épithéliale, suivi d’une formation de colonie compacte délimitée, a été observé (Figure 2a). Les TSiPSC ont acquis une morphologie semblable à celle des cellules souches embryonnaires humaines avec des bords distincts et un rapport noyau/cytoplasme élevé vers le 20e jour après la transfection (Figure 2b). En revanche, les cellules incomplètement reprogrammées acquièrent des morphologies épithéliales mais ne parviennent pas à former des colonies compactes. (Figure 2c).
Caractérisation des TSiPSC
Le caryotypage des TSiPSC a révélé le caryotype 45XO associé au syndrome de Turner(Figure 3a). L’immunofluorescence des TSiPSC a montré l’expression des facteurs de transcription de pluripotence OCT4, NANOG, SOX2 et des marqueurs de surface cellulaire SSEA4, E-Cadherin et TRA-1-81. Les cellules souches embryonnaires humaines sont l’étalon-or des cellules souches pluripotentes. Nous avons simultanément effectué une immunofluorescence de HUES 1 qui a été utilisé comme témoin positif pour la comparaison de l’expression des biomarqueurs de pluripotence par TSiPSC (Figure 3b). Le statut sans transgène des TSiPSC a été démontré par une PCR génomique de l’ADN pour les marqueurs plasmidiques épisomiques OriP et EBNA. Au passage 15, les gènes OriP et EBNA ont été perdus dans les TSiPSC.Les gènes épisomaux OriP et EBNA ont été amplifiés et ont montré des bandes dans le passage 9 TSiPSC indiquant la présence des plasmides épisomiques à ce stade. Cependant, ces gènes n’ont pas été amplifiés dans le passage 15 des TSiPSC indiquant une perte des plasmides épisomiques et donc un état libre transgénique (Figure 3c).
Essais de différenciation in vitro
Le potentiel de différenciation des lignées TSiPSC a été démontré in vitro. Les TSiPSC lors de l’agrégation dans des plaques à faible fixation formaient des corps embryoïques(Figure 4a). La différenciation induite par le facteur de croissance des TSiPSC a été utilisée pour générer des types cellulaires des trois couches germinales. L’analyse d’immunofluorescence à l’aide de biomarqueurs spécifiques à la lignée a confirmé que les TSiPSC se différenciaient en dérivés représentatifs de l’endoderme (SOX17), du mésoderme (MYOSIN VENTRICULAR HEAVY CHAINα/β) et de l’ectoderme (βIII TUBULIN)(Figure 4b).
Différenciation organoïde cérébrale.
Les TSiPSC ont été différenciés en organoïdes cérébraux de manière progressive. Les suspensions unicellulaires de TSiPSC ont été agrégées en corps embryoïques pour stimuler le développement des couches germinales pendant les 6 premiers jours, suivis de l’induction du développement neuroépithélial pendant 5 jours. Les neuroépithéliales agrégés étaient intégrés dans Matrigel qui fournissait la matrice extracellulaire et les composants de la membrane basale qui soutiennent une orientation apicobasale appropriée, excroissance de bourgeons neuroépithéliaux qui se dilatent et forment des lumens. L’immunofluorescence avec le marqueur neuroépithélial NESTIN a été réalisée pour observer la morphologie globale des organoïdes (Figure 5b). Le neuroépithélium entoure une cavité semblable à un ventricule(Figure 5c - ligne blanche). Les organoïdes présentent morphologiquement des zones ventriculaires (VZ), des zones sous-ventriculaires (SVZ) et des régions semblables au cortex(Figure 5c - lignes rouges, oranges et jaunes respectivement)

Figure 1: Isolement et reprogrammation des fibroblastes par nucléofection. (a) Image microscopique des villosités chorioniques fœtales avant le traitement à la collagénase. b)Fibroblastes isolés des villosités choriales fœtales pour des expériences de reprogrammation. c)Vérification de la reprogrammation des plasmides par digestion de restriction EcoRI. (d) Schéma du protocole de transfection utilisé pour la génération d’iPSC à partir de fibroblastes de villosités chorioniques fœtales à l’aide de plasmides de reprogrammation épisomique par nucléofection. Veuillez cliquer ici pour voir une version agrandie de cette figure.
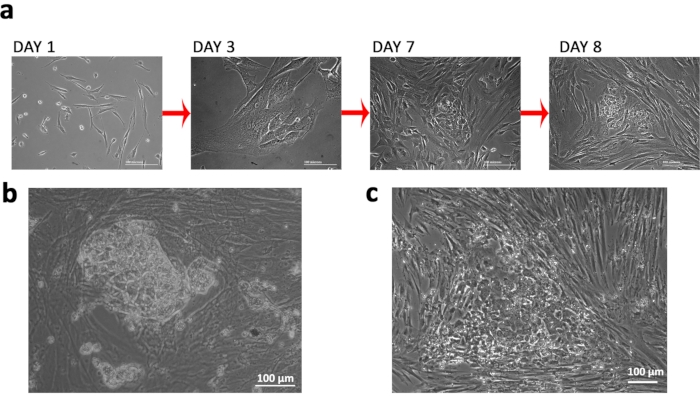
Figure 2: Établissement de cellules souches pluripotentes induites par le syndrome de Turner. (a) Changements de morphologie cellulaire observés au cours de la reprogrammation. b)Une colonie TSiPSC entièrement reprogrammée. c)Image représentative d’une colonie avec des cellules mal reprogrammées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
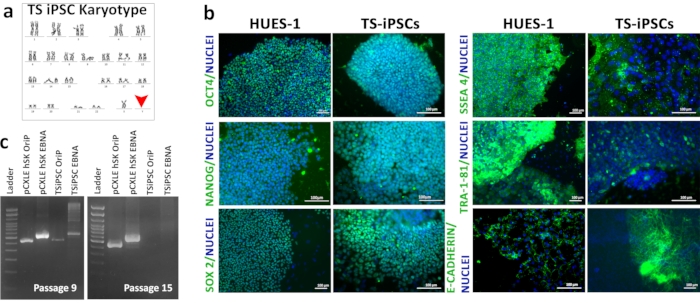
Figure 3: Caractérisation des TSiPSC. a)Caryotype des TSiPSC. b)Analyse par immunofluorescence des biomarqueurs de pluripotence OCT4, NANOG, SOX2, SSEA-4 et TRA-1-81 dans les TSiPSC par rapport aux cellules souches embryonnaires HUES1. Les noyaux sont colorés avec du 4', 6-diamidino-2-phénylindole. 3c. Démonstration du statut sans transgène des TSiPSC. Voie 1 - Échelle d’ADN, Voie 2 - Contrôle positif OriP avec pCXLE-hSK, Voie 3 - Contrôle positif EBNA avec pCXLE-hSK, Voie 4-OriP avec TSiPSC, Voie 5-EBNA avec TSiPSC. Veuillez cliquer ici pour voir une version agrandie de cette figure.
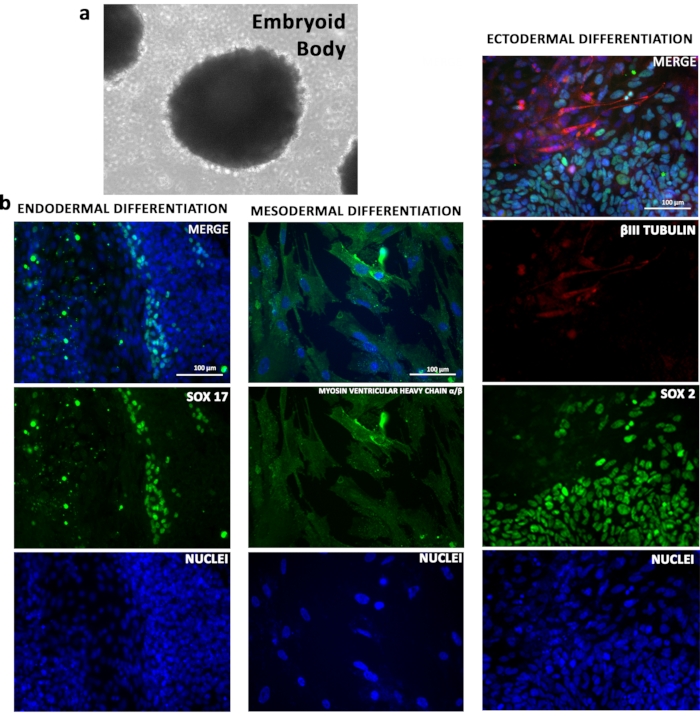
Figure 4: Potentiel de différenciation in vitro des TSiPSC. (a) TSiPSC différencié en corps embryoïques. b)Analyses d’immunofluorescence des CSIPC pour le marqueur endodermique SOX17, le marqueur mésodermique myosine ventriculaire à chaîne lourde α/β et les marqueurs ectodermiques βIII tubuline et SOX2. Les noyaux sont colorés avec du 4', 6-diamidino-2-phénylindole. Veuillez cliquer ici pour voir une version agrandie de cette figure.
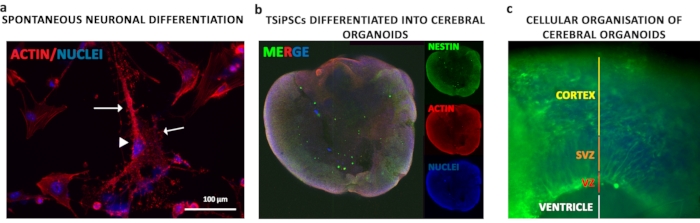
Figure 5: Différenciation organoïde neuronale et cérébrale des TSiPSC. ( a ) Pour comprendre la cytoarchitecture des neurones différenciés,unecoloration phalloïdine de l’actine a été effectuée. Les neurones dérivés de TSiPSC présentaient un soma neuronal de forme pyramidale (pointe de flèche) avec des dendrites et des axones (flèches). Les noyaux sont colorés avec du 4', 6-diamidino-2-phénylindole. Immunocoloration. (a) Immunocoloration de Nestin et d’actine pour observer la morphologie brute des organoïdes. (c) Coloration pour Nestin afin de visualiser les couches neuronales organisées de manière apicale et basale. Les noyaux sont colorés avec du 4', 6-diamidino-2-phénylindole. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Composition des supports, des tampons et des solutions Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Mélange de réactions pcr Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Programme de cyclisme PCR Veuillez cliquer ici pour télécharger ce tableau.
Discussion
La génération de modèles cellulaires stables de tissu fœtal cytogénétiquement anormal est nécessaire pour perpétuer un phénotype défectueux. La voie iPSC est la méthode la plus efficace de préparation cellulaire pour la conservation perpétuelle des propriétés défectueuses20.
Les cellules souches pluripotentes (CSE) présentent des propriétés d’auto-renouvellement et de différenciation en cellules spécialisées qui rappellent les embryons de clivage précoce21. Par conséquent, les CSE peuvent servir d’excellents modèles pour étudier les anomalies moléculaires, cellulaires et développementales précoces chez les fœtus prématurément avortés.
Dans cet article, nous avons décrit la génération humaine d’iPSC à l’aide de la nucléofection combinée aux vecteurs épisomiques améliorés. Les résultats montrent que cette combinaison comprend une méthode robuste pour générer des lignes iPSC humaines sans intégration, comme en témoigne le fait que des transfections uniques étaient suffisantes pour une reprogrammation réussie. Nous avons suivi la conversion progressive des fibroblastes FCV en cellules pluripotentes au microscope. 20 jours après la transfection, nous avons observé des colonies de TSiPSC reprogrammés entourés de fibroblastes FCV non reprogrammés. Morphologiquement, les CSP humaines dérivées ressemblaient à des cellules souches embryonnaires cultivées en laboratoire. Typiquement, les cellules agrégées comme des colonies compactes avec des bordures brillantes. Les cellules des colonies avaient de gros noyaux et étroitement emballées, ce qui suggère un contact membranaire étroit entre les cellules. Les fibroblastes non reprogrammés cambraient et entouraient ces colonies. Lors de leur transfert vers les iMEF, ils continuent de proliférer dans la culture pendant plus de 30 transferts, démontrant la propriété d’un auto-renouvellement continu.
Comme les TSiPSC ont été générés à partir de fibroblastes 45XO, nous avons caryotypé les cellules pour vérifier si elles conservaient la composition chromosomique. Les TSiPSC ont maintenu le caryotype 45XO en culture cellulaire continue suggérant une constitution génétique stable du chromosome 45XO. Pour être utiles en tant que ressource cellulaire représentant l’aneuploïdie 45XO, les TSiPSC doivent être exempts d’ADN exogène utilisé dans les expériences de reprogrammation. Nous avons vérifié la présence de plasmides épisomiques résiduels en effectuant une PCR génomique de l’ADN pour les marqueurs épisomiques spécifiques - OriP et EBNA. Nous n’avons trouvé aucune trace de ces marqueurs dans les cellules TSiPS après 15 passages suggérant que les TSiPSC perdaient progressivement des vecteurs de reprogrammation épisomique en culture prolongée.
La caractéristique d’une cellule pluripotente est son potentiel à se différencier en cellules de trois lignées germinales à la fois in vitro et in vivo. Pour tester cette capacité dans les TSiPSC dérivés, nous les avons soumis in vitro à des tests de formation et de différenciation du corps embryoïde dirigés par la lignée spécifiant les cytokines et les facteurs de croissance. Les TSiPSC ont formé des corps embryoïques et se sont différenciés en cellules ectodermiques exprimant des marqueurs neuronaux, en cellules mésodermiques exprimant des marqueurs cardiaques et en cellules endodermiques exprimant SOX17 un biomarqueur du destin endodermique. Nous avons également testé la capacité des TSiPSC à se différencier en organoïdes cérébraux 3D d’ordre supérieur en utilisant des protocoles précédemment établis22. Les TSiPSC s’auto-organisent progressivement en raison de leurs propres programmes de développement intrinsèques en mini-tissus appelés organoïdes. Les TSiPSC ont donné des organoïdes cérébraux montrant une cytoarchitecture similaire au tissu cérébral avec un neuroépithhélium entourant une cavité semblable à un ventricule. Cependant, ces organoïdes doivent être caractérisés de manière plus approfondie pour révéler les types de cellules exacts et comparés aux IPSC normaux pour distinguer les propriétés intrinsèques de modelage des tissus neuronaux des TSiPSC. Ces organoïdes cérébraux et d’autres types d’organoïdes cérébraux générés par les TSiPSC peuvent être utilisés pour modéliser les incohérences développementales et fonctionnelles qui peuvent contribuer aux symptômes de déficiences neurologiques des personnes TS. Les TSiPSC présentaient des caractéristiques biomarqueurs de pluripotence ainsi que le trait caractéristique de différenciation, soulignant ainsi le succès de la reprogrammation en pluripotence induite.
La méthode décrite ci-dessus a fonctionné efficacement dans la reprogrammation des fibroblastes dermiques et des cellules mésenchymateuses dérivées de diverses sources dans notre laboratoire (données d’autres lignées non montrées). D’après notre expérience, les étapes suivantes sont essentielles au succès de l’expérience de reprogrammation :
a) Qualité de la préparation plasmidique : les anciennes préparations ne donnent pas de CSI.
b) Qualité des cellules utilisées pour les transfections : les cellules proliférantes sont essentielles à la génération d’iPSC. 0,5 à 1 million de cellules par transfection ont donné une efficacité de reprogrammation reproductible.
c) Réactifs de nucléofectoriels fraîchement reconstitués : les réactifs de nucléofectoriel reconstitués stockés pendant plus d’un mois n’ont pas produit de CSI.
d) La maintenance de la banque de cellules maîtresses par sous-culture mécanique des IPSC a donné des lignées stables. La dissociation enzymatique a été utilisée conformément aux exigences de l’expérience.
L’objectif futur du laboratoire est d’établir un panel de CSI chromosomiquement anormales pour le développement en aval, la modélisation fonctionnelle et pathologique en utilisant cette méthode efficace. Les aneuploïdoïdes fœtales provoquent des pertes de grossesse et des malformations d’organes lors des naissances vivantes. Les CSI aneuploïdes dérivées de tissus d’avortements spontanés sont une ressource précieuse pour modéliser et étudier les événements de développement embryonnaire échoués. Les systèmes de culture in vitro 2D et 3D, y compris les corps embryoïdes et les organoïdes spécifiques aux tissus22, permettront aux chercheurs de comprendre les irrégularités moléculaires et cellulaires telles que la prolifération cellulaire aberrante et la mort cellulaire dans des cellules spécifiques à la lignée qui pourraient se manifester par des anomalies de développement et des échecs de grossesse associés à des syndromes d’aneuploïdie.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le soutien financier pour la recherche ci-dessus a été fourni par Manipal Academy of Higher Education. La caractérisation de la lignée a été réalisée en partie dans le laboratoire de M.M. Panicker au NCBS. Nous remercions Anand Diagnostic Laboratory pour son aide avec le caryotypage.
matériels
| Name | Company | Catalog Number | Comments |
| 0.15% trypsin | Thermo Fisher Scientific | 27250018 | G Banding |
| 2-mercaptoethanol | Thermo Fisher Scientific | 21985023 | Pluripotency and Embryoid body medium |
| 4', 6 diamidino-2-phenylindole | Sigma Aldrich | D8417 | Immunocytochemistry |
| Activin A | Sigma Aldrich | SRP3003 | Differentiation assays |
| Alkaline Phosphatase Live Stain | Thermo Fisher Scientific | A14353 | AP staining |
| AMAXA Nucleofector II | Lonza | - | Nucleofection |
| AmnioMAX II complete media | Thermo Fisher Scientific, Gibco | 11269016 | Medium specific for foetal chorionic villi cell cultures |
| Ampicillin | HiMedia | TC021 | Plasmid purification |
| Anti Mouse IgG (H+L) Alexa Fluor 488 | Invitrogen | A11059 | Immunocytochemistry |
| Anti Rabbit IgG (H+L) Alexa Fluor 488 | Invitrogen | A11034 | Immunocytochemistry |
| Anti Rabbit IgG (H+L) Alexa Fluor 546 | Invitrogen | A11035 | Immunocytochemistry |
| Antibiotic-Antimycotic | Thermo Fisher Scientific, Gibco | 15240096 | Contamination control |
| Anti-E-Cadherin | BD Biosciences | 610181 | Immunocytochemistry |
| Anti-Nanog | BD Biosciences | 560109 | Immunocytochemistry |
| Anti-OCT3/4 | BD Biosciences | 611202 | Immunocytochemistry |
| Anti-SOX17 | BD Biosciences | 561590 | Immunocytochemistry |
| Anti-SOX2 | BD Biosciences | 561469 | Immunocytochemistry |
| Anti-SSEA4 | BD Biosciences | 560073 | Immunocytochemistry |
| Anti-TRA 1-81 | Millipore | MAB4381 | Immunocytochemistry |
| basic Fibroblast Growth Factor[FGF2] | Sigma Aldrich | F0291 | Pluripotency medium |
| Bone Morphogenetic Factor 4 | Sigma Aldrich | SRP3016 | Differentiation assays |
| Bovine Serum Albumin | Sigma Aldrich | A3059 | Blocking |
| Collagen Human Type IV | BD Biosciences | 354245 | Differentiation assays |
| Collagenase blend | Sigma Aldrich | C8051 | Digestion of foetal chorionic villi |
| Dexamethasone | Sigma Aldrich | D4902 | Differentiation assays |
| DMEM F12 | Thermo Fisher Scientific | 11320033 | Differentiation assays |
| FastDigest EcoR1 | Thermo Scientific | FD0274 | Restriction digestion |
| Fibronectin | Sigma Aldrich | F2518 | Differentiation assays |
| Giemsa Stain | HiMedia | S011 | G Banding |
| Glacial Acetic Acid | HiMedia | AS001 | Fixative for karyotyping |
| Glucose | Sigma Aldrich | G7528 | Differentiation assays |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | Pluripotency and Embryoid body medium |
| Heparin sodium | Sigma Aldrich | H3149 | Differentiation assays |
| Insulin solution human | Sigma Aldrich | I9278 | Differentiation assays |
| Insulin Transferrin Selenite | Sigma Aldrich | I1884 | Differentiation assays |
| KAPA HiFi PCR kit | Kapa Biosystems | KR0368 | OriP, EBNA1 PCR |
| KaryoMAX Colcemid | Thermo Fisher Scientific | 15210040 | Mitotic arrest for karyotyping |
| KnockOut DMEM | Thermo Fisher Scientific | 10829018 | Pluripotency and Embryoid body medium |
| KnockOut Serum Replacement | Thermo Fisher Scientific | 10828028 | Pluripotency and Embryoid body medium |
| Luria Bertani agar | HiMedia | M1151F | Plasmid purification |
| Matrigel | BD Biosciences | 356234 | Differentiation assays |
| MEM Non-essential amino acids | Thermo Fisher Scientific | 11140035 | Pluripotency and Embryoid body medium |
| Methanol | HiMedia | MB113 | Fixative for karyotyping |
| Myosin ventricular heavy chain α/β | Millipore | MAB1552 | Immunocytochemistry |
| NHDF Nucleofector Kit | Lonza | VAPD-1001 | Nucleofection |
| Paraformaldehyde (PFA) | Sigma Aldrich | P6148 | Fixing cells |
| pCXLE-hOCT3/ 4-shp53-F | Addgene | 27077 | Episomal reprogramming Plasmid |
| pCXLE-hSK | Addgene | 27078 | Episomal reprogramming Plasmid |
| pCXLE-hUL | Addgene | 27080 | Episomal reprogramming Plasmid |
| Penicillin Streptomycin | Thermo Fisher Scientific, | 15070063 | Pluripotency and Embryoid body medium |
| Phalloidin- Tetramethylrhodamine B isothiocyanate | Sigma Aldrich | P1951 | Immunocytochemistry |
| Phosphate buffered saline | Sigma Aldrich | P4417 | 1 X PBS 1 tablet of PBS dissolved in 200mL of deionized water and sterilized by autoclaving Storage: Room temperature. PBST- 0.05% Tween 20 in 1X PBS. Storage: Room temperature. |
| Plasmid purification Kit- Midi prep | QIAGEN | 12143 | Plasmid purification |
| Potassium Chloride Solution | HiMedia | MB043 | Hypotonic solution for karyotyping |
| QIAamp DNA Blood Kit | Qiagen | 51104 | Genomic DNA isolation |
| RPMI 1640 | Thermo Fisher Scientific | 11875093 | Hepatocyte differentiation medium |
| Sodium Citrate | HiMedia | RM255 | Hypotonic solution for karyotyping |
| Triton X-100 | HiMedia | MB031 | Permeabilisation |
| Trypsin-EDTA (0.05%) | Thermo Fisher Scientific, Gibco | 25300054 | Subculture of foetal chorionic villi fibroblasts |
| Tween 20 | HiMedia | MB067 | Preparation of PBST |
| β III tubulin | Sigma Aldrich | T8578 | Immunocytochemistry |
| Y-27632 dihydrochloride | Sigma Aldrich | Y0503 | Differentiation assays |
Références
- Verlinsky, Y., et al. Human embryonic stem cell lines with genetic disorders. Reproductive BioMedicine Online. 10, 105-110 (2005).
- Eiges, R., et al. Developmental Study of Fragile X Syndrome Using Human Embryonic Stem Cells Derived from Preimplantation Genetically Diagnosed Embryos. Cell Stem Cell. 1, 568-577 (2007).
- Biancotti, J. -. C., et al. Human Embryonic Stem Cells as Models for Aneuploid Chromosomal Syndromes. STEM CELLS. 28, 1530-1540 (2010).
- Li, W., et al. Modeling abnormal early development with induced pluripotent stem cells from aneuploid syndromes. Human Molecular Genetics. 21, 32-45 (2012).
- Gravholt, C. H., Viuff, M. H., Brun, S., Stochholm, K., Andersen, N. H. Turner syndrome: mechanisms and management. Nature Reviews Endocrinology. 15, 601-614 (2019).
- Luo, Y., et al. Uniparental disomy of the entire X chromosome in Turner syndrome patient-specific induced pluripotent stem cells. Cell Discovery. 1, 15022 (2015).
- Luo, Y., et al. Generation of an induced pluripotent stem cell line from an adult male with 45,X/46,XY mosaicism. Stem Cell Research. 27, 42-45 (2018).
- Parveen, S., Panicker, M. M., Gupta, P. K. Generation of an induced pluripotent stem cell line from chorionic villi of a Turner syndrome spontaneous abortion. Stem Cell Research. 19, (2017).
- Takahashi, K., et al. Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors. Cell. 131, 861-872 (2007).
- Soldner, F., et al. Parkinson's Disease Patient-Derived Induced Pluripotent Stem Cells Free of Viral Reprogramming Factors. Cell. 136, 964-977 (2009).
- Somers, A., et al. Generation of Transgene-Free Lung Disease-Specific Human Induced Pluripotent Stem Cells Using a Single Excisable Lentiviral Stem Cell Cassette. STEM CELLS. 28, 1728-1740 (2010).
- Woltjen, K., et al. piggyBac transposition reprograms fibroblasts to induced pluripotent stem cells. Nature. 458, 766-770 (2009).
- Stadtfeld, M., Nagaya, M., Utikal, J., Weir, G., Hochedlinger, K. Induced Pluripotent Stem Cells Generated Without Viral Integration. Science. 322, 945-949 (2008).
- Fusaki, N., Ban, H., Nishiyama, A., Saeki, K., Hasegawa, M. Efficient induction of transgene-free human pluripotent stem cells using a vector based on Sendai virus, an RNA virus that does not integrate into the host genome. Proceedings of the Japan Academy, Series B. 85, 348-362 (2009).
- Warren, L., et al. Highly Efficient Reprogramming to Pluripotency and Directed Differentiation of Human Cells with Synthetic Modified mRNA. Cell Stem Cell. 7, 618-630 (2010).
- Kim, D., et al. Generation of human induced pluripotent stem cells by direct delivery of reprogramming proteins. Cell Stem Cell. 4, 472-476 (2009).
- Yu, J., et al. Human Induced Pluripotent Stem Cells Free of Vector and Transgene Sequences. Science. 324, 797-801 (2009).
- Okita, K., et al. A more efficient method to generate integration-free human iPS cells. Nature Methods. 8, 409-412 (2011).
- Martí, M., et al. Characterization of pluripotent stem cells. Nature Protocols. 8, 223-253 (2013).
- Avior, Y., Sagi, I., Benvenisty, N. Pluripotent stem cells in disease modelling and drug discovery. Nature Reviews Molecular Cell Biology. 17, 170-182 (2016).
- Ambartsumyan, G., Clark, A. T. Aneuploidy and early human embryo development. Human Molecular Genetics. 17, 10-15 (2008).
- Lancaster, M. A., Knoblich, J. A. Generation of cerebral organoids from human pluripotent stem cells. Nature Protocols. 9, 2329-2340 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon