Method Article
Generierung induzierter pluripotenter Stammzellen aus dem Turner-Syndrom (45XO) fetaler Zellen zur nachgelagerten Modellierung neurologischer Defizite im Zusammenhang mit dem Syndrom
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Erzeugung von integrationsfreien iPS-Zellen aus fetalen Gewebefibroblasten durch Abgabe episomaler Plasmide durch Nukleofektion, gefolgt von der Beschreibung von Methoden zur iPSC-Charakterisierung und neuronalen Differenzierung.
Zusammenfassung
Chromosomale Aneuploidien verursachen schwere angeborene Fehlbildungen, einschließlich Fehlbildungen des zentralen Nervensystems und fetalem Tod. Das pränatale genetische Screening ist rein diagnostisch und klärt den Krankheitsmechanismus nicht auf. Obwohl Zellen von aneuploiden Föten wertvolles biologisches Material sind, das die chromosomale Aneuploidie trägt, sind diese Zellen kurzlebig, was ihre Verwendung für nachgelagerte Forschungsexperimente einschränkt. Die Generierung von modellen für induzierte pluripotente Stammzellen (iPSC) ist eine effektive Methode der Zellvorbereitung zur ständigen Erhaltung aneuploider Merkmale. Sie erneuern sich selbst und differenzieren sich zu spezialisierten Zellen, die an die Embryonalentwicklung erinnern. Daher dienen iPS-Zellen als hervorragende Werkzeuge, um frühe Entwicklungsereignisse zu untersuchen. Das Turner-Syndrom (TS) ist eine seltene Erkrankung, die mit einem ganz oder teilweise fehlenden X-Chromosom einhergeht. Das Syndrom ist gekennzeichnet durch Unfruchtbarkeit, Kleinwuchs, endokrine, metabolische, autoimmune und kardiovaskuläre Störungen und neurokognitive Defekte. Das folgende Protokoll beschreibt die Isolierung und Kultivierung von Fibroblasten aus TS (45XO) fetalem Gewebe, die Erzeugung von integrationsfreien TSiPS-Zellen durch Abgabe von episomalen Reprogrammierungsplasmiden durch Nukleofektion mit anschließender Charakterisierung. Die reprogrammierenden TSiPS-Zellen wurden zunächst durch lebendzellige alkalische Phosphatase-Färbung gescreent, gefolgt von umfangreichen Untersuchungen nach Pluripotenz-Biomarkern. Ausgewählte Kolonien wurden mechanisch seziert, mehrmals durchquert und stabile, sich selbst erneuernde Zellen für weitere Experimente verwendet. Die Zellen exprimierten Pluripotenz-Transkriptionsfaktoren OCT4, NANOG, SOX2, Zelloberflächenmarker SSEA 4 und TRA1-81, die typisch für pluripotente Stammzellen sind. Der ursprüngliche 45XO-Karyotyp wurde nach der Neuprogrammierung beibehalten. Die TSiPS-Zellen waren in der Lage, embryoide Körper zu bilden und sich in Zellen von Endoderm, Mesoderm und Ektoderm zu differenzieren, die linienspezifische Biomarker exprimieren ((SRY BOX17), (MYOSIN VENTRICULAR HEAVY CHAINα/β), (βIII TUBULIN)). Die exogenen episomalen Plasmide gingen spontan verloren und wurden nach Passage 15 in Zellen nicht nachgewiesen. Diese TSiPS-Zellen sind eine wertvolle zelluläre Ressource für die Modellierung defekter molekularer und zellulärer Neuroentwicklung, die neurokognitive Defizite im Zusammenhang mit dem Turner-Syndrom verursachen.
Einleitung
Aneuploidien führen beim Menschen zu Geburtsfehlern/angeborenen Fehlbildungen und Schwangerschaftsverlust. ~ 50% -70% der Proben aus Schwangerschaftsverlusten zeigen zytogenetische Anomalien. Aneuploide Embryonen, die früh in der Schwangerschaft verloren gehen, können nicht einfach für experimentelle Analysen gewonnen werden, was die Notwendigkeit erhöht, andere Modelle zu entwickeln, die die menschliche Embryogenese genau repräsentieren. Induzierte pluripotente Stammzellen (iPS-Zellen), die aus Zellen gewonnen wurden, bei denen genetische Störungen diagnostiziert wurden, wurden verwendet, um die repräsentativen genetischen Unregelmäßigkeiten und ihre Folgen für die fetale Entwicklung zu modellieren1,2,3,4. Diese iPS-Zellen ähneln Epiblastenzellen des sich entwickelnden Embryos und können die frühen Ereignisse der Embryonenbildung rekapitulieren. Sie ermöglichen das Verständnis und die Charakterisierung des Entwicklungsprogramms von Zelllinien und Mustern in frühen Säugetierembryonen. iPS-Zellen, die zuvor aus Hautfibroblasten und Amniozyten aus pränatalen diagnostischen Tests von Aneuploidie-Syndromen wie Monosomie X (Turner-Syndrom), Trisomie 8 (Warkany-Syndrom 2), Trisomie 13 (Patau-Syndrom) und partieller Trisomie 11 abgeleitet wurden; 22 (Emanuel-Syndrom) haben wertvolle Erkenntnisse über gescheiterte Entwicklungen geliefert4.
Das Turner-Syndrom (TS) ist eine seltene Erkrankung, die durch weibliche Unfruchtbarkeit, Kleinwuchs, endokrine und metabolische Störungen, ein erhöhtes Risiko für Autoimmunerkrankungen und eine Prädisposition für Herz-Kreislauf-Erkrankungen gekennzeichnet ist5. Obwohl es das einzige überlebensfähige Monosomie-Syndrom ist, ist es auch tödlich für den sich entwickelnden Embryo, der spontane Abtreibungen verursacht6. Überlebende Individuen mit TS zeigen einen Grad der Veränderung von X-chromosomalem Material in ihren Zellen. Karyotypen reichen vom vollständigen Verlust eines X-Chromosoms (45,XO) bis hin zu Mosaiken wie 45,XO/46,XX; 45,XO/47,XXX, das Vorhandensein von Ringchromosomen, das Vorhandensein von Y-chromosomalem Material usw.5.
Die Diagnose des Syndroms erfolgt im Allgemeinen durch Karyotypisierung von Blut von symptomatischen Personen und Chorionzottenbiopsie (CVS), um frühe Aneuploidie-Syndrome zu erkennen. Da Aneuploidie-Syndrome für ~ 30% der spontanen Abtreibungen verantwortlich sind, ist es Routine, das Produkt der Empfängnis (POC) nach einer spontanen Abtreibung zu karyotypisieren. Diese fetalen Zellen, einschließlich der Chorionzotten, die die zytogenetische Anomalie besitzen, und die daraus abgeleiteten iPS-Zellen bieten eine wertvolle Quelle für biologisches Material zur Untersuchung der Aneuploidie-Syndrome4,6. TS iPS-Zellen wurden zuvor aus Amniozyten durch retrovirale Reprogrammierung4, Fibroblasten von Chorionzotten (erhalten durch pränatale Diagnostik) durch retrovirale Reprogrammierung6, aus mononukleären Blutzellen7 durch Sendai-Virus-Reprogrammierung und aus Hautfibroblasten von TS-Individuen durch lentivirale Reprogrammierung4 . Da das Hauptaugenmerk unseres Labors auf dem Verständnis von Entwicklungsstörungen liegt, haben wir TS iPSCs aus POC erzeugt, insbesondere die Chorionzottenkomponente einer spontanen Abtreibung8. Alle aus diesem fötalen Gewebe isolierten Zellen hatten einen 45XO-Karyotyp und ergaben iPS-Zellen mit dem gleichen Karyotyp. Diese iPS-Zellen sind einzigartig, da sie die ersten sind, die von einem abgetriebenen Fötus erzeugt werden und eine wertvolle Ressource zur Untersuchung von aneuploidiebedingten Schwangerschaftsfehlern darstellen. Dieser Artikel bietet eine detaillierte Methodik der Erzeugung von iPS-Zellen aus dieser einzigartigen Zellquelle durch episomale Reprogrammierung.
Die frühen Methoden der iPSC-Generierung verwendeten virale Transduktion und Transposons, um die Reprogrammierungsfaktoren zu liefern. Methoden zur Induktion von Zellen zur Pluripotenz haben sich von der Integration retroviralerVektoren 9,erregbaren lentiviralenVektoren 10,11 und Transposon-basierten Methoden12 zu nicht-integrierenden adenoviralenVektoren 13 und Sendai-Virus-basierten Vektoren14entwickelt. Retrovirale und lentivirale Reprogrammierung, obwohl effizient, beinhaltet die Integration der Reprogrammierungsfaktoren in die Wirtschromosomen, was zu Insertionsmutationen führt, die unvorhergesehene Auswirkungen in den iPS-Zellen haben. Darüber hinaus verhindert die virale Reprogrammierung die translationale Anwendung von iPS-Zellen. RNA-basierte Systeme15 und direkte Proteinabgabe16 wurden untersucht, um die potenziellen Risiken, die mit der Verwendung von Viren und DNA-Transfektionen verbunden sind, vollständig zu eliminieren. Diese Methoden haben sich jedoch als ineffizient erwiesen.
Im Jahr 2011 berichteten Okita et al. über eine verbesserte Effizienz der Reprogrammierung durch episomale Plasmide, die mit TP53-Unterdrückung über shRNA verstärkt wurden. Sie ersetzten cMYC auch durch nicht transformierendes LMYC (small cell lung carcinoma associated MYC), um die Sicherheit der hiPS-Zellen zu erhöhen. Diese episomalen Plasmide exprimieren 5 Reprogrammierungsfaktoren: OCT4, LIN28, SOX2, KLF4, LMYC und shRNA für TP5317,18. Diese Vektoren werden extrachromosomal beibehalten und gehen bei kontinuierlicher Kultur von den reprogrammierten Zellen verloren, wodurch die Linien innerhalb von 10-15 Passagen transgenfrei werden. Die Nukleofektion ist eine spezialisierte Form der Elektroporation, die Nukleinsäuren direkt in den Kern der Wirtszellen abgibt. Es ist eine effiziente Methode zur Abgabe der reprogrammierenden Plasmide in verschiedene Zelltypen. Episomale Plasmide sind kostengünstig und kompensieren die hohen Kosten der Nukleofektion. Diese Methode ist effizient und reproduzierbar unter optimierten Bedingungen und liefert stabile iPS-Zellen aus einer Vielzahl von somatischen Zellen. In diesem Protokoll beschreiben wir die Methode zur Erzeugung von iPS-Zellen aus Fibroblasten, die aus fetalem Gewebe durch Nukleofektion von episomalen Reprogrammierungsplasmiden isoliert wurden. Hier sind die detaillierten Protokolle für die Fibroblastenisolierung aus fetalen Chorionzotten, Plasmidreinigung, Nukleofektion, Pflücken von Kolonien von der Reprogrammierungsplatte und Etablierung stabiler iPS-Zellen.
Es ist wichtig, das Vorhandensein von Pluripotenzmerkmalen in den neu erzeugten iPS-Zellen zu bestätigen. Dazu gehören der Nachweis von pluripotenzbezogenen Faktoren (z. B. alkalische Phosphataseexpression, NANOG, SSEA4, Tra 1-80, Tra 1-81, E-Cadherin; in der Regel mit Immunfluoreszenz- oder Genexpressionsassays gezeigt), die Identifizierung der drei Keimschichten durch In-vitro-Differenzierungstests zur Validierung ihres Differenzierungspotenzials, die Karyotypisierung zur Bestimmung des Chromosomengehalts, die STR-Typisierung zur Feststellung der Identität mit Elternzellen, die Überprüfung des Verlusts exogener Gene. und strengere In-vivo-Assays wie Teratombildung und tetraploide Komplementierung. Hier beschreiben wir Charakterisierungsprotokolle der Karyotypisierung, die alkalische Phosphatasefärbung lebender Zellen, den Nachweis von pluripotenzbezogenen Biomarkern durch Immunfluoreszenz, In-vitro-Differenzierungsassays und Methoden zum Nachweis des Verlusts exogener Gene19.
Protokoll
FCV wurden vom Manipal Hospital, Bengaluru, unter der Genehmigung der Ethikkommission der Manipal Hospitals erhalten.
HINWEIS: Siehe Tabelle 1 für die Zusammensetzung aller Puffer und Lösungen.
1. Isolierung von Fibroblasten aus fetalen Chorionzotten (FCV)
-
Probenentnahme und Gewebedesintegration in kollagenase
- FCV unter sterilen Bedingungen in phosphatgepufferter Kochsalzlösung (PBS) sammeln und (bei Raumtemperatur) zur Zellkulturanlage transportieren.
- Die Zotten in eine 60 mm Petrischale geben und mehrmals (mindestens 4 mal) in PBS mit 1x Antibiotikum Antimykotika (PBS-AA) waschen. Entfernen Sie PBS-AA vollständig durch Pipettieren.
- Behandeln Sie die Chorionzotten mit 1 ml Kollagenasemischung (5 mg/ml) für 5 min bei 37 °C.
- Mit Zellkulturmedium, das 10% fötales Rinderserum (FBS) enthält, neutralisieren, in ein 15-ml-Röhrchen überführen und 5 Minuten lang bei 225 x g zentrifugieren, um die zerfallenen Zotten und freigesetzten Zellen als Pellet zu sammeln.
-
Subkultur und Bestandserweiterung
- Plattenzersinkte Zotten zusammen mit freigesetzten Zellen in einem T25-Kulturkolben, der 5 ml vollständige Medien (z. B. AmnioMAX) enthält, und wachsen, bis eine konfluente Fibroblastenkultur erhalten wird.
- Erweitern Sie Fibroblasten in Kultur, um die Bestände für die Verwendung in nachfolgenden Transfektions- und Charakterisierungsexperimenten wie folgt vorzubereiten:
- 2 ml 0,05% Trypsin in den T25-Kolben mit FCV-Fibroblasten geben und bei 37 °C für 3-5 min inkubieren, um die Zellen zu dissoziieren.
- Neutralisieren Sie Trypsin nach der Inkubation durch Zugabe von FBS (im gleichen Volumen wie Trypsin).
HINWEIS: Kulturmedium, das FBS enthält, kann auch verwendet werden, um Trypsin zu neutralisieren, wenn es in einem Trypsin:Medien-Verhältnis von 1:3 hinzugefügt wird. - Sammeln Sie die dissoziierten Zellen in einem 15-ml-Röhrchen und zentrifugieren Sie bei 225 x g für 5 min, um Zellpellets zu erhalten.
- Dekantieren Sie Überstand und resuspend Zellpellet in 1 mL komplettem Medium.
- Übertragen Sie je 500 μL auf 60 mm gewebekulturbehandelte Schalen und machen Sie das Volumen auf 5 mL auf. Dieses Spaltverhältnis von 1:2 wurde auch für nachfolgende Passagen verwendet.
-
Kryokonservierung
- Enzymatische Dissoziation unter Verwendung von 0,05% Trypsin wie in den Schritten 1.2.2.1 - 1.2.2.3 beschrieben durchführen und Zellpellet erhalten.
- Verwerfen Sie den Überstand und resuspenieren Sie das Zellpellet in 1 ml Gefriermischung, umfassend 1:9 Dimethylsulfoxid (DMSO): FBS.
- Geben Sie den Inhalt in ein steriles Kryovial und legen Sie die Durchstechflasche in einen Gefrierbehälter.
- Über Nacht bei -80 °C einfrieren und die Durchstechflaschen am nächsten Tag auf flüssigen Stickstoff (-196 °C) umfüllen.
2. DNA-Isolierung und -Verifizierung von Plasmiden
-
Bakterielle Zellvorbereitung
- Streakglycerinvorräte von E. coli enthalten die drei einzelnen Plasmide pCXLE-hOCT3/4-shp53-F, pCXLE-hSK und pCXLE-hUL (von Addgene) auf separaten Luria Bertani-Ampicillin-Agarplatten.
- Impfen Sie einzelne Kolonien in Starterkulturen von 5 ml Luria Bertani-Ampicillinmedium. 8 Stunden bei 37 °C unter Schütteln (10 x g) inkubieren.
- Impfen Sie 200 μL dieser Starterkultur in 100 ml Luria Bertani-Ampicillin-Medium. Über Nacht bei 37 °C unter Schütteln inkubieren.
- Ernte über Nacht Bakterienkultur durch Zentrifugieren bei 6000 x g für 15 min bei 4 °C.
-
Plasmidisolierung mit Midi-Plasmid-Reinigungskit
- Resuspend Bakterienpellet in 4 ml Resuspensionspuffer.
- Fügen Sie 4 ml Lysepuffer hinzu und mischen Sie gründlich, indem Sie 4-6 mal kräftig invertieren und bei Raumtemperatur für 5 min inkubieren.
- Fügen Sie 4 ml vorgekühlten Neutralisationspuffer hinzu und invertieren Sie das Röhrchen 4-6 Mal, um es gründlich zu mischen. 15 Min. auf Eis inkubieren.
- Zentrifuge bei ≥20.000 x g für 30 min bei 4 °C. Überstand in einem frischen Röhrchen auffangen und bei ≥20.000 x g für 15 min bei 4 °C erneut zentrifugieren.
- Richten Sie die Säule aus, indem Sie 4 ml Äquilibrierungspuffer auftragen.
- Wenden Sie den Überstand auf die Spalte an.
- Waschen Sie die Säule zweimal mit 10 ml Waschpuffer.
- Elute DNA mit 5 ml warmem (65 °C) Elutionspuffer.
- Niederschlag der DNA durch Zugabe von 3,5 ml Isopropanol zur eluierten DNA. Gut mischen. Zentrifuge bei ≥15.000 x g für 30 min bei 4 °C. Überstand vorsichtig dekantieren.
- Waschen Sie das DNA-Pellet mit 2 ml 70% Ethanol und zentrifugieren Sie es bei ≥15.000 x g für 10 min. Überstand vorsichtig dekantieren.
- Pellet für 5-10 min an der Luft trocknen und DNA in einem geeigneten Volumen von PCR-Wasser lösen, um eine Endkonzentration von 1 μg/ml zu erhalten.
HINWEIS: Lösen Sie DNA nicht in Puffern auf, da sie nicht für die Elektroporation geeignet ist. Alte Plasmid-DNA-Präparate ergeben keine reprogrammierten Kolonien.
-
Plasmidverifikation durch EcoRI Restriktionsaufschluss
- Kombinieren Sie 15 μL nukleasefreies Wasser, 2 μL 10x Puffer, 1 μg Plasmid-DNA und 1 μL EcoRI-Enzym. Vorsichtig mischen.
- Die Mischung bei 37 °C für 15 min in einem Hitzeblock inkubieren.
- Mischen Sie die verdauten Plasmidproben mit 6x DNA-Gelbeladungsfarbstoff und Elektrophorrese auf 1% Agarosegel in 1x TAE-Puffer mit 0,5 μg/ml Ethidiumbromid. Fügen Sie eine Standard-DNA-Leiter hinzu. Stellen Sie sich das Gel vor, nachdem sich die DNA entsprechend aufgelöst hat. Erwartete EcoRI-Bandgrößen von pCXLE-hOCT3 / 4-shp53-F sind 6.834 bp, 3.758 bp und 1.108 bp; pCXLE-hUL sind 10.200 bp und 1.900 bp; pCXLE-hSK sind 10.200 bp und 2.500 bp.
3. Nukleofektion
-
Zellpelletierung
- Kultur der isolierten fetalen Chorionzotten Fibroblasten in T25-Kolben in 5 ml vollständigem Medium bis zu 80-90% Konfluenz.
- Zellen zweimal mit PBS waschen und trypsinisieren, wie in den Schritten 1.2.2.1 - 1.2.2.3 beschrieben.
- Entfernen Sie den Überstand, resuspend Pellet in 5 ml reduziertem Serummedium (z. B. Opti-MEM). Zählen Sie Zellen mit Hämozytometer und nehmen Sie 106 Zellen für die Nukleofektion. Zentrifuge bei 225 x g für 5 min. Überstand vollständig entfernen.
-
Reagenzienpräparation und Nukleofektion
- Herstellung des Nukleofektorreagenzes durch Mischen von 0,5 ml Ergänzung und 2,25 ml Nukleofectorlösung (beide im Kit enthalten).
- Fügen Sie 100 μL Nukleofectorlösung in einem 1,5 ml Röhrchen hinzu. Fügen Sie jeweils 1μg pCXLE-hOCT3/4-shp53-F, pCXLE-hSK und pCXLE-hUL in das Röhrchen hinzu. Resuspendieren Sie vorsichtig 106 Zellen (aus Schritt 3.1.2) in dieser Mischung.
- Übertragen Sie die Zell-DNA-Suspension in die Küvette und stellen Sie sicher, dass die Probe den Boden der Küvette (im Kit enthalten) ohne Luftblasen bedeckt. Die Küvette verschließen und in die Halterung einsetzen. Wählen Sie das Nukleofector-Programm U-23 (für hohe Effizienz) und bewerben Sie sich.
- Entfernen Sie die Küvette aus der Halterung und fügen Sie 1 ml komplettes Medium hinzu. Den Inhalt schonend in eine mit 60 mm Gewebekultur behandelte Petrischale geben, die mit 4 mL kompletten Medien (auf insgesamt 5 mL Medien) gefüllt ist. Inkubieren Sie die Zellen ineinem befeuchteten CO2-Inkubator bei 37 °C.
- Überprüfen Sie nach 24 h, ob sich die Zellen angeschlossen haben. Ersetzen Sie das Medium vollständig.
HINWEIS: Die Zelltodrate ist in der Nukleofektion hoch und hinterlässt nur wenige lebensfähige Zellen, die sich anheften. - Halten Sie die Zellen für 10 Tage in einem vollständigen Medium und wechseln Sie für die nächsten 20 Tage zu Pluripotenzmedien.
HINWEIS: Visualisieren Sie Zellen regelmäßig, um morphologische Veränderungen in den reprogrammierenden Zellen (wie Epithelmorphologie und kompakte Koloniebildung) zu verfolgen, um zu bestätigen, ob das Experiment funktioniert. Rund 25 iPSC-Kolonien sind nach 20 Tagen Kultur in Pluripotenzmedien zu sehen.
4. Pflücken und Vermehren von iPSC-Kolonien
-
Pflücken der Kolonie aus der Reprogrammierplatte
- Sezieren Sie die embryonalen stammzellähnlichen Kolonien, die in der Umprogrammierschale gebildet wurden, manuell mit gezogenen Glaspipetten oder 1 ml Spritzennadeln und übertragen Sie sie mit inaktivierten embryonalen Maus-Fibroblasten-Feedern mit Pluripotenzmedium auf eine zuvor vorbereitete Platte oder legen Sie feederfreie Kulturen auf Matrigel-beschichteten Platten mit mTESR-Medium fest.
ANMERKUNG: Embryonale Fibroblasten (MEFs) der Maus wurden durch enzymatische Isolierung aus Mausembryonen (seziert von 13-14 Tagen schwangeren weiblichen Mäusen) gewonnen und durch Mitomycin C-Behandlung mitotisch inaktiviert. Etablieren Sie einzelne Klonpopulationen, indem Sie einzelne Kolonien aus der Umprogrammierung von Tellern in separaten Schalen oder gemischten Klonpopulationen züchten, indem Sie viele Kolonien von der Reprogrammierungsplatte auf eine einzige Schüssel übertragen.
- Sezieren Sie die embryonalen stammzellähnlichen Kolonien, die in der Umprogrammierschale gebildet wurden, manuell mit gezogenen Glaspipetten oder 1 ml Spritzennadeln und übertragen Sie sie mit inaktivierten embryonalen Maus-Fibroblasten-Feedern mit Pluripotenzmedium auf eine zuvor vorbereitete Platte oder legen Sie feederfreie Kulturen auf Matrigel-beschichteten Platten mit mTESR-Medium fest.
-
Mechanische Übertragung von entstehenden Kolonien auf frische Feeder und Passaging zur Etablierung stabiler iPS-Zellen
- Vermehren Sie iPS-Zellen in Pluripotenzmedium, indem Sie jeden zweiten Tag füttern und alle 5-7 Tage 1: 3 teilen. Bereiten Sie die Bestände vor, indem Sie in einer Gefriermischung aus KnockOut Serum Replacement und DMSO im Verhältnis 9: 1 kryovorservieren.
HINWEIS: KnockOut Serum Replacement wird in der Gefriermischung zur Kryokonservierung von iPS-Zellen anstelle von FBS verwendet, da Komponenten im FBS während der Langzeitkonservierung eine Differenzierung der pluripotenten Zellen induzieren könnten.
- Vermehren Sie iPS-Zellen in Pluripotenzmedium, indem Sie jeden zweiten Tag füttern und alle 5-7 Tage 1: 3 teilen. Bereiten Sie die Bestände vor, indem Sie in einer Gefriermischung aus KnockOut Serum Replacement und DMSO im Verhältnis 9: 1 kryovorservieren.
5. Charakterisierung von iPS-Zellen
HINWEIS: Charakterisierungsstudien einschließlich PCR und Immunfärbung für Pluripotenz-Biomarker wurden nach der fünften Passagenzahl durchgeführt. Die Karyotypisierung wurde bei einer späteren Passage durchgeführt.
- Karyotypisierung
- Behandeln Sie eine konfluente 60 mm Petrischale aus iPS-Zellen mit Colcemid für 45 min im befeuchteten CO2 -Inkubator bei 37 °C.
- Ernte durch 0,05% Trypsin-Behandlung und Zentrifuge. Entfernen Sie den Überstand und pipettieren Sie die übrig gebliebenen Mediumspuren, um das Zellpellet zu lösen.
- Fügen Sie 5 ml hypotonische Lösung hinzu. Mit invertierendem Röhrchen mischen und 20 Minuten bei 37 °C inkubieren. Zentrifuge bei 225 x g für 5 min.
HINWEIS: Das erhaltene Pellet sollte flauschig erscheinen. - Fügen Sie 2,5 ml Carnoys Fixierlösung langsam hinzu, während Sie klopfen, um das Pellet zu lösen.
- Bereiten Sie die Aufstriche für die Karyotypisierung vor, indem Sie die Zellsuspension auf saubere Glasobjektträger fallen lassen.
- Behandeln Sie die Objektträger 1 Minute lang mit 0,15% Trypsin und waschen Sie sie einmal mit PBS. Dann mit Giemsa-Lösung für 4 min färben und mit einer destillierten Wasserwäsche beenden. Akquise und Verarbeitung mit entsprechender Software.
- Demonstration des transgenen freien Status
-
Genomische DNA-Isolierung
- 20 μL Protease in den Boden eines 1,5 ml Mikrozentrifugenröhrchens pipettieren.
- Geben Sie 200 μL TSiPS-Zellen, die in PBS resuspendiert sind, in das Mikrozentrifugenröhrchen.
- 200 μL Buffer AL in die Probe geben und für 15 s durch Pulswirbeln mischen.
- 10 min bei 56 °C inkubieren.
- Zentrifugieren Sie kurz das Mikrozentrifugenröhrchen, um Tropfen von der Innenseite des Deckels zu entfernen.
- 200 μL Ethanol (96-100%) in die Probe geben und erneut für 15 s durch Pulswirbeln mischen. Nach dem Mischen das Röhrchen kurz zentrifugieren, um Tropfen von der Innenseite des Deckels zu entfernen.
- Tragen Sie die Mischung aus dem vorherigen Schritt vorsichtig auf die Mini-Spin-Säule (in einem 2-ml-Auffangrohr) auf, ohne den Rand zu benetzen. Schließen Sie die Kappe und zentrifugieren Sie bei 6000 x g für 1 min. Entsorgen Sie das Röhrchen, das das Filtrat enthält, und legen Sie die Mini-Spin-Säule in ein sauberes 2 ml Auffangrohr.
- Öffnen Sie vorsichtig die Mini-Spin-Säule und fügen Sie ohne Benetzung der Felge 500 μL Buffer AW1 hinzu. Verschließen Sie die Kappe und zentrifugieren Sie sie bei 6000 x g für 1 min. Legen Sie die Mini-Spin-Säule in ein sauberes 2 ml Auffangrohr und entsorgen Sie das Auffangrohr, das das Filtrat enthält.
- Öffnen Sie vorsichtig die Mini-Spin-Säule und fügen Sie 500 μL Buffer AW2 hinzu, ohne die Felge zu benetzen. Verschließen Sie die Kappe und die Zentrifuge bei voller Geschwindigkeit (20.000 x g) für 3 min.
- Legen Sie die Mini-Spin-Säule in ein neues 2-ml-Auffangrohr und entsorgen Sie das alte Auffangrohr mit dem Filtrat. Zentrifugieren Sie mit voller Geschwindigkeit für 1 min, um die Möglichkeit eines möglichen Buffer AW2-Übertrags zu eliminieren.
- Legen Sie die Mini-Spin-Säule in ein sauberes 1,5 ml Mikrozentrifugenröhrchen und entsorgen Sie das Auffangröhrchen, das das Filtrat enthält. Öffnen Sie vorsichtig die Mini-Spin-Säule und fügen Sie 200 μL Buffer AE oder destilliertes Wasser hinzu. Bei Raumtemperatur (15-25 °C) für 5 min inkubieren und dann bei 6000 x g für 1 min zentrifugieren.
-
Transgenfreie Status-PCR (mit KAPA HiFi PCR Kit KR0368)
- Stellen Sie sicher, dass alle Reagenzien ordnungsgemäß aufgetaut und gemischt sind.
- Bereiten Sie eine PCR-Mastermischung vor, die das entsprechende Volumen aller Reaktionskomponenten enthält, basierend auf Tabelle 2 (Reaktionen auf Eis einrichten).
- Übertragen Sie die entsprechenden Volumina von PCR-Master-Mix, Template und Primer auf einzelne PCR-Röhrchen.
- Einzelne Reaktionen abschließen, mischen und kurz zentrifugieren.
- Führen Sie eine PCR gemäß Tabelle 3 durch.
-
Genomische DNA-Isolierung
- Identifizierung von Pluripotenz-Biomarkern
-
Färbung der alkalischen Phosphatase (AP)
- Bereiten Sie eine 1x AP-Live-Färbe-Arbeitslösung vor, indem Sie 3 μL 500x Stammlösung in 1,5 mL DMEM/F-12 pro 10cm2 Kulturfläche verdünnen.
- Entfernen Sie das Medium aus der iPSC-Kulturschale. Waschen Sie die Kultur einmal mit DMEM/F-12. Geben Sie die 1x AP-Lebendflecklösung auf die iPSCs. Bei 37 °C für 45 min inkubieren.
- Entfernen Sie den AP-Fleck und waschen Sie ihn zweimal mit DMEM/F-12. Fügen Sie frisches DMEM / F-12 hinzu und nehmen Sie es unter dem Fluoreszenzmikroskop mit einem Standard-FITC-Filter innerhalb von 30-90 Minuten nach der Färbung auf.
-
Immunostaining für Pluripotenz-Biomarker
- Konfluente iPSC-Kulturen mit 4% Paraformaldehyd über Nacht bei 4 °C fixieren. Dreimal mit PBS Tween 20 (PBST) waschen, jeweils 5 min waschen.
- Permeabilisieren Sie die Zellen mit 0,3% Triton X-100 in PBST für 15 Minuten bei Raumtemperatur. Dreimal mit PBST waschen.
HINWEIS: Die Permeabilisierung sollte nur für intrazelluläre Antigene durchgeführt werden. - Blockzellen mit 3% Rinderserumalbumin (BSA) in PBST für 30 min bei Raumtemperatur. Färben Sie die Zellen über Nacht mit primären Antikörpern (verdünnt 1:1000 in 1% BSA). Nach der primären Antikörperinkubation dreimal mit PBST waschen.
- Inkubieren Sie Zellen mit dem sekundären Antikörper (verdünnt 1:1000 in 1% BSA) für 5 h bei Raumtemperatur. Dreimal mit PBST waschen.
- Markieren Sie die Kerne mit 0,5 μg/ml 4',6-Diamidino-2-phenylindol (DAPI) für 1 Minute. Waschen Sie die Zellen einmal mit PBST.
- Erfassen Sie Bilder unter dem Fluoreszenzmikroskop.
-
Färbung der alkalischen Phosphatase (AP)
6.In-vitro-Differenzierungstests
- Embryoide Körper (EB) Differenzierung
- Die iPSC-Kolonien in kleine Stücke schneiden, sammeln und in Petrischalen mit niedrigem Aufsatz im embryoiden Körpermedium aufzeichnen. Züchten Sie die Zellen für 15 Tage, indem Sie das Medium alle 3 Tage ersetzen.
HINWEIS: Die Tag-15-EBs können direkt für den Nachweis der drei Keimschicht-Biomarker verwendet werden. Alternativ können spezifische Zelllinien mit Wachstumsfaktoren induziert werden, gefolgt von einem Biomarker-Nachweis.
- Die iPSC-Kolonien in kleine Stücke schneiden, sammeln und in Petrischalen mit niedrigem Aufsatz im embryoiden Körpermedium aufzeichnen. Züchten Sie die Zellen für 15 Tage, indem Sie das Medium alle 3 Tage ersetzen.
- Endoderm (Hepatozyten) Differenzierung
- Züchten Sie die iPS-Zellen in Monolayer-Kulturen in Pluripotenzmedium.
- Nach dem Konfluent wechseln Sie für 2 Tage zu RPMI 1640-Medien mit 1x Insulin Transferrin Selenite und 100 ng / ml Aktivin A, gefolgt von einem Wachstum der RPMI 1640-Medien mit 30 ng / ml bFGF und 20 ng / ml BMP4 für 9 Tage. Ersetzen Sie medium alle 2 Tage.
- Ab Tag 10 ergänzen Medien mit 0,1 μM Dexamethason. Beenden Sie das Experiment am 20. Tag.
- Mesoderm (Kardiomyozyten) Differenzierung
- Platte Tag 8 EBs auf 0,5% Matrigel-beschichteten Platten in embryoidem Körpermedium. Lassen Sie die EBs anfügen und reduzieren.
- Ergänzen Sie Medien mit 20 ng / ml BMP4 und wachsen Sie für 20 Tage. Ersetzen Sie das Medium alle 2-3 Tage. Beenden Sie das Experiment am 20. Tag.
- Ektoderm (neuronale) Differenzierung
- Platte Tag 4 EBs auf 2 μg/cm2 Kollagen Typ IV-beschichteten Platten in embryoidem Körpermedium. Lassen Sie die EBs anfügen und reduzieren.
- Wechseln Sie am nächsten Tag zu DMEM F-12 mit 2 mg / ml Glukose, 1x Insulin Transferrin Selenit und 2,5 μg / ml Fibronektin. Beenden Sie den Test an Tag 15.
- Bildung von zerebralen Organoiden
- Züchten Sie TSiPS-Zellen in einer 35-mm-Gewebekulturschale auf MEFs bis zu 70-80% konfluent. Schneiden Sie die Kolonien ab und sammeln Sie sie in einer 15 ml Röhre. Zentrifugieren Sie die Zellen bei 225 x g für 5 min. Verwerfen Sie den Überstand.
- Waschen Sie die Koloniestücke, indem Sie sie in 2 ml PBS wiederverwenden und zentrifugieren, um den Überstand zu entfernen.
- Fügen Sie 1 ml 0,05% Trypsin hinzu und klopfen Sie auf die Röhre, um die Zellen zu entfernen. Inkubieren Sie das Röhrchen bei 37 °C für 3-4 min, um die Koloniestücke in eine einzelne Zellsuspension zu dissoziieren.
- Neutralisieren Sie das Trypsin durch Verdünnung mit 4 ml Pluripotenzmedien, die 10 μg/ml Rho-assoziierte Proteinkinase (ROCK) -Inhibitor Y-27632 Dihydrochlorid (ROCKi) enthalten, um den dissoziationsinduzierten Zelltod zu verhindern.
- Zentrifuge, um ein Pellet zu erhalten. Verwerfen Sie den Überstand und resuspendieren Sie die Zellen in 2 ml embryoidem Körpermedium, das 10 μg / ml ROCKi enthält.
- Entfernen Sie 10 μL Zellsuspension für die Zellzählung. Fügen Sie 10 μL Trypanblau hinzu, um abgestorbene Zellen zu erkennen. Zählen Sie die Zellen mit einem Hämozytometer.
- Fügen Sie der Zellsuspension ein geeignetes Volumen des embryoiden Körpermediums mit ROCKi hinzu, um 9.000 lebende Zellen pro 150 μL zu erhalten.
- Platte 150 μL in jeder Vertiefung einer Low-Attachment 96-Well-Platte und inkubieren in einem befeuchteten CO2-Inkubator bei 37 °C. Überprüfen Sie die Platten nach 24 Stunden auf Aggregation. Am Tag 2 das Medium vorsichtig entfernen und durch frisches embryoides Medium ohne ROCKi ersetzen.
- Übertragen Sie am Tag 6 EBs auf Vertiefungen mit einer niedrigen Attachment 24 Well Plate, die 500 μL neuronales Induktionsmedium enthält, bestehend aus DMEM-F12 mit 1% N2-Ergänzung, 2 mM GlutaMAX-Ergänzung und 1 mM nicht-essentiellen Aminosäuren und 1 μg / ml Heparin. Wechseln Sie das Medium alle 2 Tage.
- Nach 5 Tagen in neuronales Induktionsmedium die neuroepithelialen Aggregate in Matrigel einbetten, indem ein 2 cm x 2 cm großes Quadrat Parafilm über eine leere Spitzenschale mit 200 μL-Spitzen geschichtet wird. Drücken Sie parafilm mit behandschuhten Fingern über jedes Loch in der Spitzenschale, um kleine Dellen zu machen. Reinigen Sie parafilm mit 70% Ethanol zum Sterilisieren.
- Übertragen Sie das Parafilmquadrat in eine 60-mm-Schale. Verwenden Sie geschnittene 200 μL-Spitzen, um die neuroepithelialen Aggregate auf die Dellen im Parafilm zu übertragen. Entfernen Sie überschüssiges Medium durch Pipettieren.
- 30 μL aufgetautes Matrigel auf die neuroepithlialen Aggregate geben und das Aggregat mit einer Pipettenspitze in der Mitte des Matrigels positionieren. Stellen Sie die 60-mm-Schale für 20-30 min in einen 37 °C-Inkubator, damit das Matrigel polymerisieren kann.
- Fügen Sie 5 ml zerebrales Organoid-Differenzierungsmedium hinzu, bestehend aus 1:1 DMEM-F12: Neurobasales Medium, 0,5% N2-Präparat, 2,5 μg/ml Insulin, 2 mM GlutaMAX-Präparat, 0,5 mM NEAA, 1% B27-Präparat und 2,5 ml Penicillin-Streptomycin.
- Drehen Sie mit einer sterilen Pinzette die Parafilmfolie um und rühren Sie die Schale, bis die in Matrigel eingebetteten Aggregate von der Folie in das Medium fallen. Züchten Sie die eingebetteten Aggregate in einem befeuchteten CO2 -Inkubator bei 37 ° C für 4 Tage und geben Sie an wechselnden Tagen Medienwechsel.
- Nach 4 Tagen statischer Kultur legen Sie die 60-mm-Schalen auf einen orbitalen Shaker, der im Inkubator installiert ist und mit 50 U / min schüttelt. Kultivieren Sie die Organoide für 3 Monate und geben Sie alle 3 Tage vollständige Medienveränderungen mit zerebralem Organoid-Differenzierungsmedium.
Ergebnisse
Generierung von integrationsfreien iPS-Zellen aus einem spontan abgetriebenen Fötus mit 45XO-Karyotyp
Wir isolierten Fibroblasten aus FCV mit einem Turner-Syndrom (TS)-spezifischen 45XO-Karyotyp und nukleofizierten sie mit episomalen Reprogrammierungsplasmiden, um TSiPS-Zellen zu erzeugen, die für die nachgeschaltete Modellierung des Syndroms, insbesondere der damit verbundenen neurologischen Defizite, verwendet werden können (Abbildung 1a& b). Für die Transfektionsexperimente verwendeten wir nicht-integrierende episomale Vektoren und Nukleofektion (Abbildung 1 c&d). Wir verfolgten morphologische Veränderungen von Zellen, um den Erfolg der Reprogrammierung zu überwachen. Die Verschiebung vom Fibroblasten zur Epithelmorphologie, gefolgt von einer abgegrenzten kompakten Koloniebildung, wurde beobachtet (Abbildung 2a). TSiPS-Zellen erfassten eine humane embryonale Stammzell-ähnliche Morphologie mit unterschiedlichen Kanten und einem hohen Zellkern-zytoplasma-Verhältnis um den Tag 20 nach der Transfektion(Abbildung 2b). Im Gegensatz dazu erwerben unvollständig reprogrammierte Zellen epitheliale Morphologien, bilden aber keine kompakten Kolonien. (Abbildung 2c).
Charakterisierung von TSiPS-Zellen
Die Karyotypisierung von TSiPS-Zellen zeigte den 45XO-Karyotyp, der mit dem Turner-Syndrom assoziiert ist (Abbildung 3a). Die Immunfluoreszenz von TSiPS-Zellen zeigte die Expression der Pluripotenz-Transkriptionsfaktoren OCT4, NANOG, SOX2 und der Zelloberflächenmarker SSEA4, E-Cadherin und TRA-1-81. Humane embryonale Stammzellen sind der Goldstandard pluripotenter Stammzellen. Wir führten gleichzeitig die Immunfluoreszenz von HUES 1 durch, die als Positivkontrolle für den Vergleich der Pluripotenz-Biomarkerexpression durch TSiPSC verwendet wurde (Abbildung 3b). Der transgene freie Status der TSiPS-Zellen wurde durch eine genomische DNA-PCR für die episomalen Plasmidmarker OriP und EBNA nachgewiesen. In Passage 15 gingen das OriP- und EBNA-Gen in den TSiPS-Zellen verloren.Die episomalen Gene OriP und EBNA wurden amplifiziert und zeigten in Passage 9 TSiPS-Zellen Banden, die auf das Vorhandensein der episomalen Plasmide in diesem Stadium hinweisen. Diese Gene wurden jedoch in Passage 15 TSiPS-Zellen nicht amplifiziert, was auf einen Verlust der episomalen Plasmide und damit auf einen transgenen freien Zustand hinweist (Abbildung 3c).
In-vitro-Differenzierungstests
Das Differenzierungspotenzial von TSiPSC-Linien wurde in vitronachgewiesen. TSiPS-Zellen bildeten bei Aggregation in platten mit niedriger Anhaftung embryoide Körper (Abbildung 4a). Wachstumsfaktorinduzierte Differenzierung von TSiPS-Zellen wurde verwendet, um Zelltypen der drei Keimschichten zu erzeugen. Die Immunfluoreszenzanalyse unter Verwendung linienspezifischer Biomarker bestätigte, dass TSiPS-Zellen in repräsentative Derivate von Endoderm (SOX17), Mesoderm (MYOSIN VENTRICULAR HEAVY CHAINα/β) und Ektoderm (βIII TUBULIN) differenzierten (Abbildung 4b).
Zerebrale Organoid-Differenzierung.
TSiPS-Zellen wurden als zerebrale Organoide stufenweise unterschieden. Einzelzellsuspensionen von TSiPS-Zellen wurden in embryoide Körper aggregiert, um die Entwicklung von Keimschichten für die ersten 6 Tage zu stimulieren, gefolgt von der Induktion der neuroepithelialen Entwicklung für 5 Tage. Die neuroepithelial aggregierten waren sie in Matrigel eingebettet, das die extrazelluläre Matrix und die Basalmembrankomponenten lieferte, die die richtige apikobasale Orientierung, das Auswachsen neuroepithelialer Knospen, die sich ausdehnen und Lumen bilden, unterstützen. Die Immunfluoreszenz mit dem neuroepithelialen Marker NESTIN wurde durchgeführt, um die Gesamtmorphologie der Organoide zu beobachten (Abbildung 5b). Das Neuroepithel umgibt eine ventrikelartige Höhle(Abbildung 5c - weiße Linie). Die Organoide zeigen morphologisch ventrikuläre Zonen (VZ), subventrikuläre Zonen (SVZ) und kortexartige Regionen(Abbildung 5c - rote, orange bzw. gelbe Linien)

Abbildung 1: Isolierung und Reprogrammierung von Fibroblasten durch Nukleofektion. (a) Mikroskopische Aufnahme von fetalen Chorionzotten vor der Kollagenasebehandlung. b)Fibroblasten, die aus fetalen Chorionzotten für Reprogrammierungsexperimente isoliert wurden. c) Nachweis von Reprogrammierungsplasmiden durch EcoRI-Restriktionsaufschluss. ( d) Schematische Darstellung des Transfektionsprotokolls, das für die iPSC-Erzeugung aus fetalen Chorionzottenfibroblasten unter Verwendung episomaler Reprogrammierungsplasmide durch Nukleofektion verwendet wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
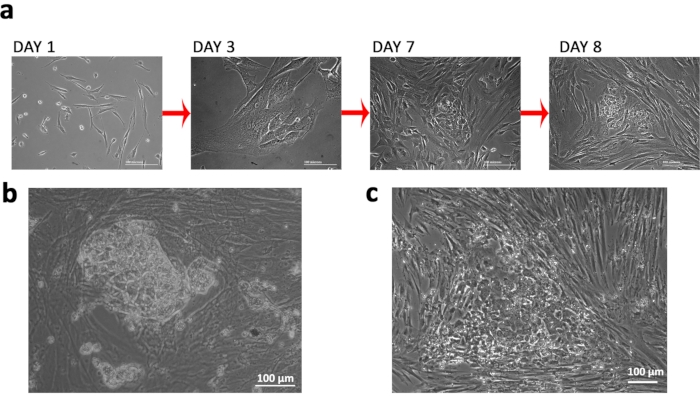
Abbildung 2: Etablierung von Turner-Syndrom-induzierten pluripotenten Stammzellen. (a) Veränderungen der Zellmorphologie, die während des Zeitablaufs der Reprogrammierung beobachtet werden. b)Eine vollständig umprogrammierte TSiPSC-Kolonie. (c) Ein repräsentatives Bild einer Kolonie mit unsachgemäß umprogrammierten Zellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
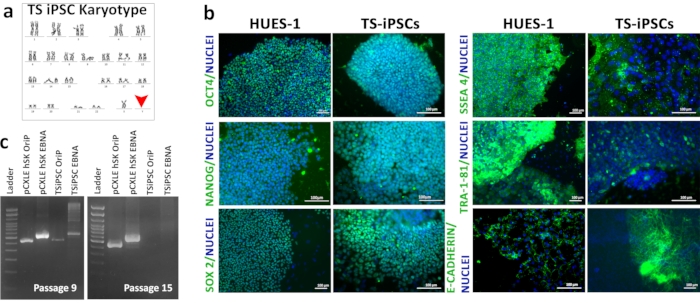
Abbildung 3: Charakterisierung von TSiPS-Zellen. a)Karyotyp von TSiPS-Zellen. b)Immunfluoreszenzanalyse der Pluripotenz-Biomarker OCT4, NANOG, SOX2, SSEA-4 und TRA-1-81 in TSiPS-Zellen im Vergleich zu embryonalen Stammzellen HUES1. Kerne werden mit 4', 6-Diamidino-2-phenylindol angefärbt. 3c. Nachweis des transgenfreien Status von TSiPS-Zellen. Lane 1- DNA Ladder, Lane 2- OriP Positivkontrolle mit pCXLE-hSK, Lane 3- EBNA Positivkontrolle mit pCXLE-hSK, Lane 4-OriP mit TSiPSCs, Lane 5-EBNA mit TSiPSCs. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
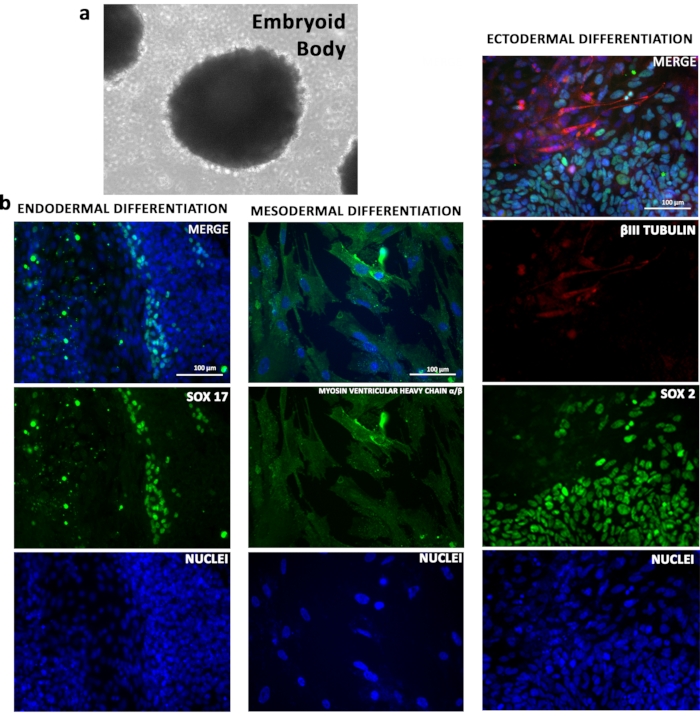
Abbildung 4: In-vitro-Differenzierungspotential von TSiPS-Zellen. ( a) TSiPSC differenziert zu embryoiden Körpern. b)Immunfluoreszenzanalysen von TSiPS-Zellen für den endodermalen Marker SOX17, den mesodermalen Marker Myosin ventrikuläre schwere Kette α/β und die ektodermalen Marker βIII Tubulin und SOX2. Kerne werden mit 4', 6-Diamidino-2-phenylindol angefärbt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
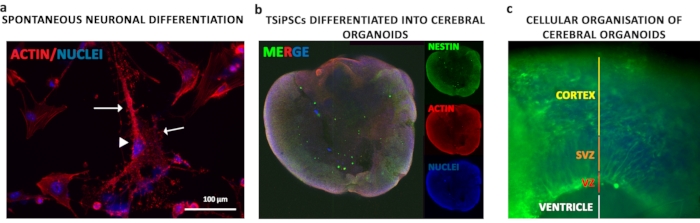
Abbildung 5: Neuronale und zerebrale Organoiddifferenzierung von TSiPS-Zellen. (a) Um die Zytoarchitektur differenzierter Neuronen zu verstehen, wurde eine Phalloidinfärbung von Aktin durchgeführt. TSiPSC-abgeleitete Neuronen zeigten pyramidenförmige neuronale Soma (Pfeilspitze) mit Dendriten und Axonen (Pfeile). Kerne werden mit 4', 6-Diamidino-2-phenylindol angefärbt. Immunfärbung. (a) Immunfärbung für Nestin und Aktin zur Beobachtung der groben Morphologie der Organoide. (c) Färbung für Nestin zur Visualisierung der apikal und basal organisierten neuronalen Schichten. Kerne werden mit 4', 6-Diamidino-2-phenylindol angefärbt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Zusammensetzung von Medien, Puffern und Lösungen Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: PCR Reaction Mix Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: PCR Cycling Program Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die Erzeugung stabiler zellulärer Modelle zytogenetisch abnormalen fetalen Gewebes ist notwendig, um einen defekten Phänotyp aufrechtzuerhalten. Der iPSC-Weg ist die effektivste Methode der Zellvorbereitung zur ständigen Erhaltung defekter Eigenschaften20.
Pluripotente Stammzellen (PSC) zeigen Eigenschaften der Selbsterneuerung und Differenzierung in spezialisierte Zellen, die an frühe Spaltungsembryonen erinnern21. Daher können PSCs als hervorragende Modelle dienen, um frühe molekulare, zelluläre und Entwicklungsdefekte bei vorzeitig abgetriebenen Föten zu untersuchen.
In diesem Artikel haben wir die menschliche iPSC-Erzeugung mittels Nukleofektion in Kombination mit den verbesserten episomalen Vektoren beschrieben. Die Ergebnisse zeigen, dass diese Kombination eine robuste Methode zur Erzeugung integrationsfreier menschlicher iPSC-Linien darstellt, was durch die Tatsache belegt wird, dass einzelne Transfektionen für eine erfolgreiche Reprogrammierung ausreichten. Wir haben die fortschreitende Umwandlung von FCV-Fibroblasten in pluripotente Zellen mikroskopisch verfolgt. 20 Tage nach der Transfektion beobachteten wir Kolonien von reprogrammierten TSiPS-Zellen, die von nicht reprogrammierten FCV-Fibroblasten umgeben waren. Morphologisch ähnelten die abgeleiteten humanen iPS-Zellen embryonalen Stammzellen, die im Labor gezüchtet wurden. Typischerweise aggregierten die Zellen als kompakte Kolonien mit glänzenden Rändern. Die Zellen der Kolonien hatten große Kerne und dicht gepackt, was auf einen engen Membrankontakt zwischen den Zellen hindeutet. Die nicht reprogrammierten Fibroblasten wölbten und umgaben diese Kolonien. Nach der Übertragung auf iMEFs vermehren sie sich in der Kultur für über 30 Transfers, die die Eigenschaft der fortgesetzten Selbsterneuerung demonstrieren.
Da TSiPS-Zellen aus 45XO-Fibroblasten erzeugt wurden, karyotypisierten wir die Zellen, um zu überprüfen, ob sie die Chromosomenzusammensetzung beibehielten. Die TSiPS-Zellen behielten den 45XO-Karyotyp in kontinuierlicher Zellkultur bei, was auf ein stabiles 45XO-Chromosomen-Erbgut hindeutet. Um als zelluläre Ressource, die die 45XO-Aneuploidie darstellt, nützlich zu sein, sollten die TSiPS-Zellen frei von exogener DNA sein, die in den Reprogrammierungsexperimenten verwendet wird. Wir überprüften das Vorhandensein von episomalen Restplasmiden, indem wir eine genomische DNA-PCR für episomale spezifische Marker -OriP und EBNA durchführten. Wir fanden keine Spur dieser Marker in TSiPS-Zellen nach 15 Passagen, was darauf hindeutet, dass die TSiPS-Zellen bei längerer Kultur schrittweise episomale Reprogrammierungsvektoren verloren.
Das Kennzeichen einer pluripotenten Zelle ist ihr Potenzial, sich sowohl in vitro als auch in vivo zu Zellen mit drei Keimlinien zu differenzieren. Um diese Fähigkeit in den abgeleiteten TSiPS-Zellen zu testen, haben wir sie in vitro embryoiden Körperbildungs- und Differenzierungsassays unterzogen, die nach Abstammung geleitet werden und Zytokine und Wachstumsfaktoren spezifizieren. TSiPS-Zellen bildeten embryoide Körper und differenzierten sich in ektodermale Zellen, die neuronale Marker exprimierten, mesodermale Zellen, die Herzmarker exprimierten, und endodermale Zellen, die SOX17, einen Biomarker für das Schicksal von Endodermen, exprimierten. Wir testeten auch die Fähigkeit von TSiPS-Zellen, sich in 3D-Zerebralorganoide höherer Ordnung zu differenzieren, indem wir zuvor etablierte Protokolle22verwendeten. TSiPS-Zellen organisieren sich aufgrund ihrer eigenen intrinsischen Entwicklungsprogramme allmählich selbst in Minigewebe, die Organoide genannt werden. TSiPS-Zellen ergaben zerebrale Organoide, die eine Zytoarchitektur ähnlich dem Hirngewebe mit Neuroepithel zeigten, das eine ventrikelartige Höhle umgibt. Diese Organoide müssen jedoch weiter umfassend charakterisiert werden, um die genauen Zelltypen aufzudecken, und mit normalen iPS-Zellen verglichen werden, um die intrinsischen neuronalen Gewebemustereigenschaften von TSiPS-Zellen zu unterscheiden. Diese zerebralen Organoide und andere Arten von Gehirnorganoiden, die aus TSiPS-Zellen erzeugt werden, können verwendet werden, um Entwicklungs- und Funktionsinkonsistenzen zu modellieren, die zu den Symptomen neurologischer Defizite von TS-Personen beitragen können. TSiPS-Zellen wiesen Biomarkereigenschaften der Pluripotenz sowie das charakteristische Merkmal der Differenzierung auf und hoben damit den Erfolg der Reprogrammierung auf induzierte Pluripotenz hervor.
Die oben beschriebene Methode hat effizient bei der Reprogrammierung von dermalen Fibroblasten und mesenchymalen Zellen aus verschiedenen Quellen in unserem Labor gearbeitet (Daten anderer Linien nicht gezeigt). Nach unserer Erfahrung sind die folgenden Schritte entscheidend für den Erfolg des Reprogrammierungsexperiments:
a) Qualität der Plasmidpräparation: Alte Präparate ergeben keine iPS-Zellen.
b) Qualität der für Transfektionen verwendeten Zellen: Proliferierende Zellen sind essentiell für die iPSC-Erzeugung. 0,5 bis 1 Million Zellen pro Transfektion ergaben eine reproduzierbare Reprogrammierungseffizienz.
c) Frisch rekonstituierte Nukleofektorreagenzien: Rekonstituierte Nukleofektorreagenzien, die über einen Monat gelagert wurden, ergaben keine iPS-Zellen.
d) Die Pflege der Master-Zellbank durch mechanische Subkultur der iPS-Zellen ergab stabile Linien. Die enzymatische Dissoziation wurde gemäß den Anforderungen des Experiments verwendet.
Das zukünftige Ziel des Labors ist es, ein Panel von chromosomal abnormalen iPS-Zellen für die nachgelagerte Entwicklung, Funktions- und Krankheitsmodellierung mit dieser effizienten Methode zu etablieren. Fetale Aneuploidien verursachen Schwangerschaftsverlust und Organfehlbildungen bei Lebendgeburten. Aneuploide iPS-Zellen, die aus Geweben spontaner Abortus gewonnen werden, sind eine wertvolle Ressource, um fehlgeschlagene embryonale Entwicklungsereignisse zu modellieren und zu untersuchen. In-vitro-2D- und 3D-Kultursysteme, einschließlich embryoider Körper und gewebespezifischer Organoide22, werden es den Forschern ermöglichen, molekulare und zelluläre Unregelmäßigkeiten wie aberrante Zellproliferation und Zelltod in linienspezifischen Zellen zu verstehen, die sich als Entwicklungsanomalien und Schwangerschaftsversagen im Zusammenhang mit Aneuploidie-Syndromen manifestieren könnten.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Finanzielle Unterstützung für die oben genannte Forschung wurde von der Manipal Academy of Higher Education bereitgestellt. Die Charakterisierung der Linie wurde teilweise im Labor von M.M. Panicker am NCBS durchgeführt. Wir danken Anand Diagnostic Laboratory für die Unterstützung bei der Karyotypisierung.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.15% trypsin | Thermo Fisher Scientific | 27250018 | G Banding |
| 2-mercaptoethanol | Thermo Fisher Scientific | 21985023 | Pluripotency and Embryoid body medium |
| 4', 6 diamidino-2-phenylindole | Sigma Aldrich | D8417 | Immunocytochemistry |
| Activin A | Sigma Aldrich | SRP3003 | Differentiation assays |
| Alkaline Phosphatase Live Stain | Thermo Fisher Scientific | A14353 | AP staining |
| AMAXA Nucleofector II | Lonza | - | Nucleofection |
| AmnioMAX II complete media | Thermo Fisher Scientific, Gibco | 11269016 | Medium specific for foetal chorionic villi cell cultures |
| Ampicillin | HiMedia | TC021 | Plasmid purification |
| Anti Mouse IgG (H+L) Alexa Fluor 488 | Invitrogen | A11059 | Immunocytochemistry |
| Anti Rabbit IgG (H+L) Alexa Fluor 488 | Invitrogen | A11034 | Immunocytochemistry |
| Anti Rabbit IgG (H+L) Alexa Fluor 546 | Invitrogen | A11035 | Immunocytochemistry |
| Antibiotic-Antimycotic | Thermo Fisher Scientific, Gibco | 15240096 | Contamination control |
| Anti-E-Cadherin | BD Biosciences | 610181 | Immunocytochemistry |
| Anti-Nanog | BD Biosciences | 560109 | Immunocytochemistry |
| Anti-OCT3/4 | BD Biosciences | 611202 | Immunocytochemistry |
| Anti-SOX17 | BD Biosciences | 561590 | Immunocytochemistry |
| Anti-SOX2 | BD Biosciences | 561469 | Immunocytochemistry |
| Anti-SSEA4 | BD Biosciences | 560073 | Immunocytochemistry |
| Anti-TRA 1-81 | Millipore | MAB4381 | Immunocytochemistry |
| basic Fibroblast Growth Factor[FGF2] | Sigma Aldrich | F0291 | Pluripotency medium |
| Bone Morphogenetic Factor 4 | Sigma Aldrich | SRP3016 | Differentiation assays |
| Bovine Serum Albumin | Sigma Aldrich | A3059 | Blocking |
| Collagen Human Type IV | BD Biosciences | 354245 | Differentiation assays |
| Collagenase blend | Sigma Aldrich | C8051 | Digestion of foetal chorionic villi |
| Dexamethasone | Sigma Aldrich | D4902 | Differentiation assays |
| DMEM F12 | Thermo Fisher Scientific | 11320033 | Differentiation assays |
| FastDigest EcoR1 | Thermo Scientific | FD0274 | Restriction digestion |
| Fibronectin | Sigma Aldrich | F2518 | Differentiation assays |
| Giemsa Stain | HiMedia | S011 | G Banding |
| Glacial Acetic Acid | HiMedia | AS001 | Fixative for karyotyping |
| Glucose | Sigma Aldrich | G7528 | Differentiation assays |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | Pluripotency and Embryoid body medium |
| Heparin sodium | Sigma Aldrich | H3149 | Differentiation assays |
| Insulin solution human | Sigma Aldrich | I9278 | Differentiation assays |
| Insulin Transferrin Selenite | Sigma Aldrich | I1884 | Differentiation assays |
| KAPA HiFi PCR kit | Kapa Biosystems | KR0368 | OriP, EBNA1 PCR |
| KaryoMAX Colcemid | Thermo Fisher Scientific | 15210040 | Mitotic arrest for karyotyping |
| KnockOut DMEM | Thermo Fisher Scientific | 10829018 | Pluripotency and Embryoid body medium |
| KnockOut Serum Replacement | Thermo Fisher Scientific | 10828028 | Pluripotency and Embryoid body medium |
| Luria Bertani agar | HiMedia | M1151F | Plasmid purification |
| Matrigel | BD Biosciences | 356234 | Differentiation assays |
| MEM Non-essential amino acids | Thermo Fisher Scientific | 11140035 | Pluripotency and Embryoid body medium |
| Methanol | HiMedia | MB113 | Fixative for karyotyping |
| Myosin ventricular heavy chain α/β | Millipore | MAB1552 | Immunocytochemistry |
| NHDF Nucleofector Kit | Lonza | VAPD-1001 | Nucleofection |
| Paraformaldehyde (PFA) | Sigma Aldrich | P6148 | Fixing cells |
| pCXLE-hOCT3/ 4-shp53-F | Addgene | 27077 | Episomal reprogramming Plasmid |
| pCXLE-hSK | Addgene | 27078 | Episomal reprogramming Plasmid |
| pCXLE-hUL | Addgene | 27080 | Episomal reprogramming Plasmid |
| Penicillin Streptomycin | Thermo Fisher Scientific, | 15070063 | Pluripotency and Embryoid body medium |
| Phalloidin- Tetramethylrhodamine B isothiocyanate | Sigma Aldrich | P1951 | Immunocytochemistry |
| Phosphate buffered saline | Sigma Aldrich | P4417 | 1 X PBS 1 tablet of PBS dissolved in 200mL of deionized water and sterilized by autoclaving Storage: Room temperature. PBST- 0.05% Tween 20 in 1X PBS. Storage: Room temperature. |
| Plasmid purification Kit- Midi prep | QIAGEN | 12143 | Plasmid purification |
| Potassium Chloride Solution | HiMedia | MB043 | Hypotonic solution for karyotyping |
| QIAamp DNA Blood Kit | Qiagen | 51104 | Genomic DNA isolation |
| RPMI 1640 | Thermo Fisher Scientific | 11875093 | Hepatocyte differentiation medium |
| Sodium Citrate | HiMedia | RM255 | Hypotonic solution for karyotyping |
| Triton X-100 | HiMedia | MB031 | Permeabilisation |
| Trypsin-EDTA (0.05%) | Thermo Fisher Scientific, Gibco | 25300054 | Subculture of foetal chorionic villi fibroblasts |
| Tween 20 | HiMedia | MB067 | Preparation of PBST |
| β III tubulin | Sigma Aldrich | T8578 | Immunocytochemistry |
| Y-27632 dihydrochloride | Sigma Aldrich | Y0503 | Differentiation assays |
Referenzen
- Verlinsky, Y., et al. Human embryonic stem cell lines with genetic disorders. Reproductive BioMedicine Online. 10, 105-110 (2005).
- Eiges, R., et al. Developmental Study of Fragile X Syndrome Using Human Embryonic Stem Cells Derived from Preimplantation Genetically Diagnosed Embryos. Cell Stem Cell. 1, 568-577 (2007).
- Biancotti, J. -. C., et al. Human Embryonic Stem Cells as Models for Aneuploid Chromosomal Syndromes. STEM CELLS. 28, 1530-1540 (2010).
- Li, W., et al. Modeling abnormal early development with induced pluripotent stem cells from aneuploid syndromes. Human Molecular Genetics. 21, 32-45 (2012).
- Gravholt, C. H., Viuff, M. H., Brun, S., Stochholm, K., Andersen, N. H. Turner syndrome: mechanisms and management. Nature Reviews Endocrinology. 15, 601-614 (2019).
- Luo, Y., et al. Uniparental disomy of the entire X chromosome in Turner syndrome patient-specific induced pluripotent stem cells. Cell Discovery. 1, 15022 (2015).
- Luo, Y., et al. Generation of an induced pluripotent stem cell line from an adult male with 45,X/46,XY mosaicism. Stem Cell Research. 27, 42-45 (2018).
- Parveen, S., Panicker, M. M., Gupta, P. K. Generation of an induced pluripotent stem cell line from chorionic villi of a Turner syndrome spontaneous abortion. Stem Cell Research. 19, (2017).
- Takahashi, K., et al. Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors. Cell. 131, 861-872 (2007).
- Soldner, F., et al. Parkinson's Disease Patient-Derived Induced Pluripotent Stem Cells Free of Viral Reprogramming Factors. Cell. 136, 964-977 (2009).
- Somers, A., et al. Generation of Transgene-Free Lung Disease-Specific Human Induced Pluripotent Stem Cells Using a Single Excisable Lentiviral Stem Cell Cassette. STEM CELLS. 28, 1728-1740 (2010).
- Woltjen, K., et al. piggyBac transposition reprograms fibroblasts to induced pluripotent stem cells. Nature. 458, 766-770 (2009).
- Stadtfeld, M., Nagaya, M., Utikal, J., Weir, G., Hochedlinger, K. Induced Pluripotent Stem Cells Generated Without Viral Integration. Science. 322, 945-949 (2008).
- Fusaki, N., Ban, H., Nishiyama, A., Saeki, K., Hasegawa, M. Efficient induction of transgene-free human pluripotent stem cells using a vector based on Sendai virus, an RNA virus that does not integrate into the host genome. Proceedings of the Japan Academy, Series B. 85, 348-362 (2009).
- Warren, L., et al. Highly Efficient Reprogramming to Pluripotency and Directed Differentiation of Human Cells with Synthetic Modified mRNA. Cell Stem Cell. 7, 618-630 (2010).
- Kim, D., et al. Generation of human induced pluripotent stem cells by direct delivery of reprogramming proteins. Cell Stem Cell. 4, 472-476 (2009).
- Yu, J., et al. Human Induced Pluripotent Stem Cells Free of Vector and Transgene Sequences. Science. 324, 797-801 (2009).
- Okita, K., et al. A more efficient method to generate integration-free human iPS cells. Nature Methods. 8, 409-412 (2011).
- Martí, M., et al. Characterization of pluripotent stem cells. Nature Protocols. 8, 223-253 (2013).
- Avior, Y., Sagi, I., Benvenisty, N. Pluripotent stem cells in disease modelling and drug discovery. Nature Reviews Molecular Cell Biology. 17, 170-182 (2016).
- Ambartsumyan, G., Clark, A. T. Aneuploidy and early human embryo development. Human Molecular Genetics. 17, 10-15 (2008).
- Lancaster, M. A., Knoblich, J. A. Generation of cerebral organoids from human pluripotent stem cells. Nature Protocols. 9, 2329-2340 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten