Method Article
Analyse du contenu des gouttelettes lipidiques dans les levures de fission et les levures en herbe à l'aide du traitement automatisé de l'image
Dans cet article
Résumé
Ici, nous présentons une implémentation de MATLAB de la détection automatisée et de la description quantitative des gouttelettes de lipide dans les images de microscopie de fluorescence des cellules de fission et de levure en herbe.
Résumé
Le métabolisme des lipides et sa régulation sont d'intérêt pour les sciences de la vie de base et appliquées et la biotechnologie. À cet égard, diverses espèces de levures sont utilisées comme modèles dans la recherche métabolique lipidique ou pour la production industrielle de lipides. Les gouttelettes lipidiques sont des corps de stockage très dynamiques et leur contenu cellulaire représente une lecture pratique de l'état métabolique des lipides. La microscopie fluorescence est une méthode de choix pour l'analyse quantitative des gouttelettes lipidiques cellulaires, car elle repose sur un équipement largement disponible et permet l'analyse des gouttelettes lipidiques individuelles. En outre, l'analyse microscopique de l'image peut être automatisée, ce qui augmente considérablement le débit global de l'analyse. Ici, nous décrivons un flux de travail expérimental et analytique pour la détection automatisée et la description quantitative des gouttelettes lipidiques individuelles dans trois espèces différentes de levure modèle : les levures de fission Schizosaccharomyces pombe et Schizosaccharomyces japonicus, et la levure en herbe Saccharomyces cerevisiae. Les gouttelettes lipidiques sont visualisées avec BODIPY 493/503, et le dextran fluorescent à l'imperméabilité cellulaire est ajouté au support culturel pour aider à identifier les limites cellulaires. Les cellules sont soumises à une microscopie d'épifluorescence 3D dans des canaux verts et bleus et les images z-stack qui en résultent sont traitées automatiquement par un pipeline MATLAB. La procédure produit de riches données quantitatives sur la teneur en gouttelettes lipidiques cellulaires et les caractéristiques individuelles des gouttelettes lipidiques dans un format tabulaire adapté aux analyses en aval dans les principaux paquets de tabletur ou statistiques. Nous fournissons des analyses d'exemple de la teneur en gouttelettes lipidiques dans diverses conditions qui affectent le métabolisme des lipides cellulaires.
Introduction
Les lipides jouent un rôle crucial dans l'énergie cellulaire et le métabolisme du carbone, la synthèse des composants membranaires et la production de substances bioactives. Le métabolisme des lipides est affiné en fonction des conditions environnementales, de la disponibilité des nutriments et de la phase1du cycle cellulaire. Chez l'homme, le métabolisme des lipides a été relié à des maladies telles que l'obésité, le diabète de type II et le cancer2. Dans l'industrie, les lipides produits par les micro-organismes, comme les levures, représentent une source prometteuse de carburants diesel renouvelables3. Les cellules stockent les lipides neutres dans ce qu'on appelle les gouttelettes lipidiques (LD). Ces corps conservés de façon évolutive sont composés de triacylglycérols, d'esters de stéroyles, d'une monocouche phospholipide externe et de protéines associées1. Les LD proviennent du réticulum endoplasmique, exercent une dynamique du cycle cellulaire ou de la phase de croissance, et sont importants pour l'homéostasie lipidique cellulaire1. Le nombre De LD et la morphologie peuvent être utilisés comme un proxy commode en assérant le métabolisme de lipide dans diverses conditions de croissance ou en examinant un panneau de mutants. Compte tenu de leur nature dynamique, les techniques capables d'analyser les propriétés des LD individuels sont d'un intérêt particulier dans les études du métabolisme des lipides.
Diverses espèces de levures ont été utilisées pour décrire les voies métaboliques liées aux lipides et leur régulation, ou utilisées en biotechnologie pour produire des composés ou des carburants intéressants1. En outre, pour les levures modèles, telles que la levure en herbe Saccharomyces cerevisiae ou la levure de fission lointaine ment liée Schizosaccharomyces pombe, bibliothèques de souches de suppression à l'échelle du génome sont disponibles qui peuvent être utilisés pour le haut débit écrans4,5. Récemment la composition et la dynamique de LD ont été décrites dans S. pombe6,7,8,9, et les mutants liés au métabolisme de lipide ont été isolés dans la levure modèle émergente Schizosaccharomyces japonicus10.
De nombreuses techniques sont disponibles pour étudier le contenu et la dynamique LD. La plupart emploient une sorte de coloration de LD avec des colorants lipophiles tels que nile Red ou BODIPY 493/503. Ce dernier montre des spectres d'excitation et d'émission plus étroits, et une plus grande spécificité vers les lipides neutres (LD) par opposition aux phospholipides (membranes)11. Les méthodes fluormétriques et de flux-cytométrie ont été utilisées avec succès dans diverses espèces fongiques pour découvrir des gènes et des conditions de croissance qui affectent la teneur en lipides de stockage12,13,14,15. Bien que ces méthodes conviennent aux applications à haut débit, elles ne peuvent pas mesurer le nombre et la morphologie des LD individuels dans les cellules, qui peuvent différer considérablement entre les conditions de croissance et les génotypes. La diffusion cohérente de Raman ou la microscopie holographique numérique sont des méthodes sans étiquette qui produisent des données de niveau LD, mais exigent l'équipement coûteux spécialisé16,17,18. La microscopie fluorescence, d'autre part, peut fournir des données détaillées sur le contenu LD, tout en utilisant des instruments couramment disponibles et des outils logiciels d'analyse d'images. Plusieurs workflows d'analyse existent qui comportent divers degrés de sophistication et d'automatisation dans la détection de cellules/LD à partir des données d'image, et sont optimisés pour différents types de cellules, tels que les cellules métazoaires avec de grands LD19,20 , 21, ou levures en herbe17,22,23. Certaines de ces approches ne fonctionnent qu'en 2D (p. ex., sur des images de projection maximale), ce qui peut ne pas décrire de façon fiable le contenu LD cellulaire. À notre connaissance, aucun outil n'existe pour déterminer le contenu et la morphologie de LD à partir des données microscopiques de levure de fission. Le développement d'analyses automatisées et robustes au niveau de la LD apporterait une sensibilité accrue et une puissance statistique accrue, et fournirait des informations riches sur la teneur en lipides neutres, idéalement chez plusieurs espèces de levures.
Nous avons développé un flux de travail pour l'analyse du contenu LD à partir d'images de microscopie de fluorescence 3D de cellules de levure. Les cellules vivantes sont tachées de BODIPY 493/503 et de Dextran Cascade Blue pour visualiser les LD et déterminer les limites cellulaires, respectivement. Les cellules sont immobilisées sur des lames de verre et soumises à l'imagerie par la pile z à l'aide d'un microscope à épifluorescence standard. Les images sont ensuite traitées par un pipeline automatisé mis en œuvre dans MATLAB, un paquet (commercial) largement utilisé pour les analyses statistiques. Le pipeline effectue le prétraitement de l'image, la segmentation (cellules par rapport à l'arrière-plan, l'élimination des cellules mortes) et l'identification LD. Les données riches au niveau LD, telles que la taille du LD et l'intensité de la fluorescence, sont ensuite fournies dans un format tabulaire compatible avec les principaux outils logiciels de feuilles de calcul. Le flux de travail a été utilisé avec succès pour déterminer l'impact de la disponibilité des sources d'azote sur le métabolisme des lipides dans S. pombe24. Nous démontrons maintenant la fonctionnalité du flux de travail dans S. pombe, S. japonicus et S. cerevisiae, en utilisant des conditions de croissance ou des mutants qui affectent le contenu lD cellulaire.
Protocole
1. Préparation de solutions et de médias
-
Préparer la solution de coloration lipidique.
- Pour préparer la solution de coloration lipidique de stock dissoudre 10 mg de BODIPY 493/503 dans 10 ml de DMSO anhydre (concentration finale 1 mg/mL). Dissoudre la teneur entière d'une flacon BODIPY 493/503 de 10 mg pour éviter la perte de matière pendant la pesée.
MISE EN GARDE: DMSO peut passer à travers la peau. Portez l'équipement de protection individuelle approprié. - Préparer la solution de coloration lipidique de travail en mélangeant 100 'L de la solution de 1 mg/mL BODIPY 493/503 stock et 900 'L de DMSO anhydre (concentration finale 0.1 mg/mL).
- Aliquot le stock et les solutions de travail, et stocker à -20 oC.
REMARQUE: Le BODIPY 493/503 dissous est stable depuis plusieurs années à -20 oC. Cependant, la solution doit être protégée de l'humidité et de la lumière.
- Pour préparer la solution de coloration lipidique de stock dissoudre 10 mg de BODIPY 493/503 dans 10 ml de DMSO anhydre (concentration finale 1 mg/mL). Dissoudre la teneur entière d'une flacon BODIPY 493/503 de 10 mg pour éviter la perte de matière pendant la pesée.
- Pour préparer la solution de stock pour la visualisation de limite cellulaire, dissoudre 25 mg de dextran bleu de cascade (flacon entier) dans 2,5 ml d'eau déionisée (concentration finale 10 mg/mL). Aliquot la solution de stock et le stocker à -20 oC protégé de la lumière.
- Pour préparer la solution de revêtement de glissement de microscope, dissoudre 5 mg de lectine de soja dans 5 ml d'eau déionisée (concentration finale 1 mg/ml). Aliquot la solution de lectine et stocker à -80 oC.
REMARQUE: La solution de lectine est stable depuis plusieurs années à -80 oC. Les Aliquots actuellement en service peuvent être stockés à -20 oC. -
Préparer les supports de culture.
- Pour préparer 400 ml de milieu de culture OUI complexe pour S. pombe et S. japonicus, dissoudre 2 g d'extrait de levure et 0,1 g de suppléments SP (si nécessaire pour les mutants auxotrophiques) dans 340 mL d'eau déionisée dans une bouteille de 500 ml et autoclave. Ajouter 60 ml de 20 % (w/v) de glucose séparément autoclave ou stérilisé par filtre dans des conditions aseptiques.
- Pour préparer 400 ml de milieu de culture EMM défini pour S. pombe et S. japonicus, dissoudre 4,9 g de bouillon EMM sans dextrose dans 360 mL d'eau déionisée dans une bouteille de 500 ml et autoclave. Ajouter 40 ml de 20 % (w/v) de glucose séparément autoclave ou stérilisé par filtre dans des conditions aseptiques.
REMARQUE: Pour les lignes directrices générales sur S. pombe et S. japonicus culture voir25 et26, respectivement. - Préparer 300 ml de milieu de culture Complexe de YPAD pour Saccharomyces cerevisiae,dissoudre 3 g d'extrait de levure, 6 g de peptone et 30 mg de sulfate d'adénine dans 270 mL d'eau déionisée dans une bouteille de 500 ml et autoclave. Ajouter 30 ml de 20 % (w/v) de glucose séparément autoclave ou stérilisé par filtre dans des conditions aseptiques.
- Pour préparer 300 ml de milieu minimal défini pour S. cerevisiae, dissoudre 2 g de base d'azote de levure (sans acides aminés) dans 270 mL d'eau déionisée dans une bouteille de 500 ml et autoclave. Ajouter 30 ml de 20 % (w/v) de glucose séparément autoclave ou stérilisé par filtre dans des conditions aseptiques.
REMARQUE: Pour les lignes directrices générales sur la culture S. cerevisiae voir27.
2. Cultivation cellulaire
-
Croissance de S. pombe ou S. japonicus à la phase exponentielle ou stationnaire précoce.
- Le matin, inoculer 5 ml de OUI moyen avec de la biomasse de levure de fission fraîche. Incuber à 32 oC en secouant (180 tr/min) pendant plusieurs heures.
REMARQUE: Pour toutes les cultures, utilisez des flacons Erlenmeyer ayant 10 fois le volume de culture pour assurer une bonne aération. Certains laboratoires préfèrent cultiver des levures de fission à 30 oC, mais la température de culture de 32 oC entraîne des temps de doublement plus courts sans effets néfastes sur les cellules, réduisant ainsi le temps total requis pour effectuer une expérience25,28 . - En fin d'après-midi du même jour (après au moins 6 heures de culture), diluer la culture avec un oui frais moyen à un volume de culture final de 10 ml afin qu'il atteigne la densité optique désirée (OD) (ou le nombre de cellules/mL) le lendemain matin, et couver à 32 oC avec s haking (180 tr/min). Il est avantageux de connaître le temps de doublement de chaque souche utilisée pour déterminer avec précision le facteur de dilution (utiliser l'équation 1).

Là où laculture Vest le volume de préculture nécessaire à la dilution, Vfinal est le volume total de la nouvelle culture (10 ml pour les cultures standard), oDfinale est l'OD souhaitée à atteindre le le lendemain matin, lecourant oDest l'OD actuellement mesuré de la préculture, t est le temps de la croissance cellulaire jusqu'à la récolte, tlag est la durée de la phase de décalage (dépend des conditions de laboratoire, doit être empiriquement définies) et tDT est le temps de doublement de la souche.
REMARQUE: Lorsque les cellules de phase exponentielle doivent être analysées, ne laissez pas les précultures atteindre la phase stationnaire, car cela modifie considérablement la physiologie cellulaire (y compris la teneur en LD) pour plusieurs générations suivantes. - Le matin du jour de l'imagerie, si la culture atteint une OD légèrement plus élevée que nécessaire (en cas de cellules de phase exponentielle), diluez-la avec un OUI frais et continuez l'incubation pendant au moins deux fois de plus avant de les colorer. Autrement, procéder directement à coloration (section 3).
- Le matin, inoculer 5 ml de OUI moyen avec de la biomasse de levure de fission fraîche. Incuber à 32 oC en secouant (180 tr/min) pendant plusieurs heures.
-
Croissance de S. cerevisiae à la phase exponentielle et stationnaire.
- Dans l'après-midi, inoculer 10 ml de YPAD moyen avec une petite quantité de biomasse de levure fraîche en herbe et incuber toute la nuit à 30 oC avec des secousses (180 tr/min).
- Le matin du jour d'imagerie, diluer la culture à OD 0.1 dans 10 mL de milieu YPAD et de croître à l'OD requis (par exemple, OD 1 pour la phase exponentielle). Effectuer toutes les dilutions de culture telles que décrites à l'étape 2.1.2. Procéder à la coloration (section 3).
3. Taches de gouttelettes de lipide
- Préparer un bordereau de couvercle de microscope pour chaque échantillon à être photographié. Étendre 1 ll de solution de revêtement coulissant sur un bordereau de couverture propre à l'aide du long côté d'une pointe de pipette positionnée horizontalement. Laissez sécher complètement la solution de revêtement et conservez les bordereaux de couverture dans un environnement sans poussière.
REMARQUE: Les glissières et les lames de verre peuvent être nettoyés avant d'être utilisés si nécessaire. La procédure de nettoyage consiste à laver avec du détergent à vaisselle, le rinçant à l'eau, le trempage de nuit dans l'acide chlorhydrique de 3%, et le lavage avec de l'eau distillée. Les glissières et les couvercles nettoyés sont stockés dans de l'éthanol pur jusqu'à leur utilisation. - Mesurer l'OD de la culture cellulaire ou le nombre de cellules/mL, au besoin. Essayez d'atteindre des valeurs similaires parmi toutes les souches testées pour assurer des conditions expérimentales comparables.
- Pipette 1 ml de chaque culture cellulaire à un tube microcentrifuge de 1,5 ml. Pour S. cerevisiae seulement, ajouter 5 l de la solution de revêtement de la diapositive, vortex brièvement, et incuber à 30 oC en secouant pendant 5 min.
- Ajouter brièvement 1 L de la solution de coloration lipidique à chaque culture aliquot et vortex. Ensuite, ajoutez brièvement 10 L de la solution de visualisation de la limite cellulaire et vortex.
REMARQUE: Ne préparez pas de solutions prémélangées des deux taches, car cela conduit à l'étanchéité de la fluorescence de BODIPY 493/503. - Recueillir les cellules par centrifugation (1 000 x g, 3 min, RT) et enlever presque tous les supernatants (950 l). Resuspendre les cellules du supernatant restant.
- Pipette 2 -L de la suspension cellulaire dense sur un couvercle enduit de lectine et placez-la sur une lame de microscope propre. Les cellules doivent former une monocouche. Procéder à la microscopie (section 4) le plus rapidement possible afin de minimiser les artefacts en imagerie; traiter un maximum de deux échantillons à la fois.
4. Mise en place du microscope et de l'imagerie
-
Optimiser les conditions d'imagerie.
REMARQUE : La mise en place du microscope nécessite de longues expositions à de fortes sources de lumière qui pourraient endommager l'échantillon et fausser les résultats. Par conséquent, configurez les conditions d'imagerie à l'aide d'une diapositive d'échantillon dédiée qui ne sera pas utilisée pour la quantification de LD.- Concentrez-vous sur les cellules à l'aide d'un contraste de phase ou d'un contraste d'interférence différentiel (DIC).
REMARQUE: Les images de contraste de phase ou de DIC peuvent être prises pour référence, mais elles ne sont pas utilisées pendant l'étape automatisée d'analyse d'image. - Définir les paramètres de la pile z pour couvrir l'ensemble du volume de la cellule. La distance verticale totale dépend de la taille de la cellule; le nombre de tranches optiques dépend de l'ouverture numérique de l'objectif (fonction de propagation de points dans l'axe Z). Définir la mise au point pour se déplacer par rapport au plan focal central.
REMARQUE: Le nombre optimal de tranches est souvent fixé par le logiciel de contrôle au microscope et n'a pas besoin d'être calculé manuellement. Les largeurs de cellules typiques sont de 3-5 m pour S. pombe,4-7 'm pour S. japonicus, et 3-7 'm pour S. cerevisiae. - Pour imager les LD, définir l'intensité lumineuse et le temps d'exposition dans le canal vert (les maxima d'excitation et d'émission de BODIPY sont respectivement de 493 et 503 nm).
REMARQUE: BODIPY 493/503 est un fluorochrome très lumineux; cependant, il peut obtenir blanchi rapidement avec l'intensité lumineuse excessivement forte. En outre, les LD sont mobiles dans les cellules vivantes, minimisant ainsi le temps d'exposition et capturent d'abord la pile z à canal vert (avant de passer au canal bleu) pour éviter de brouiller les artefacts. En outre, prendre en compte la portée linéaire de la caméra pour l'intensité du signal pour éviter les pixels saturés. - Pour imager les limites cellulaires, fixez l'intensité de la lumière et le temps d'exposition dans le canal bleu (les maxima d'excitation et d'émission de Cascade Blue dextran sont de 400 et 420 nm, respectivement).
REMARQUE: L'intensité du signal dans le canal bleu est nécessaire pour la segmentation de l'image, mais elle n'est pas utilisée pour la quantification LD elle-même. Par conséquent, les paramètres optimaux dans ce canal ne sont pas cruciaux pour l'analyse. - Si possible, créez un flux de travail expérimental automatisé dans le logiciel de contrôle au microscope pour faciliter l'imagerie de plusieurs échantillons dans des conditions normalisées.
- Concentrez-vous sur les cellules à l'aide d'un contraste de phase ou d'un contraste d'interférence différentiel (DIC).
- Une fois que les conditions d'imagerie ont été optimisées, des échantillons d'images doivent être utilisés pour la quantification. Concentrez-vous sur les cellules et imagez-les en canaux verts et bleus tels que décrits à l'étape 4.1.
REMARQUE: Toutes les images doivent être acquises en utilisant les mêmes paramètres pour permettre la comparaison entre les échantillons. Imagez plusieurs champs de vue par échantillon pour obtenir des données fiables et représentatives. - Enregistrez les images de z-stack de canaux bleus et verts sous forme de fichiers TIFF multicouches 16 bits (c.-à-d. deux fichiers par champ de vision). Inclure les mots « vert » ou « bleu » dans les noms de fichiers correspondants. Procéder à l'analyse d'image (section 5).
5. Analyse d'image
-
Vérifier visuellement la qualité des images acquises.
- Ouvrez des images microscopiques dans ImageJ29,30 ou tout autre logiciel d'analyse d'image approprié.
- Supprimer toutes les piles d'images contenant un nombre considérable de cellules qui se sont déplacées lors de l'acquisition (et ont ainsi créé des artefacts flous).
- Enlever toutes les piles d'images contenant des particules non cellulaires très fluorescentes dans le canal bleu (p. ex., saleté sur la glissière ou le glissement de couverture au microscope, impuretés dans le milieu de culture).
REMARQUE: Les objets non cellulaires très brillants dans le canal bleu peuvent créer des artefacts de détection cellulaire ou interférer avec la détection des cellules dans leur voisinage. - Enlevez toutes les piles d'images contenant une grande proportion de cellules mortes (c.-à-d. cellules avec fluorescence bleue accrue par rapport aux cellules vivantes).
REMARQUE: Bien que la présence d'une petite proportion de cellules mortes dans l'échantillon ne soit généralement pas un problème et que ces cellules soient automatiquement rejetées au cours de l'analyse, certaines cellules mortes ou mourantes peuvent parfois être reconnues comme cellules vivantes par l'algorithme de segmentation et biaiser les résultats rapportés.
-
Analysez les images dans le logiciel MATLAB.
- Créez un dossier principal et copiez tous les scripts MATLAB à cet emplacement.
- Créez un sous-dossier ("pombe", "cerevisiae" ou "japonicus") et copiez les fichiers d'images TIFF à cet endroit.
- Démarrer MATLAB, ouvrir le script MAIN.m et l'exécuter. Dans le menu, sélectionnez les espèces de levures à analyser et commencez le traitement de l'image.
REMARQUE: Certains des paramètres requis pour la détection cellulaire et LD sont pré-réglés pour l'espèce particulière, d'autres sont déterminés automatiquement pendant le traitement de l'image. Les valeurs préétablies ont été déterminées empiriquement et dépendent de plusieurs facteurs tels que le grossissement objectif, le type et la sensibilité de la caméra, et les paramètres d'imagerie. Si nécessaire, les utilisateurs peuvent modifier les fichiers de script pour modifier les préréglages spécifiques à l'organisme afin de mieux refléter leur configuration expérimentale. À savoir, lors de la reconnaissance cellulaire, les tailles d'objets acceptables sont données par les paramètres "minArea" et "maxArea", et la fraction minimale du volume rempli dans les limites de l'objet est donnée par le paramètre "Solidity". Pour la reconnaissance LD, le seuil de luminosité est donné par le paramètre "th" (sa valeur est principalement affectée par la profondeur du bit d'image et l'intensité du signal de fluorescence), et la taille maximale acceptable LD est donnée par le paramètre "MaxArea". - Inspecter et traiter les fichiers de sortie au besoin à l'aide d'un éditeur de feuilles de calcul ou d'un paquet statistique; le flux de travail produit des fichiers CSV séparés par les points-virgules et segmenté les fichiers TIFF avec des objets cellulaires et des LD détectés.
REMARQUE: Le flux de travail segmente les images en arrière-plan et en objets cellulaires, où chaque objet cellulaire peut être composé de plusieurs cellules adjacentes. Par conséquent, la sortie dans les fichiers "xxxx-cells.csv" ne représente pas les données unicellulaires et ne doit être utilisée que pour calculer les métriques par unité de volume de cellule.
Résultats
L'ensemble de la procédure est résumé e à la figure 1 pour les levures de fission (le flux de travail de levure en herbe est analogue), et ci-dessous nous fournissons des exemples de la façon dont le flux de travail peut être utilisé pour étudier la teneur en LD dans trois espèces de levures différentes dans diverses conditions connues d'affecter le contenu LD cellulaire. Chaque exemple représente une seule expérience biologique.
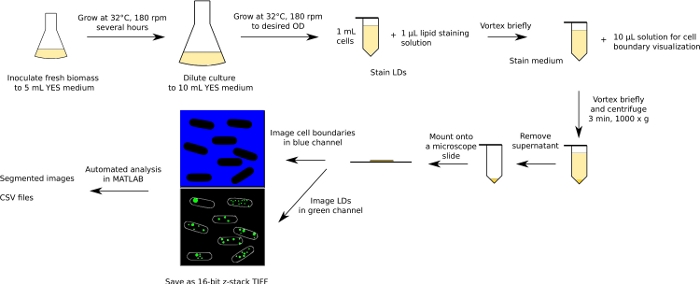
Figure 1 : Diagramme schématique du flux de travail expérimental et analytique. Le flux de travail pour les levures de fission est montré à titre d'exemple. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Tout d'abord, nous avons analysé les cellules de S. pombe (Figure 2). Type sauvage (WT; h (en) (en) les cellules ont été développées à la phase exponentielle dans le milieu complexe de OUI ou le milieu défini d'EMM. Par rapport à OUI, moins de LD et une intensité de coloration LD plus élevée par unité de volume cellulaire ont été détectées dans eMM (Figure 2A-C). De plus, les LD individuels formés dans le milieu EMM étaient plus grands et montraient une intensité de coloration totale accrue (Figure 2D, E). Ceci est en accord avec les résultats précédents de l'augmentation de la teneur en lipides de stockage dans les cellules cultivées dans EMM24. Le gène ppc1 code une ligase de phosphopantothenate-cystéine exigée pour la synthèse de coenzyme A. Le mutant ppc1-88 sensible à la température montre une diminution marquée de la teneur en LD lorsqu'il est cultivé à la température restrictive31, fournissant un exemple de cellules avec un faible signal BODIPY 493/503 (Figure 2A). Par conséquent, par rapport au type sauvage (cultivé à 32 oC), des LD plus petits ayant une intensité totale de coloration plus faible ont été détectés dans des cellules ppc1-88 cultivées en OUI à la suite d'un passage à 36 oC (figure2D,E), sans aucun changement apparent du nombre de LD par unité de volume cellulaire (Figure 2B).
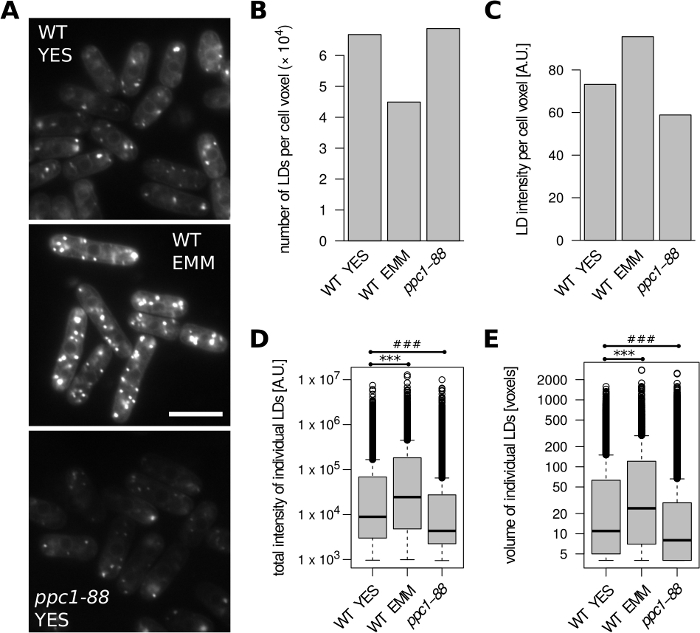
Figure 2 : Impact de la mutation des médias de croissance et du métabolisme lipidique sur le contenu lD dans S. pombe. Les cellules de type sauvage (WT) et de ppc1-88 ont été cultivées à une phase exponentielle dans le système complexe OUI ou le milieu EMM défini, comme indiqué. Les cellules WT ont été cultivées à 32 oC. Les cellules ppc1-88 sensibles à la température ont été cultivées à 25 oC et sont passées à 36 oC pendant 2 heures avant l'analyse. (A) Représentation d'images microscopiques non traitées de LD tachés de BODIPY 493/503. Une seule tranche optique est indiquée pour chaque condition; 10% de la pareille avec canal bleu inversé a été ajouté pour mieux visualiser les limites cellulaires. Barre d'échelle de 10 m . (B) Nombre de LD identifiés par unité de volume cellulaire. (C) Intensité de fluorescence des LD identifiés par unité de volume cellulaire. (D) Distributions des intensités totales de fluorescence de tous les LD identifiés. ' , '' ' ' ' ' ' '' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' (E) Distributions des volumes de tous les LD identifiés. ' ' ' ' '' ' ' ' '' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' Les données des panneaux B-E ont été tirées de 242, 124 et 191 objets cellulaires pour les échantillons WT YES, WT EMM et ppc1-88, respectivement. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Ensuite, nous avons quantifié la teneur en LD dans les cellules De S. japonicus (h-matsj-2017)32 à partir de cultures exponentielles et précoces cultivées en OUI (Figure 3A). Les cellules entrant dans la phase stationnaire ont montré un nombre nettement diminué de LDpar unité de volume cellulaire par rapport aux cellules en croissance exponentielle (Figure 3B), tandis que l'intensité de fluorescence lD normalisée en volume a légèrement diminué entre les deux conditions ( Figure 3C). Les LD de phase stationnaire précoce étaient généralement modérément plus grandes en taille et avaient une intensité de fluorescence totale modérément plus élevée que les LD provenant de cellules en croissance exponentielle (figure3D, E).

Figure 3 : Contenu LD dans Les cellules S. japonicus changent avec la phase de croissance. Des cellules de croissance exponentielle (LOG) et de phase stationnaire précoce (STAT) ont été analysées. (A) Représentation d'images microscopiques non traitées de LD tachés de BODIPY 493/503. Une seule tranche optique est indiquée pour chaque condition; 10% de la pareille avec canal bleu inversé a été ajouté pour mieux visualiser les limites cellulaires. La barre d'échelle représente 10 m. (B) Nombre de LD identifiés par unité de volume cellulaire. (C) Intensité de fluorescence des LD identifiés par unité de volume cellulaire. (D) Distributions des intensités totales de fluorescence de tous les LD identifiés. '' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' (E) Distributions des volumes de tous les LD identifiés. ' test Wilcoxon non apparié p ' 2,4 x 10-85. Les données des panneaux B-E ont été tirées de 274 et 187 objets cellulaires pour les échantillons LOG et STAT, respectivement. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Enfin, nous avons analysé les cellules De S. cerevisiae de la souche de laboratoire BY4741 largement utilisée (MATahis3-1 leu20 met15-0 ura3-0)cultivée en phase exponentielle et stationnaire, respectivement, dans le milieu complexe yPAD. Les cellules de levure en herbe accumulent typiquement des lipides de stockage à l'entrée dans la phasestationnaire1, et nous avons pu récapituler ces résultats (figure 4). Les cellules stationnaires contenaient un peu moins de LD par unité de volume que les cellules en croissance exponentielle (figure4B),mais leur intensité de fluorescence lD normalisée en volume a presque doublé (Figure 4C). Cette forte augmentation de la teneur globale en LD est attribuable à l'intensité et au volume beaucoup plus élevés de fluorescence des LD individuels en phase stationnaire (Figure 4D, E).
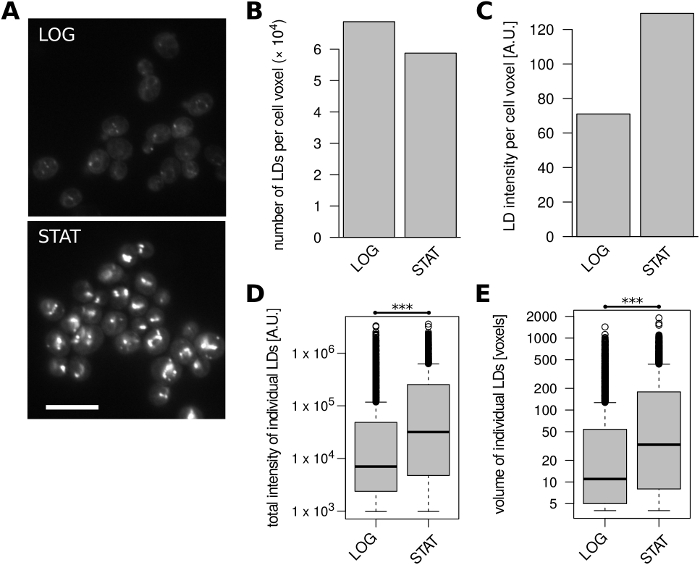
Figure 4 : Contenu LD dans Les cellules S. cerevisiae changent avec la phase de croissance. Des cellules de croissance exponentielle (LOG) et de phase stationnaire (STAT) ont été analysées. (A) Représentation d'images microscopiques non traitées de LD tachés de BODIPY 493/503. Une seule tranche optique est indiquée pour chaque condition; 10% de la pareille avec canal bleu inversé a été ajouté pour mieux visualiser les limites cellulaires. La barre d'échelle représente 10 m. (B) Nombre de LD identifiés par unité de volume cellulaire. (C) Intensité de fluorescence des LD identifiés par unité de volume cellulaire. (D) Distributions des intensités totales de fluorescence de tous les LD identifiés. '' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' (E) Distributions des volumes de tous les LD identifiés. ' test Wilcoxon non apparié p ' 3,7 x 10-63. Les données des panneaux B-E ont été dérivées de 430 et 441 objets cellulaires pour les échantillons LOG et STAT, respectivement. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Ainsi, notre workflow d'analyse peut détecter des changements dans le nombre de LD, la taille et la teneur en lipides dans trois espèces différentes et morphologiquement distinctes de levure dans diverses conditions qui affectent positivement ou négativement la teneur en LD cellulaire.
Discussion
La compréhension du métabolisme des lipides et de sa régulation est importante à la fois pour la biologie de base et pour les applications cliniques et biotechnologiques. Le contenu de LD représente une lecture commode de l'état de métabolisme de lipide de la cellule, avec la microscopie de fluorescence étant l'une des méthodes principales employées pour la détermination de contenu de LD. Le protocole présenté permet la détection automatisée et la description quantitative des LD individuels chez trois espèces de levures différentes et morphologiquement distinctes. À notre connaissance, il n'existe pas d'outils similaires pour les levures de fission. Les scripts MATLAB requis pour le traitement d'image sont inclus dans les fichiers supplémentaires, et sont également disponibles dans le référentiel Figshare (DOI 10.6084/m9.figshare.7745738) ainsi que toutes les images brutes et traitées et les données tabulaires de ce manuscrit, descriptions détaillées des fichiers de sortie CSV et des scripts R pour l'analyse et la visualisation des données en aval. En outre, la dernière version des scripts MATLAB est disponible chez GitHub (https://github.com/MartinSchatzCZ/LipidDots-analysis).
L'analyse LD réussie dépend en grande partie de la qualité des images de fluorescence brute obtenues. Pour une performance optimale des algorithmes de segmentation, des diapositives de verre propres dépourvues de particules de poussière doivent être utilisées pour la microscopie, les cellules doivent former une monocouche (le nombre réel de cellules par champ de vision n'est pas un paramètre critique), et ne doivent pas contenir un grande proportion de cellules mortes. En outre, l'imagerie z-pile devrait commencer légèrement en dessous et se terminer légèrement au-dessus des cellules. Selon la configuration microscopique particulière, les utilisateurs peuvent avoir besoin d'ajuster certains des paramètres dans les scripts de traitement d'image (tels que "th" pour le seuil d'intensité de fond d'image). Bien que la méthode actuelle soit capable de détecter et de décrire des LD individuels dans les objets cellulaires segmentés, le flux de travail ne produit pas vraiment de données unicellulaires en raison de difficultés de séparation automatisée de toutes les cellules individuelles. Au lieu de cela, le contenu LD par unité de volume cellulaire généralisé pour l'ensemble de l'échantillon est signalé. Cette limitation peut entraver l'interprétation des données dans les analyses des populations cellulaires hétérogènes. En outre, il faut prendre soin de travailler avec des cellules avec un transport altéré de petites molécules (par exemple, mutants pompe efflux), car cela pourrait affecter la concentration intracellulaire BODIPY 493/503 et la coloration LD, comme observé pour le colorant lipophile rouge du Nil33 , 34.
La coloration du milieu avec le dextran fluorescent Cascade Blue à l'imperméable est un moyen pratique de distinguer les cellules de l'arrière-plan35,qui peut être appliquée à de nombreuses espèces de levures (si ce n'est la totalité). Il contribue également à l'élimination automatisée des cellules mortes de l'analyse car celles-ci deviendront bleues lors de la coloration. Toute cellule mourante ou malade (et donc partiellement perméable pour le dextran) détectée comme vivante peut être supprimée au cours des étapes d'analyse de données en fonction de la valeur "IntensityMedianBlue" des objets cellulaires détectés. En principe, l'ensemble du flux de travail peut être utilisé pour détecter diverses autres structures cellulaires, telles que les foyers de réparation de l'ADN, à condition que les structures peuvent être étiquetées avec des fluorophores appropriés. Le flux de travail devrait également s'appliquer aux cellules d'autres espèces (de levure), ce qui élargit encore son utilité.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par les subventions de l'Université Charles PRIMUS/MED/26, GAUK 1308217 et SVV 260310. Nous remercions Ondôej Ebesta pour son aide dans le développement de la microscopie et le développement du pipeline d'analyse d'images. Nous remercions le laboratoire ReGenEx pour les souches De Cervisiae, et le laboratoire de JapoNet et Hironori Niki pour les souches de S. japonicus. La souche ppc1-88 a été fournie par The Yeast Genetic Resource Center Japan. La microscopie a été réalisée dans le Laboratoire de microscopie Confocal et Fluorescence cofinancé par le Fonds européen de développement régional et le budget de l'Etat de la République tchèque (Projet no. CZ.1.05/4.1.00/16.0347 et CZ.2.16/3.1.00/21515).
matériels
| Name | Company | Catalog Number | Comments |
| 12-bit monochromatic CCD camera Hamamatsu ORCA C4742-80-12AG | Hamamatsu | or equivalent | |
| Adenine hemisulfate salt, ≥99% | Merck | A9126-25G | |
| BODIPY 493/503 (4,4-Difluoro-1,3,5,7,8-Pentamethyl-4-Bora-3a,4a-Diaza-s-Indacene) | Thermo Fisher Scientific | D3922 | for neutral lipid staining |
| D-(+) - Glucose, ≥99.5% | Merck | G7021 | |
| Dextran, Cascade Blue, 10,000 MW, Anionic, Lysine Fixable | Thermo Fisher Scientific | D1976 | for negative staining of cells |
| Dimethyl sulfoxide, ≥99.5% | Merck | D4540 | or higher purity, keep anhydrous on molecular sieves |
| EMM broth without dextrose | Formedium | PMD0405 | medium may also be prepared from individual components |
| Fiji/ImageJ software | NIH | or equivalent; for visual inspection of microscopic data | |
| High precision cover glasses, 22 mm x 22 mm, No 1.5 | VWR | 630-2186 | use any # 1.5 cover glass |
| Image Processing Toolbox for MATLAB, version 10.0 | Mathworks | ||
| Lectin from Glycine max (soybean) | Merck | L1395 | for cell immobilization on slides |
| MATLAB software, version 9.2 | Mathworks | ||
| Microscope slide, 26 mm x 76 mm, 1 mm thickness | Knittel Glass | L762601.2 | use any microscope slide fitting your microscope stage, clean thoroughly before loading cells |
| Olympus CellR microscope with automatic z-axis objective movement | Olympus | or equivalent | |
| Pentaband filter set | Semrock | F66-985 | brightfield, green and blue channels are sufficient |
| Signal Processing Toolbox for MATLAB, version 7.4 | Mathworks | ||
| SP supplements | Formedium | PSU0101 | |
| Standard office computer capable of running MATLAB | |||
| Statistics and Machine Learning Toolbox for MATLAB, version 11.1 | Mathworks | ||
| Universal peptone M66 for microbiology | Merck | 1070431000 | |
| UPLSAPO 60XO objective | Olympus | or equivalent | |
| Yeast extract | Formedium | YEA03 | |
| Yeast nitrogen base without amino acids | Formedium | CYN0405 |
Références
- Koch, B., Schmidt, C., Daum, G. Storage lipids of yeasts: a survey of nonpolar lipid metabolism in Saccharomyces cerevisiae, Pichia pastoris, and Yarrowia lipolytica. FEMS Microbiology Reviews. 38 (5), 892-915 (2014).
- Krahmer, N., Farese, R. V., Walther, T. C. Balancing the fat: lipid droplets and human disease. EMBO Molecular Medicine. 5 (7), 973-983 (2013).
- Lazar, Z., Liu, N., Stephanopoulos, G. Holistic Approaches in Lipid Production by Yarrowia lipolytica. Trends in Biotechnology. 36 (11), 1157-1170 (2018).
- Kim, D. U., et al. Analysis of a genome-wide set of gene deletions in the fission yeast Schizosaccharomyces pombe. Nature Biotechnology. 28 (6), 1628-1629 (2010).
- Giaever, G., Nislow, C. The yeast deletion collection: a decade of functional genomics. Genetics. 197 (2), 451-465 (2014).
- Meyers, A., et al. The protein and neutral lipid composition of lipid droplets isolated from the fission yeast, Schizosaccharomyces pombe. Journal of Microbiology (Seoul, Korea). 55 (2), 112-122 (2017).
- Meyers, A., et al. Lipid Droplets Form from Distinct Regions of the Cell in the Fission Yeast Schizosaccharomyces pombe. Traffic (Copenhagen, Denmark). 17 (6), 657-659 (2016).
- Long, A. P., et al. Lipid droplet de novo formation and fission are linked to the cell cycle in fission yeast. Traffic (Copenhagen, Denmark). 13 (5), 705-714 (2012).
- Yang, H. J., Osakada, H., Kojidani, T., Haraguchi, T., Hiraoka, Y. Lipid droplet dynamics during Schizosaccharomyces pombe sporulation and their role in spore survival. Biology Open. , 8(2016).
- Aoki, K., Shiwa, Y., Takada, H., Yoshikawa, H., Niki, H. Regulation of nuclear envelope dynamics via APC/C is necessary for the progression of semi-open mitosis in Schizosaccharomyces japonicus. Genes To Cells: Devoted To Molecular & Cellular Mechanisms. 18 (9), 733-752 (2013).
- Karolin, J., Johansson, L. B. A., Strandberg, L., Ny, T. Fluorescence and Absorption Spectroscopic Properties of Dipyrrometheneboron Difluoride (BODIPY) Derivatives in Liquids, Lipid Membranes, and Proteins. Journal of the American Chemical Society. 116 (17), 7801-7806 (1994).
- Bozaquel-Morais, B. L., Madeira, J. B., Maya-Monteiro, C. M., Masuda, C. A., Montero-Lomeli, M. A new fluorescence-based method identifies protein phosphatases regulating lipid droplet metabolism. PloS One. 5 (10), e13692(2010).
- Sitepu, I. R., et al. An improved high-throughput Nile red fluorescence assay for estimating intracellular lipids in a variety of yeast species. Journal of Microbiological Methods. 91 (2), 321-328 (2012).
- Rostron, K. A., Lawrence, C. L. Nile Red Staining of Neutral Lipids in Yeast. Methods in Molecular Biology (Clifton, N.J.). 1560, 219-229 (2017).
- Romero-Aguilar, L., Montero-Lomeli, M., Pardo, J. P., Guerra-Sánchez, G. Lipid Index Determination by Liquid Fluorescence Recovery in the Fungal Pathogen Ustilago Maydis. Journal of Visualized Experiments. (134), 1-6 (2018).
- Gupta, A., Dorlhiac, G. F., Streets, A. M. Quantitative imaging of lipid droplets in single cells. The Analyst. , (2018).
- Wolinski, H., Bredies, K., Kohlwein, S. D. Quantitative imaging of lipid metabolism in yeast: from 4D analysis to high content screens of mutant libraries. Methods in Cell Biology. , 108-365 (2012).
- Campos, V., Rappaz, B., Kuttler, F., Turcatti, G., Naveiras, O. High-throughput, nonperturbing quantification of lipid droplets with digital holographic microscopy. Journal of Lipid Research. 59 (7), 1301-1310 (2018).
- Ranall, M. V., Gabrielli, B. G., Gonda, T. J. High-content imaging of neutral lipid droplets with 1,6-diphenylhexatriene. BioTechniques. 51 (1), 35-42 (2011).
- Schnitzler, J. G., et al. Nile Red Quantifier: a novel and quantitative tool to study lipid accumulation in patient-derived circulating monocytes using confocal microscopy. Journal of Lipid Research. 58 (11), 2210-2219 (2017).
- Bombrun, M., Gao, H., Ranefall, P., Mejhert, N., Arner, P., Wählby, C. Quantitative high-content/high-throughput microscopy analysis of lipid droplets in subject-specific adipogenesis models. Cytometry. Part A the journal of the International Society for Analytical Cytology. 91 (11), 1068-1077 (2017).
- Capus, A., Monnerat, M., Ribeiro, L. C., de Souza, W., Martins, J. L., Sant’Anna, C. Application of high-content image analysis for quantitatively estimating lipid accumulation in oleaginous yeasts with potential for use in biodiesel production. Bioresource Technology. 203, 309-317 (2016).
- Lv, X., et al. Identification of gene products that control lipid droplet size in yeast using a high-throughput quantitative image analysis. Biochimica et biophysica acta. Molecular and Cell Biology Of Lipids. 1864 (2), 113-127 (2018).
- Zach, R., Tvarůžková, J., Schätz, M., Ťupa, O., Grallert, B., Převorovský, M. Mitotic defects in fission yeast lipid metabolism “cut” mutants are suppressed by ammonium chloride. FEMS Yeast Research. 18 (6), 1-7 (2018).
- Petersen, J., Russell, P. Growth and the Environment of Schizosaccharomyces pombe. Cold Spring Harbor Protocols. 2016 (3), (2016).
- Aoki, K., Furuya, K., Niki, H. Schizosaccharomyces japonicus: A Distinct Dimorphic Yeast among the Fission Yeasts. Cold Spring Harbor Protocols. (12), (2017).
- Curran, B. P. G., Bugeja, V. Basic investigations in Saccharomyces cerevisiae. Methods in Molecular Biology (Clifton, N.J.). , 1-14 (2014).
- Sabatinos, S. A., Forsburg, S. L. Molecular genetics of Schizosaccharomyces pombe. Methods in Enzymology. 470 (10), 759-795 (2010).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82 (7-8), 518-529 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Nakamura, T., Pluskal, T., Nakaseko, Y., Yanagida, M. Impaired coenzyme A synthesis in fission yeast causes defective mitosis, quiescence-exit failure, histone hypoacetylation and fragile DNA. Open Biology. 2 (9), 120117(2012).
- Furuya, K., Niki, H. Isolation of heterothallic haploid and auxotrophic mutants of Schizosaccharomyces japonicus. Yeast. 26 (4), 221-233 (2009).
- Ivnitski-Steele, I., et al. Identification of Nile red as a fluorescent substrate of the Candida albicans ATP-binding cassette transporters Cdr1p and Cdr2p and the major facilitator superfamily transporter Mdr1p. Analytical Biochemistry. 394 (1), 87-91 (2009).
- Wolinski, H., Kohlwein, S. D. Microscopic analysis of lipid droplet metabolism and dynamics in yeast. Methods in Molecular Biology (Clifton, N.J.). 457 (1), 151-163 (2008).
- Graml, V., et al. A genomic Multiprocess survey of machineries that control and link cell shape, microtubule organization, and cell-cycle progression. Developmental Cell. 31 (2), 227-239 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon