Method Article
Analyse des Lipidtröpfchengehalts in Spalt- und Budding-Hefen mittels automatisierter Bildverarbeitung
In diesem Artikel
Zusammenfassung
Hier stellen wir eine MATLAB-Implementierung des automatisierten Nachweises und der quantitativen Beschreibung von Lipidtröpfchen in Fluoreszenzmikroskopiebildern von Spalt- und angehenden Hefezellen vor.
Zusammenfassung
Der Fettstoffwechsel und seine Regulierung sind sowohl für die Grundlagen- als auch für die angewandten Biowissenschaften und die Biotechnologie von Interesse. In dieser Hinsicht werden verschiedene Hefearten als Modelle in der Fettstoffwechselforschung oder für die industrielle Lipidproduktion verwendet. Lipidtröpfchen sind hochdynamische Speicherkörper und ihr zellulärer Inhalt stellt eine bequeme Auslesung des Fettstoffwechselzustands dar. Die Fluoreszenzmikroskopie ist eine Methode der Wahl für die quantitative Analyse zellulärer Lipidtröpfchen, da sie auf weit verbreitete Geräte angewiesen ist und die Analyse einzelner Lipidtröpfchen ermöglicht. Darüber hinaus kann die mikroskopische Bildanalyse automatisiert werden, was den Gesamtanalysedurchsatz erheblich erhöht. Hier beschreiben wir einen experimentellen und analytischen Workflow zur automatisierten Detektion und quantitativen Beschreibung einzelner Lipidtröpfchen in drei verschiedenen Modellhefearten: den Spalthefen Schizosaccharomyces pombe und Schizosaccharomyces japonicus, und die aufkeimende Hefe Saccharomyces cerevisiae. Lipidtröpfchen werden mit BODIPY 493/503 visualisiert, und zellunpermeablefluoreszierende Dextran wird den Kulturmedien hinzugefügt, um Zellgrenzen zu identifizieren. Zellen werden in grünen und blauen Kanälen der 3D-Epifluoreszenzmikroskopie unterzogen und die resultierenden Z-Stack-Bilder werden automatisch von einer MATLAB-Pipeline verarbeitet. Das Verfahren gibt umfangreiche quantitative Daten über den zellulären Lipidtröpfchengehalt und die individuellen Lipidtröpfcheneigenschaften in einem tabellarischen Format aus, das für nachgelagerte Analysen in großen Tabellen- oder statistischen Paketen geeignet ist. Wir bieten Beispielanalysen des Fetttröpfchengehalts unter verschiedenen Bedingungen, die den zellulären Fettstoffwechsel beeinflussen.
Einleitung
Lipide spielen eine entscheidende Rolle im Zellenergie- und Kohlenstoffstoffwechsel, bei der Synthese von Membrankomponenten und bei der Produktion bioaktiver Substanzen. Der Fettstoffwechsel wird entsprechend den Umweltbedingungen, der Nährstoffverfügbarkeit und der Zellzyklusphase1abgestimmt. Beim Menschen ist der Fettstoffwechsel mit Krankheiten wie Fettleibigkeit, Typ-II-Diabetes und Krebs verbunden2. In der Industrie stellen Lipide, die von Mikroorganismen wie Hefen hergestellt werden, eine vielversprechende Quelle für erneuerbare Dieselkraftstoffedar 3. Zellen speichern neutrale Lipide in sogenannten Lipidtröpfchen (LDs). Diese evolutionär konservierten Körper bestehen aus Triacylglycerolen, Sterylestern, einer äußeren Phospholipid-Monoschicht und assoziierten Proteinen1. LDs stammen aus dem endoplasmatischen Retikulum, üben zellzyklus- oder Wachstumsphasendynamik aus und sind wichtig für die zelluläre Lipidhomöostase1. LD-Nummer und Morphologie können als bequemer Proxy verwendet werden, wenn sie den Fettstoffwechsel unter verschiedenen Wachstumsbedingungen untersuchen oder eine Gruppe von Mutanten untersuchen. Aufgrund ihrer dynamischen Natur sind Techniken, die in der Lage sind, die Eigenschaften einzelner LDs zu analysieren, von besonderem Interesse in Studien des Fettstoffwechsels.
Verschiedene Hefearten wurden verwendet, um lipidbedingte Stoffwechselwege und ihre Regulierung zu beschreiben, oder in der Biotechnologie verwendet, um interessante Verbindungen oder Brennstoffe zu produzieren1. Darüber hinaus stehen für Modellhefen, wie die angehende Hefe Saccharomyces cerevisiae oder die entfernt verwandte Spalthefe Schizosaccharomyces pombe,genomweite Deletionsstammbibliotheken zur Verfügung, die für Hochdurchsatz verwendet werden können. Bildschirme4,5. Kürzlich wurden LD-Zusammensetzung und -Dynamik in S. pombe6,7,8,9beschrieben und Mutanten im Zusammenhang mit dem Fettstoffwechsel wurden in der entstehenden Modellhefe isoliert Schizosaccharomyces japonicus10.
Es stehen zahlreiche Techniken zur Verfügung, um LD-Inhalte und -Dynamiken zu untersuchen. Die meisten verwenden eine Art von Färbung von LDs mit lipophilen Farbstoffen wie Nile Red oder BODIPY 493/503. Letzteres zeigt engere Anregungs- und Emissionsspektren und eine erhöhte Spezifität gegenüber neutralen Lipiden (LDs) im Gegensatz zu Phospholipiden (Membranen)11. Fluorimetrische und Strömungszytometrie-Methoden wurden erfolgreich in verschiedenen Pilzarten verwendet, um Gene und Wachstumsbedingungen aufzudecken, die den Speicherfettgehalt12,13,14,15beeinflussen. Obwohl diese Methoden für Anwendungen mit hohem Durchsatz geeignet sind, können sie die Anzahl und Morphologie einzelner LDs in Zellen nicht messen, die sich zwischen Wachstumsbedingungen und Genotypen dramatisch unterscheiden können. Kohärente Raman-Streuung oder digitale holographische Mikroskopie sind etikettenfreie Methoden, die Daten auf LD-Ebene liefern, aber spezielle teure Geräte erfordern16,17,18. Die Fluoreszenzmikroskopie hingegen kann detaillierte Daten zu LD-Inhalten liefern und gleichzeitig allgemein verfügbare Instrumente und Softwaretools zur Bildanalyse verwenden. Es gibt mehrere Analyse-Workflows, die unterschiedliche Raffinesse und Automatisierung in der Zell-/LD-Erkennung aus Bilddaten aufweisen und für verschiedene Zelltypen optimiert sind, z. B. metazoanische Zellen mit großen LDs19,20 , 21, oder angehende Hefen17,22,23. Einige dieser Ansätze funktionieren nur in 2D (z. B. bei maximalen Projektionsbildern), die den zellulären LD-Inhalt möglicherweise nicht zuverlässig beschreiben. Unserer Kenntnis nach gibt es keine Werkzeuge zur Bestimmung des LD-Gehalts und der Morphologie aus mikroskopischen Spalthefedaten. Die Entwicklung automatisierter und robuster Analysen auf LD-Ebene würde eine erhöhte Empfindlichkeit und erhöhte statistische Leistung bringen und umfassende Informationen über neutralen Lipidgehalt liefern, idealerweise bei mehreren Hefearten.
Wir haben einen Workflow für die LD-Inhaltsanalyse aus 3D-Fluoreszenzmikroskopiebildern von Hefezellen entwickelt. Lebende Zellen werden mit BODIPY 493/503 und Cascade Blue dextran gebeizt, um LDs zu visualisieren bzw. Zellgrenzen zu bestimmen. Zellen werden auf Glasdias immobilisiert und mit einem Standard-Epifluoreszenzmikroskop einer Z-Stack-Bildgebung unterzogen. Die Bilder werden dann von einer automatisierten Pipeline verarbeitet, die in MATLAB, einem weit verbreiteten (kommerziellen) Paket für statistische Analysen, implementiert ist. Die Pipeline führt Bildvorverarbeitung, Segmentierung (Zellen vs. Hintergrund, Entfernen abgestorbener Zellen) und LD-Identifikation durch. Umfangreiche Daten auf LD-Ebene, wie LD-Größe und Fluoreszenzintensität, werden dann in einem tabellarischen Format bereitgestellt, das mit wichtigen Tabellenkalkulationssoftware-Tools kompatibel ist. Der Workflow wurde erfolgreich eingesetzt, um die Auswirkungen der Stickstoffquellenverfügbarkeit auf den Fettstoffwechsel in S. pombe24zu bestimmen. Wir demonstrieren nun die Funktionalität des Workflows in S. pombe, S. japonicus und S. cerevisiae, mit Wachstumsbedingungen oder Mutanten, die zellulären LD-Gehalt beeinflussen.
Protokoll
1. Vorbereitung von Lösungen und Medien
-
Bereiten Sie Lipid-Färbunglösung.
- Zur Herstellung von Lagerlipid-Färbunglösung lösen 10 mg BODIPY 493/503 in 10 ml wasserfreien DMSO (Endkonzentration 1 mg/ml). Lösen Sie den gesamten Gehalt einer 10 mg BODIPY 493/503 Durchstechflasche auf, um Materialverluste beim Wiegen zu verhindern.
ACHTUNG: DMSO kann durch die Haut passieren. Tragen Sie eine geeignete persönliche Schutzausrüstung. - Bereiten Sie die arbeitsfähige Lipid-Färbungslösung vor, indem Sie 100 l der 1 mg/ml BODIPY 493/503-Stammlösung und 900 l wasserfreien DMSO (Endkonzentration 0,1 mg/ml) mischen.
- Aliquot die Lager- und Arbeitslösungen, und lagern Bei -20 °C.
HINWEIS: Gelöste BODIPY 493/503 ist für mehrere Jahre bei -20 °C stabil. Die Lösung muss jedoch vor Feuchtigkeit und Licht geschützt werden.
- Zur Herstellung von Lagerlipid-Färbunglösung lösen 10 mg BODIPY 493/503 in 10 ml wasserfreien DMSO (Endkonzentration 1 mg/ml). Lösen Sie den gesamten Gehalt einer 10 mg BODIPY 493/503 Durchstechflasche auf, um Materialverluste beim Wiegen zu verhindern.
- Um eine Stammlösung für die Visualisierung der Zellgrenze vorzubereiten, lösen Sie 25 mg Cascade Blue dextran (ganze Durchstechflasche) in 2,5 ml entionisiertem Wasser (Endkonzentration 10 mg/ml). Aliquot die Lagerlösung und lagern Sie bei -20 °C vor Licht geschützt.
- Zur Herstellung der Mikroskop-Diabeschichtungslösung 5 mg Sojalectin in 5 ml entionisiertem Wasser (Endkonzentration 1 mg/ml) auflösen. Aliquot die Lektinlösung und lagern Sie bei -80 °C.
HINWEIS: Die Lektinlösung ist bei -80 °C mehrere Jahre stabil. Aliquots, die derzeit verwendet werden, können bei -20 °C gelagert werden. -
Bereiten Sie Kultivierungsmedien vor.
- Um 400 ml komplexes JA-Anbaumedium für S. Pombe und S. japonicus zuzubereiten, 2 g Hefeextrakt und 0,1 g SP-Ergänzungen (falls für auxotrophe Mutanten erforderlich) in 340 ml entionisiertem Wasser in einer 500 ml Flasche und Autoklaven auflösen. Fügen Sie 60 ml 20% (w/v) separat autoklavte oder filtersterilisierte Glukose unter aseptischen Bedingungen hinzu.
- Um 400 ml definiertes EMM-Anbaumedium für S. pombe und S. japonicus vorzubereiten, 4,9 g EMM-Brühe ohne Dextrose in 360 ml entionisiertem Wasser in einer 500 ml Flasche und einem Autoklaven auflösen. 40 ml 20 % (w/v) separat autoklavte oder filtersterilisierte Glukose unter aseptischen Bedingungen hinzufügen.
HINWEIS: Allgemeine Richtlinien zur Kultivierung von S. pombe und S. japonicus finden Sie unter25 bzw.26. - Um 300 ml komplexes YPAD-Anbaumedium für Saccharomyces cerevisiaevorzubereiten, lösen Sie 3 g Hefeextrakt, 6 g Pepton und 30 mg Adeninsulfat in 270 ml entionisiertem Wasser in einer 500 ml Flasche und Einem Autoklaven auf. Fügen Sie 30 ml 20% (w/v) separat autoklavte oder filtersterilisierte Glukose unter aseptischen Bedingungen hinzu.
- Um 300 ml definiertes Minimalmedium für S. cerevisiae vorzubereiten, lösen Sie 2 g Hefestickstoffbasis (ohne Aminosäuren) in 270 ml entionisiertem Wasser in einer 500 ml Flasche und einem Autoklaven auf. Fügen Sie 30 ml 20% (w/v) separat autoklavte oder filtersterilisierte Glukose unter aseptischen Bedingungen hinzu.
HINWEIS: Allgemeine Richtlinien zum Anbau von S. cerevisiae finden Sie unter27.
2. Zellkultivierung
-
Wachsende S. pombe oder S. japonicus zu exponentiellen oder frühen stationären Phase.
- Am Morgen 5 ml YES-Medium mit frischer Spalthefe-Biomasse impfen. Bei 32 °C mit Schütteln (180 Rpm) mehrere Stunden bebrüten.
HINWEIS: Verwenden Sie für alle Kultivierungen Erlenmeyerkolben mit dem 10-fachen Kulturvolumen, um eine ordnungsgemäße Belüftung zu gewährleisten. Einige Laboratorien ziehen es vor, Spalthefen bei 30 °C anzubauen, aber die Kultivierungstemperatur von 32 °C führt zu kürzeren Verdoppelungszeiten ohne nachteilige Auswirkungen auf die Zellen, wodurch die Gesamtzeit für die Durchführung eines Experiments verringert wird25,28 . - Am späten Nachmittag desselben Tages (nach mindestens 6 Stunden Kultivierung) die Kultur mit frischem JA-Medium auf ein 10 ml Endkulturvolumen verdünnen, so dass sie am nächsten Morgen die gewünschte optische Dichte (OD) (oder Anzahl der Zellen/ml) erreicht, und bei 32 °C mit s Haking (180 Rpm). Es ist von Vorteil, die Verdoppelungszeit jedes verwendeten Stammes zu kennen, um den Verdünnungsfaktor genau zu bestimmen (Verwenden Sie Gleichung 1).

Wenn V-Kultur das für die Verdünnung benötigte Präkulturvolumen ist, ist Vfinal das Gesamtvolumen der neuen Kultur (10 ml für Standardkulturen), ODfinal ist das gewünschte OD, das erreicht wird, am nächsten Morgen ist DEROD-Strom die derzeit gemessene OD der Präkultur, t ist die Zeit des Zellwachstums bis zur Ernte, tVerzögerung ist Die Dauer der Verzögerungsphase (abhängig von den Laborbedingungen, muss empirisch definiert werden) und tDT ist die Verdoppelungszeit der Sorte.
HINWEIS: Wenn exponentielle Phasenzellen analysiert werden sollen, lassen Sie die Präkulturen nicht in die stationäre Phase gelangen, da dies die Zellphysiologie (einschließlich LD-Gehalt) für mehrere nachfolgende Generationen dramatisch verändert. - Am Morgen des Bildtages, wenn die Kultur etwas höher OD als erforderlich erreicht (im Falle von exponentiellen Phasenzellen), verdünnen Sie es mit frischem JA und setzen Sie die Inkubation für mindestens zwei weitere Verdoppelungszeiten vor der Färbung von LDs fort. Färbung (Abschnitt 3).
- Am Morgen 5 ml YES-Medium mit frischer Spalthefe-Biomasse impfen. Bei 32 °C mit Schütteln (180 Rpm) mehrere Stunden bebrüten.
-
Wachsende S. cerevisiae zu exponentiellen und stationären Phase.
- Am Nachmittag 10 ml YPAD-Medium mit einer kleinen Menge an frisch aufkeimender Hefebiomasse impfen und über Nacht bei 30 °C mit Schütteln (180 U/min) inkubieren.
- Am Morgen des Bildtages, verdünnen Sie die Kultur auf OD 0,1 in 10 ml YPAD Medium und wachsen auf die erforderliche OD (z.B. OD 1 für exponentielle Phase). Führen Sie alle Kulturverdünnungen durch, wie in Schritt 2.1.2 beschrieben. Fahren Sie mit der Färbung fort (Abschnitt 3).
3. Lipid Droplet Färbung
- Bereiten Sie einen Mikroskop-Abdeckungsschlupf für jede zu bebilderte Probe vor. Mit der langen Seite einer horizontal positionierten Pipettenspitze 1 L Gleitbeschichtungslösung auf einen sauberen Deckelschlupf verteilen. Lassen Sie die Beschichtungslösung vollständig trocknen und lagern Sie die Abdeckschlupf in einer staubfreien Umgebung.
HINWEIS: Glasschlitten und Abdeckungen können bei Bedarf vor dem Gebrauch gereinigt werden. Das Reinigungsverfahren besteht aus dem Waschen mit Geschirrspülmittel, dem Spülen mit Wasser, dem Einweichen von 3% Salzsäure über Nacht und dem Waschen mit destilliertem Wasser. Gereinigte Dias und Abdeckungen werden bis zur Verwendung in reinem Ethanol gelagert. - Messen Sie die OD der Zellkultur oder die Anzahl der Zellen/ml, je nach Bedarf. Versuchen Sie, ähnliche Werte unter allen getesteten Stämmen zu erreichen, um vergleichbare experimentelle Bedingungen zu gewährleisten.
- Pipette 1 ml jeder Zellkultur zu einem 1,5 ml Mikrozentrifugenrohr. Nur für S. cerevisiae 5 l der Gleitbeschichtungslösung hinzufügen, kurz wirbeln und bei 30 °C mit Schütteln für 5 min inkubieren.
- Fügen Sie jeder Kultur-Aliquot und Wirbel kurz 1 l der Lipid-Färbungslösung hinzu. Fügen Sie dann kurz 10 L der Zellgrenzvisualisierungslösung und wirbeln sie hinzu.
HINWEIS: Bereiten Sie keine vorgemischten Lösungen beider Flecken vor, da dies zu einer Fluoreszenzabschreckung von BODIPY 493/503 führt. - Sammeln Sie die Zellen durch Zentrifugation (1.000 x g, 3 min, RT) und entfernen Sie fast alle Überstand (ca. 950 l). Setzen Sie die Zellen im verbleibenden Überstand wieder aus.
- Pipette 2 l der dichten Zellsuspension auf einem mit Lektin beschichteten Deckel schlupf und auf ein sauberes Mikroskopschlitten legen. Die Zellen sollten eine Monoschicht bilden. Fahren Sie so schnell wie möglich mit der Mikroskopie (Abschnitt 4) fort, um Artefakte in der Bildgebung zu minimieren; maximal zwei Proben gleichzeitig verarbeiten.
4. Einrichten des Mikroskops und der Bildgebung
-
Optimieren Sie die Bildgebungsbedingungen.
HINWEIS: Das Einrichten des Mikroskops erfordert lange Expositionen gegenüber starken Lichtquellen, die Schäden an der Probe und schiefe Ergebnisse verursachen können. Richten Sie daher die Bildbedingungen mithilfe eines dedizierten Beispieldias ein, das nicht weiter für die LD-Quantifizierung verwendet wird.- Konzentrieren Sie sich auf die Zellen mit Phasenkontrast oder Differential interferenzkontrast (DIC).
HINWEIS: Phasenkontrast- oder DIC-Bilder können als Referenz verwendet werden, werden jedoch während des automatischen Bildanalyseschritts nicht verwendet. - Legen Sie die Z-Stack-Einstellungen so fest, dass sie das gesamte Zellenvolumen umfassen. Die gesamte vertikale Entfernung hängt von der Zellengröße ab. die Anzahl der optischen Scheiben hängt von der numerischen Blende des Objektivs ab (Punktstreufunktion in z-Achse). Legen Sie den Fokus so fest, dass er relativ zur zentralen Fokusebene verschoben wird.
HINWEIS: Die optimale Anzahl von Scheiben wird oft von der Mikroskopsteuerungssoftware eingestellt und muss nicht manuell berechnet werden. Die typischen Zellbreiten sind 3-5 m für S. pombe,4-7 m für S. japonicusund 3-7 m für S. cerevisiae. - Um LDs abzubilden, stellen Sie Lichtintensität und Belichtungszeit im grünen Kanal ein (Erregungs- und Emissionsmaxima von BODIPY sind 493 bzw. 503 nm).
HINWEIS: BODIPY 493/503 ist ein sehr helles Fluorchrom; jedoch kann es schnell mit zu starker Lichtintensität gebleicht werden. Darüber hinaus sind LDs in live Zellen mobil, minimieren so die Belichtungszeit und erfassen zuerst den vollständigen Grünen-Kanal-Z-Stack (vor dem Wechsel zum blauen Kanal), um unschärfende Artefakte zu verhindern. Berücksichtigen Sie auch den linearen Bereich der Kamera für die Signalintensität, um gesättigte Pixel zu vermeiden. - Um Zellgrenzen abzubilden, legen Sie Lichtintensität und Belichtungszeit im blauen Kanal fest (Erregungs- und Emissionsmaxima von Cascade Blue dextran sind 400 bzw. 420 nm).
HINWEIS: Die Signalintensität im blauen Kanal ist für die Bildsegmentierung erforderlich, wird aber nicht für die LD-Quantifizierung selbst verwendet. Daher sind optimale Einstellungen in diesem Kanal für die Analyse nicht entscheidend. - Erstellen Sie nach Möglichkeit einen automatisierten experimentellen Workflow in der Mikroskopsteuerungssoftware, um die Bildgebung mehrerer Proben unter standardisierten Bedingungen zu erleichtern.
- Konzentrieren Sie sich auf die Zellen mit Phasenkontrast oder Differential interferenzkontrast (DIC).
- Sobald die Bildbedingungen optimiert wurden, werden Bildproben für die Quantifizierung verwendet. Konzentrieren Sie sich auf die Zellen und stellen Sie sie in grünen und blauen Kanälen ab, wie in Schritt 4.1 beschrieben.
HINWEIS: Alle Bilder müssen mit den gleichen Einstellungen erfasst werden, um einen Vergleich zwischen Samples zu ermöglichen. Bildner mehrere Sichtfelder pro Beispiel, um robuste, repräsentative Daten zu erhalten. - Speichern Sie die z-Stack-Bilder mit blauem und grünem Kanal als 16-Bit-Mehrschicht-TIFF-Dateien (d. h. zwei Dateien pro Sichtfeld). Fügen Sie die Wörter "grün" oder "blau" in die entsprechenden Dateinamen ein. Fahren Sie mit der Bildanalyse fort (Abschnitt 5).
5. Bildanalyse
-
Überprüfen Sie visuell die Qualität der aufgenommenen Bilder.
- Öffnen Sie mikroskopische Bilder in ImageJ29,30 oder einer anderen geeigneten Bildanalysesoftware.
- Entfernen Sie alle Bildstapel, die eine beträchtliche Anzahl von Zellen enthalten, die während der Erfassung verschoben wurden (und dadurch unscharfe Artefakte erzeugt enden).
- Entfernen Sie alle Bildstapel, die hochfluoreszierende nichtzellige Partikel im blauen Kanal enthalten (z. B. Schmutz auf Mikroskopschlitten oder Deckschlupf, Verunreinigungen im Anbaumedium).
HINWEIS: Sehr helle Nicht-Zell-Objekte im blauen Kanal können Zellerkennungsartefakte erzeugen oder die Erkennung von Zellen in ihrer Nähe beeinträchtigen. - Entfernen Sie alle Bildstapel, die einen großen Anteil abgestorbener Zellen enthalten (d. h. Zellen mit erhöhter blauer Fluoreszenz im Vergleich zu lebenden Zellen).
HINWEIS: Während das Vorhandensein eines kleinen Anteils abgestorbener Zellen in der Probe in der Regel kein Problem darstellt und diese Zellen während der Analyse automatisch verworfen werden, können einige abgestorbene oder absterbende Zellen gelegentlich vom Segmentierungsalgorithmus als lebende Zellen erkannt werden und somit die gemeldeten Ergebnisse verzerren.
-
Analysieren Sie Bilder in der MATLAB-Software.
- Erstellen Sie einen Hauptordner, und kopieren Sie alle MATLAB-Skripts an diesen Speicherort.
- Erstellen Sie einen Unterordner ("pombe", "cerevisiae" oder "japonicus") und kopieren Sie Eingabe-TIFF-Bilddateien an diesen Speicherort.
- Starten Sie MATLAB, öffnen Sie das Skript MAIN.m und führen Sie es aus. Wählen Sie im Menü die zu analysierende Hefeart aus und starten Sie die Bildverarbeitung.
HINWEIS: Einige der für die Zell- und LD-Erkennung erforderlichen Parameter sind für die jeweilige Art voreingestellt, andere werden bei der Bildverarbeitung automatisch ermittelt. Die voreingestellten Werte wurden empirisch ermittelt und hängen von mehreren Faktoren wie objektiver Vergrößerung, Kameratyp und Empfindlichkeit sowie Bildgebungseinstellungen ab. Bei Bedarf können Benutzer die Skriptdateien bearbeiten, um die organismusspezifischen Voreinstellungen zu ändern, um ihre experimentellen Einstellungen besser widerzuspiegeln. Nämlich werden bei der Zellerkennung akzeptable Objektgrößen durch die Parameter "minArea" und "maxArea" angegeben, und der minimale Anteil des gefüllten Volumens innerhalb der Objektgrenzen wird durch den Parameter "Solidity" angegeben. Für die LD-Erkennung wird der Helligkeitsschwellenwert durch den Parameter "th" angegeben (sein Wert wird hauptsächlich durch die Bildbittiefe und die Fluoreszenzsignalintensität beeinflusst), und die maximal akzeptable LD-Größe wird durch den Parameter "MaxArea" angegeben. - Überprüfen und verarbeiten Sie die Ausgabedateien nach Bedarf mit einem Tabelleneditor oder einem statistischen Paket; Der Workflow erzeugt semikolongetrennte CSV-Dateien und segmentierte TIFF-Dateien mit erkannten Zellobjekten und LDs.
HINWEIS: Der Workflow segmentiert Bilder in Hintergrund- und Zellobjekte, wobei jedes Zellobjekt aus mehreren benachbarten Zellen bestehen kann. Daher stellt die Ausgabe in "xxxx_cells.csv"-Dateien keine Einzelzellendaten dar und sollte nur zur Berechnung von Metriken pro Einheit des Zellenvolumens verwendet werden.
Ergebnisse
Das gesamte Verfahren ist in Abbildung 1 für die Spalthefen zusammengefasst (der angehende Hefe-Workflow ist analog), und unten finden Sie Beispiele dafür, wie der Workflow verwendet werden kann, um LD-Inhalte in drei verschiedenen Hefearten unter verschiedenen bekannten Bedingungen zu untersuchen. um den Zell-LD-Inhalt zu beeinflussen. Jedes Beispiel stellt ein einziges biologisches Experiment dar.
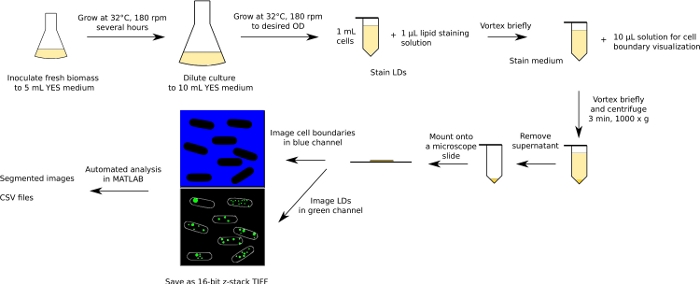
Abbildung 1: Schematisches Diagramm des experimentellen und analytischen Workflows. Der Workflow für Spalthefen wird als Beispiel dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Zuerst analysierten wir S. Pombe-Zellen (Abbildung 2). Wild-Type (WT; h +s) Zellen wurden entweder im komplexen JA-Medium oder im definierten EMM-Medium zur exponentiellen Phase herangewachsen. Im Vergleich zu JA wurden in EMM weniger LDs und eine höhere LD-Färbungsintensität pro Zellvolumeneinheit nachgewiesen (Abbildung 2A-C). Darüber hinaus waren einzelne LDs, die im EMM-Medium gebildet wurden, größer und zeigten eine erhöhte Gesamtfärbeintensität (Abbildung 2D, E). Dies steht im Widerspruch zu früheren Erkenntnissen über einen erhöhten Speicherfettgehalt in Zellen, die in EMM24angebaut werden. Das ppc1-Gen kodiert eine Phosphopantothen-Cysteinligase, die für die Coenzym-A-Synthese erforderlich ist. Die temperaturempfindliche Ppc1-88 Mutante zeigt einen deutlichen Rückgang des LD-Gehalts, wenn sie bei der restriktiven Temperatur31angebaut wird, und liefert ein Beispiel für Zellen mit niedrigem BODIPY 493/503-Signal (Abbildung 2A). Dementsprechend wurden im Vergleich zum Wildtyp (bei 32°C) kleinere LDs mit geringerer Gesamtfärbeintensität in ppc1-88-Zellen nachgewiesen, die in JA nach einer Verschiebung auf 36 °C angebaut wurden (Abbildung2D, E), ohne dass sich die LD-Zahl pro Einheit des Zellvolumens (Abbildung 2B).
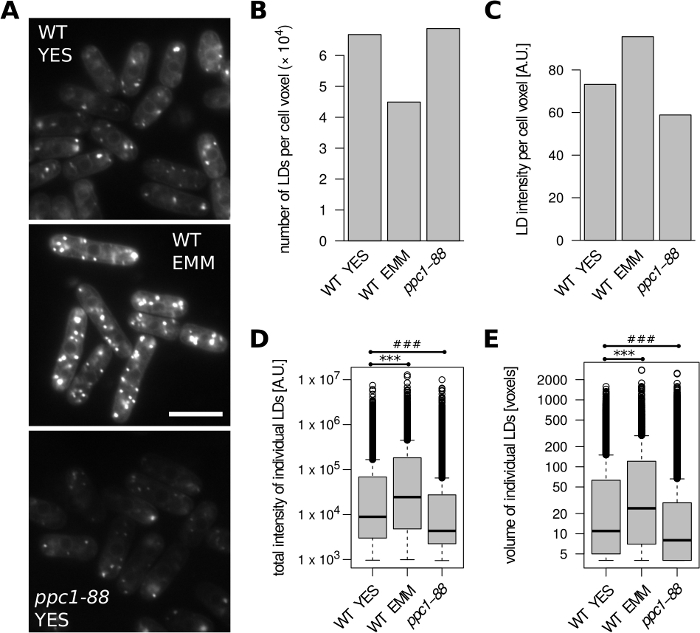
Abbildung 2: Auswirkungen der Wachstumsmedien und der Lipidstoffwechselmutation auf den LD-Gehalt in S. pombe. Wildtyp (WT) und ppc1-88 Zellen wurden zu exponentiellen Phase in der komplexen JA oder definierten EMM Medium angebaut, wie angegeben. WT-Zellen wurden bei 32 °C angebaut. Die temperaturempfindlichen ppc1-88-Zellen wurden bei 25°C angebaut und vor der Analyse für 2 Stunden auf 36°C verschoben. (A) Repräsentative unverarbeitete mikroskopische Bilder von LDs, die mit BODIPY 493/503 gefärbt sind. Für jede Bedingung wird ein einzelnes optisches Slice angezeigt. 10% Overlay mit invertiertem blauen Kanal wurde hinzugefügt, um Zellgrenzen besser zu visualisieren. Skala bar = 10 m . (B) Anzahl der identifizierten LDs pro Einheit des Zellvolumens. (C) Fluoreszenzintensität der identifizierten LDs pro Zellvolumeneinheit. (D) Verteilung der Gesamtfluoreszenzintensitäten aller identifizierten LDs. ***, ungepaarter Wilcoxon-Test p = 1,7 x 10-107, p = 3,7 x 10-132, bzw. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . (E) Verteilung enkundungen von allen identifizierten LDs. ***, ungepaarter Wilcoxon-Test p = 6,8 x 10-71, p = 1 x 10-64, bzw. Die Daten in den Panels B-E wurden aus 242, 124 und 191 Zellobjekten für die Proben WT YES, WT EMM und ppc1-88 abgeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Als nächstes quantifizierten wir den LD-Gehalt in S. japonicus-Zellen (h+matsj-2017)32 aus exponentiellen und frühstationären Kulturen, die in JA angebaut wurden (Abbildung 3A). Zellen, die in die stationäre Phase eintraten, zeigten eine deutlich verringerte Anzahl von LDs pro Zellvolumeneinheit im Vergleich zu exponentiell wachsenden Zellen (Abbildung 3B), während die volumennormalisierte LD-Fluoreszenzintensität zwischen den beiden Bedingungen leicht abnahm ( Abbildung 3C). Die frühen stationären LDs waren in der Regel mäßig größer und hatten eine mäßig höhere Gesamtfluoreszenzintensität im Vergleich zu LDs aus exponentiell wachsenden Zellen (Abbildung 3D, E).

Abbildung 3: LD-Inhalt in S. japonicus Zellen ändert sich mit Derwachstumsphase. Exponentiell wachsende (LOG) und frühe stationäre Phase (STAT) Zellen wurden analysiert. (A) Repräsentative unverarbeitete mikroskopische Bilder von LDs, die mit BODIPY 493/503 gefärbt sind. Für jede Bedingung wird ein einzelnes optisches Slice angezeigt. 10% Overlay mit invertiertem blauen Kanal wurde hinzugefügt, um Zellgrenzen besser zu visualisieren. Maßstabsbalken steht für 10 m (B) Anzahl der identifizierten LDs pro Einheit des Zellvolumens. (C) Fluoreszenzintensität der identifizierten LDs pro Zellvolumeneinheit. (D) Verteilung der Gesamtfluoreszenzintensitäten aller identifizierten LDs. *** ungepaarter Wilcoxon-Test p = 1,3 x 10-114. (E) Verteilung der Volumina aller identifizierten LDs. *** ungepaarter Wilcoxon-Test p = 2,4 x 10-85. Die Daten in den Panels B-E wurden aus 274 bzw. 187 Zellobjekten für die LOG- bzw. STAT-Proben abgeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Schließlich analysierten wir S. cerevisiae Zellen des weit verbreiteten BY4741 Laborstamms (MATa his3'1 leu2'0 met15'0 ura3'0), die im komplexen YPAD-Medium zu exponentiellen bzw. stationären Phasen herangewachsen sind. Angehende Hefezellen akkumulieren in der Regel Speicherlipide beim Eintritt in die stationäre Phase1, und wir konnten diese Befunde rekapitulieren (Abbildung 4). Stationäre Zellen enthielten etwas weniger LDs pro Volumeneinheit im Vergleich zu exponentiell wachsenden Zellen (Abbildung 4B), aber ihre volumennormalisierte LD-Fluoreszenzintensität verdoppelte sich fast (Abbildung 4C). Dieser starke Anstieg des GesamtenLD-Gehalts war auf die viel höhere Fluoreszenzintensität und das deutlich höhere Volumen einzelner LDs in stationärer Phase zurückzuführen (Abbildung 4D, E).
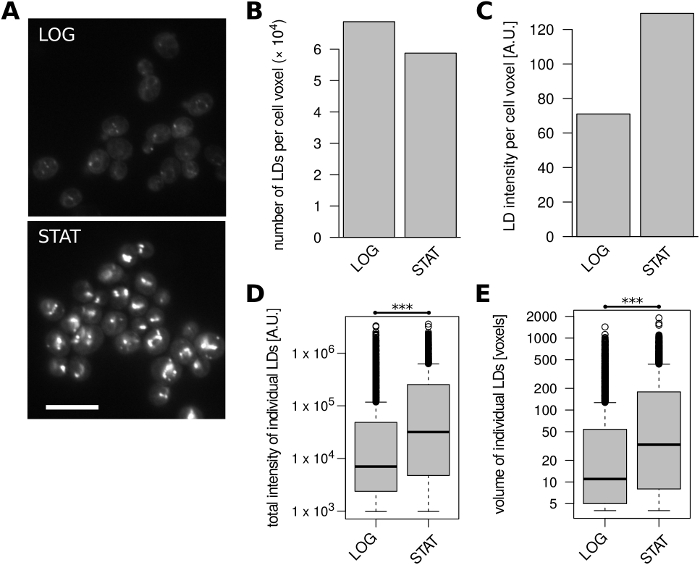
Abbildung 4: LD-Inhalt in S. cerevisiae Zellen ändert sich mit Wachstumsphase. Exponentiell wachsende (LOG) und stationäre Phase (STAT) Zellen wurden analysiert. (A) Repräsentative unverarbeitete mikroskopische Bilder von LDs, die mit BODIPY 493/503 gefärbt sind. Für jede Bedingung wird ein einzelnes optisches Slice angezeigt. 10% Overlay mit invertiertem blauen Kanal wurde hinzugefügt, um Zellgrenzen besser zu visualisieren. Maßstabsbalken steht für 10 m (B) Anzahl der identifizierten LDs pro Einheit des Zellvolumens. (C) Fluoreszenzintensität der identifizierten LDs pro Zellvolumeneinheit. (D) Verteilung der Gesamtfluoreszenzintensitäten aller identifizierten LDs. *** ungepaarter Wilcoxon-Test p = 4,6 x 10-78. (E) Verteilung der Volumina aller identifizierten LDs. *** ungepaarter Wilcoxon-Test p = 3,7 x 10-63. Die Daten in den Panels B-E wurden von 430 bzw. 441 Zellobjekten für die LOG- bzw. STAT-Proben abgeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
So kann unser Analyse-Workflow Veränderungen der LD-Zahl, -Größe und -Lipidgehalte in drei verschiedenen und morphologisch unterschiedlichen Hefearten unter verschiedenen Bedingungen erkennen, die sich positiv oder negativ auf den zellulären LD-Gehalt auswirken.
Diskussion
Das Verständnis des Fettstoffwechsels und seiner Regulierung ist sowohl für die Grundbiologie als auch für klinische und biotechnologische Anwendungen wichtig. Der LD-Gehalt stellt eine bequeme Auslesung des Lipidstoffwechselzustands der Zelle dar, wobei die Fluoreszenzmikroskopie eine der wichtigsten Methoden zur Bestimmung des LD-Gehalts ist. Das vorgestellte Protokoll ermöglicht die automatisierte Detektion und quantitative Beschreibung einzelner LDs in drei verschiedenen und morphologisch unterschiedlichen Hefearten. Unserer Kenntnis nach gibt es keine ähnlichen Werkzeuge für die Spalthefen. Die für die Bildverarbeitung erforderlichen MATLAB-Skripte sind als Zusatzdateien enthalten und sind auch im Figshare-Repository (DOI 10.6084/m9.figshare.7745738) zusammen mit allen Roh- und verarbeiteten Bild- und Tabellardaten aus diesem Manuskript verfügbar, detaillierte Beschreibungen der CSV-Ausgabedateien und R-Skripte für die nachgelagerte Datenanalyse und -visualisierung. Außerdem ist die neueste Version der MATLAB-Skripte bei GitHub (https://github.com/MartinSchatzCZ/LipidDots-analysis) verfügbar.
Eine erfolgreiche LD-Analyse hängt weitgehend von der Qualität der erhaltenen Rohfluoreszenzbilder ab. Für eine optimale Leistung der Segmentierungsalgorithmen sollten saubere Glasgdias ohne Staubpartikel für die Mikroskopie verwendet werden, die Zellen sollten eine Monoschicht bilden (die tatsächliche Anzahl der Zellen pro Sichtfeld ist kein kritischer Parameter) und sollten keine einen großen Teil der abgestorbenen Zellen. Außerdem sollte die Z-Stack-Bildgebung leicht unterhalb beginnen und leicht über den Zellen enden. Je nach mikroskopischem Setup müssen Benutzer möglicherweise einige der Parameter in den Bildverarbeitungsskripts anpassen (z. B. "th" für den Schwellenwert für die Bildhintergrundintensität). Während die aktuelle Methode in der Lage ist, einzelne LDs in den segmentierten Zellobjekten zu erkennen und zu beschreiben, erzeugt der Workflow aufgrund von Schwierigkeiten bei der automatisierten Trennung aller einzelnen Zellen keine wirklich einzelzelligen Daten. Stattdessen wird der LD-Gehalt pro Einheit des Zellvolumens, der für die gesamte Stichprobe verallgemeinert ist, gemeldet. Diese Einschränkung kann die Dateninterpretation bei Analysen heterogener Zellpopulationen behindern. Auch bei der Arbeit mit Zellen mit verändertem Transport kleiner Moleküle (z. B. Efflux-Pumpenmutanten) sollte Vorsicht geboten sein, da dies die intrazelluläre BODIPY 493/503-Konzentration und LD-Färbung beeinflussen könnte, wie bei dem Nil-Rot-Lipophilen Farbstoff33 beobachtet. , 34.
Das Beflecken des Mediums mit dem zellunperinierbaren Kaskadenblau fluoreszierenden Dextran ist eine bequeme Möglichkeit, Zellen vom Hintergrund35zu unterscheiden, die auf viele (wenn nicht alle) Hefearten angewendet werden können. Es hilft auch bei der automatisierten Entfernung von abgestorbenen Zellen aus der Analyse, da diese bei der Färbung blau werden. Alle sterbenden oder kranken (und damit teilweise durchlässigen Dextran)-Zellen, die als lebendig erkannt wurden, können während der Datenanalyseschritte auf der Grundlage des "IntensityMedianBlue"-Werts der erkannten Zellobjekte entfernt werden. Grundsätzlich kann der gesamte Workflow genutzt werden, um verschiedene andere zelluläre Strukturen, wie DNA-Reparatur-Brennpunkte, zu erkennen, sofern die Strukturen mit geeigneten Fluorophoren gekennzeichnet werden können. Der Workflow sollte auch auf Zellen anderer (Hefe-)Arten anwendbar sein, wodurch sein Nutzen weiter erweitert wird.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch die Stipendien der Karlsuniversität PRIMUS/MED/26, GAUK 1308217 und SVV 260310 unterstützt. Wir danken Ond'ej ebesta für die Hilfe bei der Mikroskopie und der Entwicklung der Bildanalyse-Pipeline. Wir danken dem ReGenEx-Labor für S. cerevisiae-Stämme und JapoNet und Hironori Nikis Labor für S. japonicus-Stämme. Der ppc1-88 Stamm wurde vom Yeast Genetic Resource Center Japan bereitgestellt. Die Mikroskopie wurde im Labor für Konfokal- und Fluoreszenzmikroskopie durchgeführt, das vom Europäischen Fonds für regionale Entwicklung und dem Staatshaushalt der Tschechischen Republik kofinanziert wurde (Projekt Nr. CZ.1.05/4.1.00/16.0347 und CZ.2.16/3.1.00/21515).
Materialien
| Name | Company | Catalog Number | Comments |
| 12-bit monochromatic CCD camera Hamamatsu ORCA C4742-80-12AG | Hamamatsu | or equivalent | |
| Adenine hemisulfate salt, ≥99% | Merck | A9126-25G | |
| BODIPY 493/503 (4,4-Difluoro-1,3,5,7,8-Pentamethyl-4-Bora-3a,4a-Diaza-s-Indacene) | Thermo Fisher Scientific | D3922 | for neutral lipid staining |
| D-(+) - Glucose, ≥99.5% | Merck | G7021 | |
| Dextran, Cascade Blue, 10,000 MW, Anionic, Lysine Fixable | Thermo Fisher Scientific | D1976 | for negative staining of cells |
| Dimethyl sulfoxide, ≥99.5% | Merck | D4540 | or higher purity, keep anhydrous on molecular sieves |
| EMM broth without dextrose | Formedium | PMD0405 | medium may also be prepared from individual components |
| Fiji/ImageJ software | NIH | or equivalent; for visual inspection of microscopic data | |
| High precision cover glasses, 22 mm x 22 mm, No 1.5 | VWR | 630-2186 | use any # 1.5 cover glass |
| Image Processing Toolbox for MATLAB, version 10.0 | Mathworks | ||
| Lectin from Glycine max (soybean) | Merck | L1395 | for cell immobilization on slides |
| MATLAB software, version 9.2 | Mathworks | ||
| Microscope slide, 26 mm x 76 mm, 1 mm thickness | Knittel Glass | L762601.2 | use any microscope slide fitting your microscope stage, clean thoroughly before loading cells |
| Olympus CellR microscope with automatic z-axis objective movement | Olympus | or equivalent | |
| Pentaband filter set | Semrock | F66-985 | brightfield, green and blue channels are sufficient |
| Signal Processing Toolbox for MATLAB, version 7.4 | Mathworks | ||
| SP supplements | Formedium | PSU0101 | |
| Standard office computer capable of running MATLAB | |||
| Statistics and Machine Learning Toolbox for MATLAB, version 11.1 | Mathworks | ||
| Universal peptone M66 for microbiology | Merck | 1070431000 | |
| UPLSAPO 60XO objective | Olympus | or equivalent | |
| Yeast extract | Formedium | YEA03 | |
| Yeast nitrogen base without amino acids | Formedium | CYN0405 |
Referenzen
- Koch, B., Schmidt, C., Daum, G. Storage lipids of yeasts: a survey of nonpolar lipid metabolism in Saccharomyces cerevisiae, Pichia pastoris, and Yarrowia lipolytica. FEMS Microbiology Reviews. 38 (5), 892-915 (2014).
- Krahmer, N., Farese, R. V., Walther, T. C. Balancing the fat: lipid droplets and human disease. EMBO Molecular Medicine. 5 (7), 973-983 (2013).
- Lazar, Z., Liu, N., Stephanopoulos, G. Holistic Approaches in Lipid Production by Yarrowia lipolytica. Trends in Biotechnology. 36 (11), 1157-1170 (2018).
- Kim, D. U., et al. Analysis of a genome-wide set of gene deletions in the fission yeast Schizosaccharomyces pombe. Nature Biotechnology. 28 (6), 1628-1629 (2010).
- Giaever, G., Nislow, C. The yeast deletion collection: a decade of functional genomics. Genetics. 197 (2), 451-465 (2014).
- Meyers, A., et al. The protein and neutral lipid composition of lipid droplets isolated from the fission yeast, Schizosaccharomyces pombe. Journal of Microbiology (Seoul, Korea). 55 (2), 112-122 (2017).
- Meyers, A., et al. Lipid Droplets Form from Distinct Regions of the Cell in the Fission Yeast Schizosaccharomyces pombe. Traffic (Copenhagen, Denmark). 17 (6), 657-659 (2016).
- Long, A. P., et al. Lipid droplet de novo formation and fission are linked to the cell cycle in fission yeast. Traffic (Copenhagen, Denmark). 13 (5), 705-714 (2012).
- Yang, H. J., Osakada, H., Kojidani, T., Haraguchi, T., Hiraoka, Y. Lipid droplet dynamics during Schizosaccharomyces pombe sporulation and their role in spore survival. Biology Open. , 8(2016).
- Aoki, K., Shiwa, Y., Takada, H., Yoshikawa, H., Niki, H. Regulation of nuclear envelope dynamics via APC/C is necessary for the progression of semi-open mitosis in Schizosaccharomyces japonicus. Genes To Cells: Devoted To Molecular & Cellular Mechanisms. 18 (9), 733-752 (2013).
- Karolin, J., Johansson, L. B. A., Strandberg, L., Ny, T. Fluorescence and Absorption Spectroscopic Properties of Dipyrrometheneboron Difluoride (BODIPY) Derivatives in Liquids, Lipid Membranes, and Proteins. Journal of the American Chemical Society. 116 (17), 7801-7806 (1994).
- Bozaquel-Morais, B. L., Madeira, J. B., Maya-Monteiro, C. M., Masuda, C. A., Montero-Lomeli, M. A new fluorescence-based method identifies protein phosphatases regulating lipid droplet metabolism. PloS One. 5 (10), e13692(2010).
- Sitepu, I. R., et al. An improved high-throughput Nile red fluorescence assay for estimating intracellular lipids in a variety of yeast species. Journal of Microbiological Methods. 91 (2), 321-328 (2012).
- Rostron, K. A., Lawrence, C. L. Nile Red Staining of Neutral Lipids in Yeast. Methods in Molecular Biology (Clifton, N.J.). 1560, 219-229 (2017).
- Romero-Aguilar, L., Montero-Lomeli, M., Pardo, J. P., Guerra-Sánchez, G. Lipid Index Determination by Liquid Fluorescence Recovery in the Fungal Pathogen Ustilago Maydis. Journal of Visualized Experiments. (134), 1-6 (2018).
- Gupta, A., Dorlhiac, G. F., Streets, A. M. Quantitative imaging of lipid droplets in single cells. The Analyst. , (2018).
- Wolinski, H., Bredies, K., Kohlwein, S. D. Quantitative imaging of lipid metabolism in yeast: from 4D analysis to high content screens of mutant libraries. Methods in Cell Biology. , 108-365 (2012).
- Campos, V., Rappaz, B., Kuttler, F., Turcatti, G., Naveiras, O. High-throughput, nonperturbing quantification of lipid droplets with digital holographic microscopy. Journal of Lipid Research. 59 (7), 1301-1310 (2018).
- Ranall, M. V., Gabrielli, B. G., Gonda, T. J. High-content imaging of neutral lipid droplets with 1,6-diphenylhexatriene. BioTechniques. 51 (1), 35-42 (2011).
- Schnitzler, J. G., et al. Nile Red Quantifier: a novel and quantitative tool to study lipid accumulation in patient-derived circulating monocytes using confocal microscopy. Journal of Lipid Research. 58 (11), 2210-2219 (2017).
- Bombrun, M., Gao, H., Ranefall, P., Mejhert, N., Arner, P., Wählby, C. Quantitative high-content/high-throughput microscopy analysis of lipid droplets in subject-specific adipogenesis models. Cytometry. Part A the journal of the International Society for Analytical Cytology. 91 (11), 1068-1077 (2017).
- Capus, A., Monnerat, M., Ribeiro, L. C., de Souza, W., Martins, J. L., Sant’Anna, C. Application of high-content image analysis for quantitatively estimating lipid accumulation in oleaginous yeasts with potential for use in biodiesel production. Bioresource Technology. 203, 309-317 (2016).
- Lv, X., et al. Identification of gene products that control lipid droplet size in yeast using a high-throughput quantitative image analysis. Biochimica et biophysica acta. Molecular and Cell Biology Of Lipids. 1864 (2), 113-127 (2018).
- Zach, R., Tvarůžková, J., Schätz, M., Ťupa, O., Grallert, B., Převorovský, M. Mitotic defects in fission yeast lipid metabolism “cut” mutants are suppressed by ammonium chloride. FEMS Yeast Research. 18 (6), 1-7 (2018).
- Petersen, J., Russell, P. Growth and the Environment of Schizosaccharomyces pombe. Cold Spring Harbor Protocols. 2016 (3), (2016).
- Aoki, K., Furuya, K., Niki, H. Schizosaccharomyces japonicus: A Distinct Dimorphic Yeast among the Fission Yeasts. Cold Spring Harbor Protocols. (12), (2017).
- Curran, B. P. G., Bugeja, V. Basic investigations in Saccharomyces cerevisiae. Methods in Molecular Biology (Clifton, N.J.). , 1-14 (2014).
- Sabatinos, S. A., Forsburg, S. L. Molecular genetics of Schizosaccharomyces pombe. Methods in Enzymology. 470 (10), 759-795 (2010).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82 (7-8), 518-529 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Nakamura, T., Pluskal, T., Nakaseko, Y., Yanagida, M. Impaired coenzyme A synthesis in fission yeast causes defective mitosis, quiescence-exit failure, histone hypoacetylation and fragile DNA. Open Biology. 2 (9), 120117(2012).
- Furuya, K., Niki, H. Isolation of heterothallic haploid and auxotrophic mutants of Schizosaccharomyces japonicus. Yeast. 26 (4), 221-233 (2009).
- Ivnitski-Steele, I., et al. Identification of Nile red as a fluorescent substrate of the Candida albicans ATP-binding cassette transporters Cdr1p and Cdr2p and the major facilitator superfamily transporter Mdr1p. Analytical Biochemistry. 394 (1), 87-91 (2009).
- Wolinski, H., Kohlwein, S. D. Microscopic analysis of lipid droplet metabolism and dynamics in yeast. Methods in Molecular Biology (Clifton, N.J.). 457 (1), 151-163 (2008).
- Graml, V., et al. A genomic Multiprocess survey of machineries that control and link cell shape, microtubule organization, and cell-cycle progression. Developmental Cell. 31 (2), 227-239 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten