Method Article
Comment quantifier la Fraction des protéines fluorescentes photoactivation en vrac et dans des cellules vivantes
Dans cet article
Résumé
Nous présentons ici un protocole qui implique de photoactivatable spectralement distincte génétiquement couplé et des protéines fluorescentes. Ces chimères de la protéine fluorescente permettent de quantification de la fraction de PA-FP est photoactivation être fluorescents, c'est-à-dire l’efficacité de photoactivation. Le protocole indique que les différents modes de photoactivation rendement photoactivation différentes efficacités.
Résumé
Photoactivatable et - protéines fluorescentes convertibles (PA-FPs) ont été utilisés en microscopie de fluorescence cellules vivantes pour analyser la dynamique des cellules et des ensembles de protéines. Jusqu’ici, aucune méthode n’a été disponible à quantifier en vrac et en live cellules exprimé combien du PA-SFP sont photoactivation de fluorescence.
Ici, nous présentons un protocole mettant en cause des règles internes, c'est-à-dire génétiquement couplés protéines fluorescentes spectralement distinctes (photoactivatable), à ratiometrically quantifier la fraction de tous les PA-MF intégré à une cellule qui s’allument pour être fluorescent. Utilisant ce protocole, nous montrons que les différents modes de photoactivation donné photoactivation différentes efficacités. Courte photoactivation haute puissance avec un confocal laser scanning microscope (CLSM) a entraîné jusqu'à quatre fois plus faible photoactivation efficacité que des centaines d’expositions bas niveau appliquées par CLSM ou une brève impulsion appliquée par widefield illumination. Alors que le protocole a été exemplifié ici pour GFP (PA-) et Cherry (PA-), il peut en principe être appliqué à n’importe quelle paire de protéine fluorescente photoactivatable ou et spectralement distinctes et tout montage expérimental.
Introduction
En 2002, le premier largement applicable photoactivatable (PA-GFP1) et des protéines fluorescentes et (Kaede2) ont été décrits. Ces protéines fluorescentes surligneur optique modifier leurs propriétés spectrales lors de l’irradiation UV-lumière, c'est-à-dire, ils deviennent lumineux (protéines fluorescentes photoactivatable, c.-à-d., PA-FPs), ou changer leur couleur (et Images/s). A ce jour, plusieurs réversibles et irréversibles photoactivatable et et protéines fluorescentes ont été développés3,4. Dans les études d’ensemble ou en vrac, surligneurs optiques ont été utilisés pour étudier la dynamique des cellules entières ou des protéines et la connectivité des compartiments subcellulaires. En outre, surligneurs optique activées seule molécule basée superrésolution imaging techniques telles que PALM5 et6de la FPALM.
Bien que la photochimique traite pendant photoactivation ou - conversion ont été décrits pour de nombreuses optiques surligneurs et structures cristallographiques même avant et après photoactivation / - conversion ont disponible7 , 8, le mécanismephysique sous-jacent de photo de photoactivation et - conversion n’est pas complètement élucidé. En outre, jusqu’ici seulement brutes estimations existent de l’efficacité de photoactivation et - conversion, c'est-à-dire la fraction des protéines fluorescentes exprimées autrement dit effectivement photoconverted ou photoactivation être fluorescent. Des études in vitro ensemble ont été signalés à quantifier la Maj dans les spectres d’absorption et de la quantité de natif et activé des protéines dans un gel9,10,11.
Nous présentons ici un protocole impliquant les chimères de la protéine fluorescente afin d’évaluer la fraction des protéines fluorescentes photoactivation en vrac et dans des cellules vivantes. Chaque fois que travaillant avec des protéines fluorescentes génétiquement encodés, la quantité absolue de la protéine exprimée varie de cellule en cellule et ne connaît pas. Si une cellule exprimant une PA-FP présente un signal plus clair après photoactivation qu’une autre cellule, on ne peut pas différencier si ce signal lumineux est due à une expression plus élevée de la PA-FP ou une photoactivation plus efficace du PA-FP. Pour uniformiser le niveau d’expression dans les cellules, nous introduisons des règles internes de protéines fluorescentes spectralement distinctes génétiquement couplés. Par couplage de l’information génétique d’une protéine fluorescente photoactivatable à un spectralement distincts toujours en protéines fluorescentes, règles internes sont créés qui sera toujours exprimé pour un montant inconnu, mais dans une quantité relative fixe et connue de 1 : 1. cette stratégie permet la caractérisation quantitative des différents régimes photoactivation lumière UV, c'est-à-dire l’évaluation de la quantité relative de PA-FPs pouvant être photoactivées avec différents modes de photoactivation, et permet donc pour définir des schémas de photoactivation qui sont plus efficaces que d’autres. En outre, cette stratégie permet en principe de l’évaluation de la quantification absolue de la fraction de photoactivation PA-FP. À cette fin, il est important de réaliser que les études d’ensemble présenté sont fondés sur l’intensité qui rend l’analyse plus complexe comme décrit dans le présent protocole. Paramètres déterminant l’intensité de fluorescence mesurée, c'est-à-dire différentes luminosité moléculaire, absorbance et spectres d’émission et effets de la frette, doivent être examinées lors de la comparaison des intensités de fluorescence de différentes protéines fluorescentes.
La quantification de basé sur l’intensité ratiométrique présenté de photoactivation efficacité est illustrée pour PA-GFP et PA-Cherry dans des cellules vivantes, mais est en principe largement applicables et peut être utilisée pour n’importe quelle protéine fluorescente photoactivatable sous aucune conditions expérimentales.
Protocole
1. Construction de plasmide
- Générer des sondes de la fusion de deux couleurs. Utiliser un vecteur d’expression de cellules de mammifères (voir Table des matières) dans lequel mCherry112 et PA-mCherry1,13 ont été insérés avec la restriction sites AgeI et BsrGI.
- Commande personnalisée oligo-nucléotides pour amplifier les variantes monomères d’eGFP et PA-eGFP contenant l’A206K mutation, c'est-à-dire, mEGFP PA-mEGFP14 sans un codon d’arrêt comme un fragment de SalI-BamHI . Utiliser le primaire N-terminal 5'-AAT TAA CAG TCG ACG ATG GTG AGC AAG GGC GAG G 3' et le C-terminal apprêt 5'-AAT ATA TGG ATC GCC CTT GTA CAG CCT GTC CAT GC 3' et insérez ce fragment SalI-BamHI dans le site multiple de clonage du vecteur d’expression. Cela va créer l’éditeur de liens de cinq acides aminés RNPPV entre la protéine fluorescente verte et rouge. Nous appellerons les chimères du fluorophore comme GFP — cerise, PA-GFP — cerise et GFP — PA-cerise pour le reste de cet article.
Remarque : Le couplage des fluorophores et création des souverains internes pour évaluer l’efficacité de photoactivation peuvent être faits avec n’importe quelle paire de fluorophore spectralement distinctes, par exemple superfolder PA-GFP 15/ PA-TagRFP 16, etc. Bon nombre des protéines fluorescentes photoactivatable proviennent de GFP. Par conséquent, la séquence d’amorce fournie ci-dessus peut être utilisée pour plusieurs protéines fluorescentes pour construire une chimère fluorophore dans un vecteur d’expression de cellules de mammifères.
2. Culture et Transfection des cellules
- Utilisez soit n’importe quelle ligne de cellule standard tels que HeLa, NRK, Cos-7 ou une lignée cellulaire spécifique à utiliser pour des expériences de photoactivation spécifiques. Utilisez DMEM (additionné de sérum de veau fœtal 10 % et de 2 mM de glutamine) et la trypsine sans rouge de phénol (voir Table des matières) pour réduire la fluorescence de fond.
- Détacher les cellules d’une culture anastomosée à la trypsine, compter le nombre de cellules en suspension de cellules à l’aide d’une chambre de Neubauer et semences 5 000 – 10 000 cellules par puits. Alternativement, utilisez 1 goutte d’une pipette de 2 mL d’une suspension cellulaire de 10 mL d’une culture de cellules confluentes cultivée dans un flacon de culture cellulaire-25 T ou 3 gouttes d’une suspension de cellules de 5 mL d’une culture confluente, cultivée dans un flacon de culture cellulaire-12,5 T.
- La croissance de cellules dans des chambres de 8 puits à 1.0 # couvercle en verre (voir Table des matières) pour la microscopie de fluorescence cellules vivantes.
- Transfecter cellules 24h après ensemencement à l’aide de réactifs commerciaux (voir la Table des matières) selon le protocole du distributeur avec la GFP — cerise, PA-GFP — cerise et GFP — chimères PA-Cherry.
- Cellules d’image après un total de 20 h après transfection permettant l’expression de la protéine, le pliage et la maturation.
3. imagerie et Photoactivation
- Cellules d’image dans une chambre environnementale humidifiée et chauffage à 37 degrés Celsius. Pour les médias de la cellule de la mémoire tampon à pH physiologique et à le rendre CO2-indépendant, ajouter 20 mM HEPES, ou fonctionnant au gaz de2 CO 5 % débit la valeur.
- Tout d’abord, l’image des cellules exprimant la GFP — construction de cerise. Définir les paramètres qui définissent l’intensité du laser intégrée dans le temps par pixel dans une image confocale, c'est-à-dire, les temps de pause pixel en microsecondes, transmission filtre accordable acousto-optique (AOTF) en pour cent et un zoom numérique.
Remarque : En utilisant un objectif 60 x et un zoom numérique 3 x permet l’imagerie d’une cellule dans son intégralité, tout en offrant un grossissement suffisant. Valeur temps de pause de pixel 2-4 μs et transmission AOTF pour le laser 488 nm et 561 nm tel que les images montrent un bon rapport signal sur bruit sans aucun blanchiment et aucun pixel indiquant la saturation intensité de fluorescence. - L’image, à l’aide de set laser puissance, transmission AOTF, temps de pause de pixel et zoom numérique, 15-20 cellules exprimant GFP — cerise.
- Ensuite, l’image avec la même puissance de laser de réglage et temporisation pixel, transmission AOTF zoom numérique cellules exprimant GFP — PA-Cherry et PA-GFP — cerise. Recherche pour exprimer des cellules de la couche verte ou le canal rouge, respectivement. Éviter la longue exposition des cellules au cours de la recherche pour exprimer des cellules afin de ne pas javelliser les protéines fluorescentes.
- Mettre en place une série de mini-temps avec une seule image de préactivation et trois images après activation. Les images après activation permettra d’identifier les éventuels états transitoires de sombres en raison de l’exposition aux rayons UV.
Remarque : Dans nos mains, changements dans l’intensité de la fluorescence détectée en raison des États sombres transitoires étaient < 1 % et pourrait être négligé, mais ces États sombres devraient être évalués pour chaque montage expérimental. - Pour déterminer l’efficacité de photoactivation, c'est-à-dire, la fraction de PA-FPs qui est allumé pour être fluorescent, dans les expériences de photoactivation spécifiques qui ont déjà été établis, appliquer les mêmes paramètres de photoactivation de 15 à 20 cellules qui expriment les dirigeants internes introduites ici et de procéder à l’analyse d’image (section 4). Si commence à mettre en place des expériences de photoactivation, trouver ici quelques réglages différents basés sur notre expérience expérimentale avec PA-GFP et PA-Cherry ; modifier au besoin.
- Pour les instantanés photoactivation de PA-GFP et PA-Cherry utilisant un confocal laser scanning microscope (CLSM), s’appliquent μW 90 de 405 nm lumière laser dans 3 ou 5 itérations, respectivement.
Remarque : Avec notre système microscopique, à l’aide d’une transmission de AOTM 38 % et un temps de pause de pixel 2 μs, nous avons mesuré cette puissance de laser de 405 nm en mode de balayage linéaire à la lentille de l’objectif. Avec ces paramètres, environ 8 % et 16 % des PA-GFP et PA-Cherry exprimées peuvent être photoactivation être fluorescent. La série de titrage complet à l’aide de CLSM de photoactivation instantanée a été publiée précédemment17. - Si une fraction plus élevée de photoactivation PA-GFP et PA-Cherry fluorophores est avantageux par exemple pour obtenir un rapport signal-bruit plus élevé, et photoactivation n’a pas à être immédiat, appliquez 40 μW de 405 nm laser avec un temps de pause pour le pixel μs et 6 % 2 Transmission de AOTF pour 450 itérations. Puis, photoactivation prendra jusqu'à 4 min par opposition à seulement 1-2 secondes, mais efficacité de photoactivation pour PA-GFP sera de 29 % au lieu de 8 %, ce qui permet un rapport signal-bruit plus élevé.
- Pour les instantanés photoactivation de PA-GFP et PA-Cherry utilisant un confocal laser scanning microscope (CLSM), s’appliquent μW 90 de 405 nm lumière laser dans 3 ou 5 itérations, respectivement.
- S’il existe un système d’éclairage numérique de mosaïque qui contient des tableaux de micro miroir dans un modulateur spatial de lumière, lumière de 405 nm laser peut être utilisée pour widefield-photoactivation. Cela permet de photoactivation efficace quelques millisecondes.
Remarque : Avec la puissance de laser de mW en 1,6 telle que mesurée à la lentille d’objectif et un temps d’exposition de 250 ms, 29 % des PA-GFP peut être photoactivation être fluorescent. - Image avec tous les paramètres set photoactivation de 15 à 20 cellules exprimant PA-GFP — cerise et GFP — PA-Cherry, respectivement.
- Étant donné que le pH, les espèces réactives de l’oxygène et les autres facteurs environnementaux peuvent influencer l’efficacité de photoactivation, il peut être important d’évaluer les efficacités de photoactivation du PA-SFP dans le micromilieu subcellulaire respectif où la protéine d’intérêt est situé. En couplant les chimères du fluorophore à la protéine d’intérêt, comme les auteurs l’ont fait avec la membrane plasmique protéine VSVG 18, photoactivation efficacité peut être évaluée dans le compartiment subcellulaire spécifique d’intérêt.
4. image Analysis and algorithme pour ratiométrique fondés sur l’intensité de Quantification de Photoactivation efficacité
- Analyse d’image peut être fait avec le plates-formes ImageJ ou Fidji de traitement d’image open source. Déterminer l’intensité de fluorescence de fond dans les cellules non transfectées dans le vert (B1) et le canal (B2) rouge. Évitez périnucléaire ou toutes les zones montrant une augmentation automatique-fluorescence.
- Pour déterminer l’intensité de fluorescence dans une cellules transfectées, décrire le corps de la cellule avec l’outil de sélection à main levée. Encore une fois, éviter périnucléaire ou toute autre zone liste auto-fluorescence.
- Soustraire l’arrière-plan de l’intensité de la fluorescence mesurée dans chaque canal.
J’aiG = IGreen_measured – B1
J’aiR = IRed_measured – B2 - Utilisation de la GFP — cerise construct pour calculer le ratio de rouge-à-vert (RtoGr) et de corriger les donateurs-piégeage en raison du transfert d’énergie de fluorescence par résonance (FRET). L’efficacité de la frette E a été établie à 0,3 dans des expériences antérieures de la GFP — construction de cerise à l’aide de l’éditeur de liens du même acide aminé entre les deux fluorophores18.
RtoGr = (j’aiRed_measured – B2) / (j’aiGreen_measured – B1)
RtoGrcorr = RtoGr * (1-E)
Remarque : Dans cette approche de base intensité, donneur trempe pour mEGFP et PA-mGFP peut être différent compte tenu des propriétés spectrales distinctes possibles qui n’ont pas été caractérisées. Le taux de FRET (kET) et la distance de Foerster (R0) dépendent du rendement quantique du donneur qui n’a pas été déterminé pour mEGFP et PA-mGFP. - Utilisation de la GFP — construction de PA-Cherry afin d’évaluer la fraction de photoactivation PA-Cherry. Déterminer l’intensité de fluorescence attendue de PA-Cherry en multipliant l’intensité de la fluorescence verte inassouvie mesurée de la GFP — PA-Cherry construire préalable de photoactivation avec le corrigé rouge-à-vert-ratio (RtoGrcorr ).
J’aiRed_expected (IGreen_measured -B,1) = * RtoGrcorr
Remarque : Comme indiqué ci-dessus, la luminosité moléculaire de la FP toujours sur et la photoactivatable FP peut être différente. Voir la Discussion pour plus d’informations sur la façon de tenir compte de ces différences. - Calculer l’efficacité de photoactivation de PA-Cherry comme une fraction de l’intensité de fluorescence rouge mesurée après photoactivation et l’intensité de fluorescence attendus dans le canal rouge.
(FPA-Cherry) = (j’aiRed_measured – B2) / j’aiRed_expected - Utiliser le PA-GFP — cerise construct afin d’évaluer la fraction de photoactivation PA-GFP. Déterminer l’intensité de fluorescence attendue de PA-GFP en divisant l’intensité de la fluorescence rouge mesurée de la PA-GFP — cerise construire préalable de photoactivation par le rouge-à-vert-ratio (RtoGr). Ici, le RtoGr n’a pas besoin d’être corrigé pour la trempe, des donneurs car GFP et PA-GFP sont passibles de donateurs trempe au même montant.
J’aiGreen_expected = (j’aiRed_measured – B2) / RtoGr - Calculer l’efficacité de photoactivation de PA-GFP comme une fraction de l’intensité de fluorescence verte mesurée après photoactivation et l’intensité de fluorescence attendus dans le canal vert.
(FPA-GFP) = (j’aiGreen_measured – B1) / j’aiGreen_expected
Résultats
Le protocole présenté ici montre la quantification logométrique de la fraction des protéines fluorescentes qui sont photoactivées d’être fluorescente (Figure 1). Cette fraction est différent selon le mode de photoactivation.
Le résultat typique à l’aide de photoactivation de haute puissance peu de temps avec un confocal laser scanning microscope (CLSM) est affiché à la Figure 2C. Après titration de la puissance du laser telle que mesurée à la lentille de l’objectif par pixel s’attarder temps et transmission ATOF, l’efficacité maximale de photoactivation PA-GFP était d’environ 8 % et les PA-cerise environ 16 %. L’efficacité de photoactivation comparativement faible des PA-GFP et PA-Cherry peut s’expliquer par photoactivation simultanée et - destruction lorsqu’il est exposé à un flux continu de déterministe des photons par CLSM. Les durées de vie de fluorescence plus courtes et les spectres d’absorption différentes, tronqué à droite de la rouge PA-FPs peut contribuer à l’efficacité supérieure de photoactivation de PA-cerise par rapport aux PA-GFP.
À l’aide de laser de faible puissance et des centaines d’itérations, une plus grande efficacité de photoactivation est possible. 450 itérations de la lumière UV livré par un CLSM sur un total de 4 min a donné un rendement de photoactivation de 29 % pour PA-GFP (Figure 3C). L’efficacité supérieure de photoactivation avec exposition répétée aux photons de la lumière UV peut suggérer un processus multipas photoactivation. Par ailleurs, la lumière UV appliquée est assez forte pour photoactivate mais pas assez pour photodestruct qui conduit cumulatifs au fil du temps à une fraction plus élevée de photoactivation protéines fluorescentes.
Avec widefield d’illumination, les fluorophores stochastique et répétitivement affleurent à 405 nm photons. Ici, l’exposition pour seulement 250 ms a donné un rendement de photoactivation de 29 % pour PA-GFP.

Figure 1 : Concept de la façon de déterminer l’efficacité de photoactivation en vrac et dans des cellules vivantes. En couplant des protéines fluorescentes spectralement distinctes, règles internes sont créés qui permettre l’évaluation de base intensité ratiométrique de photoactivation efficacité. Les intensités mesurées de PA-cerise et PA-GFP étaient liées aux intensités attendues. Intensités attendues provenaient de déterminer l’intensité de la fluorescence des protéines fluorescentes toujours sur GFP-Cherry, GFP-PA-Cherry ou chimères GFP-PA-Cherry avant photoactivation. Figure de Renz et Wunder 201717. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : en vrac GFP-PA-cerise et photoactivation de PA-GFP-Cherry (a) (b), instantanément et complètement que possible à l’aide d’un microscope confocal à balayage laser dans des cellules vivantes. 8 % de PA-GFP exprimé était photoactivation avec un temps de pause de pixel de 2 µs et une transmission AOTF de 38 %, entraînant dans la puissance du laser de 90 μW, telle que mesurée à la lentille de l’objectif et 3 itérations (c). Augmentant la puissance de 405 nm laser n’a pas augmenté l’efficacité de photoactivation. Figure de Renz et Wunder 201717. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
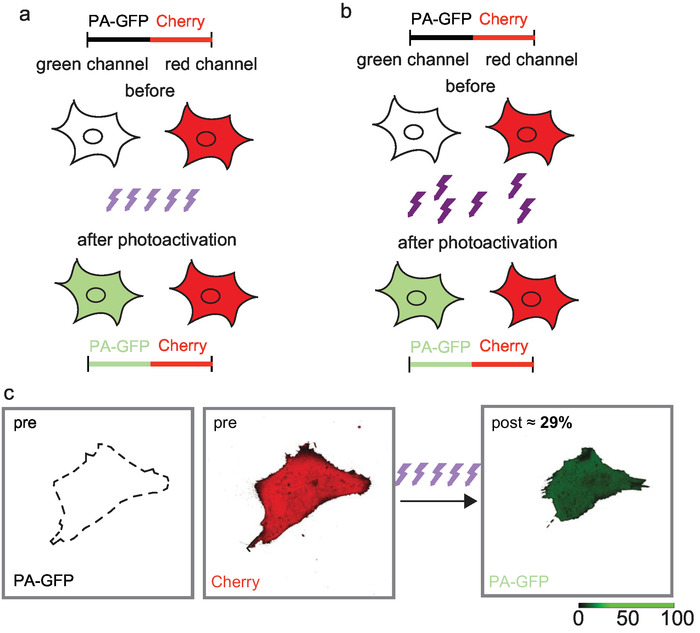
Figure 3 : itérative photoactivation de faible puissance avec un microscope confocal à balayage laser (a) et le rendement de photoactivation élevé court widefield haute puissance éclairage b rendement. 29 % des PA-GFP a été photoactivées avec un temps de pause de pixel de 2 µs et une transmission AOTF de 6 %, entraînant dans la puissance du laser de 40 μW, telle que mesurée à 450 itérations (c) et l’objectif. Figure de Renz et Wunder 201717. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Jusqu’ici, aucune méthode n’existait pour déterminer, en vrac, la fraction de PA-FPs exprimées dans des cellules vivantes qui est photoactivées d’être fluorescente. Le protocole présenté peut être utilisé pour n’importe quelle paire de protéine fluorescente spectralement distinctes. Bien qu’illustrée ici pour l’irréversible PA-FPs PA-GFP et PA-cerise, cette approche est en principe applicable aux protéines et ainsi. La protéine fluorescente spectralement distincte, toutefois, doit être sélectionnée avec soin afin de minimiser le chevauchement spectral étant donné que les protéines fluorescentes et déplacement leur absorption et les spectres d’émission, par exemple du vert à fluorescence rouge.
Comme indiqué ci-dessus, il est important de préciser que l’approche présentée est ratiométrique et fondés sur l’intensité. Il peut être utilisé pour normaliser le niveau d’expression inconnue dans les cellules et définir les différences relatives de photoactivation efficacité de différents modes de photoactivation. Le protocole permet également d’évaluer la fraction absolue de photoactivation PA-FPs. Ensuite, différentes propriétés spectrales de différents FPs doivent être prises en compte.
La luminosité moléculaire (MB) est le produit du rendement quantique (QY), coefficient d’extinction (EC) et pourcentage absorbance à la longueur d’onde d’excitation donnée par rapport à la pointe de l’absorbance. Pour cerise12 et13de la PA-Cherry, des valeurs respectives de QY et EC ont été publiées. Le pourcentage absorbance à la longueur d’onde d’excitation donnée de 543 nm par rapport au pic d’absorbance est de 0,5 et 0,7, respectivement.
MBcerise = 0,22 * 72 000 * 0,5 = 7 920
MoPA-cerise = 0,46 * 18 000 * 0,7 = 5 796
Ainsi, la luminosité moléculaire inférieure de PA-Cherry par rapport à la cerise peut tenir compte en divisant j’aiRed_expected de 1,37 (dérivé de 7 920/5 796).
Cependant, on ne sait pas dans quelles conditions de photoactivation la luminosité moléculaire publiée de PA-Cherry a été déterminée. C’est important, étant donné que nous montrons ici que le mode de photoactivation passe la fraction mesurée de photoactivation PA-FPs. En outre, pour les versions monomères comprenant la mutation A206K. par exemple, mEGFP et PA-mEGFP, aucun luminosité moléculaire n’a été publié.
Dans cette approche ratiométrique intensité, la luminosité moléculaire du PA-SFP et les homologues FP toujours actif dans une première approximation ont été considérés identiques. Nous avons décidé sur cette approche, puisque (i) pour certains FPs aucun moléculaire de la luminosité n’a été signalée, et (ii) on ignore jusqu'à présent dans la rubrique loin différents modes de photoactivation peuvent influer sur la luminosité moléculaire du PA-SFP rapportée dans la littérature. En outre, (iii) pour une analyse comparative de la connaissance de la luminosité moléculaire n’est pas nécessaire ; Il est seulement nécessaire pour la détermination fondés sur l’intensité de la fraction absolue de photoactivation PA-FPs, ce qui peut être calculée comme indiqué ci-dessus.
Notre approche impliquant les chimères de la protéine fluorescente comme dirigeants internes montre qu’une exposition différente à la lumière UV donne photoactivation différentes efficacités. Ainsi, il définit des options comme la fraction photoactivate une plus grande PA-FP et atteindre un meilleur rapport signal sur bruit. En outre, elle ouvre des possibilités de façon différentielle photoactivate PA-FPs différents dans la même cellule donnée leur réponse différentielle à lumière UV ou à différentiellement photoactivate le même PA-FP dans différents compartiments subcellulaires par exposant différemment à la lumière UV. En résumé, notre protocole aidera davantage la compréhension quantitative des processus cellulaires à l’aide de PA-FPs en microscopie des cellules vivantes.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous tenons à remercier le laboratoire Dorigo et le Service d’imagerie de Neuroscience à la Stanford University School of Medicine pour la fourniture d’équipements et espace pour ce projet.
matériels
| Name | Company | Catalog Number | Comments |
| pEGFP-N1 mammalian cell expression vector | Clontech | ||
| DMEM w/o phenol red | Thermo Fisher Scientific | 11054020 | |
| Trypsin w/o phenol red | Thermo Fisher Scientific | 15400054 | |
| L-Glutamine (200 mM) | Thermo Fisher Scientific | 25030081 | |
| HEPES | Thermo Fisher Scientific | 15630080 | |
| LabTek 8-well chambers #1.0 | Thermo Fisher Scientific | 12565470 | |
| Fugene 6 | Promega | E2691 |
Références
- Patterson, G. H., Lippincott-Schwartz, J. A photoactivatable GFP for selective photolabeling of proteins and cells. Science. 297 (5588), 1873-1877 (2002).
- Ando, R., Hama, H., Yamamoto-Hino, M., Mizuno, H., Miyawaki, A. An optical marker based on the UV-induced green-to-red photoconversion of a fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (20), 12651-12656 (2002).
- Renz, M., Lippincott-Schwartz, J. Ch. 9. The Fluorescent Protein Revolution Series in Cellular and Clinical Imaging. Day, R. N., Davidson, M. W. , CRC Press. 201-228 (2014).
- Shcherbakova, D. M., Sengupta, P., Lippincott-Schwartz, J., Verkhusha, V. V. Photocontrollable fluorescent proteins for superresolution imaging. Annual Review of Biophysics. , 303-329 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Henderson, J. N., et al. Structure and mechanism of the photoactivatable green fluorescent protein. Journal of the American Chemical Society. 131 (12), 4176-4177 (2009).
- Subach, F. V., et al. Photoactivation mechanism of PAmCherry based on crystal structures of the protein in the dark and fluorescent states. Proceedings of the National Academy of Sciences of the United States of America. 106 (50), 21097-21102 (2009).
- Wiedenmann, J., et al. EosFP, a fluorescent marker protein with UV-inducible green-to-red fluorescence conversion. Proceedings of the National Academy of Sciences of the United States of America. 101 (45), 15905-15910 (2004).
- Habuchi, S., Tsutsui, H., Kochaniak, A. B., Miyawaki, A., van Oijen, A. M. mKikGR, a monomeric photoswitchable fluorescent protein. PLoS One. 3 (12), e3944(2008).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein. Nature Methods. 6 (2), 131-133 (2009).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nature Biotechnology. 22 (12), 1567-1572 (2004).
- Subach, F. V., et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nature Methods. 6 (2), 153-159 (2009).
- Zacharias, D. A., Violin, J. D., Newton, A. C., Tsien, R. Y. Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells. Science. 296 (5569), 913-916 (2002).
- Slocum, J. D., Webb, L. J. A Double Decarboxylation in Superfolder Green Fluorescent Protein Leads to High Contrast Photoactivation. Journal of Physical Chemistry Letters. 8 (13), 2862-2868 (2017).
- Subach, F. V., Patterson, G. H., Renz, M., Lippincott-Schwartz, J., Verkhusha, V. V. Bright monomeric photoactivatable red fluorescent protein for two-color super-resolution sptPALM of live cells. Journal of the American Chemical Society. 132 (18), 6481-6491 (2010).
- Renz, M., Wunder, C. Internal rulers to assess fluorescent protein photoactivation efficiency. Cytometry A. , (2017).
- Renz, M., Daniels, B. R., Vamosi, G., Arias, I. M., Lippincott-Schwartz, J. Plasticity of the asialoglycoprotein receptor deciphered by ensemble FRET imaging and single-molecule counting PALM imaging. Proceedings of the National Academy of Sciences of the United States of America. 109 (44), E2989-E2997 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon